Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Système de culture automatisé destiné à être utilisé dans les essais précliniques de thérapies dirigées par l’hôte pour la tuberculose
Dans cet article
Résumé
Une quantification rapide et efficace de la croissance intracellulaire de M. tuberculosis est cruciale pour poursuivre des thérapies améliorées contre la tuberculose (TB). Ce protocole décrit un test de détection colorimétrique à base de bouillon utilisant un système automatisé de culture liquide pour quantifier la croissance de Mtb dans les macrophages traités avec des thérapies candidates dirigées par l’hôte.
Résumé
Mycobacterium tuberculosis (Mtb), l’agent causal de la tuberculose (TB), était la maladie infectieuse la plus meurtrière dans le monde jusqu’à l’avènement de la COVID-19. Le VTT a évolué pour persister dans son environnement intracellulaire, échapper aux défenses de l’hôte et a développé une résistance à de nombreux médicaments antituberculeux. Une approche pour résoudre la résistance consiste à identifier les médicaments approuvés existants qui stimuleront la réponse immunitaire de l’hôte au Mtb. Ces médicaments pourraient ensuite être réutilisés en tant que thérapies d’appoint dirigées par l’hôte (HDT) pour raccourcir la durée du traitement et aider à surmonter la résistance aux antibiotiques.
La quantification de la croissance intracellulaire du Mtb dans les macrophages est un aspect crucial de l’évaluation du potentiel HDT. L’étalon-or pour mesurer la croissance du Mtb est le comptage des unités formant colonie (UFC) sur des plaques de gélose. Il s’agit d’un test lent et laborieux qui ne se prête pas à un dépistage rapide des médicaments. Dans ce protocole, un système de culture automatisé à base de bouillon, qui est plus couramment utilisé pour détecter le Mtb dans les échantillons cliniques, a été adapté pour le dépistage préclinique des thérapies dirigées par l’hôte. La capacité du système d’essai de culture liquide à étudier la croissance intracellulaire du Mtb dans les macrophages traités par HDT a été évaluée. Les HDT testés pour leur capacité à inhiber la croissance de Mtb étaient de l’acide rétinoïque tout-trans (AtRA), à la fois en solution et encapsulé dans des microparticules de poly(acide lactique-co-glycolique) (PLGA) et la combinaison d’interféron-gamma et de linézolide. Les avantages de cette technique automatisée basée sur la culture liquide par rapport à la méthode CFU comprennent la simplicité de configuration, une préparation moins laborieuse et un délai d’obtention des résultats plus rapide (5-12 jours contre 21 jours ou plus pour les plaques de gélose).
Introduction
Mycobacterium tuberculosis (Mtb), l’agent causal de la tuberculose, était la maladie infectieuse la plus meurtrière dans le monde en 20191. Pour échapper aux défenses de l’hôte, le Mtb subvertit l’activité mycobactéricide des cellules immunitaires innées telles que les macrophages et les cellules dendritiques (DC), ce qui lui permet de persister intracellulaire et de se répliquer2. L’absence d’un vaccin efficace pour prévenir la tuberculose pulmonaire chez l’adulte et l’émergence croissante de souches pharmacorésistantes soulignent le besoin urgent de nouvelles thérapies.
Les thérapies d’appoint dirigées par l’hôte (HDT) pourraient raccourcir la durée du traitement et aider à surmonter la résistance3. L’évaluation préclinique des candidats HDT in vitro pour déterminer l’activité mycobactéricide dans les macrophages repose souvent sur la quantification de la croissance du Mtb par unités formant colonies (UFC) sur des plaques de gélose solide. Il s’agit d’un test lent et laborieux qui ne se prête pas à un dépistage rapide des médicaments. Les systèmes automatisés de détection microbienne à base de bouillon disponibles dans le commerce sont plus couramment utilisés dans les laboratoires de microbiologie clinique pour la détection et les tests de sensibilité aux médicaments du Mtb et d’autres espèces mycobactériennes dans les échantillons cliniques4. Ces instruments mesurent indirectement la croissance en fonction de l’activité métabolique bactérienne entraînant des modifications physiques dans les milieux de culture (modification des niveaux de CO2 ou d’O2 ou de la pression) surveillée au fil du temps5. La lecture est le temps de positivité (TPP), dont il a déjà été démontré qu’il était corrélé avec l’UFC Mtb dans les échantillons d’expectorations de patients tuberculeux en réponse au traitement 6,7 et dans les lysats de poumon murin et de rate8 infectés. En outre, des systèmes de détection par culture liquide ont été utilisés pour mesurer l’effet des thérapies conventionnelles dirigées contre les agents pathogènes sur la croissance des mycobactéries en culture axénique et en culturede macrophages 9,10. L’instrument a également été utilisé pour étudier la capacité innée des cellules dendritiques et des macrophages alvéolaires à contrôler la croissance intracellulaire de Mtb11,12. Ce protocole expérimental démontre qu’un système de diagnostic par culture liquide peut être adapté pour effectuer le dépistage préclinique de la tuberculose HDT dans des macrophages en culture. Par rapport au dénombrement des UFC, le principal avantage de cette technique est qu’elle réduit considérablement le travail expérimental et le temps nécessaire pour quantifier la croissance/survie des mycobactéries intracellulaires. Cette technique repose sur l’accès à un instrument de culture automatisé qui peut être utilisé pour évaluer la survie mycobactérienne intracellulaire dans les cellules immunitaires traitées avec une large gamme de réactifs pharmacologiques ciblant les fonctions cellulaires pour renforcer l’immunité de l’hôte.
Protocole
Les expériences décrites dans ce protocole ont été réalisées en utilisant la souche H37Ra atténuée de Mtb, qui peut être manipulée dans un laboratoire de niveau de confinement 2. Toutes les manipulations de mycobactéries vivantes ont été effectuées dans une enceinte de sécurité biologique (ESB) de classe II. Des procédures expérimentales ont été conçues pour minimiser la production d’aérosols. Une culture de cellules eucaryotes (cellules THP-1) a également été réalisée dans une ESB de classe II. Le laboratoire a procédé à une évaluation des risques et s’est assuré que toutes les procédures étaient conformes aux réglementations institutionnelles et nationales en matière de sécurité biologique. La lignée cellulaire monocytaire humaine THP-1 a été utilisée pour exécuter la méthode décrite (étape 1). Les cellules sont différenciées en macrophages après stimulation avec du phorbol 12-myristate 13-acétate (PMA) avant l’infection par des mycobactéries.
1. Culture cellulaire
- Multiplier le stock de semences H37Ra à la phase logarithmique dans un bouillon Middlebrook 7H9 (MB) complété par un enrichissement albumine-dextrose-catalase (ADC) (10%) et 0,05% de polysorbate 80. Conservez le matériel de H37Ra dans des aliquotes de 1 mL dans un congélateur à -80 °C pendant une période allant jusqu’à 1 an.
- Décongeler un flacon de 1 mL de Mtb-H37Ra et le transférer dans une fiole T25 munie d’un bouchon filtrant contenant 9 mL de bouillon supplémenté en MB environ 1 semaine avant l’expérience prévue. Incuber à 37 °C pendant 5 à 7 jours dans un incubateur statique.
- Cultiver des cellules THP-1 dans RPMI-1640 complétées par du sérum de veau fœtal à 10 % non tué par la chaleur ((c)RPMI) complet dans une fiole T75 dans un incubateur humidifié au CO 2 à 5 % de CO2/37 °C et une sous-culture deux fois par semaine pour maintenir une densité inférieure à 1 x 106 cellules/mL.
- Différencier les cellules THP-1 en macrophages 3 jours avant l’infection en pipetant doucement les cellules plusieurs fois à l’aide d’une pipette sérologique dans des flacons T75 pour disperser les amas et les placer dans un tube conique de 50 mL.
- Centrifuger les cellules à 300 x g pendant 10 minutes à température ambiante, décanter le surnageant et remettre doucement la pastille en suspension dans 2 ml de RPMI. Effectuer le comptage des cellules pour estimer les cellules/mL.
- Ensemencer 2 mL de macrophages THP-1 dans des plaques de culture tissulaire à 12 puits à une densité de 100 000 cellules/mL dans une cRPMI avec 100 ng/mL de PMA pendant 72 h. Retirer le milieu contenant du PMA des cellules et reconstituer avec un cRPMI frais avant l’infection à Mtb.
- Configurez des plaques individuelles pour chaque point de temps requis.
- Cellules de semence à la même densité (100 000 cellules/mL) dans des lames de chambre en verre à 2 puits pour déterminer la multiplicité de l’infection (MOI).
- Placer dans un incubateur humidifié à 5% de CO2 pendant 3 jours à 37 °C.
2. Quantification de l’absorption du VTT
- Détermination de l’absorption du Mtb par les macrophages (MOI)
- Installez l’enceinte de sécurité biologique (ESB) de classe II le jour de l’infection et travaillez sur deux couches de papier de soie pour détecter tout déversement. Mettez en place des conteneurs de déchets conformément à la réglementation locale.
- Prélever 6 à 8 mL de culture mycobactérienne du ballon T25 et le transférer dans un tube en polypropylène de 15 mL.
REMARQUE: Des tubes de plus petit volume peuvent être utilisés pour des expériences plus petites. - Centrifuger le tube dans une centrifugeuse de paillasse à température ambiante pendant 10 min à 2890 x g.
- Retirez délicatement le tube de la centrifugeuse et transférez-le dans l’enceinte de sécurité biologique. Attendez 1 min pour permettre aux bactéries de s’installer.
- Versez le surnageant dans le récipient de désinfectant jeté, reboucher le tube et remettre en suspension les bactéries dans le milieu restant en tapotant le côté du tube. Attendez 1 min pour permettre aux bactéries de s’installer.
- Ajouter 2 mL de cRPMI préchauffé, mélanger doucement et transférer dans un tube conique de 50 mL.
- Remettez les mycobactéries en suspension très soigneusement à l’aide d’une aiguille de 25 G et d’une seringue de 5 mL. Pour remettre en suspension, aspirez la suspension de mycobactéries dans la seringue et éjectez-la très doucement le long de la paroi latérale du tube pour minimiser la production d’aérosols. Répétez 6-8 fois.
REMARQUE: Faites preuve de la plus grande prudence car il s’agit d’une culture à haute densité de mycobactéries. Pour éviter le risque de blessure par piqûre d’aiguille, utilisez des aiguilles contondantes dans la mesure du possible et des seringues Luer. - Jeter l’aiguille et la seringue dans un contenant pour objets pointus ou tranchants dans l’ESB.
- Transférer la suspension dans un tube à microfuger de 2 ml (muni d’un bouchon à visser) et centrifuger à température ambiante pendant 3 min à 100 x g pour enfouir les touffes restantes. Remettez le tube dans l’armoire de sécurité et attendez 1 min pour permettre aux bactéries de se déposer.
- Transférer les 1 à 1,5 mL supérieurs du surnageant dans un nouveau tube. Jetez le tube d’origine dans le seau à déchets contenant du désinfectant. Bien mélanger et ajouter diverses quantités de suspension mycobactérienne (p. ex. 5, 25, 50, 150 μL) aux lames de la chambre de verre à 2 puits et incuber pendant 3 h dans un incubateur de CO2 à 37 °C.
- Coloration pour les bactéries acido-résistantes (AFB)
NOTE: Après 3 h d’incubation, les macrophages sont lavés et fixés avec du paraformaldéhyde pour inactiver les mycobactéries. Les lames sont ensuite colorées à l’aide d’un kit Auramine O modifié (voir le tableau des matériaux) pour estimer les mycobactéries phagocytées par cellule. En raison de leur paroi cellulaire cireuse, les mycobactéries retiennent le colorant Auramine après un lavage acido-alcoolique. Les noyaux des macrophages sont ensuite contre-colorés avec Hoechst. Cette méthode permet de compter le nombre de bactéries phagocytées par cellule et est utilisée pour déterminer la multiplicité de l’infection (MOI) des macrophages.- Retirez le milieu de la lame de la chambre de verre après avoir pipeté de haut en bas trois fois pour déloger les bactéries qui n’ont pas été phagocytées.
- Laver une fois avec 2 mL de PBS à température ambiante.
- Stocker des stocks de paraformaldéhyde à 4 %, dissous dans du PBS dans des aliquotes à -20 °C pendant 6 mois au maximum. Décongeler une partie aliquote de 4 % de paraformaldéhyde immédiatement avant utilisation. Diluer à 2 % de paraformaldéhyde avec du PBS et ajouter 2 mL par puits.
- Incuber pendant 10 min à température ambiante. La glissière de la chambre en verre peut être retirée de l’armoire de sécurité à ce stade pour la coloration.
- Lavez la lame sous un léger jet d’eau du robinet.
- Distribuer suffisamment d’auramine sur la lame pour couvrir les cellules à l’aide d’une pipette de transfert en plastique et incuber pendant 1 min à température ambiante dans l’obscurité (couvrir avec du papier d’aluminium).
- Lavez l’excès de colorant de la lame avec de l’eau du robinet et ajoutez le décolorant/désinfectant pendant 1 min dans l’obscurité.
- Laver l’excès avec de l’eau du robinet et incuber pendant 15 minutes à température ambiante avec Hoechst 33342 (10 μg/mL dans PBS) dans l’obscurité.
- Lavez la tache Hoechst avec de l’eau du robinet, retirez les chambres, drainez l’excès d’eau de la glissière, ajoutez une goutte d’antidécoloration et de capseau et séchez à l’air.
- Examinez la lame au microscope fluorescent à l’aide de l’objectif d’huile 100x. Les mycobactéries deviennent vertes sous le filtre FITC. Les noyaux sont fluorescents en bleu sous le filtre DAPI (Figure 1C).
- Déterminer le MOI en comptant le nombre de mycobactéries phagocytées par cellule et le pourcentage de cellules infectées.
- Calculer le volume de suspension mycobactérienne nécessaire pour obtenir l’MOI requis en fonction de la surface d’un puits dans la plaque; Par exemple, la surface des lames de chambre en verre utilisées dans cette expérience est de 4 cm2. Un faible MOI (environ 1-2 bacilles/cellule) est souhaitable pour les expériences menées sur plusieurs jours (p. ex. 5 jours).
- Infection des macrophages
- Bien mélanger la suspension de mycobactéries et ajouter la quantité nécessaire aux cellules sur des plaques à 12 puits une fois que le volume requis pour obtenir la MOI désirée a été déterminé.
- Incuber à 37 °C pendant 3 h pour permettre la phagocytose des mycobactéries.
- Éliminez les bactéries extracellulaires en lavant les puits avec un RPMI chaud ou HBSS plusieurs fois.
- Lyser les macrophages dans un puits (échantillon de 3 h) pour déterminer le pourcentage de temps jusqu’à positivité (PTT) de l’inoculum initial (échantillon de 3 h), comme indiqué à l’étape 3 ci-dessous.
- Ajouter le cRPMI frais et les doses de médicament requises ou le contrôle du véhicule aux puits restants, incuber les plaques dans l’incubateur de CO2 à 37 °C pendant le temps nécessaire (selon le plan expérimental, mais généralement à plusieurs intervalles entre 1 et 8 jours).
3. Prélèvement d’échantillons pour le système de détection de culture liquide
NOTE: Le jour de l’infection, les mycobactéries extracellulaires sont éliminées par lavage, et les mycobactéries intracellulaires sont récoltées par lyse d’un puits de macrophages (échantillon de 3 h) pour déterminer la quantité initiale phagocytée comme contrôle de base de l’infection. Par la suite, le milieu, le lysat cellulaire et les lavages sont combinés pour mesurer la croissance mycobactérienne totale. La croissance extracellulaire et intracellulaire peut également être évaluée séparément si vous le souhaitez.
- Prélèvement d’un échantillon de 3 heures pour déterminer le PTT
- Éliminer les mycobactéries extracellulaires de tous les puits après les 3 premières heures d’infection, comme indiqué à l’étape 2.3.3. Ajouter 1 mL de média frais au puits de contrôle de 3 h pour égaliser le volume de lysat avec ceux des points temporels ultérieurs.
REMARQUE : Voir l’étape 3.2.7 si les mycobactéries extracellulaires doivent être exclues de l’analyse.
- Éliminer les mycobactéries extracellulaires de tous les puits après les 3 premières heures d’infection, comme indiqué à l’étape 2.3.3. Ajouter 1 mL de média frais au puits de contrôle de 3 h pour égaliser le volume de lysat avec ceux des points temporels ultérieurs.
- Prélèvement d’échantillons
- Bouillon MB chaud et bouteilles de culture d’instruments pour les amener à température ambiante.
- Transférer le milieu de la plaque de 12 puits aux tubes coniques marqués correspondants.
- Ajouter 500 μL de tampon de lyse stérile (0,1% Triton x-100 dans PBS filtré à travers un filtre stérile de 0,2 μm) à chaque puits pendant 10 min.
- Grattez délicatement les cellules du puits avec un grattoir stérile et combinez avec le milieu dans le tube conique approprié.
- Laver le puits avec 0,5 mL de PBS stérile et transférer dans le tube approprié.
- Passer délicatement chaque échantillon à travers une aiguille et une seringue (25 G) 6-8 fois pour briser les touffes. Diluer les échantillons 1:10 dans un bouillon MB; 100 μL d’échantillon + 900 μL de milieu MB.
- Aux heures/jours requis (habituellement entre 1 et 8 jours), prélever les échantillons restants en suivant les étapes 3.2.1 à 3.2.6 ci-dessus.
NOTE: Les chercheurs peuvent préférer exclure les mycobactéries extracellulaires de leur analyse, auquel cas, à l’étape 3.2.2 ci-dessus, le milieu de chaque puits est jeté et les macrophages sont lavés plusieurs fois avant d’ajouter un tampon de lyse.
- Inoculation et chargement de bouteilles de culture d’instruments
NOTE: Les détails de l’instrument de culture liquide et des consommables connexes sont fournis dans le tableau des matériaux.- Stérilisez le bouchon en caoutchouc du flacon de culture instrumentale avec du papier de soie imbibé d’alcool à 70% et laissez-le sécher à l’air.
REMARQUE : Cette étape doit être effectuée dans l’ESB. - Préparez les bouteilles en transférant suffisamment de suppléments nutritifs pour tous les échantillons dans un tube conique (0,5 mL/bouteille). Utilisez une aiguille et une seringue pour injecter 0,5 mL de supplément nutritif dans le flacon de culture instrumentale.
- Pipeter 500 μL de l’échantillon dilué (1:10) dans un tube à fond en V de 1 mL.
- Utilisez une aiguille et une seringue pour injecter les 500 μL d’échantillon dans le flacon de culture d’instruments assigné.
- Stérilisez le bouchon en caoutchouc du flacon de culture instrumentale avec du papier de soie imbibé d’alcool à 70% et laissez-le sécher à l’air. Essuyez les bouteilles avec du papier de soie imbibé d’alcool à 70% avant de les retirer de l’ESB.
- Transportez soigneusement les bouteilles de l’armoire de biosécurité à l’instrument pour le chargement.
- Appuyez sur le bouton de chargement du système automatisé de détection microbienne.
- Scannez les codes-barres sur les bouteilles de culture instrumentale et placez les bouteilles dans l’incubateur du système de détection à 37 °C pendant 42 jours. Lisez et notez le temps nécessaire pour atteindre la positivité à partir de l’écran de l’instrument.
REMARQUE: Le code à barres permet à l’instrument d’identifier la bouteille et de relier les lectures de réflectance à une bouteille particulière. - Calculer le pourcentage de temps jusqu’à la positivité (TTP) en comparant le TTP de l’inoculum intracellulaire initial (Jour 0) à celui des macrophages cultivés pour les temps indiqués. Un changement positif du pourcentage de PTT signifie une croissance mycobactérienne13.
Par exemple, pour le jour 3 :
Variation en pourcentage du délai jusqu’à la positivité =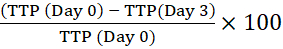
- Stérilisez le bouchon en caoutchouc du flacon de culture instrumentale avec du papier de soie imbibé d’alcool à 70% et laissez-le sécher à l’air.
Résultats
L’instrument automatisé de culture liquide utilisé dans cette étude surveille les niveaux de CO2 toutes les 10 minutes. Un changement de couleur dans le capteur au fond de la bouteille de l’instrument est mesuré colorimétriquement et exprimé en unités de réflectance. Le logiciel de l’instrument applique ensuite des algorithmes de détection pour calculer le temps jusqu’à la positivité (TTP), c’est-à-dire le nombre de jours entre l’inoculation et le marquage des cultures comme positives (...
Discussion
Les auteurs ont utilisé la méthode de culture liquide décrite dans ce protocole pour surveiller la croissance de Mtb dans les macrophages dérivés de monocytes et les macrophages alvéolaires et les cellules THP-1 différenciées avec PMA11,16,17. Cette technique peut également être modifiée pour une utilisation avec des cellules non adhérentes12. Plus récemment, l’instrument a également été...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été financé par la Science Foundation Ireland (SFI 08/RFP/BMT1689), le Health Research Board in Ireland [HRA-POR/2012/4 et HRA-POR-2015-1145] et le Royal City of Dublin Hospital Trust.
matériels
| Name | Company | Catalog Number | Comments |
| IX51 Fluorescent Microscope | Olympus, Japan | N/A | AFB detection and imaging |
| 2 mL microtube, flat bottom, screw cap, sterile | Sarstedt, North Carolina, USA | 72.694.006 | Mtb infection of macropahges |
| 5 mL syringe, Luer lock | BD Biosciences, San Jose, CA, USA | SZR-150-031K | Mtb infection of macropahges/CFU |
| 50 mL tube, sterile | Sarstedt, North Carolina, USA | 62.547.254 | Mtb infection of macropahges |
| all-trans-Retinoic Acid (ATRA) ≥98% (HPLC) | Sigma Aldrich, Missouri, USA | R2625 | Host directed therapy candidate |
| BacT/ALERT 3D Microbial Detection System | Biomerieux ( Hampshire, UK) | 247001 | Broth-based colormetric detection system |
| BACT/ALERT MP BACT/ALERT MP Nutrient Supplement | Biomerieux ( Hampshire, UK) | 414997 | Broth-based colormetric detection assay |
| BACT/ALERT MP culture bottles | Biomerieux ( Hampshire, UK) | 419744 | Broth-based colormetric detection assay |
| BD BBL Middlebrook ADC Enrichment, 20 mL | BD Biosciences, San Jose, CA, USA | M0553 | Mycobacterium liquid culture |
| BD BBL Middlebrook OADC Enrichment, 20mL | BD Biosciences, San Jose, CA, USA | M0678 | Colony Forming Units |
| Cell scraper, 25 cm | Sarstedt, North Carolina, USA | 83.1830 | Harvest of lmacrophage lysates |
| Corning Syringe Filter, 0.2 µm | Corning Incorporated, Germany | 431219 | Sterilization of lysis buffer |
| Cover glass (borosilicate), 24 x 50 mm, #1.5 thickness | VWR International Limited | 631 - 0147 | |
| Cycloheximide, from microbial | Sigma Aldrich, Missouri, USA | C7698 | Colony Forming Units |
| Dako Fluorescent Mounting Medium | Agilent Technologies Ireland Limited | S3023 | Antifade mounting medium |
| Dulbecco’s Phosphate Buffered Saline | Sigma Aldrich, Missouri, USA | D8537 | Mtb infection of macropahges |
| Fetal Bovine Serum, Gibco | Thermo Fisher, Massachusetts, USA | 10270106 | Macrophage cell culture |
| Glycerol, Difco | BD Biosciences, San Jose, CA, USA | 228220 | Colony Forming Units |
| Hoescht 33342 (bisBenzimide H 33342 trihydrochloride) | Sigma Aldrich, Missouri, USA | B2261 | Nuclear stain |
| IFNγ, recombinant human | R&D Systems Inc, Minnesota, USA | 285-IF | Host directed therapy candidate |
| Labtek 2-well chamber slide, sterile, Nunc | Thermo Fisher, Massachusetts, USA | TKT-210-150R | Mtb infection of macropahges |
| L-Asparagine, anhydrous | Sigma Aldrich, Missouri, USA | A4159 | Colony Forming Units |
| Linezolid | Sigma Aldrich, Missouri, USA | PZ0014 | Antibiotic |
| Microlance Hypodermic Needle 25 G | BD Biosciences, San Jose, CA, USA | 300400 | Mtb infection of macropahges/CFU |
| Middlebrook 7H10 Agar Base | BD Biosciences, San Jose, CA, USA | M0303 | Colony Forming Units |
| Middlebrook 7H9 Broth Base | BD Biosciences, San Jose, CA, USA | M0178 | Mycobacterium liquid culture |
| Modified Auramine O Stain and Decolourizer | Scientific Device Laboratory, IL, USA | 345-250 | AFB stain |
| Paraformaldehyde | Sigma Aldrich, Missouri, USA | 158127 | Mtb infection of macropahges |
| Petri dishes, 92 x 16mm (20/bag) | Sarstedt, North Carolina, USA | 82.1473.001 | Colony Forming Units |
| Phorbol 12-myristate 13-acetate (PMA) | Sigma Aldrich, Missouri, USA | P8139 | Macrophage cell culture |
| Polysorbate 80, Difco | BD Biosciences, San Jose, CA, USA | 231181 | Mycobacterium liquid culture |
| RPMI-1640, Gibco | Thermo Fisher, Massachusetts, USA | 52400025 | Macrophage cell culture |
| Sterile Cell Spreader, L-Shaped | Fisherbrand, Thermo Fisher, MA, USA | RB-44103 | Colony Forming Units |
| T25 TC flask, angled neck, filter cap, sterile, Nunc | Thermo Fisher, Massachusetts, USA | 156367 | Mycobacterium liquid culture |
| THP-1 cell line | ATCC, Virginia, USA | ATCC TIB-202 | Macrophage cell culture |
Références
- WHO. Global Tuberculosis Report. World Health Organization. , (2020).
- Russell, D. G. Mycobacterium tuberculosis and the intimate discourse of a chronic infection. Immunological Reviews. 240 (1), 252-268 (2011).
- Young, C., Walzl, G., Du Plessis, N. Therapeutic host-directed strategies to improve outcome in tuberculosis. Mucosal Immunology. 13 (2), 190-204 (2020).
- Angeby, K. A., et al. Evaluation of the BacT/ALERT 3D system for recovery and drug susceptibility testing of Mycobacterium tuberculosis. Clinical Microbiology and Infection. 9 (11), 1148-1152 (2003).
- Asmar, S., Drancourt, M. Rapid culture-based diagnosis of pulmonary tuberculosis in developed and developing countries. Frontiers in Microbiology. 6, 1184 (2015).
- Diacon, A. H., et al. Time to liquid culture positivity can substitute for colony counting on agar plates in early bactericidal activity studies of antituberculosis agents. Clinical Microbiology and Infection. 18 (7), 711-717 (2012).
- Diacon, A. H., et al. Time to positivity in liquid culture predicts colony forming unit counts of Mycobacterium tuberculosis in sputum specimens. Tuberculosis. 94 (2), 148-151 (2014).
- O'Sullivan, D. M., et al. Evaluation of liquid culture for quantitation of Mycobacterium tuberculosis in murine models. Vaccine. 25 (49), 8203-8205 (2007).
- Heinrichs, M., et al. Mycobacterium tuberculosis Strains H37ra and H37rv have equivalent minimum inhibitory concentrations to most antituberculosis drugs. International Journal of Mycobacteriology. 7 (2), 156-161 (2018).
- Sorrentino, F., et al. Development of an intracellular screen for new compounds able to inhibit mycobacterium tuberculosis growth in human macrophages. Antimicrobial Agents and Chemotherapy. 60 (1), 640-645 (2016).
- O'Leary, S. M., et al. Cigarette smoking impairs human pulmonary immunity to Mycobacterium tuberculosis. American Journal of Respiratory and Critical Care Medicine. 190 (12), 1430-1436 (2014).
- Ryan, R. C., O'Sullivan, M. P., Keane, J. Mycobacterium tuberculosis infection induces non-apoptotic cell death of human dendritic cells. BMC Microbiology. 11, 237 (2011).
- Keane, J., Remold, H. G., Kornfeld, H. Virulent mycobacterium tuberculosis strains evade apoptosis of infected alveolar macrophages. The Journal of Immunology. 164 (4), 2016-2020 (2000).
- Gao, X. F., Yang, Z. W., Li, J. Adjunctive therapy with interferon-gamma for the treatment of pulmonary tuberculosis: a systematic review. International Journal of Infectious Diseases. 15 (9), 594-600 (2011).
- Thorpe, T. C., et al. BacT/Alert: an automated colorimetric microbial detection system. Journal of Clinical Microbiology. 28 (7), 1608-1612 (1990).
- Gleeson, L. E., et al. Cutting edge: Mycobacterium tuberculosis induces aerobic glycolysis in human alveolar macrophages that is required for control of intracellular bacillary replication. The Journal of Immunology. 196 (6), 2444-2449 (2016).
- O'Connor, G., et al. Inhalable poly(lactic-co-glycolic acid) (PLGA) microparticles encapsulating all-trans-Retinoic acid (ATRA) as a host-directed, adjunctive treatment for Mycobacterium tuberculosis infection. European Journal of Pharmaceutics and Biopharmaceutics. 134, 153-165 (2019).
- O'Connor, G., et al. Sharpening nature's tools for efficient tuberculosis control: A review of the potential role and development of host-directed therapies and strategies for targeted respiratory delivery. Advanced Drug Delivery Reviews. 102, 33-54 (2016).
- Rodriguez, D. C., Ocampo, M., Salazar, L. M., Patarroyo, M. A. Quantifying intracellular Mycobacterium tuberculosis: An essential issue for in vitro assays. Microbiologyopen. 7 (2), 00588 (2018).
- Ortalo-Magne, A., et al. Molecular composition of the outermost capsular material of the tubercle bacillus. Microbiology. 141, 1609-1620 (1995).
- Stokes, R. W., et al. The glycan-rich outer layer of the cell wall of Mycobacterium tuberculosis acts as an antiphagocytic capsule limiting the association of the bacterium with macrophages. Infection and Immunity. 72 (10), 5676-5686 (2004).
- O'Sullivan, M. P., O'Leary, S., Kelly, D. M., Keane, J. A caspase-independent pathway mediates macrophage cell death in response to Mycobacterium tuberculosis infection. Infection and Immunity. 75 (4), 1984-1993 (2007).
- Cheon, S. H., et al. Bactericidal activity in whole blood as a potential surrogate marker of immunity after vaccination against tuberculosis. Clinical and Diagnostic Laboratory Immunology. 9 (4), 901-907 (2002).
- Nathan, C., Barry, C. E. TB drug development: immunology at the table. Immunological Reviews. 264 (1), 308-318 (2015).
- Bowness, R., et al. The relationship between Mycobacterium tuberculosis MGIT time to positivity and cfu in sputum samples demonstrates changing bacterial phenotypes potentially reflecting the impact of chemotherapy on critical sub-populations. Journal of Antimicrobial Chemotherapy. 70 (2), 448-455 (2015).
- Basu Roy, R., et al. An auto-luminescent fluorescent BCG whole blood assay to enable evaluation of paediatric mycobacterial responses using minimal blood volumes. Frontiers in Pediatrics. 7, 151 (2019).
- Christophe, T., et al. High content screening identifies decaprenyl-phosphoribose 2' epimerase as a target for intracellular antimycobacterial inhibitors. PLoS Pathogens. 5 (10), 1000645 (2009).
- Franzblau, S. G., et al. Comprehensive analysis of methods used for the evaluation of compounds against Mycobacterium tuberculosis. Tuberculosis. 92 (6), 453-488 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon