Method Article
Effiziente und skalierbare Produktion von humanen Huntingtin-Varianten in voller Länge in Säugetierzellen unter Verwendung eines transienten Expressionssystems
In diesem Artikel
Zusammenfassung
Wir bieten skalierbare Protokolle, die das Konstruktdesign, die transiente Transfektion sowie die Expression und Reinigung von humanen Huntingtin-Proteinvarianten in voller Länge in HEK293-Zellen abdecken.
Zusammenfassung
Full-length Huntingtin (FL HTT) ist ein großes (aa 1-3,144), ubiquitär exprimiertes, Polyglutamin (polyQ)-haltiges Protein mit einer Masse von etwa 350 kDa. Während die zelluläre Funktion von FL HTT nicht vollständig verstanden wird, ist eine mutierte Expansion des PolyQ-Trakts über ~ 36 Wiederholungen mit der Huntington-Krankheit (HD) verbunden, wobei die PolyQ-Länge ungefähr mit dem Erkrankungsalter korreliert. Um den Einfluss der Struktur auf die Funktion des mutierten HTT (mHTT) besser zu verstehen, werden große Mengen des Proteins benötigt. Die Submilligrammproduktion von FL HTT in Säugetierzellen wurde unter Verwendung der Doxycyclin-induzierbaren stabilen Zelllinienexpression erreicht. Die Proteinproduktion aus stabilen Zelllinien hat jedoch Einschränkungen, die mit transienten Transfektionsmethoden überwunden werden können.
Dieser Artikel stellt eine robuste Methode zur Herstellung von FL HTT und seinen Varianten aus Codon-optimierten Plasmiden durch transiente Transfektion mit Polyethylenimin (PEI) vor. Die Methode ist skalierbar (>10 mg) und liefert konstant 1-2 mg/L Zellkultur von hochgereinigtem FL HTT. In Übereinstimmung mit früheren Berichten wurde festgestellt, dass der Zustand der gereinigten Lösung von FL HTT sehr dynamisch ist. Das Protein neigt zur Bildung von Dimeren und Oligomeren hoher Ordnung. Ein Schlüssel zur Verlangsamung der Oligomerbildung ist die schnelle Isolierung der monomeren Fraktionen aus den dimeren und oligomeren Fraktionen hoher Ordnung während der Größenausschlusschromatographie.
Die Größenausschlusschromatographie mit Mehrwinkellichtstreuung (SEC-MALS) wurde verwendet, um den Dimer- und oligomeren Gehalt höherer Ordnung von gereinigtem HTT zu analysieren. Es wurde keine Korrelation zwischen der FL HTT polyQ-Länge (Q23, Q48 und Q73) und dem Oligomergehalt beobachtet. Das exon1-gelöschte Konstrukt (aa 91-3,144) zeigte eine vergleichbare Oligomerisierungsneigung zu FL HTT (aa 1-3,144). Herstellungs-, Reinigungs- und Charakterisierungsmethoden mittels SEC/MALS-Brechungsindex (RI), Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE), Western Blot, Native PAGE und Blue Native PAGE werden hierin beschrieben.
Einleitung
Die Huntington-Krankheit (HD) ist eine seltene neurodegenerative Erkrankung, die hauptsächlich durch instabile und unwillkürliche motorische Bewegungen sowie kognitive und psychiatrische Veränderungen wie Persönlichkeitsveränderungen und Apathie gekennzeichnetist 1,2. Die Huntington-Krankheit ist mit einer Erweiterung des CAG-Wiederholungstrakts im Exon 1 des Huntingtin-Gens (HTT) auf mehr als 35 Wiederholungen verbunden, wobei eine höhere Anzahl von CAG-Wiederholungen mit einem früheren Ausbruch der Krankheit korreliert 3,4. Das translationale Produkt von HTT, das Huntingtin-Protein (HTT), ist an der neuronalen Lebensfähigkeit und der Gehirnentwicklung beteiligt 5,6,7,8,9.
HTT ist ein Gerüstprotein, von dem berichtet wird, dass es an einer Vielzahl von zellulären Prozessen, Vesikeltransport, Zellteilung, Ziliogenese und Autophagie beteiligt ist10,11. Die molekulare Pathogenese der Huntington-Krankheit ist jedoch nicht ganz klar, und die Identifizierung von Schlüsselproteininteraktoren, die die pathologischen Auswirkungen von polyQ-expandiertem mHTT vermitteln, fehlt. Einige Forschungsergebnisse deuten auf einen Gewinn der toxischen Funktion durch mHTT hin, der durch die Oligomerisierungsneigung des expandierten HTT-Proteins angetrieben wird, da HTT-Aggregate in Neuronen und Gliazellen bei Huntington-Patienten und Tiermodellen der Krankheit identifiziert wurden 12,13,14,15,16,17 . Um die Untersuchung der Funktion und Struktur von FL-HTT- und mHTT-Varianten voranzutreiben und Forscher mit hochwertigen Proteinstandards für die Assay-Entwicklung zu versorgen, ist eine robuste und skalierbare Versorgung mit homogenen rekombinanten Proteinen erforderlich.
Aufgrund seiner Größe (aa 1-3.144, Nummerierung basierend auf der PolyQ-Länge Q23), proteolytischer Instabilität und Aggregationsneigung hat sich FL HTT als schwierig erwiesen, als lösliches Protein zu exprimieren und zu isolieren. Zuvor wurde die Exon-1-Region (aa 2-90) von HTT exprimiert und in großem Maßstab mit verschiedenen Tags gereinigt, die die Löslichkeit des Proteins in Escherichia coli18,19,20 erhöhen können. FL HTT wurde zuerst in einem Insektenzellexpressionssystem unter Verwendung von Baculovirus 21,22 exprimiert und gereinigt, und niedrig aufgelöste 30 Å Elektronenmikroskopie (EM) Strukturen chemisch vernetzter FL Q23-HTT und Q78-HTT wurden berichtet23. Die Untersuchung der HTT-Struktur wurde weiter vorangetrieben, als die Produktion von FL Q17, Q46 und Q128-HTT mit nativen posttranslationalen Modifikationen (PTMs) in menschlichen Zellen unter Verwendung stabiler Zelllinien oder Adenovirus-Expressionssysteme erreicht wurde24. Diese Studien deuten darauf hin, dass, obwohl gereinigtes HTT hauptsächlich im monomeren Zustand existiert, es auch dazu neigt, Oligomere und Aggregate hoher Ordnung zu bilden.
Die analytische Ultrazentrifugation von FL Q128-HTT mit einer stark expandierten PolyQ-Region lieferte mehr oligomere und aggregierte Fraktionen als das Protein mit der nicht expandierten polyQ-Region24. Unter Verwendung einer stabilen Zelllinie wurde erfolgreich eine Strategie zur Stabilisierung von FL HTT durch Co-Expression mit dem Interaktionspartner HAP40 angepasst. Eine Kryo-EM-Struktur des FL HTT- und HAP40-Komplexes wurde mit einer durchschnittlichen Auflösung von 4 Å unter Verwendung des gereinigten Proteinkomplexes (PDB:6EZ8)25 gelöst. Diese Co-Expressionsstrategie wurde erfolgreich an ein Baculovirus-System angepasst, und eine Reihe hochwertiger HTT-Varianten mit unterschiedlichen PolyQ-Längen wurden aus Insektenzellen exprimiert und gereinigt26. Seitdem wurden weitere Kryo-EM-Strukturen des HTT-Komplexes mit variablen PolyQ-Längen und HAP40- und höher aufgelösten Strukturen gelöst und in der Protein Data Base27,28 (PDB: 7DXK, 7DXH, 6X9O) hinterlegt.
Wir optimierten eine Transfektions- und Expressionsmethode in HEK293-Zellen unter Verwendung von Polyethylenimin (PEI) für eine schnelle transiente Expression von FL HTT. Als Proof-of-principle wurden FL-HTT-Varianten mit 23 Glutaminen (FL Q23-HTT) zunächst gereinigt und unter Verwendung einer Modifikation einer zuvor beschriebenen Reinigungsmethodecharakterisiert 24. Diese transiente Transfektionsmethode ist bequem, hocheffizient und skalierbar. Es kann gereinigtes HTT mit Ausbeuten von 1-2 mg/L produzieren, vergleichbar mit der beschriebenen stabilen Zelllinienmethode24. Da das Protein in einer menschlichen Zelllinie produziert wird, ist es wahrscheinlicher, dass das produzierte HTT native menschliche PTMs aufweist, wenn es einer massenspektrometrischen Proteomikanalyse 11,29,30,31 unterzogen wird. Milligramm-Mengen der FL Q48-HTT, FL Q73-HTT und exon1-deleted (ΔExon1-HTT) Varianten von FL HTT wurden produziert, was zeigt, dass die transiente Expressionsmethode besonders nützlich ist, um schnell alternative Varianten von HTT herzustellen, ohne den zeitaufwändigen Aufwand zu benötigen, der erforderlich ist, um stabile Zelllinien für die Produktion zu etablieren.
Das folgende Protokoll veranschaulicht die Standardmethode, die im Labor dieser Autoren zur Zellkultur, Transfektion, Proteinreinigung und Proteincharakterisierung nach der Reinigung verwendet wird, um FL Q23-HTT aus einer 2-L-Zellkultur herzustellen. Das Protokoll kann auf größere Kulturen skaliert oder angepasst werden, um andere HTT-Varianten zu reinigen. Bis zu 10 L Zellkulturen von FL HTT und verschiedene Orts- oder Trunkierungsmutationen von HTT und HTT-Homologen wurden erfolgreich im Labor mit dem gleichen Protokoll durchgeführt. Gereinigtes FL HTT enthält einen hohen Anteil an Monomeren sowie Dimere und Oligomere höherer Ordnung. Das gleiche Aggregatprofil wird bei den produzierten Varianten beobachtet (Q23, Q48, Q73 und gelöschtes Exon1). Da eine Aggregation auftreten kann, wenn nicht die richtige Sorgfalt angewendet wird, wurde eine Formulierungs- und Gefrier-Tau-Stabilitätsstudie durchgeführt, um die besten Bedingungen für die Proteinhandhabung zu ermitteln. Methoden wie Blue Native PAGE und SEC/MALS-RI werden ebenfalls beschrieben, um den HTT-Oligomergehalt im Rahmen des Qualitätskontrollprozesses zu analysieren. Zum Nutzen der Huntington-Forschungsgemeinschaft werden die in dieser Studie beschriebenen Plasmide und HTT-Proteine auch im HD Community Repository am Coriell Institute (www.coriell.org/1/CHDI) deponiert.
Protokoll
1. Design und Herstellung von Konstrukten für FLAG-getaggte HTT-Säugetierexpression
- Rufen Sie die menschliche HTT-Proteinsequenz in voller Länge (P42858) vom National Center for Biotechnology Information (https://www.ncbi.nlm.nih.gov/) ab.
HINWEIS: Forscher müssen mit den Domänenorganisationen von HTT vertraut sein und die HTT-Kern-3D-Struktur beibehalten, wenn sie Konstrukte für HTT-Mutanten entwerfen. - Fordern Sie einen Gensynthesedienst an, um eine Codonoptimierung für die menschliche Zellexpression basierend auf der Sequenz von P42858 durchzuführen. Ändern Sie die PolyQ-Zahl von Q16 auf die gewünschte Q-Länge (Q23 wurde hier als erstes Konstrukt gewählt) und synthetisieren Sie das HTT-Gen in voller Länge.
HINWEIS: Das synthetisierte Codon-optimierte Q23-HTT-Konstrukt in voller Länge wurde in dieser Studie als Insert in das pUC18-Plasmid geliefert. - Optional: Fügen Sie Funktionen hinzu, um das Klonen verschiedener Q-Längen und die Reinigung in den Konstrukten zu erleichtern.
HINWEIS: Eine Spaltungsstelle des Tabakätzvirus (TEV) und ein FLAG-Reinigungstag (AAAENLYFQGDYKDDDDK) wurden dem C-terminalen Ende der Konstrukte hinzugefügt. Zwei HindIII-Stellen wurden in den Konstrukten entworfen, um die polyQ-Region zu umfassen (die übersetzte Proteinsequenz wird durch die Einführung von HindIII-Stellen nicht verändert). Dies ermöglicht es dem Forscher, die Q-Länge von HTT durch Einschränkung der Enzymverdauung und -ligation zu verändern, ohne das vollständige HTT-Gen neu zu synthetisieren.
2. Klonen Sie die synthetisierten HTT-Konstrukte in pcDNA3.1.
- Aufschluss von 5 μg pUC18-Q23-HTT und 5 μg pcDNA3.1 unter Verwendung von 2 μL NheI und PmeI bei jeweils 37 °C für 2 h.
- Führen Sie ein 0,5% w/v Agarosegel aus und reinigen Sie das Q23-HTT-Fragment und den verdauten pcDNA3.1-Vektor mit einem Agarose-Gel-Extraktionskit. Quantifizieren Sie die Konzentrationen gereinigter DNA mit OD280 mit einem UV-Spektrometer, das Mikroliter von Proben messen kann.
HINWEIS: OD260/280 im Bereich von 1,8 bis 2,0 wird typischerweise beobachtet. Das synthetisierte FL HTT wird als Insert mit NheI und PmeI an beiden Enden in einem pUC18-Plasmid geliefert. Verwenden Sie andere Restriktionsenzyme, wenn HTT anders synthetisiert wird. - Verwenden Sie 10 ng des verdauten pcDNA3.1-Vektors in der Reaktion. Ligieren Sie die gereinigten DNAs bei einem Molverhältnis von 1:1 (HTT:pcDNA3.1) in einer 10 μL-Reaktion bei Raumtemperatur für 5 min unter Verwendung von T4-DNA-Ligase.
- Transformieren Sie das ligierte Produkt in kompetente E. coli-Zellen (siehe Materialtabelle) unter Verwendung des vom Ligasehersteller angegebenen Protokolls.
- Nehmen Sie 6 einzelne Kolonien auf und machen Sie über Nacht Kulturen in 4-6 ml LB, ergänzt mit 100 μg / ml Carbenicillin bei 37 ° C.
- Weisen Sie 1 ml von jeder Nachtkultur zu. Fügen Sie Glycerin zu 25% v/v hinzu und speichern Sie die Glycerinbrühe bei -80 °C. Reinigen Sie die verbleibende Übernachtkultur mit einem Mini-Prep-Kit gemäß den im Benutzerhandbuch angegebenen Schritten.
- Sequenzieren Sie alle Plasmide mit Sequenzierungsprimern, die sich über den Transkriptionsbereich des Plasmids erstrecken. Wählen Sie eine Glycerinbrühe, die die richtige Reihenfolge als Master-Glycerinbrühe hat, und verwerfen Sie den Rest.
- Optional: Fordern Sie einen Gensynthesedienst an, um DNA mit den verschiedenen Q-Längen (Q48, Q73 und Exon1) zu synthetisieren, die die beiden HindIII-Stellen im pcDNA3.1-Q23-HTT-Plasmid umfassen. Verdauen Sie pcDNA3.1-Q23-HTT und die neu synthetisierten DNAs unter Verwendung von HindIII und religieren Sie sie mit T4-Ligase wie in den Schritten 2.2-2.7, um FL HTT mit verschiedenen PolyQ-Längen im pcDNA3.1-Plasmid herzustellen.
HINWEIS: Die in dieser Studie verwendeten Plasmidkonstrukte sind auch direkt im HD Community Repository am Coriell Institute verfügbar (www.coriell.org/1/CHDI); siehe Materialtabelle.
3. GIGA bereitet endotoxinfreie Plasmid-DNA für großflächige Transfektion vor
- Streifen Sie die bakteriellen Glycerinbestände von pcDNA3.1-Q23-HTT-TEV-FLAG auf einer LB-Agarplatte mit Carbenicillin (100 μg/ml). Die Platte bei 37 °C für 16-24 h inkubieren, bis einzelne Kolonien entstehen.
- Nehmen Sie eine einzelne Kolonie auf, inokulieren Sie eine 5-ml-Starterkultur in einem reichhaltigen Medium, das für die Plasmidamplifikation mit Carbenicillin (100 μg / ml) formuliert ist, und wachsen Sie bei 37 ° C für 8 h.
- Wählen Sie ein endotoxinfreies GIGA Plasmid-Reinigungskit. Folgen Sie den Schritten im Handbuch des Plasmid-GIGA-Kits, um das pcDNA3.1-Q23-HTT-TEV-FLAG-Plasmid zu reinigen.
- Messen Sie die Plasmid-Endotoxin-Spiegel mit einem Limulus-Amöbozyten-Lysat (LAL)-basierten Endotoxin-Quantifizierungskit. Befolgen Sie das im Handbuch des Herstellers beschriebene Verfahren.
HINWEIS: Eine qualitativ hochwertige Plasmidreinigung mit niedrigem Endotoxingehalt ist unerlässlich, um eine gute Transfektionseffizienz zu erzielen. Mit diesem Protokoll können 20-40 mg Plasmid (supercoiled form >80%) pro L Bakterienkultur bei Plasmidkonzentrationen > 4 mg/ml erhalten werden. Ein ordnungsgemäß gereinigtes Plasmid sollte einen Endotoxingehalt < 30 EU/mg aufweisen. OD260/280 im Bereich von 1,8 bis 2,0 wird typischerweise beobachtet.
4. Großflächige Transfektion von 2 L HEK293-Zellen durch Polyethylenimin (PEI)
- 1 g PEI 25K auf 1 L endotoxinfreies Wasser unter Rühren geben. Stellen Sie den pH-Wert mit 100 mM HCl auf 2,0 ein und rühren Sie, bis sich das gesamte PEI 25K auflöst. Stellen Sie den pH-Wert mit 100 mM NaOH-Lösung auf 7,0 ein und filtern Sie durch einen 0,2-μm-Filter. Aliquot und bis zu einem Jahr bei -20 °C lagern.
HINWEIS: Aliquots von PEI können bis zu zwei Wochen bei 4 °C aufbewahrt werden, sollten aber nach dem Auftauen niemals wieder eingefroren werden. - Vermehrung von HEK293-Zellen im Wachstumsmedium (siehe Materialtabelle), ergänzt mit Penicillin-Streptomycin (Endkonzentration bei 5 U/ml für Penicillin und 5 μg/ml für Streptomycin) in einem befeuchteten Shaker-Inkubator bei 37 °C, 90 U/min, 5%CO2 für 18-24 h. Verdünnen Sie die Zellen auf 2 L bei einer Dichte von ~1,2 × 106 Zellen/ml mit dem Wachstumsmedium in 5 L Erlenmeyerkolben einen Tag vor der Transfektion.
- Die Zellen bei 37 °C, 90 U/min, 5%CO2 für 18-24 h weiter wachsen lassen. Messen Sie die Zellparameter mit einem automatischen Zellzähler, der die Zelldichte und Lebensfähigkeit gemäß dem Benutzerhandbuch messen kann.
HINWEIS: Die Zelldichte sollte sich verdoppeln und die Lebensfähigkeit sollte >95% betragen. Die Zelldichte vor der Transfektion sollte ca. 2,0 × 10 6-2,4 × 106 Zellen/ml betragen. Verdünnen Sie die Zellen vor der Transfektion bei Bedarf auf die gewünschte Dichte. - Berechnen Sie die Mengen an Plasmid und PEI, die für die Transfektion erforderlich sind; Verwenden Sie 1 mg Plasmid und 3 mg PEI für die Transfektion jedes Liters Zellkultur. Weisen Sie 2 mg Plasmid und 6 mg PEI zu, die für eine 2-L-Transfektion benötigt werden.
- Das Plasmid und PEI werden einzeln zu einem Volumen phosphatgepufferter Kochsalzlösung verdünnt, das 1/20des Gesamtvolumens der Zellkultur entspricht (jeweils 100 ml für eine 2 L Transfektion) und bei Raumtemperatur für 5 min inkubieren. Mischen Sie das verdünnte Plasmid und PEI durch leichtes Schwenken und inkubieren Sie die Mischung bei Raumtemperatur für 30 min.
HINWEIS: Die Mischung erscheint nach der Inkubation leicht trüb. - Fügen Sie die Mischung in die Zellkultur und schwenken Sie sie vorsichtig, um sie zu mischen.
- Die Zellen wachsen bei 37 °C, 5% CO2, 90 U/min für 24 h.
- 2 M Natriumbutyratlösung zu einer Endkonzentration von 2 mM zugeben. 1:1000 (v/v) Antiklumpungsmittel und 1:1000 (v/v) Antischaummittel in die Kultur geben.
- Der Kolben wird in einen befeuchteten Schüttelbrutschrank bei 32 °C, 90 U/min, 5 % CO2 gegeben und 48 h lang weiter wachsen lassen.
- Messen Sie die Zellparameter, einschließlich Zelldichte und Lebensfähigkeit, mit dem automatischen Zellzähler gemäß der Bedienungsanleitung.
- Transfer 2,0 × 10 6 Zellen (Vol = 2,0 × 106/Zelldichte) in ein Mikrozentrifugenröhrchen. Pelletieren Sie die Zellen bei 2.000 × g für 1 min in einer Zentrifuge zum Western Blotting in Abschnitt 5.
- Ernten Sie die Zellen durch Zentrifugation bei 2.000 × g für 30 min und lagern Sie das Zellpellet vor der Reinigung bei -80 °C.
5. SDS-PAGE und Western Blot von HEK293-Zelllysat zur Abschätzung des HTT-Expressionsniveaus
- Nehmen Sie ein Aliquot von 2,0 × 106 zuvor eingefrorenen Zellen (Schritt 4.11) aus der groß angelegten Transfektion der HEK293-Zellkultur. Fügen Sie 250 μL Tris-gepufferte Kochsalzlösung (TBS) hinzu, ergänzt mit 50 μg / ml Digonin, 5 mM EDTA und 1x Proteaseinhibitor-Cocktail, und suspendieren Sie das Zellpellet durch mehrmaliges Absaugen mit einer Pipette.
- Drehen Sie die Röhrchen vorsichtig für 30 min bei 4 °C mit einem Minirotator, um die Zellen zu lysieren. Pelletieren Sie das unlösliche Material durch Zentrifugieren bei 17.000 × g für 5 min.
- 1/3des Volumens des 4x reduzierenden Lithiumdodecylsulfat (LDS) Ladepuffers zum Überstand geben und bei 70 °C für 10 min erhitzen.
- Laden Sie 5-20 μL Zelllysat auf ein vorgefertigtes 3-8% Tris-acetat PAGE Gel. Unter Verwendung des gelkompatiblen 1x Tris-acetate SDS-Laufpuffers läuft das Gel in einem Konstantspannungsmodus bei 150 V für 60 min.
HINWEIS: Tris-acetat SDS-PAGE wurde für die FL-HTT-Analyse verwendet, da es für Proteine mit einem Molekulargewicht über 300 kDa eine höhere Auflösung als andere Arten von SDS-PAGE erzeugt. Proteine, die in dieser Studie verwendet werden, sind auch direkt aus dem HD Community Repository am Coriell Institute erhältlich (www.coriell.org/1/CHDI); siehe Materialtabelle. - Um Western Blotting durchzuführen, montieren Sie ein Transfersandwich mit einem Transferpuffer-äquilibrierten dicken Transferpapier, einer Methanol-aktivierten Polyvinylidenfluorid-Membran (PVDF) und einem SDS-PAGE-Gel. Übertragen Sie die Proteine auf die PVDF-Membran mit einem halbtrockenen Western Blotter gemäß der Bedienungsanleitung des Herstellers.
HINWEIS: Typischerweise sind 20-30 min bei 135 mA für eine 10 cm x 10 cm große Membran ausreichend. - Zerlegen Sie das Transfersandwich und blockieren Sie die Membran in TBST (20 mM Tris pH 7,4, 150 mM NaCl und 0,1% v/v Tween-20), ergänzt mit 5% w/v fettfreier Milch.
- Inkubieren Sie die Membran auf einer Wippe für 1 h bei Raumtemperatur mit 15 ml primärem Antikörper (1:2.500 Verdünnung für monoklonale Anti-FLAG-Antikörper und 1:2.000 für alle anderen primären Antikörper).
HINWEIS: Primäre Antikörper, die in dieser Studie verwendet werden, sind Anti-FLAG M2, MAB5492, MAB5490, MAB2166, MAB3E10, MAB4E10, MAB2168, MAB8A4 (siehe Materialtabelle). - Waschen Sie die Membran 3 x 5 min mit 30-50 ml TBST.
- Inkubieren Sie die Membran auf einer Wippe mit einem fluoreszierenden farbstoffkonjugierten Ziegen-Anti-Maus-IgG-Sekundärantikörper bei 1:15.000, Raumtemperatur, in 15 ml TBST, der 5% w/v Trockenmilch enthält.
- Visualisieren Sie die Western-Blot-Bänder auf einem fluoreszierenden Imager unter Verwendung der Wellenlänge, die für den sekundären Antikörper spezifisch ist. Quantifizieren Sie das Bandsignal mit der Software, die dem Imager gemäß der Bedienungsanleitung beiliegt.
HINWEIS: Quantitatives Western Blotting kann standardmäßig mit gereinigtem HTT durchgeführt werden. Ein linearer Standardbereich von HTT ist gerätespezifisch und wurde in diesem Labor von 25 ng bis 250 ng HTT pro Spur unter Verwendung eines Anti-FLAG-Antikörpers festgelegt. Der Western Blot von HTT sollte frei von Abbau sein; Typischerweise wird ein Gesamt-HTT-Expressionsniveau von 2-4 pg/Zelle beobachtet. Einzelheiten zur Durchführung eines quantitativen Western Blots finden Sie in einem zuvor veröffentlichten Protokoll32 .
6. Schnelle Proteinflüssigkeitschromatographie (FPLC) Reinigung von HTT mit Anti-FLAG-Säule und SEC
- Anti-FLAG-Reinigung
- Schätzen Sie die Menge an FLAG-Harz, die für die Reinigung benötigt wird (typischerweise 12 ml Anti-FLAG M2-Affinitätsharz zur Reinigung von 2-4 L transfizierter Zellkultur). Packen Sie 12-25 ml Anti-FLAG-Harz auf eine leere Säule (siehe Materialtabelle) mit FPLC bei einer Durchflussrate von 4 ml/min unter Verwendung von Puffer A (Tabelle 1). Stellen Sie die Höhe des Kolbens so ein, dass keine Lücke zwischen dem Ende des Kolbens und dem Harzbett besteht.
- Bei einem Verhältnis von 10 ml Lysepuffer pro 1 g Zellpellet wird das Zellpellet aufgetaut und in kaltem Lysepuffer suspendiert (Tabelle 1).
- Führen Sie die Zellsuspension einmal durch einen High-Shear-Homogenisator bei 10.000 psi. Das Lysat wird durch Zentrifugieren bei 20.000 × g für 1 h in einer Zentrifuge mit einem kompatiblen Festwinkelrotor geklärt.
- Programmieren Sie das FPLC (siehe Materialtabelle für die in der Studie verwendete Software) und führen Sie die folgenden Sequenzen aus.
- Laden Sie das geklärte Lysat über die Probenpumpe.
- Waschen Sie mit 4 Spaltenvolumina (CVs) von Puffer A (Tabelle 1).
- Mit 4 CVs Puffer B waschen (Tabelle 1).
- Mit 8 CVs Puffer C waschen (Tabelle 1).
- Mit 3 CVs Puffer D waschen (Tabelle 1).
- Mit 3 CVs Elution Buffer waschen (Tabelle 1).
- Analysieren Sie 10 μL der Peakanteile mit SDS-PAGE. Sammeln und kombinieren Sie die Spitzenfraktionen mit der gewünschten Reinheit. Sparen Sie ~50 μL der kombinierten Eluate für die SDS-PAGE-Analyse.
HINWEIS: Normalerweise erscheint ein einzelner Peak, und alle im Peak eluierten Brüche enthalten ~90% reines HTT. - Regenerieren Sie eine Anti-FLAG-Spalte mit 5 CVs Regenerationspuffer (Tabelle 1) und gleichen Sie die Spalte mit 5 CVs von Puffer A wieder aus.
HINWEIS: Anti-FLAG-Harz kann bis zu fünfmal wiederverwendet werden oder bis die relative Ausbeute / Liter auf 50% der ersten Reinigung fällt.
- Aufreinigung von Größenausschlusssäulen (SEC) mithilfe einer SEC-Spalte
- Preequilibrieren Sie eine SEC-Säule, die die Trennung von Proteinen mit einem Molekulargewicht (MW) > 500 kDa ermöglicht (siehe Materialtabelle für die verwendete Säule) unter Verwendung von 2 × CV SEC-Puffer (Tabelle 1).
- Laden Sie den Anti-FLAG-Eluat (ab Schritt 6.1.5) direkt über einen 50-ml-Superloop. Führen Sie 1,2 × CV SEC-Puffer pro Injektion aus. Führen Sie die SEC-Trennung über Nacht bei 4 °C durch.
HINWEIS: Maximal 5 ml oder 15 ml Proteinprobe können auf die in dieser Studie ausgewählten SEC-Spalten geladen werden. Programmieren Sie den FPLC so, dass mehrere Injektionen automatisch durchgeführt werden können. Beispielmethodenskripts sind auch als Zusatzdatei 1 und Zusatzdatei 2 enthalten. - Vergleichen Sie das Elutionsprofil mit dem Standard-HTT-Elutionsprofil, um die Monomer-, Dimer- und höhergeordneten oligomeren Peaks zu unterscheiden. Poolen Sie die monomeren HTT-Fraktionen basierend auf dem Elutionprofil der SEC-Säule. Falls gewünscht, bündeln Sie die höhergeordneten oligomeren und dimeren HTT-Fraktionen getrennt.
- Das gepoolte HTT-Protein wird mit einem 100-kDa-Zentrifugalkonzentrator bei 4 °C konzentriert. Berechnen Sie die Proteinkonzentrationen, indem Sie ihre OD280-Werte durch die jeweiligen Extinktionskoeffizienten dividieren (die theoretischen Extinktionskoeffizienten von Q23-HTT, Q48-HTT, Q73-HTT und ΔExon1-HTT betragen 0,776, 0,769, 0,762 bzw. 0,798 (mg / ml) - 1 cm-1für die Berechnung). Halten Sie die HTT-Konzentration ≤ 1,0 mg / ml.
HINWEIS: Es ist wichtig, den Konzentrationsprozess zu überwachen, da eine Überkonzentration zu einer Aggregation führt. - Aliquot das gereinigte HTT-Protein in kryosicheren Mikrozentrifugenröhrchen in einem Volumen < 100 μL. Die Aliquots mit flüssigem Stickstoff schockgefrieren und bei -80 °C lagern.
7. Analytische HPLC SEC-MALS-dRI zur Analyse der HTT-Polydispersität
- Führen Sie alle analytischen SEC-MALS bei 4 °C auf einem Hochleistungsflüssigkeitschromatographiesystem (HPLC) in Verbindung mit einem UV-Detektor, einem Mehrwinkel-Lichtstreudetektor und einem Detektor für den differentiellen Brechungsindex (dRI) durch.
- Bevor Sie die UHPLC-Säule an das System anschließen, reinigen Sie die Pumpe und die Detektoren mit gefiltertem (0,1 μm) HPLC-Wasser.
- Verbinden Sie die UHPLC-Säule (siehe Materialtabelle für die verwendete Säule) mit dem System. Die Säule wird mit gefiltertem (0,1 μm) Wasser und dann dem SEC-MALS-Puffer (Tabelle 1) ausgeglichen, bis alle Detektorsignale die Basislinie erreichen.
- Injizieren Sie 2 μL 6 mg/ml Rinderserumalbumin (BSA) mit einer Flussrate von 0,3 ml/min für 15 min pro Injektion und überprüfen Sie die Datenqualität. Führen Sie Normalisierung, Spitzenausrichtung und Banderweiterungskorrektur basierend auf dem BSA-Profil durch und erstellen Sie eine Vorlage für die folgenden HTT-Stichprobenläufe.
- Eine Durchstechflasche der FL Q23-HTT-Probe in einem Wasserbad bei Raumtemperatur mit einem Schwimmer schnell auftauen. Filtern Sie das HTT durch einen 0,1 μm Spinfilter. Injizieren Sie 2-4 μL der HTT-Probe und lassen Sie sie 15 min bei 4 °C bei einer Flussrate von 0,3 ml/min laufen.
- Analysieren Sie die chromatographischen und lichtstreuenden Daten mit der mitgelieferten Software (siehe Materialtabelle). Verwenden Sie den dRI-Detektor als Konzentrationsdetektor und verwenden Sie 0,185 als Brechungsindex-Inkrement (dn/dc) für HTT. Generieren Sie ein Zimm-Diagramm, um die gewichtsgemittelte Molekülmasse für jeden Peak33,34 zu bestimmen.
HINWEIS: Das Brechungsindex-Inkrement von HTT wird als 0,185 unter Verwendung der SEDFIT-Software35 und der primären Aminosäuresequenz von HTT als Eingabe berechnet.
HINWEIS: HTT-Monomer MW wird durch SEC-MALS bei ~370 kDa ± 30 kDa bestimmt. Gereinigtes HTT hat typischerweise einen Monomergehalt zwischen 60 und 75% (in diesem Labor). Ein niedriger Monomergehalt kann darauf hindeuten, dass bei der Handhabung mehr Vorsicht geboten ist, um eine Aggregation zu verhindern.
8. Blue Native PAGE zur Analyse der HTT-Polydispersität
- Bereiten Sie 1 l Anodenpuffer vor, indem Sie 50 ml 20x Blue Native PAGE Laufpuffer (siehe Materialtabelle) mit 950 ml H2 O mischen. Bereiten Sie 2 l dunkelblauen Kathodenpuffer vor, indem Sie 100 ml 20x Blue Native PAGE Laufpuffer und 100 mL Blue Native PAGE Kathodenadditiv (20x) mit 1.800 mLH2O mischen. Kühlen Sie die Puffer vor Gebrauch auf 4 °C.
- Eine Durchstechflasche mit FL Q23-HTT-Probe in einem Wasserbad bei Raumtemperatur mit einem Schwimmer schnell auftauen. Bewahren Sie das aufgetaute Protein vor Gebrauch auf Eis auf.
- Mischen Sie 5 μg FL Q23-HTT (~1 mg/ml), 1 μL 0,5% G250-Additiv, 2,5 μL 4x Blue Native PAGE-Probenpuffer und Wasser, um das Endvolumen auf 10 μL zu bringen.
- Laden Sie die gemischte FL Q23-HTT-Probe auf ein 3-12% vorgefertigtes Bis-Tris-Gel. Laden Sie 7,5 μL des ungefärbten Proteinstandards in dasselbe Gel wie das Standard.
- Füllen Sie die Vorderseite des Tanks mit dunkelblauem Kathodenpuffer und die Rückseite des Tanks mit Anodenpuffer.
HINWEIS: Füllen Sie die Puffer nach dem Laden der Probe, um eine einfache Visualisierung beim Laden der Proben zu ermöglichen. - Lassen Sie das Gel bei 150 V für 120 Minuten in einem kalten Raum laufen.
- Das Gel mit Entfärbungslösung (Tabelle 1) färben, bis Banden beobachtet werden; Übertragen Sie das Gel auf Wasser. Visualisieren und dokumentieren Sie das Gel auf einer Bildgebungsstation.
HINWEIS: Blue Native PAGE wurde ursprünglich zur Analyse von Membranproteinen entwickelt. Es wurde in diesem Labor als alternative Methode zur Abschätzung des monomeren Gehalts von HTT angepasst. Es bindet an die hydrophoben Bereiche von HTT und verhindert, dass es unter Pufferbedingungen ohne Reinigungsmittel Aggregate bildet. Traditionelles natives PAGE ohne Verwendung von Coomassie-Blau G250 bewirkt, dass HTT lösliche Oligomere und Aggregate bildet, wahrscheinlich aufgrund der vielen hydrophoben Taschen, die in HTT vorhanden sind.
9. SDS PAGE gefolgt von Coomassie oder Silberfärbung zur Analyse der HTT-Reinheit
- Fügen Sie 4x LDS-Probenpuffer und 10x reduzierendes Reagenz zu gereinigtem FL Q23-HTT hinzu, um die Endkonzentration des Ladepuffers und des Reagenzes auf 1x zu erhöhen.
- Die Probe wird 10 min lang auf einem trockenen Heizblock bei 70 °C erhitzt.
- Laden Sie maximal 1 μg Protein pro Vertiefung auf ein 3-8% Trisacetat-Gel und laufen Sie bei 150 V für 1 h mit Tris-acetate SDS-Laufpuffer.
HINWEIS: Proteine, die in dieser Studie verwendet werden, sind auch direkt aus dem HD Community Repository am Coriell Institute erhältlich (www.coriell.org/1/CHDI); siehe Materialtabelle. - Coomassie-Färbung
- Waschen Sie das Gel mit H2O für 5 min.
- Färben Sie das Gel in der Coomassie-Färbelösung (Tabelle 1), indem Sie das Gel in 30 ml Färbelösung für 15 min schaukeln.
- De-Färben, indem Sie das Gel in 50 mlH2Ofür 5 min schaukeln. Visualisieren und dokumentieren Sie das Coomassie-gefärbte Gel auf einer Bildgebungsstation.
- Silberfleck mit einem handelsüblichen Silberflecken-Kit.
- Nach SDS-PAGE fixieren Sie das Gel mit Fixierlösung (Tabelle 1) für 1 h bis über Nacht bei Raumtemperatur.
- Führen Sie die Färbung durch, waschen und entwickeln Sie gemäß den Anweisungen des Kits.
- Stoppen Sie den Entwicklungsschritt sofort, sobald die Bänder die gewünschte Intensität erreicht haben.
- Dokumentieren Sie das Gel in einem Gel-Dokumentationssystem, das mit einer sichtbaren Lichtquelle ausgestattet ist.
HINWEIS: HTT, gereinigt bei >95%, kann durch Coomassie und Silberfärbung mit diesem Protokoll nachgewiesen werden. Einzelheiten zur Durchführung der quantitativen Proteinanalyse finden Sie in einem zuvor veröffentlichten Protokoll32 .
Ergebnisse
Ein transienter Expressionsvektor (pcDNA3.1-Q23-HTT-TEV-FLAG, Abbildung 1A) ist für die schnelle Produktion von FL Q23-HTT in Säugetierzellen konstruiert (aa 1-3,144, basierend auf Q23-Nummerierung). Dieses Konstrukt verfügt über die Funktionen, die entwickelt wurden, um schnell verschiedene HTT-Mutationskonstrukte durch Kassettenklonen zu erzeugen, die Reinigung des HTT-Proteins auf hohe Qualität und Homogenität mit minimalen chromatographischen Schritten zu erleichtern und die Möglichkeit zu haben, unmarkiertes FL-HTT herzustellen. Die Liste der Funktionen umfasst 1. HindIII-Restriktionsverdauungsstellen, die die CAG-Wiederholung im HTT-Exon 1 umgeben, können verwendet werden, um FL-HTT-Mutanten mit einer PolyQ-Strecke unterschiedlicher Länge durch Restriktionsenzymverdauung und Ligation zu erzeugen; 2. Das C-terminale Ende von FL HTT ist mit einem FLAG-Epitop mit einer TEV-Protease-Erkennungsstelle für eine einstufige Affinitätsreinigung von FL HTT mit hoher Reinheit und optionaler Erzeugung eines markierungsfreien FL-HTT-Proteins unter Verwendung der TEV-Proteasespaltung markiert; 3. Codon-optimierte FL-HTT-Sequenz für die Verwendung von menschlichen Zellcodons zur High-Level-Expression in HEK293-Zellen. Der pcDNA 3.1 (+)-Vektor wird als Rückgrat des Konstrukts verwendet, um die hohe transkriptionelle Aktivierungsaktivität des CMV-Promotors in Säugetierzelllinien zu nutzen.
Unter Verwendung von pcDNA3.1-Q23-HTT-TEV-FLAG als Ausgangsvorlage wurden die HTT-Konstrukte Q48 und Q73 FL erzeugt, indem DNA-Fragmente mit der richtigen Q-Länge über zwei HindIII-Restriktionsenzymstellen synthetisiert und die gleiche Region in der Vorlage ausgetauscht wurden. Die ΔExon1-Mutante von FL HTT (aa 91-3,144) (Abbildung 1B) wurde unter Verwendung von Primern hergestellt, die auf gelöschte Rückstände gerichtet waren, die den Exon-1-Bereich in der Vorlage überspannen. HEK293-Zellen, die mit pcDNA3.1-Q23-HTT-TEV-FLAG unter PEI transfiziert wurden, wurden in 5-L-Schüttelkolben unter 5%CO2 gezüchtet. Eine typische großflächige Reinigung verwendet ein 2-10 L Zellpellet mit 6,0 × 10 9-3,0 × 1010 Zellen. Vor der Reinigung wurde das HTT-Expressionsniveau jeder Transfektion durch quantitatives Western Blotting unter Verwendung von gereinigtem rekombinantem FLAG-markiertem HTT als Standard und Anti-FLAG-Antikörper als erster Antikörper geschätzt. Zur Reinigung wurden Pellets mit einem geschätzten HTT-Expressionsgrad von ≥2 pg HTT/Zelle verwendet.
Die Reinigung von FL HTT besteht aus einem 2-stufigen Säulenprozess, zuerst mit Anti-FLAG-Affinitätsreinigung und dann mit SEC auf einer Gelfiltrationskolonne mit einem geeigneten Trennbereich für HTT (Abbildung 2A; siehe Materialtabelle für Beispiele). Nach beiden Schritten wurde HTT mit >95% Probenreinheit erhalten, wie von SDS-PAGE mit Coomassieblau und >65% Monomergehalt basierend auf analytischem SEC-MALS bestimmt. Da sich sowohl eine verlängerte Reinigungszeit als auch eine längere Temperatur negativ auf den endgültigen HTT-Monomergehalt auswirken, wurde FPLC in beiden Reinigungsschritten verwendet, um die Handhabung zu minimieren und eine gleichbleibende Probenqualität zu erzielen. Die Hauptverunreinigung während der Anti-FLAG-Reinigung war das Chaperon Hsp70, das durch Massenspektrometrie bestimmt wurde (Abbildung 2B, Bahn 2). Dies steht im Einklang mit dem Befund, dass Hsp70 zusammen mit FL HTT in humanen Zelllinien24 stabil exprimiert wird, was darauf hindeutet, dass Hsp70 ein häufiger Stabilisator für FL HTT in vivo sein könnte.
Hsp70-Verunreinigungen können durch ausgiebiges Waschen mit Magnesiumchlorid und ATP während des Anti-FLAG-Affinitätsreinigungsschritts eliminiert werden (Abbildung 2B, Spur 1). Nach Entfernung von Hsp70 neigt FL HTT zur Bildung von höhergeordneten Oligomeren24 und muss in einer Konzentration ≤ 1 mg/ml gehalten werden. Der Konzentrationsschritt vor SEC kann oft zu einer signifikanten Aggregation führen. Daher ist es am besten, Spitzenfraktionen aus der Anti-FLAG-Reinigung direkt auf die Größenausschlusssäule zu laden, ohne sich zu konzentrieren. Nach SEC wurde die Probe auf ≤1 mg/ml konzentriert, um eine maximale Wiederfindung der monomeren FL-HTT zu erreichen. Die Menge an HTT, die aus jedem Reinigungsschritt gewonnen wurde, wurde entweder durch Coomassie-Blau oder quantitatives Western Blotting unter Verwendung von gereinigtem FL-HTT als Quantifizierungsstandard geschätzt (Tabelle 2). Die typische Ausbeute an gereinigten FL-HTT-Proteinen, die nach dem beschriebenen Verfahren hergestellt werden, beträgt etwa 1 mg/L Zellkultur, kann aber aufgrund der Variabilität von Charge zu Charge deutlich darunter liegen (Tabelle 3), oder wenn das Anti-FLAG-Reinigungsharz mehrmals wiederverwendet wird.
Eine Überexpression von FL HTT kann zu einer Fragmentierung des Proteinsführen 22. FL Q23-HTT, hergestellt nach dem hier beschriebenen Verfahren, aufgelöst als ein einzelnes Band mit dem korrekten MW von 350 kDa durch SDS PAGE, gefärbt entweder durch Coomassie G250 oder durch Silberfärbung (Abbildung 2C). Durch Western Blotting reagierte FL Q23-HTT mit Antikörpern, die gegen Epitope an den N-terminalen, C-terminalen und mehreren Zwischendomänen angehoben wurden, ohne dass zusätzliche fragmentbezogene Banden beobachtet wurden, was darauf hindeutet, dass das Protein ohne signifikante nachweisbare Trunkierungen isoliert wurde (Abbildung 3A). FL HTT polyQ Längenvarianten Q23, Q48 und Q73 reagierten wie erwartet im Western Blot und zeigten ein progressiv stärkeres Signal für polyQ-gerichtetes mAb MW1, das mit zunehmender Q-Länge korreliert: Q23-HTT < Q48-HTT < Q73-HTT (Abbildung 3B). Für ΔExon1-HTT (aa 91-3,144) wurde kein Signal beobachtet, wenn es mit den Antikörpern MW1 und MAB549 untersucht wurde, die auf das N-terminale Exon 1 abzielen (Abbildung 3B).
SEC-MALS wurde eingesetzt, um den Aggregationszustand und die Molekülmasse des gereinigten HTT-Proteins zu analysieren. Die Proben wurden von analytischen SEC-Detektoren analysiert, die von UV-, MALS- und dRI-Detektoren überwacht wurden. Die aus SEC-MALS erhaltene absolute molare Masse hängt nicht von der Form der Moleküle33,34 ab; Daher bietet SEC-MALS eine unverzerrte Schätzung von MW für monomere und oligomere Fraktionen, wenn sie gut getrennt sind. Unter den getesteten HPLC-Säulen zeigte die SEC-Säule (siehe Materialtabelle) eine ausreichende Auflösung zwischen dem HTT-Monomer und dem Dimer, so dass molare Massen unterschieden werden konnten (Abbildung 4). Die Proteinkonzentration wurde durch dRI-Detektion bestimmt. Brechungsindexschritte (dn/dc) von FL HTT betragen 0,1853 ml/g, berechnet von der SEDFIT-Software35. Ähnliche analytische SEC-Elutionsmuster wurden für ΔExon1 HTT (91-3.144), FL Q23, Q48 und Q73 HTT (1-3.144) beobachtet, die jeweils aus einem Hauptmonomerpeak mit geringen dimeren und oligomeren Peaks bestehen (Tabelle 4). Die berechnete MW für die monomere Form ist größer als die theoretische MW. Dies wird wahrscheinlich durch überlappende Spezies aus höhergeordneten oligomeren Peaks und Fehler verursacht, die sich aus schwachen dRI-Signalen ergeben, da HTT-Proteine in niedriger Konzentration gehalten werden, um die Bildung von höhergeordneten Oligomeren zu vermeiden. Durch die Integration der UV-Peaks mehrerer Chargen gereinigter FL-HTT-Varianten wurde keine eindeutige Korrelation zwischen PolyQ-Länge und Aggregatprofil beobachtet (Tabelle 4).
Zusätzlich zur analytischen SEC wurde die traditionelle native PAGE durchgeführt, um festzustellen, ob sie als ergänzende Methode zur Charakterisierung des oligomeren Zustands FL HTT verwendet werden kann. Oligomere höherer Ordnung wurden durch 3-8% Tris-acetat-Gele unter Verwendung von nativem Puffer ohne Detergens gelöst. Gereinigtes FL HTT von SEC zeigte mehrere Banden, die den Oligomerisierungszuständen entsprechen (Abbildung 5A). Die niedrigste Bande befand sich zwischen dem nativen Marker 480 kDa und 720 kDa, ähnlich wie frühere Ergebnisse für FL HTT, das aus Insektenzellen gereinigt wurde22. Das HTT-Monomer war jedoch nicht die am häufigsten vorkommende Bande bei Verwendung von herkömmlichem nativem PAGE, und die Ergebnisse korrelieren nicht mit dem durch analytische SEC-MALS bestimmten Aggregatprofil. Mehrere hydrophobe Patches, die in FL HTT36,37,38 vorhanden sind, insbesondere die hydrophobe Grenzfläche zwischen HAP40 und FL HTT25, tragen wahrscheinlich zur Bildung von höhergeordneten Oligomeren während der Migration innerhalb des Gels bei. Dies liegt daran, dass bekannt ist, dass hydrophobe Regionen in Abwesenheit von Detergens- oder stabilisierenden Protein-Protein-Wechselwirkungen miteinander interagieren. In Übereinstimmung mit den hydrophoben Eigenschaften von HTT bildet FL HTT während des SEC-Reinigungsschritts zunehmende Mengen an höhergeordneten oligomeren Fraktionen in Abwesenheit von CHAPS.
Blue Native PAGE, das häufig zur Untersuchung von Membranproteinen und großen Proteinkomplexen mit hydrophoben Patches39 verwendet wird, wurde mit herkömmlichem nativem PAGE verglichen. Gereinigtes HTT zeigte drei Hauptbänder auf Blue Native PAGE mit geschätzten MW von 643, 927 und 1070 kDa (Abbildung 5B), die wahrscheinlich die monomeren, dimeren und trimeren Arten von HTT darstellen. Die Monomerbande blieb die am häufigsten vorkommende Bande in der Blue Native PAGE und entsprach gut dem analytischen SEC-Profil derselben Proben. Die Überschätzung von MW des HTT-Monomers durch Blue Native PAGE kann auf die einzigartige hohle sphärische Struktur oder hydrophobe Bereiche von HTT zurückzuführen sein, die eine langsamere Migration im Vergleich zu den entsprechenden Molekulargewichtsmarkern 11,23,25 verursachen. Insgesamt haben FL Q23-HTT, FL Q48-HTT, FL Q73-HTT und ΔExon1-HTT ähnliche Blue Native PAGE-Profile mit nur geringen Unterschieden in der Proteinbandmigration aufgrund ihrer Molekulargewichtsunterschiede.
Als zusätzliche Überprüfung der Qualität der gereinigten Proteine kann der C-terminale FLAG-Tag durch Behandlung mit TEV-Protease aus FL HTT entfernt werden. Nach proteolytischer Spaltung wurden die Proben mittels Western Blot unter Verwendung von vier Antikörpern analysiert, um die Entfernung des FLAG-Tags zu bestätigen und den HTT-Abbau nachzuweisen. Die Immunreaktivität gegen Anti-FLAG M2 und drei Huntingtin-spezifische Antikörper mit Epitopen zum N-Terminus, zu Zwischendomänen und C-Terminus von HTT zeigte eine erfolgreiche Entfernung von FLAG-Tags und keine HTT-spezifischen Abbauprodukte (ergänzende Abbildung S1).
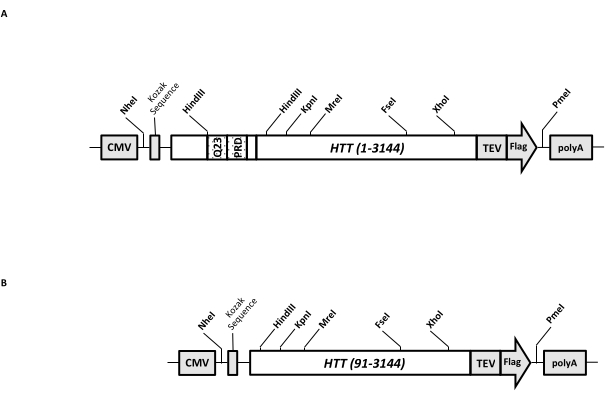
Abbildung 1: Konstrukt für HTT-Ausdruck in voller Länge. (A) Q23 HTT in voller Länge wurde Codon-optimiert und in pcDNA3.1 (+)-Plasmid kloniert. Das 3'-Ende von HTT wurde mit Flag-Epitop und TEV-Protease-Spaltungsstelle markiert, um tag-freies HTT-Protein zu produzieren. Die Polyglutamindehnung und die Prolin-reiche Domäne wurden mit flankierten HindIII-Restriktionsendonukleasestellen entwickelt, um zusätzliche CAG-Wiederholungen mittels Kassettenklonen, d.h. Q48 und Q73, einzufügen, um HTT-Varianten mit unterschiedlichen PolyQ-Längen herzustellen. (B) Das ΔExon1-Konstrukt wurde mittels PCR-Mutagenese unter Verwendung von pcDNA3.1-Q23-HTT als Vorlage erstellt. Die Reste 91-3,144 von HTT verblieben im ΔExon1-Konstrukt zur Expression. Abkürzungen: HTT = huntingtin; CMV = Cytomegalovirus; Q23 = Polyglutamindehnung; PRD = Prolin-reiche Domain; TEV = Tabakätzvirus-Spaltungsstelle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
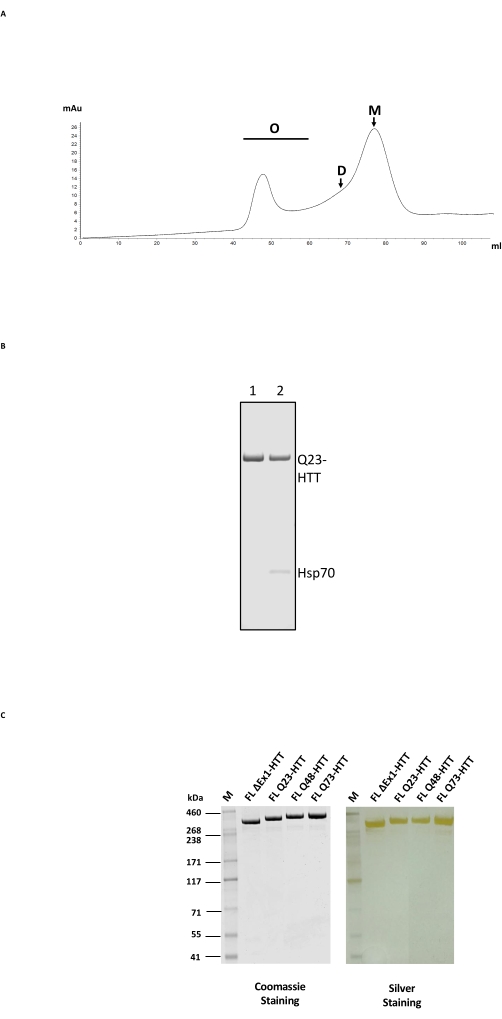
Abbildung 2: Großflächige Reinigung von HTT. (A) SEC-Profil des Anti-Flag-gereinigten Q23-HTT in voller Länge auf einer FPLC-Säule. Hochgeordnete Oligomere, Dimere und Monomerpeaks von Q23-HTT sind markiert. Monomerhaltige Fraktionen wurden als endgültige HTT-Probe gesammelt. (B) SDS-PAGE von gereinigtem Q23-HTT mit ATP/Magnesium-Waschschritt (Bahn 1) oder ohne ATP/Magnesium-Waschen ergibt eine Hsp70-Co-Elution (Spur 2). (C) Endgereinigte HTT-Varianten in voller Länge auf SDS-PAGE, gefärbt mit Coomassieblau G-250 oder Silberfleck. Abkürzungen: FL = volle Länge; HTT = Huntingtin; SEC = Größenausschlusschromatographie; FPLC = schnelle Proteinflüssigkeitschromatographie; O = Oligomer; D = Dimer; M = Monomer; SDS-PAGE = Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese; Hsp70 = Hitzeschockprotein 70. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
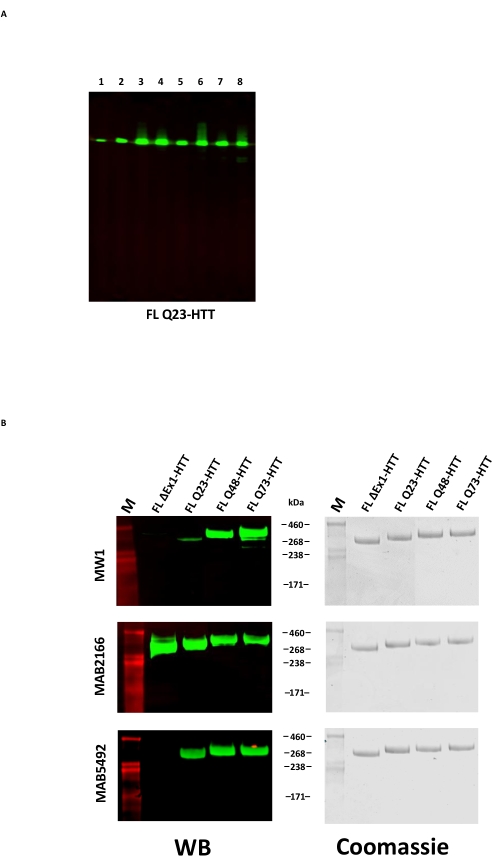
Abbildung 3: Western-Blot-Analyse von gereinigten HTT-Varianten . (A) Gereinigtes FL Q23-HTT wurde auf SDS-PAGE ausgeführt und auf PVDF-Membran übertragen. Die primären Antikörper und interagierenden Epitope sind Lane 1, α-FLAG M2, FLAG-Tag; Bahn 2, MAB5492, HTT aa. 1-82; Bahn 3, MAB5490, HTT aa 115-129; Bahn 4, MAB2166, HTT AA 181-810; Bahn 5, MAB3E10, HTT aa 1.171-1.177; Bahn 6, MAB4E10, HTT aa 1.844-2.131; Bahn 7, MAB2168, HTT aa 2,146-2,541; Bahn 8, MAB8A4, HTT aa 2,703-2,911. (B) 1 μg gereinigte FL-HTT-Varianten wurden auf SDS-PAGE ausgeführt und auf PVDF übertragen (links), und ein doppeltes SDS-Gel wurde ausgeführt und mit Coomassie Blue (rechts) angefärbt. Die primären Antikörper und interagierenden Epitope sind Reihe 1, MW1, erweiterte PolyQ-Wiederholungen; Reihe 2, MAB2166, HTT AA 181-810; Reihe 3, MAB5492, HTT aa 1-82. Abkürzungen: FLL Q23-HTT = Huntingtin-Protein in voller Länge mit 23 Glutaminresten; SDS-PAGE = Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese; WB = Western Blot; M = Marker; PVDF = Polyvinylidenfluorid. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
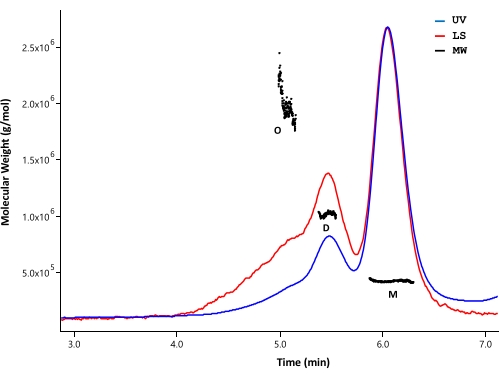
Abbildung 4: SEC-MALS-Analyse von HTT in voller Länge. Gereinigtes Q23-HTT in voller Länge wurde auf einer UPLC-Säule eluiert. Spitzenpositionen von vorhergesagtem Monomer, Dimer und Oligomer werden angezeigt. Die Molekulargewichte wurden für Monomer-, Dimer- und Trimerpeaks berechnet und in Tabelle 5 aufgeführt. Ähnliche Elutionsprofile werden für Q48, Q73 und ΔExon1 HTT beobachtet, mit variablen Monomer-, Dimer- und Oligomergehalten in jeder Reinigung. Abkürzungen: SEC-MALS = Größenausschlusschromatographie mit Mehrwinkellichtstreuung; UV = Ultraviolett; LS = Lichtstreuung; MW = Molekulargewicht; Q23-HTT = Huntingtin-Protein mit 23 Glutaminresten; M = Monomer; D = Dimer; O = Oligomer. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
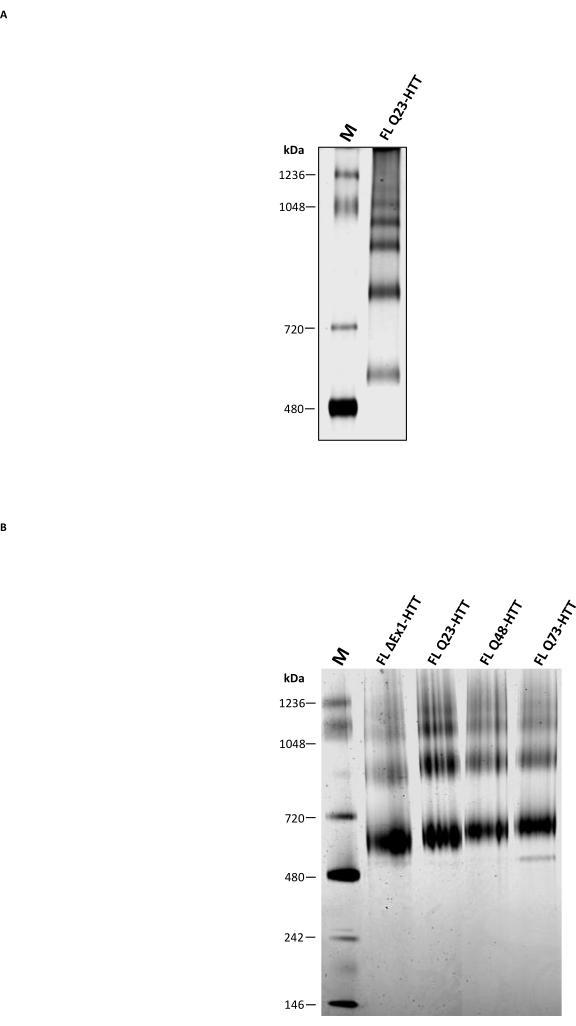
Abbildung 5: Charakterisierung von gereinigtem HTT unter Verwendung von klarem nativem PAGE oder blauem nativem PAGE-Gel. Nativer Marker und scheinbares monomeres Q23-HTT aus SEC wurden auf 3-8% Tris-acetat-Gelen in einem nicht denaturierenden PAGE-System (A) und einem Blue Native PAGE-System (B) aufgelöst. Abkürzungen: FL = Full-Length; Q23-HTT = Huntingtin-Protein mit 23 Glutaminresten; PAGE = Polyacrylamid-Gelelektrophorese; M = Marker. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Schritt | Name | Zusammensetzung | ||
| 6.1.1 | Puffer A | 50 mM Tris, 500 mM NaCl, 5% v/v Glycerin, 5 mM EDTA, 0,01% v/v Tween-20, pH 8,0. | ||
| 6.1.2 | Lyse-Puffer | 50 mM Tris, 500 mM NaCl, 5% v/v Glycerin, 5 mM EDTA und 1x Proteaseinhibitor-Cocktail | ||
| 6.1.4.2 | Puffer A | 50 mM Tris, 500 mM NaCl, 5% v/v Glycerin, 5 mM EDTA, 0,01% v/v Tween-20, pH 8,0. | ||
| 6.1.4.3 | Puffer B | 50 mM Tris; 500 mM KCl; 5 mM MgCl2; 5% v/v Glycerin; 0,01% v/v Tween-20, pH 8,0 | ||
| 6.1.4.4 | Puffer C | 20 mM Tris; 200 mM KCl; 5 mM MgCl2; 5 mM ATP; 0,01% v/v Tween-20; 5% v/v Glycerin, pH 8,0 | ||
| 6.1.4.5 | Puffer D | 50 mM Tris; 500 mM NaCl; 5% v/v Glycerin; 5 mM EDTA; 0,5 % w/v CHAPS, pH 8,0 | ||
| 6.1.4.6 | Elution Puffer | 50 mM Tris; 500 mM NaCl; 5% v/v Glycerin; 0,5 % w/v CHAPS; 0,2 mg/ml DYKDDDDK-Peptid, pH 8,0 | ||
| 6.1.6 | Regenerationspuffer | 0,1 M Glycin HCl, pH 3,5; 0,01% v/v Tween-20 | ||
| 6.2.1 | SEC-Puffer | 50 mM Tris, 500 mM NaCl, 5 % v/v Glycerin, 0,5 % w/v CHAPS, 1 mM TCEP | ||
| 7.3 | SEC-MALS Puffer | 50 mM HEPES, pH 7,2, 500 mM NaCl, 5% v/v Glycerin, 0,5% w/v CHAPS | ||
| 8.7 | Entfärbungslösung | 40% v/v Methanol und 7% v/v Essigsäure | ||
| 9.4.2 | Coomassie Färbelösung | 0,01% w / v Coomassie G250, 50% v / v / Methanol, 10% v / v Essigsäure | ||
| 9.5.1 | Reparaturlösung | 50% v/v Methanol, 10% v/v Essigsäure, 50 μL Formaldehyd/100 mL Lösung | ||
Tabelle 1: Zusammensetzung der Puffer und Lösungen
| Schritte | HTT-Konzentration (mg/ml) | Gesamtvolumen (ml) | HTT-Gehalt (mg) | HTT-Ausbeute pro Zelle (pg/Zelle) | % Ertrag |
| Überstand | 0.1792 | 220 | 39.4 | 4.4 | 100 |
| Anti-Flagge | 1.524 | 8.6 | 13.1 | 1.47 | 33.4 |
| SEC | 0.91 | 3.9 | 3.54 | 0.4 | 9.1 |
Tabelle 2: HTT-Ausbeute aus einem 2 L HEK293 Pellet, transfiziert mit pcDNA3.1-Q23-HTT-TEV-Flag. Abkürzungen: FL Q23-HTT = Huntingtin-Protein in voller Länge mit 23 Glutaminresten; TEV = Tobacco Etch Virus Cleavage Site; SEC = Größenausschlusschromatographie.
| HTT-Beispiel | HTT-Ausbeute (mg/L) | Durchschnittliche Reinheit (%) | ||
| BCA | A280 | |||
| 1 | FL DEx1-HTT (N=3) | 0.67-1.30 | 0.69-1.18 | 99.3 |
| 2 | FL Q23-HTT (N=3) | 0.25-0.92 | 0.28-0.98 | 96.9 |
| 3 | FL Q48-HTT (N=3) | 0.28-1.15 | 0.38-1.16 | 97.4 |
| 4 | FL Q73-HTT (N=3) | 0.58-1.05 | 0.57-0.97 | 98.8 |
Tabelle 3: Zusammenfassung der Proteinausbeute von vier FL-HTT-Variantenreinigungen und deren Endreinheit. Abkürzung: FLL HTT = Full-length Huntingtin Protein.
| HTT-Beispiel | Ein | D | M |
| FL Q23-HTT | 4.2-6.9% | 18.7-29.3% | 66.5-76.0% |
| FL Q48-HTT | 4.0-9.4% | 10.6-17.8% | 73.6-85.4% |
| FL Q73-HTT | 2.0-14.0% | 16.9-24.6% | 65.1-81.1% |
Tabelle 4: Zusammenfassung des repräsentativen Aggregat-, Dimer- und Monomergehalts von FL-HTT-Varianten aus der Reinigung. Abkürzungen: FL HTT = Full-length Huntingtin Protein; A = Aggregat; D = Dimer; M = Monomer; SEC = Größenausschlusschromatographie.
Ergänzende Abbildung S1: Western-Blot-Analyse nach TEV-Protease-Verdauung. Gereinigtes FL Q23-HTT und FL Q48-HTT wurden auf SDS-PAGE ausgeführt, auf PVDF-Membranen übertragen und nach dem TEV-Aufschluss durch Western Blotting analysiert. Primäre Antikörper waren Anti-Flag M2 (Flag-Tag), MAB5492 (HTT aa 1-82), MAB3E10 (HTT aa 997-1,276) und MAB2168 (HTT aa 2,146-2,541). Bahn 1, Proteinstandard; Bahn 2, Q23-HTT-TEV-Flag; Bahn 3, Q48-HTT-TEV-Flagge; Bahn 4, Q23-HTT-TEV-Flag behandelt mit TEV-Protease bei 1:5, über Nacht bei 4 °C; Bahn 5, Q48-HTT-TEV-Flag behandelt mit TEV-Protease bei 1:5, über Nacht bei 4 °C. Abkürzungen: FL HTT = Full-length Huntingtin Protein; SDS-PAGE = Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese; TEV = Tabakätzvirus; PVDF = Polyvinylidenfluorid. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S2: SEC-MALS-Analyse von FL-HTT-Varianten, die Gefrier-Tau-Zyklen unterzogen wurden. Gereinigtes Q23-HTT (A) und Q48-HTT (B) wurden bei -80 °C eingefroren und bis zu 6-mal bei Raumtemperatur aufgetaut. Q23-HTT und Q48-HTT nach dem ersten Gefrier-Tau-Zyklus und dem sechsten Gefrier-Tau-Zyklus wurden dann von SEC-MALS analysiert. Eine leichte Abnahme der Monomerfraktion und eine Zunahme der Dimer- und Oligomerfraktionen hoher Ordnung wurden durch Lichtstreuung nach wiederholten Gefrier-Tau-Zyklen beobachtet. Spitzenpositionen von vorhergesagtem Monomer, Dimer und Oligomer hoher Ordnung werden angezeigt. Abkürzungen: FL HTT = Full-length Huntingtin Protein; O = Oligomer; D = Dimer; M = Monomer; SEC-MALS = Größenausschlusschromatographie mit Mehrwinkel-Lichtstreuung. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung S3: SDS PAGE der FL HTT-Varianten, die Gefrier-Tau-Zyklen unterzogen wurden. Gereinigte Q23-HTT (Bahnen 2-7) und Q48-HTT (Bahnen 9-14) wurden bei -80 °C eingefroren und bis zu 6-mal bei Raumtemperatur aufgetaut. Aliquots von Q23-HTT und Q48-HTT wurden nach jedem Gefrier-Tau-Zyklus gespeichert und dann von SDS PAGE analysiert. Es wurde kein Anstieg der Zuschlagstoffe oder Abbauprodukte beobachtet; Die Proben wurden durch Banddensitometrie als stabil und >95% rein eingestuft. Abkürzungen: FL HTT = Full-length Huntingtin Protein; SDS-PAGE = Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 1: FPLC 15 mL Anti-FLAG HTT Skript. Abkürzungen = FPLC = Fast Protein Liquid Chromatographie; HTT = Huntingtin-Protein. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: FPLC SEC_MALS HTT-Skript. Abkürzungen: SEC-MALS = Größenausschlusschromatographie mit Mehrwinkellichtstreuung; FPLC = schnelle Proteinflüssigkeitschromatographie; HTT = Huntingtin-Protein. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Wir beschreiben hier eine transiente Transfektions-, Expressions- und Reinigungsmethode zur Erzeugung mehrerer FL-HTT-Proteinkonstrukte mit geeigneter Reinheit und Homogenität für die Verwendung als Standards für die Immunoassay- und MS-Assay-Entwicklung, Kontrollen für die Western-Blot-Analyse und für Struktur-Funktions-Studien. Diese transiente Expressionsmethode ist skalierbar und vielseitig und ermöglicht es dem Anwender, geringe Milligrammmengen von FL-HTT-Varianten effizienter zu erzeugen als mit stabilen Zelllinien oder virusbasierten Methoden, die zuvorbeschrieben wurden 21,22,23,24. Routinemäßig können 2-5 mg hochgereinigtes FL HTT aus einer Proteinproduktion im 2-L-Maßstab in weniger als einer Woche mit der transienten Expressionsmethode erzeugt werden, sobald das Plasmid konstruiert ist, mit einer typischen Ausbeute von 1-2,5 mg FL HTT pro Liter Zellkultur.
Die hier beschriebene transiente Expressionsmethode überwindet viele Hindernisse in der stabilen Zelllinienexpression, wie z.B. die lange Zeit, die benötigt wird, um Zelllinien zu etablieren und Schwierigkeiten bei der Lagerung und Aufrechterhaltung stabiler Zelllinien. PEI ist auch relativ kostengünstig im Vergleich zu anderen Transfektionsreagenzien auf dem Markt, was eine groß angelegte Transfektion wirtschaftlich rentabel macht. Es gibt auch Einschränkungen im Protokoll: Die Transfektionseffizienz hängt weitgehend von der Qualität der Plasmide, dem optimalen Zellwachstum und der Art und Weise ab, wie gut PEI gespeichert und hergestellt wird. Die Betreiber müssen bei diesen kritischen Schritten besondere Vorsicht walten lassen und Qualitätskontrollen durchführen, um einen drastischen Rückgang der Proteinausbeute zu vermeiden. Das im Protokoll verwendete Anti-FLAG-Harz ist ebenfalls relativ teuer und zeigt eine reduzierte Erfassung von FL HTT nach mehreren Reinigungs- und Regenerationsvorgängen. Einige Forscher finden es möglicherweise praktischer, zu einem anderen Tag zu wechseln, um eine robustere Regeneration des Affinitätsharzes zu ermöglichen.
Verschiedene Zelllinien und Expressionsbedingungen wurden getestet, um die FL-HTT-Expressionsniveaus zu optimieren. HEK293-Zellen wurden aufgrund der hohen Proteinexpression und der einfachen Handhabung in einem Suspensionskulturformat für die Expression von FL HTT ausgewählt, wodurch die Methode für die großflächige Expression in Schüttlern oder Bioreaktoren geeignet ist. Ein höherer FL-HTT-Proteinexpressionsgrad kann bei niedrigeren Kulturtemperaturen wie 32 °C erreicht werden, anstatt die übliche Temperatur von 37 °C zu verwenden. Es ist möglich, dass die niedrigere Temperatur die Proteinsynthese verlangsamt und die korrekte Faltung von FL HTT40 fördert. Dieses Phänomen ist jedoch nicht spezifisch für FL HTT oder die getesteten Zelllinien. Die reduzierte Posttransfektionstemperatur wurde häufig in der pharmazeutischen Proteinexpression in CHO-Zellen verwendet. Obwohl der Mechanismus nicht vollständig verstanden ist, wird angenommen, dass niedrige Temperaturen den Zellzyklus in der G1-Phase stoppen und zelluläre Energie zur Proteinproduktion umleiten41.
HTT in voller Länge, das aus Säugetierzellen gereinigt wird, eluiert mit dem Chaperon Hsp7024, und Mg-ATP-Waschschritte können das Hsp70-Protein entfernen. Interessanterweise wird co-eluiertes Hsp70 nicht in FL HTT beobachtet, das aus einem Insektenzellexpressionssystem21,22,23 gereinigt wurde. Dies kann einen Unterschied in den PTMs von FL HTT oder Hitzeschockproteinantworten auf die Überexpression von FL HTT in Säugetier- und Insektenzellen widerspiegeln. Sobald das rekombinante Protein von Hsp70 befreit wurde, werden nichtionische Detergenzien wie CHAPS oder DDM benötigt, um die monomere Form von FL HTT zu stabilisieren.
Die Oligomerisierungszustände von FL-HTT-Varianten wurden mit Blue Native PAGE und SEC-MALS analysiert. Ein kleiner Anteil an dimerem und oligomerem HTT höherer Ordnung war vorhanden, wenn es entweder von Blue Native PAGE oder SEC-MALS analysiert wurde. Bemerkenswert ist, dass höhergeordnete Oligomere, die von FL HTT gebildet werden, nicht mit der PolyQ-Länge zu korrelieren scheinen, und selbst die Exon1-Deletionsmutante zeigt ein ähnliches Oligomer-Dimer-Monomer-Verhältnis. Die tatsächlichen Schwankungen des Oligomergehalts zwischen diesen Konstrukten sind wahrscheinlich auf geringfügige Unterschiede in der Herstellung und Handhabung jeder Charge zurückzuführen. Im Gegensatz zu Aggregaten und Fibrillen, die von HTT Exon140,41 gebildet wurden, blieben die höhergeordneten Oligomere von FL HTT löslich und konnten von SEC und Native PAGE analysiert werden.
Gereinigtes monomeres FL HTT ist nur relativ stabil. Längere Lagerung bei 4 °C, kurze Inkubationen bei Raumtemperatur oder Konzentrationen > 1 mg/ml wandeln monomere FL HTT in dimere und oligomere Formen höherer Ordnung um, obwohl unter diesen Bedingungen kein sichtbarer Niederschlag beobachtet wird. Gereinigtes monomeres FL HTT, das bei ≤1 mg/ml gehalten wurde, blieb bei -80 °C im Speicherpuffer (50 mM Tris, pH 8,0, 500 mM NaCl, 5% v/v Glycerin, 0,5% w/v CHAPS und 5 mM DTT) relativ stabil24. Bis zu 6 Gefrier-Tau-Zyklen von FL HTT, die auf diese Weise hergestellt und gelagert wurden, verursachten keine sichtbare Ausfällung des Proteins, obwohl eine leichte Verschiebung in einen höheren oligomeren Zustand von SEC-MALS beobachtet wurde (ergänzende Abbildung S2). Die Proben wurden auch von SDS PAGE nach wiederholten Gefrier-Tau-Zyklen analysiert. Es wurden keine sichtbaren Niederschläge beobachtet; von SDS-PAGE wurden keine Aggregate oder zusätzliche Abbauprodukte gesehen (ergänzende Abbildung S3). Die Langzeitstabilität von gereinigtem FL HTT wird noch untersucht. In Ermangelung aussagekräftiger Langzeitdaten empfehlen wir, gereinigtes FL HTT bei -80 °C nicht länger als 6 Monate zu lagern.
Hochwertige, rekombinante FL-HTT-Proteinvarianten und die Methoden, um sie herzustellen, sind in der Huntington-Forschungsgemeinschaft sehr gefragt. Diese Proteine werden als Immunoassay- und MS-Analysestandards, in Strukturstudien und für die Entwicklung neuartiger FL-HTT-spezifischer Assays verwendet. Die hier beschriebenen großflächigen transienten Expressionsmethoden haben durchweg Milligrammmengen von FL-HTT-Varianten mit >95% Reinheit erzeugt und liefern wesentliche Werkzeuge für HTT-Studien. Die Produktion von Dutzenden Milligramm hochgereinigter FL HTT polyQ-Varianten und anderer Mutanten zur Unterstützung der Huntington-Forschung ist zur Routine geworden.
Offenlegungen
Die Autoren erklären, dass sie keine Interessenkonflikte mit dem Inhalt dieses Artikels haben.
Danksagungen
Wir danken dem Department of Pharmaceutical Sciences, The State University of New York in Buffalo, für die Durchführung der MS-Analyse von HTT. Diese Arbeit war eine Zusammenarbeit mit der CHDI Foundation. Wir danken besonders Elizabeth M. Doherty; Ignacio Munoz-Sanjuan; Douglas Macdonald, CHDI Foundation; und Rory Curtis, Curia, für ihren unschätzbaren Beitrag während der Vorbereitung dieses Manuskripts. Wir danken auch Michele Luche, Mithra Mahmoudi und Stephanie Fox für ihre Unterstützung dieser Forschungsbemühungen.
Materialien
| Name | Company | Catalog Number | Comments |
| 100 kDa concentrator-Amicon | Millipore | UFC910096 | Protocol Section Number-6.2.4 |
| 20x blue native PAGE running buffer | Invitrogen | BN2001 | Protocol Section Number-8.1 |
| 20x TBS | Thermo Fisher | PI28358 | Protocol Section Number-5.1 |
| 4x blue native PAGE sample buffer | Invitrogen | BN2003 | Protocol Section Number-8.3 |
| 4x LDS loading buffer | Invitrogen | NP0007 | Protocol Section Number-5.3 |
| 5 L Erlenmeyer flasks | Corning | 431685 | Protocol Section Number-4.2 |
| Agarose gel extraction kit | Qiagen | 28704 | Protocol Section Number-2.2 |
| Anti-clumping agent | Thermo Fisher | 0010057AE | Protocol Section Number-4.8 |
| anti-FLAG M2 affinity gel | Sigma | A2220 | Protocol Section Number-6.1.1 |
| anti-FLAG M2 | Sigma | F3165 | Protocol Section Number-5.7 |
| Anti foam-Excell anti foam | Sigma | 59920C-1B | Protocol Section Number-4.8 |
| ATP | Sigma | A6419 | Protocol Section Number-6.1.4.4 |
| BEH 450 SEC | Waters | 186006851 | 2.5 µm x 4.6 mm x 150 mm Protocol Section Number-7.3 |
| blue native PAGE 5% G-250 sample additive | Invitrogen | BN2004 | Protocol Section Number-8.3 |
| carbenicillin | Thermo Fisher | 10177012 | Protocol Section Number-2.5 |
| centrifuge - Sorvall Lynx 6000 | Thermo Fisher | 75006590 | Protocol Section Number-6.1.3 |
| Cell Counter - ViCELL | BECKMAN COULTER | Protocol Section Number-4.3 | |
| CHAPS | Anatrace | C316S | Protocol Section Number-6.1.4.6 |
| Competent E. coli cells-TOP10 | Invitrogen | C404010 | Protocol Section Number-2.4 |
| digitonin | Sigma | D141 | Protocol Section Number-5.1 |
| differential refractive index detector | Wyatt | Protocol Section Number-7.1 | |
| DYKDDDDK peptide | Genscript | Peptide synthesis service Protocol Section Number-6.1.4.6 | |
| EDTA | Sigma | EDS | Protocol Section Number-5.1 |
| EndoFree Plasmid Giga Kit | Qiagen | 12391 | Protocol Section Number-3.3 |
| Endotoxin free water | Cytiva | SH30529.03 | Protocol Section Number-4.1 |
| endotoxin quantification kit-CRL Endosafe Nexgen-PTS detection system | Charles River | PTS150K | Protocol Section Number-3.4 |
| fixed angle rotor A23-6x100 rotor | Thermo Fisher | 75003006 | Protocol Section Number-6.1.3 |
| FPLC software- Unicorn 6.2 | Cytiva | Protocol Section Number-6.1.4 | |
| Gene synthesis | Genscript | Gene synthesis service Protocol Section Number-1.2 | |
| Glycerol | Fisher Scientific Glycerol (Certified ACS) | G33-4 | Protocol Section Number-5.6 |
| Growth Medium-Expi293 expression medium | Thermo Fisher | A1435102 | Protocol Section Number-4.2 |
| HEK293 cells | Thermo Fisher | R79007 | Protocol Section Number-4 |
| high shear homogenizer-Microfluidizer | MicroFluidics | LM10 | Protocol Section Number-6.1.3 |
| HPLC - 1260 infinity II Bio-Insert HPLC | Agilent | Protocol Section Number-7.1 | |
| Image Studio | LiCor | Image analysis software Protocol Section Number-5.1 | |
| MAB2166 | Sigma | MAB2166 | Protocol Section Number-5.7 |
| MAB2168 | EMD | MAB2168 | Protocol Section Number-5.7 |
| MAB3E10 | Santa Cruz | SC-47757 | Protocol Section Number-5.7 |
| MAB4E10 | Santa Cruz | SC-7757 | Protocol Section Number-5.7 |
| MAB5490 | Sigma | MAB5490 | Protocol Section Number-5.7 |
| MAB5492 | Sigma | MAB5492 | Protocol Section Number-5.7 |
| MAB8A4 | Santa Cruz | SC-47759 | Protocol Section Number-5.7 |
| multi-angle light scattering detector | Wyatt | Protocol Section Number-7.1 | |
| NativeMark Unstained Protein Standard | Invitrogen | LC0725 | Protocol Section Number-8.4 |
| NaCl | Sigma | S9888 | Protocol Section Number-5.6 |
| NheI | New England Biolab | R0131S | Hi-Fi version available Protocol Section Number-2.2 |
| NuPAGE 3–8% Tris acetate gels | Invitrogen | EA0375PK2 | Protocol Section Number-5.4 |
| NuPAGE Tris-Acetate SDS Running buffer | Invitrogen | LA0041 | Protocol Section Number-5.4 |
| PEI 25K | Polysciences | 23966-1 | Protocol Section Number-4.1 |
| Penicillin-Streptomycin | Thermo Fisher | 15070063 | Protocol Section Number-4.2 |
| Phosphate Buffered Saline (PBS) | Cytiva | SH30256.02 | Protocol Section Number-4.5 |
| plasmid miniprep kit | Qiagen | 27104 | Protocol Section Number-2.6 |
| PmeI | New England Biolab | R0560S | Protocol Section Number-2.2 |
| precast Bis-tris gel- 3-12% NativePAGE Novex Bis-Tris Gel | Invitrogen | BN1003BOX | Protocol Section Number-8.4 |
| protease inhibitor cocktail | GoldBio | GB-331-1 | Protocol Section Number-5.1 |
| SEC-MALS analysis software - Astra 7 | Wyatt Technology | Protocol Section Number-7.6 | |
| secondary antibody -IRdye 800 CW goat anti-mouse IgG | LiCor | 926-32210 | Protocol Section Number-5.9 |
| Superose 6 pg XK 16/70 | Cytiva | 90100042 | Protocol Section Number-6.2 |
| Tris base | Fisher | BP152 | Protocol Section Number-5.6 |
| Tween-20 | Thermo Fisher | AAJ20605AP | Protocol Section Number-6.1.1 |
| UV spectrometer - Nanodrop 8000 | Thermo Fisher | ND-8000-GL | Protocol Section Number-2.2 |
| XK26/100 | Cytiva | 28988951 | Protocol Section Number-6.1.1 |
Referenzen
- Walker, F. O. Huntington's disease. Lancet. 369 (9557), 218-228 (2007).
- McColgan, P., Tabrizi, S. J. Huntington's disease: a clinical review. European Journal of Neurology. 25 (1), 24-34 (2018).
- Duyao, M., et al. Trinucleotide repeat length instability and age of onset in Huntington's disease. Nature Genetics. 4 (4), 387-392 (1993).
- MacDonald, M. E., et al. A novel gene containing a trinucleotide repeat that is expanded and unstable on Huntington's disease chromosomes. Cell. 72 (6), 971-983 (1993).
- Nasir, J., et al. Targeted disruption of the Huntington's disease gene results in embryonic lethality and behavioral and morphological changes in heterozygotes. Cell. 81 (5), 811-823 (1995).
- Dragatsis, I., Levine, M. S., Zeitlin, S. Inactivation of Hdh in the brain and testis results in progressive neurodegeneration and sterility in mice. Nature Genetics. 26 (3), 300-306 (2000).
- Anne, S. L., Saudou, F., Humbert, S. Phosphorylation of huntingtin by cyclin-dependent kinase 5 is induced by DNA damage and regulates wild-type and mutant huntingtin toxicity in neurons. Journal of Neuroscience. 27 (27), 7318-7328 (2007).
- Dietrich, P., Johnson, I. M., Alli, S., Dragatsis, I. Elimination of huntingtin in the adult mouse leads to progressive behavioral deficits, bilateral thalamic calcification, and altered brain iron homeostasis. PLoS Genetics. 13 (7), 1006846(2017).
- Dragatsis, I., et al. Effect of early embryonic deletion of huntingtin from pyramidal neurons on the development and long-term survival of neurons in cerebral cortex and striatum. Neurobiology of Disease. 111, 102-117 (2018).
- Benn, C. L., et al. Huntingtin modulates transcription, occupies gene promoters in vivo, and binds directly to DNA in a polyglutamine-dependent manner. Journal of Neuroscience. 28 (42), 10720-10733 (2008).
- Saudou, F., Humbert, S. The biology of huntingtin. Neuron. 89 (5), 910-926 (2016).
- Davies, S. W., et al. Formation of neuronal intranuclear inclusions underlies the neurological dysfunction in mice transgenic for the HD mutation. Cell. 90 (3), 537-548 (1997).
- DiFiglia, M., et al. Aggregation of huntingtin in neuronal intranuclear inclusions and dystrophic neurites in brain. Science. 277 (5334), 1990-1993 (1997).
- Gutekunst, C. A., et al. Nuclear and neuropil aggregates in Huntington's disease: Relationship to neuropathology. Journal of Neuroscience. 19 (7), 2522-2534 (1999).
- Hodgson, J. G., et al. A YAC mouse model for Huntington's disease with full-length mutant huntingtin, cytoplasmic toxicity, and selective striatal neurodegeneration. Neuron. 23 (1), 181-192 (1999).
- Hoffner, G., Djian, P. Polyglutamine aggregation in Huntington disease: does structure determine toxicity. Molecular Neurobiology. 52 (3), 1297-1314 (2015).
- Waldvogel, H. J., Kim, E. H., Tippett, L. J., Vonsattel, J. P. G., Faull, R. L. M. The neuropathology of Huntington's disease. Current Topics in Behavioral Neurosciences. 22, 33-80 (2014).
- Kim, M. Beta conformation of polyglutamine track revealed by a crystal structure of huntingtin N-terminal region with insertion of three histidine residues. Prion. 7 (3), 221-228 (2013).
- Hoop, C. L., et al. Huntingtin exon 1 fibrils feature an interdigitated β-hairpin-based polyglutamine core. Proceedings of the National Academy of Sciences of the United States of America. 113 (6), 1546-1551 (2016).
- Vieweg, S., Ansaloni, A., Wang, Z. M., Warner, J. B., Lashuel, H. A. An intein-based strategy for the production of tag-free huntingtin exon 1 proteins enables new insights into the polyglutamine dependence of Httex1 aggregation and fibril formation. Journal of Biological Chemistry. 291 (23), 12074-12086 (2016).
- Seong, I. S., et al. Huntingtin facilitates polycomb repressive complex 2. Human Molecular Genetics. 19 (4), 573-583 (2009).
- Li, W., Serpell, L. C., Carter, W. J., Rubinsztein, D. C., Huntington, J. A. Expression and characterization of full-length human huntingtin, an elongated HEAT repeat protein. Journal of Biological Chemistry. 281 (23), 15916-15922 (2006).
- Vijayvargia, R., et al. Huntingtin's spherical solenoid structure enables polyglutamine tract-dependent modulation of its structure and function. eLife. 5, 11184(2016).
- Huang, B., et al. Scalable production in human cells and biochemical characterization of full-length normal and mutant huntingtin. PLoS ONE. 10 (3), 0121055(2015).
- Guo, Q., et al. The cryo-electron microscopy structure of huntingtin. Nature. 555 (7694), 117-120 (2018).
- Harding, R. J., et al. Design and characterization of mutant and wildtype huntingtin proteins produced from a toolkit of scalable eukaryotic expression systems. Journal of Biological Chemistry. 294 (17), 6986-7001 (2019).
- Harding, R. J., et al. HAP40 orchestrates huntingtin structure for 1 differential interaction with polyglutamine 2 expanded exon 1. bioRxiv. , (2021).
- Huang, B., et al. Pathological polyQ expansion does not alter the conformation of the Huntingtin-HAP40 complex. Structure. 29 (8), 804-809 (2021).
- Colin, E., et al. Huntingtin phosphorylation acts as a molecular switch for anterograde/retrograde transport in neurons. EMBO Journal. 27 (15), 2124-2134 (2008).
- Thompson, L. M., et al. IKK phosphorylates Huntingtin and targets it for degradation by the proteasome and lysosome. Journal of Cell Biology. 187 (7), 1083-1099 (2009).
- Ratovitski, T., et al. Post-translational modifications (PTMs), identified on endogenous Huntingtin, cluster within proteolytic domains between HEAT repeats. Journal of Proteome Research. 16 (8), 2692-2708 (2017).
- Taylor, S. C., Berkelman, T., Yadav, G., Hammond, M. A defined methodology for reliable quantification of western blot data. Molecular Biotechnology. 55 (3), 217-226 (2013).
- Tarazona, M. P., Saiz, E. Combination of SEC/MALS experimental procedures and theoretical analysis for studying the solution properties of macromolecules. Journal of Biochemical and Biophysical Methods. 56 (1-3), 95-116 (2003).
- Folta-Stogniew, E. Oligomeric states of proteins determined by size-exclusion chromatography coupled with light scattering, absorbance, and refractive index detectors. Methods in Molecular Biology. 328, Clifton, N.J. 97-112 (2006).
- McMeekin, T. L., Wilensky, M., Groves, M. L. Refractive indices of proteins in relation to amino acid composition and specific volume. Biochemical and Biophysical Research Communications. 7 (2), 151-156 (1962).
- Atwal, R. S., et al. Huntingtin has a membrane association signal that can modulate huntingtin aggregation, nuclear entry and toxicity. Human Molecular Genetics. 16 (21), 2600-2615 (2007).
- Kegel-Gleason, K. B. Huntingtin interactions with membrane phospholipids: Strategic targets for therapeutic intervention. Journal of Huntington's Disease. 2 (3), 239-250 (2013).
- Michalek, M., Salnikov, E. S., Werten, S., Bechinger, B. Membrane interactions of the amphipathic amino terminus of huntingtin. Biochemistry. 52 (5), 847-858 (2013).
- Wittig, I., Braun, H. P., Schägger, H. Blue native PAGE. Nature Protocols. 1 (1), 418-428 (2006).
- Nissley, D. A., O'Brien, E. P. Altered co-translational processing plays a role in huntington's pathogenesis-A hypothesis. Frontiers in Molecular Neuroscience. 9, 54(2016).
- Kumar, N., Gammell, P., Clynes, M. Proliferation control strategies to improve productivity and survival during CHO based production culture: A summary of recent methods employed and the effects of proliferation control in product secreting CHO cell lines. Cytotechnology. 53 (1-3), 33-46 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten