Method Article
Produzione efficiente e scalabile di varianti di huntingtina umana a lunghezza intera in cellule di mammifero utilizzando un sistema di espressione transitoria
In questo articolo
Riepilogo
Forniamo protocolli scalabili che coprono la progettazione di costrutti, la trasfezione transitoria, l'espressione e la purificazione di varianti proteiche di huntingtina umana a lunghezza intera in cellule HEK293.
Abstract
L'huntingtina a lunghezza intera (FL HTT) è una proteina di grandi dimensioni (aa 1-3.144), ubiquitariamente espressa, contenente poliglutammina (polyQ) con una massa di circa 350 kDa. Mentre la funzione cellulare della FL HTT non è completamente compresa, un'espansione mutante del tratto polyQ sopra ~36 ripetizioni è associata alla malattia di Huntington (HD), con la lunghezza del polyQ correlata approssimativamente con l'età di esordio. Per comprendere meglio l'effetto della struttura sulla funzione dell'HTT mutante (mHTT), sono necessarie grandi quantità di proteina. La produzione submilligramma di FL HTT nelle cellule di mammifero è stata ottenuta utilizzando l'espressione stabile della linea cellulare inducibile dalla doxiciclina. Tuttavia, la produzione di proteine da linee cellulari stabili ha limitazioni che possono essere superate con metodi di trasfezione transitoria.
Questo documento presenta un metodo robusto per la produzione di quantità di basso milligrammo di FL HTT e delle sue varianti da plasmidi ottimizzati per codone mediante trasfezione transitoria utilizzando polietilenimina (PEI). Il metodo è scalabile (>10 mg) e produce costantemente 1-2 mg/L di coltura cellulare di FL HTT altamente purificata. Coerentemente con i rapporti precedenti, lo stato della soluzione purificata di FL HTT è risultato altamente dinamico; La proteina ha una propensione a formare dimeri e oligomeri di alto ordine. Una chiave per rallentare la formazione di oligomeri è lavorare rapidamente per isolare le frazioni monomeriche dalle frazioni dimeriche e oligomeriche di alto ordine durante la cromatografia di esclusione dimensionale.
La cromatografia ad esclusione dimensionale con diffusione della luce multiangolo (SEC-MALS) è stata utilizzata per analizzare il dimero e il contenuto oligomerico di ordine superiore dell'HTT purificato. Non è stata osservata alcuna correlazione tra la lunghezza del poliQ HTT FL (Q23, Q48 e Q73) e il contenuto di oligomeri. Il costrutto esone1-cancellato (aa 91-3,144) ha mostrato una propensione all'oligomerizzazione comparabile a FL HTT (aa 1-3,144). I metodi di produzione, purificazione e caratterizzazione mediante indice di rifrazione SEC/MALS (RI), elettroforesi su gel di sodio dodecilsolfato-poliacrilammide (SDS-PAGE), western blot, Native PAGE e Blue Native PAGE sono descritti nel presente documento.
Introduzione
La malattia di Huntington (HD) è una rara malattia neurodegenerativa caratterizzata principalmente da movimenti motori instabili e involontari, nonché alterazioni cognitive e psichiatriche, come cambiamenti di personalità e apatia 1,2. La MH è associata ad un'espansione del tratto di ripetizione CAG localizzato nell'esone 1 del gene huntingtina (HTT) a più di 35 ripetizioni, con un numero maggiore di ripetizioni CAG correlate ad un esordio precoce della malattia 3,4. Il prodotto traslazionale di HTT, la proteina huntingtina (HTT), è implicato nella vitalità neuronale e nello sviluppo del cervello 5,6,7,8,9.
HTT è una proteina di impalcatura segnalata per partecipare a una vasta gamma di processi cellulari, trasporto di vescicole, divisione cellulare, ciliogenesi e autofagia10,11. Tuttavia, la patogenesi molecolare della MH non è del tutto chiara, e manca l'identificazione di interattori proteici chiave che mediano l'impatto patologico della mHTT espansa con polyQ. Alcune ricerche suggeriscono un guadagno della funzione tossica da mHTT guidato dalla propensione all'oligomerizzazione della proteina HTT espansa, poiché gli aggregati HTT sono stati identificati nei neuroni e nella glia in pazienti MH e modelli animali della malattia 12,13,14,15,16,17 . Per alimentare lo studio della funzione e della struttura delle varianti FL HTT e mHTT e fornire ai ricercatori standard proteici di alta qualità per lo sviluppo del test, è necessario un apporto robusto e scalabile di proteine ricombinanti omogenee.
A causa delle sue dimensioni (aa 1-3.144, numerazione basata sulla lunghezza del poliQ Q23), dell'instabilità proteolitica e della propensione all'aggregazione, FL HTT si è dimostrato difficile da esprimere e isolare come proteina solubile. In precedenza, la regione dell'esone 1 (aa 2-90) di HTT è stata espressa e purificata su larga scala utilizzando vari tag che possono aumentare la solubilità della proteina in Escherichia coli18,19,20. FL HTT è stato prima espresso e purificato in un sistema di espressione di cellule di insetti utilizzando baculovirus 21,22 e sono state riportate strutture di microscopia elettronica (EM) a bassa risoluzione 30 Å di FL Q23-HTT e Q78-HTT chimicamente reticolato23. Lo studio della struttura HTT è stato ulteriormente avanzato quando la produzione di FL Q17, Q46 e Q128-HTT con modificazioni post-traduzionali native (PTM) è stata raggiunta in cellule umane utilizzando linee cellulari stabili o sistemi di espressione di adenovirus24. Questi studi suggeriscono che, sebbene l'HTT purificato esista principalmente allo stato monomerico, tende anche a formare oligomeri e aggregati di alto ordine.
L'ultracentrifugazione analitica di FL Q128-HTT, con una regione polyQ altamente espansa, ha fornito più frazioni oligomeriche e aggregate rispetto alla proteina con la regione polyQ non espansa24. Utilizzando una linea cellulare stabile, una strategia è stata adattata con successo per stabilizzare FL HTT attraverso la co-espressione con il partner di interazione HAP40. Una struttura crio-EM del complesso FL HTT e HAP40 è stata risolta con una risoluzione media di 4 Å utilizzando il complesso proteico purificato (PDB:6EZ8)25. Questa strategia di co-espressione è stata adattata con successo a un sistema di baculovirus e una serie di varianti HTT di alta qualità con diverse lunghezze di polyQ sono state espresse e purificate da cellule di insetti26. Da allora, altre strutture crio-EM del complesso di HTT con lunghezze variabili di polyQ e HAP40 e strutture ad alta risoluzione sono state risolte e depositate nel Protein Data Base27,28 (PDB: 7DXK, 7DXH, 6X9O).
Abbiamo ottimizzato un metodo di trasfezione ed espressione in cellule HEK293, utilizzando polietilenimina (PEI), per una rapida espressione transitoria di FL HTT. Come prova di principio, le varianti FL HTT contenenti 23 glutammine (FL Q23-HTT) sono state prima purificate e caratterizzate utilizzando una modifica di un metodo di purificazione descritto in precedenza24. Questo metodo di trasfezione transitoria è conveniente, altamente efficiente e scalabile; può produrre HTT purificato con rese di 1-2 mg/L, paragonabili al metodo della linea cellulare stabile riportato24. Poiché la proteina è prodotta in una linea cellulare umana, l'HTT prodotto ha maggiori probabilità di avere PTM umani nativi quando sottoposto ad analisi proteomica della spettrometria di massa 11,29,30,31. Sono state prodotte quantità di milligrammi delle varianti FL Q48-HTT, FL Q73-HTT ed exon1-deleted (ΔExon1-HTT) di FL HTT, dimostrando che il metodo di espressione transitoria è particolarmente utile nella produzione rapida di varianti alternative di HTT senza dipendere dallo sforzo dispendioso in termini di tempo richiesto per stabilire linee cellulari stabili per la produzione.
Il seguente protocollo esemplifica il metodo standard utilizzato nel laboratorio di questi autori per la coltura cellulare, la trasfezione, la purificazione delle proteine e la caratterizzazione proteica postpurificazione per produrre FL Q23-HTT da una coltura cellulare da 2 L. Il protocollo può essere scalato fino a colture più grandi o adattato per purificare altre varianti HTT. Fino a 10 colture cellulari L di FL HTT e varie mutazioni del sito o del troncamento di omologhi HTT e HTT sono state eseguite con successo in laboratorio utilizzando lo stesso protocollo. FL HTT purificato contiene un'alta percentuale di monomeri insieme a dimeri e oligomeri di ordine superiore. Lo stesso profilo aggregato è osservato tra le varianti prodotte (Q23, Q48, Q73 e Exon1 cancellato). Poiché l'aggregazione può verificarsi quando non viene presa la dovuta attenzione, è stato condotto uno studio di formulazione e stabilità gelo-scongelamento per identificare le migliori condizioni per la gestione delle proteine. Vengono inoltre descritti metodi quali Blue Native PAGE e SEC/MALS-RI per analizzare il contenuto di oligomeri HTT come parte del processo di controllo qualità. A beneficio della comunità di ricerca sulla MH, i plasmidi e le proteine HTT descritti in questo studio sono anche depositati nel MH Community Repository presso il Coriell Institute (www.coriell.org/1/CHDI).
Protocollo
1. Progettazione e produzione di costrutti per l'espressione di mammiferi HTT marcati FLAG
- Recuperare la sequenza proteica HTT umana a lunghezza intera (P42858) dal National Center for Biotechnology Information (https://www.ncbi.nlm.nih.gov/).
NOTA: I ricercatori devono avere familiarità con le organizzazioni di dominio HTT e mantenere la struttura 3D di base HTT quando progettano costrutti per mutanti HTT. - Richiedi un servizio di sintesi genica per eseguire l'ottimizzazione del codone per l'espressione cellulare umana basata sulla sequenza di P42858. Cambiare il numero polyQ da Q16 alla lunghezza Q desiderata (Q23 è stato scelto come primo costrutto qui) e sintetizzare il gene HTT a lunghezza intera.
NOTA: Il costrutto Q23-HTT a lunghezza intera ottimizzato per il codone sintetizzato è stato consegnato come inserto nel plasmide pUC18 in questo studio. - Facoltativo: aggiungere funzionalità per facilitare la clonazione di diverse lunghezze Q e la purificazione nei costrutti.
NOTA: Un sito di scissione del virus dell'incisione del tabacco (TEV) e un tag di purificazione FLAG (AAAENLYFQGDYKDDDDK) sono stati aggiunti all'estremità C-terminale dei costrutti. Due siti HindIII sono stati progettati nei costrutti per comprendere la regione polyQ (la sequenza proteica tradotta non viene modificata introducendo siti HindIII). Ciò consente al ricercatore di modificare la lunghezza Q di HTT limitando la digestione enzimatica e la legatura senza risintetizzare l'intero gene HTT .
2. Clonare i costrutti HTT sintetizzati in pcDNA3.1.
- Digerire 5 μg di pUC18-Q23-HTT e 5 μg di pcDNA3.1 utilizzando 2 μL di NheI e PmeI ciascuno a 37 °C per 2 ore.
- Eseguire un gel di agarosio allo 0,5% p/v e purificare il frammento Q23-HTT e il vettore pcDNA3.1 digerito utilizzando un kit di estrazione del gel di agarosio. Quantificare le concentrazioni di DNA purificato mediante OD280 utilizzando uno spettrometro UV in grado di misurare microlitri di campioni.
NOTA: OD260/280 compreso tra 1,8 e 2,0 è tipicamente osservato. L'HTT FL sintetizzato viene fornito come inserto con NheI e PmeI ad entrambe le estremità in un plasmide pUC18. Utilizzare altri enzimi di restrizione se HTT è sintetizzato in modo diverso. - Utilizzare 10 ng di vettore pcDNA3.1 digerito nella reazione. Ligare il DNA purificato al rapporto molare 1:1 (HTT:pcDNA3.1) in una reazione di 10 μL a temperatura ambiente per 5 minuti utilizzando la DNA ligasi T4.
- Trasformare il prodotto legato in cellule E. coli competenti (vedi Tabella dei Materiali) utilizzando il protocollo specificato dal produttore della ligasi.
- Prelevare 6 colonie singole ed effettuare colture notturne in 4-6 mL di LB integrate con 100 μg/mL di carbenicillina a 37 °C.
- Allocare 1 mL da ogni coltura notturna. Aggiungere glicerolo al 25% v/v e conservare il brodo di glicerolo a -80 °C. Purificare la coltura notturna rimanente utilizzando un kit mini-preparazione secondo i passaggi specificati nel manuale dell'utente.
- Sequenziare tutti i plasmidi utilizzando primer di sequenziamento che attraversano la regione di trascrizione del plasmide. Scegli uno stock di glicerolo che ha la sequenza corretta come stock di glicerolo principale e scarta il resto.
- Opzionale: Richiedi un servizio di sintesi genica per sintetizzare il DNA con le diverse lunghezze Q (Q48, Q73 ed Exon1) che si estendono sui due siti HindIII nel plasmide pcDNA3.1-Q23-HTT. Digerire pcDNA3.1-Q23-HTT e i DNA appena sintetizzati usando HindIII e riprodurli con T4 ligasi come nei passaggi 2.2-2.7 per rendere FL HTT con diverse lunghezze di polyQ nel plasmide pcDNA3.1.
NOTA: I costrutti plasmidi utilizzati in questo studio sono disponibili anche direttamente presso l'HD Community Repository presso il Coriell Institute (www.coriell.org/1/CHDI); vedere la tabella dei materiali.
3. DNA plasmidico privo di endotossine per la trasfezione su larga scala
- Strisciare le scorte batteriche di glicerolo di pcDNA3.1-Q23-HTT-TEV-FLAG su una piastra di agar LB con carbenicillina (100 μg/ml). Incubare la piastra a 37 °C per 16-24 ore fino alla comparsa di singole colonie.
- Prelevare una singola colonia, inoculare una coltura starter da 5 mL in un mezzo ricco formulato per l'amplificazione plasmidica con carbenicillina (100 μg/ml) e crescere a 37 °C per 8 ore.
- Scegli un kit di purificazione del plasmide GIGA privo di endotossine. Seguire i passaggi descritti nel manuale del kit plasmide GIGA per purificare il plasmide pcDNA3.1-Q23-HTT-TEV-FLAG.
- Misurare i livelli di endotossina plasmidica utilizzando un kit di quantificazione dell'endotossina basato su limulus amoebocyte lysate (LAL). Seguire la procedura specificata nel manuale del produttore.
NOTA: Una purificazione plasmidica di alta qualità e a basso livello di endotossine è essenziale per ottenere una buona efficienza di trasfezione. Utilizzando questo protocollo, è possibile ottenere 20-40 mg di plasmide (forma superarrotolata >80%) per L di coltura batterica a concentrazioni plasmidiche > 4 mg/ml. Un plasmide adeguatamente purificato dovrebbe avere un livello di endotossina < 30 EU/mg. OD260/280 compreso tra 1,8 e 2,0 è tipicamente osservato.
4. Trasfezione su larga scala di 2 L di cellule HEK293 mediante polietilenimmina (PEI)
- Aggiungere 1 g di PEI 25K a 1 L di acqua senza endotossine agitando. Regolare il pH a 2,0 utilizzando 100 mM HCl e mescolare fino a quando tutto il PEI 25K si dissolve. Regolare il pH a 7,0 utilizzando una soluzione di NaOH da 100 mM e filtrare attraverso un filtro da 0,2 μm. Aliquote e conservare a -20 °C per un massimo di un anno.
NOTA: Le aliquote di PEI possono essere conservate a 4 °C per un massimo di due settimane, ma non devono mai essere ricongelate dopo lo scongelamento. - Propagare le cellule HEK293 nel terreno di crescita (vedere la Tabella dei Materiali) integrate con penicillina-streptomicina (concentrazione finale a 5 U/mL per la penicillina e 5 μg/mL per la streptomicina) in un incubatore umidificato a 37 °C, 90 rpm, 5% CO2 per 18-24 h. Diluire le cellule a 2 L ad una densità di ~1,2 × 106 cellule/ml utilizzando il terreno di coltura in 5 L di palloni Erlenmeyer un giorno prima della trasfezione.
- Continuare a far crescere le cellule a 37 °C, 90 rpm, 5% CO2 per 18-24 h. Misurare i parametri delle celle utilizzando un contatore automatico delle celle in grado di misurare la densità e la vitalità delle celle seguendo il manuale dell'utente.
NOTA: La densità cellulare dovrebbe raddoppiare e la vitalità dovrebbe essere >95%. La densità cellulare prima della trasfezione dovrebbe essere di circa 2,0 × 10 6-2,4 × 106 cellule/ml. Diluire le cellule alla densità desiderata prima della trasfezione quando necessario. - Calcolare le quantità di plasmide e PEI necessarie per la trasfezione; utilizzare 1 mg di plasmide e 3 mg di PEI per la trasfezione di ogni litro di coltura cellulare. Assegnare 2 mg di plasmide e 6 mg di PEI necessari per una trasfezione da 2 L.
- Diluire singolarmente il plasmide e il PEI in un volume di soluzione salina tamponata fosfato pari a 1/20del volume totale della coltura cellulare (100 ml ciascuno per una trasfezione da 2 L) e incubare a temperatura ambiente per 5 minuti. Mescolare il plasmide diluito e il PEI ruotando delicatamente e incubare la miscela a temperatura ambiente per 30 minuti.
NOTA: La miscela apparirà leggermente torbida dopo l'incubazione. - Aggiungere la miscela alla coltura cellulare e ruotare delicatamente per mescolarli.
- Coltivare le cellule a 37 °C, 5% CO2, 90 rpm per 24 h.
- Aggiungere una soluzione di butirrato di sodio 2 M ad una concentrazione finale di 2 mM. Aggiungere alla coltura 1:1000 (v/v) antiagglomerante e 1:1000 (v/v) antischiuma.
- Spostare il pallone in un incubatore umidificato a 32 °C, 90 giri/min, 5% CO2 e continuare a crescere per 48 ore.
- Misurare i parametri delle celle, tra cui la densità e la vitalità delle celle, utilizzando il contatore automatico delle celle seguendo il manuale dell'utente.
- Trasferire 2,0 × 10 6 cellule (Vol = 2,0 × 106/densità cellulare) in una provetta per microcentrifuga. Pellettare le cellule a 2.000 × g per 1 minuto in una centrifuga per l'estrazione occidentale nella sezione 5.
- Raccogliere le celle mediante centrifugazione a 2.000 × g per 30 minuti e conservare il pellet cellulare a -80 °C prima della purificazione.
5. SDS-PAGE e western blot del lisato cellulare HEK293 per stimare il livello di espressione di HTT
- Prelevare un'aliquota di 2,0 × 106 cellule precedentemente congelate (fase 4.11) dalla trasfezione su larga scala della coltura cellulare HEK293. Aggiungere 250 μL di soluzione salina tamponata Tris (TBS) integrata con 50 μg/mL di digitonina, 5 mM EDTA e 1x cocktail di inibitori della proteasi, e risospendere il pellet cellulare aspirando più volte utilizzando una pipetta.
- Ruotare delicatamente i tubi per 30 minuti a 4 °C utilizzando un minirotatore per lisare le celle. Pellettare il materiale insolubile centrifugando a 17.000 × g per 5 min.
- Aggiungere 1/3 delvolume del tampone di carico riducente di litio dodecilsolfato (LDS) 4x al surnatante e riscaldare a 70 °C per 10 minuti.
- Caricare 5-20 μL di lisato cellulare su un gel prefabbricato Tris-acetato PAGE al 3-8%. Utilizzando il tampone di funzionamento 1x Tris-acetate SDS compatibile con il gel, eseguire il gel in modalità a tensione costante a 150 V per 60 minuti.
NOTA: Tris-acetato SDS-PAGE è stato utilizzato per l'analisi FL HTT perché genera una risoluzione più elevata rispetto ad altri tipi di SDS-PAGE per le proteine con peso molecolare superiore a 300 kDa. Le proteine utilizzate in questo studio sono anche disponibili direttamente presso l'HD Community Repository presso il Coriell Institute (www.coriell.org/1/CHDI); vedere la tabella dei materiali. - Per eseguire il western blotting, assemblare un sandwich di trasferimento utilizzando una carta transfer spessa con tampone di trasferimento, una membrana di polivinilidenfluoruro (PVDF) attivata da metanolo e un gel SDS-PAGE. Trasferire le proteine alla membrana PVDF utilizzando un blotter occidentale semisecco secondo il manuale utente del produttore.
NOTA: In genere, 20-30 minuti a 135 mA sono sufficienti per una membrana di 10 cm x 10 cm. - Smontare il sandwich di trasferimento e bloccare la membrana in TBST (20 mM Tris pH 7,4, 150 mM NaCl e 0,1% v/v Tween-20) integrato con 5% p/v di latte non grasso.
- Incubare la membrana su un bilanciere per 1 ora a temperatura ambiente con 15 mL di anticorpo primario (1:2.500 diluizione per l'anticorpo monoclonale anti-FLAG e 1:2.000 per tutti gli altri anticorpi primari).
NOTA: Gli anticorpi primari utilizzati in questo studio sono anti-FLAG M2, MAB5492, MAB5490, MAB2166, MAB3E10, MAB4E10, MAB2168, MAB8A4 (vedere la tabella dei materiali). - Lavare la membrana 3 x 5 minuti utilizzando 30-50 ml di TBST.
- Incubare la membrana su un bilanciere con un anticorpo secondario IgG anti-topo coniugato con colorante fluorescente a 1:15.000, temperatura ambiente, in 15 ml di TBST contenente il 5% p/v di latte in polvere.
- Visualizza le bande di western blot su un imager fluorescente usando la lunghezza d'onda specifica per l'anticorpo secondario. Quantificare il segnale di banda utilizzando il software che accompagna l'imager secondo il manuale dell'utente.
NOTA: Il western blotting quantitativo può essere eseguito utilizzando HTT purificato come standard. Una gamma standard lineare di HTT è specifica per lo strumento ed è stata stabilita in questo laboratorio da 25 ng a 250 ng di HTT per corsia utilizzando un anticorpo anti-FLAG. La macchia occidentale di HTT dovrebbe essere priva di degrado; si osserva tipicamente un livello di espressione totale di HTT di 2-4 pg/cellula. Fare riferimento a un protocollo32 pubblicato in precedenza per i dettagli su come eseguire un western blot quantitativo.
6. Cromatografia liquida rapida delle proteine (FPLC) purificazione dell'HTT utilizzando colonna anti-FLAG e SEC
- Purificazione Anti-FLAG
- Stimare la quantità di resina FLAG necessaria per la purificazione (tipicamente, 12 ml di resina di affinità anti-FLAG M2 per la purificazione di 2-4 L di coltura cellulare trasfettata). Imballare 12-25 mL di resina anti-FLAG su una colonna vuota (vedere la Tabella dei Materiali) utilizzando FPLC ad una portata di 4 mL/min utilizzando il Buffer A (Tabella 1). Regolare l'altezza dello stantuffo, in modo che non vi sia spazio tra l'estremità dello stantuffo e il letto di resina.
- Utilizzando un rapporto di 10 mL di tampone di lisi per 1 g di pellet cellulare, scongelare e sospendere il pellet cellulare in tampone di lisi freddo (Tabella 1).
- Far passare la sospensione cellulare una volta attraverso un omogeneizzatore ad alto taglio a 10.000 psi. Chiarificazione del lisato mediante centrifugazione a 20.000 × g per 1 h in una centrifuga dotata di rotore ad angolo fisso compatibile.
- Programmare l'FPLC (vedere la tabella dei materiali per il software utilizzato nello studio) ed eseguire le seguenti sequenze.
- Caricare il lisato chiarificato tramite la pompa del campione.
- Lavare con 4 volumi di colonna (CV) del buffer A (tabella 1).
- Lavare con 4 CV di tampone B (tabella 1).
- Lavare con 8 CV di tampone C (tabella 1).
- Lavare con 3 CV di tampone D (Tabella 1).
- Lavare con 3 CV di Elution Buffer (Tabella 1).
- Analizzare 10 μL delle frazioni di picco utilizzando SDS-PAGE. Raccogliere e combinare le frazioni di picco con la purezza desiderata. Risparmiare ~50 μL degli eluati combinati per l'analisi SDS-PAGE.
NOTA: Normalmente, apparirà un singolo picco e tutte le frazioni eluite nel picco contengono ~ 90% HTT puro. - Rigenerare una colonna anti-FLAG utilizzando 5 CV di Regeneration Buffer (Tabella 1) e riequilibrare la colonna utilizzando 5 CV di Buffer A.
NOTA: La resina Anti-FLAG può essere riutilizzata fino a cinque volte o fino a quando la relativa resa/litro scende al 50% della prima purificazione.
- Purificazione della colonna di esclusione dimensionale (SEC) mediante una colonna SEC
- Preequilibrare una colonna SEC che consenta la separazione di proteine con peso molecolare (MW) > 500 kDa (vedere la Tabella dei Materiali per la colonna utilizzata) utilizzando 2 × CV di SEC Buffer (Tabella 1).
- Caricare direttamente l'eluato anti-FLAG (dal punto 6.1.5) tramite un superloop da 50 ml. Eseguire 1,2 × CV di buffer SEC per iniezione. Eseguire la separazione SEC durante la notte a 4 °C.
NOTA: Un massimo di 5 ml o 15 ml di campione proteico può essere caricato sulle colonne SEC selezionate in questo studio. Programmare l'FPLC in modo che più iniezioni possano essere eseguite automaticamente. Gli script dei metodi di esempio sono inclusi anche come file supplementare 1 e file supplementare 2. - Confrontare il profilo di eluizione con il profilo di eluizione HTT standard per distinguere i picchi monomerici, dimeri e oligomeri di ordine superiore. Raggruppare le frazioni monomeriche HTT in base al profilo di eluizione della colonna SEC. Se lo si desidera, raggruppare separatamente le frazioni HTT oligomeriche e dimeriche di ordine superiore.
- Concentrare la proteina HTT aggregata utilizzando un concentratore centrifugo da 100 kDa a 4 °C. Calcolare le concentrazioni proteiche dividendo i loro valori OD280 per i rispettivi coefficienti di estinzione (i coefficienti di estinzione teorici di Q23-HTT, Q48-HTT, Q73-HTT e ΔExon1-HTT sono 0,776, 0,769, 0,762 e 0,798 (mg / ml)-1 cm-1, rispettivamente, per il calcolo). Mantenere la concentrazione di HTT ≤ 1,0 mg/ml.
NOTA: È essenziale monitorare il processo di concentrazione poiché la concentrazione eccessiva comporterà l'aggregazione. - Aliquot la proteina HTT purificata in provette da microcentrifuga crio-sicure in un volume < 100 μL. Congelare le aliquote utilizzando azoto liquido e conservarle a -80 °C.
7. HPLC SEC-MALS-dRI analitico per analizzare la polidispersità HTT
- Eseguire tutte le analisi SEC-MALS a 4 °C su un sistema di cromatografia liquida (HPLC) ad alte prestazioni abbinato a un rilevatore UV, un rilevatore multiangolo di diffusione della luce e un rilevatore a indice di rifrazione differenziale (dRI).
- Prima di collegare la colonna UHPLC al sistema, spurgare la pompa e i rilevatori con acqua filtrata (0,1 μm) di grado HPLC.
- Collegare la colonna UHPLC (vedere la tabella dei materiali per la colonna utilizzata) al sistema. Equilibrare la colonna con acqua filtrata (0,1 μm) e quindi buffer SEC-MALS (Tabella 1) fino a quando tutti i segnali del rivelatore raggiungono la linea di base.
- Iniettare 2 μL di 6 mg/ml di albumina sierica bovina (BSA) ad una velocità di flusso di 0,3 ml/min per 15 minuti per iniezione e ispezionare la qualità dei dati. Eseguire la normalizzazione, l'allineamento dei picchi e la correzione dell'ampliamento della banda in base al profilo BSA e creare un modello per le seguenti esecuzioni di esempio HTT.
- Scongelare rapidamente una fiala del campione FL Q23-HTT in un bagno d'acqua a temperatura ambiente usando un galleggiante. Filtrare l'HTT attraverso un filtro di spin da 0,1 μm. Iniettare 2-4 μL del campione HTT e farlo funzionare per 15 minuti a 4 °C ad una portata di 0,3 mL/min.
- Analizzare i dati cromatografici e di diffusione della luce utilizzando il software di accompagnamento (vedere la tabella dei materiali). Utilizzare il rilevatore dRI come rilevatore di concentrazione e utilizzare 0,185 come incremento dell'indice di rifrazione (dn/dc) per HTT. Generare un grafico Zimm per determinare la massa molecolare media in peso per ogni picco33,34.
NOTA: L'incremento dell'indice di rifrazione di HTT viene calcolato come 0,185 utilizzando il software SEDFIT35 e la sequenza di aminoacidi primari di HTT come input.
NOTA: Il monomero HTT MW è determinato da SEC-MALS a ~370 kDa ± 30 kDa. L'HTT purificato ha tipicamente un contenuto di monomeri compreso tra il 60 e il 75% (in questo laboratorio). Un basso contenuto di monomeri può indicare che è necessario prestare maggiore attenzione nella manipolazione per prevenire l'aggregazione.
8. Blue Native PAGE per analizzare la polidispersità HTT
- Preparare 1 L di tampone anodico miscelando 50 mL di 20x Blue Native PAGE running buffer (vedere la tabella dei materiali) con 950 mL di H 2 O. Preparare 2 L di tampone catodico bluscuro mescolando 100 mL di 20x Blue Native PAGE running buffer e 100 mL di additivo catodico Blue Native PAGE (20x) con 1.800 mL di H2O. Raffreddare i tamponi a 4 °C prima dell'uso.
- Scongelare rapidamente una fiala di campione FL Q23-HTT in un bagno d'acqua a temperatura ambiente usando un galleggiante. Conservare la proteina scongelata sul ghiaccio prima dell'uso.
- Mescolare 5 μg di FL Q23-HTT (~1 mg/ml), 1 μL di additivo G250 allo 0,5%, 2,5 μL di 4x tampone campione Blue Native PAGE e acqua per portare il volume finale a 10 μL.
- Caricare il campione misto FL Q23-HTT su un gel prefabbricato Bis-Tris al 3-12%. Caricare 7,5 μL dello standard proteico non colorato nello stesso gel dello standard.
- Riempire la parte anteriore del serbatoio con tampone catodico blu scuro e la parte posteriore del serbatoio con tampone anodico.
NOTA: riempire i buffer dopo il caricamento del campione per consentire una facile visualizzazione durante il caricamento dei campioni. - Eseguire il gel a 150 V per 120 minuti in una cella frigorifera.
- Colorare il gel con una soluzione decolorante (Tabella 1) fino a quando non si osservano bande; Trasferire il gel in acqua. Visualizza e documenta il gel su una stazione di imaging.
NOTA: Blue Native PAGE è stato originariamente progettato per analizzare le proteine di membrana. È stato adattato in questo laboratorio come metodo alternativo per stimare il contenuto monomerico di HTT. Si lega alle regioni idrofobiche di HTT e impedisce la formazione di aggregati in condizioni tampone prive di detergente. La tradizionale PAGE nativa senza l'utilizzo di Coomassie blue G250 fa sì che HTT formi oligomeri solubili e aggregati, probabilmente a causa delle numerose sacche idrofobiche esistenti in HTT.
9. SDS PAGE seguita da colorazione Coomassie o argento per analizzare la purezza HTT
- Aggiungere 4x LDS sample buffer e 10x reagente riducente a FL Q23-HTT purificato per rendere la concentrazione finale del tampone di carico e reagente riducente a 1x.
- Riscaldare il campione su un blocco riscaldante a secco a 70 °C per 10 minuti.
- Caricare un massimo di 1 μg di proteine per pozzetto su un gel di acetato di Tris al 3-8% e correre a 150 V per 1 ora utilizzando il tampone di funzionamento Tris-acetato SDS.
NOTA: Le proteine utilizzate in questo studio sono disponibili anche direttamente presso l'HD Community Repository presso il Coriell Institute (www.coriell.org/1/CHDI); vedere la tabella dei materiali. - Macchia di Coomassie
- Lavare il gel con H2O per 5 min.
- Colorare il gel nella soluzione colorante di Coomassie (Tabella 1) facendo oscillare il gel in 30 ml di soluzione colorante per 15 minuti.
- Rimuovere la macchia facendo oscillare il gel in 50 ml di H2O per 5 minuti. Ripetere due volte. Visualizza e documenta il gel colorato con Coomassie su una stazione di imaging.
- Macchia d'argento utilizzando un kit di colorazione argento commerciale.
- Dopo SDS-PAGE, fissare il gel utilizzando la soluzione di fissaggio (Tabella 1) per 1 ora fino a notte a temperatura ambiente.
- Eseguire la macchia, lavare e sviluppare secondo le istruzioni del kit.
- Interrompere immediatamente la fase di sviluppo una volta che le bande raggiungono l'intensità desiderata.
- Documentare il gel in un sistema di documentazione del gel dotato di una fonte di luce visibile.
NOTA: HTT purificato al >95% può essere rilevato da Coomassie e colorazione dell'argento con questo protocollo. Fare riferimento a un protocollo32 pubblicato in precedenza per i dettagli su come eseguire l'analisi quantitativa delle proteine.
Risultati
Un vettore di espressione transitoria (pcDNA3.1-Q23-HTT-TEV-FLAG, Figura 1A) è progettato per la produzione rapida in cellule di mammifero di FL Q23-HTT (aa 1-3,144, basato sulla numerazione Q23). Questo costrutto ha le caratteristiche progettate per generare rapidamente vari costrutti di mutazione HTT mediante clonazione a cassetta, facilitare la purificazione della proteina HTT ad alta qualità e omogeneità con passaggi cromatografici minimi e avere la possibilità di produrre FL HTT senza tag. L'elenco delle funzionalità include 1. I siti di digestione di restrizione HindIII, che circondano la ripetizione CAG nell'esone 1 HTT, possono essere utilizzati per generare mutanti FL HTT con un tratto polyQ di varie lunghezze mediante digestione enzimatica di restrizione e legatura; 2. l'estremità C-terminale di FL HTT è marcata con un epitopo FLAG con un sito di riconoscimento della proteasi TEV per la purificazione di affinità in un'unica fase di FL HTT con elevata purezza e generazione opzionale di proteina FL HTT senza tag utilizzando la scissione della proteasi TEV; 3. Sequenza FL HTT ottimizzata per Codone per l'utilizzo del codone cellulare umano per l'espressione di alto livello nelle cellule HEK293. Il vettore pcDNA 3.1 (+) viene utilizzato come spina dorsale del costrutto per sfruttare l'elevata attività di attivazione trascrizionale del promotore CMV in linee cellulari di mammifero.
Utilizzando pcDNA3.1-Q23-HTT-TEV-FLAG come modello di partenza, i costrutti Q48 e Q73 FL HTT sono stati prodotti sintetizzando frammenti di DNA con lunghezza Q appropriata che copre due siti enzimatici di restrizione HindIII e scambiando la stessa regione nel modello. Il mutante ΔExon1 di FL HTT (aa 91-3,144) (Figura 1B) è stato prodotto utilizzando primer diretti a residui cancellati che attraversano la regione dell'esone 1 nel modello. Le cellule HEK293 trasfettate con pcDNA3.1-Q23-HTT-TEV-FLAG utilizzando PEI sono state coltivate in matraccio da 5 litri di CO2 inferiore al 5%. Una tipica purificazione su larga scala utilizza un pellet di celle da 2-10 litri contenente 6,0 × 10 9-3,0 × 1010 celle. Prima di procedere alla purificazione, il livello di espressione di HTT da ciascuna trasfezione è stato stimato mediante western blotting quantitativo utilizzando HTT ricombinante purificato marcato FLAG come standard e anticorpo anti-FLAG come primo anticorpo. Per la purificazione sono stati utilizzati pellet con un livello di espressione HTT stimato a ≥2 pg HTT/cella.
La purificazione di FL HTT consiste in un processo a colonna in 2 fasi, prima con purificazione di affinità anti-FLAG e poi con SEC su una colonna di filtrazione in gel con un intervallo di separazione adatto per HTT (Figura 2A; vedi Tabella dei materiali per esempi). Dopo entrambe le fasi, l'HTT è stato ottenuto con una purezza del campione del >95%, come determinato da SDS-PAGE con blu di Coomassie e contenuto di monomeri del >65% basato su SEC-MALS analitico. Poiché sia il tempo di purificazione prolungato che la temperatura hanno un impatto negativo sul contenuto finale di monomeri HTT, FPLC è stato utilizzato in entrambe le fasi di purificazione per ridurre al minimo la manipolazione e ottenere una qualità costante del campione. Il principale contaminante durante la purificazione anti-FLAG è stato lo chaperone Hsp70 determinato mediante spettrometria di massa (Figura 2B, corsia 2). Ciò è coerente con la scoperta che Hsp70 è co-purificato con FL HTT stabilmente espresso nelle linee cellulari umane24, suggerendo che Hsp70 può essere uno stabilizzatore comune per FL HTT in vivo.
La contaminazione da Hsp70 può essere eliminata mediante un ampio lavaggio con cloruro di magnesio e ATP durante la fase di purificazione dell'affinità anti-FLAG (Figura 2B, corsia 1). Dopo la rimozione di Hsp70, FL HTT è incline a formare oligomeri di ordine superiore24 e deve essere mantenuto ad una concentrazione ≤ 1 mg/ml. La fase di concentrazione prima della SEC può spesso tradursi in un'aggregazione significativa. Pertanto, la migliore pratica consiste nel caricare direttamente le frazioni di picco dalla purificazione anti-FLAG sulla colonna di esclusione dimensionale senza concentrazione. Dopo SEC, il campione è stato concentrato a ≤1 mg/ml per il massimo recupero di FL HTT monomerico. La quantità di HTT recuperata da ciascuna fase di purificazione è stata stimata mediante blu di Coomassie o western blotting quantitativo utilizzando FL HTT purificato come standard di quantificazione (Tabella 2). La resa tipica delle proteine FL HTT purificate prodotte con il metodo descritto è di circa 1 mg/L di coltura cellulare, ma può scendere ben al di sotto di quella (Tabella 3) a causa della variabilità da lotto a lotto, o se la resina purificante anti-FLAG viene riutilizzata più volte.
La sovraespressione di FL HTT può provocare la frammentazione della proteina22. FL Q23-HTT prodotto con il metodo qui descritto risolto come una singola banda con il corretto MW di 350 kDa da SDS PAGE, colorato con Coomassie G250 o con colorazione d'argento (Figura 2C). Con il western blotting, FL Q23-HTT ha reagito con anticorpi generati contro epitopi a N-terminale, C-terminale e diversi domini intermedi, senza ulteriori bande correlate ai frammenti osservate, indicando che la proteina è stata isolata senza significativi troncamenti rilevabili (Figura 3A). Le varianti di lunghezza PolyQ di FL HTT Q23, Q48 e Q73 hanno reagito come previsto nel western blot, mostrando un segnale progressivamente più forte per mAb MW1 diretto da polyQ correlato all'aumento della lunghezza Q: Q23-HTT < Q48-HTT < Q73-HTT (Figura 3B). Non è stato osservato alcun segnale per ΔExon1-HTT (aa 91-3,144) quando sondato con gli anticorpi MW1 e MAB549, che colpiscono l'esone terminale N 1 (Figura 3B).
SEC-MALS è stato impiegato per analizzare lo stato di aggregazione e la massa molecolare della proteina HTT purificata. I campioni sono stati analizzati da SEC analitici monitorati da rilevatori UV, MALES e dRI. La massa molare assoluta ottenuta da SEC-MALS non dipende dalla forma delle molecole33,34; pertanto, SEC-MALS fornisce una stima imparziale di MW per le frazioni monomeriche e oligomeriche quando sono ben separate. Tra le colonne HPLC testate, la colonna SEC (vedi la tabella dei materiali) ha mostrato una risoluzione sufficiente tra il monomero HTT e il dimero in modo tale da poter distinguere le masse molari (Figura 4). La concentrazione proteica è stata determinata mediante rilevazione di dRI. Gli incrementi dell'indice di rifrazione (dn/dc) di FL HTT sono 0,1853 mL/g calcolati dal software SEDFIT35. Modelli analitici di eluizione SEC simili sono stati osservati per ΔExon1 HTT (91-3,144), FL Q23, Q48 e Q73 HTT (1-3,144), ciascuno costituito da un picco monomero maggiore con picchi dimerici e oligomerici minori (Tabella 4). Il MW calcolato per la forma monomerica è maggiore del MW teorico. Ciò è probabilmente causato dalla sovrapposizione di specie da picchi oligomerici di ordine superiore e da errori derivanti da deboli segnali dRI poiché le proteine HTT sono mantenute in bassa concentrazione per evitare la formazione di oligomeri di ordine superiore. Integrando i picchi UV di diversi lotti di varianti FL HTT purificate, non è stata osservata una chiara correlazione tra la lunghezza del polyQ e il profilo aggregato (Tabella 4).
Oltre alla SEC analitica, è stata eseguita la tradizionale PAGE nativa per determinare se può essere utilizzata come metodo complementare per caratterizzare lo stato oligomerico FL HTT. Gli oligomeri di ordine superiore sono stati risolti attraverso gel di Tris-acetato al 3-8% utilizzando tampone nativo senza detergente. FL HTT purificato da SEC ha mostrato bande multiple corrispondenti agli stati di oligomerizzazione (Figura 5A). La banda più bassa era situata tra il marcatore nativo 480 kDa e 720 kDa, simile ai risultati precedenti riportati per FL HTT purificato da cellule di insetti22. Tuttavia, il monomero HTT non era la banda più abbondante quando si utilizzava il tradizionale PAGE nativo e i risultati non sono correlati con il profilo aggregato determinato dal SEC-MALS analitico. Diversi cerotti idrofobici presenti in FL HTT36,37,38, in particolare l'interfaccia idrofobica tra HAP40 e FL HTT25, possono contribuire alla formazione di oligomeri di ordine superiore durante la migrazione all'interno del gel. Questo perché le regioni idrofobiche sono note per interagire tra loro in assenza di interazioni detergenti o stabilizzanti proteina-proteina. Coerentemente con le proprietà idrofobiche dell'HTT, FL HTT forma quantità crescenti di frazioni oligomeriche di ordine superiore in assenza di CHAPS durante la fase di purificazione SEC.
Blue Native PAGE, che è ampiamente utilizzato per studiare proteine di membrana e grandi complessi proteici contenenti cerotti idrofobici39, è stato confrontato con il tradizionale PAGE nativo. L'HTT purificato ha mostrato tre bande principali su Blue Native PAGE con MW stimati di 643, 927 e 1070 kDa (Figura 5B) che probabilmente rappresentano rispettivamente le specie monomeriche, dimeriche e trimeriche di HTT. La banda monomerica è rimasta la banda più abbondante nella Blue Native PAGE, corrispondendo bene al profilo analitico SEC degli stessi campioni. La sovrastima di MW del monomero HTT da parte di Blue Native PAGE può derivare dall'unica struttura sferica cava o dalle regioni idrofobiche di HTT che causano una migrazione più lenta rispetto ai corrispondenti marcatori di peso molecolare 11,23,25. Nel complesso, FL Q23-HTT, FL Q48-HTT, FL Q73-HTT e ΔExon1-HTT hanno profili PAGE nativi blu simili con solo lievi differenze nella migrazione della banda proteica a causa delle loro differenze di peso molecolare.
Come ulteriore controllo della qualità delle proteine purificate, il tag C-terminale FLAG può essere rimosso da FL HTT mediante trattamento con proteasi TEV. Dopo la scissione proteolitica, i campioni sono stati analizzati mediante western blot utilizzando quattro anticorpi per confermare la rimozione del tag FLAG e rilevare la degradazione HTT. L'immunoreattività all'anti-FLAG M2 e tre anticorpi specifici per l'huntingtina con epitopi al N-terminale, ai domini intermedi e al C-terminale dell'HTT ha mostrato una rimozione efficace del tag FLAG e nessun prodotto di degradazione specifico per HTT (Figura supplementare S1).
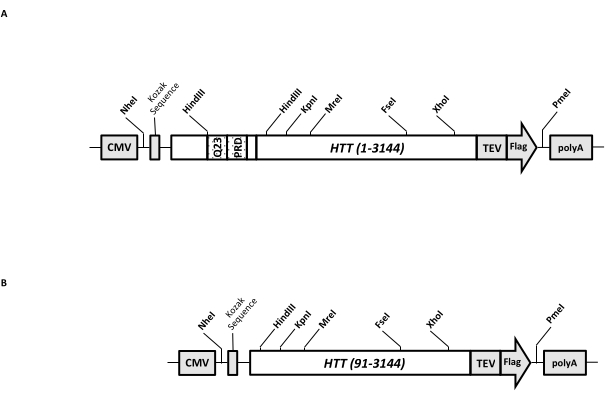
Figura 1: Costrutto per l'espressione HTT a lunghezza intera. (A) L'HTT Q23 a lunghezza intera è stato ottimizzato per il codone e clonato nel plasmide pcDNA3.1 (+). L'estremità 3' di HTT è stata marcata con epitopo Flag e sito di scissione della proteasi TEV per produrre proteine HTT prive di tag. L'allungamento della poliglutammina e il dominio ricco di prolina sono stati progettati con siti endonucleasi di restrizione HindIII affiancati per inserire ulteriori ripetizioni CAG utilizzando la clonazione a cassetta, cioè Q48 e Q73, per produrre varianti HTT con diverse lunghezze di polyQ. (B) Il costrutto ΔExon1 è stato realizzato con mutagenesi PCR usando pcDNA3.1-Q23-HTT come modello. I residui 91-3.144 di HTT sono rimasti nel costrutto ΔExon1 per l'espressione. Abbreviazioni: HTT = huntingtin; CMV = citomegalovirus; Q23 = allungamento della poliglutammina; PRD = dominio ricco di prolina; TEV = sito di scissione del virus dell'incisione del tabacco. Fare clic qui per visualizzare una versione ingrandita di questa figura.
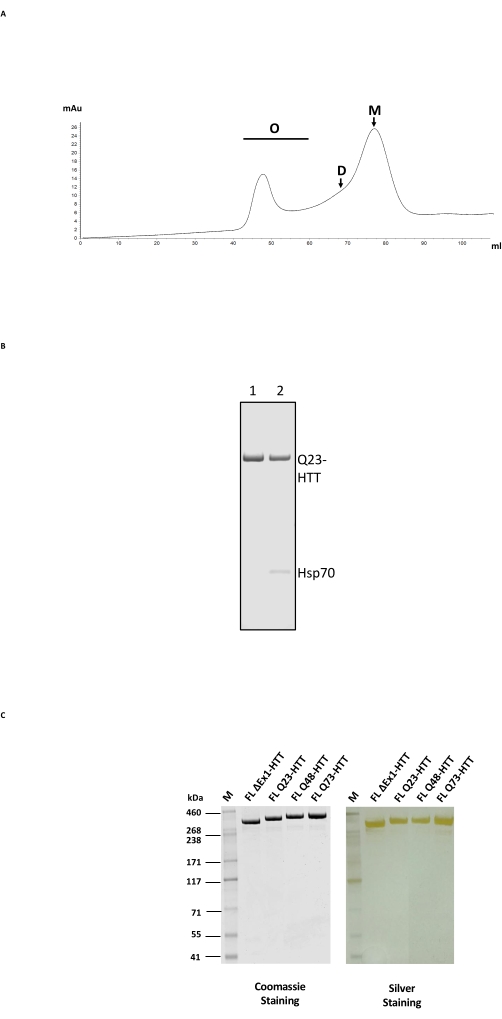
Figura 2: Purificazione su larga scala di HTT. (A) Profilo SEC di Q23-HTT a lunghezza intera purificato anti-Flag su una colonna FPLC. Gli oligomeri di alto ordine, i picchi di dimero e monomeri di Q23-HTT sono etichettati. Le frazioni contenenti monomero sono state raccolte come campione HTT finale. (B) SDS-PAGE di Q23-HTT purificato con fase di lavaggio ATP/magnesio (corsia 1) o senza lavaggio ATP/magnesio dà luogo a co-eluizione Hsp70 (corsia 2). (C) Varianti HTT finali purificate a figura intera su SDS-PAGE colorate con blu Coomassie G-250 o colorazione argento. Abbreviazioni: FL = lunghezza intera; HTT = huntingtina; SEC = cromatografia ad esclusione dimensionale; FPLC = cromatografia liquida proteica veloce; O = oligomero; D = dimero; M = monomero; SDS-PAGE = elettroforesi su gel di sodio dodecilsolfato poliacrilammide; Hsp70 = proteina da shock termico 70. Fare clic qui per visualizzare una versione ingrandita di questa figura.
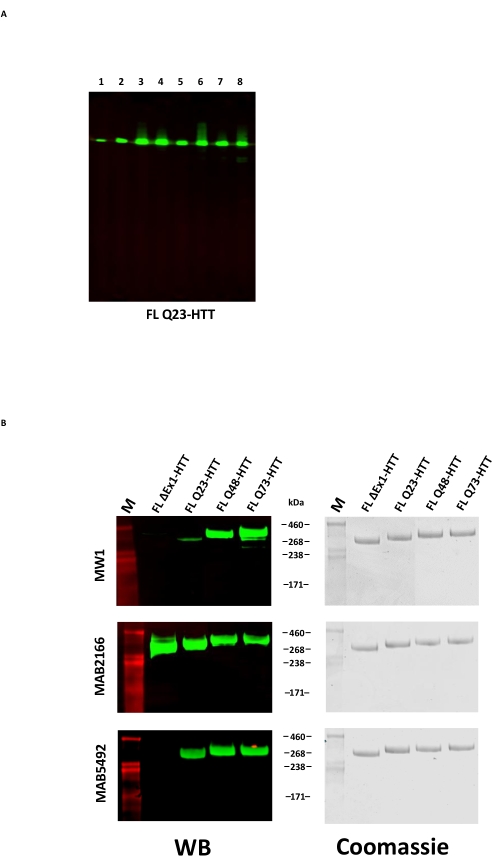
Figura 3: Analisi Western blot delle varianti HTT purificate . (A) FL Q23-HTT purificato è stato eseguito su SDS-PAGE e trasferito su membrana PVDF. Gli anticorpi primari e gli epitopi interagenti sono Lane 1, α-FLAG M2, tag FLAG; Corsia 2, MAB5492, HTT aa. 1-82; Corsia 3, MAB5490, HTT AA 115-129; Corsia 4, MAB2166, HTT AA 181-810; Corsia 5, MAB3E10, HTT aa 1.171-1.177; Corsia 6, MAB4E10, HTT aa 1.844-2.131; Corsia 7, MAB2168, HTT aa 2.146-2.541; Corsia 8, MAB8A4, HTT aa 2.703-2.911. (B) 1 μg di varianti FL HTT purificate sono state eseguite su SDS-PAGE e trasferite in PVDF (a sinistra), e un doppio gel SDS è stato eseguito e colorato con Coomassie Blue (a destra). Gli anticorpi primari e gli epitopi interagenti sono Row 1, MW1, ripetizioni PolyQ espanse; Fila 2, MAB2166, HTT aa 181-810; Fila 3, MAB5492, HTT aa 1-82. Abbreviazioni: FLL Q23-HTT = proteina huntingtina a lunghezza intera contenente 23 residui di glutammina; SDS-PAGE = elettroforesi su gel di sodio dodecilsolfato poliacrilammide; WB = macchia occidentale; M = marcatore; PVDF = fluoruro di polivinilidene. Fare clic qui per visualizzare una versione ingrandita di questa figura.
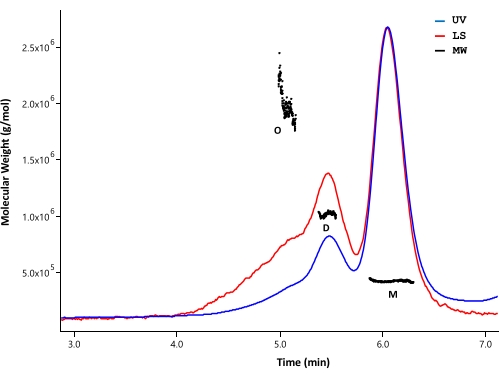
Figura 4: Analisi SEC-MALS dell'HTT full-length. Il Q23-HTT purificato a lunghezza intera è stato eluito su una colonna UPLC. Sono indicate le posizioni di picco del monomero, del dimero e dell'oligomero previsti. I pesi molecolari sono stati calcolati per i picchi di monomeri, dimeri e trimeri ed elencati nella Tabella 5. Profili di eluizione simili sono osservati per Q48, Q73 e ΔExon1 HTT, con contenuti variabili di monomeri, dimeri e oligomeri in ogni purificazione. Abbreviazioni: SEC-MALS = Cromatografia ad esclusione dimensionale con diffusione della luce multi-angolo; UV = ultravioletti; LS = diffusione della luce; MW = Peso molecolare; Q23-HTT = proteina huntingtina contenente 23 residui di glutammina; M = monomero; D = dimero; O = oligomero. Fare clic qui per visualizzare una versione ingrandita di questa figura.
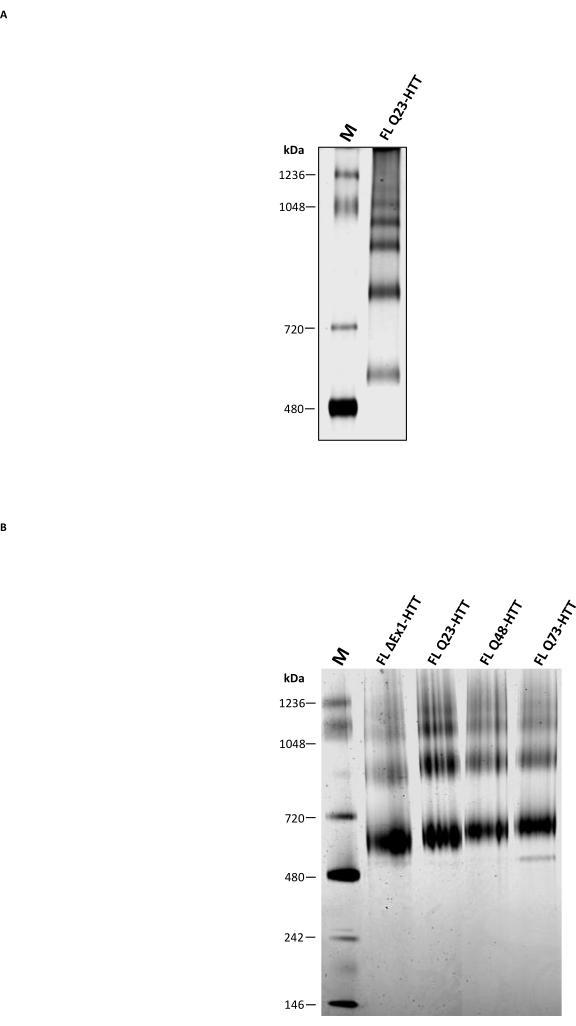
Figura 5: Caratterizzazione dell'HTT purificato utilizzando gel PAGE nativo trasparente o gel PAGE nativo blu. Il marcatore nativo e l'apparente Q23-HTT monomerico di SEC sono stati risolti su gel di tris-acetato al 3-8% in un sistema PAGE non denaturante (A) e in un sistema Blue Native PAGE (B). Abbreviazioni: FL = full-length; Q23-HTT = proteina huntingtina contenente 23 residui di glutammina; PAGE = elettroforesi su gel di poliacrilammide; M = marcatore. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Passo | Nome | Composizione | ||
| 6.1.1 | Buffer A | 50 mM Tris, 500 mM NaCl, 5% v/v glicerolo, 5 mM EDTA, 0,01% v/v Tween-20, pH 8,0. | ||
| 6.1.2 | Tampone di lisi | 50 mM Tris, 500 mM NaCl, 5% v/v glicerolo, 5 mM EDTA e 1x cocktail inibitore della proteasi | ||
| 6.1.4.2 | Buffer A | 50 mM Tris, 500 mM NaCl, 5% v/v glicerolo, 5 mM EDTA, 0,01% v/v Tween-20, pH 8,0. | ||
| 6.1.4.3 | Buffer B | 50 mM Tris; 500 mM KCl; 5 mM MgCl2; 5% v/v glicerolo; 0,01% v/v Tween-20, pH 8,0 | ||
| 6.1.4.4 | Buffer C | Tris 20 mM; 200 mM KCl; 5 mM MgCl2; 5 mM ATP; 0,01% v/v Tween-20; 5% v/v glicerolo, pH 8,0 | ||
| 6.1.4.5 | Buffer D | 50 mM Tris; 500 mM NaCl; 5% v/v glicerolo; 5 mM EDTA; 0,5% p/v CHAPS, pH 8,0 | ||
| 6.1.4.6 | Buffer di eluizione | 50 mM Tris; 500 mM NaCl; 5% v/v glicerolo; 0,5% w/v CHAPS; 0,2 mg/mL di peptide DYKDDDDK, pH 8,0 | ||
| 6.1.6 | Buffer di rigenerazione | 0,1 M glicina HCl, pH 3,5; 0,01% v/v Tween-20 | ||
| 6.2.1 | SEC Buffer | 50 mM Tris, 500 mM NaCl, 5% v/v glicerolo, 0,5% p/v CHAPS, 1 mM TCEP | ||
| 7.3 | SEC-MALS Buffer | 50 mM HEPES, pH 7,2, 500 mM NaCl, 5% v/v glicerolo, 0,5% p/v CHAPS | ||
| 8.7 | Soluzione decolorante | 40% v/v metanolo e 7% v/v acido acetico | ||
| 9.4.2 | Soluzione colorante Coomassie | 0,01% p/v Coomassie G250, 50% v/v/metanolo, 10% v/v acido acetico | ||
| 9.5.1 | Soluzione di fissaggio | 50% v/v metanolo, 10% v/v acido acetico, 50 μL di formaldeide/100 mL di soluzione | ||
Tabella 1: Composizione dei tamponi e delle soluzioni
| Passi | Concentrazione HTT (mg/ml) | Volume totale (ml) | Contenuto di HTT (mg) | Resa HTT per cella (pg/cella) | % Rendimento |
| Supernatante | 0.1792 | 220 | 39.4 | 4.4 | 100 |
| Anti-bandiera | 1.524 | 8.6 | 13.1 | 1.47 | 33.4 |
| SECONDO | 0.91 | 3.9 | 3.54 | 0.4 | 9.1 |
Tabella 2: Resa HTT da un pellet HEK293 da 2 L trasfettato con pcDNA3.1-Q23-HTT-TEV-Flag. Abbreviazioni: FL Q23-HTT = proteina huntingtina a lunghezza intera contenente 23 residui di glutammina; TEV = sito di scissione del virus dell'incisione del tabacco; SEC = cromatografia ad esclusione dimensionale.
| Esempio HTT | Resa HTT (mg/L) | Purezza media (%) | ||
| BCA | A280 | |||
| 1 | FL DEx1-HTT (N=3) | 0.67-1.30 | 0.69-1.18 | 99.3 |
| 2 | FL Q23-HTT (N=3) | 0.25-0.92 | 0.28-0.98 | 96.9 |
| 3 | FL Q48-HTT (N=3) | 0.28-1.15 | 0.38-1.16 | 97.4 |
| 4 | FL Q73-HTT (N=3) | 0.58-1.05 | 0.57-0.97 | 98.8 |
Tabella 3: Riepilogo della resa proteica di quattro purificazioni della variante FL HTT e della loro purezza finale. Abbreviazione: FLL HTT = proteina huntingtina a lunghezza completa.
| Esempio HTT | Un | D | M |
| FL Q23-HTT | 4.2-6.9% | 18.7-29.3% | 66.5-76.0% |
| FL Q48-HTT | 4.0-9.4% | 10.6-17.8% | 73.6-85.4% |
| FL Q73-HTT | 2.0-14.0% | 16.9-24.6% | 65.1-81.1% |
Tabella 4: Riepilogo del contenuto rappresentativo di aggregati, dimeri e monomeri delle varianti FL HTT della purificazione. Abbreviazioni: FL HTT = proteina huntingtina a lunghezza completa; A = aggregato; D = dimero; M = monomero; SEC = cromatografia ad esclusione dimensionale.
Figura supplementare S1: Analisi Western blot dopo digestione della proteasi TEV. FL Q23-HTT e FL Q48-HTT purificati sono stati eseguiti su SDS-PAGE, trasferiti su membrane PVDF e analizzati mediante western blotting dopo il digest TEV. Gli anticorpi primari utilizzati erano anti-Flag M2 (Flag tag), MAB5492 (HTT aa 1-82), MAB3E10 (HTT aa 997-1,276) e MAB2168 (HTT aa 2,146-2,541). Corsia 1, Protein Standard; Corsia 2, Q23-HTT-TEV-Bandiera; Corsia 3, Q48-HTT-TEV-Bandiera; Corsia 4, Q23-HTT-TEV-Flag trattata con proteasi TEV a 1:5, durante la notte a 4 °C; Corsia 5, Q48-HTT-TEV-Flag trattata con proteasi TEV a 1:5, durante la notte a 4 °C. Abbreviazioni: FL HTT = proteina huntingtina a lunghezza completa; SDS-PAGE = elettroforesi su gel di sodio dodecilsolfato poliacrilammide; TEV = virus dell'incisione del tabacco; PVDF = fluoruro di polivinilidene. Clicca qui per scaricare questo file.
Figura supplementare S2: Analisi SEC-MALS delle varianti FL HTT sottoposte a cicli di congelamento-scongelamento. Q23-HTT (A) e Q48-HTT (B) purificati sono stati congelati a -80 °C e scongelati a temperatura ambiente per un massimo di 6 volte. Q23-HTT e Q48-HTT dopo il primo ciclo di congelamento-disgelo e il sesto ciclo di congelamento-disgelo sono stati quindi analizzati da SEC-MALS. Una leggera diminuzione della frazione monomerica e un aumento delle frazioni di dimero e oligomeri di alto ordine sono stati osservati dalla diffusione della luce dopo ripetuti cicli di congelamento-disgelo. Sono indicate le posizioni di picco del monomero, del dimero e dell'oligomero di ordine elevato previsti. Abbreviazioni: FL HTT = proteina huntingtina a lunghezza completa; O = oligomero; D = dimero; M = monomero; SEC-MALS = Cromatografia ad esclusione dimensionale con diffusione della luce multi-angolo. Clicca qui per scaricare questo file.
Figura supplementare S3: PAGINA SDS delle varianti FL HTT sottoposte a cicli di congelamento-scongelamento. I Q23-HTT purificati (corsie 2-7) e Q48-HTT (corsie 9-14) sono stati congelati a -80 °C e scongelati a temperatura ambiente fino a 6 volte. Le aliquote di Q23-HTT e Q48-HTT sono state conservate dopo ogni ciclo di congelamento-scongelamento e quindi analizzate da SDS PAGE. Non è stato osservato alcun aumento dei prodotti aggregati o di degradazione; I campioni sono stati considerati stabili e puri al >95% mediante densitometria a banda. Abbreviazioni: FL HTT = proteina huntingtina a lunghezza completa; SDS-PAGE = elettroforesi su gel di sodio dodecilsolfato poliacrilammide. Clicca qui per scaricare questo file.
File supplementare 1: script FPLC 15 mL anti-FLAG HTT. Abbreviazioni = FPLC = cromatografia liquida a proteine veloci; HTT = proteina huntingtina. Clicca qui per scaricare questo file.
File supplementare 2: script FPLC SEC_MALS HTT. Abbreviazioni: SEC-MALS = Cromatografia ad esclusione dimensionale con diffusione della luce multi-angolo; FPLC = cromatografia liquida proteica veloce; HTT = proteina huntingtina. Clicca qui per scaricare questo file.
Discussione
Descriviamo qui un metodo di trasfezione, espressione e purificazione transitoria per generare più costrutti proteici FL HTT con purezza e omogeneità adeguate per l'uso come standard per lo sviluppo di saggi immunologici e MS, controlli per l'analisi western blot e per studi struttura-funzione. Questo metodo di espressione transitoria è scalabile e versatile e consente all'utente di generare quantità di basso milligrammo di varianti FL HTT in modo più efficiente rispetto all'utilizzo di linee cellulari stabili o metodi basati su virus descritti in precedenza21,22,23,24. Di routine, 2-5 mg di FL HTT altamente purificato possono essere generati da una produzione di proteine su scala 2 L in meno di una settimana utilizzando il metodo di espressione transitoria una volta costruito il plasmide, con una resa tipica di 1-2,5 mg di FL HTT per litro di coltura cellulare.
Il metodo di espressione transitoria qui descritto supera molti ostacoli nell'espressione stabile delle linee cellulari, come il lungo tempo necessario per stabilire linee cellulari e le difficoltà nello stoccaggio e nel mantenimento di linee cellulari stabili. Il PEI è anche relativamente economico rispetto ad altri reagenti di trasfezione sul mercato, rendendo economicamente sostenibile la trasfezione su larga scala. Ci sono anche limitazioni nel protocollo: l'efficienza della trasfezione dipende in gran parte dalla qualità dei plasmidi, dalla crescita ottimale delle cellule e dal modo in cui il PEI viene conservato e preparato. Gli operatori devono prestare particolare attenzione ed eseguire controlli di qualità in questi passaggi critici per evitare un drastico calo delle rese proteiche. Anche la resina Anti-FLAG utilizzata nel protocollo è relativamente costosa e mostra una ridotta cattura di FL HTT dopo diverse purificazioni e rigenerazioni. Alcuni ricercatori potrebbero trovare più pratico passare a un tag diverso per consentire una rigenerazione più robusta della resina di affinità.
Sono state testate varie linee cellulari e condizioni di espressione per ottimizzare i livelli di espressione di FL HTT. Le cellule HEK293 sono state scelte per l'espressione di FL HTT a causa dell'elevata espressione delle proteine e della facilità di manipolazione in un formato di coltura in sospensione, rendendo il metodo adatto per l'espressione su larga scala in agitatori o bioreattori. Un livello di espressione proteica FL HTT più elevato può essere raggiunto a temperature di coltura più basse come 32 °C piuttosto che utilizzare la temperatura abituale di 37 °C. È possibile che la temperatura più bassa possa rallentare la sintesi proteica e promuovere il corretto ripiegamento di FL HTT40. Tuttavia, questo fenomeno non è specifico per FL HTT o le linee cellulari testate. La ridotta temperatura post-trasfezione è stata ampiamente utilizzata nell'espressione proteica farmaceutica nelle cellule CHO. Sebbene il meccanismo non sia completamente compreso, si pensa che le basse temperature arrestino il ciclo cellulare nella fase G1 e deviino l'energia cellulare verso la produzione di proteine41.
HTT a lunghezza intera purificato da cellule di mammifero co-eluisce con lo chaperone Hsp7024 e le fasi di lavaggio Mg-ATP possono rimuovere la proteina Hsp70. È interessante notare che Hsp70 co-eluito non è osservato in FL HTT purificato da un sistema di espressione cellulare di insetti21,22,23. Ciò può riflettere una differenza nei PTM di FL HTT o nelle risposte delle proteine da shock termico alla sovraespressione di FL HTT nelle cellule di mammifero e di insetti. Una volta che la proteina ricombinante è stata spogliata di Hsp70, sono necessari detergenti non ionici come CHAPS o DDM per stabilizzare la forma monomerica di FL HTT.
Gli stati di oligomerizzazione delle varianti FL HTT sono stati analizzati utilizzando Blue Native PAGE e SEC-MALS. Una piccola frazione di HTT dimerico e oligomerico di ordine superiore era presente quando analizzata da Blue Native PAGE o SEC-MALS. Da notare che gli oligomeri di ordine superiore formati da FL HTT non sembrano essere correlati con la lunghezza del polyQ, e anche il mutante di delezione Exon1 mostra un rapporto oligomero-dimero-monomero simile. Le variazioni effettive nel contenuto di oligomeri tra questi costrutti sono probabilmente dovute a piccole differenze nella produzione e nella gestione di ciascun lotto. In contrasto con gli aggregati e le fibrille formati da HTT Exon140,41, gli oligomeri di ordine superiore di FL HTT sono rimasti solubili e hanno potuto essere analizzati da SEC e Native PAGE.
La FL HTT monomerica purificata è solo relativamente stabile. La conservazione prolungata a 4 °C, brevi incubazioni a temperatura ambiente o concentrazioni > 1 mg/ml convertiranno tutti la FL HTT monomerica in forme dimeriche e oligomeriche di ordine superiore, anche se non vi è alcuna precipitazione visibile osservata in tali condizioni. FL HTT monomerico purificato mantenuto a ≤1 mg/mL è rimasto relativamente stabile a -80 °C nel tampone di stoccaggio (50 mM Tris, pH 8,0, 500 mM NaCl, 5% v/v glicerolo, 0,5% p/v CHAPS e 5 mM DTT) come descritto in precedenza24. Fino a 6 cicli di congelamento-scongelamento di FL HTT preparati e conservati in questo modo non hanno causato precipitazioni visibili della proteina, sebbene un leggero spostamento verso uno stato oligomerico superiore sia stato osservato da SEC-MALS (Figura supplementare S2). I campioni sono stati analizzati anche da SDS PAGE dopo ripetuti cicli di congelamento-disgelo. Non sono stati osservati precipitati visibili; nessun aggregato o prodotto di degradazione aggiuntivo è stato visto da SDS-PAGE (Figura supplementare S3). La stabilità a lungo termine della FL HTT purificata è ancora oggetto di studio. In assenza di dati conclusivi a lungo termine, si consiglia di conservare FL HTT purificato a -80 °C per non più di 6 mesi.
Le varianti della proteina FL HTT ricombinante di alta qualità e i metodi per produrle sono molto richiesti dalla comunità di ricerca sulla MH. Queste proteine sono in uso come test immunologici e standard analitici per la SM, in studi strutturali e per lo sviluppo di nuovi saggi specifici per FL HTT. I metodi di espressione transitoria su larga scala qui descritti hanno costantemente prodotto quantità di milligrammi di varianti FL HTT con purezza del >95%, fornendo strumenti essenziali per gli studi HTT. La produzione di decine di milligrammi di varianti altamente purificate di FL HTT polyQ e di altri mutanti a supporto della ricerca sulla MH è diventata routine.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse con i contenuti di questo articolo.
Riconoscimenti
Ringraziamo il Dipartimento di Scienze Farmaceutiche, The State University of New York a Buffalo, per aver eseguito l'analisi MS di HTT. Questo lavoro è stato uno sforzo collaborativo con la Fondazione CHDI. Ringraziamo in particolare Elizabeth M. Doherty; Ignacio Munoz-Sanjuan; Douglas Macdonald, Fondazione CHDI; e Rory Curtis, Curia, per il loro prezioso contributo durante la preparazione di questo manoscritto. Siamo anche grati a Michele Luche, Mithra Mahmoudi e Stephanie Fox per il loro sostegno a questo sforzo di ricerca.
Materiali
| Name | Company | Catalog Number | Comments |
| 100 kDa concentrator-Amicon | Millipore | UFC910096 | Protocol Section Number-6.2.4 |
| 20x blue native PAGE running buffer | Invitrogen | BN2001 | Protocol Section Number-8.1 |
| 20x TBS | Thermo Fisher | PI28358 | Protocol Section Number-5.1 |
| 4x blue native PAGE sample buffer | Invitrogen | BN2003 | Protocol Section Number-8.3 |
| 4x LDS loading buffer | Invitrogen | NP0007 | Protocol Section Number-5.3 |
| 5 L Erlenmeyer flasks | Corning | 431685 | Protocol Section Number-4.2 |
| Agarose gel extraction kit | Qiagen | 28704 | Protocol Section Number-2.2 |
| Anti-clumping agent | Thermo Fisher | 0010057AE | Protocol Section Number-4.8 |
| anti-FLAG M2 affinity gel | Sigma | A2220 | Protocol Section Number-6.1.1 |
| anti-FLAG M2 | Sigma | F3165 | Protocol Section Number-5.7 |
| Anti foam-Excell anti foam | Sigma | 59920C-1B | Protocol Section Number-4.8 |
| ATP | Sigma | A6419 | Protocol Section Number-6.1.4.4 |
| BEH 450 SEC | Waters | 186006851 | 2.5 µm x 4.6 mm x 150 mm Protocol Section Number-7.3 |
| blue native PAGE 5% G-250 sample additive | Invitrogen | BN2004 | Protocol Section Number-8.3 |
| carbenicillin | Thermo Fisher | 10177012 | Protocol Section Number-2.5 |
| centrifuge - Sorvall Lynx 6000 | Thermo Fisher | 75006590 | Protocol Section Number-6.1.3 |
| Cell Counter - ViCELL | BECKMAN COULTER | Protocol Section Number-4.3 | |
| CHAPS | Anatrace | C316S | Protocol Section Number-6.1.4.6 |
| Competent E. coli cells-TOP10 | Invitrogen | C404010 | Protocol Section Number-2.4 |
| digitonin | Sigma | D141 | Protocol Section Number-5.1 |
| differential refractive index detector | Wyatt | Protocol Section Number-7.1 | |
| DYKDDDDK peptide | Genscript | Peptide synthesis service Protocol Section Number-6.1.4.6 | |
| EDTA | Sigma | EDS | Protocol Section Number-5.1 |
| EndoFree Plasmid Giga Kit | Qiagen | 12391 | Protocol Section Number-3.3 |
| Endotoxin free water | Cytiva | SH30529.03 | Protocol Section Number-4.1 |
| endotoxin quantification kit-CRL Endosafe Nexgen-PTS detection system | Charles River | PTS150K | Protocol Section Number-3.4 |
| fixed angle rotor A23-6x100 rotor | Thermo Fisher | 75003006 | Protocol Section Number-6.1.3 |
| FPLC software- Unicorn 6.2 | Cytiva | Protocol Section Number-6.1.4 | |
| Gene synthesis | Genscript | Gene synthesis service Protocol Section Number-1.2 | |
| Glycerol | Fisher Scientific Glycerol (Certified ACS) | G33-4 | Protocol Section Number-5.6 |
| Growth Medium-Expi293 expression medium | Thermo Fisher | A1435102 | Protocol Section Number-4.2 |
| HEK293 cells | Thermo Fisher | R79007 | Protocol Section Number-4 |
| high shear homogenizer-Microfluidizer | MicroFluidics | LM10 | Protocol Section Number-6.1.3 |
| HPLC - 1260 infinity II Bio-Insert HPLC | Agilent | Protocol Section Number-7.1 | |
| Image Studio | LiCor | Image analysis software Protocol Section Number-5.1 | |
| MAB2166 | Sigma | MAB2166 | Protocol Section Number-5.7 |
| MAB2168 | EMD | MAB2168 | Protocol Section Number-5.7 |
| MAB3E10 | Santa Cruz | SC-47757 | Protocol Section Number-5.7 |
| MAB4E10 | Santa Cruz | SC-7757 | Protocol Section Number-5.7 |
| MAB5490 | Sigma | MAB5490 | Protocol Section Number-5.7 |
| MAB5492 | Sigma | MAB5492 | Protocol Section Number-5.7 |
| MAB8A4 | Santa Cruz | SC-47759 | Protocol Section Number-5.7 |
| multi-angle light scattering detector | Wyatt | Protocol Section Number-7.1 | |
| NativeMark Unstained Protein Standard | Invitrogen | LC0725 | Protocol Section Number-8.4 |
| NaCl | Sigma | S9888 | Protocol Section Number-5.6 |
| NheI | New England Biolab | R0131S | Hi-Fi version available Protocol Section Number-2.2 |
| NuPAGE 3–8% Tris acetate gels | Invitrogen | EA0375PK2 | Protocol Section Number-5.4 |
| NuPAGE Tris-Acetate SDS Running buffer | Invitrogen | LA0041 | Protocol Section Number-5.4 |
| PEI 25K | Polysciences | 23966-1 | Protocol Section Number-4.1 |
| Penicillin-Streptomycin | Thermo Fisher | 15070063 | Protocol Section Number-4.2 |
| Phosphate Buffered Saline (PBS) | Cytiva | SH30256.02 | Protocol Section Number-4.5 |
| plasmid miniprep kit | Qiagen | 27104 | Protocol Section Number-2.6 |
| PmeI | New England Biolab | R0560S | Protocol Section Number-2.2 |
| precast Bis-tris gel- 3-12% NativePAGE Novex Bis-Tris Gel | Invitrogen | BN1003BOX | Protocol Section Number-8.4 |
| protease inhibitor cocktail | GoldBio | GB-331-1 | Protocol Section Number-5.1 |
| SEC-MALS analysis software - Astra 7 | Wyatt Technology | Protocol Section Number-7.6 | |
| secondary antibody -IRdye 800 CW goat anti-mouse IgG | LiCor | 926-32210 | Protocol Section Number-5.9 |
| Superose 6 pg XK 16/70 | Cytiva | 90100042 | Protocol Section Number-6.2 |
| Tris base | Fisher | BP152 | Protocol Section Number-5.6 |
| Tween-20 | Thermo Fisher | AAJ20605AP | Protocol Section Number-6.1.1 |
| UV spectrometer - Nanodrop 8000 | Thermo Fisher | ND-8000-GL | Protocol Section Number-2.2 |
| XK26/100 | Cytiva | 28988951 | Protocol Section Number-6.1.1 |
Riferimenti
- Walker, F. O. Huntington's disease. Lancet. 369 (9557), 218-228 (2007).
- McColgan, P., Tabrizi, S. J. Huntington's disease: a clinical review. European Journal of Neurology. 25 (1), 24-34 (2018).
- Duyao, M., et al. Trinucleotide repeat length instability and age of onset in Huntington's disease. Nature Genetics. 4 (4), 387-392 (1993).
- MacDonald, M. E., et al. A novel gene containing a trinucleotide repeat that is expanded and unstable on Huntington's disease chromosomes. Cell. 72 (6), 971-983 (1993).
- Nasir, J., et al. Targeted disruption of the Huntington's disease gene results in embryonic lethality and behavioral and morphological changes in heterozygotes. Cell. 81 (5), 811-823 (1995).
- Dragatsis, I., Levine, M. S., Zeitlin, S. Inactivation of Hdh in the brain and testis results in progressive neurodegeneration and sterility in mice. Nature Genetics. 26 (3), 300-306 (2000).
- Anne, S. L., Saudou, F., Humbert, S. Phosphorylation of huntingtin by cyclin-dependent kinase 5 is induced by DNA damage and regulates wild-type and mutant huntingtin toxicity in neurons. Journal of Neuroscience. 27 (27), 7318-7328 (2007).
- Dietrich, P., Johnson, I. M., Alli, S., Dragatsis, I. Elimination of huntingtin in the adult mouse leads to progressive behavioral deficits, bilateral thalamic calcification, and altered brain iron homeostasis. PLoS Genetics. 13 (7), 1006846(2017).
- Dragatsis, I., et al. Effect of early embryonic deletion of huntingtin from pyramidal neurons on the development and long-term survival of neurons in cerebral cortex and striatum. Neurobiology of Disease. 111, 102-117 (2018).
- Benn, C. L., et al. Huntingtin modulates transcription, occupies gene promoters in vivo, and binds directly to DNA in a polyglutamine-dependent manner. Journal of Neuroscience. 28 (42), 10720-10733 (2008).
- Saudou, F., Humbert, S. The biology of huntingtin. Neuron. 89 (5), 910-926 (2016).
- Davies, S. W., et al. Formation of neuronal intranuclear inclusions underlies the neurological dysfunction in mice transgenic for the HD mutation. Cell. 90 (3), 537-548 (1997).
- DiFiglia, M., et al. Aggregation of huntingtin in neuronal intranuclear inclusions and dystrophic neurites in brain. Science. 277 (5334), 1990-1993 (1997).
- Gutekunst, C. A., et al. Nuclear and neuropil aggregates in Huntington's disease: Relationship to neuropathology. Journal of Neuroscience. 19 (7), 2522-2534 (1999).
- Hodgson, J. G., et al. A YAC mouse model for Huntington's disease with full-length mutant huntingtin, cytoplasmic toxicity, and selective striatal neurodegeneration. Neuron. 23 (1), 181-192 (1999).
- Hoffner, G., Djian, P. Polyglutamine aggregation in Huntington disease: does structure determine toxicity. Molecular Neurobiology. 52 (3), 1297-1314 (2015).
- Waldvogel, H. J., Kim, E. H., Tippett, L. J., Vonsattel, J. P. G., Faull, R. L. M. The neuropathology of Huntington's disease. Current Topics in Behavioral Neurosciences. 22, 33-80 (2014).
- Kim, M. Beta conformation of polyglutamine track revealed by a crystal structure of huntingtin N-terminal region with insertion of three histidine residues. Prion. 7 (3), 221-228 (2013).
- Hoop, C. L., et al. Huntingtin exon 1 fibrils feature an interdigitated β-hairpin-based polyglutamine core. Proceedings of the National Academy of Sciences of the United States of America. 113 (6), 1546-1551 (2016).
- Vieweg, S., Ansaloni, A., Wang, Z. M., Warner, J. B., Lashuel, H. A. An intein-based strategy for the production of tag-free huntingtin exon 1 proteins enables new insights into the polyglutamine dependence of Httex1 aggregation and fibril formation. Journal of Biological Chemistry. 291 (23), 12074-12086 (2016).
- Seong, I. S., et al. Huntingtin facilitates polycomb repressive complex 2. Human Molecular Genetics. 19 (4), 573-583 (2009).
- Li, W., Serpell, L. C., Carter, W. J., Rubinsztein, D. C., Huntington, J. A. Expression and characterization of full-length human huntingtin, an elongated HEAT repeat protein. Journal of Biological Chemistry. 281 (23), 15916-15922 (2006).
- Vijayvargia, R., et al. Huntingtin's spherical solenoid structure enables polyglutamine tract-dependent modulation of its structure and function. eLife. 5, 11184(2016).
- Huang, B., et al. Scalable production in human cells and biochemical characterization of full-length normal and mutant huntingtin. PLoS ONE. 10 (3), 0121055(2015).
- Guo, Q., et al. The cryo-electron microscopy structure of huntingtin. Nature. 555 (7694), 117-120 (2018).
- Harding, R. J., et al. Design and characterization of mutant and wildtype huntingtin proteins produced from a toolkit of scalable eukaryotic expression systems. Journal of Biological Chemistry. 294 (17), 6986-7001 (2019).
- Harding, R. J., et al. HAP40 orchestrates huntingtin structure for 1 differential interaction with polyglutamine 2 expanded exon 1. bioRxiv. , (2021).
- Huang, B., et al. Pathological polyQ expansion does not alter the conformation of the Huntingtin-HAP40 complex. Structure. 29 (8), 804-809 (2021).
- Colin, E., et al. Huntingtin phosphorylation acts as a molecular switch for anterograde/retrograde transport in neurons. EMBO Journal. 27 (15), 2124-2134 (2008).
- Thompson, L. M., et al. IKK phosphorylates Huntingtin and targets it for degradation by the proteasome and lysosome. Journal of Cell Biology. 187 (7), 1083-1099 (2009).
- Ratovitski, T., et al. Post-translational modifications (PTMs), identified on endogenous Huntingtin, cluster within proteolytic domains between HEAT repeats. Journal of Proteome Research. 16 (8), 2692-2708 (2017).
- Taylor, S. C., Berkelman, T., Yadav, G., Hammond, M. A defined methodology for reliable quantification of western blot data. Molecular Biotechnology. 55 (3), 217-226 (2013).
- Tarazona, M. P., Saiz, E. Combination of SEC/MALS experimental procedures and theoretical analysis for studying the solution properties of macromolecules. Journal of Biochemical and Biophysical Methods. 56 (1-3), 95-116 (2003).
- Folta-Stogniew, E. Oligomeric states of proteins determined by size-exclusion chromatography coupled with light scattering, absorbance, and refractive index detectors. Methods in Molecular Biology. 328, Clifton, N.J. 97-112 (2006).
- McMeekin, T. L., Wilensky, M., Groves, M. L. Refractive indices of proteins in relation to amino acid composition and specific volume. Biochemical and Biophysical Research Communications. 7 (2), 151-156 (1962).
- Atwal, R. S., et al. Huntingtin has a membrane association signal that can modulate huntingtin aggregation, nuclear entry and toxicity. Human Molecular Genetics. 16 (21), 2600-2615 (2007).
- Kegel-Gleason, K. B. Huntingtin interactions with membrane phospholipids: Strategic targets for therapeutic intervention. Journal of Huntington's Disease. 2 (3), 239-250 (2013).
- Michalek, M., Salnikov, E. S., Werten, S., Bechinger, B. Membrane interactions of the amphipathic amino terminus of huntingtin. Biochemistry. 52 (5), 847-858 (2013).
- Wittig, I., Braun, H. P., Schägger, H. Blue native PAGE. Nature Protocols. 1 (1), 418-428 (2006).
- Nissley, D. A., O'Brien, E. P. Altered co-translational processing plays a role in huntington's pathogenesis-A hypothesis. Frontiers in Molecular Neuroscience. 9, 54(2016).
- Kumar, N., Gammell, P., Clynes, M. Proliferation control strategies to improve productivity and survival during CHO based production culture: A summary of recent methods employed and the effects of proliferation control in product secreting CHO cell lines. Cytotechnology. 53 (1-3), 33-46 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon