Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Analyse der Wechselwirkung fluoreszierend markierter Proteine mit künstlichen Phospholipid-Mikrovesikeln mittels quantitativer Durchflusszytometrie
In diesem Artikel
Zusammenfassung
Hier beschreiben wir eine Reihe von Methoden zur Charakterisierung der Interaktion von Proteinen mit Membranen von Zellen oder Mikrovesikeln.
Zusammenfassung
Im menschlichen Körper verlaufen die meisten der wichtigsten physiologischen Reaktionen, die an der Immunantwort und der Blutgerinnung beteiligt sind, auf den Membranen der Zellen. Ein wichtiger erster Schritt in jeder membranabhängigen Reaktion ist die Bindung von Protein an die Phospholipidmembran. Ein Ansatz zur Untersuchung der Proteinwechselwirkung mit Lipidmembranen wurde unter Verwendung fluoreszierend markierter Proteine und Durchflusszytometrie entwickelt. Diese Methode ermöglicht die Untersuchung von Protein-Membran-Interaktionen mit lebenden Zellen und natürlichen oder künstlichen Phospholipid-Vesikeln. Der Vorteil dieser Methode ist die Einfachheit und Verfügbarkeit von Reagenzien und Geräten. Bei dieser Methode werden Proteine mit fluoreszierenden Farbstoffen markiert. Es können jedoch sowohl selbst hergestellte als auch kommerziell erhältliche, fluoreszierend markierte Proteine verwendet werden. Nach der Konjugation mit einem fluoreszierenden Farbstoff werden die Proteine mit einer Quelle der Phospholipidmembran (Mikrovesikel oder Zellen) inkubiert und die Proben mittels Durchflusszytometrie analysiert. Die erhaltenen Daten können zur Berechnung der kinetischen Konstanten und des Gleichgewichts Kd verwendet werden. Darüber hinaus ist es möglich, die ungefähre Anzahl der Proteinbindungsstellen auf der Phospholipidmembran mit speziellen Kalibrierperlen abzuschätzen.
Einleitung
Biomembranen trennen den inneren Inhalt tierischer Zellen und des extrazellulären Raums. Beachten Sie, dass Membranen auch Mikrovesikel umgeben, die während des Lebenszyklus der Zelle gebildet wurden, und Organellen. Die Zellmembran besteht überwiegend aus Lipiden und Proteinen. Membranproteine übernehmen Signal-, Struktur-, Transport- und Klebefunktionen. Die Lipiddoppelschicht ist aber auch essentiell für die Wechselbeziehung der tierischen Zelle mit dem extrazellulären Raum. Diese Arbeit schlägt eine Methode vor, um die periphere Wechselwirkung externer Proteine mit der Lipidmembran zu untersuchen.
Das auffälligste Beispiel für Reaktionen, die auf der äußeren Membranschicht einer tierischen Zelle auftreten, ist die Blutgerinnungsreaktion. Ein wichtiges Merkmal der Blutgerinnung ist, dass alle Hauptreaktionen auf den Phospholipidmembranen von Zellen und Mikrovesikeln ablaufen, die aus diesen Zellen entstehen, und nicht im Plasma 1,2,3. Membranabhängige Reaktionen umfassen den Prozess des Startens der Koagulation (auf den Zellmembranen des Subendothels, des entzündeten Endothels oder der aktivierten Immunzellen, unter Beteiligung eines Gewebefaktors), alle Reaktionen der Hauptkaskadenaktivierung der Faktoren IX, X, Prothrombin; Aktivierung von Faktor XI durch Thrombin (auf den Membranen von aktivierten Blutplättchen, Erythrozyten, Lipoproteinen und Mikrovesikeln); Reaktionen des Protein-C-Signalwegs; Inaktivierung von Gerinnungsenzymen (auf den Membranen von Endothelzellen unter Beteiligung von Thrombomodulin-Cofaktoren, Endothelprotein-C-Rezeptor, Heparansulfat); und Kontaktwegreaktionen (auf Membranen von Blutplättchen und einigen Mikrovesikeln unter Beteiligung unbekannter Cofaktoren). Daher ist es unmöglich, die Blutgerinnung zu untersuchen, ohne die Wechselwirkung verschiedener Plasmaproteine mit der Membran von Blutzellen zu untersuchen.
Dieser Beitrag beschreibt eine auf Durchflusszytometrie basierende Methode zur Charakterisierung der Interaktion von Proteinen mit Lipidmembranen von Zellen oder Mikrovesikeln. Dieser Ansatz wurde ursprünglich vorgeschlagen, um die Wechselwirkung von Blutplasma mit Blutplättchen und künstlichen Phospholipidvesikeln zu untersuchen. Darüber hinaus interagieren die meisten der untersuchten Proteine direkt mit negativ geladenen Membranphospholipiden, insbesondere mit Phosphatidylserin 4,5. Hinzu kommen Proteine, deren Interaktion mit der Membran durch spezielle Rezeptoren vermitteltwird 6.
Eine wichtige Fähigkeit der Durchflusszytometrie ist die Unterscheidung zwischen freien und gebundenen Liganden ohne zusätzliche Trennung. Diese Eigenschaft der Zytometrie ermöglicht die Untersuchung der Ligandengleichgewichtsbindung am Endpunkt und hilft bei der Durchführung kontinuierlicher kinetischer Messungen. Die Technik ist unausgereift und erfordert keine komplexe Probenvorbereitung. Die Durchflusszytometrie wird aktiv eingesetzt, um die Dynamik der Wechselwirkung zwischen fluoreszierenden Peptiden, Rezeptoren und G-Proteinen in intakten und waschmitteldurchlässigen Neutrophilenquantitativ zu untersuchen 7. Dieser Ansatz eignet sich auch für die Erforschung von Protein-DNA-Interaktionen und der Kinetik der Endonukleaseaktivitätin Echtzeit 8. Im Laufe der Zeit wurde diese Methode verwendet, um hochaffine Protein-Protein-Interaktionen mit gereinigten Lipidvesikeln9 oder, allgemeiner, mit Membranproteinen, die in einem hocheffizienten Sf9-Zellexpressionssystem10 exprimiert werden, quantitativ zu untersuchen. Quantitative Methoden wurden auch zur Charakterisierung von Protein-Liposom-Interaktionen mittels Durchflusszytometrie für Transmembranproteinebeschrieben 11.
Diese Technik verwendet selbst hergestellte Kalibrierperlen, um die Verwendung von kommerziell erhältlichen Perlenzu vermeiden 7. Die zuvorverwendeten Kalibrierperlen 7 sollten mit Fluorescein arbeiten, was das Sortiment an zugänglichen fluoreszierenden Liganden auf den Proteinen erheblich einschränkte. Darüber hinaus bietet dieses Dokument eine neue Möglichkeit, kinetische Daten für eine angemessene Zeitauflösung zu erfassen und zu analysieren. Obwohl diese Methode für künstliche Phospholipidvesikel beschrieben wird, gibt es keine offensichtlichen Einschränkungen für ihre Anpassungsfähigkeit an Zellen, natürliche Vesikel oder künstliche Phospholipidvesikel mit einer anderen Lipidzusammensetzung. Das hierin beschriebene Verfahren ermöglicht die Abschätzung der Parameter Wechselwirkung (kon, koff) und Gleichgewicht (Kd) und erleichtert die quantitative Charakterisierung der Anzahl der Proteinbindungsstellen auf der Membran. Beachten Sie, dass diese Technik eine ungefähre Schätzung der Anzahl der Bindungsstellen liefert. Die Vorteile der Methode sind ihre relative Einfachheit, Zugänglichkeit und Anpassungsfähigkeit an native Zellen und natürliche und künstliche Mikrovesikel.
Protokoll
1. Markierung fluoreszierender Proteine
- Materialaufbereitung
- Bereiten Sie 1 M Natriumbicarbonatpuffer mit einem pH-Wert von 9,0 vor, lagern Sie ihn bei 4 °C und verwenden Sie ihn innerhalb einer Woche.
- Bereiten Sie 1,5 m Hydroxylaminhydrochloridpuffer, pH 8,5, unmittelbar vor Gebrauch vor.
- Bereiten Sie eine 10 mg / ml Lösung mit Fluoreszenzfarbstoff (siehe Materialtabelle) in Dimethylsulfoxid vor.
HINWEIS: Diese Lösung kann einen Monat lang bei -20 °C im Dunkeln gelagert werden. - Bereiten Sie Lösungen von gereinigten Antikörpern oder anderen Proteinen bei 1-10 mg / ml vor.
HINWEIS: Vermeiden Sie Puffer, die Ammoniumionen oder primäre Amine enthalten. Ersetzen Sie die Puffer, die Tris oder Glycerin enthalten, durch phosphatgepufferte Kochsalzlösung (PBS) durch Dialyse. Weder Natriumazid (≤3 mM) noch Thimerosal (≤1 mM) beeinflussen die Konjugationsreaktion signifikant. - Inkubieren Sie das Gelfiltrationsmedium (siehe Materialtabelle) zur Proteinreinigung in PBS über Nacht bei Raumtemperatur oder für 2 h bei 60 °C. Tragen Sie das Gelfiltrationsmedium auf Spinsäulen mit 0,2 μm-Membranen auf.
- Berechnen Sie die Menge an reaktivem Farbstoff, die für jede Reaktion verwendet werden soll, entsprechend der Konzentration des zu markierenden Proteins unter Verwendung von Eq (1).
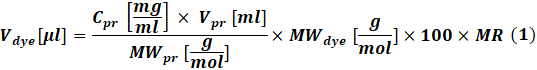
wobeiV-Farbstoff das Volumen der Farbstoffmateriallösung ist; C pr, V pr und MWpr ist die Konzentration, das Volumen und das Molgewicht des Proteins; MW-Farbstoff ist das molare Gewicht des Farbstoffs; 100 ist ein Umrechnungsfaktor für Einheiten; MR ist das molare Verhältnis von Farbstoff zu Protein in der Reaktionsmischung.
HINWEIS: Die folgenden MRs werden für IgG-Markierungsreaktionen empfohlen: MR = 40, wenn der Antikörper bei 1-3 mg/ml liegt oder MR = 30, wenn der Antikörper bei 4-10 mg/ml liegt. Für Gerinnungsfaktoren wird in der Regel MR = 5 verwendet. - Konjugationsreaktion
- Mischen Sie in einem Reaktionsrohr die Proteinlösung mit einem 10x geringeren Volumen von 1 M Bicarbonatlösung.
- Die erforderliche Menge an fluoreszierendem Farbstoff (siehe Schritt 1.2) wird unter kontinuierlichem Rühren zugegeben.
- Inkubieren Sie das Reaktionsgemisch bei Raumtemperatur für ca. 1 h, lichtgeschützt und unter kontinuierlichem Rühren.
- Für jeweils 200 μL Proteinlösung werden 5 μL 1,5 M Hydroxylaminhydrochlorid zugegeben.
- Das Reaktionsgemisch bei Raumtemperatur ca. 30 min lichtgeschützt und unter kontinuierlichem Rühren inkubieren.
- Bereiten Sie die Spinspalte vor.
- Geben Sie 500 μL Gelfiltrationsmedium zur Proteinreinigung zu einer Säule. Zentrifen Sie die Säule für 3 min bei 1.000 × g.
- Entsorgen Sie den Puffer aus dem Auffangrohr. Wenn die Säule nicht voll ist, fügen Sie mehr Gelfiltrationsmedium hinzu und zentrifieren Sie die Säule für 3 Minuten bei 1.000 × g. Wiederholen Sie diesen Schritt, bis die Spalte voll ist.
- Reinigung
- Das Reaktionsgemisch (aus Schritt 1.3.5) wird 5 min lang bei 17.000 × g zentrifugiert und der Niederschlag entfernt.
- Überstand mit Gelfiltrationsmedium auf die Schleuderkolonne übertragen. Lassen Sie die Lösung in das Gelbett einziehen.
- Verwenden Sie ein leeres Auffangrohr für die Schleudersäule und zentrifen Sie es für 5 min bei 1.000 × g. Nach der Zentrifugation das markierte Protein aus dem Auffangröhrchen entnehmen.
- Bestimmung des Kennzeichnungsgrades
- Korrekt für den Beitrag des Farbstoffs zur Absorption bei A 280 durch Messung der Absorption von freiem Farbstoff bei 280 nm (A280) und der λ max für den Farbstoff (Amax) (siehe Eq (2)).
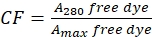 (2)
(2) - Messen Sie die Absorption des Protein-Farbstoff-Konjugats bei 280 nm (A 280) und dem λ max für den Farbstoff (Amax) und berechnen Sie die Proteinkonzetration mit Eq (3).
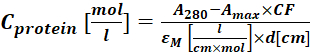 (3)
(3)
wobei εM der molare Extinktionskoeffizient des Proteins bei 280 nm ist; d ist die optische Weglänge während der Messung der Absorption; CF ist der Beitrag des Farbstoffs zur Absorption bei A280 (Schritt 1.6.1). - Berechnen Sie den Grad der Beschriftung (DOL) mit Eq (4).
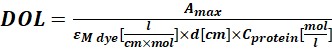 (4)
(4)
wobei ε M-Farbstoff der molare Extinktionskoeffizient des Farbstoffs bei λmax nm ist; d ist die optische Weglänge während der Messung der Absorption; C-Protein ist die Konzentration des Proteins (Schritt 1.6.2).
- Korrekt für den Beitrag des Farbstoffs zur Absorption bei A 280 durch Messung der Absorption von freiem Farbstoff bei 280 nm (A280) und der λ max für den Farbstoff (Amax) (siehe Eq (2)).
2. Herstellung von Phospholipid-Vesikeln
- Aufbereitung und Speicherung der Lipidmischung
- Kombinieren Sie die Lipide im entsprechenden Verhältnis (Phosphatidylserin/Phosphatidylcholin im Verhältnis 20 Mol-% zu 80 Mol-%).
- Trocknen Sie die Lipidmischung nach der Lyophilisation oder Verdampfung und lagern Sie sie unter einer inerten Atmosphäre in Glasampullen.
- Herstellung von Lipidfolien
- Öffnen Sie die Ampulle und resuspendieren Sie die Lipidmischung in einer kleinen Menge (~ 100 μL) Chloroform.
HINWEIS: Verwenden Sie nicht zu viel Chloroform, da es nicht vollständig verdunstet. - DiIC16(3) in Ethanol bei 0,2 mol% zugeben. Die Lipidmischung in einen runden Bodenkolben geben. Verteilen Sie die Mischung dünn über die Seiten des Kolbens, indem Sie sie drehen. Trocknen Sie die Lipidmischung 30 min unter einem Argonstrom.
- Öffnen Sie die Ampulle und resuspendieren Sie die Lipidmischung in einer kleinen Menge (~ 100 μL) Chloroform.
- Hydratation der Lipidmischung
- Einen entsprechend warmen (~55 °C) wässrigen Puffer (HEPES 20 mM, NaCl 140 mM, pH 7,4) in einem der erwarteten Lipidkonzentration entsprechenden Volumen in den Kolben mit dem Lipidfilm geben. Inkubieren Sie die Mischung mit Vortexing bei 55 ° C für 30 min für eine vollständige Hydratation.
- Legen Sie das Probenröhrchen in einen Gefrierschrank oder warmen Thermostat, damit die Lipidsuspension 3-5 Frost-Tau-Zyklen durchläuft.
- Bildung von Lipidvesikeln durch Extrusion
- Bereiten Sie den Extruder gemäß den Anweisungen des Herstellers vor. Alle Extruderkomponenten auf die Phasenübergangstemperatur des Lipidgemisches erwärmen.
- Füllen Sie eine der Extruderspritzen mit der hydratisierten Lipidmischung. Warten Sie 5-10 Minuten, bis sich die Temperatur der Lipidsuspension mit der Temperatur des Extruders ausgeglichen hat.
- Extrudieren Sie die Lipidmischung mindestens 10 Mal durch die Membran. Für die endgültige Extrusion legen Sie die Lipidsuspension in die alternative Spritze und achten Sie auf eine Veränderung des Aussehens von einer leicht nebulösen zu einer klaren Lösung.
- Lagern Sie die resultierende Mischung von Lipidvesikeln bei 4 °C, vorzugsweise in einer inerten Atmosphäre aus Argon oder Stickstoff, für 3-4 Tage. Nicht einfrieren.
3. Isolierung von Blutplättchen aus Vollblut
- Sammeln Sie Vollblut von gesunden Spendern in Tuben, die 3,2% Natriumcitrat enthalten.
- Prostaglandin E1 (PGE1) (1 μM) und Apyrase (0,1 U/ml) in das Blut geben, gefolgt von einer Zentrifugation bei Raumtemperatur bei 100 × g für 8 min.
- Nach der Zentrifugation nehmen Sie das plättchenreiche Plasma und fügen Sie Natriumcitratlösung (106 mM, pH 5,5) zu einem Plasma/Citrat-Verhältnis von 3:1 hinzu. Das Plasma bei Raumtemperatur bei 400 × g für 5 min zentrifugieren.
- Entfernen Sie den Überstand und suspendieren Sie das Pellet in 300 μL Tyrodes Puffer ohne BSA (20 mM HEPES, 150 mM NaCl, 2,7 mM KCl, 1 mMMgCl 2, 0,4 mM NaH 2 PO 4, 2,5 mM CaCl2, 5 mM Glukose, pH7,4). Reinigen Sie die Blutplättchen von Plasmaproteinen durch Gelchromatographie auf dem Gelfiltrationsmedium zur Thrombozytenreinigung (siehe Materialtabelle).
4. Nachweis von Protein-Lipid-Wechselwirkung durch Durchflusszytometrie
- Kinetische Bindungsexperimente
- Verdünnte Phospholipidvesikel (ab Schritt 2.4.4) im Tyrode-Puffer (20 mM HEPES, 150 mM NaCl, 2,7 mM KCl, 1 mM MgCl 2, 0,4 mM NaH 2 PO 4, 2,5 mM CaCl2, 5 mMGlukose, 0,5% BSA, pH7,4) auf eine Konzentration von 1 μM und ein Gesamtvolumen von 250 μL.
- Fluoreszenzmarkierter Gerinnungsfaktor X (fX-fd) aus Schritt 1 bei einer Konzentration von 500 nM mit den Phospholipidvesikeln aus Schritt 4.1.1 im Verhältnis 1:1 (Endvesikelkonzentration 0,5 μM, fX-fd-Konzentration beträgt 250 nM fX) auf ein Gesamtvolumen von 500 μL mischen.
- Injizieren Sie sofort die 500 μL der gemischten Suspension (~ 20 min für die Analyse mit einer niedrigen Fließrate) in das Durchflusszytometer. Verwenden Sie eine niedrige Durchflussrate und stellen Sie sicher, dass der Schwellenwert für Kanal FL2 (Ex-488 nm, Emissionsfilter 585/42 nm) als Wert 200 beträgt. Messen Sie die mittlere Fluoreszenz im Kanal FL4 (exсitation 633 nm, Emissionsfilter 660/20) für den Fluoreszenzfarbstoff aus der Materialtabelle.
HINWEIS: Wählen Sie ein Zytometer ohne Autosampler. Dies beschleunigt den Prozess der Injektion der Probe in die Messzelle. - Wenn eine Sättigung der Bindung erreicht ist (kein signifikanter Anstieg der Fluoreszenz innerhalb von 5 min), verdünnen Sie die Probe schnell 20-fach mit dem Puffer von Tyrode und überwachen Sie die Dissoziation, bis die Basisfluoreszenz erreicht ist (vollständige Dissoziation) oder bis ein Plateau erreicht ist (keine signifikante Abnahme der Fluoreszenz innerhalb von 5 min).
HINWEIS: Fügen Sie als Kontrolle 10 μM EDTA hinzu und überwachen Sie die vollständige Dissoziation für 5 Minuten.
- Gleichgewichtsbindungsexperimente
HINWEIS: Verwenden Sie die kinetische Bindungskurve, um die Zeit bis zum Erreichen der Sättigung zu bestimmen. die Sättigungszeit für fX-fd und künstliche Vesikel beträgt 20 min.- Inkubieren Sie künstliche Phospholipidvesikel (5 μM) für den Bindungsassay mit unterschiedlichen Konzentrationen von fX-fd (von 0 bis 1.000 nM) im Tyrode-Puffer für 20 min.
- Verdünnen Sie jede Probe von Schritt 4.2.1 um das 20-fache auf ein Endvolumen von 200 μL mit dem Puffer von Tyrode. Analysieren Sie die verdünnte Probe sofort durch Durchflusszytometrie innerhalb von 30 s. Verwenden Sie die Einstellungen aus Schritt 4.1.3.
HINWEIS: Verwenden Sie als Kontrolle für unspezifische Bindungen ähnliche Proben mit EDTA (10 μM) und inkubieren Sie sie für 5 min.
5. Analyse von Durchflusszytometriedaten
- Exportieren Sie Experimente im FSC-Format aus der Zytometrie-Datenerfassungssoftware zur Datenanalyse in die Zytometer-Software (siehe Materialtabelle). Wählen Sie Datei | | exportieren FCS-Dateien. Öffnen Sie FSC-Dateien in der Zytometersoftware zur Datenanalyse, indem Sie die Dateien auf dem Computer auswählen und in den Arbeitsbereich des Programms ziehen.
- Für das Gating der Mikrovesikel identifizieren Sie den Bereich der Mikrovesikel durch die Fluoreszenz des lipophilen Farbstoffs DiIC16 (3). Verwenden Sie Menübefehle oder die Schaltfläche "Plot" im Arbeitsblatt, um Punktplot-SSC aus FL2 (Farbstoff DilC16(3)) in Protokollkoordinaten zu erstellen. Klicken Sie auf die Schaltfläche Rechteckiges Tor, um einen Gating-Bereich zu zeichnen, sodass Ereignisse aus einer Probe ohne Vesikel nicht in diesen Bereich einbezogen werden (Abbildung 1B,C).
- Analysieren Sie die kinetischen Experimente.
- Erstellen Sie ein Punktdiagramm mit den Koordinaten der Fluoreszenz (FL4) im Laufe der Zeit für den Bereich der Vesikel (Doppelklicken Sie in Schritt 5.2 auf den Bereich der Vesikel)
- Exportieren Sie die Daten über die Änderung der Fluoreszenz im Laufe der Zeit im CSV-Format. Wählen Sie Beispiel| Klicken Sie mit der rechten Maustaste auf | | exportieren Wählen Sie FL4 und Zeit in Parameter | Verzeichnis zum Speichern | auswählen CSV-Format | auswählen Ausfuhr.
- Öffnen Sie die CSV-Datei in einer beliebigen Statistiksoftware (siehe Materialtabelle). Berechnen Sie einen einfachen gleitenden Durchschnitt der Fluoreszenz und Zeit für jeweils 1.000 Ereignisse.
- Nähern Sie sich einem Diagramm der Abhängigkeit der einfachen gleitenden durchschnittlichen Fluoreszenz von der Zeit unter der Annahme der exponentiellen Abhängigkeit (Analysis > Fitting >Nonlinear Curve Fit) und verwenden Sie dieses, um die kinetische Assoziationskonstante mit Eq (5) zu berechnen.
 (5)
(5)
wobei [XB] die gebundene Faktorkonzentration zu jedem Zeitpunkt (benutzerdefinierte Einheiten) gemäß dem einfachen gleitenden Durchschnitt aus Schritt 5.3.3 ist; [X] ist die addierte Faktorkonzentration; [X]max ist die maximale gebundene Faktorkonzentration; k ist die Assoziationskonstante; t ist die Zeit. - Wiederholen Sie die gleichen Aktionen (5.3.1-5.3.4), um die kinetische Dissoziationskonstante mit Eq (6) zu berechnen.
 (6)
(6)
wobei [XB] die gebundene Faktorkonzentration zu jedem Zeitpunkt ist; [X]0 ist die gebundene Faktorkonzentration im Anfangsmoment der Zeit; k ist die Dissoziationskonstante; t ist die Zeit.
- Gleichgewichtsbindungs-Assay
- Bestimmen Sie die mittlere Fluoreszenz von fX-fd im Bereich der Vesikel für jede ausgewählte Konzentration von fX-fd.
- Nähert sich der Abhängigkeit der gebundenen Faktorfluoreszenz von der Konzentration des hinzugefügten Faktors unter der Annahme einer einfachen Einzelplatzbindung an. Berechnen Sie die durchschnittlichen Bindungsparameter mit Eq (7) aus mindestens drei unabhängigen Wiederholungen.
 (7)
(7)
wobei [XB] die gebundene Faktorkonzentration ist; [X] ist die addierte Faktorkonzentration; nx ist die scheinbare Anzahl der Bindungsstellen pro Vesikel; Kd ist die scheinbare Dissoziationskonstante.
6. Umrechnung der Fluoreszenzintensität in die mittlere Anzahl der Bindungsstellen
- Bereiten Sie kalibrierte Perlen vor.
- Inkubieren Sie gelfiltrierte Blutplättchen (siehe Schritt 3.3) mit A23187 (10 μM) in Gegenwart von CaCl 2 (2,5 mM) für 10 min bei Raumtemperatur.
- Zu den aktivierten Blutplättchen kommen die verschiedenen Konzentrationen von fX-fd (0 bis 1.000 nM). 2% v/v Formaldehyd hinzufügen und 1 h inkubieren. Stoppen Sie die Reaktion, indem Sie die Blutplättchen mit 3 M Glycin und 5% BSA für 30 min bei Raumtemperatur inkubieren.
- Reinigen Sie die Mischung von dem nicht umgesetzten Farbstoff. Zentrifugieren Sie die Blutplättchen für 5 min bei 400 × g, entfernen Sie den Überstand und suspendieren Sie das Pellet im Puffer von Tyrode (mit 0,5% BSA).
HINWEIS: Wiederholen Sie Schritt 6.1.3 dreimal.
- Messen Sie den Fluoreszenzpegel der Kalibrierkügelchen in jeder Probe zuerst mit einem Spektrofluorometer (für Fluoreszenzfarbstoff aus der Materialtabelle, Anregung 633 nm, Emission 670 nm) und dann mit dem Durchflusszytometer (in Kanal FL4: Anregung 633 nm, Emissionsfilter 660/20). Bestimmen Sie mithilfe eines Zellzählers die Anzahl der Perlen in jeder Probe.
- Wandeln Sie die Fluoreszenzintensität der jeweiligen Perlenprobe mit einem Spektrofluorometer in die Konzentration des löslichen Fluoreszenzfarbstoffs um. Berechnen Sie die Fluoreszenzfarbstoffkonzentration für die Anzahl der Fluorophormoleküle mit Eq (8) neu.
 (8)
(8)
wobei Nx die Anzahl der Fluorophormoleküle ist; C ist die Fluoreszenzfarbstoffkonzentration; NA ist die Avogadro-Konstante; NA = 6,02214076×1023 mol -1. - Erstellen Sie ein Abhängigkeitsdiagramm der durchschnittlichen Fluoreszenz der Kügelchen in einem Durchflusszytometer (Schritt 6.2) über die Anzahl der Fluorophormoleküle (siehe Schritt 6.3) für jede Probe mit einer beliebigen statistischen Software (siehe Materialtabelle). Approximieren Sie diese Abhängigkeit durch die Linienproportionalität (Analyse | Anpassung | Linear anpassen). Berechnen Sie aus der Näherung in Eq (9) den Umrechnungsfaktor der mittleren Fluoreszenz zu Bindungsstellen.
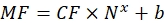 (9)
(9)
wobei MF die mittlere Fluoreszenz von Perlen durch Durchflusszytometrie ist; Nx ist die Anzahl der Fluorophormoleküle pro Perle; CF stellt den Umrechnungsfaktor der mittleren Fluoreszenz zu Bindungsstellen dar. CF und b werden aus den Ergebnissen der Anpassung des Graphen durch lineare Proportionalität erhalten. - Berechnen Sie die scheinbare Anzahl der Bindungsstellen pro Zinsvesikel mit Eq (10).
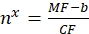 (10)
(10)
wobei nx die scheinbare Anzahl der Bindungsstellen pro Vesikel von Interesse ist; MF ist die mittlere Fluoreszenz der interessierenden Vesikel durch Durchflusszytometrie; CF und b sind Umrechnungsfaktoren aus dem Eq (8).
Ergebnisse
Das hierin beschriebene Durchflusszytometrieverfahren wird verwendet, um die Bindung von Plasmagerinnungsproteinen an aktivierte Blutplättchen zu charakterisieren. Zusätzlich wurden Phospholipidvesikel PS:PC 20:80 als Modellsystem eingesetzt. Diese Arbeit konzentriert sich hauptsächlich auf künstliche Phospholipidvesikel als Beispiel. Die Parameter des Zytometers, insbesondere die Photomultiplierröhrenspannung (PMT) und die Kompensation müssen für jedes spezifische Gerät, den Untersuchungsgegenstand (Zellen, kün...
Diskussion
Die vorgeschlagene Methode kann für eine grobe Charakterisierung der Wechselwirkung von Proteinen mit Phospholipidmembranen aus verschiedenen Quellen und Zusammensetzungen angepasst werden. Die hier beschriebene quantitative Durchflusszytometrie räumt der Oberflächenplasmonenresonanz (SPR) in mehreren Parametern zu. Insbesondere hat es eine geringere Empfindlichkeit und Zeitauflösung und erfordert eine fluoreszierende Markierung von Proteinen. Die fluoreszierende Markierung kann bei vielen Proteinen zu einer Verände...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Autoren wurden durch ein Stipendium der Russian Science Foundation 20-74-00133 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| A23187 | Sigma Aldrich | C7522-10MG | |
| Alexa Fluor 647 NHS Ester (Succinimidyl Ester) | Thermo Fisher Scientific | A37573 | fluorescent dye |
| Apyrase from potatoes | Sigma Aldrich | A2230 | |
| BD FACSCantoII | BD Bioscience | ||
| bovine serum albumin | VWR Life Science AMRESCO | Am-O332-0.1 | |
| Calcium chloride, anhydrous, powder, ≥97% | Sigma Aldrich | C4901-100G | |
| Cary Eclipse Fluorescence Spectrometer | Agilent | ||
| D-(+)-Glucose | Sigma Aldrich | G7528-1KG | |
| DiIC16(3) (1,1'-Dihexadecyl-3,3,3',3'-Tetramethylindocarbocyanine Perchlorate) | Thermo Fisher Scientific | D384 | |
| DMSO | Sigma Aldrich | D8418 | |
| EDTA disodium salt | VWR Life Science AMRESCO | Am-O105B-0.1 | |
| FACSDiva | BD Bioscience | cytometry data acquisition software | |
| FlowJo | Tree Star | cytometer software for data analysis | |
| HEPES | Sigma Aldrich | H4034-500G | |
| Human Factor X | Enzyme research | HFX 1010 | |
| Hydroxylamine hydrochloride | Panreac | 141914.1209 | |
| L-α-phosphatidylcholine (Brain, Porcine) | Avanti Polar Lipids | 840053P | |
| L-α-phosphatidylserine (Brain, Porcine) (sodium salt) | Avanti Polar Lipids | 840032P | |
| Magnesium chloride | Sigma Aldrich | M8266-100G | |
| Mini-Extruder | Avanti Polar Lipids | 610020-1EA | |
| OriginPro 8 SR4 v8.0951 | OriginLab Corporation | Statistical software | |
| Phosphate Buffered Saline (PBS) Tablets, Biotechnology Grade | VWR Life Science AMRESCO | 97062-732 | |
| Potassium chloride | Sigma Aldrich | P9541-500G | |
| Prostaglandin E1 | Cayman Chemical | 13010 | |
| Sephadex G25 | GE Healthcare | GE17-0033-01 | gel filtration medium for protein purification |
| Sepharose CL-2B | Sigma Aldrich | CL2B300-500ML | gel filtration medium for platelet purification |
| Sodium bicarbonate | Corning | 61-065-RO | |
| Sodium chloride | Sigma Aldrich | S3014-500G | |
| Sodium phosphate monobasic | Sigma Aldrich | S3139-250G | |
| Spin collumns with membrane 0.2 µm | Sartorius | VS0171 | |
| Trisodium citrate dihydrate | Sigma Aldrich | S1804-1KG |
Referenzen
- Hoffman, M., Monroe, D. M. A cell-based model of hemostasis. Thrombosis and haemostasis. 85 (6), 958-965 (2001).
- Roberts, H. R., Hoffman, M., Monroe, D. M. A cell-based model of thrombin generation. Seminars in Thrombosis and Hemostasis. 32, 32-38 (2006).
- Panteleev, M. A., Dashkevich, N. M., Ataullakhanov, F. I. Hemostasis and thrombosis beyond biochemistry: roles of geometry, flow and diffusion. Thrombosis Research. 136 (4), 699-711 (2015).
- Podoplelova, N. A., et al. Hysteresis-like binding of coagulation factors X/Xa to procoagulant activated platelets and phospholipids results from multistep association and membrane-dependent multimerization. Biochimica et Biophysica Acta. 1858 (6), 1216-1227 (2016).
- Panteleev, M. A., Ananyeva, N. M., Greco, N. J., Ataullakhanov, F. I., Saenko, E. L. Two subpopulations of thrombin-activated platelets differ in their binding of the components of the intrinsic factor X-activating complex. Journal of Thrombosis and Haemostasis. 3 (11), 2545-2553 (2005).
- Kotova, Y., et al. Binding of coagulation factor XIII zymogen to activated platelet subpopulations: roles of integrin αIIbβ3 and fibrinogen. Thrombosis and Haemostasis. 119 (6), 906-915 (2019).
- Fay, S. P., Posner, R. G., Swann, W. N., Sklar, L. A. Real-time analysis of the assembly of ligand, receptor, and G protein by quantitative fluorescence flow cytometry. Biochemistry. 30 (20), 5066-5075 (2002).
- Nolan, J. P., Shen, B., Park, M. S., Sklar, L. A. Kinetic analysis of human flap endonuclease-1 by flow cytometry. Biochemistry. 35 (36), 11668-11676 (1996).
- Sarvazyan, N. A., Lim, W. K., Neubig, R. R. Fluorescence analysis of receptor−G protein interactions in cell membranes. Biochemistry. 41 (42), 12858-12867 (2002).
- Sarvazyan, N. A., Neubig, R. R. Analysis of molecular assemblies by flow cytometry: determinants of Gi1 and by binding. Advances in Optical Biophysics. 3256, 122-131 (1998).
- De Franceschi, N., et al. ProLIF - Quantitative integrin protein-protein interactions and synergistic membrane effects on proteoliposomes. Journal of Cell Science. 132 (4), (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten