Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse de l’interaction des protéines marquées par fluorescence avec des microvésicules de phospholipides artificielles à l’aide de la cytométrie quantitative en flux
Dans cet article
Résumé
Ici, nous décrivons un ensemble de méthodes pour caractériser l’interaction des protéines avec les membranes de cellules ou de microvésicules.
Résumé
Dans le corps humain, la plupart des principales réactions physiologiques impliquées dans la réponse immunitaire et la coagulation sanguine se déroulent sur les membranes des cellules. Une première étape importante dans toute réaction dépendante de la membrane est la liaison des protéines sur la membrane des phospholipides. Une approche pour étudier l’interaction des protéines avec les membranes lipidiques a été développée en utilisant des protéines marquées par fluorescence et la cytométrie en flux. Cette méthode permet d’étudier les interactions protéine-membrane à l’aide de cellules vivantes et de vésicules phospholipides naturelles ou artificielles. L’avantage de cette méthode est la simplicité et la disponibilité des réactifs et des équipements. Dans cette méthode, les protéines sont marquées à l’aide de colorants fluorescents. Cependant, des protéines auto-fabriquées et disponibles dans le commerce, marquées par fluorescence, peuvent être utilisées. Après conjugaison avec un colorant fluorescent, les protéines sont incubées avec une source de la membrane phospholipidique (microvésicules ou cellules), et les échantillons sont analysés par cytométrie en flux. Les données obtenues peuvent être utilisées pour calculer les constantes cinétiques et l’équilibre Kd. En outre, il est possible d’estimer le nombre approximatif de sites de liaison aux protéines sur la membrane phospholipidique à l’aide de billes d’étalonnage spéciales.
Introduction
Les biomembranes séparent le contenu interne des cellules animales et de l’espace extracellulaire. Notez que les membranes entourent également les microvésicules formées au cours du cycle de vie de la cellule et les organites. La membrane cellulaire est principalement composée de lipides et de protéines. Les protéines membranaires remplissent des fonctions de signalisation, structurelles, de transport et d’adhésif. Cependant, la bicouche lipidique est également essentielle pour l’interrelation de la cellule animale avec l’espace extracellulaire. Cet article propose une méthode pour étudier l’interaction périphérique des protéines externes avec la membrane lipidique.
L’exemple le plus frappant de réactions se produisant sur la couche membranaire externe d’une cellule animale est la réaction de coagulation sanguine. Une caractéristique importante de la coagulation sanguine est que toutes les réactions principales se déroulent sur les membranes phospholipides des cellules et des microvésicules provenant de ces cellules et non dans le plasma 1,2,3. Les réactions membranaires dépendantes comprennent le processus de début de la coagulation (sur les membranes cellulaires du sous-endothhélium, l’endothélium enflammé ou les cellules immunitaires activées, avec la participation d’un facteur tissulaire), toutes les réactions de la principale activation en cascade des facteurs IX, X, prothrombine; activation du facteur XI par la thrombine (sur les membranes des plaquettes activées, des érythrocytes, des lipoprotéines et des microvésicules); réactions de la voie de la protéine C; inactivation des enzymes de coagulation (sur les membranes des cellules endothéliales avec la participation de cofacteurs de thrombomoduline, récepteur de la protéine endothéliale C, sulfate d’héparane); et les réactions de la voie de contact (sur les membranes des plaquettes et de certaines microvésicules avec la participation de cofacteurs inconnus). Ainsi, il est impossible d’étudier la coagulation sanguine sans étudier l’interaction de diverses protéines plasmatiques avec la membrane des cellules sanguines.
Cet article décrit une méthode basée sur la cytométrie en flux pour caractériser l’interaction des protéines avec les membranes lipidiques des cellules ou des microvésicules. Cette approche a été initialement proposée pour étudier l’interaction du plasma sanguin avec les plaquettes et les vésicules phospholipides artificielles. De plus, la plupart des protéines étudiées interagissent directement avec les phospholipides membranaires chargés négativement, en particulier avec la phosphatidylsérine 4,5. De plus, il existe des protéines dont l’interaction avec la membrane est médiée par des récepteurs spéciaux6.
Une capacité importante de la cytométrie en flux est la discrimination entre les ligands libres et liés sans séparation supplémentaire. Cette caractéristique de la cytométrie permet l’étude de la liaison à l’équilibre des ligands à l’extrémité et aide à effectuer des mesures cinétiques continues. La technique n’est pas sophistiquée et ne nécessite pas de préparation d’échantillon complexe. La cytométrie en flux est activement utilisée pour étudier quantitativement la dynamique d’interaction entre les peptides fluorescents, les récepteurs et les protéines G dans les neutrophiles intacts et perméables aux détergents7. Cette approche est également applicable pour explorer les interactions protéine-ADN et la cinétique de l’activité des endonucléases en temps réel8. Au fil du temps, cette méthode a été utilisée pour étudier quantitativement les interactions protéine-protéine à haute affinité avec les vésicules lipidiques purifiées9, ou, plus généralement, avec les protéines membranaires exprimées dans un système d’expression cellulaire Sf9 très efficace10. Des méthodes quantitatives ont également été décrites pour caractériser les interactions protéine-liposome en utilisant la cytométrie en flux pour les protéines transmembranaires11.
Cette technique utilise des perles d’étalonnage auto-fabriquées pour éviter d’utiliser des perles disponibles dans le commerce7. Les billes d’étalonnage utilisées précédemment7 étaient destinées à fonctionner avec la fluorescéine, ce qui limitait considérablement l’assortiment de ligands fluorescents accessibles sur les protéines. En outre, cet article offre une nouvelle façon d’acquérir et d’analyser des données cinétiques pour une résolution temporelle raisonnable. Bien que cette méthode soit décrite pour les vésicules phospholipides artificielles, il n’y a pas de limites évidentes à son adaptabilité aux cellules, aux vésicules naturelles ou aux vésicules phospholipides artificielles avec une composition lipidique différente. La méthode décrite ici permet d’estimer les paramètres d’interaction (kon, koff) etd’équilibre (K d) et facilite la caractérisation quantitative du nombre de sites de liaison aux protéines sur la membrane. Notez que cette technique fournit une estimation approximative du nombre de sites de liaison. Les avantages de la méthode sont sa simplicité relative, son accessibilité et son adaptabilité aux cellules natives et aux microvésicules naturelles et artificielles.
Protocole
1. Marquage des protéines fluorescentes
- Préparation du matériel
- Préparez 1 M de tampon de bicarbonate de sodium, pH 9,0, conservez-le à 4 °C et utilisez-le dans un délai d’une semaine.
- Préparer un tampon de chlorhydrate d’hydroxylamine de 1,5 M, pH 8,5, immédiatement avant utilisation.
- Préparer une solution de colorant fluorescent à 10 mg/mL (voir la Table des matériaux) dans du sulfure de diméthyle.
REMARQUE: Cette solution peut être conservée pendant un mois à -20 ° C dans l’obscurité. - Préparer des solutions d’anticorps purifiés ou d’autres protéines à 1-10 mg / mL.
REMARQUE: Évitez les tampons contenant des ions ammonium ou des amines primaires. Remplacer les tampons contenant du Tris ou du glycérol par une solution saline tamponnée au phosphate (PBS) par dialyse. Ni l’azoture de sodium (≤3 mM) ni le thimérosal (≤1 mM) n’affecteront de manière significative la réaction de conjugaison. - Incuber le milieu de filtration du gel (voir le tableau des matériaux) pour la purification des protéines dans le PBS pendant la nuit à température ambiante ou pendant 2 h à 60 °C. Appliquer le milieu de filtration du gel sur des colonnes de spin avec des membranes de 0,2 μm.
- Calculer la quantité de colorant réactif à utiliser pour chaque réaction en fonction de la concentration de protéine à marquer à l’aide de Eq (1).
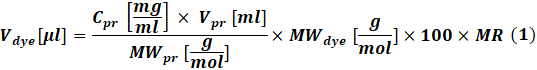
Où lecolorant V est le volume de la solution mère de colorant; Cpr, Vpr et MWpr sont la concentration, le volume et le poids molaire de la protéine; Lecolorant MW est le poids molaire du colorant; 100 est un facteur de conversion unitaire; MR est le rapport molaire du colorant à la protéine dans le mélange réactionnel.
REMARQUE: Les MR suivants sont recommandés pour les réactions de marquage IgG: MR = 40 si l’anticorps est à 1-3 mg / mL ou MR = 30 si l’anticorps est à 4-10 mg / mL. Pour les facteurs de coagulation, MR = 5 est généralement utilisé. - Réaction de conjugaison
- Dans un tube réactionnel, mélanger la solution protéique avec un volume 10x inférieur de solution de bicarbonate 1 M.
- Ajouter la quantité requise de colorant fluorescent (voir étape 1.2) en remuant continuellement.
- Incuber le mélange réactionnel à température ambiante pendant environ 1 h, à l’abri de la lumière et sous agitation continue.
- Pour chaque 200 μL de solution protéique, ajouter 5 μL de 1,5 M de chlorhydrate d’hydroxylamine.
- Incuber le mélange réactionnel à température ambiante pendant environ 30 min, à l’abri de la lumière et sous agitation continue.
- Préparez la colonne de rotation.
- Ajouter 500 μL de milieu filtrant en gel pour la purification des protéines à une colonne. Centrifuger la colonne pendant 3 min à 1 000 × g.
- Jetez le tampon du tube de collecte. Si la colonne n’est pas pleine, ajoutez plus de milieu de filtration en gel et centrifugez la colonne pendant 3 min à 1 000 × g. Répétez cette étape jusqu’à ce que la colonne soit pleine.
- Purification
- Centrifuger le mélange réactionnel (à partir de l’étape 1.3.5) pendant 5 min à 17 000 × g et retirer le précipité.
- Transférer le surnageant dans la colonne de spin avec un milieu de filtration en gel. Laissez la solution absorber dans le lit de gel.
- Utilisez un tube de collecte vide pour la colonne de spin et centrifugez-le pendant 5 min à 1 000 × g. Après centrifugation, prélever la protéine marquée dans le tube de collecte.
- Détermination du degré d’étiquetage
- Corriger la contribution du colorant à l’absorbance à A280 en mesurant l’absorbance du colorant libre à 280 nm (A280) et le λmax pour le colorant (Amax) (voir Eq (2)).
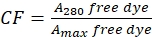 (2)
(2) - Mesurer l’absorbance du conjugué protéine-colorant à 280 nm (A280) et le λmax pour le colorant (Amax) et calculer la concétration protéique en utilisant Eq (3).
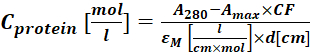 (3)
(3)
Où εM est le coefficient d’extinction molaire de la protéine à 280 nm; d est la longueur du chemin optique pendant la mesure de l’absorbance; La mucoviscidose est la contribution du colorant à l’absorbance à A280 (étape 1.6.1). - Calculez le degré d’étiquetage (DOL) à l’aide de Eq (4).
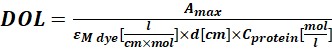 (4)
(4)
Où ε colorant M est le coefficient d’extinction molaire du colorant à λmax nm; d est la longueur du chemin optique pendant la mesure de l’absorbance; Laprotéine C est la concentration de la protéine (étape 1.6.2).
- Corriger la contribution du colorant à l’absorbance à A280 en mesurant l’absorbance du colorant libre à 280 nm (A280) et le λmax pour le colorant (Amax) (voir Eq (2)).
2. Préparation des vésicules de phospholipides
- Préparation et stockage du mélange lipidique
- Combiner les lipides dans le rapport approprié (phosphatidylsérine/phosphatidylcholine à un rapport de 20 mol% à 80 mol%).
- Sécher le mélange lipidique après lyophilisation ou évaporation et le stocker sous atmosphère inerte dans des ampoules de verre.
- Production de films lipidiques
- Ouvrez l’ampoule et remettez en suspension le mélange lipidique en une petite quantité (~100 μL) de chloroforme.
REMARQUE: N’utilisez pas trop de chloroforme car il ne s’évapore pas complètement. - Ajouter DiIC16(3) dans l’éthanol à 0,2 mol%. Transférer le mélange lipidique dans une fiole à fond rond. Étalez finement le mélange sur les côtés de la fiole en la faisant pivoter. Sécher le mélange lipidique pendant 30 min sous un jet d’argon.
- Ouvrez l’ampoule et remettez en suspension le mélange lipidique en une petite quantité (~100 μL) de chloroforme.
- Hydratation du mélange lipidique
- Ajouter un tampon aqueux convenablement chaud (~55 °C) (HEPES 20 mM, NaCl 140 mM, pH 7,4) dans un volume correspondant à la concentration lipidique attendue dans la fiole avec le film lipidique. Incuber le mélange avec un vortex à 55 °C pendant 30 min pour une hydratation complète.
- Placer le tube d’échantillonnage dans un congélateur ou un thermostat chaud pour que la suspension lipidique subisse 3 à 5 cycles de gel-dégel.
- Formation de vésicules lipidiques par extrusion
- Préparez l’extrudeuse conformément aux instructions du fabricant. Réchauffez tous les composants de l’extrudeuse à la température de transition de phase du mélange lipidique.
- Remplissez l’une des seringues de l’extrudeuse avec le mélange lipidique hydraté. Attendez 5-10 min pour que la température de la suspension lipidique s’équilibre avec la température de l’extrudeuse.
- Extruder le mélange lipidique à travers la membrane au moins 10 fois. Pour l’extrusion finale, placez la suspension lipidique dans la seringue alternative et recherchez un changement d’apparence d’une solution légèrement nébuleuse à une solution claire.
- Conserver le mélange résultant de vésicules lipidiques à 4 °C, de préférence dans une atmosphère inerte d’argon ou d’azote, pendant 3-4 jours. Ne pas congeler.
3. Isolement des plaquettes du sang total
- Prélever le sang total de donneurs sains dans des tubes contenant 3,2% de citrate de sodium.
- Ajouter la prostaglandine E1 (PGE1) (1 μM) et l’apyrase (0,1 U/mL) dans le sang, puis la centrifugation à température ambiante à 100 × g pendant 8 min.
- Après centrifugation, prendre le plasma riche en plaquettes et ajouter la solution de citrate de sodium (106 mM, pH 5,5) à un rapport plasma/citrate de 3:1. Centrifuger le plasma à température ambiante à 400 × g pendant 5 min.
- Retirer le surnageant et remettre la pastille dans 300 μL de tampon de Tyrode sans BSA (20 mM HEPES, 150 mM NaCl, 2,7 mM KCl, 1 mM MgCl2, 0,4 mM NaH2PO4, 2,5 mM CaCl2, 5 mM de glucose, pH 7,4). Purifier les plaquettes des protéines plasmatiques par chromatographie sur gel sur le milieu de filtration du gel pour la purification des plaquettes (voir la Table des matériaux).
4. Détection de l’interaction protéine-lipide par cytométrie en flux
- Expériences de liaison cinétique
- Diluer les vésicules phospholipides (à partir de l’étape 2.4.4) dans le tampon de Tyrode (20 mM HEPES, 150 mM NaCl, 2,7 mM KCl, 1 mM MgCl2, 0,4 mM NaH2PO4, 2,5 mM CaCl2, 5 mM de glucose, 0,5 % BSA, pH 7,4) à une concentration de 1 μM et un volume total de 250 μL.
- Mélanger le facteur de coagulation X marqué par fluorescence (fX-fd) de l’étape 1 à une concentration de 500 nM avec les vésicules phospholipides de l’étape 4.1.1 dans un rapport de 1:1 (concentration finale de vésicules 0,5 μM, concentration fX-fd est de 250 nM de fX) à un volume total de 500 μL.
- Injecter immédiatement les 500 μL de la suspension mixte (~20 min pour l’analyse avec un faible débit) dans le cytomètre en flux. Utilisez un faible débit et assurez-vous que le seuil pour le canal FL2 (exсitation 488 nm, filtre d’émission 585/42 nm) est la valeur 200. Mesurer la fluorescence moyenne dans le canal FL4 (exсitation 633 nm, filtre d’émission 660/20) pour le colorant de fluorescence de la table des matériaux.
REMARQUE: Choisissez un cytomètre sans échantillonneur automatique. Cela accélérera le processus d’injection de l’échantillon dans la cellule de mesure. - Lorsque la saturation de la liaison est atteinte (pas d’augmentation significative de la fluorescence dans les 5 minutes), diluer rapidement l’échantillon 20 fois avec le tampon de Tyrode, et surveiller la dissociation jusqu’à ce que la fluorescence de base soit atteinte (dissociation complète) ou jusqu’à ce qu’un plateau soit atteint (pas de diminution significative de la fluorescence dans les 5 minutes).
REMARQUE: Comme contrôle, ajoutez 10 μM EDTA et surveillez la dissociation complète pendant 5 min.
- Expériences de liaison à l’équilibre
REMARQUE: Utilisez la courbe cinétique de liaison pour déterminer le temps nécessaire pour atteindre la saturation; le temps de saturation pour fX-fd et les vésicules artificielles est de 20 min.- Incuber des vésicules phospholipides artificielles (5 μM) pour le dosage de liaison avec différentes concentrations de fX-fd (de 0 à 1 000 nM) dans le tampon de Tyrode pendant 20 min.
- Diluer chaque échantillon de l’étape 4.2.1 par 20x jusqu’à un volume final de 200 μL avec le tampon de Tyrode. Analyser immédiatement l’échantillon dilué par cytométrie en flux dans les 30 s. Utilisez les paramètres de l’étape 4.1.3.
REMARQUE: Comme contrôle de la liaison non spécifique, utilisez des échantillons similaires avec de l’EDTA (10 μM) et incubés pendant 5 min.
5. Analyse des données de cytométrie en flux
- Exportez des expériences au format FSC du logiciel d’acquisition de données cytométriques vers le logiciel cytométrique pour l’analyse des données (voir la table des matériaux). Choisissez | fichier | d’exportation FCS fichiers. Ouvrez les fichiers FSC dans le logiciel cytométrique pour l’analyse des données en sélectionnant les fichiers sur l’ordinateur et en les faisant glisser vers l’espace de travail du programme.
- Pour le contrôle des microvésicules, identifier la région des microvésicules par la fluorescence du colorant lipophile DiIC16(3). Utilisez les commandes de menu ou le bouton de traçage dans la feuille de calcul pour créer un diagramme à points SSC à partir de FL2 (colorant DilC16(3)) dans les coordonnées du journal. Choisissez le bouton Porte rectangulaire pour dessiner une région de contrôle afin que les événements d’un échantillon sans vésicules ne soient pas inclus dans cette région (Figure 1B,C).
- Analysez les expériences cinétiques.
- Créer un diagramme de points en utilisant les coordonnées de fluorescence (FL4) au fil du temps pour la région des vésicules (double-cliquez dans la région des vésicules à l’étape 5.2)
- Exportez les données sur le changement de fluorescence au fil du temps au format csv. Choisissez un exemple | Cliquez avec le bouton droit sur | | d’exportation Choisissez FL4 et Heure dans paramètres | Sélectionnez le répertoire pour enregistrer | Sélectionnez le format CSV | Exporter.
- Ouvrez le fichier CSV dans n’importe quel logiciel statistique (voir la table des matériaux). Calculez une moyenne mobile simple de fluorescence et de temps pour chaque 1 000 événements.
- Approximer un graphique de la dépendance de la fluorescence de la moyenne mobile simple sur le temps sous l’hypothèse de la dépendance exponentielle (Analysis > Fitting >Nonlinear Curve Fit) et l’utiliser pour calculer la constante d’association cinétique en utilisant Eq (5).
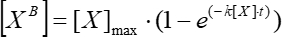 (5)
(5)
Où [XB] est la concentration du facteur lié à chaque moment du temps (unités définies par l’utilisateur) selon la moyenne mobile simple de l’étape 5.3.3; [X] est la concentration en facteur ajouté; [X]max est la concentration maximale du facteur lié; k est la constante d’association; t est le moment. - Répétez le même ensemble d’actions (5.3.1-5.3.4) pour calculer la constante de dissociation cinétique à l’aide de Eq (6).
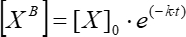 (6)
(6)
Où [XB] est la concentration du facteur lié à chaque moment du temps; [X]0 est la concentration du facteur lié au moment initial; k est la constante de dissociation ; t est le moment.
- Essai de liaison à l’équilibre
- Déterminer la fluorescence moyenne de fX-fd dans la région des vésicules pour chaque concentration sélectionnée de fX-fd.
- Approximation de la dépendance de la fluorescence du facteur lié par rapport à la concentration du facteur ajouté dans l’hypothèse d’une liaison simple à site unique. Calculez les paramètres de liaison moyens à l’aide de Eq (7) à partir de trois répétitions indépendantes au minimum.
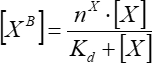 (7)
(7)
Où [XB] est la concentration du facteur lié; [X] est la concentration en facteur ajouté; nx est le nombre apparent de sites de liaison par vésicule; Kd est la constante de dissociation apparente.
6. Conversion de l’intensité de fluorescence en nombre moyen de sites de liaison
- Préparez des perles calibrées.
- Incuber des plaquettes filtrées au gel (voir étape 3.3) avec de l’A23187 (10 μM) en présence de CaCl2 (2,5 mM) pendant 10 min à température ambiante.
- Ajoutez aux plaquettes activées les différentes concentrations de fX-fd (0 à 1 000 nM). Ajouter 2% v/v de formaldéhyde et incuber pendant 1 h. Arrêtez la réaction en incubant les plaquettes avec 3 M de glycine et 5% de BSA pendant 30 min à température ambiante.
- Purifier le mélange du colorant non réagi. Centrifuger les plaquettes pendant 5 min à 400 × g, retirer le surnageant et remettre la pastille dans le tampon de Tyrode (contenant 0,5% de BSA).
REMARQUE: Répétez l’étape 6.1.3 trois fois.
- Mesurez le niveau de fluorescence des billes d’étalonnage dans chaque échantillon d’abord à l’aide d’un spectrofluoromètre (pour le colorant fluorescent de la Table des matériaux, excitation 633 nm, émission 670 nm), puis à l’aide du cytomètre en flux (dans le canal FL4: excitation 633 nm, filtre d’émission 660/20). À l’aide d’un compteur de cellules, déterminez le nombre de perles dans chaque échantillon.
- Convertissez l’intensité de fluorescence de chaque échantillon de billes respectif en concentration de colorant fluorescent soluble à l’aide d’un spectrofluoromètre. Recalculer la concentration de colorant fluorescent pour le nombre de molécules de fluorophore à l’aide d’Eq (8).
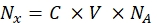 (8)
(8)
Où Nx est le nombre de molécules de fluorophore; C est la concentration de colorant fluorescent; NA est la constante d’Avogadro; NA = 6,02214076×1023 mol -1. - Créer un graphique de dépendance de la fluorescence moyenne des billes dans un cytomètre en flux (étape 6.2) sur le nombre de molécules de fluorophore (voir étape 6.3) pour chaque échantillon en utilisant n’importe quel logiciel statistique (voir le Tableau des matériaux). Approximation de cette dépendance par proportionnalité linéaire (Analyse | | de montage Ajustement linéaire). À partir de l’approximation en Eq (9), calculer le facteur de conversion de la fluorescence moyenne en sites de liaison.
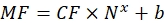 (9)
(9)
Où MF est la fluorescence moyenne des billes par cytométrie en flux; Nx est le nombre de molécules de fluorophore par perle; La mucoviscidose représente le facteur de conversion de la fluorescence moyenne en sites de liaison. CF et b sont obtenus à partir des résultats de l’ajustement du graphique par proportionnalité linéaire. - Calculer le nombre apparent de sites de liaison par vésicule d’intérêt en utilisant Eq (10).
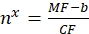 (10)
(10)
Où nx est le nombre apparent de sites de liaison par vésicule d’intérêt; MF est la fluorescence moyenne des vésicules d’intérêt par cytométrie en flux; CF et b sont des facteurs de conversion de l’Eq (8).
Résultats
La méthode de cytométrie en flux décrite ici est utilisée pour caractériser la liaison des protéines de coagulation plasmatique aux plaquettes activées. En outre, les vésicules phospholipides PS:PC 20:80 ont été appliquées comme système modèle. Cet article se concentre principalement sur les vésicules phospholipides artificielles à titre d’exemple. Les paramètres du cytomètre, en particulier la tension du tube photomultiplicateur (PMT) et la compensation doivent être sélectionnés pour chaque disposi...
Discussion
La méthode proposée peut être adaptée pour une caractérisation approximative de l’interaction des protéines avec les membranes phospholipides provenant de diverses sources et compositions. La cytométrie quantitative en flux décrite ici concède à la résonance plasmonique de surface (SPR) dans plusieurs paramètres. En particulier, il a une sensibilité et une résolution temporelle plus faibles et nécessite un marquage fluorescent des protéines. Le marquage fluorescent peut entraîner un changement de confo...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs ont été soutenus par une subvention de la Fondation russe pour la science 20-74-00133.
matériels
| Name | Company | Catalog Number | Comments |
| A23187 | Sigma Aldrich | C7522-10MG | |
| Alexa Fluor 647 NHS Ester (Succinimidyl Ester) | Thermo Fisher Scientific | A37573 | fluorescent dye |
| Apyrase from potatoes | Sigma Aldrich | A2230 | |
| BD FACSCantoII | BD Bioscience | ||
| bovine serum albumin | VWR Life Science AMRESCO | Am-O332-0.1 | |
| Calcium chloride, anhydrous, powder, ≥97% | Sigma Aldrich | C4901-100G | |
| Cary Eclipse Fluorescence Spectrometer | Agilent | ||
| D-(+)-Glucose | Sigma Aldrich | G7528-1KG | |
| DiIC16(3) (1,1'-Dihexadecyl-3,3,3',3'-Tetramethylindocarbocyanine Perchlorate) | Thermo Fisher Scientific | D384 | |
| DMSO | Sigma Aldrich | D8418 | |
| EDTA disodium salt | VWR Life Science AMRESCO | Am-O105B-0.1 | |
| FACSDiva | BD Bioscience | cytometry data acquisition software | |
| FlowJo | Tree Star | cytometer software for data analysis | |
| HEPES | Sigma Aldrich | H4034-500G | |
| Human Factor X | Enzyme research | HFX 1010 | |
| Hydroxylamine hydrochloride | Panreac | 141914.1209 | |
| L-α-phosphatidylcholine (Brain, Porcine) | Avanti Polar Lipids | 840053P | |
| L-α-phosphatidylserine (Brain, Porcine) (sodium salt) | Avanti Polar Lipids | 840032P | |
| Magnesium chloride | Sigma Aldrich | M8266-100G | |
| Mini-Extruder | Avanti Polar Lipids | 610020-1EA | |
| OriginPro 8 SR4 v8.0951 | OriginLab Corporation | Statistical software | |
| Phosphate Buffered Saline (PBS) Tablets, Biotechnology Grade | VWR Life Science AMRESCO | 97062-732 | |
| Potassium chloride | Sigma Aldrich | P9541-500G | |
| Prostaglandin E1 | Cayman Chemical | 13010 | |
| Sephadex G25 | GE Healthcare | GE17-0033-01 | gel filtration medium for protein purification |
| Sepharose CL-2B | Sigma Aldrich | CL2B300-500ML | gel filtration medium for platelet purification |
| Sodium bicarbonate | Corning | 61-065-RO | |
| Sodium chloride | Sigma Aldrich | S3014-500G | |
| Sodium phosphate monobasic | Sigma Aldrich | S3139-250G | |
| Spin collumns with membrane 0.2 µm | Sartorius | VS0171 | |
| Trisodium citrate dihydrate | Sigma Aldrich | S1804-1KG |
Références
- Hoffman, M., Monroe, D. M. A cell-based model of hemostasis. Thrombosis and haemostasis. 85 (6), 958-965 (2001).
- Roberts, H. R., Hoffman, M., Monroe, D. M. A cell-based model of thrombin generation. Seminars in Thrombosis and Hemostasis. 32, 32-38 (2006).
- Panteleev, M. A., Dashkevich, N. M., Ataullakhanov, F. I. Hemostasis and thrombosis beyond biochemistry: roles of geometry, flow and diffusion. Thrombosis Research. 136 (4), 699-711 (2015).
- Podoplelova, N. A., et al. Hysteresis-like binding of coagulation factors X/Xa to procoagulant activated platelets and phospholipids results from multistep association and membrane-dependent multimerization. Biochimica et Biophysica Acta. 1858 (6), 1216-1227 (2016).
- Panteleev, M. A., Ananyeva, N. M., Greco, N. J., Ataullakhanov, F. I., Saenko, E. L. Two subpopulations of thrombin-activated platelets differ in their binding of the components of the intrinsic factor X-activating complex. Journal of Thrombosis and Haemostasis. 3 (11), 2545-2553 (2005).
- Kotova, Y., et al. Binding of coagulation factor XIII zymogen to activated platelet subpopulations: roles of integrin αIIbβ3 and fibrinogen. Thrombosis and Haemostasis. 119 (6), 906-915 (2019).
- Fay, S. P., Posner, R. G., Swann, W. N., Sklar, L. A. Real-time analysis of the assembly of ligand, receptor, and G protein by quantitative fluorescence flow cytometry. Biochemistry. 30 (20), 5066-5075 (2002).
- Nolan, J. P., Shen, B., Park, M. S., Sklar, L. A. Kinetic analysis of human flap endonuclease-1 by flow cytometry. Biochemistry. 35 (36), 11668-11676 (1996).
- Sarvazyan, N. A., Lim, W. K., Neubig, R. R. Fluorescence analysis of receptor−G protein interactions in cell membranes. Biochemistry. 41 (42), 12858-12867 (2002).
- Sarvazyan, N. A., Neubig, R. R. Analysis of molecular assemblies by flow cytometry: determinants of Gi1 and by binding. Advances in Optical Biophysics. 3256, 122-131 (1998).
- De Franceschi, N., et al. ProLIF - Quantitative integrin protein-protein interactions and synergistic membrane effects on proteoliposomes. Journal of Cell Science. 132 (4), (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon