Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Análisis de la interacción de proteínas marcadas con fluorescentes con microvesículas de fosfolípidos artificiales utilizando citometría de flujo cuantitativa
En este artículo
Resumen
Aquí, describimos un conjunto de métodos para caracterizar la interacción de proteínas con membranas de células o microvesículas.
Resumen
En el cuerpo humano, la mayoría de las principales reacciones fisiológicas involucradas en la respuesta inmune y la coagulación de la sangre proceden en las membranas de las células. Un primer paso importante en cualquier reacción dependiente de la membrana es la unión de la proteína en la membrana de fosfolípidos. Se ha desarrollado un enfoque para estudiar la interacción de las proteínas con las membranas lipídicas utilizando proteínas marcadas fluorescentemente y citometría de flujo. Este método permite el estudio de las interacciones proteína-membrana utilizando células vivas y vesículas de fosfolípidos naturales o artificiales. La ventaja de este método es la simplicidad y disponibilidad de reactivos y equipos. En este método, las proteínas se etiquetan utilizando colorantes fluorescentes. Sin embargo, se pueden utilizar proteínas autofabricadas y disponibles comercialmente, marcadas fluorescentemente. Después de la conjugación con un colorante fluorescente, las proteínas se incuban con una fuente de la membrana de fosfolípidos (microvesículas o células), y las muestras se analizan por citometría de flujo. Los datos obtenidos se pueden utilizar para calcular las constantes cinéticas y el equilibrio Kd. Además, es posible estimar el número aproximado de sitios de unión a proteínas en la membrana de fosfolípidos utilizando perlas de calibración especiales.
Introducción
Las biomembranas separan el contenido interno de las células animales y el espacio extracelular. Tenga en cuenta que las membranas también rodean las microvesículas formadas durante el ciclo de vida de la célula y los orgánulos. La membrana celular está compuesta predominantemente de lípidos y proteínas. Las proteínas de membrana realizan funciones de señalización, estructurales, de transporte y adhesivas. Sin embargo, la bicapa lipídica también es esencial para la interrelación de la célula animal con el espacio extracelular. Este artículo propone un método para estudiar la interacción periférica de las proteínas externas con la membrana lipídica.
El ejemplo más sorprendente de reacciones que ocurren en la capa de membrana externa de una célula animal es la reacción de coagulación de la sangre. Una característica importante de la coagulación sanguínea es que todas las reacciones principales proceden sobre las membranas fosfolípidas de las células y microvesículas que surgen de estas células y no en el plasma 1,2,3. Las reacciones dependientes de la membrana incluyen el proceso de inicio de la coagulación (en las membranas celulares del subendotelio, el endotelio inflamado o las células inmunes activadas, con la participación de un factor tisular), todas las reacciones de la principal activación en cascada de los factores IX, X, protrombina; activación del factor XI por trombina (en las membranas de plaquetas, eritrocitos, lipoproteínas y microvesículas activadas); reacciones de la vía de la proteína C; inactivación de enzimas de coagulación (en las membranas de las células endoteliales con la participación de cofactores de trombomodulina, receptor de proteína C endotelial, sulfato de heparán); y reacciones de vía de contacto (en membranas de plaquetas y algunas microvesículas con la participación de cofactores desconocidos). Por lo tanto, es imposible investigar la coagulación de la sangre sin estudiar la interacción de varias proteínas plasmáticas con la membrana de las células sanguíneas.
Este artículo describe un método basado en la citometría de flujo para caracterizar la interacción de las proteínas con las membranas lipídicas de las células o microvesículas. Este enfoque se propuso inicialmente para estudiar la interacción del plasma sanguíneo con plaquetas y vesículas artificiales de fosfolípidos. Además, la mayoría de las proteínas estudiadas interactúan directamente con fosfolípidos de membrana cargados negativamente, particularmente con fosfatidilserina 4,5. Además, hay proteínas cuya interacción con la membrana está mediada por receptores especiales6.
Una capacidad importante de la citometría de flujo es discriminar entre ligandos libres y unidos sin separación adicional. Esta característica de la citometría permite el estudio de la unión al equilibrio del ligando en el punto final y ayuda a realizar mediciones cinéticas continuas. La técnica es poco sofisticada y no requiere una preparación de muestras compleja. La citometría de flujo se utiliza activamente para estudiar cuantitativamente la dinámica de la interacción entre péptidos fluorescentes, receptores y proteínas G en neutrófilos intactos y permeables a detergente7. Este enfoque también es aplicable para explorar las interacciones proteína-ADN y la cinética de la actividad de la endonucleasa en tiempo real8. Con el tiempo, este método se utilizó para estudiar cuantitativamente las interacciones proteína-proteína de alta afinidad con vesículas lipídicas purificadas9, o, más generalmente, con proteínas de membrana expresadas en un sistema de expresión celular Sf9 altamente eficiente10. También se han descrito métodos cuantitativos para caracterizar las interacciones proteína-liposoma mediante citometría de flujo para proteínas transmembrana11.
Esta técnica utiliza perlas de calibración hechas por ellos mismos para evitar el uso de cuentas disponibles comercialmente7. Las perlas de calibración utilizadas anteriormente7 estaban destinadas a trabajar con fluoresceína, lo que restringía sustancialmente la variedad de ligandos fluorescentes accesibles en las proteínas. Además, este documento ofrece una nueva forma de adquirir y analizar datos cinéticos para una resolución de tiempo razonable. Aunque este método se describe para vesículas de fosfolípidos artificiales, no hay limitaciones obvias para su adaptabilidad a células, vesículas naturales o vesículas de fosfolípidos artificiales con una composición lipídica diferente. El método aquí descrito permite la estimación de los parámetros de interacción (kon, koff) y equilibrio (Kd) y facilita la caracterización cuantitativa del número de sitios de unión a proteínas en la membrana. Tenga en cuenta que esta técnica proporciona una estimación aproximada del número de sitios de unión. Las ventajas del método son su relativa simplicidad, accesibilidad y adaptabilidad a células nativas y microvesículas naturales y artificiales.
Protocolo
1. Etiquetado de proteínas fluorescentes
- Preparación de materiales
- Prepare 1 M Tampón de bicarbonato de sodio, pH 9.0, guárdelo a 4 ° C y úselo dentro de una semana.
- Prepare 1.5 M de tampón de clorhidrato de hidroxilamina, pH 8.5, inmediatamente antes de su uso.
- Preparar una solución de 10 mg/ml de colorante fluorescente (ver la Tabla de Materiales) en dimetilsulfóxido.
NOTA: Esta solución se puede almacenar durante un mes a -20 °C en la oscuridad. - Preparar soluciones de anticuerpos purificados u otras proteínas a 1-10 mg/ml.
NOTA: Evite los tampones que contengan iones de amonio o aminas primarias. Reemplace los tampones que contienen Tris o glicerol con solución salina tamponada con fosfato (PBS) por diálisis. Ni la azida de sodio (≤3 mM) ni el timerosal (≤1 mM) afectarán significativamente la reacción de conjugación. - Incubar el medio de filtración en gel (ver la Tabla de Materiales) para la purificación de proteínas en PBS durante la noche a temperatura ambiente o durante 2 h a 60 °C. Aplique el medio de filtración de gel sobre columnas de espín con membranas de 0,2 μm.
- Calcule la cantidad de colorante reactivo que se utilizará para cada reacción de acuerdo con la concentración de proteína a etiquetar mediante Eq (1).
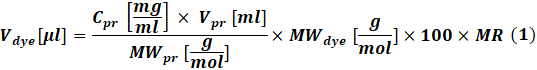
Dondeel tinte V es el volumen de la solución madre del tinte; Cpr, Vpr y MWpr es la concentración, volumen y peso molar de la proteína; Eltinte MW es el peso molar del tinte; 100 es un factor de conversión unitario; La RM es la relación molar de colorante a proteína en la mezcla de reacción.
NOTA: Se recomiendan los siguientes MR para las reacciones de marcado de IgG: MR = 40 si el anticuerpo está en 1-3 mg / ml o MR = 30 si el anticuerpo está en 4-10 mg / ml. Para los factores de coagulación, generalmente se usa MR = 5. - Reacción de conjugación
- En un tubo de reacción, mezcle la solución de proteína con un volumen 10 veces menor de solución de bicarbonato de 1 M.
- Agregue la cantidad requerida de colorante fluorescente (ver paso 1.2) con agitación continua.
- Incubar la mezcla de reacción a temperatura ambiente durante aproximadamente 1 h, protegida de la luz y con agitación continua.
- Por cada 200 μL de solución proteica, añadir 5 μL de clorhidrato de hidroxilamina de 1,5 M.
- Incubar la mezcla de reacción a temperatura ambiente durante aproximadamente 30 min, protegida de la luz y con agitación continua.
- Prepare la columna de giro.
- Agregue 500 μL de medio de filtración en gel para la purificación de proteínas a una columna. Centrifugar la columna durante 3 min a 1.000 × g.
- Deseche el búfer del tubo de recogida. Si la columna no está llena, agregue más medio de filtración de gel y centrífique la columna durante 3 minutos a 1,000 × g. Repita este paso hasta que la columna esté llena.
- Purificación
- Centrifugar la mezcla de reacción (a partir de la etapa 1.3.5) durante 5 min a 17.000 × g y retirar el precipitado.
- Transfiera el sobrenadante a la columna de centrifugado con medio de filtración en gel. Deje que la solución se absorba en la cama de gel.
- Utilice un tubo de recolección vacío para la columna de centrifugado y centrímelo durante 5 minutos a 1.000 × g. Después de la centrifugación, recoja la proteína marcada del tubo de recolección.
- Determinación del grado de etiquetado
- Corregir la contribución del tinte a la absorbancia a A280 midiendo la absorbancia del tinte libre a 280 nm (A280) y la λmáx para el tinte (Amax) (ver Eq (2)).
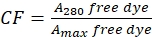 (2)
(2) - Mida la absorbancia del conjugado proteína-colorante a 280 nm (A280) y el máximo λ para el colorante (Amáx.) y calcule la concetración de proteínas utilizando Eq (3).
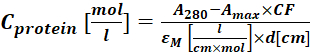 (3)
(3)
Donde εM es el coeficiente de extinción molar de la proteína a 280 nm; d es la longitud de la trayectoria óptica durante la medición de la absorbancia; La FQ es la contribución del colorante a la absorbancia a280 (paso 1.6.1). - Calcule el grado de etiquetado (DOL) utilizando Eq (4).
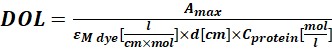 (4)
(4)
Donde ε M es el coeficiente de extinción molar del colorante a λmax nm; d es la longitud de la trayectoria óptica durante la medición de la absorbancia; Laproteína C es la concentración de la proteína (paso 1.6.2).
- Corregir la contribución del tinte a la absorbancia a A280 midiendo la absorbancia del tinte libre a 280 nm (A280) y la λmáx para el tinte (Amax) (ver Eq (2)).
2. Preparación de vesículas de fosfolípidos
- Preparación y almacenamiento de la mezcla lipídica
- Combine los lípidos en la proporción adecuada (fosfatidilserina/fosfatidilcolina en una proporción de 20 mol% a 80 mol%).
- Seque la mezcla de lípidos después de la liofilización o evaporación y guárdela bajo una atmósfera inerte en ampollas de vidrio.
- Producción de película lipídica
- Abra la ampolla y vuelva a suspender la mezcla de lípidos en una pequeña cantidad (~ 100 μL) de cloroformo.
NOTA: No use demasiado cloroformo ya que no se evapora por completo. - Añadir DiIC16(3) en etanol al 0,2 mol%. Transfiera la mezcla de lípidos a un matraz de fondo redondo. Extienda la mezcla finamente sobre los lados del matraz girándolo. Seque la mezcla de lípidos durante 30 minutos bajo una corriente de argón.
- Abra la ampolla y vuelva a suspender la mezcla de lípidos en una pequeña cantidad (~ 100 μL) de cloroformo.
- Hidratación de la mezcla lipídica
- Añadir un tampón acuoso adecuadamente caliente (~55 °C) (HEPES 20 mM, NaCl 140 mM, pH 7,4) en un volumen correspondiente a la concentración de lípidos esperada al matraz con la película lipídica. Incubar la mezcla con vórtice a 55 °С durante 30 min para una hidratación completa.
- Colocar el tubo de muestra en un congelador o termostato caliente para que la suspensión lipídica se someta a 3-5 ciclos de congelación-descongelación.
- Formación de vesículas lipídicas por extrusión
- Prepare la extrusora de acuerdo con las instrucciones del fabricante. Calentar todos los componentes del extrusor a la temperatura de transición de fase de la mezcla lipídica.
- Llene una de las jeringas extrusoras con la mezcla de lípidos hidratados. Espere 5-10 min para que la temperatura de la suspensión lipídica se equilibre con la temperatura del extrusor.
- Extruir la mezcla de lípidos a través de la membrana al menos 10 veces. Para la extrusión final, coloque la suspensión lipídica en la jeringa alternativa y busque un cambio en la apariencia de una solución ligeramente nebulosa a una solución transparente.
- Conservar la mezcla resultante de vesículas lipídicas a 4 °C, preferiblemente en una atmósfera inerte de argón o nitrógeno, durante 3-4 días. No congelar.
3. Aislamiento de plaquetas de sangre total
- Recolectar sangre entera de donantes sanos en tubos que contengan citrato de sodio al 3,2%.
- Añadir prostaglandina E1 (PGE1) (1 μM) y apirasa (0,1 U/mL) a la sangre, seguido de centrifugación a temperatura ambiente a 100 × g durante 8 min.
- Después de la centrifugación, tome el plasma rico en plaquetas y agregue la solución de citrato de sodio (106 mM, pH 5.5) a una relación plasma/citrato de 3: 1. Centrifugar el plasma a temperatura ambiente a 400 × g durante 5 min.
- Retire el sobrenadante y vuelva a suspender el pellet en 300 μL del tampón de Tyrode sin BSA (20 mM HEPES, 150 mM NaCl, 2.7 mM KCl, 1 mM MgCl2, 0.4 mM NaH2PO4, 2.5 mM CaCl2, 5 mM glucosa, pH 7.4). Purificar las plaquetas de las proteínas plasmáticas mediante cromatografía en gel en el medio de filtración de gel para la purificación de plaquetas (ver la Tabla de Materiales).
4. Detección de interacción proteína-lípido por citometría de flujo
- Experimentos de unión cinética
- Diluir vesículas de fosfolípidos (a partir del paso 2.4.4) en el tampón de Tyrode (20 mM HEPES, 150 mM NaCl, 2.7 mM KCl, 1 mM MgCl2, 0.4 mM NaH2PO4, 2.5 mM CaCl2, 5 mM glucosa, 0.5% BSA, pH 7.4) a una concentración de 1 μM y volumen total de 250 μL.
- Mezcle el factor de coagulación X (fX-fd) marcado con fluorescente del paso 1 a una concentración de 500 nM con las vesículas de fosfolípidos del paso 4.1.1 en una proporción de 1:1 (concentración final de vesícula 0.5 μM, la concentración de fX-fd es de 250 nM de fX) a un volumen total de 500 μL.
- Inyecte inmediatamente los 500 μL de la suspensión mixta (~ 20 min para análisis con una tasa de flujo baja) en el citómetro de flujo. Utilice un caudal bajo y asegúrese de que el umbral para el canal FL2 (exсitación 488 nm, filtro de emisión 585/42 nm) sea el valor 200. Mida la fluorescencia media en el canal FL4 (exсitación 633 nm, filtro de emisión 660/20) para el colorante de fluorescencia de la Tabla de Materiales.
NOTA: Elija un citómetro sin un muestreador automático. Esto acelerará el proceso de inyección de la muestra en la celda de medición. - Cuando se logre la saturación de la unión (sin aumento significativo de la fluorescencia en 5 min), diluya rápidamente la muestra 20 veces con el tampón de Tyrode y controle la disociación hasta que se alcance la fluorescencia basal (disociación completa) o hasta que se alcance una meseta (sin disminución significativa de la fluorescencia dentro de los 5 min).
NOTA: Como control, agregue 10 μM EDTA y supervise la disociación completa durante 5 min.
- Experimentos de unión al equilibrio
NOTA: Utilice la curva cinética de unión para determinar el tiempo de saturación; el tiempo de saturación para fX-fd y vesículas artificiales es de 20 min.- Incubar vesículas artificiales de fosfolípidos (5 μM) para el ensayo de unión con diferentes concentraciones de fX-fd (de 0 a 1.000 nM) en el tampón de Tyrode durante 20 min.
- Diluya cada muestra desde el paso 4.2.1 por 20x hasta un volumen final de 200 μL con el tampón de Tyrode. Analice inmediatamente la muestra diluida por citometría de flujo dentro de los 30 s. Utilice la configuración del paso 4.1.3.
NOTA: Como control para la unión inespecífica, utilice muestras similares con EDTA (10 μM) e incube durante 5 min.
5. Análisis de datos de citometría de flujo
- Exporte experimentos en formato FSC desde el software de adquisición de datos de citometría al software de citómetro para el análisis de datos (consulte la Tabla de materiales). Elija file | exportación | FCS archivos. Abra los archivos FSC en el software del citómetro para el análisis de datos seleccionando los archivos en la computadora y arrastrándolos al espacio de trabajo del programa.
- Para el gating de las microvesículas, identificar la región de las microvesículas por la fluorescencia del colorante lipofílico DiIC16(3). Utilice comandos de menú o el botón de trazado en la hoja de cálculo para crear SSC de trazado de puntos a partir de FL2 (teñir DilC16(3)) en coordenadas de registro. Elija el botón Puerta rectangular para dibujar una región de cierre de modo que los eventos de una muestra sin vesículas no se incluyan en esta región (Figura 1B,C).
- Analizar los experimentos cinéticos.
- Cree un diagrama de puntos utilizando las coordenadas de fluorescencia (FL4) a lo largo del tiempo para la región de las vesículas (haga doble clic en la región de las vesículas en el paso 5.2)
- Exporte los datos sobre el cambio en la fluorescencia a lo largo del tiempo en formato csv. Elija | de muestra Haga clic con el botón secundario en | exportación | Elija FL4 y tiempo en parámetros | Seleccione el directorio para guardar | Seleccione el formato CSV | Exportación.
- Abra el archivo CSV en cualquier software estadístico (consulte la Tabla de materiales). Calcule una media móvil simple de fluorescencia y tiempo por cada 1.000 eventos.
- Aproximar un gráfico de la dependencia de la fluorescencia de la media móvil simple en el tiempo bajo el supuesto de dependencia exponencial (Analysis > Fitting >Nonlinear Curve Fit) y utilizar esto para calcular la constante de asociación cinética utilizando Eq (5).
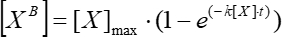 (5)
(5)
Donde [XB] es la concentración del factor enlazado en cada momento del tiempo (unidades definidas por el usuario) de acuerdo con la media móvil simple del paso 5.3.3; [X] es la concentración de factor añadido; [X]max es la concentración máxima del factor enlazado; k es la constante de asociación; t es el momento. - Repita el mismo conjunto de acciones (5.3.1-5.3.4) para calcular la constante de disociación cinética utilizando Eq (6).
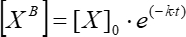 (6)
(6)
Donde [XB] es la concentración del factor enlazado en cada momento del tiempo; [X]0 es la concentración del factor enlazado en el momento inicial del tiempo; k es la constante de disociación; t es el momento.
- Ensayo de unión al equilibrio
- Determinar la fluorescencia media de fX-fd en la región de las vesículas para cada concentración seleccionada de fX-fd.
- Aproximar la dependencia de la fluorescencia del factor unido de la concentración del factor añadido en el supuesto de la unión simple de un solo sitio. Calcule los parámetros de enlace promedio usando Eq (7) a partir de tres repeticiones independientes como mínimo.
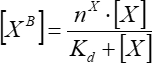 (7)
(7)
Donde [XB] es la concentración del factor enlazado; [X] es la concentración de factor añadido; nx es el número aparente de sitios de unión por vesícula; Kd es la constante de disociación aparente.
6. Conversión de la intensidad de la fluorescencia al número medio de sitios de unión
- Preparar cuentas calibradas.
- Incubar plaquetas filtradas en gel (ver paso 3.3) con A23187 (10 μM) en presencia de CaCl2 (2,5 mM) durante 10 min a temperatura ambiente.
- Añadir a las plaquetas activadas las diversas concentraciones de fX-fd (0 a 1.000 nM). Añadir 2% v/v formaldehído e incubar durante 1 h. Detenga la reacción incubando las plaquetas con 3 M de glicina y 5% de BSA durante 30 min a temperatura ambiente.
- Purifique la mezcla del tinte no reaccionado. Centrifugar las plaquetas durante 5 min a 400 × g, retirar el sobrenadante y volver a suspender el pellet en el tampón de Tyrode (que contiene 0,5% de BSA).
NOTA: Repita el paso 6.1.3 tres veces.
- Mida el nivel de fluorescencia de las perlas de calibración en cada muestra primero usando un espectrofluorómetro (para colorante fluorescente de la Tabla de Materiales, excitación 633 nm, emisión 670 nm) y luego usando el citómetro de flujo (en el canal FL4: excitación 633 nm, filtro de emisión 660/20). Usando un contador de celdas, determine el número de cuentas en cada muestra.
- Convierta la intensidad de fluorescencia de cada muestra de perla respectiva a la concentración de colorante fluorescente soluble utilizando un espectrofluorómetro. Recalcule la concentración de colorante fluorescente para el número de moléculas de fluoróforo utilizando Eq (8).
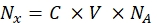 (8)
(8)
Donde Nx es el número de moléculas de fluoróforo; C es la concentración de colorante fluorescente; NA es la constante de Avogadro; NA = 6.02214076×1023 mol -1. - Cree un gráfico de dependencia de la fluorescencia promedio de las perlas en un citómetro de flujo (paso 6.2) en el número de moléculas de fluoróforo (ver paso 6.3) para cada muestra utilizando cualquier software estadístico (ver la Tabla de Materiales). Aproximar esta dependencia por proporcionalidad de línea (Análisis | | de ajuste Ajuste lineal). A partir de la aproximación en Eq (9), calcule el factor de conversión de la fluorescencia media a los sitios de unión.
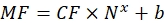 (9)
(9)
Donde MF es la fluorescencia media de las perlas por citometría de flujo; Nx es el número de moléculas de fluoróforo por perla; La FQ representa el factor de conversión de la fluorescencia media a los sitios de unión. CF y b se obtienen de los resultados de ajuste del gráfico por proporcionalidad lineal. - Calcule el número aparente de sitios de unión por vesícula de interés utilizando Eq (10).
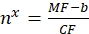 (10)
(10)
Donde nx es el número aparente de sitios de unión por vesícula de interés; MF es la fluorescencia media de las vesículas de interés por citometría de flujo; CF y b son factores de conversión del Eq (8).
Resultados
El método de citometría de flujo descrito en este documento se utiliza para caracterizar la unión de las proteínas de coagulación plasmática a las plaquetas activadas. Además, se aplicaron vesículas de fosfolípidos PS:PC 20:80 como sistema modelo. Este artículo se centra principalmente en las vesículas artificiales de fosfolípidos como ejemplo. Los parámetros del citómetro, en particular, la tensión del tubo fotomultiplicador (PMT) y la compensación deben seleccionarse para cada dispositivo específico, e...
Discusión
El método propuesto se puede adaptar para una caracterización aproximada de la interacción de proteínas con membranas de fosfolípidos de diversas fuentes y composiciones. La citometría de flujo cuantitativa descrita aquí concede a la resonancia de plasmón de superficie (SPR) en varios parámetros. En particular, tiene una menor sensibilidad y resolución de tiempo y requiere un etiquetado fluorescente de proteínas. El etiquetado fluorescente puede conducir a un cambio en la conformación y la pérdida de activid...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Los autores fueron apoyados por una subvención de la Fundación Rusa de Ciencia 20-74-00133.
Materiales
| Name | Company | Catalog Number | Comments |
| A23187 | Sigma Aldrich | C7522-10MG | |
| Alexa Fluor 647 NHS Ester (Succinimidyl Ester) | Thermo Fisher Scientific | A37573 | fluorescent dye |
| Apyrase from potatoes | Sigma Aldrich | A2230 | |
| BD FACSCantoII | BD Bioscience | ||
| bovine serum albumin | VWR Life Science AMRESCO | Am-O332-0.1 | |
| Calcium chloride, anhydrous, powder, ≥97% | Sigma Aldrich | C4901-100G | |
| Cary Eclipse Fluorescence Spectrometer | Agilent | ||
| D-(+)-Glucose | Sigma Aldrich | G7528-1KG | |
| DiIC16(3) (1,1'-Dihexadecyl-3,3,3',3'-Tetramethylindocarbocyanine Perchlorate) | Thermo Fisher Scientific | D384 | |
| DMSO | Sigma Aldrich | D8418 | |
| EDTA disodium salt | VWR Life Science AMRESCO | Am-O105B-0.1 | |
| FACSDiva | BD Bioscience | cytometry data acquisition software | |
| FlowJo | Tree Star | cytometer software for data analysis | |
| HEPES | Sigma Aldrich | H4034-500G | |
| Human Factor X | Enzyme research | HFX 1010 | |
| Hydroxylamine hydrochloride | Panreac | 141914.1209 | |
| L-α-phosphatidylcholine (Brain, Porcine) | Avanti Polar Lipids | 840053P | |
| L-α-phosphatidylserine (Brain, Porcine) (sodium salt) | Avanti Polar Lipids | 840032P | |
| Magnesium chloride | Sigma Aldrich | M8266-100G | |
| Mini-Extruder | Avanti Polar Lipids | 610020-1EA | |
| OriginPro 8 SR4 v8.0951 | OriginLab Corporation | Statistical software | |
| Phosphate Buffered Saline (PBS) Tablets, Biotechnology Grade | VWR Life Science AMRESCO | 97062-732 | |
| Potassium chloride | Sigma Aldrich | P9541-500G | |
| Prostaglandin E1 | Cayman Chemical | 13010 | |
| Sephadex G25 | GE Healthcare | GE17-0033-01 | gel filtration medium for protein purification |
| Sepharose CL-2B | Sigma Aldrich | CL2B300-500ML | gel filtration medium for platelet purification |
| Sodium bicarbonate | Corning | 61-065-RO | |
| Sodium chloride | Sigma Aldrich | S3014-500G | |
| Sodium phosphate monobasic | Sigma Aldrich | S3139-250G | |
| Spin collumns with membrane 0.2 µm | Sartorius | VS0171 | |
| Trisodium citrate dihydrate | Sigma Aldrich | S1804-1KG |
Referencias
- Hoffman, M., Monroe, D. M. A cell-based model of hemostasis. Thrombosis and haemostasis. 85 (6), 958-965 (2001).
- Roberts, H. R., Hoffman, M., Monroe, D. M. A cell-based model of thrombin generation. Seminars in Thrombosis and Hemostasis. 32, 32-38 (2006).
- Panteleev, M. A., Dashkevich, N. M., Ataullakhanov, F. I. Hemostasis and thrombosis beyond biochemistry: roles of geometry, flow and diffusion. Thrombosis Research. 136 (4), 699-711 (2015).
- Podoplelova, N. A., et al. Hysteresis-like binding of coagulation factors X/Xa to procoagulant activated platelets and phospholipids results from multistep association and membrane-dependent multimerization. Biochimica et Biophysica Acta. 1858 (6), 1216-1227 (2016).
- Panteleev, M. A., Ananyeva, N. M., Greco, N. J., Ataullakhanov, F. I., Saenko, E. L. Two subpopulations of thrombin-activated platelets differ in their binding of the components of the intrinsic factor X-activating complex. Journal of Thrombosis and Haemostasis. 3 (11), 2545-2553 (2005).
- Kotova, Y., et al. Binding of coagulation factor XIII zymogen to activated platelet subpopulations: roles of integrin αIIbβ3 and fibrinogen. Thrombosis and Haemostasis. 119 (6), 906-915 (2019).
- Fay, S. P., Posner, R. G., Swann, W. N., Sklar, L. A. Real-time analysis of the assembly of ligand, receptor, and G protein by quantitative fluorescence flow cytometry. Biochemistry. 30 (20), 5066-5075 (2002).
- Nolan, J. P., Shen, B., Park, M. S., Sklar, L. A. Kinetic analysis of human flap endonuclease-1 by flow cytometry. Biochemistry. 35 (36), 11668-11676 (1996).
- Sarvazyan, N. A., Lim, W. K., Neubig, R. R. Fluorescence analysis of receptor−G protein interactions in cell membranes. Biochemistry. 41 (42), 12858-12867 (2002).
- Sarvazyan, N. A., Neubig, R. R. Analysis of molecular assemblies by flow cytometry: determinants of Gi1 and by binding. Advances in Optical Biophysics. 3256, 122-131 (1998).
- De Franceschi, N., et al. ProLIF - Quantitative integrin protein-protein interactions and synergistic membrane effects on proteoliposomes. Journal of Cell Science. 132 (4), (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados