Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Charakterisierung der Salmonella Typhimurium-induzierten septischen Peritonitis bei Mäusen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Induktion einer gramnegativen monobakteriellen Sepsis in einem Mausmodellsystem. Das Modell ist nützlich bei der Untersuchung der entzündlichen und tödlichen Wirtsreaktionen während der Sepsis.
Zusammenfassung
Sepsis ist eine fehlregulierte Immunantwort des Wirts auf mikrobielle Invasion oder Gewebeschäden, die zu Organverletzungen an einer Stelle führt, die von der der Infektion oder des Schadens entfernt ist. Derzeit umfassen die weit verbreiteten Mäusemodelle der Sepsis Lipopolysaccharid (LPS)-induzierte Endotoxämie, cecal ligation and puncture (CLP) und monobakterielle Infektionsmodellsysteme. Dieses Protokoll beschreibt eine Methode zur Untersuchung der Wirtsreaktionen während der durch Salmonella Typhimurium-Infektion induzierten septischen Peritonitis bei Mäusen. S. Typhimurium, ein gramnegativer intrazellulärer Erreger, verursacht bei Mäusen eine typhusähnliche Erkrankung.
Dieses Protokoll entwickelt die Kulturvorbereitung, die Induktion einer septischen Peritonitis bei Mäusen durch intraperitoneale Injektion und Methoden zur Untersuchung systemischer Wirtsreaktionen. Weiterhin wird die Beurteilung der Keimbelastung in verschiedenen Organen und die durchflusszytometrische Analyse erhöhter Neutrophilenzahlen in der Peritonealspülung vorgestellt. Salmonelle Typhimurium-induzierte Sepsis bei Mäusen führt zu einem Anstieg der proinflammatorischen Zytokine und einer schnellen Infiltration von Neutrophilen in der Peritonealhöhle, was zu einem geringeren Überleben führt.
Jeder Schritt in diesem Protokoll wurde optimiert, was zu einer hohen Reproduzierbarkeit der Pathogenese der septischen Peritonitis führt. Dieses Modell ist nützlich für die Untersuchung immunologischer Reaktionen während bakterieller Sepsis, der Rolle verschiedener Gene im Krankheitsverlauf und der Auswirkungen von Medikamenten zur Abschwächung der Sepsis.
Einleitung
Sepsis ist definiert als eine fehlregulierte systemische Entzündungs- und Immunantwort auf mikrobielle Invasion oder Gewebeschäden, die zu Organverletzungen führen, die weit vom Ort der Infektion oder Schädigung entfernt sind. Der septische Schock ist eine Untergruppe der Sepsis, die durch eine Hypotonie gekennzeichnet ist, die während der Volumenreanimation anhält, mit einem wesentlich erhöhten Mortalitätsrisiko1. Die breite Öffentlichkeit ist sich dieser Störung während der COVID-19-Pandemie bewusster geworden. Trotz der hohen assoziierten Mortalität fehlen aufgrund der Komplexität der Diagnose umfassende epidemiologische Daten zur globalen Belastung durch Sepsis. Im Jahr 2017 gab es weltweit 48,9 Millionen Sepsis-Inzidenzen und 11 Millionen Todesfälle, was 19,7% aller weltweiten Todesfälleentspricht2. Darüber hinaus ergab eine Studie über die erweiterte Prävalenz von Infektionen und damit verbundenen Sepsis bei Patienten auf der Intensivstation, dass 62% der positiven Isolate von Patienten gramnegative Organismenwaren 3.
Zunächst konzentrierten sich die Untersuchungen zur Sepsis auf die Abgrenzung der mikrobiellen Pathogenese. Das Verständnis der "Gefahrenhypothese", die diktiert, wie der Wirt das Selbst und das Nicht-Selbst unterscheidet, führte jedoch dazu, dass das Gleichgewicht der Sepsisforschung in Richtung Verständnis der Reaktion des Wirts auf einen eindringenden Krankheitserreger kippte. Zu den weit verbreiteten Mäusemodellen der Sepsis gehören das Lipopolysaccharid (LPS)-induzierte Endotoxämiemodell, polymikrobielle Sepsismodelle, cecal ligation and puncture (CLP) und colon ascendens stent peritonitis (CASP) und monobakterielle Infektionsmodelle4.
Wir haben ein Mausmodellsystem standardisiert, indem wir eine Peritoneussepsis mit Salmonella Typhimurium induzieren. Dieses Modell ist vorteilhaft gegenüber anderen, da Salmonella Typhimurium ein intrazellulärer Erreger ist, der den klinisch relevanten Zustand der gramnegativen Sepsis nachahmt. Das Ergebnis der Peritonitis-Sepsis in diesem Modell ist systemisch, mit 100% Mortalität innerhalb von 96 h nach der Infektion. Daher ist dieses Modell maßgeblich an der Untersuchung der entzündlichen und tödlichen Wirtsreaktionen beteiligt. In diesem Modell wird Sepsis induziert, indem intraperitoneal 0,5 Millionen koloniebildende Einheiten (CFU) Salmonella Typhimurium in eine 8-10 Wochen alte C57BL / 6-Maus injiziert werden. Eine systemische Infektion kann durch die Beurteilung der Organbakterienbelastung ~ 16 h nach der Infektion bestätigt werden. Dieser Artikel zeigt die Salmonella Typhimurium-induzierte Peritonitis-Sepsis bei Mäusen, charakterisiert die daraus resultierenden Veränderungen in der peritonealen Zellzusammensetzung und quantifiziert die bakterielle Belastung in verschiedenen Organen.
Protokoll
Alle Experimente mit Salmonella Typhimurium wurden in Einrichtungen der Biosicherheitsstufe 2 (BSL-2) durchgeführt. Es muss darauf geachtet werden, dass eine geeignete persönliche Schutzausrüstung (PSA) verwendet, die Sicherheit gewährleistet und die Standardmethoden zur Entsorgung von BSL-2-Biogefahren befolgt werden. Alle Mäuseexperimente wurden nach den Richtlinien des Institutional Animal Ethics Committee, IISc, durchgeführt. Mäuse wurden in der Central Animal Facility von IISc (Registrierungsnummer: 48/1999/CPCSEA, vom 1.3.1999) gezüchtet und gepflegt, die vom Ministerium für Umwelt und Wald der indischen Regierung genehmigt wurde. Die Versuchsprotokolle wurden vom Committee for Purpose and Control and Supervision of Experiments on Animals mit der genehmigten Genehmigungsnummer CAF/Ethics/797/2020 genehmigt.
BSL2-Definition: Eine BSL2-Einstufung bedeutet, dass die biogefährlichen Wirkstoffe eine moderate Bedrohung für die Umwelt und das Laborpersonal darstellen5.
1. Kulturzubereitung von Salmonella Typhimurium
- 100 μL Salmonella Typhimurium NCTC 12023 Glycerinbrühe in 3 ml Luria Bertani (LB) Brühe geben. Inkubieren Sie die Kultur bei 160 U / min bei 37 ° C über Nacht.
- 50 μL der über Nacht angebauten Kultur in LB-Brühe auf eine Salmonella Shigella (SS) Agarplatte streichen und bei 37 °C für ~12 h inkubieren. Lagern Sie die SS-Agarplatte mit den Bakterienkolonien vor dem In-vivo-Infektionsexperiment mehrere Tage bei 4 °C.
- Wählen Sie eine einzelne Kolonie von der gestreiften SS-Agarplatte mit einer Mikrospitze aus. Werfen Sie die Mikrospitze über Nacht in 3 ml LB-Brühe und Kultur bei 160 U / min bei 37 ° C.
- Fügen Sie 0,1 ml der Bakterienkultur zu 50 ml LB-Brühe hinzu und inkubieren Sie bei 37 ° C in einem Shaker-Inkubator bei 160 U / min für 3-4 h, um die logarithmische Wachstumsphase zu erreichen. Verdünnen Sie die Kultur um den Faktor 2 mit LB-Brühe.
HINWEIS: Während der logarithmischen Phase sind Bakterienzellen bei bester Gesundheit und teilen sich aktiv. - Messen Sie die optische Dichte (OD) der Kultur bei 600 nm Wellenlänge des Lichts in einem Spektralphotometer oder Mikroplattenleser. Sobald die OD 1,0 erreicht hat, machen Sie zwei Aliquots von 1 ml Kultur in 1,5 ml Mikrofugenröhrchen.
- Zentrieren Sie die Röhrchen bei 7.750 × g für 15 min. Entsorgen Sie den Überstand und waschen Sie das Pellet mit 1 ml 1x PBS 2x. Zentrifen Sie die Röhrchen bei 7.750 × g für 15 min.
- Resuspendiert das Pellet in 0,5 ml von 1x PBS in zwei verschiedenen 1,5 ml Mikrofugenröhrchen. Kombinieren Sie die Suspensionen aus beiden Rohren zu einem 1,5-ml-Rohr, das jetzt ~ 2 × 108 koloniebildende Einheiten (CFU) / ml enthält.
- Bereiten Sie eine bakterielle Zellsuspension von 1 × 106 KBE/ml vor, indem Sie diese Stammlösung verdünnen.
ACHTUNG: Optimieren Sie die CFU, die OD entspricht, unter bestimmten Laborbedingungen, um die CFU für OD 1.0 zu bestimmen, bevor Sie mit den Experimenten beginnen.
2. Mäuse und Infektionen
- Haus 8-10 Wochen alte männliche C57BL / 6 Mäuse mit einem Gewicht von ~ 20 g im Reinluftraum der Tieranlage für mehrere Tage zur Akklimatisierung.
- Halten Sie am Tag der Infektion die Maus mit einer Hand, wischen Sie die Bauchhaut mit 70% Ethanol ab und spreizen Sie die Hinterbeine für eine bessere Zugänglichkeit der Bauchdecke.
- Injizieren Sie 0,5 ml 1 × 106 KBE/ml Bakteriensuspension intraperitoneal mit Hilfe einer 1 ml Spritze. Daher erhält jede Maus 5 × 105 KBE. Kontrollieren Sie, nicht infizierte Mäuse erhalten allein 0,5 ml PBS. Nach der Infektion die Kultur zur Überprüfung der tatsächlich injizierten CFU auffüllen, die zwischen 0,2 und 0,8 Millionen KBE / 0,5 ml variieren kann.
- Legen Sie die Mäuse wie zugewiesen wieder in die Käfige.
- Opfern Sie die Mäuse mit CO2 Asphyxiation ~ 12-18 h nach der Infektion für die beste Reaktion. Normalerweise sterben alle infizierten Mäuse innerhalb von 96 h. Unter einigen experimentellen Interventionen können einige Mäuse überleben. Euthanasieren Sie diese Mäuse nach 96 h. Euthanasieren Sie auch jede Maus mit einer Körpertemperatur unter 33,2 ° C und akutem Stress bei 96 h als humanen Endpunkt.
HINWEIS: In diesem Modell können einige Mäuse nach 12 Stunden Salmonelleninjektion zu sterben beginnen. Planen Sie daher ordnungsgemäß Experimente mit mehreren Zeitpunkten.
3. Bewertung von Organen durch die KBE
- Opfern Sie die infizierte Maus durch CO 2-Ersticken und wischen Sie den Bauch mit einem Stück Baumwolle ab, das in 70% Ethanol getaucht ist. Schneiden Sie die Bauchhaut auf. Im Artikel von Ray und Dittel finden Sie ein Videoprotokoll zum Sammeln von Peritonealspülflüssigkeit6. Schneiden Sie die Peritonealhöhle auf und sammeln Sie die interessierenden Organe in Mikrofugenröhrchen. Da das Blut bei Mäusen mit Sepsis schnell koaguliert wird und die Mengen niedrig sind, sammeln Sie es nach dem Opfer schnell.
HINWEIS: Dieses Video zeigt die Aufzählung von Organ-CFU aus der Leber, da die Leber in diesem Sepsismodell umfangreiche histopathologische Schäden erleidet. - Schneiden Sie ein kleines Stück der Leber und legen Sie es in einen Mikrofugenschlauch.
HINWEIS: Diese kann maximal 2-3 h auf Eis gelagert werden, bevor mit dem nächsten Schritt fortgefahren wird. - Wiegen Sie die Teile und geben Sie sie in Mikrozentrifugenröhrchen. Vorzugsweise schneiden Sie Stücke mit einem Gewicht von ~ 10-15 mg für eine ordnungsgemäße Homogenisierung. Verwenden Sie ganze Organe bei kleineren wie den mesenterialen Lymphknoten (MLNs) oder dem Thymus.
- Geben Sie 0,5 ml 1x PBS in das Röhrchen und homogenisieren Sie die Organe mit einem Handhomogenisator. Stellen Sie sicher, dass die Organe vollständig homogenisiert sind. Erhöhen Sie die Lautstärke auf 1 ml, indem Sie 0,5 ml von 1x PBS hinzufügen.
- Die Röhrchen bei 200 × g für 5 min bei 4 °C zentrifugieren.
- Sammeln Sie den Überstand in frischen Mikrofugenröhrchen und bereiten Sie Verdünnungen von 1 × 10−1 und 10−2 in einer 96-Well-Platte vor.
- 50 μL des Verdünnungsmittels auf frische SS-Agarplatten verteilen und die Platten bei 37 °C für 12 h inkubieren.
- Zählen Sie die Anzahl der Kolonien, die in jeder Bedingung auftreten, und normalisieren Sie die Daten mit dem Organgewicht mit Gleichung (1):
KBE/mg =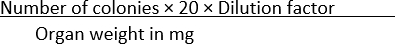 (1)
(1)
HINWEIS: Die Zahl 20 wird in der Formel verwendet, um die Kolonien pro Platte in CFU/ml umzurechnen. Diese Zahl ergibt sich, indem man 1 ml durch die Menge eines gegebenen Volumens der Kulturplattierung - in diesem Fall 50 μL - dividiert.
Wenn beispielsweise 100 Kolonien in einer SS-Agarplatte gefunden werden, in der 50 μL einer 1 × 10-1 Verdünnung eines homogenisierten Organs mit einem Gewicht von 10 mg verteilt sind, dann gilt
KBE/mg =
4. Durchflusszytometrische Analyse verschiedener Immunzellpopulationen in Peritonealexsudat
- Sammeln Sie die Peritonealzellen, wie zuvor von Ray und Dittel6 beschrieben.
- Resuspendiert das Zellpellet aus peritonealer Lavageflüssigkeit in 1 ml RPMI, ergänzt mit 10% fetalem Rinderserum (FBS). Zählen Sie die Gesamtzahl der Zellen in der Peritonealspülung mit einem Hämozytometer auf. Passen Sie die Zellzahl so an, dass jede Röhre 0,2-0,5 Millionen Zellen erhält.
HINWEIS: Peritoneale Lavage bei Mäusen mit Sepsis kann RBCs enthalten, die wahrscheinlich aufgrund von Blutungen auftreten. Achten Sie darauf, RBCs auszuschließen, während Sie Peritonealzellen zählen. Im Hellfeldmikroskop erscheinen Erythrozyten viel kleiner als die Immunzellen. Diese erscheinen als flache Scheiben oder Donuts, rund, mit einer Vertiefung in der Mitte, aber nicht hohl. Ein Schritt zur Lyse von RBCs kannhinzugefügt werden 7. - Drehen Sie die Zellen bei 200 × g bei 4 °C für 10 min nach unten, entsorgen Sie den Überstand und waschen Sie die Zellen 1x mit 1x kaltem PBS. Zentrifen Sie die Zellen bei 200 × g bei 4 °C für 10 min.
- Blockieren Sie die FC-Rezeptoren auf PECs mit FcR-Blocker (1:400 Verdünnung), hergestellt in Blockpuffer, bestehend aus 5% FBS und 0,02% Natriumazid in PBS. 15 min auf Eis inkubieren.
- Zentrifugieren Sie die Zellen bei 200 × g bei 4 °C für 10 min. Verwerfen Sie den Überstand. Verdünnen Sie die fluorochromkonjugierten Antikörper, die im Blockpuffer von Interesse sind. Verwenden Sie eine 1:500-Verdünnung von Anti-Maus-LY6G, um die Neutrophilen zu färben.
HINWEIS: Andere Immunzellpopulationen können ebenfalls nachgewiesen werden, z. B. mit Anti-Maus-B220 für B-Zellen, Anti-Maus-CD3 für T-Zellen und Anti-Maus-F4/80 für Makrophagen. - Inkubieren Sie ~ 0,2 Millionen Zellen in 200 μL der verdünnten Lösungen von Antikörpern in separaten Röhrchen. Stellen Sie als Negativkontrolle ein Röhrchen in jedem Fluorochromtyp für die ungefärbte Kontrolle zur Verfügung, um die Zellen mit 200 μL blockierendem Puffer ohne Antikörper zu inkubieren.
- Brüten Sie die Proben auf Eis für 45 Minuten mit intermittierendem Klopfen alle 15 Minuten aus.
- Zentrifugieren Sie die Zellen bei 200 × g für 10 min bei 4 °C. Verwerfen Sie den Überstand. Fixieren Sie die Zellen mit 4% Paraformaldehyd für ~ 15 min bei Raumtemperatur, wenn sie mehrere Tage gelagert werden müssen. Am besten ist es jedoch, Daten von frisch gefärbten Proben in einem Durchflusszytometer zu erfassen.
- Resuspendieren Sie die Zellen in 200 μL FACS-Färbepuffer (2% FBS in PBS). Erfassen Sie die Daten in einem Durchflusszytometer.
Ergebnisse
Eine detaillierte Charakterisierung der Immunantwort des Wirts mit diesem speziellen Modell ist in früheren Publikationengezeigt 8,9. Einige repräsentative Ergebnisse des beschriebenen Protokolls werden in diesem Abschnitt dargestellt. Dieses Modell zielt darauf ab, eine systemische Infektion von S zu induzieren. Typhimurium durch intraperitoneale Injektion der Bakterienkultur zur Induktion einer Sepsis. Um die Infektion zu bestätigen, wurden die Lysa...
Diskussion
Dieser Artikel beschreibt eine Methode zur Induktion einer schweren Form der bakteriellen Sepsis durch intraperitoneale Injektion von Salmonella Typhimurium. Dieses Modell ist vorteilhaft gegenüber anderen, da Salmonella Typhimurium ein intrazellulärer Erreger und daher hoch pathogen ist und den klinisch relevanten Zustand der gramnegativen Sepsis nachahmt. Das Ergebnis der Peritonitis-Sepsis in diesem Modell ist systemisch, mit 100% Mortalität innerhalb von 96 h nach der Infektion. Daher ist dieses ...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken der Central Animal Facility, IISc, für die Bereitstellung von Mäusen für die Forschung. Diese Studie wurde durch Zuschüsse an DpN vom Department of Biotechnology and Science and Engineering Research Board der indischen Regierung finanziert. Die infrastrukturelle Unterstützung durch das DBT-IISc-Programm und die DST-FIST-Zuschüsse werden sehr anerkannt. Wir danken allen bisherigen und aktuellen Mitgliedern des DpN-Labors für ihre Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| 1 mL Sterile Syringe with 26 G needle | Beckton Dickinson, Singapore | 303060 | |
| 1.5 mL Microcentrifuge Tube | Tarsons, USA | 500010 | |
| 10 mL Sterile Syringe with 21 G needle | Beckton Dickinson, Spain | 307758 | |
| 50 mL Conical Flask | Tarsons, USA | 441150 | |
| 50 mL Graduated Centrifuge Tube | Tarsons, USA | 546041 | |
| 50 mL Graduated Centrifuge Tube | Tarsons, USA | 546021 | |
| Cell spreader | VWR, USA | VWRU60828-680 | |
| Dulbecco’s Phosphate Buffered Saline | HiMedia, Mumbai, India | TS1006 | |
| Ethanol | Merck | 100983 | |
| FcR blocker | BD Biosciences | 553142 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| FITC Rat anti-mouse Ly6G (Clone 1A8) | BD Pharmingen | 551460 | |
| Glycerol | Sigma-Aldrich | G9012 | |
| Hand based Homogenizer | - | - | |
| Hemocytometer (Neubauer counting chamber) | Rohem, India | I.S. 10269 | |
| Luria Bertani Broth | HiMedia, Mumbai, India | M1245 | |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Petriplates | Tarsons, USA | 460091 | |
| RPMI | Himedia, Mumbai, India | AT060-10X1L | |
| Salmonella-Shigella Agar | HiMedia, Mumbai, India | M108 | |
| Sodium azide | Sigma-Aldrich | S2002 | |
| Equipments | |||
| Centrifuge | Kubota | ||
| Flow cytometer | BD FACSverse | ||
| Incubator | N-biotek | ||
| Spectrophotometer | Shimadzu | ||
| Weighing machine | Sartorius |
Referenzen
- Hotchkiss, R. S., et al. Sepsis and septic shock. Nature Reviews Disease Primers. 2 (1), 1-21 (2016).
- Rudd, K. E., et al. regional, and national sepsis incidence and mortality, 1990-2017: Analysis for the Global Burden of Disease Study. The Lancet. 395 (10219), 200-211 (2020).
- Vincent, J. L., et al. International study of the prevalence and outcomes of infection in intensive care units. JAMA. 302 (21), 2323-2329 (2009).
- Lewis, A. J., Seymour, C. W., Rosengart, M. R. Current murine models of sepsis. Surgical Infections. 17 (4), 385-393 (2016).
- Ta, L., Gosa, L., Nathanson, D. A. Biosafety and biohazards: Understanding biosafety levels and meeting safety requirements of a biobank. Biobanking. 1897, 213-225 (2019).
- Ray, A., Dittel, D. N. Isolation of Mouse Peritoneal Cavity Cells. Journal of Visualized Experiments: JoVE. (35), e1488 (2010).
- Liu, X., Quan, N. Immune cell isolation from mouse femur bone marrow. Bio-protocol. 5 (20), 1631 (2015).
- Yadav, S., et al. Nitric oxide synthase 2 enhances the survival of mice during Salmonella Typhimurium infection-induced sepsis by increasing reactive oxygen species, inflammatory cytokines and recruitment of neutrophils to the peritoneal cavity. Free Radical Biology & Medicine. 116, 73-87 (2018).
- Verma, T., et al. Cell-free hemoglobin is a marker of systemic inflammation in mouse models of sepsis: A Raman spectroscopic study. Analyst. 146 (12), 4022-4032 (2021).
- Cassado, A. D. A., Lima, M. R. D., Bortoluci, K. R. Revisiting mouse peritoneal macrophages: Heterogeneity, development, and function. Frontiers in Immunology. 6, 225 (2015).
- Yadav, S., Verma, T., Chattopadhyay, A., Nandi, D. Factors affecting the pathophysiology of sepsis, an inflammatory disorder: Key roles of oxidative and nitrosative stress. Indian Journal of Inflammation Research. 3 (1), 2 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten