É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Caracterizando Salmonella Typhimurium-induced Septic Peritonitis em camundongos
Neste Artigo
Resumo
Este protocolo descreve a indução da sepse monobacteriana Gram-negativa em um sistema modelo de rato. O modelo é útil na investigação das respostas inflamatórias e letais do hospedeiro durante a sepse.
Resumo
A sepse é uma resposta imune disregulada ao dano microbiano ou de tecido, levando a lesões de órgãos em um local distante do da infecção ou dano. Atualmente, os modelos de sepse amplamente utilizados incluem endotoxemia induzida por lipopolípido (LPS), ligante cecal e punção (CLP), e sistemas modelo de infecção monobacteriana. Este protocolo descreve um método para estudar as respostas do hospedeiro durante a peritonite séptica induzida por infecção por Salmonella Typhimurium em camundongos. S. Typhimurium, um patógeno intracelular gram-negativo, causa doença semelhante a tifoide em camundongos.
Este protocolo elabora a preparação da cultura, a indução de peritonite séptica em camundongos através de injeção intraperitoneal e métodos para estudar respostas sistêmicas do hospedeiro. Além disso, apresenta-se a avaliação da carga bacteriana em diferentes órgãos e a análise citométrica do fluxo do aumento do número de neutrófilos na lavagem peritoneal. Salmonella A sepse induzida por titriério em camundongos leva a um aumento de citocinas proinflamatórias e à rápida infiltração de neutrófilos na cavidade peritoneal, levando a uma menor sobrevivência.
Cada passo deste protocolo foi otimizado, resultando em alta reprodutibilidade da patogênese da peritonite séptica. Este modelo é útil para estudar respostas imunológicas durante a sepse bacteriana, os papéis de diferentes genes na progressão da doença e os efeitos das drogas para atenuar a sepse.
Introdução
A sepse é definida como uma resposta inflamatória sistêmica disregulada e imune à invasão microbiana ou danos teciduais, levando à lesão de órgãos distante do local da infecção ou dano. O choque séptico é um subconjunto de sepse caracterizado pela hipotensão que persiste durante a ressuscitação do volume, com um risco substancialmente aumentado de mortalidade1. O público em geral tornou-se mais consciente desse transtorno durante a pandemia COVID-19. Apesar de sua alta mortalidade associada, faltam dados epidemiológicos abrangentes sobre a carga global da sepse devido à complexidade de seu diagnóstico. Em 2017, houve 48,9 milhões de incidências de sepse e 11 milhões de mortes em todo o mundo, representando 19,7% de todas as mortes globais2. Além disso, um estudo sobre a prevalência prolongada de infecção e sepse relacionada em pacientes de unidade de terapia intensiva constatou que 62% dos isolados positivos dos pacientes eram organismos Gram-negativos3.
Inicialmente, as investigações sobre sepse se concentraram na delineamento da patogênese microbiana. No entanto, compreender a "hipótese de perigo", que dita como o hospedeiro distingue a si mesmo e a si mesmo, levou à inclinação do equilíbrio da pesquisa de sepse para entender a resposta do hospedeiro a um patógeno invasor. Os modelos de sepse amplamente utilizados incluem o modelo de endotoxemia induzido por lipopolípido (LPS), modelos de sepse polimicrobiana, ligação cecal e punção (CLP) e peritonite stent ascendentes de cólon (CASP), e modelos de infecção monobacteriana4.
Padronizamos um sistema de modelo de camundongos induzindo sepse peritoneal usando Salmonella Typhimurium. Este modelo é vantajoso em relação aos outros porque Salmonella Typhimurium é um patógeno intracelular que imita a condição clinicamente relevante da sepse Gram-negativa. O resultado da peritonite sepse nesse modelo é sistêmico, com 100% de mortalidade dentro de 96 h pós infecção. Portanto, este modelo é fundamental no estudo das respostas inflamatórias e letais do hospedeiro. Neste modelo, a sepse é induzida por injeção intraperitoneal de 0,5 milhões de unidades formadoras de colônias (UFC) de Salmonella Typhimurium em um rato C57BL/6 de 8-10 semanas de idade. A infecção sistêmica pode ser confirmada avaliando a carga bacteriana dos órgãos ~16 h após a infecção. Este artigo demonstra salmonella typhimurium-induced peritonitis sepse em camundongos, caracteriza as alterações resultantes na composição celular peritoneal, e quantifica a carga bacteriana em diferentes órgãos.
Protocolo
Todos os experimentos utilizando Salmonella Typhimurium foram realizados nas instalações do Nível de Bio Segurança 2 (BSL-2). Deve-se tomar cuidado para utilizar equipamentos de proteção individual (EPI) adequados, garantir a segurança e seguir os métodos padrão de descarte de risco biológico BSL-2. Todos os experimentos com camundongos foram realizados seguindo diretrizes do Comitê de Ética Animal Institucional, IISc. Os camundongos foram criados e mantidos no Centro de Animais do IISc (Número de registro: 48/1999/CPCSEA, datado de 3/1/1999), aprovado pelo Ministério do Meio Ambiente e Floresta, Governo da Índia. Os protocolos experimentais foram aprovados pela Comissão de Propósito e Controle e Supervisão de Experimentos em Animais com o número de autorização aprovado CAF/Ética/797/2020.
Definição BSL2: Uma classificação BSL2 representa que os agentes de risco biológico representam uma ameaça moderada ao meio ambiente e à equipe de laboratório5.
1. Preparação cultural de Salmonella Typhimurium
- Adicione 100 μL de Salmonella Typhimurium NCTC 12023 glicerol a 3 mL de caldo de Luria Bertani (LB). Incubar a cultura a 160 rpm a 37 °C durante a noite.
- Raia 50 μL da cultura cultivada durante a noite em caldo LB em uma placa de ágar Salmonella Shigella (SS) e incubar a 37 °C por ~12 h. Armazene a placa de ágar SS com as colônias bacterianas a 4 °C por vários dias antes do experimento de infecção in vivo .
- Escolha uma única colônia da placa de ágar SS listrada usando uma microtipa. Ejete a microfina em 3 mL de caldo LB e cultura a 160 rpm a 37 °C durante a noite.
- Adicione 0,1 mL da cultura bacteriana a 50 mL de caldo LB e incubar a 37 °C em uma incubadora shaker a 160 rpm por 3-4 h para alcançar a fase logarítmica de crescimento. Diluir a cultura por um fator de 2 usando caldo LB.
NOTA: Durante a fase logarítmica, as células bacterianas estão em sua melhor saúde e estão se dividindo ativamente. - Meça a densidade óptica (OD) da cultura a 600 nm de comprimento de onda de luz em um leitor de espectrofotômetro ou microplaca. Uma vez que o OD atinja 1.0, faça duas alíquotas de 1 mL de cultura em tubos de microfuge de 1,5 mL.
- Centrifugar os tubos a 7.750 × g por 15 min. Descarte o supernatante e lave a pelota com 1 mL de 1x PBS 2x. Centrifugar os tubos a 7.750 × g por 15 min.
- Resuspenja a pelota em 0,5 mL de 1x PBS em dois tubos diferentes de microfuça de 1,5 mL. Combine as suspensões de ambos os tubos em um tubo de 1,5 mL agora contendo ~2 × 108 unidades formadoras de colônias (CFU)/mL.
- Prepare uma suspensão celular bacteriana de 1 × 106 UFC/mL diluindo esta solução de estoque.
ATENÇÃO: Otimize a UFC correspondente ao OD em condições laboratoriais específicas para determinar a UFC para OD 1.0 antes de iniciar os experimentos.
2. Camundongos e infecções
- Casa de 8-10 semanas macho C57BL/6 ratos pesando ~20 g na sala de ar limpo da instalação animal por vários dias para aclimatação.
- No dia da infecção, segure o rato com uma mão, limpe a pele abdominal com 70% de etanol e espalhe as patas traseiras para melhor acessibilidade da parede abdominal.
- Injete 0,5 mL de 1 × suspensão bacteriana 106 CFU/mL intraperitonealmente com a ajuda de uma seringa de 1 mL. Portanto, cada rato recebe 5 × 105 UFC. Controle, camundongos não infectados recebem 0,5 mL apenas de PBS. Pós-infecção, emplaque a cultura para verificar a CFU real injetada, que pode variar de 0,2-0,8 milhões de UFC/0,5 mL.
- Coloque os ratos de volta nas gaiolas como atribuído.
- Sacrifique os camundongos usando asfixia de CO2 ~12-18 h de infecção pós para obter a melhor resposta. Normalmente, todos os camundongos infectados morrem dentro de 96 h. Sob algumas intervenções experimentais, alguns ratos podem sobreviver. Eutanize esses ratos depois das 96 h. Além disso, eutanize qualquer rato com uma temperatura corporal abaixo de 33,2 °C e angústia aguda a 96 h como um ponto final humano.
NOTA: Neste modelo, alguns camundongos podem começar a morrer após 12 h de injeção de Salmonella . Portanto, planeje experimentos adequadamente envolvendo vários pontos de tempo.
3. Avaliação da UFC dos órgãos
- Sacrifique o rato infectado por asfixia de CO2 , e limpe o abdômen com um pedaço de algodão mergulhado em 70% de etanol. Abra a pele abdominal. Consulte o artigo de Ray e Dittel para um protocolo de vídeo sobre como coletar fluido de lavage peritoneal6. Abra a cavidade peritoneal e colete os órgãos de interesse em tubos de microfuge. Além disso, à medida que o sangue em camundongos com sepse é coagulado rapidamente e as quantidades são baixas, colecione-o rapidamente após o sacrifício.
NOTA: Este vídeo demonstra a enumeração do órgão CFU do fígado à medida que o fígado sofre extensos danos histopatológicos neste modelo de sepse. - Corte um pequeno pedaço do fígado e coloque-o em um tubo de microfuça.
NOTA: Isso pode ser armazenado no gelo por um máximo de 2-3 horas antes de prosseguir para a próxima etapa. - Pesar e transferir as peças para tubos de microcentrifuuagem. De preferência, corte peças pesando ~10-15 mgs para homogeneização adequada. Use órgãos inteiros no caso de menores, como os linfonodos mesentéricos (MLNs) ou timo.
- Adicione 0,5 mL de 1x PBS ao tubo e homogeneize os órgãos usando um homogeneizador de mão. Certifique-se de que os órgãos estão completamente homogeneizados. Coma o volume de 1 mL adicionando 0,5 mL de 1x PBS.
- Centrifugar os tubos a 200 × g por 5 min a 4 °C.
- Colete o supernatante em tubos de microfuge frescos e prepare diluições de 1 × 10−1 e 10-2 em uma placa de 96 poços.
- Espalhe 50 μL do diluído em placas frescas de ágar SS e incubar as placas a 37 °C por 12 h.
- Conte o número de colônias que aparecem em cada condição e normalize os dados com o peso do órgão usando Equação (1):
CFU/mg =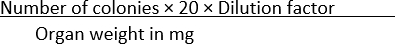 (1)
(1)
NOTA: O número 20 é usado na fórmula para converter as colônias por placa em CFU/mL. Este número é chegado dividindo 1 mL pela quantidade de um determinado volume da cultura banhada neste caso, 50 μL.
Por exemplo, se 100 colônias forem encontradas em uma placa de ágar SS, onde 50 μL de uma diluição de 1 × 10-1 de um órgão homogeneizado pesando 10 mgs é espalhado, então
CFU/mg =
4. Análise citométrica de fluxo de várias populações de células imunes em exsudato peritoneal
- Coletar as células peritoneal como descrito anteriormente por Ray e Dittel6.
- Resuspend a pelota celular do fluido de lavage peritoneal em 1 mL de RPMI suplementado com 10% de soro bovino fetal (FBS). Enumerar os números totais de células na lavage peritoneal usando um hemócito. Ajuste o número da célula para que cada tubo receba 0,2-0,5 milhões de células.
NOTA: O lavado peritoneal em camundongos com sepse pode conter RBCs, que provavelmente aparecem devido à hemorragia. Tenha cuidado para excluir RBCs enquanto conta as células peritoneais. No microscópio brightfield, os RBCs parecem muito menores do que as células imunes. Estes aparecem como discos planos ou donuts, redondos, com um recuo no centro, mas não oco. Um passo para lise RBCs pode ser adicionado7. - Gire as células a 200 × g a 4 °C por 10 minutos, descarte o sobrenante e lave as células 1x com PBS 1x frio. Centrifugar as células a 200 × g a 4 °C por 10 min.
- Bloqueie os receptores Fc em PECs usando bloqueador de FcR (diluição 1:400), preparado no buffer de bloqueio composto por 5% de FBS e 0,02% de azida de sódio no PBS. Incubar no gelo por 15 minutos.
- Centrifugar as células a 200 × g a 4 °C por 10 min. Descarte o supernatante. Diluir os anticorpos conjugados fluorocromo de interesse em bloquear o buffer. Use uma diluição de 1:500 do anti-rato LY6G para manchar os neutrófilos.
NOTA: Outras populações de células imunes também podem ser detectadas usando, por exemplo, o anti-rato B220 para células B, CD3 anti-rato para células T e f4/80 anti-rato para macrófagos. - Incubar ~0,2 milhões de células em 200 μL das soluções diluídas de anticorpos em tubos separados. Como controle negativo, reserve um tubo em cada tipo fluorocromo para o controle não manchado para incubar as células com 200 μL de tampão de bloqueio sem anticorpo.
- Incubar as amostras no gelo por 45 minutos com toques intermitentes a cada 15 minutos.
- Centrifugar as células a 200 × g por 10 min a 4 °C. Descarte o supernatante. Fixar as células com 4% de paraformaldeído por ~15 min em temperatura ambiente se precisarem ser armazenadas por vários dias. No entanto, é melhor adquirir dados de amostras recém-manchadas em um citômetro de fluxo.
- Resuspensar as células em 200 μL de tampão de coloração FACS (2% FBS em PBS). Adquira os dados em um citômetro de fluxo.
Resultados
Uma caracterização detalhada da resposta imune do hospedeiro usando este modelo em particular é mostrada em publicações anteriores 8,9. Alguns resultados representativos do protocolo descrito são retratados nesta seção. Este modelo visa induzir a infecção sistêmica de S. Tiquitário por injeção intraperitoneal da cultura bacteriana para induzir a sepse. Para confirmar a infecção, os lises do fígado e do baço de camundongos sépticos fora...
Discussão
Este artigo descreve um método de induzir uma forma grave de sepse bacteriana por injeção intraperitoneal de Salmonella Typhimurium. Este modelo é vantajoso em relação aos outros, pois Salmonella Typhimurium é um patógeno intracelular e, portanto, altamente patogênico, imitando a condição clinicamente relevante da sepse Gram-negativa. O resultado da peritonite sepse nesse modelo é sistêmico, com 100% de mortalidade dentro de 96 h pós infecção. Portanto, esse modelo é fundamental no estu...
Divulgações
Os autores não têm conflitos de interesse para divulgar.
Agradecimentos
Agradecemos ao Centro de Animais, IISc, por nos fornecer ratos para pesquisa. Este estudo foi financiado por subvenções ao DpN do Departamento de Biotecnologia e Ciência e Pesquisa de Engenharia, Governo da Índia. O apoio infra-estrutural do programa DBT-IISc e das subvenções DST-FIST são muito reconhecidos. Agradecemos a todos os membros anteriores e atuais do laboratório dpn pelo apoio.
Materiais
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| 1 mL Sterile Syringe with 26 G needle | Beckton Dickinson, Singapore | 303060 | |
| 1.5 mL Microcentrifuge Tube | Tarsons, USA | 500010 | |
| 10 mL Sterile Syringe with 21 G needle | Beckton Dickinson, Spain | 307758 | |
| 50 mL Conical Flask | Tarsons, USA | 441150 | |
| 50 mL Graduated Centrifuge Tube | Tarsons, USA | 546041 | |
| 50 mL Graduated Centrifuge Tube | Tarsons, USA | 546021 | |
| Cell spreader | VWR, USA | VWRU60828-680 | |
| Dulbecco’s Phosphate Buffered Saline | HiMedia, Mumbai, India | TS1006 | |
| Ethanol | Merck | 100983 | |
| FcR blocker | BD Biosciences | 553142 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| FITC Rat anti-mouse Ly6G (Clone 1A8) | BD Pharmingen | 551460 | |
| Glycerol | Sigma-Aldrich | G9012 | |
| Hand based Homogenizer | - | - | |
| Hemocytometer (Neubauer counting chamber) | Rohem, India | I.S. 10269 | |
| Luria Bertani Broth | HiMedia, Mumbai, India | M1245 | |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Petriplates | Tarsons, USA | 460091 | |
| RPMI | Himedia, Mumbai, India | AT060-10X1L | |
| Salmonella-Shigella Agar | HiMedia, Mumbai, India | M108 | |
| Sodium azide | Sigma-Aldrich | S2002 | |
| Equipments | |||
| Centrifuge | Kubota | ||
| Flow cytometer | BD FACSverse | ||
| Incubator | N-biotek | ||
| Spectrophotometer | Shimadzu | ||
| Weighing machine | Sartorius |
Referências
- Hotchkiss, R. S., et al. Sepsis and septic shock. Nature Reviews Disease Primers. 2 (1), 1-21 (2016).
- Rudd, K. E., et al. regional, and national sepsis incidence and mortality, 1990-2017: Analysis for the Global Burden of Disease Study. The Lancet. 395 (10219), 200-211 (2020).
- Vincent, J. L., et al. International study of the prevalence and outcomes of infection in intensive care units. JAMA. 302 (21), 2323-2329 (2009).
- Lewis, A. J., Seymour, C. W., Rosengart, M. R. Current murine models of sepsis. Surgical Infections. 17 (4), 385-393 (2016).
- Ta, L., Gosa, L., Nathanson, D. A. Biosafety and biohazards: Understanding biosafety levels and meeting safety requirements of a biobank. Biobanking. 1897, 213-225 (2019).
- Ray, A., Dittel, D. N. Isolation of Mouse Peritoneal Cavity Cells. Journal of Visualized Experiments: JoVE. (35), e1488 (2010).
- Liu, X., Quan, N. Immune cell isolation from mouse femur bone marrow. Bio-protocol. 5 (20), 1631 (2015).
- Yadav, S., et al. Nitric oxide synthase 2 enhances the survival of mice during Salmonella Typhimurium infection-induced sepsis by increasing reactive oxygen species, inflammatory cytokines and recruitment of neutrophils to the peritoneal cavity. Free Radical Biology & Medicine. 116, 73-87 (2018).
- Verma, T., et al. Cell-free hemoglobin is a marker of systemic inflammation in mouse models of sepsis: A Raman spectroscopic study. Analyst. 146 (12), 4022-4032 (2021).
- Cassado, A. D. A., Lima, M. R. D., Bortoluci, K. R. Revisiting mouse peritoneal macrophages: Heterogeneity, development, and function. Frontiers in Immunology. 6, 225 (2015).
- Yadav, S., Verma, T., Chattopadhyay, A., Nandi, D. Factors affecting the pathophysiology of sepsis, an inflammatory disorder: Key roles of oxidative and nitrosative stress. Indian Journal of Inflammation Research. 3 (1), 2 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados