Method Article
Überwachung der Leucin-reichen Repeat-haltigen 8-Kanal-Aktivität (LRRC8/VRAC) mittels sensibilisiertem Emissions-Förster-Resonanz-Energietransfer (SE-FRET)
In diesem Artikel
Zusammenfassung
Die Elektrophysiologie ist der Goldstandard für die Untersuchung der Aktivität von Ionenkanälen. Es gibt jedoch viele alternative Ansätze, einschließlich optischer Methoden. In dieser Arbeit beschreiben wir eine Methode zur Überwachung der Aktivität des leucinreichen Repeats mit 8 Kanälen (LRRC8)-gebildeten Anionenkanälen unter Verwendung einer auf Förster-Resonanzenergietransfer (FRET) basierenden Methode zwischen den Untereinheiten.
Zusammenfassung
Mitglieder der LRRC8-Proteinfamilie bilden heteromere Ionen- und Osmolytkanäle, die in zahlreichen physiologischen Prozessen eine Rolle spielen. Als volumenregulierte Anionenkanäle (VRACs)/volumenempfindliche nach außen gleichrichtende Kanäle (VSORs) werden sie bei osmotischer Zellschwellung aktiviert und vermitteln die Extrusion von Chlorid und organischen Osmolyten, was zum Ausfluss von Wasser und damit zur Zellschrumpfung führt. Neben ihrer Rolle bei der osmotischen Volumenregulation wurden VRACs mit zellulären Prozessen wie Differenzierung, Migration und Apoptose in Verbindung gebracht. Durch ihre Wirkung auf das Membranpotential und ihren Transport verschiedener Signalmoleküle spielen leucinreiche Repeat-haltige 8 (LRRC8) Kanäle eine Rolle bei der Neuron-Glia-Kommunikation, der Insulinsekretion und der Immunantwort. Der Aktivierungsmechanismus ist bisher schwer fassbar geblieben. LRRC8-Kanäle werden, wie andere Ionenkanäle auch, typischerweise mit elektrophysiologischen Methoden untersucht. In dieser Arbeit beschreiben wir eine Methode zum Nachweis der LRRC8-Kanalaktivierung durch Messung des intra-komplexen sensibilisierten Förster-Resonanzenergietransfers (SE-FRET) zwischen fluoreszierenden Proteinen, die mit den C-terminalen leucinreichen Repeatdomänen von LRRC8-Untereinheiten fusioniert sind. Diese Methode bietet die Möglichkeit, die Kanalaktivierung in situ ohne Austausch der zytosolischen Umgebung und während Prozessen wie Zelldifferenzierung und Apoptose zu untersuchen.
Einleitung
Ionenkanäle, die aus Heteromeren von Proteinen der leucinreichen Repeat-8-Familie (LRRC8) bestehen, sind in allen Wirbeltierzellen zu finden und an einer Vielzahl physiologischer Funktionen beteiligt 1,2. Diese LRRC8-Kanäle, die zunächst als volumenregulierte Anionenkanäle (VRACs) oder volumensensitive nach außen gleichrichtende Kanäle (VSOR) identifiziert wurden, spielen eine entscheidende Rolle bei der zellulären regulatorischen Volumenabnahme 3,4. Sie erleichtern den Ausstoß von Chloridionen und organischen Osmolyten, woraufhin als Reaktion auf osmotische Quellung Wasserausfluss folgt. Neben ihrer Rolle bei der osmotischen Stressreaktion wurde ihre Rolle bei der zellulären Volumenregulation mit Zellproliferation und -migration, Apoptose, Spermiogenese und epithelialer Integrität in Verbindung gebracht 5,6,7. Es wurde gezeigt, dass die Veränderung des Membranpotentials bei LRRC8/VRAC-Aktivierung zur Differenzierung der Myotuben8 und zur Insulinsekretion durch Pankreas-β-Zellenbeiträgt 9,10,11. Darüber hinaus leiten LRRC8-Kanäle eine Vielzahl organischer Osmolyte wie die purinergen Signalmoleküle ATP und cGAMP oder die exzitatorische Aminosäure Glutamat, wodurch diese Kanäle in der Zell-Zell-Kommunikation im Immunsystem oder in der Glia-Neuronen-Interaktion platziertwerden 12,13,14,15,16,17,18,19,20,21,22. Auch Xenobiotika, wie der Farbstoff Fluorescein, das Antibiotikum Blasticidin S oder das Krebsmedikament Cisplatin, werden über die LRRC8-Kanälegeleitet 23,24,25.
Es gibt zahlreiche Berichte über die Signaltransduktion, die zur Aktivierung von LRRC8/VRAC führte 26,27,28. Der Mechanismus bleibt jedoch unklar, und die Literatur präsentiert ein breites Spektrum potenzieller Mechanismen, die von dem spezifischen physiologischen Prozess abhängen könnten. Dazu gehören Änderungen der zytosolischen Ionenstärke, der Wechselwirkung mit dem Zytoskelett, der Membranzusammensetzung, der G-Proteine, des Redoxzustands und der Phosphorylierungskaskaden 2,27,29,30,31.
LRRC8/VRAC-Kanäle enthalten LRRC8A als essentielle Untereinheit 3,4, die mit mindestens einem ihrer Paraloge LRRC8B-E heteromerisieren muss, um physiologisch funktionelle Kanäle 4,14,32 zu bilden. Die Zusammensetzung der Untereinheiten bestimmt die biophysikalischen Eigenschaften des Kanals, wie z. B. die rektifikations- und depolarisationsabhängige Inaktivierung 4,29,32,33,34, die Substratspezifität 15,17,20,21,24,35 und einige Aktivierungswege 36,37. Strukturen der Kryo-Elektronenmikroskopie (Kryo-EM) zeigen, dass LRRC8A-Homomere sowie Heteromere als Hexamere anlagern 38,39,40, während LRRC8A/LRRC8C-Chimären, die funktionelle Kanäle bilden, Heptamere sind 41. Der N-terminale Teil aller LRRC8-Proteine besteht aus vier Transmembran-Helices, und der C-terminale Teil enthält eine Domäne mit leucinreichen Repeats (LRRD). Die verfügbaren LRRC8-Komplexstrukturen liefern Hinweise darauf, dass die LRRDs, die sich in das Zytosol 3,4,23 erstrecken, während des Kanal-Gating Konformationsumlagerungen erfahren können 34,42,43. Diese Annahme wird durch die Erkenntnis bestätigt, dass die C-terminale Fusion fluoreszierender Proteine zu einer basalen Kanalaktivitätführt 14 und dass die Bindung von Nanobodies an die Domänen die Kanalaktivität modulieren kann44. Darüber hinaus wurden Konformationsänderungen der C-Termini durch den intra-komplexen Förster-Resonanzenergietransfer (FRET)45 gezeigt.
Die gebräuchlichste Methode zur Untersuchung der Ionenkanalaktivität sind elektrophysiologische Messungen46, die bei der Untersuchung von VRACs vor ihrer molekularen Identifizierung ausgiebig angewendet wurden47. Es gibt jedoch verschiedene zusätzliche Möglichkeiten, die VRAC-Aktivität indirekt zu überwachen, einschließlich der Messung seiner leitungsgebundenen Substrate - Halogenidionen oder organische Osmolyte - oder seiner Wirkung auf das Zellvolumen48. Tatsächlich beruhte die Identifizierung von LRRC8-Proteinen als VRAC auf einem Assay, der auf dem Quenchen eines halogenidempfindlichen fluoreszierenden Proteins49 durch Jodid basierte, das durch aktivierte VRACs 3,4 in die Zelle gelangte. Eine andere Methode zur Überwachung der LRRC8/VRAC-Kanalaktivität nutzt die Bewegung der zytosolischen Domänen, die wie in anderen Ionenkanälen 50,51,52,53 durch Änderungen in FRET 45 beobachtet werden kann. Zu diesem Zweck wurden fluoreszierende Proteine, die als FRET-Paare dienen, wie das cyan-fluoreszierende Protein (CFP)/mCerulean3 als Donor und das gelb-fluoreszierende Protein (YFP)/mVenus als Akzeptor, mit den C-Termini der LRRC8-Proteine fusioniert (Abbildung 1). Intra-komplexe FRET zwischen LRRC8-Untereinheiten wurde durch Akzeptor-Photobleichexperimentegezeigt 45. Unter Vermeidung des destruktiven Photobleichverfahrens wurden FRET-Veränderungen im Laufe der Zeit durch sensilizierte Emissions-FRET (SE-FRET) überwacht, wobei grundsätzlich die sensibilisierte Emission des Akzeptors bei Anregung des Donors aufgrund der Überlappung des Emissionsspektrums des Donors mit dem Anregungsspektrum des Akzeptors gemessen wird. Die Anwendung der extrazellulären Hypotonie, einem Stimulus für die LRRC8/VRAC-Aktivierung, führte zu einer reversiblen Verringerung der SE-FRET-Intensität45. Wichtig ist, dass gleichzeitige Ganzzell-Patch-Clamp-Messungen und FRET-Überwachung während der hypotonen Behandlung zeigten, dass diese Verringerung der FRET tatsächlich die LRRC8/VRAC-Aktivierung widerspiegelte45. Diese Methode, die eine Störung der Plasmamembran oder eine Veränderung der intrazellulären Umgebung durch Pipettenlösung vermeidet, bietet eine Alternative zur Überwachung der LRRC8/VRAC-Aktivität. Es ist besonders nützlich in physiologischen Umgebungen, in denen die Aufrechterhaltung des nativen Cytosols entscheidend ist, eine subzelluläre Auflösung erforderlich ist oder eine längere Beobachtung der Kanalaktivität erforderlich ist.
Hier stellen wir ein Protokoll zur Untersuchung von LRRC8/VRAC mit einer solchen FRET-basierten Auslesung vor. Das Protokoll beschreibt, wie Zellen gehandhabt und transfiziert werden, wie Proben- und Kontrollbilder erfasst werden, wie die Daten analysiert werden und wie die Werte für sensibilisierte Emissions-Fret (SE-FRET) berechnet werden.
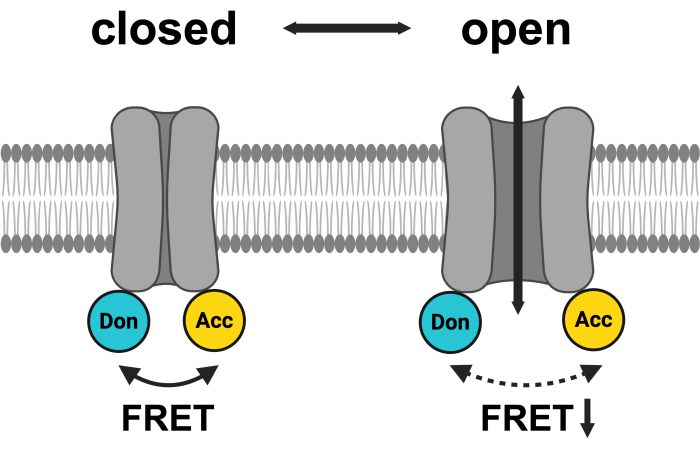
Abbildung 1: Schematische Darstellung des LRRC8 FRET-Paarsystems. mCerulean3 ist in Cyan und mVenus in Gelb dargestellt. Nach der VRAC-Öffnung ändert sich der Abstand (und/oder die räumliche Orientierung) zwischen den Fluorophoren, was zu einem verringerten Energietransfer zwischen Donor (Don) und Akzeptor (Acc) führt und damit die beobachtete FRET senkt. Erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Vorbereitung von Puffer und Reagenzien
- Bereiten Sie mittlere und isotonische, hypotonische und hypertone Puffer vor und messen Sie die Osmolarität der Puffer mit einem Osmometer, um sicherzustellen, dass die Osmolarität (Osm) mit nur einer akzeptablen Abweichung im erwarteten Bereich liegt.
- Zellkulturmedien: Bereiten Sie Dulbecco's Modified Eagle Medium (DMEM) vor, ergänzt mit 10 % fötalem Rinderserum (FBS) und 1 % Penicillin/Streptomycin
- Isotonischer Imaging-Puffer (~340 mOsm): Bereiten Sie den isotonischen Imaging-Puffer vor, indem Sie 150 mM NaCl, 6 mM KCl, 1 mM MgCl2, 1,5 mM CaCl2, 10 mM D(+)-Glucose und 10 mM 4-(2-Hydroxyethyl)-1-Piperazin-Ethansulfonsäure (HEPES) mischen.
- Hypotonischer Imaging-Puffer (~250 mOsm): Bereiten Sie den hypotonischen Imaging-Puffer vor, indem Sie 105 mM NaCl, 6 mM KCl, 1 mM MgCl2, 1,5 mM CaCl2, 10 mM D(+)-Glucose und 10 mM HEPES mischen.
- Hypertonischer Imaging-Puffer (~500 mOsm): Bereiten Sie den hypertonischen Imaging-Puffer vor, indem Sie 160 mM D(-)-Mannitol, 150 mM NaCl, 6 mM KCl, 1 mM MgCl2, 1,5 mM CaCl2, 10 mM D(+)-Glucose und 10 mM HEPES mischen.
HINWEIS: Puffer können mit bestimmten Arzneimitteln ergänzt werden, je nachdem, was untersucht wird.
2. Wachstum von adhärenten Zellen auf Glasbodenschalen
- Bereiten Sie mindestens eine Schale nur für die Transfektion vor, wobei das Protein of Interest (POI) entweder mit dem Donor- oder dem Akzeptorfluorophor fusioniert ist, und mindestens eine Schale für die Transfektion mit dem Donor-Akzeptor-Paar (Tabelle 1 und Abbildung 2). Verwenden Sie Schalen mit Glasboden, um die Fluoreszenzmikroskopie zu ermöglichen. Je nach Zelltyp die Oberfläche durch spezielle Reinigung oder Beschichtung mit Polylysin vorbehandeln.
HINWEIS: Hier werden der FRET-Donor mCerulean3 (mCer)54 und der FRET-Akzeptor mVenus (mVen)55 als FRET-Paar verwendet, aber auch andere Kombinationen von Fluorophoren sind anwendbar. Die Probe, die den POI exprimiert, der mit dem Donor oder Akzeptor fusioniert ist, wird allein verwendet, um die Korrekturfaktoren β und γ (Tabelle 1) zu bestimmen, die erforderlich sind, um das Durchbluten des Donors im Akzeptorkanal und die Queranregung des Akzeptors durch die Anregungswellenlänge des Donors zu korrigieren (Abbildung 2Ac, Bf). - Am Tag vor der Transfektion werden 1 x 105 HeLa-Zellen in 2 ml Zellkulturmedien auf 35-mm-Schalen mit einem für die Fluoreszenzmikroskopie geeigneten Glasboden ausgesät. Zellen über Nacht bei 37 °C und 5 % CO2 -Atmosphäre in einem Zellkultur-Inkubator kultivieren.
HINWEIS: Die Volumina der Reagenzien können an die Oberfläche anderer Zellkulturschalen/-kolben mit einer anderen Größe angepasst werden. Die Zellzahl kann auf eine Dichte angepasst werden, die für verschiedene Zelllinien und experimentelle Ansätze geeignet ist.- Der Zellkulturüberstand wird mit einer Vakuumpumpe aus der Monoschicht adhärenter Zellen, die auf T75-Zellkulturkolben kultiviert werden, aspiriert.
- Waschen Sie die Zellen, indem Sie 10 ml Dulbeccos phosphatgepufferte Kochsalzlösung (DPBS) in den Zellkulturkolben geben und die Lösung anschließend mit einer Vakuumpumpe aspirieren.
- Bedecken Sie die Zellen vollständig mit 1 ml 0,05 % Trypsin-EDTA und inkubieren Sie die Zellen 2 Minuten lang bei 37 °C und 5 % CO2 -Atmosphäre in einem Zellkultur-Inkubator.
- Fügen Sie 9 ml Zellkulturmedium hinzu und suspendieren Sie die Zellen durch Pipettieren.
- Die Zellsuspension in ein 15-ml-Röhrchen überführen und bei 700 x g für 3 min bei Raumtemperatur (RT) zentrifugieren.
- Der Überstand wird mit einer Vakuumpumpe abgesaugt und das Zellpellet in 10 ml Zellkulturmedium resuspendiert.
- Mischen Sie gleiche Volumina Zellsuspension und 0,4 % Trypanblau in einem Röhrchen und geben Sie 10 μl in einen Zählobjektträger. Legen Sie den Objektträger in den automatischen Zellzähler und beginnen Sie mit dem Zählen mit dem entsprechenden Programm für die Zellzählung.
- Um das Volumen der Zellsuspension zu berechnen, das für jede Schale benötigt wird, dividieren Sie die pro Schale benötigte Zellzahl (1 x 105) durch die Zellzahl pro Milliliter, die aus dem Zellzähler erhalten wurde.
- Bereiten Sie eine Zellsuspension in einem geeigneten Röhrchen vor, das die Anzahl der pro Schale benötigten Zellen in dem pro Schale benötigten Volumen (2 ml) plus die Menge für eine zusätzliche Schale enthält.
- Mischen Sie die Zellsuspension mindestens 20 Mal, indem Sie das Röhrchen umdrehen und 2 ml der Zellsuspension in jede Schale pipettieren.
- Um eine gleichmäßigere Zellverteilung zu gewährleisten, lassen Sie die Schalen 30 Minuten bei RT, bevor Sie sie in den Zellkultur-Inkubator stellen.
3. Transfektion von Zellen
HINWEIS: Hier wurde FuGENE als Transfektionsreagenz verwendet. Andere Transfektionsreagenzien und -methoden sind ebenfalls anwendbar. Das optimale Verhältnis von Plasmid-DNA (pDNA) zu Transfektionsreagenzien und die Zeit für eine optimale Expression für jeden POI und jedes Zellmodell müssen empirisch bewertet werden. Hier wurden 2 μg Gesamt-pDNA pro 35-mm-Schale verwendet. FRET-Donor- und -Akzeptorkonstrukte wurden im Verhältnis 1:1 verwendet, und das Verhältnis von pDNA zu Transfektionsreagenz betrug 1:4 (Tabelle 1).
- Vor der Transfektion ist der Zellkulturüberstand durch frische 2 ml vorgewärmtes Medium zu ersetzen.
- Verdünnen Sie die pDNA in Opti-Minimal Essential Medium (MEM) mit einem Endvolumen von 100 μL (=pDNA-Lösung).
- Verdünnen Sie das Transfektionsreagenz in Opti-MEM mit einem Endvolumen von 100 μL (=Reagenzlösung).
HINWEIS: Für eine gleichmäßigere Transfektion bereiten Sie einen Mastermix für die pDNA- und Reagenzlösung vor. - pDNA und Reagenzlösung gut mischen.
- Geben Sie die pDNA-Lösung in die Reagenzlösung (=Transfektionslösung).
- Mischen Sie die Transfektionslösung gut.
- Inkubieren Sie die Transfektionslösung 15 Minuten lang bei RT.
- Geben Sie die Transfektionslösung tropfenweise in einer spiralförmigen Bewegung in die Schale.
- Mischen Sie, indem Sie die Schüssel 5 Mal horizontal und vertikal auf der Oberfläche der Bank bewegen.
- Zellen über Nacht bei 37 °C und 5 % CO2 -Atmosphäre in einem Zellkultur-Inkubator kultivieren.
| Zustand | Konstrukt(e) | Probe | Verwendet für |
| 1 | LRRC8A-mCerulean | Nur Spenderkonstrukt | Korrekturfaktor β ermitteln |
| 2 | LRRC8E-mVenus | Nur Akzeptor-Konstrukt | Korrekturfaktor γ ermitteln |
| 3 | LRRC8A-mCerulean und LRRC8E-mVenus | BUND-Paar | SE-FRET-Quantifizierung |
Tabelle 1: Beispiel für Bedingungen, die für ein typisches SE-FRET-Experiment erforderlich sind, um die LRRC8/VRAC-Aktivität eines Kanals zu messen, der aus der LRRC8A-Untereinheit besteht, die mit dem Donor (mCerulean3) fusioniert ist, und der LRRC8E-Untereinheit, die mit dem Akzeptorfluorophor (mVenus) fusioniert ist.
4. Bildaufnahme zur Bestimmung des Korrekturfaktors
HINWEIS: Während der FRET kommt es zu einem Durchscheinen der Donoremission in die detektierte Emission des Akzeptors. Darüber hinaus kommt es zu einer Queranregung des Akzeptorfluorophors durch die Donoranregungswellenlänge. Diese Vorgänge müssen bei der Berechnung des SE-FRET kompensiert werden. Zu diesem Zweck werden Korrekturfaktoren in Zellen bestimmt, die 24 h nach der Transfektion nur den FRET-Donor oder -Akzeptor exprimieren. Hier wurde die Bildgebung auf einem Leica THUNDER Imager durchgeführt, der mit einer Leica LED8 Lampe, dem Filterwürfel CYR71010, einem HC PL APO 63x/1.40 OIL Objektiv, einem Langpassfilter für 460/80 und 553/70 und einer Leica DFC9000GTC Kamera ausgestattet war. Die Experimente wurden ohne Umweltkontrolle, aber in Gegenwart von HEPES in den Bildgebungspuffern durchgeführt, um den pH-Wert zu stabilisieren. Für Langzeitbeobachtungen/-messungen wird empfohlen, ein Umweltkontrollsystem zu verwenden. Für die Analyse wird der SE-FRET aus den aufgenommenen Rohbildern berechnet. Dies kann gleichzeitig während der Aufnahme oder danach erfolgen. Hier wurde die Leica LAS X Software mit dem SE-FRET-Plugin eingesetzt, um das experimentelle Vorgehen bei der Berechnung der Korrekturfaktoren und der Visualisierung der SE-FRET-Wertänderungen in Echtzeit während der Bildaufnahme zu vereinfachen. Für die Nacherfassung können Korrekturfaktoren und SE-FRET mit anderen Softwarepaketen (z. B. FIJI) nach der Rohdatenerfassung gemäß dem unten bereitgestellten Protokoll bestimmt werden.
- Vor der Bildaufnahme einzelner transfizierter Zellen zur Berechnung der Korrekturfaktoren verwenden Sie eine Probe, die das FRET-Paar exprimiert, um die Mikroskopieeinstellung für alle Kanäle (Donoranregung/Donoremission DD, Donor/Akzeptor DA und Akzeptor/Akzeptor AA; siehe Tabelle 1 und Abbildung 2C und Abbildung 3A) einzurichten.
HINWEIS: Für SE-FRET-Messungen mit mCerulean3- und mVenus-markierten VRAC-Untereinheiten, die wie oben beschrieben transfiziert wurden, wurden die folgenden Parameter verwendet: 8x8 Pixel-Binning, 100 ms Belichtungszeit und 10 % LED-Lampenintensität. - Entnehmen Sie die Probe, die nur das Donorkonstrukt exprimiert (Abbildung 2A), aspirieren Sie das Zellkulturmedium und waschen Sie die Zellen dreimal mit 2 ml isotonischem Puffer.
- Fügen Sie 3 ml isotonischen Puffer hinzu und legen Sie die Probe auf den Mikroskoptisch.
- Finde ein Sichtfeld (FOV) mit mindestens einer Zelle, die das Donorkonstrukt exprimiert.
- Bilden Sie alle Kanäle ab (DD, DA und AA siehe Tabelle 2 und Abbildung 2A).
- Zeichnen Sie einen Bereich of Interest (ROI) um die Zelle/Zellen und messen Sie die mittlere Intensität von DA (= IDA; Abbildung 2Ac) und DD (= IDD; Abbildung 2Aa).
- Zeichnen Sie für die Hintergrundsubtraktion einen ROI in den Kanälen DA und DD, in denen nur das Hintergrundsignal zu finden ist, und messen Sie die mittlere Intensität (IBDA und IBDD).
- Subtrahieren Sie die mittlere Intensität des Hintergrund-ROI von der mittleren Intensität, die im Zellen-ROI des entsprechenden Kanals gemessen wird (
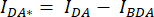 und
und 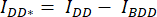 ).
). - Wiederholen Sie die Schritte 4.2 bis 4.5, wobei die Probe nur das Akzeptorkonstrukt ausdrückt (Abbildung 2B).
- Zeichnen Sie einen ROI um die Zelle(n) und messen Sie die mittlere Intensität von DA (= IDA; Abbildung 2Bf) und AA (= IAA; Abbildung 2Be).
- Für die Hintergrundsubtraktion zeichnen Sie einen ROI in den Kanälen DA und AA, in denen nur das Hintergrundsignal zu finden ist, und messen Sie die mittleren Intensitäten (IBDA und I BAA).
- Subtrahieren Sie die mittlere Intensität des Hintergrund-ROI von der mittleren Intensität, die im Zellen-ROI des entsprechenden Kanals gemessen wird (
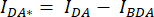 und
und 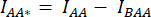 ).
). - Verwenden Sie die für IDA*, I DD* und IAA* ermittelten Werte, um die Korrekturfaktoren
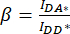 zu berechnen und
zu berechnen und 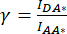 ,
,
Dabei wird der Korrekturfaktor β verwendet, um das Durchscheinen der Donoremission in den DA-Kanal zu kompensieren. Der Korrekturfaktor γ wird verwendet, um die Queranregung des Akzeptorfluorophors durch die Donoranregungswellenlänge im DA-Kanal zu kompensieren.
| Anregung von | Emission von | Name des Kanals | LED-Linie | Filterwürfel | Langpassfilter |
| Spender | Spender | DD | 440 nm | CYR71010 | 460/80 nm |
| Spender | Empfänger | DA | 440 nm | CYR71010 | 535/70 nm |
| Empfänger | Empfänger | AA | 510 nm | CYR71010 | 535/70 nm |
Tabelle 2: Zusammenfassung der für SE-FRET-Experimente benötigten Kanäle.
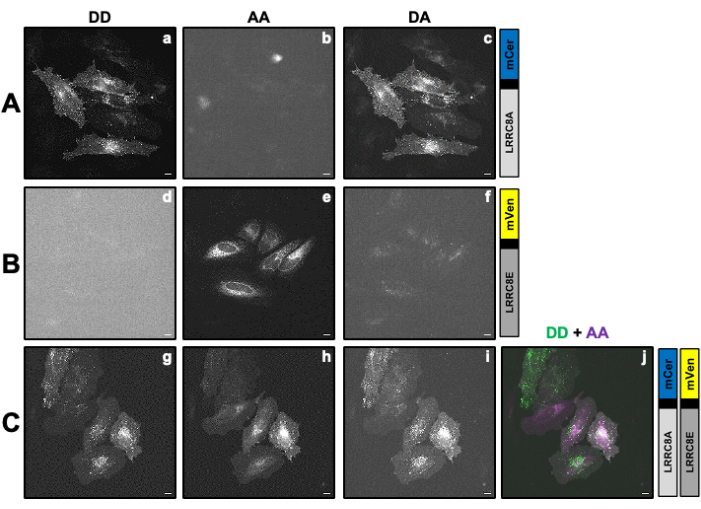
Abbildung 2: Repräsentative Fluoreszenzbilder von Proben, die zur Berechnung der Korrekturfaktoren β und γ verwendet wurden, die zur Bestimmung der VRAC-Aktivität eines Kanals erforderlich sind, der aus der Untereinheit LRRC8A, die mit dem Donor mCerulean3 (mCer) fusioniert ist, und der Untereinheit LRRC8E, die mit dem Akzeptor mVenus (mVen)-Fluorophor fusioniert ist, durch SE-FRET-Messungen verwendet wurden.(A,B) Nachweis des Donor-/Spender-DD-, des Akzeptor-/Akzeptor-AA- und des Donor/Akzeptor-DA-Kanals in HeLa-Zellen, die nur den Spender (A) LRRC8A-mCer oder den Akzeptor (B) LRRC8E-mVen exprimieren. (C) Nachweis von DD-, AA- und DA-Kanälen in HeLa-Zellen, die mit dem Donor- und Akzeptorpaar LRRC8A-mCer und LRRC8E-mVen co-transfiziert wurden. Die Panels a-i zeigen Bilder, die im Spenderdetektionskanal aufgenommen wurden (Anregung des Donators und Detektion des Donorsignals; DD; a, d und g), den Akzeptor-Detektionskanal (Anregung des Akzeptors und Detektion des Akzeptorsignals; AA; b, e und h ) und dem FRET-Signaldetektionskanal (Anregung des Donors und Detektion des Akzeptorsignals; DA; c, f und i). Feld j ist die Überlagerung der Felder g und h. Der DD-Kanal ist grün und der AA-Kanal magentafarben. Maßstabsleiste = 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Zeitraffer-Bildgebung für die SE-FRET-Quantifizierung
- Entnehmen Sie die Probe, die das Donor- und Akzeptorkonstrukt exprimiert (Tabelle 1, Abbildung 2C und Abbildung 3A), aspirieren Sie das Zellkulturmedium und waschen Sie die Zellen 24 h nach der Transfektion dreimal mit 2 ml isotonischem Puffer.
- Fügen Sie 3 ml isotonischen Puffer hinzu und legen Sie die Probe auf den Mikroskopietisch.
- Für eine spätere Aspiration des isotonischen Puffers fixieren Sie eine Schlauchkanüle und stellen Sie sie so ein, dass die Kanülenspitze den Boden der Schale erreicht.
- Um Puffer hinzuzufügen, fixieren und passen Sie die Schläuche so an, dass der durch die Schwerkraft angetriebene Puffer in die Schale fallen kann.
HINWEIS: Hier wurde ein ventilgesteuertes Schwerkraftperfusionssystem mit vier Magnetventilen zur Steuerung des Pufferflusses verwendet, aber auch andere Methoden sind anwendbar. - Finde ein FOV mit mindestens einer Zelle, die das Donor- und Akzeptorkonstrukt gleichzeitig exprimiert.
- Richten Sie ein Zeitrafferexperiment für die Kanäle DD, DA und AA mit einem Intervall von 10 s und einer Dauer ein, die alle Bedingungen der Stimulationssequenz abdeckt, z. B. 12 Zyklen der Baseline, gefolgt von 15 Zyklen pro Bedingung (Abbildung 3).
HINWEIS: Das Intervall und die Anzahl der Zyklen können je nach experimentellem Bedarf angepasst werden, sollten jedoch immer eine Basismessung zur Normalisierung enthalten, um die Daten besser zu visualisieren und zu vergleichen. - Waschen Sie die Probe nach der Basismessung im Puffer für die erste Bedingung.
- Aspirieren Sie den isotonischen Puffer über die Schlauchkanüle und legen Sie mit einer Spritze ein Vakuum an.
- Fügen Sie 3 mL des Puffers der nächsten Bedingung durch Schwerkraft hinzu.
- Aspirieren Sie den Puffer und fügen Sie erneut 3 ml desselben Puffers hinzu.
- Wiederholen Sie Schritt 5.7.3 noch einmal.
- Nach der Messung der ersten Bedingung waschen Sie die Probe im Puffer für die nächste Bedingung (Schritte 5.7.1-5.7.4).
- Wiederholen Sie die Schritte 5.5 bis 5.8, bis alle Bedingungen erfasst sind.
- Für die SE-FRET-Quantifizierung zeichnen Sie einen ROI um die Zelle(n) und messen Sie die mittlere Intensität im Kanal DD = (IDD), DA (IDA) und AA (IAA) für alle Bilder (Zeitpunkte) in der Zeitreihe (Abbildung 3A, B).
- Für die Hintergrundsubtraktion für das FRET-Signal zeichnen Sie eine ROI im DA-Kanal, wo nur das Hintergrundsignal zu finden ist, und messen Sie die mittlere Intensität (IBDA).
- Subtrahieren Sie die mittlere Intensität des Hintergrund-ROI von der mittleren Intensität, die im Zellen-ROI des DA-Kanals gemessen wurde (
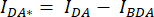 ).
). - Verwenden Sie die ermittelten Werte, um die mittleren SE-FRET-Werte für jeden ROI und alle Zeitpunkte
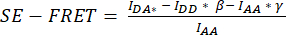 zu berechnen.
zu berechnen. - Für eine bessere Vergleichbarkeit verschiedener Bedingungen und eine Visualisierung der SE-FRET-Werte verwenden Sie den Mittelwert der Basiswerte jedes ROI, um alle Zeitpunkte des entsprechenden ROI zu normalisieren.
- Zeichnen Sie die normierten SE-FRET-Werte im Zeitverlauf auf (Abbildung 3B).
Abbildung 3: Repräsentative Fluoreszenzbilder und SE-FRET-Quantifizierung. (A) Repräsentative Fluoreszenzbilder und der scheinbare SE-FRET des ersten Zeitpunkts eines Zeitraffer-Experiments zur Messung der VRAC-Aktivität mittels SE-FRET eines Kanals, der aus LRRC8A- und LRRC8E-Untereinheiten in Abhängigkeit von der Tonizität besteht. Maßstabsleiste = 10 μm. Die gleichen Zellen sind in Abbildung 2C dargestellt. Die Felder a-i zeigen die Detektion der DD-, AA- und DA-Kanäle und den berechneten scheinbaren SE-FRET. Weiße Umrandungen stellen die ROIs dar (Zellen i-iii in Feld d), die zur Messung der mittleren Signalintensitäten in DD, AA und DA und des scheinbaren SE-FRET-Bildes verwendet werden. (B) Quantifizierung der SE-FRET-Werte im Zeitverlauf. Die Abfolge der Bedingungen bestand aus 12 Zyklen isotonischem Bildgebungspuffer (Baseline), gefolgt von 15 Zyklen hypotonem und 15 Zyklen hypertonem Bildgebungspuffer. Der rohe mittlere SE-FRET-Wert jedes ROI (Zellen i-iii) und Zeitpunkts wurde auf den Mittelwert des (isotonischen) Ausgangswerts für den entsprechenden ROI normiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Mit dieser FRET-basierten Methode kann die LRRC8/VRAC-Aktivität während der osmotischen Stimulation überwacht werden, und die Reduktion der SE-FRET korreliert mit dem Grad der extrazellulären Hypotonie45. Hier sind auch repräsentative Ergebnisse für die hypotonieinduzierte Kanalaktivierung dargestellt (Abbildung 3 und Abbildung 4). Darüber hinaus kann eine LRRC8/VRAC-Aktivierung durch verschiedene isosmotische Stimuli, wie z.B. die Manipulation des Diacylglycerin-Signalwegs45 oder während der Myozytenaktivierung56, beobachtet werden.
Abbildung 4: SE-FRET-Spuren. (A) Scheinbare SE-FRET-Spuren aus 5 unabhängigen Experimenten. Die Daten stellen den Mittelwert ± SD von N = 2 bis N = 7 Zellen pro Sichtfeld (FOV) dar. (B) Mittlere ± SD aller Zellen (N = 31 Zellen). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Da LRRC8/VRAC-Kanäle auch an der apoptotischen Volumenabnahme beteiligt sind24,57, wäre die Beobachtung der Kanalaktivität bei Induktion der Apoptose eine weitere Anwendung für das hier beschriebene Verfahren. Dementsprechend wurde das Protokoll zur Überwachung von SE-FRET in HeLa-Zellen, die LRRC8A-mCerulean3 und LRRC8E-mVenus exprimieren, unter Anwendung von Todesrezeptor-vermittelten Apoptose-induzierenden Arzneimitteln durchgeführt. Es wurde bereits gezeigt, dass Tumornekrosefaktor (TNF)-α und Cycloheximid (CHX) innerhalb weniger Minuten VRAC-Ströme hervorrufen58. Nach der Zugabe von 2 ng/ml TNF-α und 1 μg/ml CHX in isotonischem Puffer kam es zu einer robusten Abnahme des SE-FRET (Abbildung 5). Ersetzte man den Puffer durch ein hypertonisches Medium, obwohl es TNF-α und CHX enthielt, erholten sich die SE-FRET-Werte nahe dem Ausgangswert (Abbildung 5A), was einer Abnahme der VRAC-Ströme in der hypertonen Badlösung während der Behandlung mit dem Apoptose-induzierenden Fas-Liganden58 entspricht. Die Behandlung der Zellen mit DMSO, dem Lösungsmittel für TNF-α und CHX, führte nicht zu einer SE-FRET-Reduktion. TNF-α + CHX beeinflusste nicht die SE-FRET von CFP-18aa-YFP, einem EYFP- und ECFP-Tandemkonstrukt als FRET-Kontrolle59, was die Spezifität für LRRC8/VRAC demonstriert (Abbildung 5B).
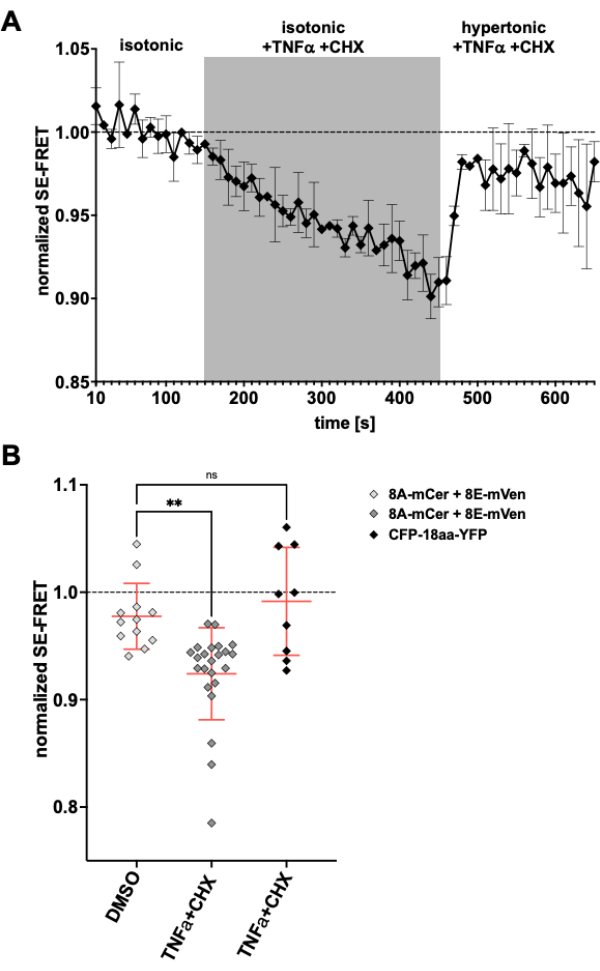
Abbildung 5: Isosmotische VRAC-Aktivierung durch Todesrezeptor-vermittelte Apoptose. (A) Normalisierte SE-FRET-Werte von LRRC8A-mCer/LRRC8E-mVen-exprimierenden HeLa-Zellen (n = 8 Schalen, 23 Zellen) über die Zeit. Nach 15 Zyklen in isotonischem Bildgebungspuffer (Baseline) wurde die Badlösung durch isotonischen Puffer ersetzt, der mit 2 ng/ml TNF-α und 1 μg/ml Cycloheximid (CHX) für 30 Zyklen ergänzt wurde, gefolgt von 20 Zyklen hypertonischem Bildgebungspuffer mit TNF-α und CHX. Der rohe mittlere SE-FRET-Wert jedes ROI und Zeitpunkts wurde auf den Mittelwert des (isotonischen) Basiswerts für den jeweiligen ROI normiert. (B) Quantifizierung der normierten SE-FRET-Werte von LRRC8A/E-exprimierenden HeLa-Zellen wie in A mit isotonischer DMSO-haltiger Lösung als Vehikelkontrolle für CHX (n = 5 Schale, 12 Zellen) oder isotonischem Puffer mit Apoptose-Induktoren wie in A (n = 8 Schale, 23 Zellen) oder von HeLa-Zellen, die CFP-18aa-YFP exprimieren, mit Apoptose-Induktoren (n = 3 Schale, 9 Zellen). Die Daten stellen den Mittelwert der letzten 10 Zeitpunkte im jeweiligen Puffer der einzelnen Zellen (Symbole) und den Mittelwert aller Zellen ± SD dar; ** p < 0,01 gewöhnliche einfache ANOVA, gefolgt von Tukeys Mehrfachvergleichs-Post-hoc-Test. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die FRET-Mikroskopie ist eine etablierte und weit verbreitete Technik, um die Wechselwirkung zwischen Proteinen zu untersuchen. Daher können FRET-basierte Methoden in vielen Laboren mit unterschiedlicher Expertise angewendet werden. Konformationsumlagerungen während des Gating wurden für ein breites Spektrum von Ionenkanälen mit FRET-basierten Assays überwacht (Beispiele siehe Referenzen 34,50,51,52,53,60,61,62,63,64,65,66,67), in einigen Fällen kombiniert mit Elektrophysiologie in der Patch-Clamp-Fluorometrie 68,69,70,71. FRET kann verwendet werden, um die Struktur-Funktions-Beziehungen dieser Ionenkanäle zu untersuchen oder ihre Aktivität unabhängig vom Ionentransport zu überwachen. Die hier vorgestellte Methode kann gegenüber der Elektrophysiologie deutliche Vorteile haben, da sie die Überwachung der Aktivität von LRRC8/VRAC-Kanälen in situ ermöglicht.
Zu den kritischen Schritten des Protokolls gehört die Beschichtung der Zellen, um eine optimale Konfluenz für die Transfektion und Bildgebung zu erreichen, was im Idealfall eine einfache Zellunterscheidung für die spätere Analyse ermöglicht. Eine effektive Co-Transfektion der verschiedenen Untereinheiten ist entscheidend für eine korrekte subzelluläre Lokalisierung; zum Beispiel führt ein Überschuss der Nicht-LRRC8A-Untereinheit zu einer verbesserten Lokalisation des endoplasmatischen Retikulums (ER)4. Daher müssen die Plasmidverhältnisse möglicherweise angepasst werden. Je nach System sollen neu erzeugte FRET-Paare z.B. durch Akzeptorbleichen verifiziert werden. Binning und Belichtungszeit müssen gegeneinander abgewogen werden, um eine optimale zeitliche und räumliche Auflösung für die Forschungsfrage zu ermöglichen. Binning ermöglicht kürzere Belichtungszeiten und reduziert somit das potenzielle Ausbleichen des FRET-Sensors bei gleichzeitiger Verringerung der räumlichen Auflösung. Wenn der Versuchsaufbau z.B. eine subzelluläre Unterscheidung der LRRC8/VRAC-Aktivität erfordert, sollte Binning daher vermieden werden. Die Forschungsfrage bestimmt gleichermaßen die Anzahl und das Intervall von Zyklen in einer Zeitrafferreihe. Das Intervall ist nur relevant, wenn die Kinetik der FRET-Änderungen (und damit die Aktivierung/Inaktivierung von LRRC8/VRAC) erforderlich ist. Ansonsten können auch einfache "Vorher-Nachher"-Aufnahmen durchgeführt werden. Die Länge des Experiments hängt vom physiologischen Prozess ab. Idealerweise sollte die LRRC8/VRAC-Aktivität nach Stimuli überwacht werden, bis sich SE-FRET stabilisiert hat. Diese Faktoren können in Pilotversuchen ermittelt werden. Korrekturfaktoren zur Berechnung des realen SE-FRET-Signals müssen für alle Bedingungen bestimmt werden. Falsch ermittelte Korrekturfaktoren können zu einer Über- oder Unterschätzung der SE-FRET-Intensitäten führen. Nach der Etablierung einer stabilen Basislinie muss das Zeitintervall zwischen den Bildern kurz genug sein, um den interessierenden physiologischen Prozess zu erfassen.
Die Methode weist einige Einschränkungen auf. Eine davon ist, dass Änderungen der inter-LRRC8-FRET-Intensitäten zwar die Bewegungen der LRRDs widerspiegeln, aber nicht unbedingt dem Ionen- oder Osmolyttransport durch die Pore entsprechen. Dies wird durch die FRET-Änderungen deutlich, die bei LRRC8A-Homomeren45 trotz ihrer minimalen Ströme 4,32,72 beobachtet wurden. Porenblocker von LRRC8/VRAC-Kanälen beeinflussen das FRET-Signal möglicherweise nicht, so dass diese Methode für die Suche nach spezifischen Kanalmodulatoren ungeeignet ist. Darüber hinaus könnten die Expressionsniveaus der überexprimierten LRRC8-Proteine die beobachteten physiologischen Prozesse beeinflussen, zumal die C-terminalen markierten LRRC8-Proteine eine basale Aktivität aufweisen14.
Ein Aspekt, der je nach Forschungsfrage als Einschränkung oder Vorteil angesehen werden kann, ist, dass bei dieser Methode nur die ektopisch exprimierten LRRC8-Untereinheiten selektiv gemessen werden. Die Hintergrundwerte endogener Proteine stören die Messungen also kaum. Andererseits verhalten sich die überexprimierten Proteine möglicherweise nicht wie die endogenen LRRC8-Kanäle mit möglicherweise unterschiedlicher Untereinheitenzusammensetzung und Stöchiometrie. Zum Beispiel können verschiedene Stimuli, wie z. B. Oxidation, gegensätzliche modulatorische Wirkungen auf unterschiedlich zusammengesetzte LRRC8-Kanälehaben 36. Durch Ändern der Verhältnisse zwischen koexprimierten Untereinheiten können ihre Stöchiometrie und die Gesamtionenleitfähigkeit eingestellt werden14,73, aber ihre native Zusammensetzung, mit wahrscheinlich oft mehr als zwei Parametern innerhalb eines Komplexes21, ist nicht klar und kann zwischen den Zelltypen variieren 74,75,76 . Darüber hinaus wurde gezeigt, dass die Fusion von fluoreszierenden Proteinen mit den zytosolischen C-Termini von LRRC8-Proteinen die basale LRRC8/VRAC-Kanalaktivität in Xenopus-Eizellen erhöht14, wahrscheinlich weil die großen Tags die Konformation der LRRDs modulieren, was die Kanalöffnung steuernkönnte 14,44,45. Daher können die Größe der fluoreszierenden Proteine, der Linker und ihre Orientierung nicht nur die FRET-Effizienz, sondern auch die Kanalaktivität beeinflussen. Wichtig ist jedoch, dass VRAC-Kanäle von LRRC8-Proteinen, die mit fluoreszierenden Proteinen fusioniert sind, weiterhin auf hypotonische Stimulationansprachen 14, was ihre Verwendung als FRET-Sensorenermöglichte 45.
Die Vorteile dieser nicht-invasiven Methode zur Überwachung der LRRC8/VRAC-Kanalaktivität durch Lichtmikroskopie im Vergleich zu anderen Methoden sind: (i) Sie ermöglicht die Beobachtung von LRRC8/VRAC in Zellen oder Kompartimenten, die für die Elektrophysiologie typischerweise unzugänglich sind. Dazu gehören intrazelluläre Organellen, auf denen LRRC8-Komplexe gefunden werden können oder auf 45,77,78 abzielen. (ii) Die zytosolische Zusammensetzung bleibt durch die Methode unverändert, während bei Ganzzell-Patch-Clamp-Messungen das Zytosol weitgehend durch Pipettenlösung ersetzt wird, was die Signalwege beeinflussen kann, wie bei der Phorbol-12-Myristat-13-acetat (PMA)-induzierten LRRC8/VRAC-Aktivierungbeobachtet wurde 45. (iii) Es bietet die Möglichkeit, die LRRC8/VRAC-Aktivierung mit subzellulärer Auflösung zu beobachten, wie z.B. die Unterscheidung der Aktivität an den Vorder- und Hinterkanten während der Zellmigration, wo - auf begrenzte Räume - VRAC beteiligt ist79,80. (iv) Es ermöglicht die kontinuierliche Überwachung der LRRC8/VRAC-Aktivität während ausgedehnter physiologischer Prozesse wie der Myozytendifferenzierung56.
Obwohl es bei dieser Methode Einschränkungen und Herausforderungen gibt, ist sie vielversprechend für weitere Forschungen, einschließlich möglicher Anwendungen in Tiermodellen. In Kombination mit anderen Methoden zur Untersuchung dieser Ionen- und Osmolytkanalfamilie kann dieser FRET-basierte Assay wesentlich dazu beitragen, Aktivierungsmechanismen zu entschlüsseln und die vielfältigen physiologischen Funktionen von LRRC8-Kanälen in ihrer natürlichen Umgebung zu erforschen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken C.F. Kaminski für die freundliche Spende des Plasmids, das das CFP-18aa-YFP-Konstrukt kodiert, A. Klemmer für die technische Unterstützung und allen aktuellen und ehemaligen Mitgliedern des Stauber-Laboratoriums, die zur Entwicklung dieser Methode beigetragen haben.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | gibco | 25300-054 | |
| Camera DFC9000GTC | Leica | 11547007 | |
| CFP-18aa-YFP | N/A | N/A | Elder et al. 2009 PMCID: PMC2706461; Gift from C.F. Kaminski (University of Cambridge, UK) |
| Cycloheximide (CHX) | Sigma-Aldrich | 66-81-9 | |
| D(-)-Mannitol | Carl Roth | 4175.1 | |
| D(+)-Glucose | Carl Roth | HN06.1 | |
| DMEM (Dulbeccos Modified Eagle Medium) | PAN-Biotech | P04-03590 | |
| DPBS (Dulbecco's Phosphate Buffer Saline) | PAN-Biotech | P04-36500 | |
| Emission filter wheel (460/80, 535/70, 590/50, 642/80, 100%) | Leica | 11525480 | |
| FBS (Fetal Bovine Serum) | PAN-Biotech | P30-3302 | |
| Filter cube CYR71010 | Leica | 11525416 | |
| FuGENE | Promega | E2691 | |
| Glas Bottom Culture Dishes 35 mm | MatTek | P35G-0-10-C | |
| HeLa cells | Leibniz Forschungsinstitut DSMZ | ACC 57 | Mammalian cervix carcinoma/ Obtained from Leibniz Forschungsinstitut DSMZ |
| HEPES | Carl Roth | 9105.4 | |
| ibidi µ-Disch 35 mm | ibidi | 81156 | |
| KCl (Potassium chloride) | Carl Roth | 6781.1 | |
| LAS X FRET Wizard | Leica | 11640862 | |
| Light source LED8 | Leica | 11504256 | |
| LRRC8A-mCerulean3 | N/A | N/A | König et al. 2019 |
| LRRC8E-mVenus | N/A | N/A | König et al. 2019 |
| Luna-II Automated Cell Counter | logos biosystems | L40002 | |
| Luna-II Cell Counter Slides | logos biosystems | L12001 | |
| MgCl2 (Magnesium chloride) | Carl Roth | KK36.1 | |
| Microscope THUNDER Imager live cell | Leica | 11525681 | |
| NaCl (Sodium chloride) | Carl Roth | 9263 | |
| Objective HC PL APO 63x/1.40 OIL | Leica | 11506349 | |
| Opti-Minimal Essential Medium (MEM) | gibco | 11058 | |
| Osmometer OM807 | Vogel | V04807 | |
| Penicillin Streptomycin (Pen Step) | gibco | 15070-063 | |
| Trypan blue solution (0,4%) | Sigma | T8154 | |
| Tumor necrosis factor (TNF)-a | Sigma-Aldrich | 94948-59-1 | |
| Valve Controlled Gravity Perfusion System | ALA Scientific Instruments | VC3-4xG |
Referenzen
- Jentsch, T. J. VRACs and other ion channels and transporters in the regulation of cell volume and beyond. Nat Rev Mol Cell Biol. 17 (5), 293-307 (2016).
- Chen, L., et al. More than just a pressure relief valve: physiological roles of volume-regulated LRRC8 anion channels. Biol Chem. 400 (11), 1481-1496 (2019).
- Qiu, Z., et al. SWELL1, a plasma membrane protein, is an essential component of volume-regulated anion channel. Cell. 157 (2), 447-458 (2014).
- Voss, F. K., et al. Identification of LRRC8 heteromers as an essential component of the volume-regulated anion channel VRAC. Science. 344 (6184), 634-638 (2014).
- Hoffmann, E. K., Schettino, T., Marshall, W. S. The role of volume-sensitive ion transport systems in regulation of epithelial transport. Comp Biochem Physiol A Mol Integr Physiol. 148 (1), 29-43 (2007).
- López-Cayuqueo, K. I., et al. Renal deletion of LRRC8/VRAC channels induces proximal tubulopathy. J Am Soc Nephrol. 33 (8), 1528-1545 (2022).
- Lück, J. C., Puchkov, D., Ullrich, F., Jentsch, T. J. LRRC8/VRAC anion channels are required for late stages of spermatid development in mice. J Biol Chem. 293 (30), 11796-11808 (2018).
- Chen, L., Becker, T. M., Koch, U., Stauber, T. The LRRC8/VRAC anion channel facilitates myogenic differentiation of murine myoblasts by promoting membrane hyperpolarization. J Biol Chem. 294 (39), 14279-14288 (2019).
- Best, L., Brown, P. D., Sener, A., Malaisse, W. J. Electrical activity in pancreatic islet cells: The VRAC hypothesis. Islets. 2 (2), 59-64 (2010).
- Kang, C., et al. SWELL1 is a glucose sensor regulating beta-cell excitability and systemic glycaemia. Nat Commun. 9 (1), 367 (2018).
- Stuhlmann, T., Planells-Cases, R., Jentsch, T. J. LRRC8/VRAC anion channels enhance beta-cell glucose sensing and insulin secretion. Nat Commun. 9 (1), 1974 (2018).
- Hisadome, K., et al. Volume-regulated anion channels serve as an auto/paracrine nucleotide release pathway in aortic endothelial cells. J Gen Physiol. 119 (6), 511-520 (2002).
- Burow, P., Klapperstück, M., Markwardt, F. Activation of ATP secretion via volume-regulated anion channels by sphingosine-1-phosphate in RAW macrophages. Pflügers Arch. 467 (6), 1215-1226 (2015).
- Gaitán-Peñas, H., et al. Investigation of LRRC8-mediated volume-regulated anion currents in Xenopus oocytes. Biophys J. 111 (7), 1429-1443 (2016).
- Lahey, L. J., et al. LRRC8A:C/E heteromeric channels are ubiquitous transporters of cGAMP. Mol Cell. 80 (4), 578-591 (2020).
- Chen, X., et al. Regulation of anion channel LRRC8 volume-regulated anion channels in transport of 2'3'-cyclic GMP-AMP and cisplatin under steady state and inflammation. J Immunol. 206 (9), 2061-2074 (2021).
- Zhou, C., et al. Transfer of cGAMP into bystander cells via LRRC8 volume-regulated anion channels augments STING-mediated interferon responses and anti-viral immunity. Immunity. 52 (5), 767-781 (2020).
- Feustel, P. J., Jin, Y., Kimelberg, H. K. Volume-regulated anion channels are the predominant contributors to release of excitatory amino acids in the ischemic cortical penumbra. Stroke. 35 (5), 1164-1168 (2004).
- Mongin, A. A. Volume-regulated anion channel--a frenemy within the brain. Pflügers Arch. 468 (3), 421-441 (2016).
- Schober, A. L., Wilson, C. S., Mongin, A. A. Molecular composition and heterogeneity of the LRRC8-containing swelling-activated osmolyte channels in primary rat astrocytes. J Physiol. 595 (22), 6939-6951 (2017).
- Lutter, D., Ullrich, F., Lueck, J. C., Kempa, S., Jentsch, T. J. Selective transport of neurotransmitters and modulators by distinct volume-regulated LRRC8 anion channels. J Cell Sci. 130 (6), 1122-1133 (2017).
- Yang, J., et al. Glutamate-releasing SWELL1 channel in astrocytes modulates synaptic transmission and promotes brain damage in stroke. Neuron. 102 (4), 813-827 (2019).
- Lee, C. C., Freinkman, E., Sabatini, D. M., Ploegh, H. L. The protein synthesis inhibitor blasticidin s enters mammalian cells via leucine-rich repeat-containing protein 8D. J Biol Chem. 289 (24), 17124-17131 (2014).
- Planells-Cases, R., et al. Subunit composition of VRAC channels determines substrate specificity and cellular resistance to Pt-based anticancer drugs. EMBO J. 34 (24), 2993-3008 (2015).
- Model, M. A., Nia, F. H., Zook, E., Hollembeak, J. E., Stauber, T. Uptake of fluorescein upon osmotic cell swelling is dependent on the volume-regulated anion channel VRAC/LRRC8. Paracelsus Proc Exp Med. 1 (1), 3-14 (2022).
- Stauber, T. The volume-regulated anion channel is formed by LRRC8 heteromers - molecular identification and roles in membrane transport and physiology. Biol Chem. 396 (9-10), 975-990 (2015).
- Bertelli, S., et al. Mechanisms of activation of LRRC8 volume regulated anion channels. Cell Physiol Biochem. 55 (S1), 41-56 (2021).
- Liu, T., Li, Y., Wang, D., Stauber, T., Zhao, J. Trends in volume-regulated anion channel (VRAC) research: visualization and bibliometric analysis from 2014 to 2022. Front Pharmacol. 14, 1234885 (2023).
- Strange, K., Yamada, T., Denton, J. S. A 30-year journey from volume-regulated anion currents to molecular structure of the LRRC8 channel. J Gen Physiol. 151 (2), 100-117 (2019).
- Osei-Owusu, J., Yang, J., Vitery, M. D. C., Qiu, Z. Molecular biology and physiology of volume-regulated anion channel (VRAC). Curr Top Membr. 81, 177-203 (2018).
- Friard, J., Laurain, A., Rubera, I., Duranton, C. LRRC8/VRAC channels and the redox balance: A complex Relationship. Cell Physiol Biochem. 55 (S1), 106-118 (2021).
- Syeda, R., et al. LRRC8 proteins form volume-regulated anion channels that sense ionic strength. Cell. 164 (3), 499-511 (2016).
- Ullrich, F., Reincke, S. M., Voss, F. K., Stauber, T., Jentsch, T. J. Inactivation and anion selectivity of volume-regulated anion channels (VRACs) depend on C-terminal residues of the first extracellular loop. J Biol Chem. 291 (33), 17040-17048 (2016).
- König, B., Stauber, T. Biophysics and structure-function relationships of LRRC8-formed volume-regulated anion channels. Biophys J. 116 (7), 1185-1193 (2019).
- Concepcion, A. R., et al. The volume-regulated anion channel LRRC8C suppresses T cell function by regulating cyclic dinucleotide transport and STING-p53 signaling. Nat Immunol. 23 (2), 287-302 (2022).
- Gradogna, A., Gavazzo, P., Boccaccio, A., Pusch, M. Subunit-dependent oxidative stress sensitivity of LRRC8 volume-regulated anion channels. J Physiol. 595 (21), 6719-6733 (2017).
- Bertelli, S., Zuccolini, P., Gavazzo, P., Pusch, M. Molecular determinants underlying volume-regulated anion channel subunit-dependent oxidation sensitivity. J Physiol. 600 (17), 3965-3982 (2022).
- Deneka, D., Sawicka, M., Lam, A. K. M., Paulino, C., Dutzler, R. Structure of a volume-regulated anion channel of the LRRC8 family. Nature. 558 (7709), 254-259 (2018).
- Kasuya, G., et al. Cryo-EM structures of the human volume-regulated anion channel LRRC8. Nat Struct Mol Biol. 25 (9), 797-804 (2018).
- Kefauver, J. M., et al. Structure of the human volume regulated anion channel. Elife. 7, e38461 (2018).
- Takahashi, H., Yamada, T., Denton, J. S., Strange, K., Karakas, E. Cryo-EM structures of an LRRC8 chimera with native functional properties reveal heptameric assembly. Elife. 12, e82431 (2023).
- Sawicka, M., Dutzler, R. Regulators of cell volume: The structural and functional properties of anion channels of the LRRC8 family. Curr Opin Struct Biol. 74, 102382 (2022).
- Kasuya, G., Nureki, O. Recent advances in the structural biology of the volume-regulated anion channel LRRC8. Front Pharmacol. 13, 896532 (2022).
- Deneka, D., et al. Allosteric modulation of LRRC8 channels by targeting their cytoplasmic domains. Nat Commun. 12 (1), 5435 (2021).
- König, B., Hao, Y., Schwartz, S., Plested, A. J., Stauber, T. A FRET sensor of C-terminal movement reveals VRAC activation by plasma membrane DAG signaling rather than ionic strength. Elife. 8, e45421 (2019).
- Hille, B. . Ion Channels of Excitable Membranes. 3rd edn. , (2001).
- Pedersen, S. F., Okada, Y., Nilius, B. Biophysics and physiology of the volume-regulated anion channel (VRAC)/volume-sensitive outwardly rectifying anion channel (VSOR). Pflügers Arch. 468 (3), 371-383 (2016).
- Kolobkova, Y., Pervaiz, S., Stauber, T. The expanding toolbox to study the LRRC8-formed volume-regulated anion channel VRAC. Curr Top Membr. 88, 119-163 (2021).
- Galietta, L. J., Haggie, P. M., Verkman, A. S. Green fluorescent protein-based halide indicators with improved chloride and iodide affinities. FEBS Lett. 499 (3), 220-224 (2001).
- Bykova, E. A., Zhang, X. D., Chen, T. Y., Zheng, J. Large movement in the C terminus of CLC-0 chloride channel during slow gating. Nat Struct Mol Biol. 13 (12), 1115-1119 (2006).
- Zheng, J., Zagotta, W. N. Gating rearrangements in cyclic nucleotide-gated channels revealed by patch-clamp fluorometry. Neuron. 28 (2), 369-374 (2000).
- Miranda, P., et al. State-dependent FRET reports calcium- and voltage-dependent gating-ring motions in BK channels. Proc Natl Acad Sci U S A. 110 (13), 5217-5222 (2013).
- Zachariassen, L. G., et al. Structural rearrangement of the intracellular domains during AMPA receptor activation. Proc Natl Acad Sci U S A. 113 (27), E3950-E3959 (2016).
- Markwardt, M. L., et al. An improved cerulean fluorescent protein with enhanced brightness and reduced reversible photoswitching. PLoS One. 6 (3), e17896 (2011).
- Nagai, T., et al. A variant of yellow fluorescent protein with fast and efficient maturation for cell-biological applications. Nat Biotechnol. 20 (1), 87-90 (2002).
- Chen, L., König, B., Stauber, T. LRRC8 channel activation and reduction in cytosolic chloride concentration during early differentiation of C2C12 myoblasts. Biochem Biophys Res Commun. 532, 482-488 (2020).
- Maeno, E., Ishizaki, Y., Kanaseki, T., Hazama, A., Okada, Y. Normotonic cell shrinkage because of disordered volume regulation is an early prerequisite to apoptosis. Proc Natl Acad Sci U S A. 97 (17), 9487-9492 (2000).
- Shimizu, T., Numata, T., Okada, Y. A role of reactive oxygen species in apoptotic activation of volume-sensitive Cl- channel. Proc Natl Acad Sci U S A. 101 (17), 6770-6773 (2004).
- Elder, A. D., et al. A quantitative protocol for dynamic measurements of protein interactions by Förster resonance energy transfer-sensitized fluorescence emission. Journal of the Royal Society Interface. 6, S59-S81 (2009).
- Glauner, K. S., Mannuzzu, L. M., Gandhi, C. S., Isacoff, E. Y. Spectroscopic mapping of voltage sensor movement in the Shaker potassium channel. Nature. 402 (6763), 813-817 (1999).
- Dai, G., Aman, T. K., DiMaio, F., Zagotta, W. N. The HCN channel voltage sensor undergoes a large downward motion during hyperpolarization. Nat Struct Mol Biol. 26 (8), 686-694 (2019).
- Renart, M. L., et al. Conformational plasticity in the KcsA potassium channel pore helix revealed by homo-FRET studies. Sci Rep. 9 (1), 6215 (2019).
- Wang, S., et al. Potassium channel selectivity filter dynamics revealed by single-molecule FRET. Nat Chem Biol. 15 (4), 377-383 (2019).
- Harley, C. A., et al. Conformation-sensitive antibody reveals an altered cytosolic PAS/CNBh assembly during hERG channel gating. Proc Natl Acad Sci U S A. 118 (44), e2108796118 (2021).
- Han, S., et al. Structural dynamics determine voltage and pH gating in human voltage-gated proton channel. Elife. 11, e73093 (2022).
- Cullinan, M. M., Klipp, R. C., Camenisch, A., Bankston, J. R. Dynamic landscape of the intracellular termini of acid-sensing ion channel 1a. Elife. 12, e90755 (2023).
- Kim, J., Won, J., Chung, D. K., Lee, H. H. FRET analysis of the temperature-induced structural changes in human TRPV3. Sci Rep. 13 (1), 10108 (2023).
- Zheng, J., Zagotta, W. N. Patch-clamp fluorometry recording of conformational rearrangements of ion channels. Sci STKE. (176), PL7 (2003).
- Kusch, J., Zifarelli, G. Patch-clamp fluorometry: electrophysiology meets fluorescence. Biophys J. 106 (6), 1250-1257 (2014).
- Cowgill, J., Chanda, B. The contribution of voltage clamp fluorometry to the understanding of channel and transporter mechanisms. J Gen Physiol. 151 (10), 1163-1172 (2019).
- Bhat, S., Blunck, R. Characterising ion channel structure and dynamics using fluorescence spectroscopy techniques. Biochem Soc Trans. 50 (5), 1427-1445 (2022).
- Yamada, T., Figueroa, E. E., Denton, J. S., Strange, K. LRRC8A homohexameric channels poorly recapitulate VRAC regulation and pharmacology. Am J Physiol Cell Physiol. 320 (3), C293-C303 (2021).
- Yamada, T., Wondergem, R., Morrison, R., Yin, V. P., Strange, K. Leucine-rich repeat containing protein LRRC8A is essential for swelling-activated Cl- currents and embryonic development in zebrafish. Physiol Rep. 4 (19), e12940 (2016).
- Pervaiz, S., Kopp, A., von Kleist, L., Stauber, T. Absolute protein amounts and relative abundance of volume-regulated anion channel (VRAC) LRRC8 subunits in cells and tissues revealed by quantitative immunoblotting. Int J Mol Sci. 20 (23), 5879 (2019).
- Kern, D. M., et al. Structural basis for assembly and lipid-mediated gating of LRRC8A:C volume-regulated anion channels. Nat Struct Mol Biol. 30 (6), 841-852 (2023).
- Rutz, S., Deneka, D., Dittmann, A., Sawicka, M., Dutzler, R. Structure of a volume-regulated heteromeric LRRC8A/C channel. Nat Struct Mol Biol. 30 (1), 52-61 (2023).
- Li, P., et al. LRRC8 family proteins within lysosomes regulate cellular osmoregulation and enhance cell survival to multiple physiological stresses. Proc Natl Acad Sci U S A. 117 (46), 29155-29165 (2020).
- Kashyap, P., et al. An optogenetic method for the controlled release of single molecules. Nat Methods. 21 (4), 666-672 (2024).
- Liu, T., Stauber, T. The volume-regulated anion channel LRRC8/VRAC is dispensable for cell proliferation and migration. Int J Mol Sci. 20 (11), e2663 (2019).
- Zhang, Y., et al. Polarized NHE1 and SWELL1 regulate migration direction, efficiency and metastasis. Nat Commun. 13 (1), 6128 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten