Method Article
Monitoraggio dell'attività di ripetizione ricca di leucina contenente 8 canali (LRRC8/VRAC) utilizzando il trasferimento di energia di risonanza di Förster a emissione sensibilizzata (SE-FRET)
In questo articolo
Riepilogo
L'elettrofisiologia è il gold standard per lo studio dell'attività dei canali ionici. Tuttavia, ci sono molti approcci alternativi, compresi i metodi ottici. Qui, descriviamo un metodo per monitorare l'attività della ripetizione ricca di leucina contenente canali anionici formati a 8 canali (LRRC8) utilizzando un metodo basato sul trasferimento di energia della risonanza di Förster (FRET) inter-subunità.
Abstract
I membri della famiglia delle proteine LRRC8 formano canali eteromerici ionici e osmolitici con ruoli in numerosi processi fisiologici. Come canali anionici regolati dal volume (VRAC)/canali raddrizzatori verso l'esterno sensibili al volume (VSOR), vengono attivati in seguito al rigonfiamento osmotico delle cellule e mediano l'estrusione di cloruro e osmoliti organici, portando all'efflusso di acqua e quindi al restringimento delle cellule. Oltre al loro ruolo nella regolazione del volume osmotico, i VRAC sono stati implicati in processi cellulari come la differenziazione, la migrazione e l'apoptosi. Attraverso il loro effetto sul potenziale di membrana e il loro trasporto di varie molecole di segnalazione, le ripetizioni ricche di leucina contenenti 8 canali (LRRC8) svolgono un ruolo nella comunicazione neurone-glia, nella secrezione di insulina e nella risposta immunitaria. Il meccanismo di attivazione è rimasto sfuggente. I canali LRRC8, come altri canali ionici, sono tipicamente studiati utilizzando metodi elettrofisiologici. Qui, descriviamo un metodo per rilevare l'attivazione del canale LRRC8 misurando il trasferimento di energia di risonanza di Förster a emissione sensibilizzata intra-complesso (SE-FRET) tra proteine fluorescenti fuse ai domini di ripetizione ricchi di leucina C-terminale delle subunità LRRC8. Questo metodo offre la possibilità di studiare l'attivazione dei canali in situ senza scambio dell'ambiente citosolico e durante processi come il differenziamento cellulare e l'apoptosi.
Introduzione
I canali ionici costituiti da eteromeri di ripetizioni ricche di leucina contenenti 8 proteine della famiglia (LRRC8) si trovano in tutte le cellule dei vertebrati, partecipando a un'ampia gamma di funzioni fisiologiche 1,2. Questi canali LRRC8, identificati per la prima volta come canali anionici regolati dal volume (VRAC) o canali raddrizzatori verso l'esterno sensibili al volume (VSOR), svolgono un ruolo cruciale nella diminuzione del volume regolatorio cellulare 3,4. Facilitano l'espulsione degli ioni cloruro e degli osmoliti organici, seguita dall'efflusso d'acqua in risposta al rigonfiamento osmotico. Oltre al loro ruolo nella risposta allo stress osmotico, il loro ruolo nella regolazione del volume cellulare è stato collegato alla proliferazione e migrazione cellulare, all'apoptosi, alla spermiogenesi e all'integrità epiteliale 5,6,7. È stato dimostrato che l'alterazione del potenziale di membrana in seguito all'attivazione di LRRC8/VRAC contribuisce al differenziamento del miotubo8 e alla secrezione di insulina da parte delle cellule β pancreatiche 9,10,11. Inoltre, i canali LRRC8 conducono una varietà di osmoliti organici come le molecole di segnalazione purinergica ATP e cGAMP o l'amminoacido eccitatorio glutammato, ponendo questi canali nella comunicazione cellula-cellula nel sistema immunitario o nell'interazione glia-neurone 12,13,14,15,16,17,18,19,20,21,22. Anche gli xenobiotici, come il colorante fluoresceina, l'antibiotico blasticidina S o il farmaco antitumorale cisplatino, sono condotti dai canali LRRC8 23,24,25.
Ci sono numerosi rapporti sulla trasduzione del segnale che porta all'attivazione di LRRC8/VRAC 26,27,28. Tuttavia, il meccanismo rimane poco chiaro e la letteratura presenta un'ampia gamma di potenziali meccanismi che potrebbero dipendere dallo specifico processo fisiologico. Questi includono cambiamenti nella forza ionica citosolica, interazione con il citoscheletro, composizione della membrana, proteine G, stato redox e cascate di fosforilazione 2,27,29,30,31.
I canali LRRC8/VRAC contengono LRRC8A come subunitàessenziale 3,4 che deve eteromerizzarsi con almeno uno dei suoi paraloghi LRRC8B-E per formare i canali fisiologicamente funzionali 4,14,32. La composizione delle subunità determina le proprietà biofisiche del canale, come la rettifica e l'inattivazione dipendente dalla depolarizzazione 4,29,32,33,34, la specificità del substrato 15,17,20,21,24,35 e alcune vie di attivazione 36,37. Le strutture di crio-microscopia elettronica (cryo-EM) mostrano che gli omomeri LRRC8A, così come gli eteromeri, si assemblano come esameri 38,39,40, mentre le chimere LRRC8A/LRRC8C che formano canali funzionali sono eptameri 41. La parte N-terminale di tutte le proteine LRRC8 comprende quattro eliche transmembrana e la parte C-terminale contiene un dominio con ripetizioni ricche di leucina (LRRD). Le strutture del complesso LRRC8 disponibili forniscono la prova che gli LRRD, che si estendono nel citosol 3,4,23, possono subire riarrangiamenti conformazionali durante il gating del canale 34,42,43. Questa nozione è corroborata dalla scoperta che la fusione C-terminale di proteine fluorescenti provoca l'attività del canale basale14 e che il legame dei nanocorpi ai domini può modulare l'attività del canale44. Inoltre, alterazioni conformazionali dei C-terminali sono state dimostrate mediante trasferimento di energia di risonanza di Förster intra-complesso (FRET)45.
Il metodo più comune per studiare l'attività dei canali ionici è la misurazione elettrofisiologica46, che è stata ampiamente applicata nello studio dei VRAC prima della loro identificazione molecolare47. Tuttavia, esistono vari modi aggiuntivi per monitorare indirettamente l'attività di VRAC, tra cui la misurazione dei suoi substrati condotti - ioni alogenuri o osmoliti organici - o il suo effetto sul volume cellulare48. Infatti, l'identificazione delle proteine LRRC8 come VRAC si è basata su un saggio basato sul quenching di una proteina fluorescente49 sensibile agli alogenuri da parte dello ioduro che entra nella cellula attraverso i VRAC attivati 3,4. Un altro metodo per monitorare l'attività dei canali LRRC8/VRAC utilizza il movimento dei domini citosolici che può essere osservato, come in altri canali ionici 50,51,52,53, da cambiamenti in FRET45. A tal fine, le proteine fluorescenti che fungono da coppie FRET, come la proteina ciano-fluorescente (CFP)/mCerulean3 come donatore e la proteina giallo-fluorescente (YFP)/mVenus come accettore, sono state fuse ai C-terminali delle proteine LRRC8 (Figura 1). Il FRET intra-complesso tra le subunità LRRC8 è stato dimostrato mediante esperimenti di fotobleaching dell'accettore45. Evitando il metodo distruttivo del fotosbiancamento, le variazioni dei FRET nel tempo sono state monitorate mediante FRET A EMISSIONE SENSIBILIZZATA (SE-FRET), dove sostanzialmente viene misurata l'emissione sensibilizzata dell'accettore in seguito all'eccitazione del donatore dovuta alla sovrapposizione dello spettro di emissione del donatore con lo spettro di eccitazione dell'accettore. L'applicazione dell'ipotonicità extracellulare, uno stimolo per l'attivazione di LRRC8/VRAC, ha determinato una riduzione reversibile dell'intensità SE-FRET45. È importante sottolineare che le misurazioni simultanee del patch-clamp su cellule intere e il monitoraggio FRET durante il trattamento ipotonico hanno dimostrato che questa riduzione di FRET rispecchia effettivamente l'attivazione di LRRC8/VRAC45. Questo metodo, che evita di interrompere la membrana plasmatica o di alterare l'ambiente intracellulare con la soluzione pipettata, offre un'alternativa per il monitoraggio dell'attività di LRRC8/VRAC. È particolarmente utile in contesti fisiologici dove il mantenimento del citosol nativo è cruciale, è necessaria la risoluzione subcellulare o è richiesta un'osservazione prolungata dell'attività del canale.
Qui, presentiamo un protocollo per studiare LRRC8/VRAC con una tale lettura basata su FRET. Il protocollo illustra come gestire e trasfettare le cellule, acquisire immagini di campioni e controlli, analizzare i dati e calcolare i valori di emissione sensibilizzata FRET (SE-FRET).
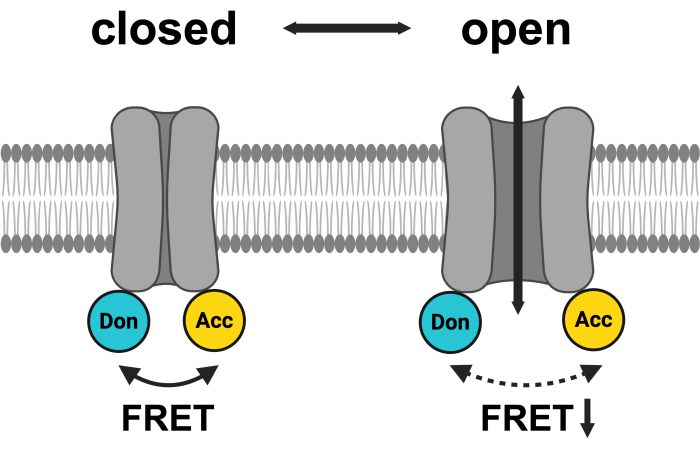
Figura 1: Schema del sistema di coppie di tasti LRRC8. mCerulean3 è mostrato in ciano e mVenus è mostrato in giallo. Dopo l'apertura del VRAC, la distanza (e/o l'orientamento spaziale) tra i fluorofori cambia, con conseguente riduzione del trasferimento di energia tra donatore (Don) e accettore (Acc) e, a sua volta, abbassamento del FRET osservato. Creato con BioRender.com. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Preparazione del tampone e dei reagenti
- Preparare tamponi medi e tamponi isotonici, ipotonici e ipertonici e misurare l'osmolarità dei tamponi con un osmometro per assicurarsi che l'osmolarità (Osm) rientri nell'intervallo previsto con solo una deviazione accettabile.
- Terreni di coltura cellulare: Preparare il Medium Eagle Modified Medium (DMEM) di Dulbecco integrato con il 10% di siero fetale bovino (FBS) e l'1% di penicillina/streptomicina
- Tampone per imaging isotonico (~340 mOsm): Preparare il tampone per imaging isotonico miscelando 150 mM di NaCl, 6 mM di KCl, 1 mM di MgCl2, 1,5 mM di CaCl2, 10 mM di D(+)-glucosio e 10 mM di 4-(2-idrossi etil)-1-piperazina etano sulfonato (HEPES).
- Tampone per imaging ipotonico (~250 mOsm): Preparare il tampone per imaging ipotonico miscelando 105 mM di NaCl, 6 mM di KCl, 1 mM di MgCl2, 1,5 mM di CaCl2, 10 mM di D(+)-glucosio e 10 mM di HEPES.
- Tampone per imaging ipertonico (~500 mOsm): Preparare il tampone per imaging ipertonico miscelando 160 mM di D(-)-Mannitolo, 150 mM di NaCl, 6 mM di KCl, 1 mM di MgCl2, 1,5 mM di CaCl2, 10 mM di D(+)-glucosio e 10 mM di HEPES.
NOTA: I tamponi possono essere integrati con farmaci specifici a seconda di ciò che verrà studiato.
2. Crescita di cellule aderenti su piastre con fondo di vetro
- Preparare almeno una piastra per la sola trasfezione con la proteina di interesse (POI) fusa al fluoroforo donatore o accettore e almeno una piastra per la trasfezione con la coppia donatore e accettore (Tabella 1 e Figura 2). Utilizzare piastre con fondo di vetro per consentire la microscopia a fluorescenza. A seconda del tipo di cella, pretrattare la superficie mediante una pulizia speciale o rivestirla con polilisina.
NOTA: In questo caso, il donatore FRET mCerulean3 (mCer)54 e l'accettore FRET mVenus (mVen)55 sono utilizzati come coppia FRET, ma sono applicabili anche altre combinazioni di fluorofori. Il campione che esprime il POI fuso con il donatore o il solo accettore viene utilizzato per determinare i fattori di correzione β e γ (Tabella 1) necessari per correggere il sanguinamento del donatore nel canale accettore e l'eccitazione crociata dell'accettore da parte della lunghezza d'onda di eccitazione del donatore (Figura 2Ac, Bf). - Il giorno prima della trasfezione, seminare 1 x 105 cellule HeLa in 2 mL di terreno di coltura cellulare su piastre da 35 mm con fondo di vetro adatte alla microscopia a fluorescenza. Colture di cellule per una notte a 37 °C e 5% di CO2 in un incubatore per colture cellulari.
NOTA: I volumi dei reagenti possono essere adattati alla superficie di altre piastre/matracci per colture cellulari di dimensioni diverse. Il numero di cellule può essere adattato a una densità adatta a diverse linee cellulari e approcci sperimentali.- Aspirare il surnatante della coltura cellulare dal monostrato di cellule aderenti coltivate su fiasche di coltura cellulare T75 utilizzando una pompa a vuoto.
- Lavare le cellule aggiungendo 10 mL di soluzione salina tamponata con fosfato (DPBS) di Dulbco al pallone di coltura cellulare e aspirando successivamente la soluzione con una pompa a vuoto.
- Coprire completamente le cellule con 1 ml di tripsina-EDTA allo 0,05% e incubare le cellule per 2 minuti a 37 °C e 5% di CO2 in un incubatore per colture cellulari.
- Aggiungere 9 mL di terreno di coltura cellulare e sospendere le cellule mediante pipettaggio.
- Trasferire la sospensione cellulare in una provetta da 15 mL e centrifugare a 700 x g per 3 minuti a temperatura ambiente (RT).
- Aspirare il surnatante con una pompa a vuoto e risospendere il pellet cellulare in 10 mL di terreno di coltura cellulare.
- Mescolare volumi uguali di sospensione cellulare e blu di tripano allo 0,4% in una provetta e aggiungere 10 μl a un vetrino di conteggio. Posizionare il vetrino nel contatore automatico di celle e iniziare il conteggio utilizzando il programma appropriato per il conteggio delle cellule.
- Per calcolare il volume di sospensione cellulare necessario per ciascuna piastra, dividere il numero di cellule necessarie per piastra (1 x 105) per il numero di cellule per millilitro ottenuto dal contatore di cellule.
- Preparare una sospensione cellulare in una provetta adatta contenente il numero di cellule necessarie per piastra nel volume necessario per piastra (2 ml) più la quantità per una piastra extra.
- Miscelare la sospensione cellulare almeno 20 volte capovolgendo la provetta e pipettare 2 mL di sospensione cellulare in ciascuna capsula.
- Per garantire una distribuzione cellulare più uniforme, lasciare le piastre per 30 minuti a RT prima di metterle nell'incubatore per colture cellulari.
3. Trasfezione cellulare
NOTA: In questo caso, FuGENE è stato utilizzato come reagente di trasfezione. Sono applicabili anche altri reagenti e metodi di trasfezione. I rapporti ottimali tra DNA plasmidico (pDNA) e reagenti di trasfezione e il tempo per l'espressione ottimale per ciascun POI e modello cellulare devono essere valutati empiricamente. In questo caso, sono stati utilizzati 2 μg di pDNA totale per piatto da 35 mm. I costrutti donatore e accettore FRET sono stati utilizzati in un rapporto 1:1 e il rapporto pDNA-reagente di trasfezione era 1:4 (Tabella 1).
- Prima della trasfezione, sostituire il surnatante della coltura cellulare con 2 mL freschi di terreno preriscaldato.
- Diluire il pDNA in Opti-Minimal Essential Medium (MEM) con un volume finale di 100 μL (=soluzione di pDNA).
- Diluire il reagente di trasfezione in Opti-MEM con un volume finale di 100 μl (=soluzione reattiva).
NOTA: Per una trasfezione più uniforme, preparare una miscela master per il pDNA e la soluzione del reagente. - Mescolare bene il pDNA e la soluzione del reagente.
- Aggiungere la soluzione di pDNA nella soluzione reagente (=soluzione di trasfezione).
- Mescolare bene la soluzione di trasfezione.
- Incubare la soluzione di trasfezione per 15 minuti a RT.
- Aggiungere la soluzione di trasfezione goccia a goccia con un movimento a spirale al piatto.
- Mescolare muovendo il piatto 5 volte in orizzontale e in verticale sulla superficie del banco.
- Colture di cellule per una notte a 37 °C e 5% di CO2 in un incubatore per colture cellulari.
| Condizione | Costrutto/i | Campione | Utilizzato per |
| 1 | LRRC8A-mCeruleo | Solo costrutto donatore | Determinare il fattore di correzione β |
| 2 | LRRC8E-mVenere | Solo costrutto accettore | determinare il fattore di correzione γ |
| 3 | LRRC8A-mCeruleo e LRRC8E-mVenere | Coppia di tasti | Quantificazione SE-FRET |
Tabella 1: Esempio di condizioni necessarie per un tipico esperimento SE-FRET per misurare l'attività LRRC8/VRAC di un canale composto dalla subunità LRRC8A fusa al donatore (mCerulean3) e dalla subunità LRRC8E fusa al fluoroforo accettore (mVenus).
4. Acquisizione dell'immagine per la determinazione del fattore di correzione
NOTA: Si verifica uno spurgo dell'emissione donatrice nell'emissione rilevata dell'accettore durante la FRET. Inoltre, c'è un'eccitazione incrociata del fluoroforo accettore da parte della lunghezza d'onda di eccitazione del donatore. Questi processi devono essere compensati durante il calcolo del SE-FRET. A tal fine, i fattori di correzione sono determinati nelle cellule che esprimono solo il donatore o accettore FRET 24 ore dopo la trasfezione. In questo caso, l'imaging è stato effettuato su un Leica THUNDER Imager dotato di una lampada Leica LED8, il CYR71010 del cubo del filtro, un obiettivo HC PL APO 63x/1.40 OIL, un filtro passa-lungo per 460/80 e 553/70 e una fotocamera Leica DFC9000GTC. Gli esperimenti sono stati eseguiti senza controllo ambientale ma in presenza di HEPES nei tamponi di imaging per stabilizzare il pH. Per l'osservazione/misurazione a lungo termine, si consiglia di utilizzare un sistema di controllo ambientale. Per l'analisi, SE-FRET viene calcolato dalle immagini grezze acquisite. Questa operazione può essere eseguita contemporaneamente durante l'acquisizione o successivamente. In questo caso, il software Leica LAS X con il plug-in SE-FRET è stato utilizzato per semplificare la procedura sperimentale relativa al calcolo dei fattori di correzione e alla visualizzazione delle variazioni del valore SE-FRET in tempo reale durante l'acquisizione dell'immagine. Per la post-acquisizione, i fattori di correzione e SE-FRET possono essere determinati con altri pacchetti software (ad es. FIJI) dopo l'acquisizione dei dati grezzi secondo il protocollo fornito di seguito.
- Prima dell'acquisizione dell'immagine di singole cellule trasfettate per calcolare i fattori di correzione, utilizzare un campione che esprime la coppia FRET per impostare l'impostazione della microscopia per tutti i canali (eccitazione del donatore/emissione DD del donatore, DA del donatore/accettore e AA accettore/accettore; vedere Tabella 1 e Figura 2C e Figura 3A).
NOTA: Per le misurazioni SE-FRET con subunità VRAC con tag mCerulean3 e mVenus trasfettate come descritto sopra, sono stati utilizzati i seguenti parametri: pixel binning 8x8, tempo di esposizione 100 ms e intensità della lampada LED del 10%. - Prelevare il campione che esprime solo il costrutto del donatore (Figura 2A), aspirare il terreno di coltura cellulare e lavare le cellule tre volte con 2 mL di tampone isotonico.
- Aggiungere 3 mL di tampone isotonico e posizionare il campione sul tavolino del microscopio.
- Trova un campo visivo (FOV) con almeno una cellula che esprime il costrutto del donatore.
- Immagine di tutti i canali (DD, DA e AA vedere la Tabella 2 e la Figura 2A).
- Disegnare una regione di interesse (ROI) attorno alla cellula o alle celle e misurare l'intensità media di DA (= IDA; Figura 2Ac) e DD (= IDD; Figura 2Aa).
- Per la sottrazione di fondo, disegnare un ROI nei canali DA e DD in cui si trova solo il segnale di fondo e misurare l'intensità media (IBDA e IBDD).
- Sottrarre l'intensità media della ROI di fondo dall'intensità media misurata nella ROI della cella del canale corrispondente (
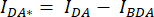 e
e 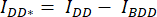 ).
). - Ripetere i passaggi 4.2-4.5 con il campione che esprime solo il costrutto accettore (Figura 2B).
- Disegna un ROI attorno alla cella o alle celle e misura l'intensità media di DA (= IDA; Figura 2Bf) e AA (= IAA; Figura 2Be).
- Per la sottrazione di fondo, tracciare un ROI nei canali DA e AA dove si trova solo il segnale di fondo e misurare le intensità medie (IBDA e IBAA).
- Sottrarre l'intensità media della ROI di fondo dall'intensità media misurata nella ROI della cella del canale corrispondente (
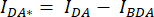 e
e 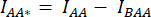 ).
). - Utilizzare i valori determinati per IDA*, IDD* e IAA* per calcolare i fattori
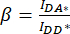 di correzione e
di correzione e 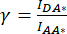 ,
,
In cui il fattore di correzione β viene utilizzato per compensare il sanguinamento dell'emissione del donatore nel canale DA. Il fattore di correzione γ viene utilizzato per compensare l'eccitazione incrociata del fluoroforo accettore da parte della lunghezza d'onda di eccitazione del donatore nel canale DA.
| Eccitazione di | Emissione di | Nome del canale | Linea LED | Cubo filtrante | Filtro passa-lungo |
| Donatore | Donatore | DD | 440 nm | CYR71010 | 460/80 nm |
| Donatore | Accettore | DA | 440 nm | CYR71010 | 535/70 nm |
| Accettore | Accettore | AA | 510 nm | CYR71010 | 535/70 nm |
Tabella 2: Riepilogo dei canali necessari per gli esperimenti SE-FRET.
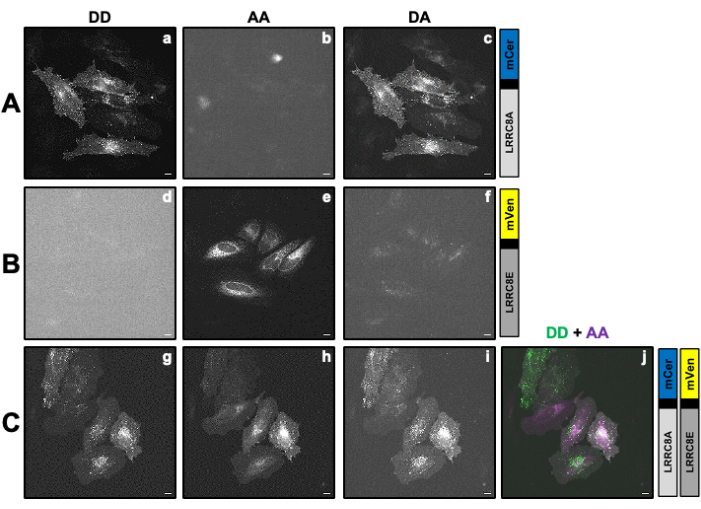
Figura 2: Immagini di fluorescenza rappresentative di campioni utilizzati per calcolare i fattori di correzione β e γ necessari per determinare l'attività VRAC di un canale composto da subunità LRRC8A fusa al donatore mCerulean3 (mCer) e subunità LRRC8E fusa al fluoroforo accettore mVenus (mVen) mediante misure SE-FRET. (A,B) Rilevamento del DD donatore/donatore, dell'AA accettore/accettore e del canale DA donatore/accettore nelle cellule HeLa che esprimono solo il donatore (A) LRRC8A-mCer o l'accettore (B) LRRC8E-mVen. (C) Rilevamento dei canali DD, AA e DA nelle cellule HeLa co-trasfettate con la coppia donatore e accettore LRRC8A-mCer e LRRC8E-mVen. I pannelli a-i mostrano le immagini scattate nel canale di rilevamento del donatore (eccitazione del donatore e rilevamento del segnale del donatore; DD; a, d e g), il canale di rilevamento dell'accettore (eccitazione dell'accettore e rilevamento del segnale dell'accettore; AA; b, e e h ) e il canale di rilevamento del segnale FRET (eccitazione del donatore e rilevamento del segnale accettore; DA; c, f e i). Il pannello j è la sovrapposizione dei pannelli g e h. Il canale DD è mostrato in verde e il canale AA in magenta. Barra della scala = 10 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
5. Imaging time-lapse per la quantificazione SE-FRET
- Prelevare il campione che esprime il costrutto donatore e accettore (Tabella 1, Figura 2C e Figura 3A), aspirare i terreni di coltura cellulare e lavare le cellule tre volte con 2 mL di tampone isotonico 24 ore dopo la trasfezione.
- Aggiungere 3 mL di tampone isotonico e posizionare il campione sul tavolino di microscopia.
- Per una successiva aspirazione del tampone isotonico, fissare e regolare una cannula del tubo flessibile in modo che la punta della cannula raggiunga il fondo del piatto.
- Per aggiungere tamponi, fissare e regolare i tubi in modo che il tampone azionato dal flusso di gravità possa cadere nella capsula.
NOTA: In questo caso è stato utilizzato un sistema di perfusione a gravità controllato da valvola con quattro valvole magnetiche per controllare il flusso tampone, ma sono applicabili anche altri metodi. - Trova un FOV con almeno una cellula che esprime contemporaneamente il costrutto donatore e accettore.
- Impostare un esperimento time-lapse per i canali DD, DA e AA con un intervallo di 10 s e una durata che copra tutte le condizioni della sequenza di stimolazione, ad esempio 12 cicli di linea di base seguiti da 15 cicli per condizione (Figura 3).
NOTA: L'intervallo e il numero di cicli possono essere adottati in base alle esigenze sperimentali, ma dovrebbero sempre includere una misurazione di base per la normalizzazione per una migliore visualizzazione e confronto dei dati. - Dopo la misurazione di base, lavare il campione nel tampone per la prima condizione.
- Aspirare il tampone isotonico attraverso la cannula del tubo, applicando un vuoto con una siringa.
- Aggiungere 3 mL del tampone della condizione successiva mediante flusso di gravità.
- Aspirare il tampone e aggiungere nuovamente 3 mL dello stesso tampone.
- Ripetere ancora una volta il passaggio 5.7.3.
- Dopo aver misurato la prima condizione, lavare il campione nel tampone per la condizione successiva (passaggi 5.7.1-5.7.4).
- Ripetere i passaggi 5.5-5.8 fino a quando tutte le condizioni non vengono acquisite.
- Per la quantificazione SE-FRET, tracciare un ROI attorno alla cellula/e alle celle e misurare l'intensità media nei canali DD = (IDD), DA (IDA) e AA (IAA) per tutte le immagini (punti temporali) nelle serie temporali (Figura 3A, B).
- Per la sottrazione di fondo per il segnale FRET, tracciare una ROI nel canale DA dove si trova solo il segnale di fondo e misurare l'intensità media (IBDA).
- Sottrarre l'intensità media della ROI di fondo dall'intensità media misurata nella ROI della cella del canale DA (
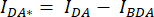 ).
). - Utilizzare i valori determinati per calcolare i valori medi SE-FRET per ciascun ROI e tutti i punti
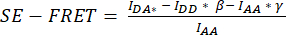 temporali.
temporali. - Per una migliore comparabilità delle diverse condizioni e la visualizzazione dei valori SE-FRET, utilizzare la media dei valori di base di ciascun ROI per normalizzare tutti i punti temporali del ROI corrispondente.
- Tracciare i valori SE-FRET normalizzati nel tempo (Figura 3B).
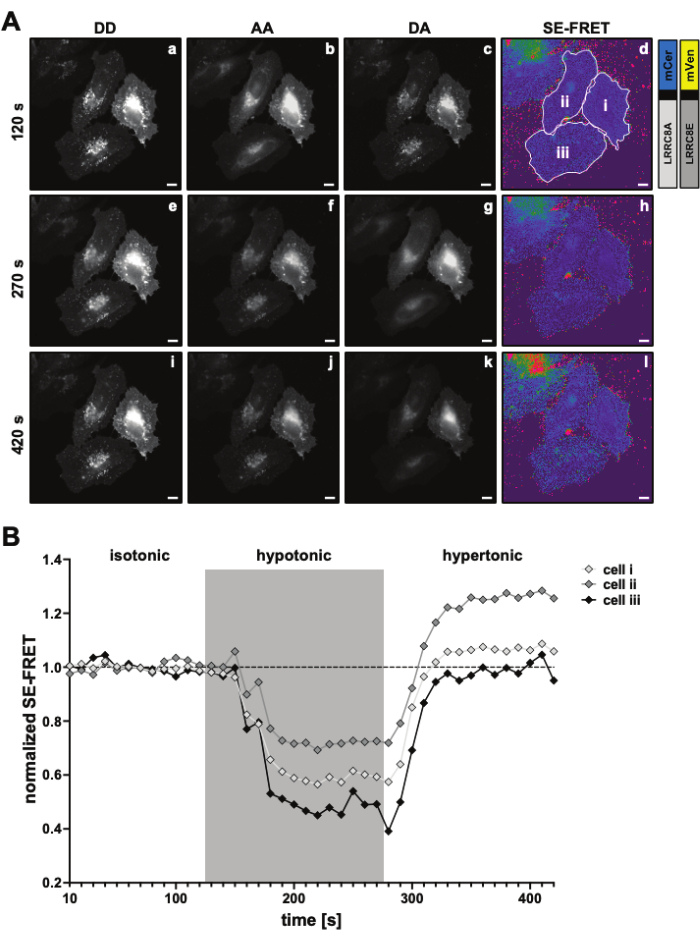
Figura 3: Immagini rappresentative della fluorescenza e quantificazione SE-FRET. (A) Immagini rappresentative della fluorescenza e l'apparente SE-FRET del primo punto temporale di un esperimento di time laps per misurare l'attività VRAC mediante SE-FRET di un canale composto da subunità LRRC8A e LRRC8E a seconda della tonicità. Barra di scala = 10 μm. Le stesse celle sono mostrate nella Figura 2C. I pannelli a-i mostrano il rilevamento dei canali DD, AA e DA e il SE-FRET apparente calcolato. I contorni bianchi rappresentano le ROI (celle i-iii nel pannello d) utilizzate per misurare le intensità medie del segnale in DD, AA e DA e l'immagine SE-FRET apparente. (B) Quantificazione dei valori SE-FRET nel tempo. La sequenza delle condizioni era di 12 cicli di tampone di imaging isotonico (basale) seguiti da 15 cicli di tampone di imaging ipotonico e 15 cicli di tampone di imaging ipertonico. Il valore grezzo medio SE-FRET di ciascun ROI (celle i-iii) e punto temporale è stato normalizzato alla media del valore basale (isotonico) per il ROI corrispondente. Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
Con questo metodo basato su FRET, l'attività di LRRC8/VRAC può essere monitorata durante la stimolazione osmotica e la riduzione di SE-FRET è correlata al grado di ipotonicità extracellulare45. Qui sono mostrati anche i risultati rappresentativi per l'attivazione del canale indotta dall'ipotonicità (Figura 3 e Figura 4). Inoltre, si può osservare l'attivazione di LRRC8/VRAC da parte di diversi stimoli isosmotici, come la manipolazione del segnale del diacilglicerolo45 o durante l'attivazione dei miociti56.
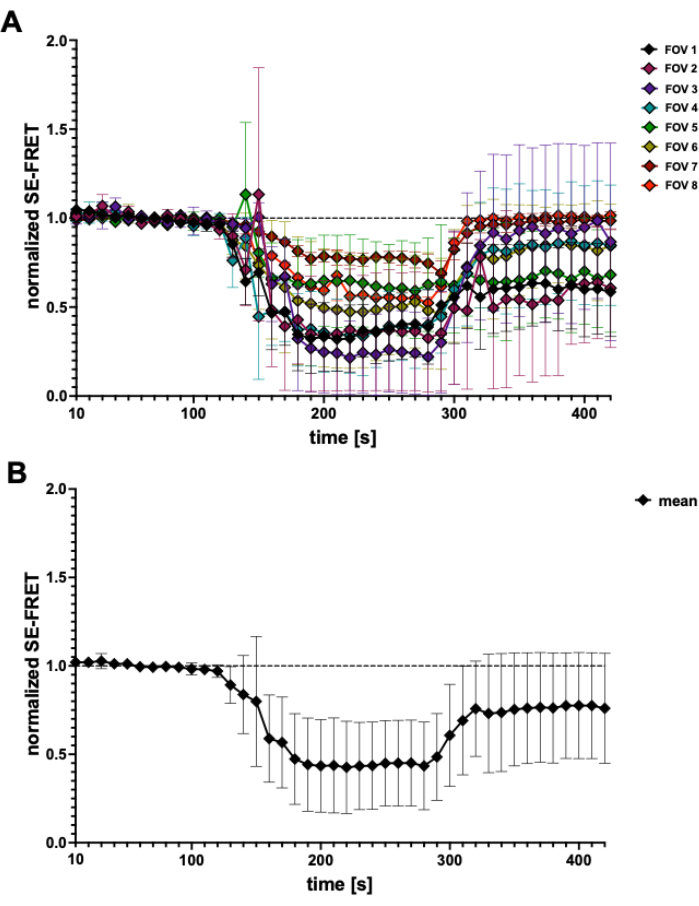
Figura 4: Tracce SE-FRET. (A) Tracce SE-FRET apparenti da 5 esperimenti indipendenti. I dati rappresentano la media ± SD da N = 2 a N = 7 celle per campo visivo (FOV). (B) Medie ± SD di tutte le celle (N = 31 cellule). Clicca qui per visualizzare una versione più grande di questa figura.
Poiché i canali LRRC8/VRAC sono anche coinvolti nella diminuzione del volume apoptotico24,57, l'osservazione dell'attività del canale dopo l'induzione dell'apoptosi sarebbe un'altra applicazione per il metodo qui descritto. Di conseguenza, il protocollo per monitorare SE-FRET nelle cellule HeLa che esprimono LRRC8A-mCerulean3 e LRRC8E-mVenus è stato eseguito applicando farmaci che inducono l'apoptosi mediata dal recettore della morte. In precedenza, era stato dimostrato che il fattore di necrosi tumorale (TNF)-α e la cicloesimide (CHX) evocavano correnti VRAC entro pochi minuti58. Dopo l'aggiunta di 2 ng/mL di TNF-α e 1 μg/mL di CHX in tampone isotonico, si è verificata una robusta diminuzione di SE-FRET (Figura 5). Sostituendo il tampone con un mezzo ipertonico, anche se contenente TNF-α e CHX, i valori di SE-FRET si sono ripresi vicino al basale (Figura 5A), corrispondenti alla diminuzione delle correnti di VRAC nella soluzione del bagno ipertonico durante il trattamento con il ligandoFas 58 che induce l'apoptosi. Il trattamento delle cellule con DMSO, il solvente per TNF-α e CHX, non ha portato alla riduzione di SE-FRET. TNF-α + CHX non hanno influenzato SE-FRET di CFP-18aa-YFP, un costrutto tandem EYFP ed ECFP come controllo FRET59, dimostrando la specificità per LRRC8/VRAC (Figura 5B).
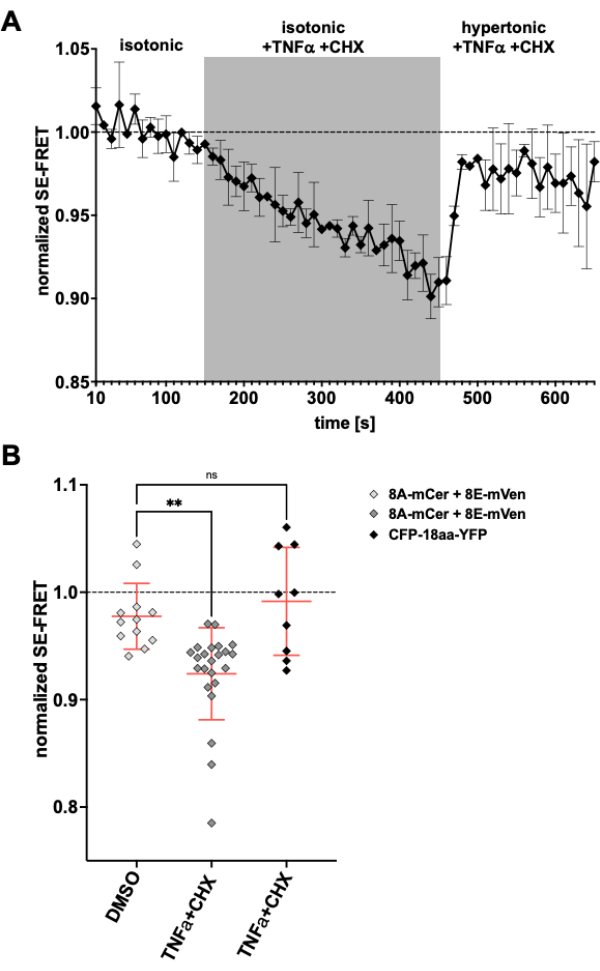
Figura 5: Attivazione isosmotica di VRAC mediante apoptosi mediata dal recettore di morte. (A) Valori normalizzati di SE-FRET da cellule HeLa che esprimono LRRC8A-mCer/LRRC8E-mVen (n = 8 piatti, 23 cellule) nel tempo. Dopo 15 cicli in tampone di imaging isotonico (basale), la soluzione del bagno è stata sostituita da un tampone isotonico integrato con 2 ng/mL di TNF-α e 1 μg/mL di cicloesimide (CHX) per 30 cicli, seguiti da 20 cicli di tampone di imaging ipertonico con TNF-α e CHX. Il valore SE-FRET medio grezzo di ciascun ROI e punto temporale è stato normalizzato alla media del valore basale (isotonico) per il rispettivo ROI. (B) Quantificazione dei valori normalizzati SE-FRET di cellule HeLa che esprimono LRRC8A/E come in A con soluzione isotonica contenente DMSO come veicolo di controllo per CHX (n = 5 piatti, 12 cellule) o tampone isotonico contenente induttori dell'apoptosi come in A (n = 8 piatti, 23 cellule), o di cellule HeLa che esprimono CFP-18aa-YFP con induttori dell'apoptosi (n = 3 piatti, 9 cellule). I dati rappresentano la media degli ultimi 10 punti temporali nel rispettivo buffer delle singole celle (simboli) e la media di tutte le celle ± SD; ** p < 0,01 ANOVA unidirezionale ordinaria seguita dal test post-hoc di confronto multiplo di Tukey. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
La microscopia FRET è una tecnica ben consolidata e ampiamente utilizzata per studiare l'interazione tra proteine. Pertanto, i metodi basati su FRET possono essere applicati in molti laboratori di competenza variabile. I riarrangiamenti conformazionali durante il gating sono stati monitorati per un'ampia gamma di canali ionici utilizzando saggi basati su FRET (per esempi, vedere riferimenti 34,50,51,52,53,60,61,62,63,64,65,66,67), in alcuni casi combinato con l'elettrofisiologia nella fluorimetria patch-clamp 68,69,70,71. FRET può essere utilizzato per studiare le relazioni struttura-funzione di questi canali ionici o per monitorare la loro attività indipendentemente dal trasporto ionico. Il metodo qui presentato può avere chiari vantaggi rispetto all'elettrofisiologia in quanto consente il monitoraggio dell'attività dei canali LRRC8/VRAC in situ.
Le fasi critiche del protocollo includono la placcatura delle cellule per raggiungere la confluenza ottimale per la trasfezione e l'imaging, il che facilita idealmente la facile distinzione delle cellule per l'analisi successiva. Un'efficace co-trasfezione delle diverse subunità è fondamentale per una corretta localizzazione subcellulare; ad esempio, un eccesso della subunità non-LRRC8A porterà a una migliore localizzazione del reticolo endoplasmatico (ER)4. Pertanto, potrebbe essere necessario aggiustare i rapporti plasmidici. A seconda del sistema, le coppie FRET appena generate devono essere verificate, ad esempio mediante sbiancamento dell'accettore. Il binning e il tempo di esposizione devono essere bilanciati l'uno con l'altro per consentire una risoluzione temporale e spaziale ottimale per la domanda di ricerca. Il binning consente tempi di esposizione più brevi e quindi riduce il potenziale sbiancamento del sensore FRET diminuendo la risoluzione spaziale. Pertanto, se la configurazione sperimentale richiede, ad esempio, la discriminazione subcellulare dell'attività di LRRC8/VRAC, il binning dovrebbe essere evitato. La domanda di ricerca determina anche il numero e l'intervallo dei cicli in una serie time-lapse. L'intervallo è rilevante solo se sono richieste le variazioni cinetiche dei FRET (e quindi l'attivazione/inattivazione di LRRC8/VRAC); In caso contrario, è possibile eseguire anche semplici registrazioni "prima e dopo". La durata dell'esperimento dipende dal processo fisiologico. Idealmente, l'attività di LRRC8/VRAC in seguito agli stimoli dovrebbe essere monitorata fino a quando SE-FRET non si è stabilizzato. Questi fattori possono essere determinati in esperimenti pilota. I fattori di correzione per calcolare il segnale SE-FRET reale devono essere determinati per tutte le condizioni. I fattori di correzione determinati in modo errato possono portare a una sovrastima o a una sottostima delle intensità SE-FRET. Infine, dopo aver stabilito una linea di base stabile, l'intervallo di tempo tra le immagini deve essere sufficientemente breve da catturare il processo fisiologico di interesse.
Il metodo presenta alcune limitazioni. Uno di questi è che i cambiamenti nelle intensità dei FRET inter-LRRC8, pur riflettendo i movimenti degli LRRD, non corrispondono necessariamente al trasporto di ioni o osmoliti attraverso il poro. Ciò è chiaro dalle variazioni di FRET osservate con gli omomeri45 di LRRC8A nonostante le loro correnti minime 4,32,72. I bloccanti dei pori dei canali LRRC8/VRAC potrebbero non influenzare il segnale FRET, rendendo questo metodo inadatto per la ricerca di modulatori di canale specifici. Inoltre, i livelli di espressione delle proteine LRRC8 sovraespresse potrebbero influenzare i processi fisiologici osservati, soprattutto perché le proteine LRRC8 marcate con C-terminale mostrano attività basale14.
Un aspetto che può essere considerato un limite o un vantaggio a seconda della particolare domanda di ricerca è che in questo metodo, solo le subunità LRRC8 espresse ectopicamente vengono misurate selettivamente. Quindi, i livelli di fondo di proteine endogene difficilmente interferiscono con le misurazioni. D'altra parte, le proteine sovraespresse potrebbero non comportarsi come i canali endogeni LRRC8 con composizione di subunità e stechiometria potenzialmente diverse. Ad esempio, vari stimoli come l'ossidazione possono avere effetti modulatori opposti su canali LRRC8 composti in modo diverso36. Modificando i rapporti tra le subunità co-espresse, la loro stechiometria e la conduttanza ionica complessiva possono essere regolate14,73, ma la loro composizione nativa, con probabilmente spesso più di due paraloghi all'interno di un complesso21, non è chiara e può variare tra i tipi di cellule 74,75,76. Inoltre, è stato dimostrato che la fusione di proteine fluorescenti con i C-terminali citosolici delle proteine LRRC8 aumenta l'attività basale del canale LRRC8/VRAC negli ovociti di Xenopus 14, probabilmente perché i grandi tag modulano la conformazione dei LRRD, che possono governare l'apertura del canale 14,44,45. Pertanto, la dimensione delle proteine fluorescenti, il linker e il loro orientamento possono non solo influenzare l'efficienza FRET, ma anche l'attività del canale. Tuttavia, è importante sottolineare che i canali VRAC delle proteine LRRC8 fuse con proteine fluorescenti sono rimasti sensibili alla stimolazione ipotonica14, consentendone l'uso come sensori FRET45.
I vantaggi di questo metodo non invasivo per monitorare l'attività del canale LRRC8/VRAC mediante microscopia ottica rispetto ad altri metodi comprendono: (i) Consente di osservare LRRC8/VRAC all'interno di cellule o compartimenti tipicamente inaccessibili per l'elettrofisiologia. Ciò include gli organelli intracellulari su cui i complessi LRRC8 possono essere trovati o mirati a 45,77,78. (ii) La composizione citosolica rimane inalterata dal metodo, mentre durante le misure di patch-clamp su cellule intere, il citosol è in gran parte sostituito da una soluzione pipetta, che può influenzare le vie di segnalazione come osservato con l'attivazione di LRRC8/VRAC indotta da forbolo-12-miristato-13-acetato (PMA)45. (iii) Offre la possibilità di osservare l'attivazione di LRRC8/VRAC con risoluzione subcellulare, come la distinzione dell'attività ai bordi di entrata e di uscita durante la migrazione cellulare, dove -limitato a spazi confinati- VRAC è stato implicato79,80. (iv) Consente il monitoraggio continuo dell'attività di LRRC8/VRAC durante processi fisiologici estesi come la differenziazione dei miociti56.
Sebbene questo metodo presenti limitazioni e sfide, è promettente per ulteriori esplorazioni, comprese le potenziali applicazioni nei modelli animali. In combinazione con altri metodi per studiare questa famiglia di canali ionici e osmolitici, questo saggio basato su FRET può contribuire in modo significativo a svelare i meccanismi di attivazione e ad esplorare le diverse funzioni fisiologiche dei canali LRRC8 nei loro ambienti nativi.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Ringraziamo C.F. Kaminski per il gentile dono del plasmide che codifica per il costrutto CFP-18aa-YFP, A. Klemmer per l'assistenza tecnica e tutti i membri attuali ed ex del laboratorio Stauber che hanno contribuito allo sviluppo di questo metodo.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | gibco | 25300-054 | |
| Camera DFC9000GTC | Leica | 11547007 | |
| CFP-18aa-YFP | N/A | N/A | Elder et al. 2009 PMCID: PMC2706461; Gift from C.F. Kaminski (University of Cambridge, UK) |
| Cycloheximide (CHX) | Sigma-Aldrich | 66-81-9 | |
| D(-)-Mannitol | Carl Roth | 4175.1 | |
| D(+)-Glucose | Carl Roth | HN06.1 | |
| DMEM (Dulbeccos Modified Eagle Medium) | PAN-Biotech | P04-03590 | |
| DPBS (Dulbecco's Phosphate Buffer Saline) | PAN-Biotech | P04-36500 | |
| Emission filter wheel (460/80, 535/70, 590/50, 642/80, 100%) | Leica | 11525480 | |
| FBS (Fetal Bovine Serum) | PAN-Biotech | P30-3302 | |
| Filter cube CYR71010 | Leica | 11525416 | |
| FuGENE | Promega | E2691 | |
| Glas Bottom Culture Dishes 35 mm | MatTek | P35G-0-10-C | |
| HeLa cells | Leibniz Forschungsinstitut DSMZ | ACC 57 | Mammalian cervix carcinoma/ Obtained from Leibniz Forschungsinstitut DSMZ |
| HEPES | Carl Roth | 9105.4 | |
| ibidi µ-Disch 35 mm | ibidi | 81156 | |
| KCl (Potassium chloride) | Carl Roth | 6781.1 | |
| LAS X FRET Wizard | Leica | 11640862 | |
| Light source LED8 | Leica | 11504256 | |
| LRRC8A-mCerulean3 | N/A | N/A | König et al. 2019 |
| LRRC8E-mVenus | N/A | N/A | König et al. 2019 |
| Luna-II Automated Cell Counter | logos biosystems | L40002 | |
| Luna-II Cell Counter Slides | logos biosystems | L12001 | |
| MgCl2 (Magnesium chloride) | Carl Roth | KK36.1 | |
| Microscope THUNDER Imager live cell | Leica | 11525681 | |
| NaCl (Sodium chloride) | Carl Roth | 9263 | |
| Objective HC PL APO 63x/1.40 OIL | Leica | 11506349 | |
| Opti-Minimal Essential Medium (MEM) | gibco | 11058 | |
| Osmometer OM807 | Vogel | V04807 | |
| Penicillin Streptomycin (Pen Step) | gibco | 15070-063 | |
| Trypan blue solution (0,4%) | Sigma | T8154 | |
| Tumor necrosis factor (TNF)-a | Sigma-Aldrich | 94948-59-1 | |
| Valve Controlled Gravity Perfusion System | ALA Scientific Instruments | VC3-4xG |
Riferimenti
- Jentsch, T. J. VRACs and other ion channels and transporters in the regulation of cell volume and beyond. Nat Rev Mol Cell Biol. 17 (5), 293-307 (2016).
- Chen, L., et al. More than just a pressure relief valve: physiological roles of volume-regulated LRRC8 anion channels. Biol Chem. 400 (11), 1481-1496 (2019).
- Qiu, Z., et al. SWELL1, a plasma membrane protein, is an essential component of volume-regulated anion channel. Cell. 157 (2), 447-458 (2014).
- Voss, F. K., et al. Identification of LRRC8 heteromers as an essential component of the volume-regulated anion channel VRAC. Science. 344 (6184), 634-638 (2014).
- Hoffmann, E. K., Schettino, T., Marshall, W. S. The role of volume-sensitive ion transport systems in regulation of epithelial transport. Comp Biochem Physiol A Mol Integr Physiol. 148 (1), 29-43 (2007).
- López-Cayuqueo, K. I., et al. Renal deletion of LRRC8/VRAC channels induces proximal tubulopathy. J Am Soc Nephrol. 33 (8), 1528-1545 (2022).
- Lück, J. C., Puchkov, D., Ullrich, F., Jentsch, T. J. LRRC8/VRAC anion channels are required for late stages of spermatid development in mice. J Biol Chem. 293 (30), 11796-11808 (2018).
- Chen, L., Becker, T. M., Koch, U., Stauber, T. The LRRC8/VRAC anion channel facilitates myogenic differentiation of murine myoblasts by promoting membrane hyperpolarization. J Biol Chem. 294 (39), 14279-14288 (2019).
- Best, L., Brown, P. D., Sener, A., Malaisse, W. J. Electrical activity in pancreatic islet cells: The VRAC hypothesis. Islets. 2 (2), 59-64 (2010).
- Kang, C., et al. SWELL1 is a glucose sensor regulating beta-cell excitability and systemic glycaemia. Nat Commun. 9 (1), 367 (2018).
- Stuhlmann, T., Planells-Cases, R., Jentsch, T. J. LRRC8/VRAC anion channels enhance beta-cell glucose sensing and insulin secretion. Nat Commun. 9 (1), 1974 (2018).
- Hisadome, K., et al. Volume-regulated anion channels serve as an auto/paracrine nucleotide release pathway in aortic endothelial cells. J Gen Physiol. 119 (6), 511-520 (2002).
- Burow, P., Klapperstück, M., Markwardt, F. Activation of ATP secretion via volume-regulated anion channels by sphingosine-1-phosphate in RAW macrophages. Pflügers Arch. 467 (6), 1215-1226 (2015).
- Gaitán-Peñas, H., et al. Investigation of LRRC8-mediated volume-regulated anion currents in Xenopus oocytes. Biophys J. 111 (7), 1429-1443 (2016).
- Lahey, L. J., et al. LRRC8A:C/E heteromeric channels are ubiquitous transporters of cGAMP. Mol Cell. 80 (4), 578-591 (2020).
- Chen, X., et al. Regulation of anion channel LRRC8 volume-regulated anion channels in transport of 2'3'-cyclic GMP-AMP and cisplatin under steady state and inflammation. J Immunol. 206 (9), 2061-2074 (2021).
- Zhou, C., et al. Transfer of cGAMP into bystander cells via LRRC8 volume-regulated anion channels augments STING-mediated interferon responses and anti-viral immunity. Immunity. 52 (5), 767-781 (2020).
- Feustel, P. J., Jin, Y., Kimelberg, H. K. Volume-regulated anion channels are the predominant contributors to release of excitatory amino acids in the ischemic cortical penumbra. Stroke. 35 (5), 1164-1168 (2004).
- Mongin, A. A. Volume-regulated anion channel--a frenemy within the brain. Pflügers Arch. 468 (3), 421-441 (2016).
- Schober, A. L., Wilson, C. S., Mongin, A. A. Molecular composition and heterogeneity of the LRRC8-containing swelling-activated osmolyte channels in primary rat astrocytes. J Physiol. 595 (22), 6939-6951 (2017).
- Lutter, D., Ullrich, F., Lueck, J. C., Kempa, S., Jentsch, T. J. Selective transport of neurotransmitters and modulators by distinct volume-regulated LRRC8 anion channels. J Cell Sci. 130 (6), 1122-1133 (2017).
- Yang, J., et al. Glutamate-releasing SWELL1 channel in astrocytes modulates synaptic transmission and promotes brain damage in stroke. Neuron. 102 (4), 813-827 (2019).
- Lee, C. C., Freinkman, E., Sabatini, D. M., Ploegh, H. L. The protein synthesis inhibitor blasticidin s enters mammalian cells via leucine-rich repeat-containing protein 8D. J Biol Chem. 289 (24), 17124-17131 (2014).
- Planells-Cases, R., et al. Subunit composition of VRAC channels determines substrate specificity and cellular resistance to Pt-based anticancer drugs. EMBO J. 34 (24), 2993-3008 (2015).
- Model, M. A., Nia, F. H., Zook, E., Hollembeak, J. E., Stauber, T. Uptake of fluorescein upon osmotic cell swelling is dependent on the volume-regulated anion channel VRAC/LRRC8. Paracelsus Proc Exp Med. 1 (1), 3-14 (2022).
- Stauber, T. The volume-regulated anion channel is formed by LRRC8 heteromers - molecular identification and roles in membrane transport and physiology. Biol Chem. 396 (9-10), 975-990 (2015).
- Bertelli, S., et al. Mechanisms of activation of LRRC8 volume regulated anion channels. Cell Physiol Biochem. 55 (S1), 41-56 (2021).
- Liu, T., Li, Y., Wang, D., Stauber, T., Zhao, J. Trends in volume-regulated anion channel (VRAC) research: visualization and bibliometric analysis from 2014 to 2022. Front Pharmacol. 14, 1234885 (2023).
- Strange, K., Yamada, T., Denton, J. S. A 30-year journey from volume-regulated anion currents to molecular structure of the LRRC8 channel. J Gen Physiol. 151 (2), 100-117 (2019).
- Osei-Owusu, J., Yang, J., Vitery, M. D. C., Qiu, Z. Molecular biology and physiology of volume-regulated anion channel (VRAC). Curr Top Membr. 81, 177-203 (2018).
- Friard, J., Laurain, A., Rubera, I., Duranton, C. LRRC8/VRAC channels and the redox balance: A complex Relationship. Cell Physiol Biochem. 55 (S1), 106-118 (2021).
- Syeda, R., et al. LRRC8 proteins form volume-regulated anion channels that sense ionic strength. Cell. 164 (3), 499-511 (2016).
- Ullrich, F., Reincke, S. M., Voss, F. K., Stauber, T., Jentsch, T. J. Inactivation and anion selectivity of volume-regulated anion channels (VRACs) depend on C-terminal residues of the first extracellular loop. J Biol Chem. 291 (33), 17040-17048 (2016).
- König, B., Stauber, T. Biophysics and structure-function relationships of LRRC8-formed volume-regulated anion channels. Biophys J. 116 (7), 1185-1193 (2019).
- Concepcion, A. R., et al. The volume-regulated anion channel LRRC8C suppresses T cell function by regulating cyclic dinucleotide transport and STING-p53 signaling. Nat Immunol. 23 (2), 287-302 (2022).
- Gradogna, A., Gavazzo, P., Boccaccio, A., Pusch, M. Subunit-dependent oxidative stress sensitivity of LRRC8 volume-regulated anion channels. J Physiol. 595 (21), 6719-6733 (2017).
- Bertelli, S., Zuccolini, P., Gavazzo, P., Pusch, M. Molecular determinants underlying volume-regulated anion channel subunit-dependent oxidation sensitivity. J Physiol. 600 (17), 3965-3982 (2022).
- Deneka, D., Sawicka, M., Lam, A. K. M., Paulino, C., Dutzler, R. Structure of a volume-regulated anion channel of the LRRC8 family. Nature. 558 (7709), 254-259 (2018).
- Kasuya, G., et al. Cryo-EM structures of the human volume-regulated anion channel LRRC8. Nat Struct Mol Biol. 25 (9), 797-804 (2018).
- Kefauver, J. M., et al. Structure of the human volume regulated anion channel. Elife. 7, e38461 (2018).
- Takahashi, H., Yamada, T., Denton, J. S., Strange, K., Karakas, E. Cryo-EM structures of an LRRC8 chimera with native functional properties reveal heptameric assembly. Elife. 12, e82431 (2023).
- Sawicka, M., Dutzler, R. Regulators of cell volume: The structural and functional properties of anion channels of the LRRC8 family. Curr Opin Struct Biol. 74, 102382 (2022).
- Kasuya, G., Nureki, O. Recent advances in the structural biology of the volume-regulated anion channel LRRC8. Front Pharmacol. 13, 896532 (2022).
- Deneka, D., et al. Allosteric modulation of LRRC8 channels by targeting their cytoplasmic domains. Nat Commun. 12 (1), 5435 (2021).
- König, B., Hao, Y., Schwartz, S., Plested, A. J., Stauber, T. A FRET sensor of C-terminal movement reveals VRAC activation by plasma membrane DAG signaling rather than ionic strength. Elife. 8, e45421 (2019).
- Hille, B. . Ion Channels of Excitable Membranes. 3rd edn. , (2001).
- Pedersen, S. F., Okada, Y., Nilius, B. Biophysics and physiology of the volume-regulated anion channel (VRAC)/volume-sensitive outwardly rectifying anion channel (VSOR). Pflügers Arch. 468 (3), 371-383 (2016).
- Kolobkova, Y., Pervaiz, S., Stauber, T. The expanding toolbox to study the LRRC8-formed volume-regulated anion channel VRAC. Curr Top Membr. 88, 119-163 (2021).
- Galietta, L. J., Haggie, P. M., Verkman, A. S. Green fluorescent protein-based halide indicators with improved chloride and iodide affinities. FEBS Lett. 499 (3), 220-224 (2001).
- Bykova, E. A., Zhang, X. D., Chen, T. Y., Zheng, J. Large movement in the C terminus of CLC-0 chloride channel during slow gating. Nat Struct Mol Biol. 13 (12), 1115-1119 (2006).
- Zheng, J., Zagotta, W. N. Gating rearrangements in cyclic nucleotide-gated channels revealed by patch-clamp fluorometry. Neuron. 28 (2), 369-374 (2000).
- Miranda, P., et al. State-dependent FRET reports calcium- and voltage-dependent gating-ring motions in BK channels. Proc Natl Acad Sci U S A. 110 (13), 5217-5222 (2013).
- Zachariassen, L. G., et al. Structural rearrangement of the intracellular domains during AMPA receptor activation. Proc Natl Acad Sci U S A. 113 (27), E3950-E3959 (2016).
- Markwardt, M. L., et al. An improved cerulean fluorescent protein with enhanced brightness and reduced reversible photoswitching. PLoS One. 6 (3), e17896 (2011).
- Nagai, T., et al. A variant of yellow fluorescent protein with fast and efficient maturation for cell-biological applications. Nat Biotechnol. 20 (1), 87-90 (2002).
- Chen, L., König, B., Stauber, T. LRRC8 channel activation and reduction in cytosolic chloride concentration during early differentiation of C2C12 myoblasts. Biochem Biophys Res Commun. 532, 482-488 (2020).
- Maeno, E., Ishizaki, Y., Kanaseki, T., Hazama, A., Okada, Y. Normotonic cell shrinkage because of disordered volume regulation is an early prerequisite to apoptosis. Proc Natl Acad Sci U S A. 97 (17), 9487-9492 (2000).
- Shimizu, T., Numata, T., Okada, Y. A role of reactive oxygen species in apoptotic activation of volume-sensitive Cl- channel. Proc Natl Acad Sci U S A. 101 (17), 6770-6773 (2004).
- Elder, A. D., et al. A quantitative protocol for dynamic measurements of protein interactions by Förster resonance energy transfer-sensitized fluorescence emission. Journal of the Royal Society Interface. 6, S59-S81 (2009).
- Glauner, K. S., Mannuzzu, L. M., Gandhi, C. S., Isacoff, E. Y. Spectroscopic mapping of voltage sensor movement in the Shaker potassium channel. Nature. 402 (6763), 813-817 (1999).
- Dai, G., Aman, T. K., DiMaio, F., Zagotta, W. N. The HCN channel voltage sensor undergoes a large downward motion during hyperpolarization. Nat Struct Mol Biol. 26 (8), 686-694 (2019).
- Renart, M. L., et al. Conformational plasticity in the KcsA potassium channel pore helix revealed by homo-FRET studies. Sci Rep. 9 (1), 6215 (2019).
- Wang, S., et al. Potassium channel selectivity filter dynamics revealed by single-molecule FRET. Nat Chem Biol. 15 (4), 377-383 (2019).
- Harley, C. A., et al. Conformation-sensitive antibody reveals an altered cytosolic PAS/CNBh assembly during hERG channel gating. Proc Natl Acad Sci U S A. 118 (44), e2108796118 (2021).
- Han, S., et al. Structural dynamics determine voltage and pH gating in human voltage-gated proton channel. Elife. 11, e73093 (2022).
- Cullinan, M. M., Klipp, R. C., Camenisch, A., Bankston, J. R. Dynamic landscape of the intracellular termini of acid-sensing ion channel 1a. Elife. 12, e90755 (2023).
- Kim, J., Won, J., Chung, D. K., Lee, H. H. FRET analysis of the temperature-induced structural changes in human TRPV3. Sci Rep. 13 (1), 10108 (2023).
- Zheng, J., Zagotta, W. N. Patch-clamp fluorometry recording of conformational rearrangements of ion channels. Sci STKE. (176), PL7 (2003).
- Kusch, J., Zifarelli, G. Patch-clamp fluorometry: electrophysiology meets fluorescence. Biophys J. 106 (6), 1250-1257 (2014).
- Cowgill, J., Chanda, B. The contribution of voltage clamp fluorometry to the understanding of channel and transporter mechanisms. J Gen Physiol. 151 (10), 1163-1172 (2019).
- Bhat, S., Blunck, R. Characterising ion channel structure and dynamics using fluorescence spectroscopy techniques. Biochem Soc Trans. 50 (5), 1427-1445 (2022).
- Yamada, T., Figueroa, E. E., Denton, J. S., Strange, K. LRRC8A homohexameric channels poorly recapitulate VRAC regulation and pharmacology. Am J Physiol Cell Physiol. 320 (3), C293-C303 (2021).
- Yamada, T., Wondergem, R., Morrison, R., Yin, V. P., Strange, K. Leucine-rich repeat containing protein LRRC8A is essential for swelling-activated Cl- currents and embryonic development in zebrafish. Physiol Rep. 4 (19), e12940 (2016).
- Pervaiz, S., Kopp, A., von Kleist, L., Stauber, T. Absolute protein amounts and relative abundance of volume-regulated anion channel (VRAC) LRRC8 subunits in cells and tissues revealed by quantitative immunoblotting. Int J Mol Sci. 20 (23), 5879 (2019).
- Kern, D. M., et al. Structural basis for assembly and lipid-mediated gating of LRRC8A:C volume-regulated anion channels. Nat Struct Mol Biol. 30 (6), 841-852 (2023).
- Rutz, S., Deneka, D., Dittmann, A., Sawicka, M., Dutzler, R. Structure of a volume-regulated heteromeric LRRC8A/C channel. Nat Struct Mol Biol. 30 (1), 52-61 (2023).
- Li, P., et al. LRRC8 family proteins within lysosomes regulate cellular osmoregulation and enhance cell survival to multiple physiological stresses. Proc Natl Acad Sci U S A. 117 (46), 29155-29165 (2020).
- Kashyap, P., et al. An optogenetic method for the controlled release of single molecules. Nat Methods. 21 (4), 666-672 (2024).
- Liu, T., Stauber, T. The volume-regulated anion channel LRRC8/VRAC is dispensable for cell proliferation and migration. Int J Mol Sci. 20 (11), e2663 (2019).
- Zhang, Y., et al. Polarized NHE1 and SWELL1 regulate migration direction, efficiency and metastasis. Nat Commun. 13 (1), 6128 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon