Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Festlegen einer erfolgreichen Sortierung für die Isolierung extrazellulärer Vesikel
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet eine detaillierte Beschreibung der Sortierung extrazellulärer Vesikel (EVs), die von mesenchymalen Stromazellen freigesetzt werden. Dabei geht es insbesondere um die Geräteeinstellung und die Optimierung der Sortierbedingungen. Ziel ist es, extrazelluläre Vesikel zu sortieren und dabei ihre Eigenschaften zu erhalten.
Zusammenfassung
Extrazelluläre Vesikel (EVs), die von mesenchymalen Stromazellen (MSCs) freigesetzt werden, enthalten eine Reihe von microRNAs mit regenerativen und entzündungshemmenden Rollen. Daher werden gereinigte MSC-EVs als therapeutische Option der nächsten Generation für eine Vielzahl von Krankheiten angesehen. In diesem Protokoll berichten wir über die Strategie zur erfolgreichen Sortierung von EVs aus dem Überstand von aus Fett gewonnenen MSCs (ASCs), die häufig in orthopädischen regenerativen Medizinanwendungen verwendet werden.
Zunächst haben wir die Probenvorbereitung beschrieben, wobei wir uns auf die EV-Isolierung und die Markierungsschritte mit Carboxyfluorescein-Succinimidylester (CFSE) für die Fluoreszenzdetektion konzentriert haben. Anschließend haben wir den Sortierprozess detailliert beschrieben, der den Hauptteil des Protokolls ausmacht.
Zusätzlich zu den in MISEV 2023 und den MIFlowCyt EV-Richtlinien definierten Regeln haben wir spezifische experimentelle Bedingungen in Bezug auf Düsengröße, Frequenz und Manteldruck angewendet. Die morphologischen Parameter werden anhand von Kügelchen mit Durchmessern ermittelt, die ausgewählt wurden, um den theoretischen Bereich der EV-Größe abzudecken. Nach der Sortierung der ASC-EVs führten wir eine Reinheitsprüfung der sortierten Fraktion durch, indem wir sie mit dem Sortierer erneut analysierten und die EV-Größenverteilung mit der Nanopartikel-Tracking-Analysetechnik überprüften.
Aufgrund der zunehmenden Bedeutung von Elektrofahrzeugen wird es immer wichtiger, eine reine Population zu untersuchen und zu charakterisieren. Hier zeigen wir eine Gewinnerstrategie, um die Sortierung so einzurichten, dass dieses Ziel erreicht wird.
Einleitung
Extrazelluläre Vesikel (EVs) sind eine heterogene Gruppe von membranstrukturierten Vesikel, die von fast allen Zellen freigesetzt werden, durch eine Lipiddoppelschicht begrenzt sind und sich nicht selbst replizieren können1. Sie sind in verschiedenen Bioflüssigkeiten wie Blutplasma, Serum, Speichel, Muttermilch, Urin, Bronchialspülflüssigkeit, Fruchtwasser, Liquor cerebrospinalis und malignem Aszitesenthalten 2. Eine der Hauptfunktionen von EVs besteht darin, verschiedene Moleküle, darunter Nukleinsäuren, Proteine, Lipide und Kohlenhydrate, zwischen einer Spender- und einer Empfängerzelle zu transportieren. Dies kann durch verschiedene Mechanismen geschehen, wie z. B. direkte Membranfusion, Rezeptor-Liganden-Interaktion, Endozytose und Phagozytose 3,4. Aus diesem Grund wurde gezeigt, dass sie eine wichtige Rolle bei vielen physiologischen und pathologischen Prozessen spielen, und sie sind vielversprechend als neuartige Biomarker für Krankheiten, als Vehikel für die Verabreichung von Medikamenten und als therapeutische Wirkstoffe 5,6.
Mesenchymale Stromazellen (MSCs) sind multipotente Zellen, die aus vielen Geweben isoliert werden können, darunter Fettgewebe, Zahnpulpa, Nabelschnurblut, Plazenta, Fruchtwasser, Wharton-Gelee und sogar Gehirn, Lunge, Thymus, Bauchspeicheldrüse, Milz, Leber und Niere. In den letzten Jahren haben sie ein erhebliches Interesse an der regenerativen Medizin geweckt7. Aus Fettgewebe gewonnene mesenchymale Stammzellen (ASCs) können im Vergleich zu anderen Quellen wie Knochenmark durch ein weniger invasives Verfahren aus Fettgewebe gewonnen werden, was zu einem geringeren Risiko schwerer Komplikationen führt und ethische Fragen vermeidet8.
Darüber hinaus enthält Fettgewebe eine signifikant höhere Konzentration an MSCs als Knochenmark (1 % gegenüber >0,01 %) und andere Quellen wie Dermis, Zahnpulpa, Nabelschnur und Plazenta. MSCs sind aufgrund ihrer Differenzierungsfähigkeit und ihrer Sekretion eines breiten Repertoires an Wachstumsfaktoren, Chemokinen und Zytokinen von entscheidender Bedeutung für die Regeneration verletzter Gewebe und Zellen; Dieser therapeutische Nutzen ist auf ihre Differenzierungsfähigkeit zurückzuführen, aber auch auf die Tatsache, dass sie ein breites Repertoire an Wachstumsfaktoren, Chemokinen und Zytokinen sezernieren. Ein markantes Beispiel ist das therapeutische Potenzial von MSCs für orthopädische Erkrankungen, wobei der Begriff "Muskel-Skelett-Erkrankungen" die höhere Anzahl registrierter klinischer Studien unter clinicaltrials.gov aufweist (abgerufen am 13. Mai 2024).
Darüber hinaus können MSCs auch EVs sezernieren, die an der Geweberegeneration beteiligt sind, indem sie Informationen auf geschädigte Zellen oder Gewebe übertragen und eine ähnliche biologische Aktivität wie die Mutterzellen ausüben 9,10. Aus diesem Grund können MSC-EVs ein wertvoller Ersatz für die Zelltherapie sein, um einen zellfreien Ansatz zu erreichen11, wobei zwei klinische Studien mit MSC-EVs bei orthopädischen Erkrankungen (NCT05261360 und NCT04998058) durchgeführt wurden. Für die klinischen Anwendungen von Elektrofahrzeugen gibt es jedoch noch einige Herausforderungen. Zum Beispiel gibt es einige Bedenken hinsichtlich der Isolationstechniken von Elektrofahrzeugen: Die meisten von ihnen garantieren keine Reinheit oder Integrität der Vesikel. Darüber hinaus sind einige Isolationstechniken komplex, zeitaufwändig und haben eine geringe Wiederholbarkeit, was sie für den klinischen Einsatz ungeeignet macht12.
Die Zellsortierung hingegen ist eine häufig verwendete Methode, die es ermöglicht, einzelne Zellen aus heterogenen Zellsuspensionen unter Verwendung spezifischer Fluoreszenzmarkerzu isolieren 13. Er kann für viele Anwendungen eingesetzt und an verschiedene Probentypen angepasst werden. Obwohl die Zellsortierung eine etablierte und weit verbreitete Technologie ist, ist die EV-Sortierung immer noch eine große Herausforderung, da die meisten EVs selbst für die empfindlichsten Durchflusszytometer unter der minimalen Nachweisschwelle liegen. Es gibt einige Merkmale, die einen Sortierer für diesen Zweck besser geeignet machen. Zunächst einmal mit einem Jet-in-Air-System, bei dem der Strom, der die Partikel suspendiert, von Lasern in der Luft und nicht in einer Durchflusszelle abgefragt wird; Dieses System schont die Probe, indem es die Spannung, der sie ausgesetzt ist, verringert. Ein zweiter wichtiger Punkt ist das Vorhandensein eines "Abdunkelungs"-Balkens zwischen dem Strahl und der Sammellinse, der das optische Hintergrundrauschen des Instruments verringert. Obwohl es niedrig ist, wird das Hintergrundrauschen nicht vollständig eliminiert und stellt eine Referenz dar, die ein teilweises Fenster in die Ereignisse bietet, die unter den Schwellenwert fallen: Es ist sehr wichtig für die Analyse von Ereignissen, die nahe der "Nachweisgrenze" des Instruments14 liegen. Schließlich verfügt der Sortierer über einen Dual-Path Forward Scatter (FSC) mit zwei verschiedenen Masken, die eine verbesserte Unterscheidung zwischen kleinen und großen Partikeln in der Probe ermöglichen.
Auf dieser Grundlage haben wir ein Protokoll entwickelt, das darauf abzielt, Carboxyfluorescein-Succinimidylester (CFSE)-markierte MSC-EVs mit Hilfe eines hochempfindlichen Zellsortierers zu trennen. Um die Manipulation von EVs zu minimieren und ihre Integrität und Menge zu erhalten, haben wir Ultrazentrifugationsschritte während der Probenvorbereitung vermieden. Darüber hinaus wurden die Sortierbedingungen angepasst, um die Belastung der Vesikel zu minimieren, einschließlich einer weiteren Optimierung unseres Instruments durch Reduzierung des mit der Düsengröße verbundenen Sortierdrucks (70 μm Düse für einen Druck von 35 psi).
Access restricted. Please log in or start a trial to view this content.
Protokoll
Das Protokoll besteht hier aus vier Teilen: (1) Probenvorbereitung, (2) Charakterisierung von ASC-EVs, (3) Sortierung von ASC-EVs und (4) Analyse nach der Sortierung. Ein Schema, das den Arbeitsablauf darstellt, ist in Abbildung 1 dargestellt.
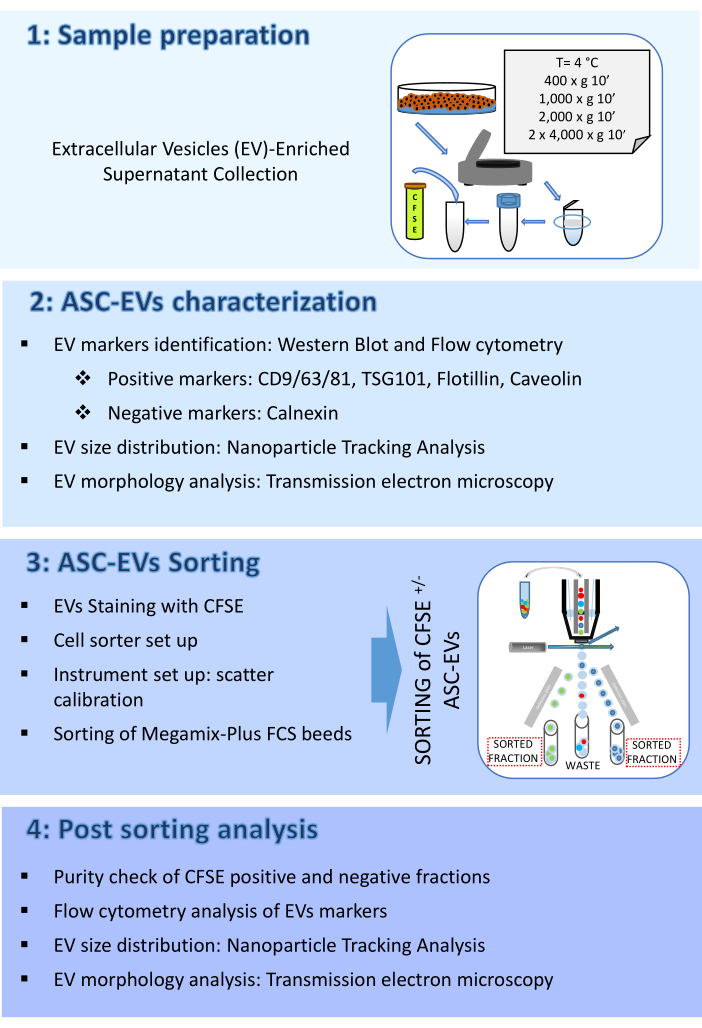
Abbildung 1: Ablaufdiagramm des Protokolls. Das Flussdiagramm zeigt die Schritte, die an dem Protokoll beteiligt sind. (1) Probenvorbereitung, (2) Charakterisierung von Vesikeln vor der Sortierung, (3) Sortierung und (4) Analyse von Vesikeln nach der Sortierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Vorbereitung der Probe
- Mit extrazellulären Vesikeln (EV) angereicherte Überstandsansammlung
- Tauen oder sammeln Sie Fett-MSCs (ASCs), die in der Passage vor der EV-Entnahme kultiviert wurden (normalerweise Passage 1 bis 5), und säen Sie pro ASC-Isolat eine identische Menge an Zellen aus (z. B. 1 Million Zellen pro 175 cm2 der Kolbenoberfläche ergibt eine Konfluenz von etwa 60 % bis 70 %).
- Züchten Sie ASCs in dem geeigneten Kulturmedium (DMEM-F12 für dieses Protokoll), ergänzt mit EV-freiem fötalem Rinderserum (FBS) oder humanem Thrombozytenlysat (hPL), gemäß dem angeforderten Protokoll, für 48-72 Stunden.
- Um EV-freies FBS oder hPL zu erhalten, ultrazentrifugieren Sie bei 120.000 x g über Nacht bei 4 °C und verwenden Sie den Überstand.
HINWEIS: Ab diesem Zeitpunkt ist die angegebene Zentrifugalkraft immer als Durchschnitt für das verwendete Instrument, den Rotor und die Rohre zu verstehen. Beachten Sie, dass verschiedene Rotoren oder Rohre eine variable g-Kraft pro U/min und einen k-Faktor haben können. Eine einfache Möglichkeit, Rotoren zu vergleichen und G-Kraft und Drehzahl einzustellen, finden Sie in einer der verfügbaren Umrechnungstabellen https://www.beckman.it/centrifuges/rotors/calculator.
- Um EV-freies FBS oder hPL zu erhalten, ultrazentrifugieren Sie bei 120.000 x g über Nacht bei 4 °C und verwenden Sie den Überstand.
- Bei 90 % Zellkonfluenz ASCs ablösen, zählen und in einem geeigneten Volumen serumfreien Mediums suspendieren, idealerweise 1 ml pro 1 x 106 ASCs. ASCs in Suspension in 24-Well-Platten mit 1 mL pro Well aussäen. Ohne Serum bleiben die Zellen in Suspension und bilden ein Sphäroid. Nach 96 h den Überstand einsammeln.
- Entfernen Sie schwimmende Zellen und Ablagerungen durch serielle Zentrifugation bei 4 °C: 400 x g für 10 min, 1.000 x g für 10 min, 2.000 x g für 10 min und zweimal 4.000 x g für 10 min.
- Filtrieren Sie den Überstand durch einen 0,22-μm-Filter, um verbleibende Partikel zu entfernen, die größer als 220 nm sind.
HINWEIS: Für nachgeschaltete Anwendungen sofort verwenden oder maximal eine Nacht bei 4 °C lagern oder bei -80 °C einfrieren, wenn weitere Schritte nicht innerhalb von 24 h durchgeführt werden.
- Färben von Elektrofahrzeugen für die Sortierung
- Bereiten Sie eine 5 mM-Lösung von 5(6)-Carboxyfluoresceindiacetat-N-Succinimidylester (CFSE) vor. Frisch zubereitet verwenden oder bei -20 °C lichtgeschützt einfrieren.
- Fahren Sie direkt mit den Schritten 1.1.1-1.1.5 fort (innerhalb von 24 h) oder tauen Sie gelagerte EVs-haltige Überstände bei -80 °C bei 4 °C auf.
- Vor der Färbung die Überstände bei 37 °C erwärmen und CFSE für eine Endkonzentration von 10 μM (500-fache Verdünnung) zugeben. 1 h bei 37 °C im Dunkeln inkubieren.
- Während der Inkubation 2 mL PBS in einen Zentrifugalkonzentrator (regenerierte Zellulosemembran, MWCO 100 kDa) geben, verschließen und 10 Minuten lang bei 4.000 x g in einem schwingenden Schaufelrotor zentrifugieren. Entfernen Sie das ungefilterte PBS vom Boden der Filtervorrichtung und saugen Sie das Filtrat aus dem Auffangrohr ab.
HINWEIS: Das beschriebene Zentrifugalultrafiltrationsprotokoll basiert auf der Verarbeitung von bis zu 15 mL der Probe (maximales Volumen). - Geben Sie bis zu 15 ml Probe in den Zentrifugalkonzentrator und verschließen Sie das Gerät. Zentrifugieren Sie bei 4.000 x g für bis zu 30 min.
HINWEIS: Dies ergibt eine Endprobe von durchschnittlich 500 μl. Abhängig von Faktoren wie der Art der Probe und der Durchflussrate kann die Zentrifugationszeit, die zum Erreichen der gewünschten Konzentration erforderlich ist, jedoch variieren. - Nehmen Sie das Gerät aus der Zentrifuge und leeren Sie das Auffangröhrchen. Geben Sie 14 mL PBS in das Filtergerät. Zentrifugieren Sie bei 4.000 x g für bis zu 30 min.
- Wiederholen Sie Schritt 1.2.6.
- Die konzentrierte CFSE-gefärbte EV-Probe von ca. 500 μl wird aus dem Filtergerät entnommen und 100 μl bei 4 °C im Dunkeln gelagert, während mit der Sortierung der verbleibenden Probe fortgefahren wird.
2. Charakterisierung von ASC-EVs
- Western-Blot-Analyse von EV-Markern
- Pelletieren Sie ASCs (1 x 106) bei 376 x g bei RT für 5 min und suspendieren Sie sie in geeignetem Lysepuffer, der mit Proteaseinhibitoren ergänzt wird. Führen Sie die Proteinquantifizierung mit dem Assay Ihrer Wahl durch.
HINWEIS: Wie in dieser Studie zu sehen ist, werden die besten Quantifizierungsergebnisse mit der Bicinchoninsäure-Assay-Technik erzielt. - Quantifizieren Sie den ASC-konzentrierten Überstand entsprechend 1 x 106 ASCs mit Bradford-Technik oder Pellet-EVs entsprechend 1 x 106 ASCs bei 100.000 x g bei 4 °C für 1 h und suspendieren Sie ihn in geeignetem Lysepuffer, der mit Proteaseinhibitoren ergänzt ist. Führen Sie die Proteinquantifizierung mit dem Assay Ihrer Wahl durch.
HINWEIS: Wie in dieser Studie erfahren, werden die besten Quantifizierungsergebnisse für EV-Pellets mit der Bicinchoninsäure-Assay-Technik erzielt. - Wenn ein konzentrierter Überstand anstelle von gereinigten EVs verwendet wird, werden die Proben in 5 % 2-Mercaptoethanol und 2x Laemmli-Puffer lysiert.
HINWEIS: Wie in dieser Studie erfahren, liefern sowohl konzentrierte Überstände als auch gereinigte EVs vergleichbare Ergebnisse für positive und negative EV-Marker. - Proben (1-10 μg) in ein 10%iges Polyacrylamid-Gel bei 110 V für 90 min laden und separieren.
- Auf eine Nitrozellulosemembran bei 250 mA für 120 min übertragen.
- Färben Sie die Membran mit Ponceau S zur Visualisierung von Proben und Leitertransfer. Ponceau S mit PBS unter leichtem Schütteln entfernen.
- Blockmembranen mit 5 % fettfreier Trockenmilch und 0,1 % Tween in PBS für 60 min.
- Sondieren Sie Membranen mit geeigneten Antikörpern bei Arbeitsverdünnungen, einschließlich positiver (z. B. in dieser Arbeit CD9, CD81, TSG101 und Flotillin) und negativer (z. B. in dieser Arbeit Calnexin) EV-Marker, bei 4 °C über Nacht.
- Mit 0,1 % Tween in PBS waschen und mit geeigneten Peroxidase-konjugierten Sekundärantikörpern 45 Minuten bei RT inkubieren, bevor die Bande mit dem ECL-System der Wahl sichtbar wird. Erfassen Sie Bilder mit einem verfügbaren Bildgebungssystem (Abbildung 2A).
- Pelletieren Sie ASCs (1 x 106) bei 376 x g bei RT für 5 min und suspendieren Sie sie in geeignetem Lysepuffer, der mit Proteaseinhibitoren ergänzt wird. Führen Sie die Proteinquantifizierung mit dem Assay Ihrer Wahl durch.
- Durchflusszytometrische Analyse von EV-Markern: Oberflächenfärbung
- Zentrifugieren Sie vor der Anwendung jeden monoklonalen Antikörper (mAb) bei 15.000-17.000 x g für 30 min bei 4 °C, um Aggregate zu eliminieren, die falsch positive Signale verursachen können. Des Weiteren werden mAbs in separate 0,22 μm Zentrifugalfilterröhrchen bei 1.000 x g bei 4 °C filtriert, bis das gesamte Gemisch den Filter passiert hat und keine Antikörperflüssigkeit mehr auf der Oberfläche des Filters verbleibt. Lagern Sie mAbs bei 4 °C.
- Bereiten Sie eine Verdünnung von CFSE-gefärbten EVs im Verhältnis 1:10 (siehe Schritt 1.2.1-1.2.8) in 0,22 μm-gefiltertem PBS vor.
- Inkubieren Sie 100 μl Proben oder 0,22 μm gefiltertes PBS mit oder ohne EV-spezifische mAbs (anti-CD9/63/81) oder MSC-spezifische mAbs (anti-CD44), die zuvor titriert wurden. Die Inkubation bei 4 °C im Dunkeln für 30 min durchführen.
HINWEIS: Dieses Protokoll, das zur Erkennung typischer EVs und MSC-Linienmarker entwickelt wurde, kann auf alle anderen Zelllinien- oder EVs-Subtyp-spezifischen Oberflächenmarker ausgeweitet werden. Verwenden Sie keine Fluorochrome, die in den Fluorescein-Isothiocyanat-Kanal (FITC) fallen, um eine Überlappung mit der CFSE-Färbung von EVs zu vermeiden. Führen Sie eine einzelne Färbung durch, indem Sie jeden mAb verwenden, der mit einem Fluorochrom, z. B. Allophycocyanin (APC), konjugiert ist. Mehrfarbige Färbung ist möglich, aber es müssen Kontrollen eingebaut werden, um potenzielle Probleme mit der sterischen Behinderung von Antikörpern zu beheben. Es ist notwendig, die einzelnen Antikörper einzeln oder innerhalb eines Mixes zu testen und sicherzustellen, dass das Signal vergleichbar ist. Darüber hinaus müssen FMOs in die Kontrollen einbezogen werden. - Richten Sie die Streukalibrierung wie in Schritt 3.2 beschrieben ein.
- Erstellen Sie ein Punktdiagramm der logarithmischen Skala der Seitenstreuung (SSC) im Vergleich zur FITC logarithmischen Skala und führen Sie ein Rohr mit 0,22 μm-gefilterten PBS oder ungefärbten EVs durch. Stellen Sie den Schwellenwert auf dem FITC-Kanal ein und stellen Sie ihn auf die höchsten Werte ein, die den Großteil der Hintergrundgeräusche ausschließen. Zeichnen Sie eine Region, die die positiven CFSE-Ereignisse identifiziert.
- Erstellen Sie ein APC-Histogrammdiagramm, das auf der CFSE-Region positiver Ereignisse abgegrenzt ist, und führen Sie eine Röhre mit 0,22 μm-gefilterten PBS- oder ungefärbten EVs durch.
HINWEIS: Verwenden Sie für die APC-Verstärkung die mit der QC des Instruments festgelegte Verstärkung, obwohl empfohlen wird, eine bestimmte Verstärkung durchzuführen, um die Leistung des Geräts zu optimieren. - 200 μl 0,22 μm gefiltertes PBS zu gefärbten Proben geben und erfassen. Verwenden Sie eine niedrige Durchflussrate (10 μl pro min) für die Erfassung und Aufzeichnung. Wenn möglich, erfassen Sie mindestens 5.000 Ereignisse im positiven FITC-Gate.
- Verwenden Sie den mAb-gefärbten PBS-Regler, um mögliche unspezifische Signale der mAbs zu erkennen. Lesen Sie alle Probenröhrchen mit der gleichen Durchflussrate ab, um die Konsistenz zwischen den Läufen zu gewährleisten.
- Um eine Kreuzkontamination der Probe zu vermeiden, lassen Sie ein Reinigungsmittel zwischen jedem analysierten Röhrchen 10 s lang laufen, gefolgt von 10 s mit deionisiertem (DI) Wasser (Abbildung 2B).
HINWEIS: Um eine Instabilität des Fluidikgeräts zu vermeiden, nehmen Sie die Probe 10 s lang auf und starten Sie dann die Aufnahme.
- Durchflusszytometrische Analyse von EV-Markern: Intrazelluläre Färbung
HINWEIS: Verwenden Sie für die intrazelluläre Färbung ein spezielles Kit mit Fixierungs- und Permeabilisierungsreagenzien. Anti-Caveolin mAb ist mit Alexa 488 konjugiert. Die IC-Färbung wurde in Abwesenheit von CFSE durchgeführt.- Vor der Verwendung ist der mAb wie zuvor beschrieben (Schritt 2.2.1) zu zentrifugieren.
- Befolgen Sie für die intrazelluläre Färbung die Anweisungen des Herstellers (Abbildung 2C).
- Charakterisierung der EV-Konzentration und -Größe durch Nanopartikel-Tracking-Analyse (NTA)
- Nehmen Sie geeignete Verdünnungen der Proben vor, um 20-120 Ereignisse im Betrachtungsrahmen des NTA-Instrumentendisplays zu erhalten.
- Injizieren Sie die Proben mit einer 1-ml-Insulinspritze in die Probenkammer des NTA-Instruments.
- Stellen Sie die NTA-Software für die Aufzeichnung wie folgt ein: 5 Standardmessungen à 60 s und eine Durchflusspumpe auf 30 eingestellt.
- Stellen Sie die Lichtintensität auf einen geeigneten Wert ein, der es ermöglicht, Partikel klar vom Hintergrund zu unterscheiden, und führen Sie das Capture-Skript auf der NTA-Software aus.
HINWEIS: Die Einstellung des Kamerapegels kann die Partikelerkennung beeinflussen. Ist der Wert zu hoch, entsteht ein milchiger Hintergrund und eine starke Partikellichtstreuung, die die meisten Teilchensignale überdeckt. Wenn der Wert zu niedrig ist, obwohl ein dunklerer Hintergrund erhalten wird, ist es möglich, dass die meisten der weniger hellen Ereignisse übersehen werden. Jede Probe sollte auf eine optimale Einstellung überwacht werden. - Passen Sie den Verdünnungsfaktor an, wenn die Partikel pro Frame kleiner als 20 oder mehr als 80 sind.
- Legen Sie den Schwellenwert auf 4 fest, um die durchschnittliche Modalgröße und Partikelkonzentration des Überstands zu analysieren. Berücksichtigen Sie die anfänglichen Verdünnungsfaktoren.
- Reinigen Sie die Instrumentenkammer nach jeder Probe mit deionisiertem Wasser, um Kreuzkontaminationen zu vermeiden.
- Öffnen Sie die NTA-Analysesoftware und führen Sie die Analyse der erfassten Proben unter Berücksichtigung des Verdünnungsfaktors durch (Abbildung 2D).
- Charakterisierung der EV-Morphologie mittels Transmissionselektronenmikroskopie (TEM)
- Sammeln Sie ASCs-konzentrierten Überstand oder isolieren Sie EVs durch Ultrazentrifugation (100.000 x g bei 4 °C für 1 h), gefolgt von einer Pelletsuspension im gleichen Volumen von PBS.
- 5 μl des konzentrierten Überstands oder der konzentrierten EVs werden 10 Minuten lang auf kohlenstoffbeschichteten Gittern absorbiert. Überschüssige Flüssigkeitstropfen mit Filterpapier abtupfen.
- Führen Sie eine negative Färbung mit 2 % Uranylacetat in wässriger Suspension für 10 Minuten mit einem identischen Volumen des Probentropfens (5 μl) durch. Entfernen Sie das überschüssige Uranylacetat, indem Sie das Gitter mit Filterpapier berühren.
- Trocknen Sie das Gitter bei Raumtemperatur (RT) und untersuchen Sie es mit einem Transmissionselektronenmikroskop bei 120 kV (Abbildung 2E).
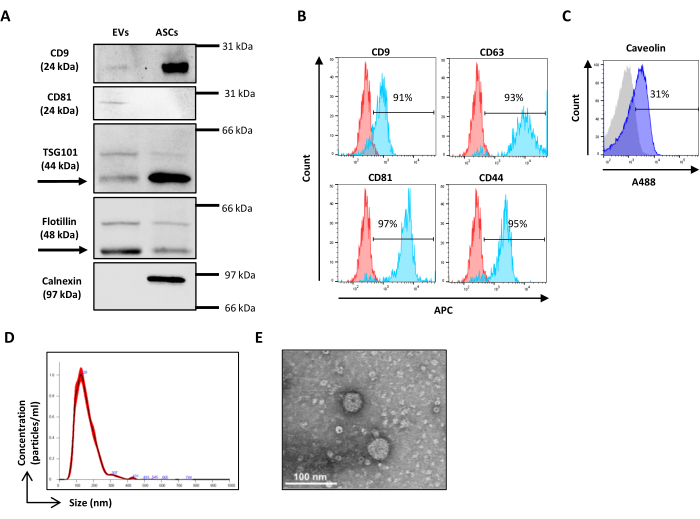
Abbildung 2: Charakterisierung der ASC-EVs. (A) Repräsentativer Western Blot der positiven (CD9, CD81, TSG101 und Flotillin) und negativen (Calnexin) Marker. Entsprechende Molekulargewichte werden angegeben, und ASC-Lysate wurden als Kontrolle verwendet. (B) Durchflusszytometrische Analyse von EVs-Markern. Die Expression der folgenden Marker wurde analysiert: CD9, CD63, CD81 und CD44. Es wurden nur CFSE-positive ASC-EVs auf Markerexpression analysiert. Histogramme stellen ungefärbte (rote Histogramme) und gefärbte (blaue Histogramme) ASC-EVs dar. (C) Durchflusszytometrie, intrazelluläre Analyse des EVs-Markers Caveolin. Histogramme stellen ungefärbte (graues Histogramm) und gefärbte (blaue Histogramme) ASC-EVs dar. (D) Charakterisierung von ASC-EVs durch NTA. Histogramme stellen die Konzentration (Partikel/ml)/Größe (nm) der Probe dar. (e) Visualisierung von ASC-EVs durch TEM. Maßstabsbalken = 100 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Sortierung von ASC-EVs
- Einrichtung des Zellsortierers
HINWEIS: Ein Zellsortierer ist ein Durchflusszytometer, das die Isolierung einer reinen Population aus einer heterogenen Ausgangsprobe ermöglicht. Der Zellsortierer trennt die Zielpartikel, indem er den Strom mit einem Piezoelektrikum bei hoher Frequenz oszilliert, um Tropfen zu erzeugen, die ein Ereignis enthalten. Die Tropfen, die die interessierenden Teilchen wie Zellen oder Vesikel enthielten, werden durch metallische Ablenkplatten geladen und abgelenkt. Die sortierte Fraktion wird für die nachgelagerte Analyse verwendet.- Öffnen Sie die Druckleitung und saugen Sie ab.
HINWEIS: Einige Sortierer verfügen über einen automatischen Start, während andere über einen manuellen Start verfügen. Ein Sortierer mit manuellem Start wird bevorzugt, da er die Optimierung einiger technischer Merkmale wie Frequenz, Druck und die Wahl der Düsenspitze ermöglicht. Insbesondere wird empfohlen, mit einer Frequenz von 66.000 Hz, einem Druck von 35 psi und einer 70-μm-Düse zu arbeiten. - Schalten Sie das Gerät ein, öffnen Sie die Biosicherheitswerkbank und starten Sie die Sortiersoftware.
- Setzen Sie die Fluidik unter Druck und schalten Sie dann das Fluidiksystem ein.
- Aktivieren Sie den Drop Drive, den piezoelektrischen Kristall in der Düse, der vibriert, um Tröpfchen zu bilden.
- Führen Sie ein Debubbling-Verfahren durch, um das Vorhandensein von Blasen im System zu beseitigen, und lassen Sie dann ein 5-ml-Röhrchen mit Reinigungslösung 5 Minuten lang bei hohem Differenzdruck und 5 ml-Röhrchen mit DI-Wasser 5 Minuten lang laufen.
- Führen Sie den manuellen Startvorgang durch.
- Drücken Sie auf der Registerkarte "Lasersteuerung" die Taste "Laser Power ", um den Laser einzuschalten.
HINWEIS: Wiederholen Sie diesen Vorgang für alle Laser: Blau 488 nm, Gelb-Grün 561 nm, Violett 405 nm und Rot 640 nm. - Untersuchen Sie die vertikale Ausrichtung des Streams.
HINWEIS: Bewegen Sie bei Bedarf den Düsenmontagetisch, insbesondere das vordere und hintere Mikrometer, das linke und rechte Mikrometer und den Gimbal, um die vertikale Ausrichtung einzustellen und den vertikalen Strom mit dem Mikrometer nach oben und unten zu überprüfen. Für eine optimale Ausrichtung muss der Strahl auf jedem Laser vertikal sein. Wenn dies das erste Mal ist, dass eine bestimmte Düse verwendet wird, muss eine Laserverzögerung durchgeführt werden. Wählen Sie auf dem Touchscreen auf der Registerkarte Laser und Stream Intercept die richtige Düsengröße aus, drücken Sie die Taste Laserverzögerung und befolgen Sie die Anweisungen zur Laserverzögerung, um den Laserverzögerungsvorgang durchzuführen. Im Allgemeinen wird bei einer Änderung des Manteldrucks und damit der Düsenspitze die Laserverzögerungsbestimmung durchgeführt, bevor die Ausrichtung gefunden wird. Am Ende des Laserverzögerungsverfahrens wird empfohlen, das Verfahren zur Subtraktion des Hintergrundbildes gemäß den Anweisungen auf dem Bildschirm durchzuführen, um das Hintergrundsignal zu minimieren. - Führen Sie die Laserpunktbestimmung durch, indem Sie auf die Registerkarte Laserstrahlabfang und die grüne Pfeiltaste drücken und den Anweisungen auf dem Touchscreen-Monitor folgen.
- Drücken Sie auf der Registerkarte "Lasersteuerung" die Taste "Laser Power ", um den Laser einzuschalten.
- Initialisieren Sie Intellisort. Legen Sie die Drop-Drive-Frequenz und einen Standardamplitudenwert fest, der bewirkt, dass der Stream ein Tröpfchen bildet.
- Nachdem Sie diesen Schritt ausgeführt haben, laden Sie eine gespeicherte Konfiguration. Um eine gespeicherte Einstellungskonfiguration zu laden, wählen Sie Sortiereinstellung laden in der Symbolleiste der Sortiersoftware. Nachdem Sie verschiedene Frequenzen und Amplituden überprüft haben, wählen Sie diejenige aus, die die beste Fallstabilität gewährleistet. Arbeiten Sie mit einer Frequenz von 66.000 Hz und einer Amplitude von etwa 40-45 V.
- Führen Sie das Verfahren zur Feinausrichtung des Lasers durch.
- Laden Sie ein 5-ml-Röhrchen mit QC-Ausrichtungsperlen in den Probenehmer und lassen Sie es laufen. Wählen Sie auf der Registerkarte Feinausrichtung den gewünschten Parameter auf der X- und Y-Achse, um die Sicken gut verdichtet und kollimiert zu visualisieren. Wählen Sie im Datenanzeigebereich zunächst 488 - 513/26-H für den Parameter Y-Achse und 488-FSC1-H für den Parameter X-Achse aus. Um dann alle Laser fein abzustimmen, wählen Sie 405-488/59-H für den Y-Achsen-Parameter und 640-795/70-H für den X-Achsen-Parameter.
HINWEIS: Die gewählten Parameter hängen von der Laserkonfiguration des Sortierers ab. Der Vorschlag für das Instrumentenhandbuch ist die Auswahl der Laser, die die größte räumliche Trennung auf dem Lochblendenstreifen aufweisen. Verwenden Sie dazu einen violetten Laser (405 nm) und einen roten Laser (640 nm). - Sobald die manuelle Ausrichtung vom Sortierspezialisten überprüft wurde, führen Sie die automatische Qualitätskontrolle durch. Wählen Sie den Durchmesser der QC-Kügelchen: 3 μm Durchmesser.
HINWEIS: Am Ende dieses Verfahrens kann das Ergebnis sein, dass die Qualitätskontrolle nicht bestanden wurde oder die Qualitätskontrolle nicht bestanden wurde. Falls die Qualitätskontrolle ausfällt, ist es notwendig, die Ausrichtung manuell zu optimieren. - Speichern Sie die QC-Erfassung im Alignment-Protokoll der Software.
- Laden Sie ein 5-ml-Röhrchen mit QC-Ausrichtungsperlen in den Probenehmer und lassen Sie es laufen. Wählen Sie auf der Registerkarte Feinausrichtung den gewünschten Parameter auf der X- und Y-Achse, um die Sicken gut verdichtet und kollimiert zu visualisieren. Wählen Sie im Datenanzeigebereich zunächst 488 - 513/26-H für den Parameter Y-Achse und 488-FSC1-H für den Parameter X-Achse aus. Um dann alle Laser fein abzustimmen, wählen Sie 405-488/59-H für den Y-Achsen-Parameter und 640-795/70-H für den X-Achsen-Parameter.
- Abschließen der Intellisort-Prozedur
- Platzieren Sie die Umlenkbleche in der richtigen Position im Sortierer und schalten Sie die Spannung ein (empfohlen wird eine Ladung von 3.000 V auf den Umlenkblechen).
- Wählen Sie die Sortierausgabe für 6 Rohrhalter, aktivieren Sie die Ströme, indem Sie sie auf der Stream-Anzeige auswählen, und führen Sie den Teststream durch.
HINWEIS: Wenn die Ströme nicht klar getrennt und definiert sind, passen Sie die Ladungsphase, die Definition an oder ändern Sie andere Parameter, um eine klar definierte und nicht oszillierende Strömung zu erhalten. - Aktivieren Sie die Schaltfläche Intellisort Automatic Drop Delay Determination (Automatische Bestimmung der Drop-Verzögerung ). In diesem Schritt können Sie die richtige Drop-Verzögerung einstellen. Überprüfen Sie nach diesem Schritt die Streams erneut.
- Aktivieren Sie den Intellisort-Wartungsmodus. Überprüfen Sie die Drop-Verzögerung manuell.
- Laden Sie das manuelle Drop Delay-Protokoll in die Sortiersoftware. Erwerben Sie fluoreszierende Kontrollperlen.
- Setzen Sie den richtigen Folienhalter ein. Wählen Sie in der Sortiersoftware Sortier- > Drop-Verzögerungs-Assistent aus. Wählen Sie Sortierlogik aus.
- Überprüfen Sie mit einem Fluoreszenzmikroskop, ob 97 % der fluoreszierenden Kügelchen auf der fünften Pfütze vorhanden sind.
- Öffnen Sie die Druckleitung und saugen Sie ab.
- Geräte-Setup: Scatter-Kalibrierung
HINWEIS: Die Streukalibrierung wird mit Hilfe von Verity Shells-Kügelchen (im Folgenden als hohle Organokieselsäurekügelchen bezeichnet) durchgeführt. Diese Organo-Siliziumdioxid-Kügelchen zeichnen sich durch eine Brechungsindexverteilung und Lichtstreueigenschaften aus, die näher an EVs liegen als Polystyrolkügelchen, wodurch der Aufbau der SSC- und FSC-Spannung ermöglicht wird, um besser zwischen EV-ähnlichen Ereignissen und dem elektronischen Rauschen zu unterscheiden. Die Organokieselsäurekügelchen bestehen aus zwei Fläschchen mit Bead-Mischungen, VER01A (189 nm Kügelchen) und VER01B (374 nm Kügelchen). Jedes Fläschchen enthält eine Mischung aus hohlen Organo-Kieselsäurekügelchen und 380 nm grünen fluoreszierenden Kügelchen, die als Referenzkügelchen bezeichnet werden. Die Konzentration der hohlen Organokieselsäurekügelchen beträgt 1 x 108 Kügelchen/ml.- Bereiten Sie eine Probe von VER01A- und VER01B-Kügelchen vor, indem Sie jeweils einen Tropfen (50 μl) in 1 mL 0,22 μm-gefiltertem PBS mit einer Endkonzentration von 5.000 Kügelchen/μl verdünnen.
HINWEIS: Wirbeln Sie die Perlen vor der Verwendung 5 s lang ein. - Erstellen Sie ein Punktdiagramm der SSC-Log-Skala im Vergleich zur FITC-Log-Skala.
- Erfassen Sie VER01B-Kügelchen, und stellen Sie die Spannung so ein, dass die Referenzperlen im oberen rechten Teil des SSC/FITC-Punktdiagramms gut unterschieden werden (Abbildung 3A).
- Legen Sie einen Bereich um diese Perlenpopulation fest.
- Erstellen Sie ein Punktdiagramm der SSC-Log-Skala im Vergleich zur FSC-Log-Skala. Identifizieren Sie die Referenzkügelchen und die VER01B-Beads in der Population. Legen Sie einen Bereich um die Population der VER01B-Perlen fest (Abbildung 3B).
- Erfassen Sie VER01A-Kügelchen, und erstellen Sie einen Bereich um diese Kügelchenpopulation (Abbildung 3C).
HINWEIS: Diese Population von Perlen überlappt sich teilweise mit dem Hintergrundrauschen des Instruments. - Erfassen Sie 0,22 μm gefiltertes PBS und passen Sie den Schwellenwert an, um das Hintergrundrauschen des Instruments zu verringern, ohne die Sichtbarkeit der kleinsten Kügelchen zu verlieren.
HINWEIS: Die Streukalibrierung kann mit FSC Megamix-Kügelchen (im Folgenden als FSC-Polystyrolkügelchen bezeichnet) durchgeführt werden, einer Mischung aus fluoreszierenden Kügelchen mit den folgenden Durchmessern: 100 nm, 300 nm, 500 nm und 900 nm. Die Verwendung der hohlen Organokieselsäurekügelchen muss FSC-Polystyrolkügelchen vorzuziehen sein, da sie eine Brechungsindexverteilung und Lichtstreueigenschaften aufweisen, die denen von Elektrofahrzeugen ähneln. Die Population der 300 nm FSC-Polystyrolkügelchen überlappt sich mit der Referenz der hohlen Organokieselsäurekügelchen. Daher wählt ein Gate auf Basis von hohlen Organo-Kieselsäurekügelchen auf dem Durchflusszytometer ähnlich große EVs aus.
- Bereiten Sie eine Probe von VER01A- und VER01B-Kügelchen vor, indem Sie jeweils einen Tropfen (50 μl) in 1 mL 0,22 μm-gefiltertem PBS mit einer Endkonzentration von 5.000 Kügelchen/μl verdünnen.
- Probenaufnahme
- Erstellen Sie ein Punktdiagramm der SSC-Log-Skala im Vergleich zur FITC-Log-Skala, erfassen Sie 0,22 μm-gefilterte PBS und definieren Sie das Referenz-Hintergrundrauschen (Abbildung 4A).
HINWEIS: Stellen Sie die Flussrate des Zytometers auf Niedrig und steuern Sie die Flussrate. Die Anzahl der Events/s darf nicht mehr als 2.000 Events/s betragen. Durchfluss Niedrig bedeutet bei geringem Differenzdruck. - Entnehmen Sie die Kontrollprobe (ungefärbte Elektrofahrzeuge) und überprüfen Sie die Durchflussrate. Stellen Sie sicher, dass die Anzahl der Ereignisse/s etwa 5.000 Ereignisse/s beträgt.
HINWEIS: Wenn die Anzahl der Ereignisse die empfohlene Anzahl überschreitet, verdünnen Sie die Probe. - Erfassen Sie PBS, gefärbt und behandelt wie die CFSE-Probe, und überprüfen Sie, ob keine positiven Ereignisse sichtbar sind (Abbildung 4B).
- Entnehmen Sie die mit CFSE gefärbte Probe. Verdünnen Sie es basierend auf der Durchflussrate. Zeichnen Sie eine Region, die die positiven CFSE-Ereignisse identifiziert, und eine Region, die die negativen CFSE-Ereignisse identifiziert. Verwenden Sie eine ungefärbte Probe als Kontrolle (Abbildung 5A, B). Dies sind die Sortierbereiche.
- Erstellen Sie ein Punktdiagramm der SSC-Log-Skala im Vergleich zur FITC-Log-Skala, erfassen Sie 0,22 μm-gefilterte PBS und definieren Sie das Referenz-Hintergrundrauschen (Abbildung 4A).
- Sortierung von Proben
- Öffnen Sie in der Sortiersoftware ein neues Protokoll (wählen Sie in der Symbolleiste Datei > Protokoll > Neu aus).
- Klicken Sie in der Sortiersoftware auf Datenerfassungseinstellung. Wählen Sie im Fenster Akquisitionsparameter die gewünschten Kanäle aus und deaktivieren Sie die anderen. Für dieses spezielle Panel sind die signalfähigen: 488-FSC1, 488-FSC2, 488-SSC und 488-513/26 (für CFSE-Signal ).
- Auf dem Probenehmer ist ein 5-ml-Röhrchen mit der Probe zu finden. Klicken Sie auf dem Touchscreen auf die Schaltfläche Laden und wählen Sie in der Symbolleiste der Sortiersoftware die Option Erfassung > Start (oder drücken Sie die Taste F2 ). Ein Fenster mit Beispieleigenschaften wird angezeigt. Geben Sie den Namen des Beispiels ein, und klicken Sie auf OK.
- Erstellen Sie das Panel mit dem Diagramm von Interesse, und erstellen Sie die Gating-Strategie.
- Wählen Sie in der Symbolleiste der Sortiersoftware Histogramm > Histogramm erstellen aus.
- Erstellen Sie im Arbeitsbereich drei Punktdiagramme, das erste mit den Parametern 488-FSC1 Height-Log auf der x-Achse und 488-SSC Height-Log auf der x-Achse, das zweite mit den Parametern 488-FSC2 Height-Log auf der x-Achse und 488-SSC Height-Log auf der x-Achse, das dritte mit den Parametern 488-513/26 CFSE Height-Log auf der x-Achse und 488-SSC Height-Log.
- Identifizieren Sie auf der Registerkarte Sortieren die Region(en), die sortiert werden sollen. Klicken Sie in der Sortiersoftware auf Sortiereinstellung. Wählen Sie im Fenster Sortierlogik und Statistik die Region aus, die im Logik-Generator sortiert werden soll.
HINWEIS: Um eine Sortierentscheidung zu treffen, müssen verschiedene Einstellungen berücksichtigt werden: Sortierlogik, Sortiermodus und Drop-Envelope. Die Sortierlogik bezieht sich auf die Gating-Strategie. Die Events, die sich außerhalb des ausgewählten Gates befinden, werden nicht sortiert. Der Sortiermodus bezieht sich auf die endgültige Ausgabe. Arbeiten Sie im Reinigungsmodus, auf diese Weise enthalten alle sortierten Tropfen nur die gewünschten positiven Ereignisse. Die Drop Envelope definiert, wie viele Tropfen aufgeladen und sortiert werden, bezogen auf die Position im Tropfen des positiven Ereignisses: Wählen Sie 1-2 Tropfen, um sicherzustellen, dass alle positiven Ereignisse sortiert sind. - Wenn die Durchflussmenge stabil ist, stoppen Sie die Erfassung (wählen Sie in der Symbolleiste der Sortiersoftware Erfassung > Stopp oder drücken Sie erneut die Taste F2 ). Starten Sie die Sortierung (wählen Sie in der Symbolleiste der Sortiersoftware Sortieren > Starten oder drücken Sie die Taste F4 ).
HINWEIS: Bevor Sie mit der Sortierung beginnen, überprüfen Sie den Teststream. Vergewissern Sie sich, dass die sortierte Fraktion korrekt in das Auffangröhrchen fällt. - Speichern Sie die Sortiereinstellung. Wählen Sie in der Symbolleiste der Sortiersoftware Sortieren > Sortiereinstellungen speichern aus.
- Reinheitsprüfung der sortierten Grundgesamtheit
- Waschen Sie das Instrument, indem Sie ein 5-ml-Röhrchen Reinigungslösung für 10 Minuten bei hohem Differenzdruck und ein 5-ml-Röhrchen mit DI-Wasser für 10 Minuten aufnehmen.
- Erfassen Sie 0,22 μm gefiltertes PBS und prüfen Sie, ob keine CFSE-positiven Ereignisse vorhanden sind.
- 5 μl der sortierten Probe werden in 100 μl 0,22 μm-filtriertem PBS verdünnt.
- Erfassen und Aufzeichnen aller Probenvolumina (Abbildung 5C, D).
HINWEIS: Es wird über eine erneute Analyse sowohl der CFSE-positiven als auch der negativ sortierten EVs berichtet.
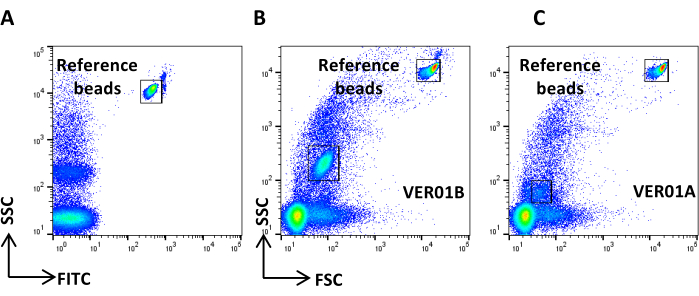
Abbildung 3: Einstellung der physikalischen Parameter mit hohlen Organo-Kieselsäurekügelchen. (A) SSC/FITC-Punktdiagramm: Referenzgrüne, fluoreszierende Kügelchen wurden verwendet, um den SSC-Parameter einzustellen. SSC/FSC-Punktdiagramm von (B) VER01B- und (C) VER01A-Kügelchen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
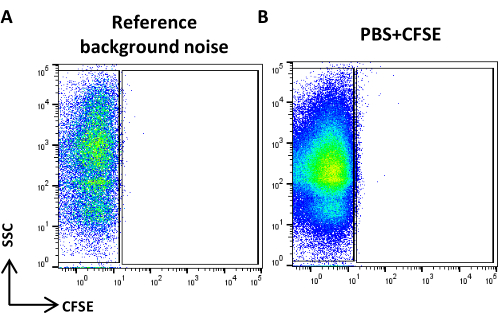
Abbildung 4: Referenz-Hintergrundrauschen. (A) SSC/CFSE-Punktdiagramm der PBS-Stichprobe. (B) SSC/CFSE-Punktdiagramm der PBS + CFSE-Stichprobe. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
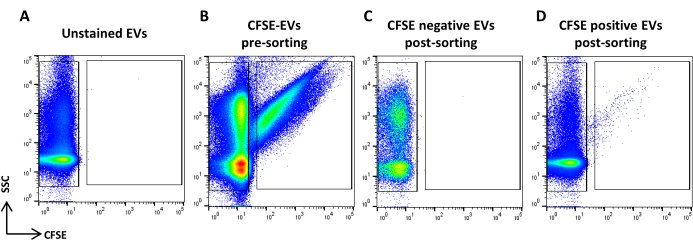
Abbildung 5: Sortierung von CFSE-gefärbten ASC-EVs. (A) SSC/CFSE-Punktdiagramm von ungefärbten EVs, (B) CFSE-gefärbten EVs, (C) CFSE-negativen EVs nach der Sortierung und (D) CFSE-positiven EVs nach der Sortierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Analysen nach der Sortierung
HINWEIS: Aufgrund der begrenzten Materialmenge nach der Sortierung kann es sein, dass nicht alle Analysen durchgeführt werden können. Mit dem erhaltenen Betrag wird folgendes ausgeführt.
- Charakterisierung von EV durch Durchflusszytometrie: Oberflächenfärbung (siehe Schritt 2.2)
HINWEIS: Wie zuvor für die Vorsortierung von Proben beschrieben, mit Ausnahme der Probenvorbereitung. Proben nach der Sortierung können unverdünnt verwendet werden.- Es werden unverdünnte sortierte Proben verwendet und wie in Schritt 2.2.3 beschrieben. (Abbildung 6A)
- Charakterisierung der EV-Konzentration und -Größe durch NTA (siehe Schritt 2.4)
HINWEIS: Wie zuvor für die Vorsortierung von Proben beschrieben, mit Ausnahme der Probenvorbereitung. Proben nach der Sortierung können unverdünnt verwendet werden.- Es werden unverdünnte sortierte Proben verwendet und wie in Schritt 2.4.2 beschrieben. (Abbildung 6B)
- Charakterisierung der EV-Morphologie mittels TEM (siehe Schritt 2.5)
HINWEIS: Wie zuvor für die Vorsortierung von Proben beschrieben, mit Ausnahme der Probenvorbereitung.- Geben Sie 2 mL PBS in einen Zentrifugalkonzentrator (regenerierte Zellulosemembran, MWCO 100 kDa), eine Kappe und zentrifugieren Sie sie 10 Minuten lang bei 4.000 x g in einem schwingenden Schaufelrotor. Entfernen Sie ungefiltertes PBS von der Unterseite des Filtergeräts.
- Filtrat aus dem Auffangröhrchen aspirieren. Das beschriebene Zentrifugalultrafiltrationsprotokoll basiert auf der Verarbeitung von bis zu 15 mL Proben (maximales Volumen).
- Geben Sie bis zu 15 ml Probe in den AU-15-Filter und verschließen Sie das Gerät. Zentrifugieren Sie bei 4.000 x g für bis zu 30 min. Konzentrieren Sie bis zu dem minimalen Volumen, das das Gerät zulässt, ca. 100-150 μl.
- Die konzentrierte sortierte EV-Probe wird aus der Filtervorrichtung entnommen und wie zuvor für die Vorsortierung von Proben TEM beschrieben (Schritt 2.5.2) verfahren ( Abbildung 6C).
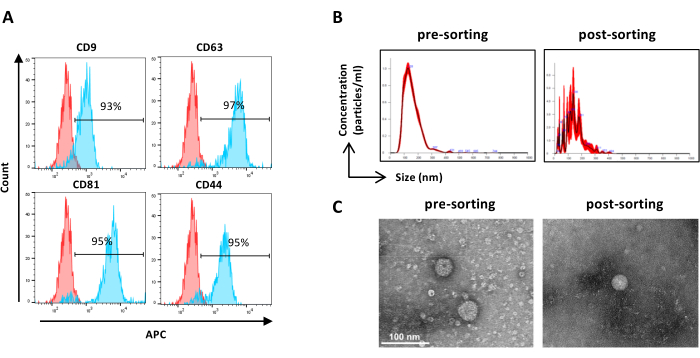
Abbildung 6: Charakterisierung von sortierten ASC-EVs. Durchflusszytometrische Analyse von EV-Markern. Die Expression der folgenden Marker wurde analysiert: CD9, CD63, CD81 und CD44. Es wurden nur CFSE-positive ASC-EVs auf Markerexpression analysiert. (A) Histogramme stellen ungefärbte (rote Histogramme) und gefärbte (blaue Histogramme) ASC-EVs dar. (B) Charakterisierung von ASC-EVs durch NTA. Histogramme stellen die Konzentration (Partikel/ml)/Größe (nm) der Proben vor der Sortierung (links) und nach der Sortierung (rechts) dar. (C) Visualisierung von ASC-EVs durch TEM der Probe vor der Sortierung (links) und nach der Sortierung (rechts). Maßstabsbalken = 100 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Die FSC-Polystyrolkügelchen wurden sortiert, um die Einrichtung des Instruments und die Sortierbedingungen zu validieren. FSC-Polystyrolkügelchen sind eine Mischung aus fluoreszierenden Kügelchen von 100 nm, 300 nm, 500 nm und 900 nm und sind auf dem FITC-Kanal sichtbar. Abbildung 7A zeigt die SSC-Log-Skala im Vergleich zum FITC-Log-Skala-Punktdiagramm mit den vier Populationen von Perlen vor der Sortierung. Die fluoreszierenden Populationen von 100 nm, 3...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die Analyse und Sortierung von Elektrofahrzeugen ist aufgrund ihrer geringen Größe und der Tatsache, dass sie nahe der Nachweisgrenze der meisten Durchflusszytometer liegen, eine Herausforderung. Unser Ziel war es, ein Protokoll für die Isolierung von Elektrofahrzeugen zu entwickeln, die von mit CFSE markierten AMSCs abgeleitet sind. CFSE wurde aufgrund der berichteten hohen Markierungseffizienz von EVs (≥90%) als Färbemethode ausgewählt, ohne dass unerwünschte Partikel wie Prote...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Wir danken Emanuele Canonico für die technische Unterstützung. Ein Teil dieser Arbeit wurde im ALEMBIC durchgeführt, einem fortschrittlichen Mikroskopielabor, das vom IRCCS Ospedale San Raffaele und der Università Vita-Salute San Raffaele eingerichtet wurde. Die Arbeit von Enrico Ragni und Laura de Girolamo wurde vom italienischen Gesundheitsministerium "Ricerca Corrente" unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 5(6)-Carboxyfluorescein diacetate N-succinimidyl ester | Merck | 150347-59-4 | |
| Adipose Mesenchymal Stromal Cells | Wepredic, Parc d'affaires, 35760 Saint-Grégoire, France | Cells used in this study | |
| Alexa 488 anti-Caveolin | R&D Systems | IC5736G | Flow cytometry antibody |
| APC anti-human CD44 | BioLegend | 338805 | Flow cytometry antibody |
| APC anti-human CD63 | BioLegend | 353007 | Flow cytometry antibody |
| APC anti-human CD81 (TAPA-1) | BioLegend | 349509 | Flow cytometry antibody |
| APC anti-human CD9 | BioLegend | 312107 | Flow cytometry antibody |
| BC CytoFLEX S | Beckman Coulter | BC CytoFLEX S equipped with 3 lasers, Blue, Red and Violet | |
| Flow-Check Pro Fluorospheres | Beckman Coulter | A63493 | Fluorescent control beads for MoFLO Astrios EQ |
| FlowJo software (version 10.8.1) | BD | version 10.8.1 | Analysis software |
| IntraSure kit | BD Biosciences | 641776 | Fixation and permeabilization for intracellular staining |
| Megamix-Plus FSC | BioCytex | 7802 | FSC polystyrene beads |
| MoFLO Astrios EQ | Beckman Coulter | MoFLO Astrios EQ equipped with 4 lasers, Blue, Yellow - Green, Violet and Red | |
| Mouse anti-FLOT1 antibody | BD Transduction Laboratories | 610820 | Western Blot antibody |
| NanoSight NS300 | Malvern | NS300 | |
| Rabbit anti-Calnexin antibody | Origene | TA336279 | Western Blot antibody |
| Rabbit anti-CD9 and CD81 antibody (ExoAb antibody kit) | System Biosciences | EXOAB-KIT-1 | Western Blot antibodies |
| Rabbit anti-TSG101 antibody | Merck | HPA006161 | Western Blot antibody |
| Triton X-100 | Merck | 9036-19-5 | |
| Ultra Rainbow Fluorescent Particles | Spherotech | URFP-30-2 | |
| Ultracel 100 kDa MWCO | Merck | UFC910024 | |
| VER01 - Verity Shells | Exometry | Organo silica beads for scatter calibration |
Referenzen
- Welsh, J. A., et al. Minimal information for studies of extracellular vesicles (misev2023): From basic to advanced approaches. J Extracell Vesicles. 13 (2), e12404(2024).
- Xu, R., Greening, D. W., Zhu, H. J., Takahashi, N., Simpson, R. J. Extracellular vesicle isolation and characterization: Toward clinical application. J Clin Invest. 126 (4), 1152-1162 (2016).
- Dixson, A. C., Dawson, T. R., Di Vizio, D., Weaver, A. M. Context-specific regulation of extracellular vesicle biogenesis and cargo selection. Nat Rev Mol Cell Biol. 24 (7), 454-476 (2023).
- Witwer, K. W., Wolfram, J. Extracellular vesicles versus synthetic nanoparticles for drug delivery. Nat Rev Mater. 6 (2), 103-106 (2021).
- Cheng, L., Hill, A. F. Therapeutically harnessing extracellular vesicles. Nat Rev Drug Discov. 21 (5), 379-399 (2022).
- Du, S., et al. Extracellular vesicles: A rising star for therapeutics and drug delivery. J Nanobiotechnology. 21 (1), 231(2023).
- Keshtkar, S., Azarpira, N., Ghahremani, M. H. Mesenchymal stem cell-derived extracellular vesicles: Novel frontiers in regenerative medicine. Stem Cell Res Ther. 9 (1), 63(2018).
- Orbay, H., Tobita, M., Mizuno, H. Mesenchymal stem cells isolated from adipose and other tissues: Basic biological properties and clinical applications. Stem Cells Int. 2012, 461718(2012).
- Caplan, A. I., Dennis, J. E. Mesenchymal stem cells as trophic mediators. J Cell Biochem. 98 (5), 1076-1084 (2006).
- Rani, S., Ryan, A. E., Griffin, M. D., Ritter, T. Mesenchymal stem cell-derived extracellular vesicles: Toward cell-free therapeutic applications. Mol Ther. 23 (5), 812-823 (2015).
- Galipeau, J., Sensébé, L. Mesenchymal stromal cells: Clinical challenges and therapeutic opportunities. Cell Stem Cell. 22 (6), 824-833 (2018).
- Jia, Y., et al. Small extracellular vesicles isolation and separation: Current techniques, pending questions and clinical applications. Theranostics. 12 (15), 6548-6575 (2022).
- Ohnuma, K., Yomo, T., Asashima, M., Kaneko, K. Sorting of cells of the same size, shape, and cell cycle stage for a single cell level assay without staining. BMC Cell Biol. 7, 25(2006).
- Morales-Kastresana, A., et al. High-fidelity detection and sorting of nanoscale vesicles in viral disease and cancer. J Extracell Vesicles. 8 (1), 1597603(2019).
- Mortati, L., et al. In vitro study of extracellular vesicles migration in cartilage-derived osteoarthritis samples using real-time quantitative multimodal nonlinear optics imaging. Pharmaceutics. 12 (8), 734(2020).
- Andreu, Z., Yanez-Mo, M. Tetraspanins in extracellular vesicle formation and function. Front Immunol. 5, 442(2014).
- Mildmay-White, A., Khan, W. Cell surface markers on adipose-derived stem cells: A systematic review. Curr Stem Cell Res Ther. 12 (6), 484-492 (2017).
- Welsh, J. A., Tang, V. A., Van Der Pol, E., Gorgens, A. MIFlowCyt-EV: The next chapter in the reporting and reliability of single extracellular vesicle flow cytometry experiments. Cytometry A. 99 (4), 365-368 (2021).
- Maia, J., et al. Employing flow cytometry to extracellular vesicles sample microvolume analysis and quality control. Front Cell Dev Biol. 8, 593750(2020).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten