Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mise en place d’un tri réussi pour l’isolement des vésicules extracellulaires
Dans cet article
Résumé
Ce protocole fournit une description détaillée du tri des vésicules extracellulaires (VE) libérées par les cellules stromales mésenchymateuses. Il se concentre en particulier sur le réglage de l’instrument et l’optimisation des conditions de tri. L’objectif est de trier les vésicules extracellulaires tout en préservant leurs caractéristiques.
Résumé
Les vésicules extracellulaires (VE) libérées par les cellules stromales mésenchymateuses (CSM) contiennent un ensemble de microARN ayant des rôles régénérateurs et anti-inflammatoires. Par conséquent, les CSM-EV purifiées sont envisagées comme une option thérapeutique de nouvelle génération pour un large éventail de maladies. Dans ce protocole, nous rapportons la stratégie pour trier avec succès les VE du surnageant des CSM dérivées du tissu adipeux (CSA), souvent utilisées dans les applications de médecine régénérative orthopédique.
Tout d’abord, nous avons décrit la préparation de l’échantillon, en nous concentrant sur les étapes d’isolement et de marquage des VE avec de l’ester de carboxyfluorescéine succinimidylique (CFSE) pour la détection de la fluorescence ; Par la suite, nous avons détaillé le processus de tri, qui constitue la partie principale du protocole.
En plus des règles définies par les directives MISEV 2023 et MIFlowCyt EV, nous avons appliqué des conditions expérimentales spécifiques concernant la taille de la buse, la fréquence et la pression de la gaine. Les paramètres morphologiques sont établis à l’aide de billes de diamètres choisis pour couvrir la plage théorique de taille des VE. Après le tri des ASC-EVs, nous avons effectué un contrôle de pureté de la fraction triée en la réanalysant avec le trieur et en vérifiant la distribution de la taille des EV avec la technique d’analyse de suivi des nanoparticules.
En raison de l’importance croissante des VE, il devient crucial de disposer d’une population pure à étudier et à caractériser. Ici, nous démontrons une stratégie gagnante pour mettre en place le tri afin d’atteindre cet objectif.
Introduction
Les vésicules extracellulaires (VE) sont un groupe hétérogène de vésicules structurées par une membrane libérées par presque toutes les cellules, délimitées par une bicouche lipidique, incapables de se répliquer par elles-mêmes1. Ils peuvent être trouvés dans plusieurs biofluides tels que le plasma sanguin, le sérum, la salive, le lait maternel, l’urine, le liquide de lavage bronchique, le liquide amniotique, le liquide céphalo-rachidien et l’ascite maligne2. L’une des principales fonctions des VE est de transporter diverses molécules, notamment des acides nucléiques, des protéines, des lipides et des glucides, entre une cellule donneuse et une cellule réceptrice. Cela peut se produire par divers mécanismes, tels que la fusion directe de la membrane, l’interaction récepteur-ligand, l’endocytose et la phagocytose 3,4. Pour cette raison, il a été démontré qu’ils jouent un rôle important dans de nombreux processus physiologiques et pathologiques, et ils sont très prometteurs en tant que nouveaux biomarqueurs de la maladie, en tant que véhicules d’administration de médicaments et en tant qu’agents thérapeutiques 5,6.
Les cellules stromales mésenchymateuses (CSM) sont des cellules multipotentes qui peuvent être isolées dans de nombreux tissus, notamment le tissu adipeux, la pulpe dentaire, le sang de cordon ombilical, le placenta, le liquide amniotique, la gelée de Wharton et même le cerveau, les poumons, le thymus, le pancréas, la rate, le foie et les reins. Ces dernières années, ils ont suscité un intérêt considérable pour la médecine régénérative7. Les cellules souches mésenchymateuses (ASC) dérivées du tissu adipeux peuvent être prélevées dans le tissu adipeux par une procédure moins invasive par rapport à d’autres sources comme la moelle osseuse, ce qui réduit les risques de complications graves et évite les problèmes éthiques8.
De plus, le tissu adipeux contient une concentration significativement plus élevée de CSM que la moelle osseuse (1 % contre >0,01 %) et d’autres sources telles que le derme, la pulpe dentaire, le cordon ombilical et le placenta. Les CSM sont cruciales dans la régénération des tissus et des cellules lésés en raison de leur capacité de différenciation et de leur sécrétion d’un large répertoire de facteurs de croissance, de chimiokines et de cytokines ; Ces avantages thérapeutiques sont attribuables à leur capacité de différenciation, mais aussi au fait qu’ils sécrètent un large répertoire de facteurs de croissance, de chimiokines et de cytokines. Un exemple frappant est donné par le potentiel thérapeutique des CSM pour les affections orthopédiques, le terme « maladies musculo-squelettiques » ayant le plus grand nombre d’études cliniques enregistrées sous clinicaltrials.gov (consultéle 13 mai 2024).
De plus, les CSM peuvent également sécréter des VE qui participent à la régénération des tissus en transférant des informations aux cellules ou aux tissus endommagés et exercent une activité biologique similaire à celle des cellules mères 9,10. Pour cette raison, les CSM-EV peuvent être un substitut précieux à la thérapie cellulaire pour parvenir à une approche acellulaire11, avec deux études cliniques portant sur les CSM-EV pour des affections orthopédiques (NCT05261360 et NCT04998058). Cependant, plusieurs défis subsistent pour les applications cliniques des VE. Par exemple, les techniques d’isolation des VE, qui suscitent des inquiétudes, ne garantissent pas la pureté ou l’intégrité des vésicules. De plus, certaines techniques d’isolement sont complexes, longues et peu répétitives, ce qui les rend inadaptées à un usage clinique12.
Le tri cellulaire, quant à lui, est une méthode couramment utilisée qui permet d’isoler des cellules uniques à partir de suspensions cellulaires hétérogènes à l’aide de marqueurs fluorescents spécifiques13. Il peut être utilisé pour de nombreuses applications et adapté à différents types d’échantillons. Cependant, bien que le tri cellulaire soit une technologie bien établie et largement utilisée, le tri des VE reste très difficile car la plupart des VE sont en dessous du seuil de détection minimum, même pour les cytomètres en flux les plus sensibles. Certaines caractéristiques rendent un trieur plus adapté à cet effet. Tout d’abord, à l’aide d’un système Jet-in-air dans lequel le flux en suspension des particules est interrogé par des lasers dans l’air, plutôt qu’à l’intérieur d’une cellule d’écoulement ; Ce système préserve l’échantillon en diminuant les contraintes auxquelles il est soumis. Un deuxième point important est la présence d’une barre d'« obscurcissement » entre le flux et la lentille collectrice qui diminue le bruit optique de fond de l’instrument. Bien qu’il soit faible, le bruit de fond n’est pas complètement éliminé et constitue une référence qui offre une fenêtre partielle sur les événements qui tombent en dessous du seuil : il est très important pour l’analyse des événements proches de la « limite de détection » de l’instrument14. Enfin, le trieur est doté d’une diffusion directe à double chemin (FSC) avec deux masques différents qui permettent une meilleure discrimination entre les petites et les grosses particules de l’échantillon.
Sur cette base, nous avons développé un protocole visant à séparer les MSC-EV marquées à l’ester succinimidylique de carboxyfluorescéine (CFSE) à l’aide d’un trieur de cellules à haute sensibilité. Pour minimiser la manipulation des VE et préserver leur intégrité et leur quantité, nous avons évité les étapes d’ultracentrifugation lors de la préparation de l’échantillon. De plus, les conditions de tri ont été ajustées pour minimiser les contraintes sur les vésicules, y compris l’optimisation supplémentaire de notre instrument en réduisant la pression de tri associée à la taille de la buse (buse de 70 μm pour une pression de 35 psi).
Access restricted. Please log in or start a trial to view this content.
Protocole
Le protocole se compose ici de quatre parties : (1) la préparation des échantillons, (2) la caractérisation des ASC-EV, (3) le tri des ASC-EV et (4) l’analyse post-tri. Un schéma représentant le flux de travail est illustré à la figure 1.
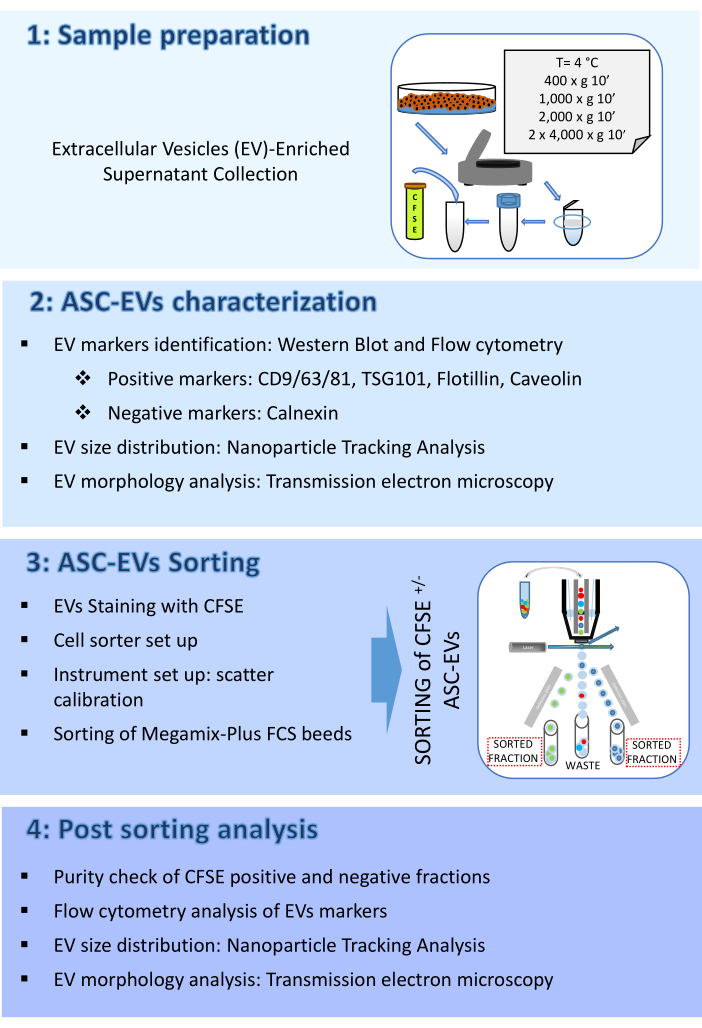
Figure 1 : organigramme du protocole. L’organigramme montre les étapes impliquées dans le protocole. (1) préparation des échantillons, (2) caractérisation des vésicules avant le tri, (3) tri et (4) analyse des vésicules après le tri. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Préparation de l’échantillon
- Collection de surnageants enrichis en vésicules extracellulaires (VE)
- Décongeler ou prélever des CSM adipeux (CSA) qui ont été cultivées avant la collecte des VE (généralement le passage 1 à 5) et semer pour chaque isolat d’ASC une quantité identique de cellules (p. ex., 1 million de cellules par 175 cm2 de surface de flacon donne une confluence d’environ 60 % à 70 %).
- Cultivez les ASC dans le milieu de culture approprié (DMEM-F12 pour ce protocole) complété par du sérum fœtal bovin (FBS) sans EV ou du lysat de plaquettes humaines (hPL), conformément au protocole demandé, pendant 48 à 72 h.
- Pour obtenir un FBS ou un hPL sans VE, ultracentrifuger à 120 000 x g pendant la nuit à 4 °C et utiliser le surnageant.
REMARQUE : À partir de ce moment, la force centrifuge indiquée est toujours considérée comme une moyenne pour l’instrument, le rotor et les tubes utilisés. Gardez à l’esprit que différents rotors ou tubes peuvent avoir une force g par régime et un facteur k variables. Pour un moyen facile de comparer les rotors et d’ajuster la force g et la vitesse, reportez-vous à l’un des tableaux de conversion disponibles https://www.beckman.it/centrifuges/rotors/calculator.
- Pour obtenir un FBS ou un hPL sans VE, ultracentrifuger à 120 000 x g pendant la nuit à 4 °C et utiliser le surnageant.
- À 90 % de confluence cellulaire, détachez et comptez les ASC et suspendez-les dans un volume approprié de milieu sans sérum, idéalement 1 mL par 1 x 106 ASC. Semences ASC en suspension dans des plaques de 24 puits avec 1 mL par puits. Sans sérum, les cellules resteront en suspension et formeront un sphéroïde. Après 96 h, récupérez le surnageant.
- Éliminer les cellules flottantes et les débris par centrifugation en série à 4 °C : 400 x g pendant 10 min, 1 000 x g pendant 10 min, 2 000 x g pendant 10 min et deux fois 4 000 x g pendant 10 min.
- Filtrez le surnageant à travers un filtre de 0,22 μm pour éliminer les particules restantes de plus de 220 nm.
REMARQUE : Utiliser immédiatement pour les applications en aval, ou stocker pendant une nuit maximum à 4 °C ou congeler à -80 °C si d’autres étapes ne sont pas effectuées dans les 24 h.
- Coloration EV pour le tri
- Préparez une solution de 5 mM de diacétate de N-succinimidyle de 5(6)-carboxyfluorescéine (CFSE). Utilisez-le fraîchement préparé ou congelez-le à -20 °C à l’abri de la lumière.
- Passer directement de l’étape 1.1.1 à 1.1.5 (dans les 24 h) ou décongeler les VE stockés à -80 °C contenant des surnageants à 4 °C.
- Avant la coloration, réchauffer les surnageants à 37 °C et ajouter du CFSE pour une concentration finale de 10 μM (dilution 500 fois). Incuber pendant 1 h à 37 °C dans l’obscurité.
- Pendant l’incubation, ajouter 2 mL de PBS dans un concentrateur centrifuge (membrane en cellulose régénérée, MWCO 100 kDa), un capuchon et une centrifugeuse à 4 000 x g pendant 10 min dans un rotor à godets oscillants. Retirez le PBS non filtré du bas du dispositif de filtration et aspirez le filtrat du tube de collecte.
REMARQUE : Le protocole d’ultrafiltration centrifuge décrit est basé sur le traitement d’un maximum de 15 ml de l’échantillon (volume maximal). - Ajoutez jusqu’à 15 ml d’échantillon dans le concentrateur centrifuge et bouchez l’appareil. Centrifugeuse à 4 000 x g pendant 30 min maximum.
REMARQUE : Cela donnera un échantillon final de 500 μL en moyenne. Cependant, en fonction de facteurs tels que la nature de l’échantillon et le débit, le temps de centrifugation nécessaire pour atteindre la concentration souhaitée peut varier. - Retirez l’appareil de la centrifugeuse et videz le tube de collecte. Ajouter 14 ml de PBS dans le dispositif de filtration. Centrifugeuse à 4 000 x g pendant 30 min maximum.
- Répétez l’étape 1.2.6.
- Récupérer l’échantillon concentré d’VE coloré au CFSE à partir du dispositif de filtration, soit environ 500 μL, et stocker 100 μL à 4 °C dans l’obscurité tout en procédant au tri de l’échantillon restant.
2. Caractérisation des ASC-EV
- Analyse par transfert Western des marqueurs EV
- ASC en pastille (1 x 106) à 376 x g à RT pendant 5 min et en suspension dans un tampon de lyse approprié complété par des inhibiteurs de protéase. Effectuez la quantification des protéines avec le test de votre choix.
REMARQUE : Comme je l’ai constaté dans cette étude, les meilleurs résultats de quantification sont obtenus avec la technique de dosage de l’acide bicchoninique. - Quantifier le surnageant concentré en ASC équivalent à 1 x 106 ASC avec la technique de Bradford ou les VE en pastille correspondant à 1 x 106 ASC à 100 000 x g à 4 °C pendant 1 h et suspendre dans un tampon de lyse approprié complété par des inhibiteurs de protéase. Effectuez la quantification des protéines avec le test de votre choix.
REMARQUE : Comme je l’ai constaté dans cette étude, les meilleurs résultats de quantification pour les pastilles EV sont obtenus avec la technique de dosage de l’acide bicchoninique. - Si l’on utilise un surnageant concentré à la place des VE purifiés, lyser les échantillons dans du 2-Mercaptoéthanol à 5 % et un tampon Laemmli 2x.
REMARQUE : Comme nous l’avons constaté dans cette étude, les VE surnageants concentrés et purifiés donnent des résultats comparables pour les marqueurs positifs et négatifs des VE. - Charger et séparer les échantillons (1-10 μg) dans un gel de polyacrylamide à 10 % à 110 V pendant 90 min.
- Transfert sur une membrane de nitrocellulose à 250 mA pendant 120 min.
- Membrane de coloration avec Ponceau S pour visualiser les échantillons et le transfert en échelle. Retirer Ponceau S avec PBS en secouant doucement.
- Bloquez les membranes avec 5 % de lait écrémé en poudre et 0,1 % de Tween dans du PBS pendant 60 min.
- Membranes de sonde avec des anticorps appropriés aux dilutions de travail, y compris des marqueurs EV positifs (par exemple, dans ce travail CD9, CD81, TSG101 et Flotillin) et négatifs (par exemple, dans ce travail Calnexin), à 4 °C pendant la nuit.
- Laver avec 0,1 % de Tween dans du PBS et incuber avec des anticorps secondaires conjugués à la peroxydase appropriés pendant 45 min à RT avant de révéler la bande avec le système ECL de votre choix. Acquérez des images à l’aide d’un système d’imagerie disponible (Figure 2A).
- ASC en pastille (1 x 106) à 376 x g à RT pendant 5 min et en suspension dans un tampon de lyse approprié complété par des inhibiteurs de protéase. Effectuez la quantification des protéines avec le test de votre choix.
- Analyse par cytométrie en flux des marqueurs EV : coloration de surface
- Avant utilisation, centrifugez chaque anticorps monoclonal (mAb) à 15 000-17 000 x g pendant 30 min à 4 °C pour éliminer les agrégats, qui peuvent provoquer des signaux faussement positifs. De plus, filtrez les anticorps monoclonaux dans des tubes filtrants centrifuges séparés de 0,22 μm à 1 000 x g à 4 °C jusqu’à ce que tout le mélange soit passé à travers le filtre et qu’il ne reste plus d’anticorps liquide à la surface du filtre. Conservez les anticorps monoclonaux à 4 °C.
- Préparer une dilution de 1:10 de VE colorés au CFSE (voir les étapes 1.2.1-1.2.8) dans du PBS filtré à 0,22 μm.
- Incuber 100 μL d’échantillons, ou du PBS filtré à 0,22 μm, avec ou sans anticorps monostatiques spécifiques aux VE (anti-CD9/63/81) ou aux anticorps monoblocs spécifiques aux CSM (anti-CD44), préalablement titrés. Effectuer l’incubation à 4 °C dans l’obscurité pendant 30 min.
REMARQUE : Ce protocole, développé pour détecter les marqueurs typiques de la lignée EV et MSC, peut être étendu à tous les autres marqueurs de surface spécifiques à la lignée cellulaire ou au sous-type de VE. N’utilisez pas de fluorochromes tombant dans le canal de l’isothiocyanate de fluorescéine (FITC) pour éviter le chevauchement avec la coloration CFSE des VE. Effectuez une seule coloration en utilisant chaque mAb conjugué à un fluorochrome, par exemple l’allophycocyanine (APC). Une coloration multicolore est possible, mais des contrôles doivent être inclus pour résoudre les problèmes potentiels d’entrave aux anticorps stériques. Il est nécessaire de tester les anticorps individuels, seuls ou au sein d’un mélange, et de s’assurer que le signal est comparable. De plus, les OGF doivent être inclus dans les contrôles. - Configurez l’étalonnage de la diffusion comme décrit à l’étape 3.2.
- Créez un diagramme à points de l’échelle logarithmique de diffusion latérale (SSC) par rapport à l’échelle logarithmique FITC et exécutez un tube de PBS ou d’EV non colorés filtrés à 0,22 μm ; définissez le seuil sur le canal FITC et ajustez-le aux valeurs les plus élevées qui excluent la majorité du bruit de fond. Dessinez une région identifiant les événements positifs au CFSE.
- Créez un histogramme APC à portes sur la région d’événements positifs CFSE et exécutez un tube de PBS ou d’EV non colorés filtrés à 0,22 μm.
REMARQUE : Pour le gain APC, utilisez le gain établi avec l’instrument QC, bien qu’il soit recommandé d’effectuer un gain spécifique afin d’optimiser les performances de l’instrument. - Ajouter 200 μL de PBS filtré à 0,22 μm aux échantillons colorés et acquérir. Utilisez un faible débit (10 μL par min) pour l’acquisition et l’enregistrement. Si possible, enregistrez au moins 5 000 événements dans la porte positive FITC.
- Utilisez la commande PBS colorée aux anticorps monoclonaux pour détecter d’éventuels signaux aspécifiques des anticorps monoclonaux. Lisez tous les tubes d’échantillonnage au même débit pour assurer la cohérence entre les cycles.
- Pour éviter la contamination croisée de l’échantillon, appliquez un détergent pendant 10 s entre chaque tube analysé, suivi de 10 s avec de l’eau désionisée (DI) (Figure 2B).
REMARQUE : Pour éviter toute instabilité de l’instrument fluidique, acquérez l’échantillon pendant 10 s, puis commencez l’enregistrement.
- Analyse par cytométrie en flux d’un marqueur EV : coloration intracellulaire
REMARQUE : Pour la coloration intracellulaire, utilisez un kit spécifique contenant des réactifs de fixation et de perméabilisation. L’anticorps monoclonal anti-cavéoline est conjugué à Alexa 488. La coloration IC a été réalisée en l’absence de CFSE.- Avant utilisation, centrifugez l’AcM comme décrit précédemment (étape 2.2.1).
- Pour la coloration intracellulaire, suivez les instructions du fabricant (Figure 2C).
- Caractérisation de la concentration et de la taille des VE par analyse de suivi des nanoparticules (NTA)
- Effectuer des dilutions appropriées des échantillons pour obtenir 20 à 120 événements dans le cadre de visualisation de l’affichage de l’instrument NTA.
- Injectez les échantillons dans la chambre d’échantillonnage de l’instrument NTA à l’aide d’une seringue à insuline de 1 ml.
- Réglez le logiciel NTA pour l’enregistrement comme suit : 5 mesures standard de 60 s chacune et une pompe de débit réglée à 30.
- Réglez l’intensité lumineuse à une valeur appropriée permettant de distinguer clairement les particules de l’arrière-plan et exécutez le script de capture sur le logiciel NTA.
REMARQUE : Le réglage du niveau de la caméra peut influencer la détection des particules. Si la valeur est trop élevée, un fond laiteux et une forte diffusion de la lumière des particules apparaissent, couvrant la plupart des signaux de particules. Si la valeur est trop faible, bien qu’un arrière-plan plus sombre soit obtenu, il est possible que la plupart des événements moins lumineux soient manqués. Chaque échantillon doit être surveillé pour un réglage optimal. - Ajustez le facteur de dilution si les particules par image sont inférieures à 20 ou supérieures à 80.
- Réglez le seuil sur 4 pour analyser la taille modale moyenne et la concentration en particules du surnageant. Tenez compte des facteurs de dilution initiaux.
- Nettoyez la chambre de l’instrument avec de l’eau désionisée après chaque échantillon pour éviter la contamination croisée.
- Ouvrez le logiciel d’analyse NTA et effectuez l’analyse sur les échantillons acquis, en tenant compte du facteur de dilution (Figure 2D).
- Caractérisation de la morphologie des VE par microscopie électronique à transmission (MET)
- Prélever les surnageants concentrés en ASC ou isoler les VE par ultracentrifugation (100 000 x g à 4 °C pendant 1 h) suivie d’une suspension de pastilles dans le même volume de PBS.
- Laisser absorber 5 μL du surnageant concentré ou des VE sur des grilles recouvertes de carbone pendant 10 min. Épongez les gouttes de liquide en excès avec du papier filtre.
- Effectuer une coloration négative avec de l’acétate d’uranyle à 2 % en suspension aqueuse pendant 10 min en utilisant un volume identique de goutte d’échantillon (5 μL). Retirez l’excès d’acétate d’uranyle en touchant la grille avec du papier filtre.
- Séchez la grille à température ambiante (RT) et examinez-la à l’aide d’un microscope électronique à transmission à 120 kV (Figure 2E).
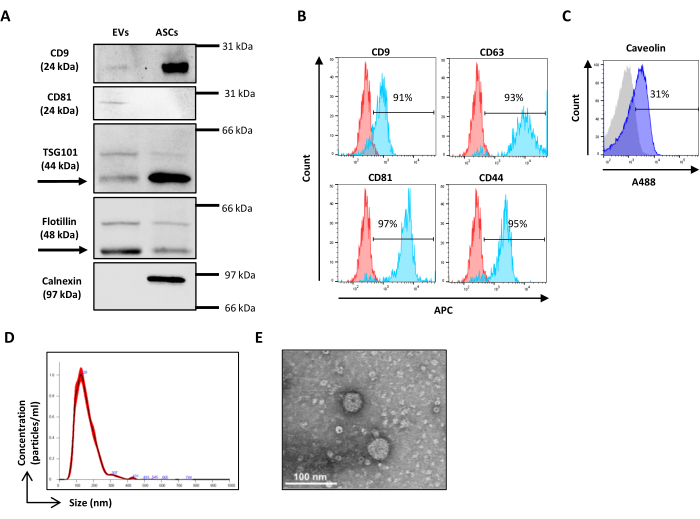
Figure 2 : Caractérisation des ASC-EV. (A) Transfert Western représentatif des marqueurs positifs (CD9, CD81, TSG101 et Flotilline) et négatifs (Calnexin). Les poids moléculaires correspondants sont rapportés, et les lysats d’ASC ont été utilisés comme témoin. (B) Analyse par cytométrie en flux des marqueurs EV. L’expression des marqueurs suivants a été analysée : CD9, CD63, CD81 et CD44. Seules les ASC-EV positives au CFSE ont été analysées pour l’expression des marqueurs. Les histogrammes représentent des ASC-EV non colorés (histogrammes rouges) et colorés (histogrammes bleus). (C) Analyse intracellulaire par cytométrie en flux du marqueur EV Cavéoline. Les histogrammes représentent des ASC-EV non colorés (histogrammes gris) et colorés (histogrammes bleus). (D) Caractérisation des ASC-EV par la NTA. Les histogrammes représentent la concentration (particules/mL)/la taille (nm) de l’échantillon. e) Visualisation des ASC-EV par TEM. Barres d’échelle = 100 nm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Tri des ASC-EV
- Configuration du trieur de cellules
REMARQUE : Un trieur de cellules est un cytomètre en flux qui permet d’isoler une population pure à partir d’un échantillon de départ hétérogène. Le trieur de cellules sépare les particules cibles en faisant osciller le flux avec un piézoélectrique à haute fréquence pour générer des gouttes contenant un événement. Les gouttes qui contenaient les particules d’intérêt, telles que les cellules ou les vésicules, sont chargées et déviées à travers des plaques de déviation métalliques. La fraction triée est utilisée pour effectuer l’analyse en aval.- Ouvrez la conduite de pression et faites l’aspirateur.
REMARQUE : Certains trieurs ont un démarrage automatique, tandis que d’autres ont un démarrage manuel. Un trieur à démarrage manuel est privilégié, car il permet d’optimiser certaines caractéristiques techniques telles que la fréquence, la pression, et le choix de l’embout de la buse. En particulier, il est recommandé de travailler avec une fréquence de 66 000 Hz, une pression de 35 psi et une buse de 70 μm. - Allumez l’instrument, ouvrez l’armoire de biosécurité et lancez le logiciel du trieur.
- Mettez sous pression la fluidique, puis allumez le système fluidique.
- Activez Drop Drive, le cristal piézoélectrique dans la buse qui vibre pour avoir des gouttelettes.
- Effectuez une procédure de débauchage pour éliminer la présence de bulles dans le système, puis faites fonctionner un tube de 5 mL de solution de nettoyage pendant 5 min à haute pression différentielle et un tube de 5 mL d’eau DI pendant 5 min.
- Effectuez la procédure de démarrage manuel.
- Dans l’onglet Contrôle laser, appuyez sur le bouton Alimentation laser pour allumer l’alimentation laser.
REMARQUE : Répétez cette procédure pour tous les lasers : Bleu 488 nm, Jaune-Vert 561 nm, Violet 405 nm et Rouge 640 nm. - Examinez l’alignement vertical du cours d’eau.
REMARQUE : En cas de besoin, déplacez l’étage d’assemblage de la buse, en particulier le micromètre avant et arrière, le micromètre gauche et droit et le cardan pour ajuster l’alignement vertical et vérifier le flux vertical avec le micromètre haut et bas ; Pour un meilleur alignement, le jet doit être vertical sur chaque laser. Si c’est la première fois que l’on utilise une buse spécifique, un délai laser doit être effectué. Sur le panneau de l’écran tactile, dans l’onglet Interception laser et flux , sélectionnez la taille de buse correcte, appuyez sur le bouton Retard laser et suivez les instructions de retard laser pour effectuer la procédure de retard laser. En général, lors de la modification de la pression de la gaine et, par conséquent, de la pointe de la buse, la détermination du retard laser est exécutée avant de trouver l’alignement. À la fin de la procédure de retard laser , il est suggéré d’effectuer la procédure de soustraction de l’image d’arrière-plan en suivant les instructions à l’écran afin de minimiser le signal de fond. - Effectuez la détermination du point laser en appuyant sur l’onglet Interception du flux laser , sur le bouton Flèche verte et en suivant les instructions sur le moniteur à écran tactile.
- Dans l’onglet Contrôle laser, appuyez sur le bouton Alimentation laser pour allumer l’alimentation laser.
- Initialisez Intellisort. Définissez la fréquence du lecteur de chute et une valeur d’amplitude par défaut, ce qui entraîne la formation d’une gouttelette dans le flux.
- Une fois cette étape effectuée, chargez une configuration enregistrée. Pour charger une configuration de paramètre enregistrée, sélectionnez Charger le paramètre de tri dans la barre d’outils du logiciel de tri. Après avoir vérifié les différentes fréquences et amplitudes, choisissez celle qui assure la meilleure stabilité de chute. Fonctionne à une fréquence de 66 000 Hz et à une amplitude d’environ 40-45 V.
- Effectuez la procédure d’alignement laser fin .
- Chargez dans l’échantillonneur un tube de 5 mL de billes d’alignement QC et passez-le ; sélectionnez dans l’onglet Alignement fin le paramètre souhaité sur les axes X et Y afin de visualiser les billes bien compactées et collimatées. Dans la zone d’affichage des données, sélectionnez d’abord 488 - 513/26-H pour le paramètre de l’axe Y et 488-FSC1-H pour le paramètre de l’axe X. Ensuite, pour affiner tous les lasers, sélectionnez 405-488/59-H pour le paramètre de l’axe Y et 640-795/70-H pour le paramètre de l’axe X.
REMARQUE : Les paramètres sélectionnés dépendent de la configuration laser du trieur. La suggestion du manuel de l’instrument est le choix des lasers qui ont la plus grande séparation spatiale sur la bande de sténopé. Pour cela, utilisez un laser Violet (405 nm) et un laser Rouge (640 nm). - Une fois l’alignement manuel vérifié par le spécialiste du trieur, effectuez le CQ automatique. Sélectionnez le diamètre des billes QC : 3 μm de diamètre.
REMARQUE : À la fin de cette procédure, le résultat peut être que le contrôle qualité n’a pas réussi ou que le contrôle qualité a réussi. En cas d’échec du QC, il est nécessaire d’optimiser manuellement l’alignement. - Enregistrez l’acquisition du contrôle qualité sur le protocole d’alignement logiciel.
- Chargez dans l’échantillonneur un tube de 5 mL de billes d’alignement QC et passez-le ; sélectionnez dans l’onglet Alignement fin le paramètre souhaité sur les axes X et Y afin de visualiser les billes bien compactées et collimatées. Dans la zone d’affichage des données, sélectionnez d’abord 488 - 513/26-H pour le paramètre de l’axe Y et 488-FSC1-H pour le paramètre de l’axe X. Ensuite, pour affiner tous les lasers, sélectionnez 405-488/59-H pour le paramètre de l’axe Y et 640-795/70-H pour le paramètre de l’axe X.
- Finalisation de la procédure Intellisort
- Placez les plaques de déviation dans la bonne position dans le trieur et allumez la tension (une charge de 3 000 V sur les plaques de déviation est recommandée).
- Choisissez 6 sorties de tri du support de tube, activez les flux en les sélectionnant sur l’indicateur de flux et effectuez le flux de test.
REMARQUE : Si les flux ne sont pas clairement séparés et définis, ajustez la phase de charge, le défanage ou modifiez d’autres paramètres afin d’obtenir un flux clairement défini et non oscillant. - Activez le bouton Intellisort Automatic Drop Delay Determination . Cette étape permet de définir le délai de chute correct. Après cette étape, vérifiez à nouveau les flux.
- Activez le mode de maintenance Intellisort. Vérifiez manuellement le délai de chute .
- Chargez le protocole manuel Drop Delay sur le logiciel de tri. Acquérir des billes de contrôle fluorescentes.
- Insérez le bon support de diapositive. Dans le logiciel de tri, sélectionnez Trier > Assistant de délai de dépôt. Sélectionnez Logique de tri.
- Vérifiez avec un microscope fluorescent la présence de 97 % de billes fluorescentes sur la cinquième flaque d’eau.
- Ouvrez la conduite de pression et faites l’aspirateur.
- Configuration de l’instrument : Étalonnage de la diffusion
REMARQUE : L’étalonnage de la diffusion est effectué à l’aide de billes Verity Shells (ci-après appelées billes creuses d’organo-silice). Ces billes d’organosilice se caractérisent par une distribution de l’indice de réfraction et des propriétés de diffusion de la lumière plus proches des VE que des billes de polystyrène, permettant ainsi la configuration de la tension SSC et FSC pour mieux distinguer les événements de type EV et le bruit électronique. Les billes d’organosilice sont constituées de deux flacons avec des mélanges de billes, VER01A (billes de 189 nm) et VER01B (billes de 374 nm). Chaque flacon contient un mélange de billes creuses d’organosilice et de billes fluorescentes vertes de 380 nm, appelées billes de référence. La concentration des billes creuses de silice organo-organique est de 1 x 108 billes/mL.- Préparez un échantillon de billes VER01A et VER01B en diluant une goutte (50 μL) de chacune dans 1 mL de PBS filtré à 0,22 μm avec une concentration finale de 5 000 billes/μL.
REMARQUE : Vortex les perles pendant 5 s avant de les utiliser. - Créez un diagramme à points de l’échelle logarithmique de SPC par rapport à l’échelle logarithmique FITC.
- Munissez-vous des billes VER01B et ajustez la tension pour bien discriminer les billes de référence dans la partie supérieure droite du graphique à points SSC/FITC (Figure 3A).
- Définissez une région autour de cette population de perles.
- Créez un diagramme à points de l’échelle logarithmique SSC par rapport à l’échelle logarithmique FSC. Identifiez les billes de référence et la population de billes VER01B. Définissez une région autour de la population de billes VER01B (Figure 3B).
- Acquérez des billes VER01A et créez une région autour de cette population de billes (Figure 3C).
REMARQUE : Cette population de perles chevauche en partie le bruit de fond de l’instrument. - Acquérez un PBS filtré de 0,22 μm et ajustez le seuil pour diminuer le bruit de fond de l’instrument sans perdre la visualisation des plus petites billes.
REMARQUE : L’étalonnage de la diffusion peut être effectué à l’aide de billes FSC Megamix (ci-après appelées billes de polystyrène FSC), un mélange de billes fluorescentes des diamètres suivants : 100 nm, 300 nm, 500 nm et 900 nm. L’utilisation des billes creuses d’organosilice doit être préférée aux billes de polystyrène FSC car elles ont des propriétés de distribution de l’indice de réfraction et de diffusion de la lumière similaires à celles des VE. La population de billes de polystyrène FSC de 300 nm chevauche la référence des billes de silice organo-organique creuses. Par conséquent, une porte basée sur des billes creuses d’organosilice sélectionne des VE de taille similaire sur le cytomètre en flux.
- Préparez un échantillon de billes VER01A et VER01B en diluant une goutte (50 μL) de chacune dans 1 mL de PBS filtré à 0,22 μm avec une concentration finale de 5 000 billes/μL.
- Acquisition d’échantillons
- Créez un diagramme à points de l’échelle logarithmique SSC par rapport à l’échelle logarithmique FITC, acquérez un PBS filtré de 0,22 μm et définissez le bruit de fond de référence (Figure 4A).
REMARQUE : Réglez le débit du cytomètre sur Faible et contrôlez le débit. Le nombre d’événements/s ne doit pas dépasser 2 000 événements/s. Débit Faible signifie à faible pression différentielle. - Procurez-vous l’échantillon de contrôle (EV non colorés) et vérifiez le débit. Assurez-vous que le nombre d’événements/s est d’environ 5 000 événements/s.
REMARQUE : Si le nombre d’événements dépasse celui suggéré, diluez l’échantillon. - Acquérir du PBS coloré et traité comme l’échantillon CFSE et vérifier qu’aucun événement positif n’est visible (Figure 4B).
- Procurez-vous l’échantillon coloré au CFSE. Diluez-le en fonction du débit. Dessinez une région identifiant les événements positifs du CFSE et une autre identifiant les événements négatifs du CFSE. Utilisez un échantillon non coloré comme témoin (figures 5A, B). Il s’agit des régions de tri.
- Créez un diagramme à points de l’échelle logarithmique SSC par rapport à l’échelle logarithmique FITC, acquérez un PBS filtré de 0,22 μm et définissez le bruit de fond de référence (Figure 4A).
- Tri des échantillons
- Sur le logiciel de tri, ouvrez un nouveau protocole (dans la barre d’outils, sélectionnez Fichier > Protocole > Nouveau).
- Sur le logiciel de tri, cliquez sur Paramètres d’acquisition de données ; dans la fenêtre Paramètre d’acquisition, sélectionnez les chaînes qui vous intéressent et désactivez les autres. Pour ce panneau spécifique, les signaux activés sont les suivants : 488-FSC1, 488-FSC2, 488-SSC et 488-513/26 (pour le signal CFSE).
- Localisez sur l’échantillonneur un tube de 5 mL d’échantillon. Sur l’écran tactile, cliquez sur le bouton Charger , puis dans la barre d’outils du logiciel de tri, sélectionnez Acquisition > Démarrer (ou appuyez sur le bouton F2 ). Une fenêtre d’exemples de propriétés s’affiche ; Insérez le nom de l’échantillon et cliquez sur OK.
- Créez le panneau, avec la parcelle d’intérêt et créez la stratégie de gating.
- Dans la barre d’outils du logiciel de tri, sélectionnez Histogramme > Créer un histogramme.
- Dans l’espace de travail, créez trois diagrammes à points, le premier avec le paramètre Height-Log sur l’axe x 488-FSC1 et le paramètre Height-Log 488-SSC sur l’axe y, le second avec le paramètre Height-Log sur l’axe x 488-FSC2 et l’axe y 488-SSC Height-Log, le troisième avec le paramètre CFSE Height-Log sur l’axe x 488-513/26 et l’axe y 488-SSC Height-Log.
- Dans l’onglet Trier, identifiez la ou les régions à trier. Sur le logiciel de tri, cliquez sur Paramètres de tri. Dans la fenêtre Logique de tri et statistiques , sélectionnez la région à trier dans le générateur de logiques.
REMARQUE : Pour prendre une décision de tri, vous devez prendre en compte différents paramètres : Logique de tri, Mode de tri et Enveloppe de dépôt. La logique de tri fait référence à la stratégie de contrôle. Les événements qui se trouvent en dehors de la porte sélectionnée ne sont pas triés. Le mode de tri est lié à la sortie finale. Travaillez en mode purification, de cette façon, toutes les gouttes triées ne contiennent que les événements positifs souhaités. L’enveloppe de largage définit le nombre de butins qui seront facturés et triés en fonction de l’emplacement dans le largage de l’événement positif : choisissez 1-2 largages afin d’être sûr que tous les événements positifs sont triés. - Lorsque le débit est stable, arrêtez l’acquisition (dans la barre d’outils du logiciel du trieur, sélectionnez Acquisition > Arrêt ou appuyez à nouveau sur le bouton F2 ). Démarrer le tri (dans la barre d’outils du logiciel de tri, sélectionnez Trier > Démarrer ou appuyez sur la touche F4 ).
REMARQUE : Avant de commencer le tri, vérifiez le flux de test. Vérifiez que la fraction triée tombe correctement dans le tube de collecte. - Enregistrez le paramètre de tri. Dans la barre d’outils du logiciel de tri, sélectionnez Trier > Enregistrer les paramètres de tri.
- Contrôle de pureté de la population triée
- Lavez l’instrument en acquérant un tube de 5 ml de solution de nettoyage pendant 10 min à haute pression différentielle et un tube de 5 ml d’eau DI pendant 10 min.
- Acquérez un PBS filtré à 0,22 μm et vérifiez qu’aucun événement CFSE-positif n’est présent.
- Diluez 5 μL de l’échantillon trié dans 100 μL de PBS filtré à 0,22 μm.
- Acquérez et enregistrez tous les volumes d’échantillons (Figure 5C, D).
REMARQUE : Une nouvelle analyse des VE triés positifs et négatifs du CFSE est rapportée.
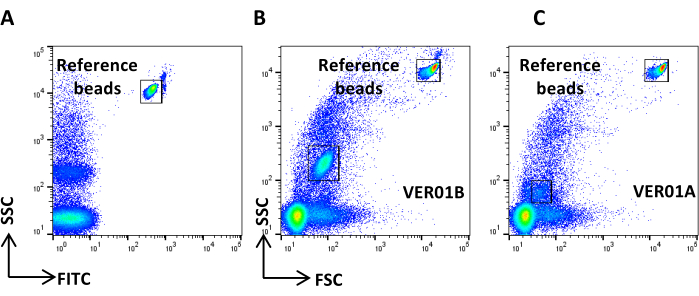
Figure 3 : Réglage des paramètres physiques avec des billes creuses d’organo-silice. (A) Graphique à points SSC/FITC : des billes fluorescentes vertes de référence ont été utilisées pour définir le paramètre SSC. Graphique en points SSC/FSC des billes (B) VER01B et (C) VER01A. Veuillez cliquer ici pour voir une version agrandie de cette figure.
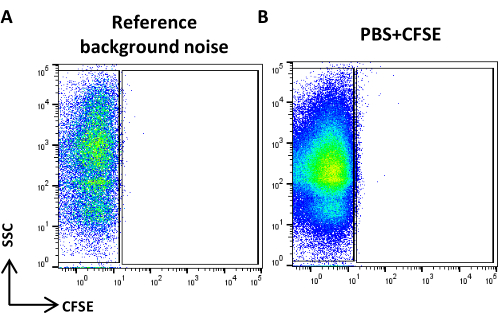
Figure 4 : Bruit de fond de référence. (A) Diagramme à points SSC/CFSE de l’échantillon PBS. (B) Graphique à points SSC/CFSE de l’échantillon PBS + CFSE. Veuillez cliquer ici pour voir une version agrandie de cette figure.
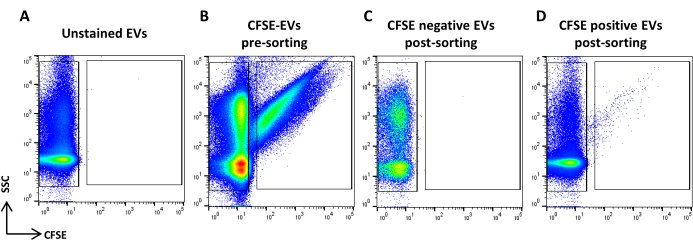
Figure 5 : Tri des ASC-EV colorés CFSE. (A) Graphique à points SSC/CFSE des EV non colorés, (B) EV colorés CFSE, (C) EV négatifs CFSE post-tri, et (D) EV positifs CFSE post-tri. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Analyses de tri des articles
REMARQUE : En raison de la quantité limitée de matériaux après le tri, il peut être impossible d’effectuer toutes les analyses. Avec le montant obtenu, les éléments suivants sont effectués.
- Caractérisation de l’EV par cytométrie en flux : coloration de surface (voir étape 2.2)
REMARQUE : Comme décrit précédemment pour le pré-tri des échantillons, à l’exception de la préparation des échantillons. Après tri, les échantillons peuvent être utilisés non dilués.- Utilisez des échantillons triés non dilués et procédez comme à l’étape 2.2.3. (Figure 6A)
- Caractérisation de la concentration et de la taille des VE par NTA (voir étape 2.4)
REMARQUE : Comme décrit précédemment pour le pré-tri des échantillons, à l’exception de la préparation des échantillons. Après tri, les échantillons peuvent être utilisés non dilués.- Utilisez des échantillons triés non dilués et procédez comme indiqué à l’étape 2.4.2. (Figure 6B)
- Caractérisation de la morphologie des VE par TEM (voir étape 2.5)
REMARQUE : Comme décrit précédemment pour le pré-tri des échantillons, à l’exception de la préparation des échantillons.- Ajouter 2 ml de PBS dans un concentrateur centrifuge (membrane en cellulose régénérée, MWCO 100 kDa), un capuchon et une centrifugeuse à 4 000 x g pendant 10 min dans un rotor à godet oscillant. Retirez le PBS non filtré du bas de l’appareil de filtration.
- Aspirer le filtrat du tube collecteur. Le protocole d’ultrafiltration centrifuge décrit est basé sur le traitement d’échantillons allant jusqu’à 15 mL (volume maximal).
- Ajoutez jusqu’à 15 ml d’échantillon dans le filtre AU-15 et bouchez l’appareil. Centrifugeuse à 4 000 x g pendant 30 min maximum. Concentrez-vous jusqu’au volume minimal autorisé par l’appareil, soit environ 100-150 μL.
- Récupérer l’échantillon EV trié concentré du dispositif de filtration et procéder comme décrit précédemment pour le pré-tri des échantillons TEM (étape 2.5.2) ( figure 6C).
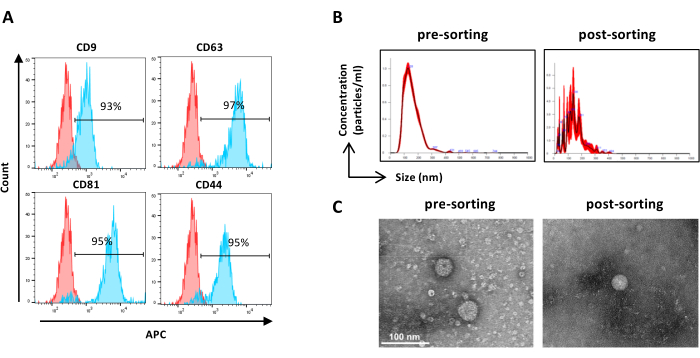
Figure 6 : Caractérisation des ASC-EV triés. Analyse par cytométrie en flux des marqueurs EV. L’expression des marqueurs suivants a été analysée : CD9, CD63, CD81 et CD44. Seules les ASC-EV positives au CFSE ont été analysées pour l’expression des marqueurs. (A) Les histogrammes représentent des ASC-EV non colorés (histogrammes rouges) et colorés (histogrammes bleus). b) Caractérisation des ASC-EV par la NTA. Les histogrammes représentent la concentration (particules/mL)/la taille (nm) des échantillons de pré-tri (à gauche) et de post-tri (à droite). (C) Visualisation des ASC-EV par TEM d’un échantillon de pré-tri (à gauche) et de post-tri (à droite). Barres d’échelle = 100 nm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
Les billes de polystyrène FSC ont été triées pour valider la configuration de l’instrument et les conditions de tri. Les billes de polystyrène FSC sont un mélange de billes fluorescentes allant de 100 nm, 300 nm, 500 nm et 900 nm et sont visibles sur le canal FITC. La figure 7A montre le diagramme logarithmique de la SSC par rapport au diagramme logarithmique de la FITC avec les quatre populations de billes avant le tri. Les populations fluorescentes...
Access restricted. Please log in or start a trial to view this content.
Discussion
L’analyse et le tri des VE sont difficiles en raison de leur petite taille et du fait qu’ils sont proches de la limite de détection de la plupart des cytomètres en flux. Notre objectif était de développer un protocole d’isolement des VE dérivés d’AMSC marqués avec CFSE. La CFSE a été choisie comme méthode de coloration en raison de son efficacité de marquage élevée des VE (≥90 %), sans formation de particules indésirables telles que les agrégats de protéines do...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Emanuele Canonico pour son soutien technique. Une partie de ce travail a été réalisée à ALEMBIC, un laboratoire de microscopie avancée établi par l’IRCCS Ospedale San Raffaele et l’Università Vita-Salute San Raffaele. Le travail d’Enrico Ragni et de Laura de Girolamo a été soutenu par le ministère italien de la Santé, « Ricerca Corrente ».
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 5(6)-Carboxyfluorescein diacetate N-succinimidyl ester | Merck | 150347-59-4 | |
| Adipose Mesenchymal Stromal Cells | Wepredic, Parc d'affaires, 35760 Saint-Grégoire, France | Cells used in this study | |
| Alexa 488 anti-Caveolin | R&D Systems | IC5736G | Flow cytometry antibody |
| APC anti-human CD44 | BioLegend | 338805 | Flow cytometry antibody |
| APC anti-human CD63 | BioLegend | 353007 | Flow cytometry antibody |
| APC anti-human CD81 (TAPA-1) | BioLegend | 349509 | Flow cytometry antibody |
| APC anti-human CD9 | BioLegend | 312107 | Flow cytometry antibody |
| BC CytoFLEX S | Beckman Coulter | BC CytoFLEX S equipped with 3 lasers, Blue, Red and Violet | |
| Flow-Check Pro Fluorospheres | Beckman Coulter | A63493 | Fluorescent control beads for MoFLO Astrios EQ |
| FlowJo software (version 10.8.1) | BD | version 10.8.1 | Analysis software |
| IntraSure kit | BD Biosciences | 641776 | Fixation and permeabilization for intracellular staining |
| Megamix-Plus FSC | BioCytex | 7802 | FSC polystyrene beads |
| MoFLO Astrios EQ | Beckman Coulter | MoFLO Astrios EQ equipped with 4 lasers, Blue, Yellow - Green, Violet and Red | |
| Mouse anti-FLOT1 antibody | BD Transduction Laboratories | 610820 | Western Blot antibody |
| NanoSight NS300 | Malvern | NS300 | |
| Rabbit anti-Calnexin antibody | Origene | TA336279 | Western Blot antibody |
| Rabbit anti-CD9 and CD81 antibody (ExoAb antibody kit) | System Biosciences | EXOAB-KIT-1 | Western Blot antibodies |
| Rabbit anti-TSG101 antibody | Merck | HPA006161 | Western Blot antibody |
| Triton X-100 | Merck | 9036-19-5 | |
| Ultra Rainbow Fluorescent Particles | Spherotech | URFP-30-2 | |
| Ultracel 100 kDa MWCO | Merck | UFC910024 | |
| VER01 - Verity Shells | Exometry | Organo silica beads for scatter calibration |
Références
- Welsh, J. A., et al. Minimal information for studies of extracellular vesicles (misev2023): From basic to advanced approaches. J Extracell Vesicles. 13 (2), e12404(2024).
- Xu, R., Greening, D. W., Zhu, H. J., Takahashi, N., Simpson, R. J. Extracellular vesicle isolation and characterization: Toward clinical application. J Clin Invest. 126 (4), 1152-1162 (2016).
- Dixson, A. C., Dawson, T. R., Di Vizio, D., Weaver, A. M. Context-specific regulation of extracellular vesicle biogenesis and cargo selection. Nat Rev Mol Cell Biol. 24 (7), 454-476 (2023).
- Witwer, K. W., Wolfram, J. Extracellular vesicles versus synthetic nanoparticles for drug delivery. Nat Rev Mater. 6 (2), 103-106 (2021).
- Cheng, L., Hill, A. F. Therapeutically harnessing extracellular vesicles. Nat Rev Drug Discov. 21 (5), 379-399 (2022).
- Du, S., et al. Extracellular vesicles: A rising star for therapeutics and drug delivery. J Nanobiotechnology. 21 (1), 231(2023).
- Keshtkar, S., Azarpira, N., Ghahremani, M. H. Mesenchymal stem cell-derived extracellular vesicles: Novel frontiers in regenerative medicine. Stem Cell Res Ther. 9 (1), 63(2018).
- Orbay, H., Tobita, M., Mizuno, H. Mesenchymal stem cells isolated from adipose and other tissues: Basic biological properties and clinical applications. Stem Cells Int. 2012, 461718(2012).
- Caplan, A. I., Dennis, J. E. Mesenchymal stem cells as trophic mediators. J Cell Biochem. 98 (5), 1076-1084 (2006).
- Rani, S., Ryan, A. E., Griffin, M. D., Ritter, T. Mesenchymal stem cell-derived extracellular vesicles: Toward cell-free therapeutic applications. Mol Ther. 23 (5), 812-823 (2015).
- Galipeau, J., Sensébé, L. Mesenchymal stromal cells: Clinical challenges and therapeutic opportunities. Cell Stem Cell. 22 (6), 824-833 (2018).
- Jia, Y., et al. Small extracellular vesicles isolation and separation: Current techniques, pending questions and clinical applications. Theranostics. 12 (15), 6548-6575 (2022).
- Ohnuma, K., Yomo, T., Asashima, M., Kaneko, K. Sorting of cells of the same size, shape, and cell cycle stage for a single cell level assay without staining. BMC Cell Biol. 7, 25(2006).
- Morales-Kastresana, A., et al. High-fidelity detection and sorting of nanoscale vesicles in viral disease and cancer. J Extracell Vesicles. 8 (1), 1597603(2019).
- Mortati, L., et al. In vitro study of extracellular vesicles migration in cartilage-derived osteoarthritis samples using real-time quantitative multimodal nonlinear optics imaging. Pharmaceutics. 12 (8), 734(2020).
- Andreu, Z., Yanez-Mo, M. Tetraspanins in extracellular vesicle formation and function. Front Immunol. 5, 442(2014).
- Mildmay-White, A., Khan, W. Cell surface markers on adipose-derived stem cells: A systematic review. Curr Stem Cell Res Ther. 12 (6), 484-492 (2017).
- Welsh, J. A., Tang, V. A., Van Der Pol, E., Gorgens, A. MIFlowCyt-EV: The next chapter in the reporting and reliability of single extracellular vesicle flow cytometry experiments. Cytometry A. 99 (4), 365-368 (2021).
- Maia, J., et al. Employing flow cytometry to extracellular vesicles sample microvolume analysis and quality control. Front Cell Dev Biol. 8, 593750(2020).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon