Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Multimodale Studie des kardiovaskulären Umbaus von Mäusen: Vierdimensionale Ultraschall- und Massenspektrometrie-Bildgebung
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir ein Protokoll zur Verwendung von vierdimensionaler In-vivo-Ultraschallbildgebung und ex vivo-Massenspektrometrie-Bildgebung zur Beurteilung biomechanischer und biomolekularer Veränderungen im kardiovaskulären System der Maus. Diese Technik wird zur Analyse des kardialen Umbaus bei chirurgisch induziertem Myokardinfarkt und vaskulären Veränderungen bei alternden Tieren eingesetzt.
Zusammenfassung
Herz-Kreislauf-Erkrankungen (CVD) sind die häufigste Todesursache in den Vereinigten Staaten. Schäden im Herz-Kreislauf-System können auf Umwelteinflüsse, Traumata, Arzneimitteltoxizität oder zahlreiche andere Faktoren zurückzuführen sein. In der Folge verändern sich das Herzgewebe und die Gefäße strukturell und weisen eine verminderte Funktion auf. Die Schädigung und der daraus resultierende Umbau können mit Ultraschall (US) auf Organebene und Massenspektrometrie (MSI) auf molekularer Ebene nachgewiesen und quantifiziert werden. Dieses Manuskript beschreibt eine innovative Methodik zur Untersuchung der kardialen Pathophysiologie von Mäusen, die in vivo vierdimensionale (4D) Ultraschallbildgebung und -analyse mit ex vivo matrixgestützter Laserdesorption/Ionisation (MADLI) MSI des Herzens koppelt. 4D-Ultraschall kann dynamische volumetrische Messungen liefern, einschließlich radialer Verschiebung, Oberflächendehnung und Längsdehnung während eines gesamten Herzzyklus. Im Gefäßsystem werden MSI und Ultraschall verwendet, um die Zusammensetzung der Gefäßwand, die Hämodynamik und die Dynamik der Gefäßwand zu beurteilen. Die Methodik kann auf die Untersuchung einer Vielzahl von Herz-Kreislauf-Erkrankungen zugeschnitten werden, indem funktionelle Metriken von Interesse und/oder das MALDI-MSI-Protokoll angepasst werden, um auf bestimmte Moleküle abzuzielen. MALDI MSI kann zur Untersuchung von Lipiden, kleinen Metaboliten, Peptiden und Glykanen verwendet werden. Dieses Protokoll beschreibt die Verwendung von MALDI MSI für die ungezielte lipidomische Analyse und die Verwendung von Ultraschallbildgebung für die kardiovaskuläre Hämodynamik und Biomechanik.
Einleitung
Herz-Kreislauf-Erkrankungen (CVD) sind weltweit eine der häufigsten Todesursachen1. Die Prävention und Behandlung von Herz-Kreislauf-Erkrankungen erfordert ein tiefgreifendes Verständnis der molekularen Anpassungen an biomechanische Kräfte und der daraus resultierenden Veränderungen der mechanischen Eigenschaften. Im gesamten Herz-Kreislauf-System spielen biomechanische Kräfte eine wichtige Rolle für die Funktion und Struktur des Gewebes2. Die mechanischen Eigenschaften des Herz-Kreislauf-Gewebes (CV) werden von diesen Kräften beeinflusst, was sie zu Indikatoren für Gesundheit und Krankheitmacht 3,4,5,6. Um Herz-Kreislauf-Erkrankungen zu verhindern, zu diagnostizieren und zu behandeln, ist es von entscheidender Bedeutung, Methoden zum Verständnis und zur Beobachtung der Prozesse der Krankheitsentstehung und -progression zu entwickeln. Die biomedizinische Bildgebung war der Schlüssel zur Generierung physiologischer und mechanistischer Erkenntnisse, und neue Bildgebungstechnologien und Analysetechniken werden ständig entwickelt. Dieses Protokoll demonstriert eine Methodik zur Kombination von zwei kardiovaskulären Bildgebungs- und Analysetechniken, um das Potenzial dieser Bildgebungsmodalitäten bei ischämischen Herzerkrankungen und vaskulärer Alterung zu validieren.
Forscher auf dem Gebiet der Biomechanik nähern sich der Erforschung der Biomechanik häufig über eine Kombination aus in vivo-, ex vivo- und in silico-Methoden . Frühere Forschungen zur molekularen Biomechanik konzentrierten sich hauptsächlich auf die Proteine7 (insbesondere die extrazellulären Matrixproteine Kollagen und Elastin aufgrund ihres Einflusses auf die biomechanischen Eigenschaften), und die Arbeit zur Kombination der In-vivo-Bildgebungsbiomechanik mit molekularen Studien beschränkte sich auf Histologie und Immunhistochemie. Obwohl diese Ansätze viele molekulare Indikatoren liefern können und vorgeschlagene Mechanismen des Umbaus von EZM und Zellen hervorgebracht haben, sind sie in der Regel auf die derzeit verfügbaren Färbungen bzw. Antikörper beschränkt. In diesem Forschungsgebiet fehlen große Klassen von Molekülen, z.B. Lipide. Obwohl diese molekularen Klassen mechanistisch beteiligt sein können oder auch nicht, sind die daraus resultierenden molekularen Anpassungen wichtig zu verstehen, da diese Moleküle potenzielle Ziele sowohl für diagnostische Marker als auch für Therapeutika sein könnten. Analytische chemische Techniken, wie z. B. die Flüssigkeitschromatographie-Massenspektrometrie (LC-MS), können angewendet werden; Allerdings geht bei diesen Techniken die räumliche Orientierung der Moleküle verloren. Mit der massenspektrometrischen Bildgebung (MSI) bleibt die räumliche Verteilung der Moleküle intakt, und mehrere Analyttypen (Klassen von Molekülen) können mit seriellen Schnitten abgebildet werden. MSI ist ein leistungsstarkes Analysewerkzeug zur Untersuchung der räumlichen Verteilung nahezu aller Arten von Molekülen in biologischem Gewebe, einschließlich Metaboliten, Lipiden, Glykanen, Peptiden und niedermolekularen Arzneimitteln8. Matrixgestützte Laserdesorption/Ionisation (MALDI) MSI ist eine Art von MSI, die sich gut für die forschungsbasierte Analyse von Molekulargewichten im Bereich von 50-8000 Da eignet. MALDI-MSI ist eine Ionisationstechnik, bei der eine laserenergieabsorbierende Matrix auf die Probe aufgebracht wird, um die Ionisation der interessierenden Analyten zu unterstützen. Dieser Ansatz verhindert, dass man sich auf ein molekulares Ziel beschränkt, und kann mit Hilfe von Bioinformatik-Werkzeugen bestimmen, welche Moleküle einen Einfluss auf die biomechanischen Eigenschaften und den Umbau haben.
Der vierdimensionale Ultraschall (4DUS) ist eine nicht-invasive in vivo-Methode, die sowohl für die zeitliche als auch für die räumliche Charakterisierung des Herzens nützlich ist. 4DUS verwendet eine Reihe von Cine-Loops mit hoher Bildrate aus verschiedenen Ebenen und kompiliert sie zu einem 3D-Datensatz, der zeitliche Informationen enthält. Dies ermöglicht die direkte Visualisierung und Quantifizierung der komplexen 3D-Formänderungen der Herzkammern im Laufe des Herzzyklus, ohne sich auf geometrische Annahmen verlassen zu müssen, wie es für die traditionelle 2D-Echokardiographie erforderlich ist. 4DUS ermöglicht die Berechnung von in vivo funktionellen Metriken aus der komplexen Form und Bewegung des Herzens 9,10, und MALDI MSI ermöglicht die räumliche Untersuchung biologischer Moleküle innerhalb des Herzgewebes ex vivo11. Um Veränderungen im Herzen mit Herz-Kreislauf-Erkrankungen vollständig zu verstehen, müssen sowohl mechanische als auch molekulare Mechanismen untersucht werden. Daher wird eine kombinierte Methodik zur Untersuchung der Pathophysiologie des Mausherzens vorgeschlagen, die die 4DUS-Bildgebung und -Analyse mit MALDI MSI von Lipiden im Herzen koppelt. Diese Methodik wird in einem murinen Modell des Myokardinfarkts demonstriert.
Die vaskuläre Biomechanik spielt auch eine entscheidende Rolle bei der Regulierung der kardiovaskulären Funktion2. Eine Gefäßversteifung, die mit dem Altern einhergeht, ist ein Risikofaktor für CVD12. Die biomechanischen und hämodynamischen Veränderungen der Gefäße können mit Ultraschall dargestellt werden. Die molekulare Zusammensetzung der Gefäßwände ist ein wichtiger Bestandteil der Biomechanik und auch äußerst empfindlich gegenüber hämodynamischen Kräften. Zum Beispiel wurde die oszillatorische Wandscherspannung mit der Entwicklung von atherosklerotischen Plaques in Verbindung gebracht3. Im Folgenden werden die vorläufigen Daten zur Gefäßmechanik und Hämodynamik bei gealterten Tieren vorgestellt.
Das Team interessiert sich für den Zusammenhang zwischen Biomechanik und molekularer Zusammensetzung in verschiedenen Krankheitszuständen. Die präklinische Ultraschallbildgebung und MSI werden eingesetzt, um die räumliche Verteilung von molekularen Veränderungen in einem Gewebe und die damit verbundenen biomechanischen Veränderungen, die während des Krankheitsverlaufs auftreten, zu bestimmen. In diesem Bericht werden diese Methoden ausführlich beschrieben und vorläufige Daten zum Herzen und zum Gefäßsystem des Kopfes/Halses vorgestellt.
Protokoll
Die beschriebenen Tierversuche werden mit Genehmigung des University of Tennessee, Knoxville Institutional Animal Care and Use Committee, durchgeführt.
1. Ultraschall13
- Aufbau und Tiervorbereitung
- Überprüfen Sie vor Beginn des Experiments den Isofluran-Anästhesiespiegel und füllen Sie ihn gegebenenfalls nach. Überprüfen Sie den Füllstand des Sauerstofftanks, bevor Sie das Gas einschalten. Wiegen Sie die Holzkohle und notieren Sie sie auf dem Behälter.
- Richten Sie den Imaging-Bereich ein, indem Sie Verbrauchsmaterialien in der Nähe der Imaging-Plattform platzieren. Zu den Verbrauchsmaterialien gehören vorgeschnittenes Klebeband, Enthaarungscreme, Gaze, Wattestäbchen, autoklaviertes Wasser, Hornhautschmiermittel, Gleitgel, rektale Temperatursonde, Elektrodenleitungen, Ultraschallgel, Elektrodengel (falls zutreffend) und Wärmelampe (Abbildung 1A).
- Schalten Sie das Ultraschallgerät ein und initialisieren Sie den Motor (das System ist auch in der Lage, 4DUS-Bilddaten zu sammeln). Richten Sie ein neues Experiment auf dem Ultraschallsystem ein: Wählen Sie Neue Studie oder Neue Serie und geben Sie die Mausinformationen ein. Schalten Sie das Tablet für die physiologische Überwachung ein.
- Überprüfen Sie den Absperrhahn, um sicherzustellen, dass der Durchfluss zum Einduktionskasten offen und zum Nasenkonus geschlossen ist. Füllen Sie die Anästhesie-Induktionsbox vor, indem Sie den Sauerstoff am Verdampfer auf 1 l/min einschalten und den Drehknopf auf 3%-5% drehen.
- Nehmen Sie die Maus aus dem Gehäusekäfig.
- Legen Sie die Maus in die Induktionsbox, bis das Tier vollständig mit Isofluran zwischen 3 % und 5 % betäubt ist.
- Schalten Sie den Absperrhahn um, um den Durchfluss zum Nasenkonus auf der Ultraschall-Speicherplatte zu ändern. Drehen Sie den Isofluran-Drehregler zwischen 1 % und 2 %.
- Nehmen Sie die Maus aus der Induktionsbox und wiegen Sie das Tier. Notieren Sie das Gewicht des Tieres.
- Platzieren Sie die Maus in Rückenlage auf der Speicherfolie.
- Tragen Sie Hornhautgleitmittel auf die Augen des Tieres auf.
- Wenn Fell vorhanden ist, entfernen Sie das Fell mit einer handelsüblichen Enthaarungscreme und Wattestäbchen aus dem Bildgebungsbereich. Wischen Sie die Creme mit Wasser auf Schwämmen von der Haut ab, um überschüssige Enthaarungscreme von der Haut zu entfernen und Verbrennungen zu vermeiden.
- Legen Sie die Elektrokardiographie-Ableitungen (EKG) subkutan auf beiden Seiten der Brust und der Hintergliedmaßen an, abhängig von der Positionierung des Tieres.
- Halten Sie die EKG-Ableitungen nach dem Einsetzen mit Klebeband fest. Wenn keine EKG-Ableitungen verwendet werden (d. h. EKG-Ableitungen nicht eingeführt werden), überwachen Sie das EKG über die tierische Speicherplatte.
- Für die elektrische Leitung von der Haut zur Platte platzieren Sie das leitende Gel auf der Platte und halten Sie die Gliedmaßen der Tiere in Position.
- Halten Sie die Gliedmaßen des Tieres entweder mit Klebeband oder einem Gummiband fest, das um die Platte und über den Gliedmaßen gelegt wird (Abbildung 1B).
- Verwenden Sie Gleitgel, um den rektalen Temperaturfühler einzuführen, und befestigen Sie den Fühler mit Klebeband.
- Überprüfen Sie EKG-, Atmungs- und Temperatursignale auf dem Tablet. Stellen Sie sicher, dass die Herzfrequenz ~350-600 Schläge pro Minute, die Atmung ~50-100 Atemzüge/min und die Rektaltemperatur zwischen 35 und 37 °C liegt. Passen Sie den Isofluranspiegel nach Bedarf an.
- Schalten Sie bei Bedarf eine Wärmelampe ein (z. B. eine 250-W-Infrarot-Wärmelampe)14 und stellen Sie die Höhe ein, um die Kerntemperatur zu halten (Abbildung 1C).
- Platzieren Sie die Wärmelampe nicht näher als 12 Zoll vom Tier entfernt. Für erste Untersuchungen zur Bestimmung des optimalen Abstands zwischen Zwiebel und Tier stellen Sie die Höhe ein und erfassen Sie die Hauttemperatur mit einem Infrarot-Thermometer, die 42 °C15 nicht überschreiten sollte.
- Kardialer 4D-Ultraschall
- Setzen Sie den Wandler in einer halb verriegelten Position in die Halterung ein.
- Richten Sie den erhabenen Punkt auf dem Wandler so aus, dass der blaue Punkt auf dem Bildschirm zu sehen ist. Die Konvention besteht darin, den Punkt auf der rechten Seite des Tieres zu platzieren.
- Drehen Sie den Schallkopf so, dass er entlang der Sagittalebene der Maus ausgerichtet ist, mit einer erhabenen Kerbe, die in kaudaler Richtung zeigt.
- Betätigen Sie den Hebel an der Unterseite der Ultraschallplatte, um das Tier zu kippen. Verwenden Sie den Hebel an der Unterseite des Schallkopfs, um den Winkel des Schallkopfs einzustellen.
- Tragen Sie eine großzügige Menge Ultraschallgel auf die ventrale Oberfläche der Brusthöhle auf, um eine akustische Kopplung zwischen der Hautoberfläche und dem Schallkopf zu gewährleisten. Stellen Sie sicher, dass vor dem Ankuppeln kein Fell übrig bleibt.
- Senken Sie den Schallkopf ab, um Kontakt mit dem Ultraschallgel aufzunehmen.
- Nehmen Sie Mikroeinstellungen mit den X/Y-Knöpfen an der Unterseite der Platte vor oder bewegen Sie die gesamte Platte für große Einstellungen.
- Stellen Sie sicher, dass eine parasternale Längsachsenansicht die Spitze, den linksventrikulären Ausflusstrakt und die Aorta horizontal auf dem Bildschirm ausrichtet, um ein genaueres Bild der kurzen Achse zu erhalten (Abbildung 2A, B).
- Wählen Sie in der unteren Ecke Bild benennen aus, um das Bild in der aktuellen Serie zu speichern.
- Drehen Sie den Schallkopf um 90° im Uhrzeigersinn, um in eine parasternale Kurzachsenansicht zu wechseln (Abbildung 2C).
HINWEIS: Für eine optimale 4D-Bildqualität sollten Benutzer versuchen, den Schrittmotor entlang der parasternalen Längsachse des Ventrikels auszurichten, die nicht immer direkt parallel zum Brustbein verläuft. Das Herz/LV sitzt oft in einem linken Abwärtswinkel. - Stellen Sie sicher, dass der linke Ventrikel auf der rechten Seite des Bildschirms und der rechte Ventrikel auf der linken Seite des Bildschirms sichtbar ist (Abbildung 2D).
- Wenn die Maus einen stabilen Atemrhythmus hat, klicken Sie auf das EKG-Feld in der oberen linken Ecke des Bildes, um das Atmungs-Gating für das 4D-Bild zu aktivieren. Wenn es nicht stabil ist, führen Sie diesen Schritt während der Nachbearbeitung aus.
- Wählen Sie den Würfel in der oberen linken Ecke des Bildschirms aus, um ein 4D-Bild einzurichten.
- Setzen Sie den Geber zurück, bevor Sie die Start - und Stopppositionen des Wandlers einstellen.
- Stellen Sie die Startposition knapp unter dem Scheitelpunkt und die Stoppposition auf den Aortenbogen ein.
- Stellen Sie die Schrittweite auf 0,08 bis 0,13 mm und die Bildrate auf 200 bis 300 Hz ein.
HINWEIS: Kleinere Schrittgrößen bieten möglicherweise eine bessere Bildauflösung für die Analyse, verlängern jedoch die Erfassungszeit. Größere Schrittweiten sind akzeptabel, wenn die Daten mit nahezu perfekter linksventrikulärer Ausrichtung erhoben werden und während der Analyse nur eine minimale/keine Neuausrichtung erforderlich ist. Erwägen Sie, die Bildrate zu erhöhen, wenn Sie mit Tieren mit höheren Herzfrequenzen (>450 Schläge pro Minute) arbeiten. - Stellen Sie sicher, dass die Vitalparameter und das EKG-Signal konstant stabil sind (>350 Schläge pro Minute und Atmung über 50 bzw. min), bevor Sie mit dem Scan beginnen.
- Sobald der Scan und die Verarbeitung abgeschlossen sind, aktivieren Sie EKV/4D-Daten für Nachbearbeitung und Atmungskontrolle speichern.
- Wählen Sie unten rechts Namensbild aus und fügen Sie die Maus-ID in den Namen ein.
- Um die Bildqualität zu überprüfen, klicken Sie auf Weitere Steuerelemente und wählen Sie In 4D laden.
- Überprüfen Sie jede Ebenenansicht des Herzens und stellen Sie sicher, dass sich das Zentrum des Herzens während des gesamten Herzzyklus nicht bewegt. Die Bewegung des Zentrums zeigt eine Variabilität im EKG und/oder im Respiratory Gating an und erschwert somit den Analyseprozess. Stellen Sie die Monitore neu ein und wiederholen Sie den Scan, falls erforderlich.
- Kardiale 4D-Ultraschallanalyse
- Navigieren Sie zum Studienbrowser. Exportieren Sie alle 2D-Bilder im B-Modus nach Vevo Lab und exportieren Sie 4D-Daten als anderen Dateityp, indem Sie das Datenformat ".raw" auf eine Festplatte auswählen.
HINWEIS: Die in dieser Arbeit verwendete 4D Toolbox für die grafische Benutzeroberfläche ist derzeit nicht öffentlich verfügbar. Um Zugang und detaillierte Anweisungen zu erhalten, wenden Sie sich bitte an Dr. Craig Goergen, Purdue University. - Führen Sie eine alternative Analyse von 4D-Daten mit der VevoLab-Software durch. Um dieses Framework zu verwenden, exportieren Sie 4D-Daten von der Maschine in Vevo Lab-Dateien.
- Navigieren Sie zum Studienbrowser. Exportieren Sie alle 2D-Bilder im B-Modus nach Vevo Lab und exportieren Sie 4D-Daten als anderen Dateityp, indem Sie das Datenformat ".raw" auf eine Festplatte auswählen.
- Vaskuläre Bildgebung
- Für B-Modus-Bilder der Halsschlagadern platzieren Sie den Ultraschallschallkopf parallel zu den Halsschlagadern in der Nähe der Mittellinie.
- Sobald die Halsschlagader sichtbar ist, bewegen Sie den Schallkopf nach oben, um die Halsschlagadergabelung zu finden.
- Wenn ein klares Bild der Karotisgabelung gefunden wird, passen Sie die Verstärkungseinstellung auf 35 dB an und nehmen Sie das B-Modus-Bild auf.
- Wenn Sie den Schallkopf nach links oder rechts neigen, kann das Bild verbessert werden. Stellen Sie den Winkel des Schallkopfs oder Tisches so ein, dass das betreffende Gefäß nicht direkt parallel zum Schallkopf ist. Dadurch kann der Dopplerwinkel unter 60° bleiben. Andernfalls kann eine Verschiebung von +/- 15° erforderlich sein.
- Wechseln Sie in den gepulsten Doppler-Modus , um die Geschwindigkeitsmessungen zu erhalten.
- Platzieren Sie das Probenvolumen in der Mitte des Gefäßes. Stellen Sie den Winkel des Cursors so ein, dass er parallel zur Halsschlagader verläuft. Drücken Sie dann auf Play, um die Geschwindigkeitsmessungen zu starten, und speichern Sie , um sie aufzuzeichnen.
- Für das B-Mode-Bild der Jugularvenen platzieren Sie den Ultraschallschallkopf parallel zu den Jugularvenen. Die Halsvenen befinden sich vorne und seitlich der Halsschlagadern.
- Sobald sich die Vena jugularis lokalisiert hat, bewegen Sie den Schallkopf an die Stelle, an der die innere und die äußere Halsvene zusammenlaufen. Wenn ein klares Bild gefunden wird, stellen Sie die Verstärkungseinstellung auf 35 dB ein und nehmen Sie das Bild im B-Modus auf.
- Wechseln Sie in den Impulse-Doppler-Modus , um die Geschwindigkeitsmessungen zu erhalten.
- Platzieren Sie das Probenvolumen in der Mitte des Gefäßes. Stellen Sie den Cursorwinkel so ein, dass er parallel zur Halsvene verläuft. Drücken Sie dann auf Play, um die Geschwindigkeitsmessungen zu starten, und speichern Sie , um sie aufzuzeichnen.
- Vaskuläre Ultraschallanalyse
- Messen Sie nach dem Sammeln der B-Mode-Bilder und der Pulswellen-Doppler-Messungen die Durchmesser und Geschwindigkeiten mit der Software.
- Sobald die Bilder in die Software geladen sind, wählen Sie das Gefäßpaket aus.
- Wählen Sie jede Seite des Gefäßes aus, um den vollen Durchmesser zu erhalten.
- Führen Sie für die Halsschlagader Durchmessermessungen in Systole und Diastole mit der Durchmesseroption in der Gefäßverpackung durch.
- Führen Sie Geschwindigkeitsmessungen mit der Geschwindigkeitsoption im Gefäßpaket durch.
- Wählen Sie für die Halsschlagader den höchsten Peak für die Geschwindigkeitsmessung in der Systole und den niedrigsten Peak für die Geschwindigkeitsmessung in der Diastole.
- Verwenden Sie die Werte der Durchmesser, um die Umfangskomponente des Dehnungstensors Green Lagrange mit der folgenden Gleichung zu berechnen:
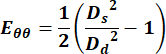 (1)
(1)
wobei Ds den Durchmesser während der Systole und Dd den Durchmesser während der Diastole darstellt. - Wählen Sie für die Vena jugularis beliebige Punkte im Zyklus aus, um die Geschwindigkeitsmessung zu erhalten.
2. Euthanasie und Gewebeentnahme
- Bereiten Sie Aluminiumfolienschiffchen für das Schockfrosten von Gewebe vor (Abbildung 3).
- Am Ende einer Studie wird das Tier durch Isofluran-Überdosierung in einer Konzentration von 5 % und entweder durch beidseitigen Pneumothorax oder Zervixluxation eingeschläfert (nur Herzstudie).
- Schneiden Sie die Haut mit einer Pinzette mit einer Schere über dem Hals für das Gefäßsystem oder direkt unter dem Brustbein für das Herzgewebe ab.
- Schneiden Sie die Haut- und Muskelschichten, um das Gefäßsystem freizulegen, oder schneiden Sie durch den Knochen, um das Herz freizulegen.
- Isolieren Sie das Herz oder das Gefäßsystem durch stumpfes Sezieren mit Wattestäbchen vom umgebenden Gewebe, einschließlich Fett. Achten Sie darauf, das Halsschlagadergefäß vom Nerv zu trennen. Entfernen Sie das Herz und die Gefäße mit chirurgischen Instrumenten.
HINWEIS: Nähte können verwendet werden, um Gefäße zu entfernen, indem proximal und distal genäht wird, bevor die Gefäße entfernt werden. - Legen Sie das Taschentuch auf ein vorbeschriftetes Alufolienboot und legen Sie das Boot in flüssigen Stickstoff (Abbildung 3).
- Lagern Sie das Gewebe bei -80 °C bis zur Kryosektion. Transportieren Sie das Gewebe mit Trockeneis, um die Temperatur zu halten.
3. Massenspektrometrische Bildgebung
- Kryosektion und Montage der optimalen Schnitttemperatur (OCT) auf Objektträgern
- Stellen Sie die Kryostatentemperatur auf -25 °C ein und setzen Sie die Klinge ein.
- Bereiten Sie ein Metallfutter vor, indem Sie OCT auftragen und es im Kryostaten einfrieren lassen.
- Befestigen Sie die Basis des Herzens mit OCT an der vorbereiteten Metallspannplatte (Abbildung 4A). Lassen Sie die OCT nicht mit dem interessierenden Bereich der Probe in Berührung kommen, da die OCT die Massenspektren verunreinigt (Kontamination mit Polyethylenglykol [PEG]) und die interessierenden Lipidsignale unterdrückt.
HINWEIS: Option zur Wassermontage, um eine Kontamination der Probe zu vermeiden. Anweisungen finden Sie im zusätzlichen Protokoll unten. - Basierend auf der Bedeutung der räumlichen Ausrichtung in dieser Methode verwenden Sie die skalierte digitale Darstellung des Herzens, die von der MATLAB-GUI bereitgestellt wird, um die Schnitterstellung zu leiten (Abbildung 4B).
- Zählen Sie jede Umdrehung (10 μm) während des Schneidens und notieren Sie die Tiefe in Millimetern für jede Schnittscheibe, die auf den Objektträger montiert werden soll.
HINWEIS: Es ist notwendig, bestimmte Stellen im Herzen oder Gefäßsystem zu verfolgen, um eine genaue Übereinstimmung mit der digitalen Darstellung der von MATLAB bereitgestellten Ultraschalldaten zu gewährleisten. - Auftauen von Gewebeschnitten (10 μm dick) auf Objektträger.
HINWEIS: Die Art der Objektträger hängt vom verwendeten Massenspektrometer ab. Jede Folie enthält einen Abschnitt aus jeder Region des Herzens. Es sind mindestens drei Objektträger erforderlich (positiver Ionenmodus, negativer Ionenmodus, extra). - Für n = 1/Gruppe wird das Herz über die lange Achse geschnitten, um molekulare Veränderungen von der Spitze zur Basis sichtbar zu machen.
- Für das Gefäßsystem das Gewebe in Gelatine einbetten und vor dem Schnitt11 schockfrosten.
- Lagern Sie die Objektträger in Objektträger-Mailern bei -80 °C bis zu massenspektrometrischen Bildgebungsexperimenten.
- Wenn Sie eine Flüssigkeitschromatographie-Massenspektrometrie (LC-MS) zur weiteren Identifizierung und Quantifizierung von Lipiden oder Metaboliten durchführen, schneiden Sie serielle Gewebeschnitte von ~60-100 μm und sammeln Sie sie vor dem Einfrieren in 0,5-2 ml-Röhrchen.
- Kryosektionen und Wassereinbetten von Gewebe auf Objektträgern
- Füllen Sie ein Becherglas mit Hochleistungsflüssigkeitschromatographie (HPLC) Wasser und stellen Sie es mit 5 mL und 1 mL Spritzen beiseite.
- Stellen Sie die Kryostatentemperatur auf -25 °C ein und setzen Sie die Klinge ein.
- Legen Sie eine Pinzette in den Kryostaten, um sie vor der Montage abzukühlen.
- Ziehen Sie 5 mL HPLC-Wasser in eine Spritze und geben Sie es in den Kryostaten.
- Kurz bevor die Spritze vollständig gefroren ist, leeren Sie das Wasser auf das Metallfutter und lassen Sie es vollständig einfrieren.
- Nehmen Sie 1 ml HPLC-Wasser auf und legen Sie die Spritze in den Kryostaten.
- Nach ca. 30-60 s geben Sie einen kleinen Punkt teilweise verfestigten Wassers auf die Mitte des Spannfutters. Fassen Sie das Herz sofort mit einer Pinzette und platzieren Sie es in den Punkt, bevor es vollständig einfriert. Halten Sie das Herz an Ort und Stelle, bis das umgebende Wasser vollständig gefroren ist.
HINWEIS: Das Herz kann je nach interessierender Region an der Spitze oder an der Basis montiert werden. Für diese Studie wird der Sockel mit dem Scheitelpunkt nach außen montiert. - Führen Sie die restlichen Schritte für diese Methode wie in Schritt 3.1 beschrieben aus.
- Matrix-Anwendung
- Nehmen Sie den Objektträger aus dem Gefrierschrank und legen Sie ihn in einen Exsikkator, bis der Objektträger trocken ist.
- Schalten Sie das HTX M3+ Sprühgerät ein, öffnen Sie die HTX-App auf dem Laptop und wählen Sie Methode in der Mitte des Bildschirms. Standard-Matrixkonzentrationen finden Sie in den gespeicherten Methoden (Methoden auf der linken Seite des Bildschirms > OI_usermethods > Matrix).
- Stellen Sie dazu die Düsentemperatur auf 75 °C, die Durchflussmenge auf 100 μl/min und den Druck auf 10 psi ein.
- Notieren Sie den Probennamen, die Polarität, die Matrix, das Lösungsmittel und die Konzentration in das Labornotizbuch. Berechnen Sie die benötigte Matrixmenge pro Konzentration (z. B. 5 ml bei 10 mg/ml = 50 mg Matrix).
- Stellen Sie ein Lösungsmittel her (z. B. 70 % MeOH).
- Für diese Arbeit wird 40 mg/ml 2,5-Dihydroxybenzoesäure (DHB)-Matrixlösung für den positiven Modus und 10 mg/ml 9-Aminoacridin (9AA)-Matrixlösung für den negativen Modus hergestellt. Stellen Sie beide Matrizen mit einem 70%igen MeOH-Lösungsmittel her. Für andere gängige Matrizen und Sprühparameter befolgen Sie bitte die Schritte 3.3.2.
- Wiegen Sie die Matrize und geben Sie die Matrize in ein konisches 15-ml-Röhrchen. Stellen Sie sicher, dass der Betrag in der Nähe des in Schritt 3.3.3 berechneten erforderlichen Betrags liegt, aber nicht exakt sein muss. Berechnen Sie die Lösungsmittelmenge basierend auf der gemessenen Masse.
- Geben Sie mit einer Pipette Lösungsmittel in das konische Röhrchen.
HINWEIS: Das Volumen basiert auf der in Schritt 3.3.5 gemessenen Masse. - Die Mischung 10 Minuten lang beschallen. Während die Matrize beschallt wird, nehmen Sie den Objektträger aus dem Exsikkator.
- Öffnen Sie die Sprühschale, setzen Sie die Folie in die untere linke Ecke und kleben Sie die Kanten fest.
HINWEIS: Falten Sie ein Ende des Klebebandes, um es leicht zu entfernen. - Wählen Sie den Sprühbereich der Probe aus. Schließen Sie das Fach.
- Verwenden Sie eine Spritze und einen Filter und gießen Sie die Matrize in die Spritze, dann filtrieren Sie die Matrize durch die Spritze in das Fläschchen mit dem schwarzen Deckel auf der linken Seite des Sprühgeräts.
- Setzen Sie das Fläschchen wieder an seinen Platz auf dem Sprühgerät ein und führen Sie den D-Leitungsschlauch in das Fläschchen ein.
HINWEIS: Stellen Sie sicher, dass das Röhrchen den Boden des Fläschchens nicht berührt und vollständig in die Flüssigkeit eingetaucht ist. - Schalten Sie das Inertgas ein und stellen Sie sicher, dass das Messgerät am Sprühgerät 10 psi anzeigt. Drücken Sie Start. Sobald das Sprühgerät die gewünschte Temperatur erreicht hat, wählen Sie den Blinkstart.
- Öffnen Sie nach Beendigung des Sprühvorgangs das Tablett, entnehmen Sie die Probe und legen Sie sie in den MALDI-Objektträgerhalter oder zurück in den Exsikkator (Abbildung 4C).
- Scannen Sie den MALDI-Diahalter und schieben Sie ihn auf den Scanner oder machen Sie ein Foto mit einem Telefon. Speichern Sie das Bild auf einem Flash-Laufwerk für die Verwendung mit MSI.
- Wählen Sie die passende Wäsche oder den nächsten Sprühstoß auf das Sprühgerät. Schieben Sie die D-Linie beim Waschen aus dem Matrixfläschchen in das Abfallbecherglas.
- Sprühen Sie Methanol auf die Schale und wischen Sie sie zur Reinigung ab. Schalten Sie den Stickstoff aus.
- MALDI MSI
- Navigieren Sie auf dem Computer zu Synapt, überprüfen Sie, ob die Polarität in den gewünschten Modus geändert wurde, und schalten Sie um, wenn nicht.
- Laden Sie den Objektträger in das Gerät auf die MALDI-Quelle und klicken Sie mit der Software auf die Schaltfläche "Laden " auf dem Computer.
- Verschieben Sie das Bild (Schritt 1.2.17) in den Ordner Bilder im Projektordner.
- Navigieren Sie zu HD Imaging und öffnen Sie das vorherige Projekt (das dieselbe Matrix verwendet).
- Erfassen Sie in einem Labornotebook den Massenbereich, die Kollisionsenergie der Falle und Übertragung, die Laserenergie und den Neg/Pos-Modus.
- Navigieren Sie in HD Imaging zum Symbol für die weiße Seite. Klicken Sie dann auf den Dropdown-Pfeil und wählen Sie Neue Platte aus.
- Suchen Sie das Bild im Projektordner > im Ordner Bilder und öffnen Sie es.
- Ändern Sie den Plattentyp in MALDI Standard und definieren Sie die Kanten der Platte (wählen Sie 4 Ecken, die unten mit roten Pluszeichen angezeigt werden).
HINWEIS: Das obere linke Feld dient zum Navigieren zu den einzelnen Ecken. Das obere rechte Feld dient zur Auswahl der Ecken. Das untere Feld dient nur zur Visualisierung der gesamten Folie. - Navigieren Sie zum kleinen Muster-Tab in der oberen linken Ecke und klicken Sie auf Bleistift oder andere Rechteck- oder Kreisoptionen, um Gewebe zu umreißen. Umreißen Sie das Gewebe mit Linksklicks und klicken Sie mit der rechten Maustaste, um den Vorgang abzuschließen.
- Die Pixelgröße beträgt standardmäßig 50 μm. Bei Bedarf wechseln.
- Klicken Sie auf Speichern unter , navigieren Sie zu Project Imaging AcqFolder und beschriften Sie als Folienname (Format JJJJMMTT). Speichern Sie dann.
- Klicken Sie auf die Schaltfläche Mass Lynx in der oberen Mitte, um nach Mass Lynx zu exportieren.
- Navigieren Sie zur Mass Lynx-Software.
- Klicken Sie auf Datei > öffnen Sie den Projektordner. Bearbeiten Sie den Dateinamen und den Textnamen auf der Folie (Format JJJJMMTT).
- Klicken Sie mit der rechten Maustaste auf die MS-Datei, durchsuchen Sie die aktuelle Folie und wählen Sie sie aus.
- Klicken Sie erneut mit der rechten Maustaste auf die MS-Datei und klicken Sie auf Bearbeiten. Doppelklicken Sie mit der linken Maustaste auf das blaue Feld.
- Navigieren Sie zu den Scanbedingungen. Suchen Sie die Quelleinstellungen, und ändern Sie sie zu benutzerdefiniert. Navigieren Sie dann zum richtigen Projektordner > abgerufenen Ordner > aktuellen Folie > OKAY.
- Speichern Sie, indem Sie auf Speichern unter klicken, und fügen Sie den Namen der Folie ein.
- Kalibrieren Sie das Gerät mit einer Referenzverbindung (z. B. rotem Phosphor)
- Navigieren Sie zu Synapt (Kalibrieren). Klicken Sie auf die Wiedergabetaste , um mit der Datenerfassung zu beginnen. Etikettieren Sie als YYYYMMDD_cal1 (z. B. 20230614_cal1).
- Stellen Sie sicher, dass es den gleichen Massenbereich von 50 bis 2000 hat. Die Laserenergie kann etwa 175 betragen.
- Drücken Sie auf Fire Laser Play und klicken Sie dann auf die Schaltfläche Weiter . Lassen Sie es bis etwa 15 oder 20 laufen. Klicken Sie auf roter Stopp.
- Navigieren Sie zu W Console.
- Erstellen Sie eine Kalibrierung und klicken Sie auf Start. Klicken Sie unter Kalibrierungsprofil-Editor auf Datei > Neu. Geben Sie YYYYMMDD_cal1 ein.
- Bearbeiten Sie den Massenbereich auf den gewünschten Massenbereich für das Projekt (z. B. 50-2000).
- Wählen Sie die manuelle Kalibrierung. Wählen Sie den Auflösungsmodus und klicken Sie auf Bearbeiten. Wählen Sie roten Phosphor als Referenzverbindung.
- Klicken Sie auf die Rohdatendatei und scrollen Sie ganz nach unten (es sollte sich um das handeln, was gerade abgerufen wurde).
- Gehen Sie unten zum Verlauf, wählen Sie Acc mass > okay > okay aus.
- Klicken Sie weiter auf Weiter und OK , bis die Startschaltfläche angezeigt wird, und klicken Sie auf Start.
- Navigieren Sie zum grünen Häkchen und sehen Sie sich an, welche Peaks nicht gefunden wurden.
- Navigieren Sie zu Mass Lynx (Starten Sie den eigentlichen Lauf für experimentelle Proben).
- Klicken Sie auf die Play-Taste und überprüfen Sie, ob die Anzahl der Samples korrekt ist
- Navigieren Sie zu HD-Bildgebung.
- Stellen Sie sicher, dass die Pixelgröße immer noch der ursprünglichen Einstellung entspricht
- Navigieren Sie zu Synapt, um die Polarität zu überprüfen.
- Klicken Sie auf die Kameraschaltfläche , um sicherzustellen, dass sie sich bewegt und den Laser abfeuert.
- Überprüfen Sie, ob Daten erfasst werden. Das erfasste Massenspektrum sollte auf der Synapt-Seite angezeigt werden.
- MS-Analyse
- Verwenden Sie die HD-Imaging-Software und Mass Lynx für die Analyse.
- Verarbeiten Sie Bilddaten über die Registerkarte "Prozess" in HD Imaging.
- Visualisieren Sie Bilder von molekularen Ionen, auch MS-Bilder genannt, in HD Imaging auf der Registerkarte Analyse . Eine Heatmap eines molekularen Ionenbildes zeigt die Intensität dieses m/z-Ions über einer Region (Abbildung 5). Verwenden Sie die Überlagerungen von MS-Bildern, um die räumliche Verteilung von Molekülen darzustellen (Abbildung 6 und Abbildung 7).
- Normalisieren Sie Daten mit TIC oder einer geeigneten Normalisierung für die Bildgebung.
- Identifizieren Sie m/z-Peaks, die räumlich mit den interessierenden Regionen korrelieren. Führen Sie eine räumliche Korrelation (R > 0,65) für HD Imaging durch, um kolokalisierte Peaks zu identifizieren.
- Suche nach einer möglichen Lipididentifikation dieser Peaks mit LipidMaps16 und METASPACE17.
- Bestätigen Sie die Lipidannotationen mit nachfolgenden LC-MS-Daten. Verwenden Sie Metaboanalyst für die statistische Analyse von LC-MS-Daten.
Ergebnisse
Die oben beschriebenen bildgebenden Verfahren wurden für zwei Vorstudien verwendet: Myokardinfarkt-Umbau (MI) und Gefäßalterung. Für die kardialen Experimente wurde eine permanente Koronararterien-Ligatur-Operation durchgeführt, um einen akuten Myokardinfarkt zu induzieren18,19. 4D-Ultraschall und MALDI MSI wurden schrittweise am selben Gewebe durchgeführt, wodurch physiologische und molekulare Veränderungen sichtbar wurde...
Diskussion
Die Bildgebung in den USA kann vom Bediener abhängen, aber die Verwendung anatomischer Orientierungspunkte und eine angemessene Schulung können die Verzerrung des Benutzers begrenzen. 2D-Ultraschall ist besonders anfällig für Variabilität zwischen den Benutzern, da die Ansichten winkelabhängig sind, während 4DUS weniger anfällig ist, da die Erfassung das gesamte Volumen umfasst und winkelunabhängig ist. Es wurde auch festgestellt, dass die Bildreproduzierbarkeit aufgrund der ver...
Offenlegungen
Craig J. Goergen ist ein bezahlter Berater von FUJIFILM VisualSonics.
Danksagungen
Allison Jones wird unterstützt durch das Graduiertenstipendium des University of Tennessee, Mechanical, Aerospace & Biomedical Engineering Department. Die in dieser Veröffentlichung (Conner Earl) berichtete Forschung wurde vom National Heart, Lung, and Blood Institute der National Institutes of Health F30HL162452 unterstützt. Der Inhalt liegt ausschließlich in der Verantwortung der Autoren und gibt nicht unbedingt die offizielle Meinung der National Institutes of Health wieder.
Materialien
| Name | Company | Catalog Number | Comments |
| 2,5-dihydroxybenzoic acid (DHB) | Supelco, >99.0% (HPLC) | 85707-10MG-F | DHB matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sigma/85707?cm_sp=Insite-_-wimsShippingEmailRecs_wims EmailAPI_wimsGruCrossEntropy-_-wimsEmailAPI10-3 |
| 9-aminoacridine (9AA) | Supelco, ≥99.5% (HPLC) | 92817-1G | 9-Aminoacridine matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sial/92817?srsltid=AfmBOooiQjQ4pWv_XxITkU 4Lkm0UnHXKekGS_ dFl7V40V9QLWoPpNLoc |
| Aquasonic Ultrasound Gel | Parker Laboratories | Parker 01-02 | Ultrasound Gel; https://www.parkerlabs.com/products/aquasonic-100-ultrasound-transmission-gel/ |
| Benchtop Dewar Flasks | ThermoScientific | 4150-2000 | Container for liquid nitrogen; https://www.thermofisher.com/order/catalog/product/4150-4000?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sQVc2f-NxN04Nb5Mv F6TZ7GLHWWDEeqDYmEvtKJSQ YHDeVgZ9qylvYaAs27EALw_wcB &source=google_shopping&ISO_ CODE=us&LANG_CODE=en&ef_id =Cj0KCQjwpvK4BhDUARIsADHt9 sQVc2f-NxN04Nb5MvF6TZ7GLHWWDE eqDYmEvtKJSQYHDeVgZ9qylvYa As27EALw_wcB:G:s&s_kwcid=AL!3652 !3!716188292869!!!g!2366243726129 !!21787513085!171591181194&ev_chn =shop&cid=0se_gaw_30092024_ PBYTXL&source=google_shopping &ISO_CODE=us&LANG_CODE= en&gad_source=1 |
| Cryostat | Leica Biosystems | CM Series | https://www.leicabiosystems.com/us/histology-equipment/cryostats/ |
| Dessicator | VWR | 89054-052 | https://us.vwr.com/store/product/9104882/desiccator-plastic-ace-glass-incorporated |
| Epredia MX35 Premier Disposable Low-profile Microtome Blades | Fisher Scientific | 3052835 | Cryostat blade; https://www.fishersci.com/shop/products/mx35-premier-disposable-low-profile-microtome-blades/3052835 |
| Falcon 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | Conical Tubes; https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/1495953A?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sSBcy5n-lhShligJUOX5KKVGn0bt87 8AB2_muOD2PPTue1phpZgeqwa AqgiEALw_wcB&ef_id=Cj0KCQjw pvK4BhDUARIsADHt9sSBcy5n-lhS hligJUOX5KKVGn0bt878AB2_muO D2PPTue1phpZgeqwaAqgiEALw_ wcB:G:s&ppc_id=PLA_goog_20861 45674_81843405034_1495953A__ 386247001345_165426395473886 37329&ev_chn=shop&s_kwcid=AL!4428!3 !386247001345!!!g!856907751004!& gad_source=1 |
| Flex-Tubes Microcentrifuge Tubes | Eppendorf | EP022364120 | Centrifuge tubes; https://www.sigmaaldrich.com/US/en/product/sigma/ep022364120?utm_source=google%2Cgoogle&utm _medium=organicshopping%2Ccpc&utm _campaign=21043330280&utm_ content=&gclid=Cj0KCQjwpv K4BhDUARIsADHt9sTHRD35k CHPtfI2A41axodnMVr6a1eBKk zM4bSUYQAyfEKo3UgTAEQa Ap7wEALw_wcB |
| Gas Nitrogen | Airgas | N/A | |
| Glass microscope slides | Electron Microscopy Sciences | 71873-02 | https://www.emsdiasum.com/positive-charge-microscope-slides |
| Liquid Nitrogen | Airgas | N/A | |
| Mass Spectrometer | Waters | Synapt G2-Si | https://www.waters.com/waters/en_US/SYNAPT-G2-Si-Mass-Spectrometry/nav.htm?locale=en_mkcid=1000251Foodety%3C/a%3E&cid=134740653&bcid= 134528734 |
| Matrix Sprayer | HTX Technologies | M3+ | https://www.htximaging.com/htx-m3-sprayer |
| Methanol (HPLC), >99.9% | Fisher Chemical | A4524 | Methanol; https://www.fishersci.com/shop/products/methanol-hplc-fisher-chemical-9/A4524?crossRef=A4524#?keyword=A4524 |
| Preclinical Ultrasound System | FUJIFILM VisualSonics | Vevo 3100 | https://www.visualsonics.com/product/imaging-systems/vevo-3100; Vevo F2 has replaced the Vevo 3100 in production. System includes isoflurane vaporizer and induction box. |
| Reynolds Wrap | N/A | N/A | Aluminum foil |
| Signagel Electrode Gel | Parker Laboratories | Parker 15-60 | Electrode Conducting Gel; https://www.parkerlabs.com/products/signagel-electrode-gel/ |
| Sterile Lubricating Jelly | Medline | MDS032273Z | Lubricating Gel; https://www.medline.com/ce/product/Sterile-Lubricating-Jelly/Lubricating-Jelly/Z05-PF03664?sku=MDPMDS032273H |
| Surgical instruments: scissors, forceps/tweezers, sutures | Fine Science Tools | 11252-00, 11050-10, 14016-14, 14084-08, 15000-08 | info@finescience.com |
| Surgical Sponges 200 Pack –Gauze Pads Non sterile -First Aid Wound Care Dressing Sponge –Woven Medical Nonstick, Non Adherent Mesh Scrubbing | Medpride | B08RZGQ5GW | Gauze; https://www.amazon.com/Medpride-Surgical-Sponges-200-Pack/dp/B08RZGQ5GW/ref=asc_df_B08RZGQ5GW/?tag=hyprod-20&linkCode=df0&h vadid=693270340506&hvpos= &hvnetw=g&hvrand=960915122 2290977669&hvpone=&hvptwo= &hvqmt=&hvdev=c&hvdvcmdl=& hvlocint=&hvlocphy=9192978&hv targid=pla-1245491514869&psc= 1&mcid=33f4d647c88630c79116 888d565a63b0 |
| Tissue-Plus O.C.T. Compound | Fisher Scientific | 23-730-571 | OCT; https://www.fishersci.com/shop/products/tissue-plus-o-c-t-compound-2/23730571 |
| Wood Handled Cotton Swabs and Applicators | Fisherbrand | 22-363-160 | Cotton swab; https://www.fishersci.com/shop/products/wood-handled-cotton-swabs-applicators-8/p-7146852 |
Referenzen
- Tsao, C. W., et al. Heart disease and stroke statistics-2022 update: A report from the American Heart Association. Circulation. 145 (8), E153-E639 (2022).
- Kassab, G. S. Biomechanics of the cardiovascular system: The aorta as an illustratory example. J R Soc Interface. 3 (11), 719-740 (2006).
- Moore, J. E., Xu, C., Glagov, S., Zarins, C. K., Ku, D. N. Fluid wall shear stress measurements in a model of the human abdominal aorta: Oscillatory behavior and relationship to atherosclerosis. Atherosclerosis. 110 (2), 225-240 (1994).
- Nerem, R. M. Vascular fluid mechanics, the arterial wall, and atherosclerosis. J Biomech Eng. 114 (3), 274 (1992).
- Cecchi, E., et al. Role of hemodynamic shear stress in cardiovascular disease. Atherosclerosis. 214 (2), 249-256 (2011).
- Good, B. The influence of blood composition and loading conditions on the behavior of embolus analogs. J Mech Behav Biomed Mater. 140, 105738 (2023).
- Bao, G., et al. Molecular biomechanics: The molecular basis of how forces regulate cellular function NIH public access. Mol Cell Biomech. 3 (2), 91-105 (2010).
- Schwamborn, K., Caprioli, R. M. Molecular imaging by mass spectrometry-looking beyond classical histology. Nat Rev Cancer. 10 (9), 639-646 (2010).
- Soepriatna, A. H., Kevin Yeh, A., Clifford, A. D., Bezci, S. E., O'Connell, G. D., Goergen, C. J. Three-dimensional myocardial strain correlates with murine left ventricular remodelling severity post-infarction. J R Soc Interface. 16 (160), 20190570 (2019).
- Damen, F. W., et al. High-frequency 4-dimensional ultrasound (4DUS): A reliable method for assessing murine cardiac function. Tomography. 3 (4), 180-187 (2017).
- McDonald, R., Poulos, D., Gutzwiller, L., Sheth, R. A., Good, B., Crouch, A. C. A MALDI mass spectrometry imaging sample preparation method for venous thrombosis with initial lipid characterization of lab made and murine clots. J Am Soc Mass Spectrom. 34 (9), 1879-1889 (2023).
- Benetos, A., et al. Influence of age, risk factors, and cardiovascular and renal disease on arterial stiffness: Clinical applications. Am J Hypertens. 15 (12), 1101-1108 (2002).
- Pistner, A., Belmonte, S., Coulthard, T., Blaxall, B. C. Murine echocardiography and ultrasound imaging. J Vis Exp. (42), v2100 (2010).
- Bellantuono, I., et al. A toolbox for the longitudinal assessment of healthspan in aging mice. Nat Protoc. 15 (2), 540-574 (2020).
- Henriques, F. C., Moritz, A. R. Studies of thermal injury: I. The conduction of heat to and through skin and the temperatures attained therein. A theoretical and an experimental investigation. Am J Pathol. 23 (4), 530 (1947).
- Conroy, M. J., et al. LIPID MAPS: Update to databases and tools for the lipidomics community. Nucleic Acids Res. 52 (D1), D1677-D1682 (2024).
- Palmer, A., et al. FDR-controlled metabolite annotation for high-resolution imaging mass spectrometry. Nat Methods. 14 (1), 57-60 (2016).
- Michael, L. H., et al. Myocardial ischemia and reperfusion: A murine model. Am J Physiol. 269 (6 Pt 2), H2147-H2154 (1995).
- Clark, J. E., Flavell, R. A., Faircloth, M. E., Davis, R. J., Heads, R. J., Marber, M. S. Post-infarction remodeling is independent of mitogen-activated protein kinase kinase 3 (MKK3). Cardiovasc Res. 75 (3), 523-530 (2007).
- Hattori, Y., Konno, H., Abe, M., Miyoshi, H., Goto, T., Makabe, H. Synthesis, determination of the absolute configuration of tonkinelin, and inhibitory action with bovine heart mitochondrial complex I. Bioorg Med Chem. 15 (8), 3026-3031 (2007).
- Golam Mostofa, M., et al. CLIP and cohibin separate rDNA from nucleolar proteins destined for degradation by nucleophagy. J Cell Biol. 217 (8), 2675-2690 (2018).
- Chan, J. N. Y., Poon, B. P. K., Salvi, J., Olsen, J. B., Emili, A., Mekhail, K. Perinuclear cohibin complexes maintain replicative life span via roles at distinct silent chromatin domains. Dev Cell. 20 (6), 867-879 (2011).
- Dann, M. M., et al. Quantification of murine myocardial infarct size using 2-D and 4-D high-frequency ultrasound. Am J Physiol Heart Circ Physiol. 322 (3), H359-H372 (2022).
- Kaya, I., Sämfors, S., Levin, M., Borén, J., Fletcher, J. S. Multimodal MALDI imaging mass spectrometry reveals spatially correlated lipid and protein changes in mouse heart with acute myocardial infarction. J Am Soc Mass Spectrom. 31 (10), 2133-2142 (2020).
- Tressler, C., et al. Factorial design to optimize matrix spraying parameters for MALDI mass spectrometry imaging. J Am Soc Mass Spectrom. 32 (12), 2728 (2021).
- Zhang, L., et al. Vascular lipidomics analysis reveals increased levels of phosphocholine and lysophosphocholine in atherosclerotic mice. Nutr Metab. 20 (1), 1-16 (2023).
- Kohno, M., et al. Induction by lysophosphatidylcholine, a major phospholipid component of atherogenic lipoproteins, of human coronary artery smooth muscle cell migration. Circulation. 98 (4), 353-359 (1998).
- Crouch, A. C., Batra, A., Greve, J. M. Hemodynamic response to thermal stress varies with sex and age: A murine MRI study. Int J Hyperthermia. 39 (1), 69-80 (2022).
- Crouch, A. C., Scheven, U. M., Greve, J. M. Cross-sectional areas of deep/core veins are smaller at lower core body temperatures. Physiol Rep. 6 (16), e13839 (2018).
- Crouch, A. C., Castle, P. E., Fitzgerald, L., Scheven, U. M., Greve, J. M. Assessing structural and functional response of murine vasculature to acute β-adrenergic stimulation in vivo during hypothermic and hyperthermic conditions. Int J Hyperthermia. 36 (1), 1136-1145 (2019).
- Crouch, A. C., Manders, A. B., Cao, A. A., Scheven, U. M., Greve, J. M. Cross-sectional area of the murine aorta linearly increases with increasing core body temperature. Int J Hyperthermia. 34 (7), 1121-1133 (2018).
- Clift, C. L., Mehta, A., Drake, R. R., Angel, P. M. Multiplexed imaging mass spectrometry of histological staining, N-glycan and extracellular matrix from one tissue section: A tool for fibrosis research. Methods Mol Biol. 2350, 313-329 (2021).
- Salehi, H. S., et al. Coregistered photoacoustic and ultrasound imaging and classification of ovarian cancer: Ex vivo and in vivo studies. J Biomed Opt. 21 (4), 046006 (2016).
- Verbeeck, N., et al. Connecting imaging mass spectrometry and magnetic resonance imaging-based anatomical atlases for automated anatomical interpretation and differential analysis. Biochim Biophys Acta. 1865 (7), 967-977 (2017).
- Yang, E. Y., et al. Real-time co-registration using novel ultrasound technology: Ex vivo validation and in vivo applications. J Am Soc Echocardiogr. 24 (7), 720 (2011).
- Maris, L., et al. Method for co-registration of high-resolution specimen PET-CT with histopathology to improve insight into radiotracer distributions. EJNMMI Phys. 11 (1), 1-20 (2024).
- Lin, B. J., et al. MSIr: Automatic registration service for mass spectrometry imaging and histology. Anal Chem. 95 (6), 3317-3324 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten