É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Estudo Multimodal da Remodelação Cardiovascular Murina: Ultrassom Quadridimensional e Imagem por Espectrometria de Massas
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, descrevemos um protocolo para usar imagens de ultrassom quadridimensionais in vivo e imagens de espectrometria de massa ex vivo para avaliar alterações biomecânicas e biomoleculares no sistema cardiovascular murino. Esta técnica é aplicada para analisar o remodelamento cardíaco no infarto do miocárdio induzido cirurgicamente e alterações vasculares em animais idosos.
Resumo
A doença cardiovascular (DCV) é a principal causa de morte nos Estados Unidos. Danos no sistema cardiovascular podem ser devidos à exposição ambiental, trauma, toxicidade de drogas ou vários outros fatores. Como resultado, o tecido cardíaco e a vasculatura sofrem alterações estruturais e apresentam função diminuída. O dano e a remodelação resultante podem ser detectados e quantificados com imagens de ultrassom (US) no nível do órgão e imagens de espectrometria de massa (MSI) no nível molecular. Este manuscrito descreve uma metodologia inovadora para estudar a fisiopatologia cardíaca murina, acoplando imagens e análises de ultrassom quadridimensionais ( 4D) in vivo com MSI de dessorção/ionização a laser assistida por matriz ( MADLI) ex vivo do coração. O ultrassom 4D pode fornecer medições volumétricas dinâmicas, incluindo deslocamento radial, tensão da área de superfície e tensão longitudinal ao longo de todo o ciclo cardíaco. Na vasculatura, MSI e ultrassom são usados para avaliar a composição da parede do vaso, hemodinâmica e dinâmica da parede do vaso. A metodologia pode ser adaptada para estudar uma infinidade de doenças cardiovasculares, ajustando métricas funcionais de interesse e/ou variando o protocolo MALDI MSI para atingir moléculas específicas. O MALDI MSI pode ser usado para estudar lipídios, pequenos metabólitos, peptídeos e glicanos. Este protocolo descreve o uso de MALDI MSI para análise lipidômica não direcionada e o uso de imagens de ultrassom para hemodinâmica e biomecânica cardiovascular.
Introdução
A doença cardiovascular (DCV) é uma das principais causas de mortalidade em todo o mundo1. A prevenção e o tratamento da DCV requerem uma compreensão profunda das adaptações moleculares às forças biomecânicas e das mudanças resultantes nas propriedades mecânicas. Em todo o sistema cardiovascular, as forças biomecânicas desempenham um papel importante na função e estrutura do tecido2. As propriedades mecânicas do tecido cardiovascular (CV) são influenciadas por essas forças, tornando-as indicadoras de saúde e doença 3,4,5,6. Para prevenir, diagnosticar e tratar DCV, é crucial desenvolver métodos para entender e observar os processos de iniciação e progressão da doença. A imagem biomédica tem sido fundamental na geração de insights fisiológicos e mecanicistas, e novas tecnologias de imagem e técnicas de análise estão sendo constantemente desenvolvidas. Este protocolo demonstra uma metodologia para combinar duas técnicas de imagem e análise cardiovascular para validar o potencial dessas modalidades de imagem na doença cardíaca isquêmica e no envelhecimento vascular.
Pesquisadores no campo da biomecânica geralmente abordam o estudo da biomecânica por meio de uma combinação de métodos in vivo, ex vivo e in silico . Pesquisas anteriores em biomecânica molecular se concentraram principalmente nas proteínas7 (particularmente as proteínas da matriz extracelular colágeno e elastina devido ao seu impacto nas propriedades biomecânicas), e o trabalho para combinar biomecânica de imagem in vivo com estudos moleculares foi limitado à histologia e imuno-histoquímica. Embora essas abordagens possam produzir muitos indicadores moleculares e tenham produzido mecanismos propostos de remodelação da ECM e das células, elas são normalmente limitadas às colorações ou anticorpos atualmente disponíveis, respectivamente. Este campo de pesquisa está faltando grandes classes de moléculas, por exemplo, lipídios. Embora essas classes moleculares possam ou não estar mecanicamente envolvidas, é importante entender as adaptações moleculares resultantes porque essas moléculas podem ser alvos potenciais para marcadores diagnósticos e terapêuticos. Técnicas de química analítica, como cromatografia líquida-espectrometria de massa (LC-MS), podem ser aplicadas; no entanto, a orientação espacial das moléculas nessas técnicas é perdida. Com a imagem por espectrometria de massa (MSI), a distribuição espacial das moléculas permanece intacta e vários tipos de analitos (classes de moléculas) podem ser visualizados com seções seriais. O MSI é uma poderosa ferramenta analítica para investigar as distribuições espaciais de quase todos os tipos de moléculas no tecido biológico, incluindo metabólitos, lipídios, glicanos, peptídeos e medicamentos de pequeno peso molecular8. Dessorção/ionização a laser assistida por matriz (MALDI) O MSI é um tipo de MSI adequado para análise baseada em descoberta de pesos moleculares na faixa de 50-8000 Da. O MALDI-MSI é uma técnica de ionização que aplica uma matriz de absorção de energia a laser à amostra para auxiliar na ionização dos analitos de interesse. Essa abordagem evita ser limitada a um alvo molecular e pode usar ferramentas de bioinformática para determinar quais moléculas têm impacto nas propriedades biomecânicas e na remodelação.
O ultrassom quadridimensional (4DUS) é um método in vivo não invasivo útil para a caracterização temporal e espacial do coração. O 4DUS utiliza uma série de loops de cinema de alta taxa de quadros de diferentes planos, compilando-os em um conjunto de dados 3D que inclui informações temporais. Isso permite a visualização direta e a quantificação das complexas mudanças de forma 3D das câmaras cardíacas ao longo do ciclo cardíaco, sem depender de suposições geométricas, conforme exigido pela ecocardiografia 2D tradicional. A 4DUS permite que métricas funcionais in vivo sejam calculadas a partir da forma e movimento complexos do coração 9,10, e a MALDI MSI permite o estudo espacial de moléculas biológicas dentro do tecido cardíaco ex vivo11. Para entender completamente as alterações no coração com DCV, mecanismos mecânicos e moleculares precisam ser investigados. Assim, uma metodologia combinada é proposta para estudar a fisiopatologia do coração murino, acoplando imagens e análises de 4DUS com MALDI MSI de lipídios no coração. Essa metodologia é demonstrada em um modelo murino de infarto do miocárdio.
A biomecânica vascular também desempenha um papel crítico na regulação da função cardiovascular2. O enrijecimento vascular, que está associado ao envelhecimento, é um fator de risco para DCV12. As alterações biomecânicas e hemodinâmicas nos vasos podem ser visualizadas por meio de ultrassom. As composições moleculares das paredes dos vasos são componentes importantes da biomecânica e também são extremamente sensíveis às forças hemodinâmicas. Por exemplo, a tensão de cisalhamento da parede oscilatória tem sido implicada no desenvolvimento de placas ateroscleróticas3. Os dados preliminares da mecânica e hemodinâmica dos vasos em animais idosos serão apresentados posteriormente.
A equipe está interessada na relação entre biomecânica e composição molecular em vários estados de doença. A ultrassonografia pré-clínica e a MSI são usadas para determinar a distribuição espacial das alterações moleculares em um tecido e as alterações biomecânicas associadas que ocorrem durante a progressão da doença. Este relatório descreve essas metodologias em detalhes e apresenta dados preliminares sobre o coração e a vasculatura da cabeça/pescoço.
Protocolo
Os experimentos com animais descritos são realizados com a aprovação do Comitê Institucional de Cuidados e Uso de Animais da Universidade do Tennessee, Knoxville.
1. Imagem de ultrassom13
- Configuração e preparação do animal
- Antes de iniciar o experimento, verifique o nível de anestesia com isoflurano e reabasteça se necessário. Verifique os níveis do tanque de oxigênio antes de ligar o gás. Pese o carvão e registre-o no recipiente.
- Configure o espaço de imagem colocando suprimentos perto da plataforma de imagem. Os suprimentos incluem fita pré-cortada, creme depilatório, gaze, cotonetes, água autoclavada, lubrificante da córnea, gel lubrificante, sonda de temperatura retal, eletrodos, gel de ultrassom, gel de eletrodo (se aplicável) e lâmpada de calor (Figura 1A).
- Ligue a máquina de ultrassom e inicialize o motor (o sistema também é capaz de coletar dados de imagem 4DUS). Configure um novo experimento no sistema de ultrassom: selecione Novo estudo ou Nova série e preencha as informações do mouse. Ligue o tablet para monitoramento fisiológico.
- Verifique a torneira para certificar-se de que o fluxo está aberto para a caixa de indução e fechado para o cone do nariz. Pré-encha a caixa de indução anestésica ligando o oxigênio a 1 L/min no vaporizador e girando o botão para 3%-5%.
- Remova o mouse do compartimento da caixa.
- Coloque o camundongo na caixa de indução até que o animal esteja totalmente anestesiado com isoflurano entre 3%-5%.
- Mude a torneira para mudar o fluxo para o cone do nariz na placa de imagem de ultrassom. Gire o botão de isoflurano entre 1% -2%.
- Remova o mouse da caixa de indução e pese o animal. Registre o peso do animal.
- Coloque o mouse em decúbito dorsal na placa de imagem.
- Aplique lubrificante da córnea nos olhos do animal.
- Se houver pelos, remova-os da área de imagem usando um creme depilatório disponível comercialmente e cotonetes. Limpe o creme da pele com água nas esponjas para remover o excesso de creme depilatório da pele e evitar queimaduras.
- Insira os eletrocardiogramas (ECG) por via subcutânea em ambos os lados do tórax e dos membros posteriores, dependendo do posicionamento do animal.
- Uma vez inserido, segure os eletrodos de ECG no lugar com fita adesiva. Se as derivações de ECG não forem usadas (ou seja, as derivações de ECG não são inseridas), monitore o ECG através da placa de imagem animal.
- Para condução elétrica da pele para a placa, coloque o gel condutor na placa e mantenha os membros dos animais na posição.
- Segure os membros do animal no lugar com fita adesiva ou um elástico colocado ao redor da placa e sobre os membros (Figura 1B).
- Use gel lubrificante para inserir a sonda de temperatura retal e prenda a sonda com fita adesiva.
- Verifique os sinais de ECG, respiração e temperatura no tablet. Certifique-se de que a frequência cardíaca seja de ~ 350-600 bpm, respiração ~ 50-100 respirações/min e temperatura retal entre 35-37 °C. Ajuste os níveis de isoflurano conforme necessário.
- Se necessário, acenda uma lâmpada de calor (por exemplo, lâmpada de aquecimento infravermelho de 250 W)14 e ajuste a altura para manter a temperatura central (Figura 1C).
- Coloque a lâmpada de calor a não menos de 12 polegadas do animal. Para estudos iniciais para determinar a distância ideal entre o bulbo e o animal, ajustar a altura e registrar a temperatura da pele usando um termômetro infravermelho, que não deve exceder 42 °C15.
- Ultrassom cardíaco 4D
- Coloque o transdutor no suporte em uma posição semi-travada.
- Oriente o ponto em relevo no transdutor com o ponto azul na tela. A convenção é colocar o ponto em direção ao lado direito do animal.
- Gire o transdutor para que fique orientado ao longo do plano sagital do mouse com um entalhe elevado apontando na direção caudal.
- Use a alavanca na base da placa de ultrassom para inclinar o animal. Use a alavanca na base do transdutor para ajustar o ângulo do transdutor.
- Aplique uma quantidade generosa de gel de ultrassom na superfície ventral da cavidade torácica para acoplamento acústico entre a superfície da pele e o transdutor. Certifique-se de que não haja pêlo restante antes do acoplamento.
- Abaixe o transdutor para fazer contato com o gel de ultrassom.
- Faça micro ajustes com botões X/Y na base da placa ou mova toda a placa para grandes ajustes.
- Certifique-se de que uma visão de eixo longo paraesternal inclua o ápice, a via de saída do ventrículo esquerdo e a aorta alinhados horizontalmente na tela para obter uma imagem de eixo curto mais precisa (Figura 2A, B).
- Selecione Nome da imagem no canto inferior para salvar a imagem na série atual.
- Gire o transdutor 90° no sentido horário para mudar para uma visão paraesternal de eixo curto (Figura 2C).
NOTA: Para obter a melhor qualidade de imagem 4D, os usuários devem tentar alinhar o motor de passo ao longo do eixo longo paraesternal do ventrículo, que nem sempre é diretamente paralelo ao esterno. O coração/VE geralmente fica em um ângulo para baixo à esquerda. - Certifique-se de que o ventrículo esquerdo esteja visível no lado direito da tela e o ventrículo direito esteja no lado esquerdo da tela (Figura 2D).
- Se o mouse tiver um ritmo respiratório estável, clique na caixa de eletrocardiograma no canto superior esquerdo da imagem para ativar o gating respiratório para a imagem 4D. Se não estiver estável, conclua esta etapa durante o pós-processamento.
- Selecione o cubo no canto superior esquerdo da tela para configurar uma imagem 4D.
- Reinicie o transdutor antes de ajustar as posições de partida e parada do transdutor.
- Ajuste a posição inicial logo abaixo do ápice e a posição Parada para o arco aórtico.
- Defina o tamanho da etapa para 0,08-0,13 mm e a taxa de quadros para 200-300 Hz.
NOTA: Tamanhos de passo menores podem fornecer melhor resolução de imagem para análise, mas aumentarão o tempo de aquisição. Tamanhos de passo maiores são aceitáveis se os dados forem coletados com alinhamento ventricular esquerdo quase perfeito e for necessária uma reorientação mínima/nenhuma durante a análise. Considere aumentar a taxa de quadros ao trabalhar com animais com frequências cardíacas mais altas (>450 bpm). - Certifique-se de que os sinais vitais e o sinal do eletrocardiograma estejam consistentemente estáveis (>350 bpm e respiração acima de 50 resp/min) antes de fazer o exame.
- Quando a varredura e o processamento estiverem concluídos, ative Salvar dados EKV/4D para pós-processamento e Respiration Gating ON.
- Selecione Imagem do nome no canto inferior direito e inclua o ID do mouse no nome.
- Para verificar a qualidade da imagem, clique em Mais controles e selecione Carregar em 4D.
- Revise cada visão plana do coração, certificando-se de que o centro do coração não se mova ao longo do ciclo cardíaco. O movimento do centro indica variabilidade no eletrocardiograma e/ou gating respiratório e, portanto, complica o processo de análise. Reajuste os monitores e repita a verificação, se necessário.
- Análise de ultrassom cardíaco 4D
- Navegue até o navegador do estudo. Exporte todas as imagens 2D do modo B para o Vevo Lab e exporte dados 4D como outro tipo de arquivo, selecionando o formato de dados ".raw" para um disco rígido.
NOTA: A caixa de ferramentas da interface gráfica do usuário 4D usada neste trabalho não está disponível publicamente no momento. Para solicitar acesso e instruções detalhadas, entre em contato com o Dr. Craig Goergen, da Purdue University. - Realize uma análise alternativa de dados 4D usando o software VevoLab. Para usar esta estrutura, exporte dados 4D da máquina para arquivos do Vevo Lab.
- Navegue até o navegador do estudo. Exporte todas as imagens 2D do modo B para o Vevo Lab e exporte dados 4D como outro tipo de arquivo, selecionando o formato de dados ".raw" para um disco rígido.
- Imagem vascular
- Para imagens em modo B das artérias carótidas, coloque o transdutor de ultrassom paralelo às artérias carótidas, próximo à linha média.
- Assim que a artéria carótida estiver visível, mova o transdutor para cima para encontrar a bifurcação carotídea.
- Quando uma imagem nítida da bifurcação carotídea for encontrada, ajuste a configuração de ganho para 35 dB e capture a imagem no modo B.
- Inclinar o transdutor para a esquerda ou para a direita pode melhorar a imagem. Ajuste o ângulo do transdutor ou stage para que o recipiente de interesse não fique diretamente paralelo ao transdutor. Isso permite que o ângulo Doppler permaneça abaixo de 60°. Caso contrário, pode ser necessário um deslocamento de +/- 15°.
- Mude para o modo Doppler de onda pulsada para obter as medições de velocidade.
- Coloque o sample volume no centro do recipiente. Ajuste o ângulo do cursor para que fique paralelo à artéria carótida. Em seguida, aperte o play para iniciar as medições de velocidade e salve para gravá-las.
- Para a imagem do modo B das veias jugulares, coloque o transdutor de ultrassom paralelo às veias jugulares. As veias jugulares são anteriores e laterais às artérias carótidas.
- Uma vez localizada a veia jugular, mova o transdutor para o local onde a jugular interna e externa se fundem. Quando uma imagem nítida for encontrada, ajuste a configuração de ganho para 35 dB e capture a imagem no modo B.
- Mude para o modo Doppler de onda pulsada para obter as medições de velocidade.
- Coloque o sample volume no centro do recipiente. Ajuste o ângulo do cursor para que fique paralelo à veia jugular. Em seguida, aperte o play para iniciar as medições de velocidade e salve para gravá-las.
- Análise de ultrassom vascular
- Depois de coletar as imagens do modo B e as medições do Doppler pulsado, meça os diâmetros e velocidades usando o software.
- Depois que as imagens forem carregadas no software, selecione o pacote vascular.
- Selecione cada lado do vaso para obter o diâmetro total.
- Para a artéria carótida, faça medições de diâmetro em sístole e diástole usando a opção de diâmetro no pacote vascular.
- Faça medições de velocidade usando a opção de velocidade no pacote vascular.
- Para a artéria carótida, selecione o pico mais alto para a medição da velocidade na sístole e o pico mais baixo para a medição da velocidade na diástole.
- Use os valores dos diâmetros para calcular o componente circunferencial do tensor de deformação de Green Lagrange usando a seguinte equação,
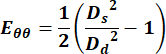 Características (1)
Características (1)
onde Ds representa o diâmetro durante a sístole e Dd representa o diâmetro durante a diástole. - Para a veia jugular, selecione quaisquer pontos ao longo do ciclo para obter a medição da velocidade.
2. Eutanásia e colheita de tecidos
- Prepare barcos de folha de alumínio para congelamento rápido de tecido (Figura 3).
- No final de um estudo, eutanasiar o animal por overdose de isoflurano na concentração de 5% e pneumotórax bilateral ou luxação cervical (somente estudo cardíaco).
- Usando uma pinça para cobrir a pele, corte a pele com uma tesoura sobre o pescoço para vasculatura ou logo abaixo do esterno para cardíaca.
- Corte a pele e as camadas musculares para expor a vasculatura ou corte o osso para expor o coração.
- Usando dissecção romba com cotonetes, isole o coração ou a vasculatura dos tecidos circundantes, incluindo a gordura. Certifique-se de separar o vaso carotídeo do nervo. Remova o coração e os vasos usando ferramentas cirúrgicas.
NOTA: As suturas podem ser usadas para remover a vasculatura suturando proximal e distal antes de remover os vasos. - Coloque o lenço de papel em um barco de folha de alumínio pré-rotulado e coloque o barco em nitrogênio líquido (Figura 3).
- Armazene o tecido a -80 °C até o momento da criosecção. Transporte o tecido com gelo seco para manter a temperatura.
3. Imagem por espectrometria de massa
- Crioseccionamento e montagem em temperatura de corte ideal (OCT) em lâminas de vidro
- Ajuste a temperatura do criostato para -25 °C e insira a lâmina.
- Prepare um mandril de metal aplicando OCT e deixando-o congelar no criostato.
- Fixe a base do coração ao mandril de metal preparado usando OCT (Figura 4A). Não permita que a OCT toque na região de amostra de interesse, pois a OCT contaminará os espectros de massa (contaminação por polietilenoglicol [PEG]) e deprimirá os sinais lipídicos de interesse.
NOTA: Opção para realizar a montagem de água para evitar a contaminação da amostra. Consulte o protocolo adicional abaixo para obter instruções. - Com base na importância do alinhamento espacial nesta metodologia, use a renderização digital em escala do coração fornecida pela GUI do MATLAB para orientar o seccionamento (Figura 4B).
- Conte cada revolução (10 μm) durante o corte e anote a profundidade em milímetros para cada fatia a ser montada na lâmina.
NOTA: É necessário acompanhar locais específicos no coração ou vasculatura para uma correspondência precisa com a renderização digital dos dados de ultrassom fornecidos pelo MATLAB. - Descongele as seções de tecido de montagem (10 μm de espessura) nas lâminas do microscópio.
NOTA: O tipo de lâminas dependerá do espectrômetro de massa usado. Cada slide inclui uma seção de cada região do coração. São necessários no mínimo três lâminas (modo de íon positivo, modo de íon negativo, extra). - Para n = 1/grupo, corte o coração através do eixo longo para visualizar as mudanças moleculares do ápice à base.
- Para vasculatura, embutir o tecido em gelatina e congelar rapidamente antes de seccionar11.
- Armazene as lâminas em envelopes de lâminas a -80 ° C até experimentos de imagem por espectrometria de massa.
- Se estiver realizando cromatografia líquida-espectrometria de massa (LC-MS) para identificar e quantificar lipídios ou metabólitos, corte seções de tecido em série de ~ 60-100 μm e colete-as em tubos de 0,5-2 mL antes de congelar.
- Crioseccionamento e montagem de tecido de água em lâminas de vidro
- Encher um copo com água de cromatografia líquida de alta resolução (HPLC) e retirar com seringas de 5 ml e 1 ml.
- Ajuste a temperatura do criostato para -25 °C e insira a lâmina.
- Coloque um par de pinças no criostato para esfriar antes da montagem.
- Retire 5 ml de água para HPLC numa seringa e coloque-a no criostato.
- Pouco antes de a seringa estar totalmente congelada, esvazie a água no mandril de metal e deixe-o congelar completamente.
- Retire 1 ml de água por HPLC e coloque a seringa no criostato.
- Após aproximadamente 30-60 s, coloque um pequeno ponto de água parcialmente solidificada no centro do mandril. Pegue imediatamente o coração com uma pinça e coloque-o no ponto antes que congele completamente. Segure o coração no lugar até que a água ao redor esteja totalmente congelada.
NOTA: O coração pode ser montado por ápice ou base, dependendo da região de interesse. Para este estudo, a base será montada com o ápice voltado para fora. - Execute o restante das etapas para esse método conforme descrito na etapa 3.1.
- Aplicação matricial
- Remova a lâmina do freezer e coloque-a em um dessecador até que a lâmina esteja seca.
- Ligue o pulverizador HTX M3+, abra o aplicativo HTX no laptop e selecione o método no meio da tela. As concentrações padrão da matriz podem ser encontradas nos métodos armazenados (métodos no lado esquerdo da tela > OI_usermethods > matriz).
- Para este trabalho, ajuste a temperatura do bico para 75 °C, a vazão para 100 μL/min e a pressão para 10 psi.
- Escreva o nome da amostra, polaridade, matriz, solvente e concentração no caderno de laboratório. Calcule a quantidade de matriz necessária por concentração (por exemplo, 5 mL a 10 mg / mL = 50 mg de matriz).
- Faça solvente (por exemplo, 70% de MeOH).
- Para este trabalho, preparar 40 mg/mL de solução de matriz de ácido 2,5-dihidroxibenzóico (DHB) para o modo positivo e 10 mg/mL de solução de matriz de 9-aminoacridina (9AA) para o modo negativo. Faça ambas as matrizes com um solvente MeOH a 70%. Para outras matrizes comuns e parâmetros de pulverização, siga as etapas 3.3.2.
- Pesar a matriz e adicioná-la a um tubo cónico de 15 ml. Certifique-se de que o valor esteja próximo do valor necessário calculado na etapa 3.3.3, mas não precisa ser exato. Calcule a quantidade de solvente com base na massa medida.
- Adicione solvente ao tubo cônico usando uma pipeta.
NOTA: O volume é baseado na massa medida na etapa 3.3.5. - Sonicar a mistura durante 10 min. Enquanto a matriz está sendo sonicada, remova a lâmina do dessecador.
- Abra a bandeja do pulverizador, coloque a corrediça no canto inferior esquerdo e prenda as bordas com fita adesiva.
NOTA: Dobre uma extremidade da fita para facilitar a remoção. - Selecione a região de pulverização da amostra . Feche a bandeja.
- Use uma seringa e um filtro e despeje a matriz na seringa e, em seguida, filtre a matriz através da seringa para o frasco com a tampa preta no lado esquerdo do pulverizador.
- Coloque o frasco de volta em seu lugar no pulverizador e insira o tubo da linha D no frasco.
NOTA: Certifique-se de que o tubo não está a tocar no fundo do frasco para injetáveis e que está totalmente imerso no líquido. - Ligue o gás inerte e certifique-se de que o medidor do pulverizador leia 10 psi. Pressione Iniciar. Quando o pulverizador estiver na temperatura desejada, selecione o início intermitente.
- Após a conclusão da pulverização, abra a bandeja, remova a amostra e coloque-a no porta-lâminas MALDI ou de volta no dessecador (Figura 4C).
- Digitalize o suporte de slides MALDI e deslize no scanner ou tire uma foto com um telefone. Salve a imagem em uma unidade flash para uso com MSI.
- Selecione a lavagem apropriada ou a próxima pulverização no pulverizador. Mova a linha D do frasco para injetáveis da matriz para o copo de resíduos se estiver lavando.
- Pulverize metanol na bandeja e limpe para limpar. Desligue o nitrogênio.
- MALDI MSI
- No computador, navegue até Synapt, verifique se a polaridade foi alterada para o modo necessário e alterne se não estiver.
- Carregue o slide no instrumento na fonte MALDI e clique no botão de carregamento no computador usando o software.
- Mova a imagem (etapa 1.2.17) para a pasta Imagens na pasta Projeto .
- Navegue até HD Imaging e abra o projeto anterior (um que usa a mesma matriz).
- Em um caderno de laboratório, registre a faixa de massa, capture e transfira energia de colisão, energia do laser, modo neg / pos.
- Em HD Imaging, navegue até o ícone de página branca. Em seguida, clique na seta suspensa e selecione nova placa.
- Navegue para encontrar a imagem na pasta Projeto > na pasta Imagens e abra-a.
- Altere o tipo de placa para o padrão MALDI e defina as bordas da placa (escolha 4 cantos mostrados abaixo com sinais de mais vermelhos).
NOTA: A caixa superior esquerda é para navegar para cada canto. A caixa superior direita é para selecionar os cantos. A caixa inferior serve apenas para visualizar o slide inteiro. - Navegue até a pequena guia de padrão no canto superior esquerdo e clique em lápis ou outras opções de retângulo ou círculo para delinear o tecido. Contorne o tecido usando os cliques esquerdo e o botão direito para finalizar.
- O tamanho do pixel é padrão para 50 μm. Mude se necessário.
- Clique em Salvar como e navegue até Project Imaging AcqFolder e rotule como nome do slide (formato AAAAMMDD). Em seguida, salve.
- Clique no botão Mass Lynx no meio superior para exportar para o Mass Lynx.
- Navegue até o software Mass Lynx.
- Clique em Arquivo > abrir a pasta do projeto. Edite o nome do arquivo e o nome do texto no slide (formato AAAAMMDD).
- Clique com o botão direito do mouse no arquivo MS, navegue e escolha o slide atual.
- Clique com o botão direito do mouse novamente no arquivo MS e clique em Editar. Clique duas vezes com o botão esquerdo na caixa azul.
- Navegue até as condições de verificação. Encontre as configurações de origem e altere para definido pelo usuário. Em seguida, navegue até a pasta correta do projeto > pasta adquirida > slide atual > OK.
- Salve clicando em Salvar como e inclua o nome do slide.
- Calibre o instrumento usando um composto de referência (por exemplo, fósforo vermelho)
- Navegue até Synapt (Calibrar). Clique no botão play para começar a adquirir dados. Rotule como YYYYMMDD_cal1 (por exemplo: 20230614_cal1).
- Certifique-se de que ele tenha a mesma faixa de massa de 50 a 2000. A energia do laser pode ser de cerca de 175.
- Clique em fire laser play e clique no botão Avançar . Deixe correr para cerca de 15 ou 20. Clique em stop vermelho.
- Navegue até o W Console.
- Crie a calibração e clique em iniciar. Em editor de perfil de calibração, clique em arquivo > novo. Digite YYYYMMDD_cal1.
- Edite a faixa de massa para a faixa de massa desejada para o projeto (por exemplo, 50-2000).
- Escolha a calibração manual. Escolha o modo de resolução e clique em editar. Selecione o fósforo vermelho como composto de referência.
- Clique no arquivo de dados brutos e role até o final (deve ser o que acabou de ser adquirido).
- Vá para o histórico na parte inferior, selecione Acc mass > okay > ok.
- Continue clicando em próximo e ok até que o botão Iniciar seja visto e clique em iniciar.
- Navegue até a verificação verde e veja quais picos não foram encontrados.
- Navegue até Mass Lynx (Inicie a execução real para amostras experimentais).
- Aperte o botão play e verifique se o número de amostras está correto
- Navegue até a imagem HD.
- Verifique se o tamanho do pixel ainda é o que foi originalmente definido como
- Navegue até o Synapt para verificar a polaridade.
- Clique no botão da câmera para certificar-se de que ele está se movendo e disparando o laser.
- Verifique e certifique-se de que está coletando dados. O espectro de massa adquirido deve ser exibido na página do Synapt.
- Análise MS
- Use o software HD Imaging e o Mass Lynx para análise.
- Processe dados de imagem usando a guia Processar em Imagens HD.
- Visualize imagens de íons moleculares, também chamadas de imagens MS, em Imagens HD na guia Análise . Um mapa de calor de uma imagem de íon molecular mostra a intensidade desse íon m / z sobre uma região (Figura 5). Use as sobreposições de imagens de MS para mostrar a distribuição espacial das moléculas (Figura 6 e Figura 7).
- Normalize os dados usando TIC ou normalização apropriada para imagens.
- Identifique picos m/z que se correlacionam espacialmente com regiões de interesse. Execute uma correlação espacial (R > 0,65) na imagem HD para identificar picos colocalizados.
- Procure a identificação lipídica putativa desses picos com LipidMaps16 e METASPACE17.
- Confirme as anotações lipídicas com dados subsequentes de LC-MS. Use o Metaboanalyst para análise estatística de dados de LC-MS.
Resultados
Os protocolos de imagem descritos acima foram utilizados para dois estudos preliminares: remodelamento do infarto do miocárdio (IM) e envelhecimento vascular. Para os experimentos cardíacos, foi realizada uma cirurgia de ligadura definitiva da artéria coronária para induzir infarto agudo do miocárdio18,19. O ultrassom 4D e o MALDI MSI foram realizados progressivamente no mesmo tecido, revelando alterações fisiológicas e m...
Discussão
A imagem de US pode ser dependente do operador, mas o uso de pontos de referência anatômicos e o treinamento adequado podem limitar o viés do usuário. O ultrassom 2D é particularmente suscetível à variabilidade entre usuários porque as visualizações dependem do ângulo, enquanto o 4DUS é menos suscetível, pois a aquisição abrange todo o volume e é independente do ângulo. Também foi determinado que a reprodutibilidade da imagem é mais fácil de alcançar devido à plataf...
Divulgações
Craig J. Goergen é consultor remunerado da FUJIFILM VisualSonics.
Agradecimentos
Allison Jones é apoiada pela bolsa de pós-graduação do Departamento de Engenharia Mecânica, Aeroespacial e Biomédica da Universidade do Tennessee. A pesquisa relatada nesta publicação (Conner Earl) foi apoiada pelo Instituto Nacional do Coração, Pulmão e Sangue dos Institutos Nacionais de Saúde F30HL162452. O conteúdo é de responsabilidade exclusiva dos autores e não representa necessariamente as opiniões oficiais dos Institutos Nacionais de Saúde.
Materiais
| Name | Company | Catalog Number | Comments |
| 2,5-dihydroxybenzoic acid (DHB) | Supelco, >99.0% (HPLC) | 85707-10MG-F | DHB matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sigma/85707?cm_sp=Insite-_-wimsShippingEmailRecs_wims EmailAPI_wimsGruCrossEntropy-_-wimsEmailAPI10-3 |
| 9-aminoacridine (9AA) | Supelco, ≥99.5% (HPLC) | 92817-1G | 9-Aminoacridine matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sial/92817?srsltid=AfmBOooiQjQ4pWv_XxITkU 4Lkm0UnHXKekGS_ dFl7V40V9QLWoPpNLoc |
| Aquasonic Ultrasound Gel | Parker Laboratories | Parker 01-02 | Ultrasound Gel; https://www.parkerlabs.com/products/aquasonic-100-ultrasound-transmission-gel/ |
| Benchtop Dewar Flasks | ThermoScientific | 4150-2000 | Container for liquid nitrogen; https://www.thermofisher.com/order/catalog/product/4150-4000?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sQVc2f-NxN04Nb5Mv F6TZ7GLHWWDEeqDYmEvtKJSQ YHDeVgZ9qylvYaAs27EALw_wcB &source=google_shopping&ISO_ CODE=us&LANG_CODE=en&ef_id =Cj0KCQjwpvK4BhDUARIsADHt9 sQVc2f-NxN04Nb5MvF6TZ7GLHWWDE eqDYmEvtKJSQYHDeVgZ9qylvYa As27EALw_wcB:G:s&s_kwcid=AL!3652 !3!716188292869!!!g!2366243726129 !!21787513085!171591181194&ev_chn =shop&cid=0se_gaw_30092024_ PBYTXL&source=google_shopping &ISO_CODE=us&LANG_CODE= en&gad_source=1 |
| Cryostat | Leica Biosystems | CM Series | https://www.leicabiosystems.com/us/histology-equipment/cryostats/ |
| Dessicator | VWR | 89054-052 | https://us.vwr.com/store/product/9104882/desiccator-plastic-ace-glass-incorporated |
| Epredia MX35 Premier Disposable Low-profile Microtome Blades | Fisher Scientific | 3052835 | Cryostat blade; https://www.fishersci.com/shop/products/mx35-premier-disposable-low-profile-microtome-blades/3052835 |
| Falcon 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | Conical Tubes; https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/1495953A?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sSBcy5n-lhShligJUOX5KKVGn0bt87 8AB2_muOD2PPTue1phpZgeqwa AqgiEALw_wcB&ef_id=Cj0KCQjw pvK4BhDUARIsADHt9sSBcy5n-lhS hligJUOX5KKVGn0bt878AB2_muO D2PPTue1phpZgeqwaAqgiEALw_ wcB:G:s&ppc_id=PLA_goog_20861 45674_81843405034_1495953A__ 386247001345_165426395473886 37329&ev_chn=shop&s_kwcid=AL!4428!3 !386247001345!!!g!856907751004!& gad_source=1 |
| Flex-Tubes Microcentrifuge Tubes | Eppendorf | EP022364120 | Centrifuge tubes; https://www.sigmaaldrich.com/US/en/product/sigma/ep022364120?utm_source=google%2Cgoogle&utm _medium=organicshopping%2Ccpc&utm _campaign=21043330280&utm_ content=&gclid=Cj0KCQjwpv K4BhDUARIsADHt9sTHRD35k CHPtfI2A41axodnMVr6a1eBKk zM4bSUYQAyfEKo3UgTAEQa Ap7wEALw_wcB |
| Gas Nitrogen | Airgas | N/A | |
| Glass microscope slides | Electron Microscopy Sciences | 71873-02 | https://www.emsdiasum.com/positive-charge-microscope-slides |
| Liquid Nitrogen | Airgas | N/A | |
| Mass Spectrometer | Waters | Synapt G2-Si | https://www.waters.com/waters/en_US/SYNAPT-G2-Si-Mass-Spectrometry/nav.htm?locale=en_mkcid=1000251Foodety%3C/a%3E&cid=134740653&bcid= 134528734 |
| Matrix Sprayer | HTX Technologies | M3+ | https://www.htximaging.com/htx-m3-sprayer |
| Methanol (HPLC), >99.9% | Fisher Chemical | A4524 | Methanol; https://www.fishersci.com/shop/products/methanol-hplc-fisher-chemical-9/A4524?crossRef=A4524#?keyword=A4524 |
| Preclinical Ultrasound System | FUJIFILM VisualSonics | Vevo 3100 | https://www.visualsonics.com/product/imaging-systems/vevo-3100; Vevo F2 has replaced the Vevo 3100 in production. System includes isoflurane vaporizer and induction box. |
| Reynolds Wrap | N/A | N/A | Aluminum foil |
| Signagel Electrode Gel | Parker Laboratories | Parker 15-60 | Electrode Conducting Gel; https://www.parkerlabs.com/products/signagel-electrode-gel/ |
| Sterile Lubricating Jelly | Medline | MDS032273Z | Lubricating Gel; https://www.medline.com/ce/product/Sterile-Lubricating-Jelly/Lubricating-Jelly/Z05-PF03664?sku=MDPMDS032273H |
| Surgical instruments: scissors, forceps/tweezers, sutures | Fine Science Tools | 11252-00, 11050-10, 14016-14, 14084-08, 15000-08 | info@finescience.com |
| Surgical Sponges 200 Pack –Gauze Pads Non sterile -First Aid Wound Care Dressing Sponge –Woven Medical Nonstick, Non Adherent Mesh Scrubbing | Medpride | B08RZGQ5GW | Gauze; https://www.amazon.com/Medpride-Surgical-Sponges-200-Pack/dp/B08RZGQ5GW/ref=asc_df_B08RZGQ5GW/?tag=hyprod-20&linkCode=df0&h vadid=693270340506&hvpos= &hvnetw=g&hvrand=960915122 2290977669&hvpone=&hvptwo= &hvqmt=&hvdev=c&hvdvcmdl=& hvlocint=&hvlocphy=9192978&hv targid=pla-1245491514869&psc= 1&mcid=33f4d647c88630c79116 888d565a63b0 |
| Tissue-Plus O.C.T. Compound | Fisher Scientific | 23-730-571 | OCT; https://www.fishersci.com/shop/products/tissue-plus-o-c-t-compound-2/23730571 |
| Wood Handled Cotton Swabs and Applicators | Fisherbrand | 22-363-160 | Cotton swab; https://www.fishersci.com/shop/products/wood-handled-cotton-swabs-applicators-8/p-7146852 |
Referências
- Tsao, C. W., et al. Heart disease and stroke statistics-2022 update: A report from the American Heart Association. Circulation. 145 (8), E153-E639 (2022).
- Kassab, G. S. Biomechanics of the cardiovascular system: The aorta as an illustratory example. J R Soc Interface. 3 (11), 719-740 (2006).
- Moore, J. E., Xu, C., Glagov, S., Zarins, C. K., Ku, D. N. Fluid wall shear stress measurements in a model of the human abdominal aorta: Oscillatory behavior and relationship to atherosclerosis. Atherosclerosis. 110 (2), 225-240 (1994).
- Nerem, R. M. Vascular fluid mechanics, the arterial wall, and atherosclerosis. J Biomech Eng. 114 (3), 274 (1992).
- Cecchi, E., et al. Role of hemodynamic shear stress in cardiovascular disease. Atherosclerosis. 214 (2), 249-256 (2011).
- Good, B. The influence of blood composition and loading conditions on the behavior of embolus analogs. J Mech Behav Biomed Mater. 140, 105738 (2023).
- Bao, G., et al. Molecular biomechanics: The molecular basis of how forces regulate cellular function NIH public access. Mol Cell Biomech. 3 (2), 91-105 (2010).
- Schwamborn, K., Caprioli, R. M. Molecular imaging by mass spectrometry-looking beyond classical histology. Nat Rev Cancer. 10 (9), 639-646 (2010).
- Soepriatna, A. H., Kevin Yeh, A., Clifford, A. D., Bezci, S. E., O'Connell, G. D., Goergen, C. J. Three-dimensional myocardial strain correlates with murine left ventricular remodelling severity post-infarction. J R Soc Interface. 16 (160), 20190570 (2019).
- Damen, F. W., et al. High-frequency 4-dimensional ultrasound (4DUS): A reliable method for assessing murine cardiac function. Tomography. 3 (4), 180-187 (2017).
- McDonald, R., Poulos, D., Gutzwiller, L., Sheth, R. A., Good, B., Crouch, A. C. A MALDI mass spectrometry imaging sample preparation method for venous thrombosis with initial lipid characterization of lab made and murine clots. J Am Soc Mass Spectrom. 34 (9), 1879-1889 (2023).
- Benetos, A., et al. Influence of age, risk factors, and cardiovascular and renal disease on arterial stiffness: Clinical applications. Am J Hypertens. 15 (12), 1101-1108 (2002).
- Pistner, A., Belmonte, S., Coulthard, T., Blaxall, B. C. Murine echocardiography and ultrasound imaging. J Vis Exp. (42), v2100 (2010).
- Bellantuono, I., et al. A toolbox for the longitudinal assessment of healthspan in aging mice. Nat Protoc. 15 (2), 540-574 (2020).
- Henriques, F. C., Moritz, A. R. Studies of thermal injury: I. The conduction of heat to and through skin and the temperatures attained therein. A theoretical and an experimental investigation. Am J Pathol. 23 (4), 530 (1947).
- Conroy, M. J., et al. LIPID MAPS: Update to databases and tools for the lipidomics community. Nucleic Acids Res. 52 (D1), D1677-D1682 (2024).
- Palmer, A., et al. FDR-controlled metabolite annotation for high-resolution imaging mass spectrometry. Nat Methods. 14 (1), 57-60 (2016).
- Michael, L. H., et al. Myocardial ischemia and reperfusion: A murine model. Am J Physiol. 269 (6 Pt 2), H2147-H2154 (1995).
- Clark, J. E., Flavell, R. A., Faircloth, M. E., Davis, R. J., Heads, R. J., Marber, M. S. Post-infarction remodeling is independent of mitogen-activated protein kinase kinase 3 (MKK3). Cardiovasc Res. 75 (3), 523-530 (2007).
- Hattori, Y., Konno, H., Abe, M., Miyoshi, H., Goto, T., Makabe, H. Synthesis, determination of the absolute configuration of tonkinelin, and inhibitory action with bovine heart mitochondrial complex I. Bioorg Med Chem. 15 (8), 3026-3031 (2007).
- Golam Mostofa, M., et al. CLIP and cohibin separate rDNA from nucleolar proteins destined for degradation by nucleophagy. J Cell Biol. 217 (8), 2675-2690 (2018).
- Chan, J. N. Y., Poon, B. P. K., Salvi, J., Olsen, J. B., Emili, A., Mekhail, K. Perinuclear cohibin complexes maintain replicative life span via roles at distinct silent chromatin domains. Dev Cell. 20 (6), 867-879 (2011).
- Dann, M. M., et al. Quantification of murine myocardial infarct size using 2-D and 4-D high-frequency ultrasound. Am J Physiol Heart Circ Physiol. 322 (3), H359-H372 (2022).
- Kaya, I., Sämfors, S., Levin, M., Borén, J., Fletcher, J. S. Multimodal MALDI imaging mass spectrometry reveals spatially correlated lipid and protein changes in mouse heart with acute myocardial infarction. J Am Soc Mass Spectrom. 31 (10), 2133-2142 (2020).
- Tressler, C., et al. Factorial design to optimize matrix spraying parameters for MALDI mass spectrometry imaging. J Am Soc Mass Spectrom. 32 (12), 2728 (2021).
- Zhang, L., et al. Vascular lipidomics analysis reveals increased levels of phosphocholine and lysophosphocholine in atherosclerotic mice. Nutr Metab. 20 (1), 1-16 (2023).
- Kohno, M., et al. Induction by lysophosphatidylcholine, a major phospholipid component of atherogenic lipoproteins, of human coronary artery smooth muscle cell migration. Circulation. 98 (4), 353-359 (1998).
- Crouch, A. C., Batra, A., Greve, J. M. Hemodynamic response to thermal stress varies with sex and age: A murine MRI study. Int J Hyperthermia. 39 (1), 69-80 (2022).
- Crouch, A. C., Scheven, U. M., Greve, J. M. Cross-sectional areas of deep/core veins are smaller at lower core body temperatures. Physiol Rep. 6 (16), e13839 (2018).
- Crouch, A. C., Castle, P. E., Fitzgerald, L., Scheven, U. M., Greve, J. M. Assessing structural and functional response of murine vasculature to acute β-adrenergic stimulation in vivo during hypothermic and hyperthermic conditions. Int J Hyperthermia. 36 (1), 1136-1145 (2019).
- Crouch, A. C., Manders, A. B., Cao, A. A., Scheven, U. M., Greve, J. M. Cross-sectional area of the murine aorta linearly increases with increasing core body temperature. Int J Hyperthermia. 34 (7), 1121-1133 (2018).
- Clift, C. L., Mehta, A., Drake, R. R., Angel, P. M. Multiplexed imaging mass spectrometry of histological staining, N-glycan and extracellular matrix from one tissue section: A tool for fibrosis research. Methods Mol Biol. 2350, 313-329 (2021).
- Salehi, H. S., et al. Coregistered photoacoustic and ultrasound imaging and classification of ovarian cancer: Ex vivo and in vivo studies. J Biomed Opt. 21 (4), 046006 (2016).
- Verbeeck, N., et al. Connecting imaging mass spectrometry and magnetic resonance imaging-based anatomical atlases for automated anatomical interpretation and differential analysis. Biochim Biophys Acta. 1865 (7), 967-977 (2017).
- Yang, E. Y., et al. Real-time co-registration using novel ultrasound technology: Ex vivo validation and in vivo applications. J Am Soc Echocardiogr. 24 (7), 720 (2011).
- Maris, L., et al. Method for co-registration of high-resolution specimen PET-CT with histopathology to improve insight into radiotracer distributions. EJNMMI Phys. 11 (1), 1-20 (2024).
- Lin, B. J., et al. MSIr: Automatic registration service for mass spectrometry imaging and histology. Anal Chem. 95 (6), 3317-3324 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados