È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studio multimodale del rimodellamento cardiovascolare murino: ecografia quadridimensionale e spettrometria di massa
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, descriviamo un protocollo per utilizzare l'ecografia quadridimensionale in vivo e la spettrometria di massa ex vivo per valutare le alterazioni biomeccaniche e biomolecolari nel sistema cardiovascolare murino. Questa tecnica viene applicata per analizzare il rimodellamento cardiaco nell'infarto miocardico indotto chirurgicamente e nelle alterazioni vascolari negli animali anziani.
Abstract
Le malattie cardiovascolari (CVD) sono la principale causa di morte negli Stati Uniti. I danni al sistema cardiovascolare possono essere dovuti all'esposizione ambientale, ai traumi, alla tossicità dei farmaci o a numerosi altri fattori. Di conseguenza, il tessuto cardiaco e il sistema vascolare subiscono cambiamenti strutturali e mostrano una funzione ridotta. Il danno e il conseguente rimodellamento possono essere rilevati e quantificati con l'imaging ecografico (US) a livello d'organo e con la spettrometria di massa (MSI) a livello molecolare. Questo manoscritto descrive una metodologia innovativa per lo studio della fisiopatologia cardiaca murina, l'accoppiamento dell'imaging e dell'analisi ecografica quadridimensionale (4D) in vivo con l'MSI del cuore di desorbimento/ionizzazione laser assistita da matrice ( MADLI) ex vivo. Gli ultrasuoni 4D possono fornire misurazioni volumetriche dinamiche, tra cui lo spostamento radiale, la deformazione superficiale e la deformazione longitudinale durante un intero ciclo cardiaco. Nel sistema vascolare, l'MSI e gli ultrasuoni vengono utilizzati per valutare la composizione della parete dei vasi, l'emodinamica e la dinamica delle pareti dei vasi. La metodologia può essere personalizzata per studiare una miriade di malattie cardiovascolari regolando le metriche funzionali di interesse e/o variando il protocollo MALDI MSI per mirare a molecole specifiche. MALDI MSI può essere utilizzato per studiare lipidi, piccoli metaboliti, peptidi e glicani. Questo protocollo delinea l'uso di MALDI MSI per l'analisi lipidomica non mirata e l'uso dell'imaging ecografico per l'emodinamica e la biomeccanica cardiovascolare.
Introduzione
Le malattie cardiovascolari (CVD) sono una delle principali cause di mortalità in tutto il mondo1. La prevenzione e il trattamento delle CVD richiedono una comprensione approfondita degli adattamenti molecolari alle forze biomeccaniche e dei conseguenti cambiamenti nelle proprietà meccaniche. In tutto il sistema cardiovascolare, le forze biomeccaniche svolgono un ruolo importante nella funzione e nella struttura del tessuto2. Le proprietà meccaniche del tessuto cardiovascolare (CV) sono influenzate da queste forze, rendendoli indicatori di salute e malattia 3,4,5,6. Per prevenire, diagnosticare e trattare le CVD, è fondamentale sviluppare metodi per comprendere e osservare i processi di inizio e progressione della malattia. L'imaging biomedico è stato fondamentale per generare intuizioni fisiologiche e meccanicistiche e vengono costantemente sviluppate nuove tecnologie di imaging e tecniche di analisi. Questo protocollo dimostra una metodologia per combinare due tecniche di imaging e analisi cardiovascolare per convalidare il potenziale di queste modalità di imaging nella cardiopatia ischemica e nell'invecchiamento vascolare.
I ricercatori nel campo della biomeccanica spesso si avvicinano allo studio della biomeccanica attraverso una combinazione di metodi in vivo, ex vivo e in silico . Precedenti ricerche nel campo della biomeccanica molecolare si sono concentrate principalmente sulle proteine7 (in particolare le proteine della matrice extracellulare collagene ed elastina a causa del loro impatto sulle proprietà biomeccaniche) e il lavoro per combinare la biomeccanica dell'imaging in vivo con gli studi molecolari è stato limitato all'istologia e all'immunoistochimica. Sebbene questi approcci possano produrre molti indicatori molecolari e abbiano prodotto la proposta di meccanismi di rimodellamento della ECM e delle cellule, sono in genere limitati rispettivamente alle colorazioni o agli anticorpi attualmente disponibili. In questo campo di ricerca mancano grandi classi di molecole, ad esempio i lipidi. Sebbene queste classi molecolari possano o meno essere coinvolte meccanicamente, gli adattamenti molecolari risultanti sono importanti da comprendere perché queste molecole potrebbero essere potenziali bersagli sia per i marcatori diagnostici che per le terapie. Possono essere applicate tecniche di chimica analitica, come la cromatografia liquida-spettrometria di massa (LC-MS); Tuttavia, l'orientamento spaziale delle molecole in queste tecniche viene perso. Con l'imaging con spettrometria di massa (MSI), la distribuzione spaziale delle molecole rimane intatta e diversi tipi di analiti (classi di molecole) possono essere visualizzati con sezioni seriali. L'MSI è un potente strumento analitico per studiare le distribuzioni spaziali di quasi tutti i tipi di molecole nei tessuti biologici, inclusi metaboliti, lipidi, glicani, peptidi e farmaci a basso peso molecolare8. Desorbimento/ionizzazione laser assistita da matrice (MALDI) MSI è un tipo di MSI che si adatta bene per l'analisi basata sulla scoperta di pesi molecolari nell'intervallo 50-8000 Da. MALDI-MSI è una tecnica di ionizzazione che applica una matrice laser che assorbe l'energia al campione per favorire la ionizzazione degli analiti di interesse. Questo approccio evita di essere limitato a un bersaglio molecolare e può utilizzare strumenti bioinformatici per determinare quali molecole hanno un impatto sulle proprietà biomeccaniche e sul rimodellamento.
L'ecografia quadridimensionale (4DUS) è una metodica non invasiva in vivo utile per la caratterizzazione sia temporale che spaziale del cuore. 4DUS utilizza una serie di loop cine ad alta frequenza di fotogrammi provenienti da diversi piani, compilandoli in un set di dati 3D che include informazioni temporali. Ciò consente la visualizzazione diretta e la quantificazione dei complessi cambiamenti di forma 3D delle camere cardiache nel corso del ciclo cardiaco senza fare affidamento su ipotesi geometriche come richiesto per l'ecocardiografia 2D tradizionale. 4DUS consente di calcolare metriche funzionali in vivo dalla forma e dal movimento complessi del cuore 9,10, mentre MALDI MSI consente lo studio spaziale di molecole biologiche all'interno del tessuto cardiaco ex vivo11. Per comprendere appieno le alterazioni nel cuore con CVD, è necessario studiare sia i meccanismi meccanici che quelli molecolari. Pertanto, viene proposta una metodologia combinata per lo studio della fisiopatologia cardiaca murina, accoppiando l'imaging e l'analisi 4DUS con MALDI MSI dei lipidi nel cuore. Questa metodologia è dimostrata in un modello murino di infarto miocardico.
Anche la biomeccanica vascolare svolge un ruolo fondamentale nella regolazione della funzione cardiovascolare2. L'irrigidimento vascolare, che è associato all'invecchiamento, è un fattore di rischio per CVD12. I cambiamenti biomeccanici ed emodinamici nei vasi possono essere visualizzati utilizzando gli ultrasuoni. Le composizioni molecolari delle pareti dei vasi sono componenti importanti della biomeccanica e sono anche squisitamente sensibili alle forze emodinamiche. Ad esempio, lo sforzo di taglio oscillatorio della parete è stato implicato nello sviluppo della placca aterosclerotica3. Successivamente verranno presentati i dati preliminari di meccanica ed emodinamica dei vasi negli animali anziani.
Il team è interessato alla relazione tra biomeccanica e composizione molecolare in vari stati patologici. L'ecografia preclinica e l'MSI vengono utilizzati per determinare la distribuzione spaziale dei cambiamenti molecolari in un tessuto e i cambiamenti biomeccanici associati che si verificano durante la progressione della malattia. Questo rapporto descrive in dettaglio queste metodologie e presenta dati preliminari sul cuore e sulla vascolarizzazione della testa/collo.
Protocollo
Gli esperimenti sugli animali descritti sono stati condotti con l'approvazione del Comitato Istituzionale per la Cura e l'Uso degli Animali dell'Università del Tennessee, Knoxville.
1. Ecografia13
- Allestimento e preparazione degli animali
- Prima di iniziare l'esperimento, controllare il livello di anestesia dell'isoflurano e rabboccare se necessario. Controllare i livelli del serbatoio di ossigeno prima di accendere il gas. Pesare il carbone e registrarlo sul contenitore.
- Configura lo spazio di imaging posizionando i materiali di consumo vicino alla piattaforma di imaging. Le forniture includono nastro pretagliato, crema depilatoria, garza, tamponi di cotone, acqua sterilizzata in autoclave, lubrificante per cornea, gel lubrificante, sonda di temperatura rettale, elettrodi, elettrodi, gel per ultrasuoni, gel per elettrodi (se applicabile) e lampada termica (Figura 1A).
- Accendere l'ecografo e inizializzare il motore (il sistema è anche in grado di raccogliere i dati delle immagini 4DUS). Imposta un nuovo esperimento sull'ecografo: seleziona Nuovo studio o Nuova serie e compila le informazioni del mouse. Accendere il tablet per il monitoraggio fisiologico.
- Controllare il rubinetto per assicurarsi che il flusso sia aperto alla scatola di induzione e chiuso al cono anteriore. Pre-riempire la scatola di induzione dell'anestesia accendendo l'ossigeno a 1 L/min sul vaporizzatore e ruotando la manopola al 3%-5%.
- Rimuovere il mouse dalla gabbia dell'alloggiamento.
- Posiziona il topo nella scatola di induzione fino a quando l'animale non è completamente anestetizzato con isoflurano tra il 3% e il 5%.
- Commutare il rubinetto per modificare il flusso verso il cono anteriore sulla piastra di imaging a ultrasuoni. Ruotare la manopola dell'isoflurano tra l'1% e il 2%.
- Rimuovi il mouse dalla scatola di induzione e pesa l'animale. Registra il peso dell'animale.
- Posizionare il mouse in posizione supina sulla piastra ai fosfori.
- Applicare lubrificante corneale sugli occhi dell'animale.
- Se è presente del pelo, rimuovere il pelo dall'area di imaging utilizzando una crema depilatoria disponibile in commercio e tamponi di cotone. Pulire la crema dalla pelle con acqua su spugne per rimuovere la crema depilatoria in eccesso dalla pelle e prevenire ustioni.
- Inserire le derivazioni dell'elettrocardiogramma (ECG) per via sottocutanea su entrambi i lati del torace e dell'arto posteriore, a seconda della posizione dell'animale.
- Una volta inserite, tenere in posizione le derivazioni ECG con del nastro adesivo. Se non si utilizzano le derivazioni ECG (cioè le derivazioni ECG non sono inserite), monitorare l'ECG tramite la piastra di imaging per animali.
- Per la conduzione elettrica dalla pelle alla piastra, individuare il gel conduttore sulla piastra e tenere in posizione gli arti dell'animale.
- Tenere gli arti dell'animale in posizione con del nastro adesivo o un elastico posizionato attorno alla piastra e sopra gli arti (Figura 1B).
- Utilizzare gel lubrificante per inserire la sonda di temperatura rettale e fissare la sonda con del nastro adesivo.
- Controllare i segnali ECG, respirazione e temperatura sul tablet. Assicurarsi che la frequenza cardiaca sia ~350-600 bpm, la respirazione ~50-100 respiri/min e la temperatura rettale compresa tra 35-37 °C. Regolare i livelli di isoflurano secondo necessità.
- Se necessario, accendere una lampada riscaldante (ad es. lampadina riscaldante a infrarossi da 250 W)14 e regolare l'altezza per mantenere la temperatura interna (Figura 1C).
- Posiziona il calore lamp a non meno di 12 pollici dall'animale. Per gli studi iniziali per determinare la distanza ottimale tra bulbo e animale, regolare l'altezza e registrare la temperatura della pelle utilizzando un termometro a infrarossi, che non deve superare i 42 °C15.
- Ecografia cardiaca 4D
- Posizionare il trasduttore nel supporto in una posizione semibloccata.
- Orientare il punto in rilievo sul trasduttore con il punto blu sullo schermo. La convenzione è quella di posizionare il punto verso il lato destro dell'animale.
- Ruotare il trasduttore in modo che sia orientato lungo il piano sagittale del mouse con una tacca rialzata rivolta in direzione caudale.
- Utilizzare la leva alla base della piastra ecografica per inclinare l'animale. Utilizzare la leva alla base del trasduttore per regolare l'angolo del trasduttore.
- Applicare una generosa quantità di gel per ultrasuoni sulla superficie ventrale della cavità toracica per l'accoppiamento acustico tra la superficie della pelle e il trasduttore. Assicurarsi che non vi siano residui di pelo prima dell'accoppiamento.
- Abbassare il trasduttore per entrare in contatto con il gel per ultrasuoni.
- Effettuare micro regolazioni con le manopole X/Y alla base della piastra o spostare l'intera piastra per regolazioni di grandi dimensioni.
- Assicurarsi che una vista dell'asse lungo parasternale includa l'apice, il tratto di efflusso ventricolare sinistro e l'aorta allineati orizzontalmente sullo schermo per un'immagine dell'asse corto più accurata (Figura 2A, B).
- Seleziona Nome immagine nell'angolo in basso per salvare l'immagine nella serie corrente.
- Ruotare il trasduttore di 90° in senso orario per passare a una vista parasternale dell'asse corto (Figura 2C).
NOTA: Per una migliore qualità dell'immagine 4D, gli utenti dovrebbero cercare di allineare il motore passo-passo lungo lungo parasternale del ventricolo che non è sempre direttamente parallelo allo sterno. Il cuore/LV si trova spesso ad un angolo sinistro verso il basso. - Assicurarsi che il ventricolo sinistro sia visibile sul lato destro dello schermo e che il ventricolo destro sia sul lato sinistro dello schermo (Figura 2D).
- Se il mouse ha un ritmo respiratorio stabile, premi la casella ECG nell'angolo in alto a sinistra dell'immagine per attivare il gating respiratorio per l'immagine 4D. Se non è stabile, completare questo passaggio durante la post-elaborazione.
- Seleziona il cubo nell'angolo in alto a sinistra dello schermo per impostare un'immagine 4D.
- Reimpostare il trasduttore prima di regolare le posizioni di avvio e arresto del trasduttore.
- Regolare la posizione di avvio appena sotto l'apice e la posizione di arresto rispetto all'arco aortico.
- Imposta la dimensione del passo su 0,08-0,13 mm e la frequenza fotogrammi su 200-300 Hz.
NOTA: Passi di dimensioni inferiori possono fornire una migliore risoluzione dell'immagine per l'analisi, ma aumenteranno il tempo di acquisizione. Passi di dimensioni maggiori sono accettabili se i dati vengono raccolti con un allineamento ventricolare sinistro quasi perfetto e se durante l'analisi è necessario un riorientamento minimo/nullo. Prendi in considerazione l'aumento della frequenza dei fotogrammi quando lavori con animali con frequenze cardiache più elevate (>450 bpm). - Assicurarsi che i segni vitali e il segnale ECG siano costantemente stabili (>350 bpm e respirazione superiore a 50 resp/min) prima di eseguire la scansione.
- Una volta completate la scansione e l'elaborazione, attivare l'opzione Salva dati EKV/4D per la post-elaborazione e il Gating della respirazione .
- Seleziona Nome immagine in basso a destra e includi l'ID del mouse nel nome.
- Per controllare la qualità dell'immagine, premi Altri controlli e seleziona Carica in 4D.
- Rivedi ogni vista piana del cuore, assicurandoti che il centro del cuore non si muova durante il ciclo cardiaco. Il movimento del centro indica variabilità nell'ECG e/o nel gating respiratorio e quindi complica il processo di analisi. Regolare nuovamente i monitor e ripetere la scansione se necessario.
- Ecografia cardiaca 4D
- Passare al browser dello studio. Esporta tutte le immagini 2D B-Mode su Vevo Lab ed esporta i dati 4D come altro tipo di file, selezionando il formato dati ".raw" su un disco rigido.
NOTA: Il toolbox dell'interfaccia utente grafica 4D utilizzato in questo lavoro non è attualmente disponibile pubblicamente. Per richiedere l'accesso e istruzioni dettagliate, contattare il Dr. Craig Goergen, Purdue University. - Realizza un'analisi alternativa dei dati 4D utilizzando il software VevoLab. Per utilizzare questo framework, esportare i dati 4D dalla macchina ai file Vevo Lab.
- Passare al browser dello studio. Esporta tutte le immagini 2D B-Mode su Vevo Lab ed esporta i dati 4D come altro tipo di file, selezionando il formato dati ".raw" su un disco rigido.
- Imaging vascolare
- Per le immagini in modalità B delle arterie carotidi, posizionare il trasduttore a ultrasuoni parallelamente alle arterie carotidi, vicino alla linea mediana.
- Una volta che l'arteria carotide è visibile, spostare il trasduttore verso la parte superiore per trovare la biforcazione carotidea.
- Quando viene trovata un'immagine chiara della biforcazione carotidea, regolare l'impostazione del guadagno su 35 dB e acquisire l'immagine in modalità B.
- Inclinare il trasduttore a sinistra o a destra può migliorare l'immagine. Regolare l'angolo del trasduttore o dello stadio in modo che l'imbarcazione di interesse non sia direttamente parallela al trasduttore. Ciò consente all'angolo Doppler di rimanere al di sotto dei 60°. In caso contrario, potrebbe essere necessario uno spostamento di +/- 15°.
- Passa alla modalità Doppler a onde pulsate per ottenere le misurazioni della velocità.
- Posizionare il volume del campione al centro del recipiente. Regolare l'angolo del cursore in modo che sia parallelo all'arteria carotide. Quindi, premi play per avviare le misurazioni della velocità e salva per registrarle.
- Per l'immagine in modalità B delle vene giugulari, posizionare il trasduttore a ultrasuoni parallelamente alle vene giugulari. Le vene giugulari sono anteriori e laterali alle arterie carotidi.
- Una volta individuata la vena giugulare, spostare il trasduttore nella posizione in cui la giugulare interna ed esterna si fondono. Quando viene trovata un'immagine nitida, regolare l'impostazione del guadagno su 35 dB e acquisire l'immagine in modalità B.
- Passa alla modalità Doppler a onde pulsate per ottenere le misurazioni della velocità.
- Posizionare il volume del campione al centro del recipiente. Regolare l'angolo del cursore in modo che sia parallelo alla vena giugulare. Quindi, premi play per avviare le misurazioni della velocità e salva per registrarle.
- Ecografia vascolare
- Dopo aver raccolto le immagini in modalità B e le misure Doppler a onde pulsate, misurare i diametri e le velocità utilizzando il software.
- Una volta caricate le immagini nel software, selezionare il pacchetto vascolare.
- Seleziona ogni lato del recipiente per ottenere l'intero diametro.
- Per l'arteria carotide, effettuare misurazioni del diametro in sistole e diastole utilizzando l'opzione del diametro nella confezione vascolare.
- Effettua misurazioni della velocità utilizzando l'opzione di velocità nel pacchetto vascolare.
- Per l'arteria carotide, selezionare il picco più alto per la misurazione della velocità in sistole e il picco più basso per la misurazione della velocità in diastole.
- Utilizzare i valori dei diametri per calcolare la componente circonferenziale del tensore di deformazione di Green Lagrange utilizzando la seguente equazione,
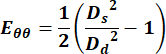 (1)
(1)
dove Ds rappresenta il diametro durante la sistole e Dd rappresenta il diametro durante la diastole. - Per la vena giugulare, selezionare qualsiasi punto del ciclo per ottenere la misurazione della velocità.
2. Eutanasia e prelievo di tessuti
- Preparare le barchette di alluminio per il congelamento rapido dei tessuti (Figura 3).
- Al termine di uno studio, sopprimere l'animale mediante sovradosaggio di isoflurano al 5% di concentrazione e pneumotorace bilaterale o lussazione cervicale (solo studio cardiaco).
- Usando una pinza per tendere la pelle, taglia la pelle a tenda con le forbici sopra il collo per la vascolarizzazione o appena sotto lo sterno per il cardio.
- Tagliare la pelle e gli strati muscolari per esporre il sistema vascolare o tagliare l'osso per esporre il cuore.
- Usando la dissezione smussata con tamponi di cotone, isolare il cuore o il sistema vascolare dai tessuti circostanti, compreso il grasso. Assicurati di separare il vaso carotideo dal nervo. Rimuovere il cuore e i vasi utilizzando strumenti chirurgici.
NOTA: Le suture possono essere utilizzate per rimuovere il sistema vascolare suturando prossimalmente e distalmente prima di rimuovere i vasi. - Posizionare il fazzoletto su una barca di alluminio pre-etichettata e immergere la barca nell'azoto liquido (Figura 3).
- Conservare il tessuto a -80 °C fino al momento della criosezione. Trasportare i tessuti con ghiaccio secco per mantenere la temperatura.
3. Imaging per spettrometria di massa
- Criosezionamento e montaggio della temperatura di taglio ottimale (OCT) su vetrini
- Impostare la temperatura del criostato a -25 °C e inserire la lama.
- Preparare un mandrino di metallo applicando l'OCT e lasciandolo congelare nel criostato.
- Fissare la base del cuore al mandrino di metallo preparato utilizzando l'OCT (Figura 4A). Non permettere all'OCT di toccare la regione del campione di interesse perché l'OCT contaminerà gli spettri di massa (contaminazione da polietilenglicole [PEG]) e deprimerà i segnali lipidici di interesse.
NOTA: Possibilità di eseguire il montaggio ad acqua per evitare la contaminazione del campione. Per istruzioni, vedere il protocollo aggiuntivo riportato di seguito. - Sulla base dell'importanza dell'allineamento spaziale in questa metodologia, utilizzare il rendering digitale in scala del cuore fornito dall'interfaccia grafica di MATLAB per guidare il sezionamento (Figura 4B).
- Contare ogni giro (10 μm) durante il sezionamento e annotare la profondità in millimetri per ogni fetta da montare sulla slitta.
NOTA: È necessario tenere traccia di posizioni specifiche nel cuore o nel sistema vascolare per una corrispondenza accurata con il rendering digitale dei dati ecografici forniti da MATLAB. - Montare le sezioni di tessuto scongelate (10 μm di spessore) sui vetrini del microscopio.
NOTA: Il tipo di vetrini dipenderà dallo spettrometro di massa utilizzato. Ogni diapositiva include una sezione di ciascuna regione del cuore. Sono necessari un minimo di tre vetrini (modalità ioni positivi, modalità ioni negativi, extra). - Per n = 1/gruppo, sezionare il cuore attraverso l'asse lungo per visualizzare i cambiamenti molecolari dall'apice alla base.
- Per la vascolarizzazione, incorporare il tessuto nella gelatina e congelare prima di sezionare11.
- Conservare i vetrini in buste postali a -80°C fino agli esperimenti di imaging con spettrometria di massa.
- Se si esegue cromatografia liquida-spettrometria di massa (LC-MS) per identificare e quantificare ulteriormente lipidi o metaboliti, sezionare sezioni seriali di tessuto di ~60-100 μm e raccoglierle in provette da 0,5-2 ml prima del congelamento.
- Criosezione e montaggio in acqua di tessuti su vetrini
- Riempire un becher con acqua per cromatografia liquida ad alta prestazione (HPLC) e mettere da parte con siringhe da 5 mL e 1 mL.
- Impostare la temperatura del criostato a -25 °C e inserire la lama.
- Posizionare un paio di pinze nel criostato per raffreddarle prima del montaggio.
- Aspirare 5 ml di acqua HPLC in una siringa e inserirla nel criostato.
- Poco prima che la siringa sia completamente congelata, svuotare l'acqua sul mandrino di metallo e lasciarlo congelare completamente.
- Aspirare 1 mL di acqua HPLC e posizionare la siringa nel criostato.
- Dopo circa 30-60 s, posizionare un puntino di acqua parzialmente solidificata al centro del mandrino. Afferra immediatamente il cuore con una pinza e posizionalo nel punto prima che si congeli completamente. Tieni il cuore in posizione fino a quando l'acqua circostante non si è completamente congelata.
NOTA: Il cuore può essere montato all'apice o alla base, a seconda della regione di interesse. Per questo studio, la base sarà montata con l'apice rivolto verso l'esterno. - Eseguire il resto dei passaggi per questo metodo come quelli descritti nel passaggio 3.1.
- Applicazione a matrice
- Togliere il vetrino dal congelatore e metterlo in un essiccatore fino a quando il vetrino non è asciutto.
- Accendi lo spruzzatore HTX M3+, apri l'app HTX sul laptop e seleziona il metodo al centro dello schermo. Le concentrazioni standard della matrice possono essere trovate nei metodi memorizzati (metodi sul lato sinistro dello schermo > matrice OI_usermethods >).
- Per questo lavoro, impostare la temperatura dell'ugello a 75 °C, la portata a 100 μL/min e la pressione a 10 psi.
- Scrivere il nome del campione, la polarità, la matrice, il solvente e la concentrazione sul quaderno di laboratorio. Calcolare la quantità di matrice necessaria per concentrazione (ad esempio, 5 mL a 10 mg/mL = 50 mg di matrice).
- Produrre solvente (ad esempio, 70% di MeOH).
- Per questo lavoro, preparare 40 mg/mL di soluzione di matrice di acido 2,5-diidrossibenzoico (DHB) per la modalità positiva e 10 mg/mL di soluzione di matrice di 9-amminoacridina (9AA) per la modalità negativa. Realizzare entrambe le matrici con un solvente MeOH al 70%. Per altre matrici e parametri di spruzzatura comuni, seguire i passaggi 3.3.2.
- Pesare la matrice e aggiungere la matrice a una provetta conica da 15 mL. Assicurarsi che l'importo sia vicino all'importo richiesto calcolato nel passaggio 3.3.3, ma non deve essere esatto. Calcolare la quantità di solvente in base alla massa misurata.
- Aggiungere il solvente al tubo conico utilizzando una pipetta.
NOTA: Il volume si basa sulla massa misurata al punto 3.3.5. - Sonicare la miscela per 10 minuti. Durante la sonicazione della matrice, rimuovere il vetrino dall'essiccatore.
- Aprire la vaschetta dello spruzzatore, posizionare il vetrino nell'angolo in basso a sinistra e fissare i bordi con del nastro adesivo.
NOTA: Piegare un'estremità del nastro per una facile rimozione. - Selezionare la regione di spruzzatura del campione . Chiudere il vassoio.
- Utilizzare una siringa e un filtro e versare la matrice nella siringa, quindi filtrare la matrice attraverso la siringa nella fiala con il coperchio nero sul lato sinistro dello spruzzatore.
- Riposizionare il flaconcino nella sua posizione sullo spruzzatore e inserire il tubo della linea D nel flaconcino.
NOTA: Assicurarsi che il tubo non tocchi il fondo del flaconcino e che sia completamente immerso nel liquido. - Accendi il gas inerte e assicurati che l'indicatore sullo spruzzatore indichi 10 psi. Premere start. Una volta che lo spruzzatore ha raggiunto la temperatura desiderata, selezionare l'inizio lampeggiante.
- Al termine dello spray, aprire il vassoio, rimuovere il campione e posizionarlo nel supporto per vetrini MALDI o reinserirlo nell'essiccatore (Figura 4C).
- Scansiona il supporto per diapositive MALDI e fai scorrere lo scanner, oppure scatta una foto con un telefono. Salva l'immagine su un'unità flash per utilizzarla con MSI.
- Selezionare il lavaggio appropriato o lo spruzzo successivo sullo spruzzatore. Spostare la linea D dal flaconcino della matrice al becher dei rifiuti durante il lavaggio.
- Spruzzare metanolo sul vassoio e strofinare per pulire. Spegni l'azoto.
- MALDI MSI
- Sul computer, vai su Synapt, controlla che la polarità sia cambiata nella modalità necessaria e cambia in caso contrario.
- Caricare il vetrino nello strumento sulla sorgente MALDI e fare clic sul pulsante di caricamento sul computer utilizzando il software.
- Spostare l'immagine (passaggio 1.2.17) nella cartella Immagini nella cartella Progetto .
- Passa a Imaging HD e apri il progetto precedente (uno che utilizza la stessa matrice).
- In un quaderno da laboratorio, registrare l'intervallo di massa, intrappolare e trasferire l'energia di collisione, l'energia laser, la modalità neg/pos.
- In Imaging HD, accedere all'icona della pagina bianca. Quindi fare clic sulla freccia del menu a discesa e selezionare nuova piastra.
- Sfoglia per trovare l'immagine nella cartella Progetto > nella cartella Immagini e aprila.
- Cambia il tipo di piastra in MALDI Standard e definisci i bordi della piastra (scegli i 4 angoli mostrati di seguito con i segni più rossi).
NOTA: La casella in alto a sinistra serve per navigare verso ogni angolo. La casella in alto a destra serve per selezionare gli angoli. La casella in basso serve solo per visualizzare l'intera diapositiva. - Vai alla piccola scheda del modello nell'angolo in alto a sinistra e fai clic su matita o altre opzioni di rettangolo o cerchio per delineare il tessuto. Delinea il tessuto facendo clic con il pulsante sinistro del mouse e facendo clic con il pulsante destro del mouse per terminare.
- La dimensione predefinita dei pixel è di 50 μm. Cambia se necessario.
- Fare clic su Salva con nome e accedere a Project Imaging AcqFolder ed etichettare come nome della diapositiva (formato AAAAMMGG). Quindi, salva.
- Fai clic sul pulsante Mass Lynx in alto al centro per esportare in Mass Lynx.
- Passare al software Mass Lynx.
- Fare clic su File > aprire la cartella del progetto. Modifica il nome del file e il nome del testo nella diapositiva (formato AAAAMMGG).
- Fare clic con il pulsante destro del mouse sul file MS, sfogliare e scegliere la diapositiva corrente.
- Fare nuovamente clic con il pulsante destro del mouse sul file MS e fare clic su Modifica. Fare doppio clic con il pulsante sinistro del mouse sulla casella blu.
- Passare alle condizioni di scansione. Trovare le impostazioni di origine e passare a quelle definite dall'utente. Quindi passare alla cartella del progetto corretta > cartella acquisita > diapositiva corrente > OKAY.
- Salva cliccando su Salva con nome e includi il nome della diapositiva.
- Calibrare lo strumento utilizzando un composto di riferimento (ad es. fosforo rosso)
- Passare a Synapt (Calibra). Fare clic sul pulsante di riproduzione per avviare l'acquisizione dei dati. Etichetta come YYYYMMDD_cal1 (es: 20230614_cal1).
- Assicurati che abbia lo stesso intervallo di massa 50 - 2000. L'energia del laser può essere di circa 175.
- Premi il fuoco laser play, quindi fai clic sul pulsante successivo . Lasciate correre fino a circa 15 o 20. Fare clic sull'arresto rosso.
- Passare a W Console.
- Creare la calibrazione e fare clic su Avvia. In Editor profilo di calibrazione, fare clic su File > nuovo. Digitare YYYYMMDD_cal1.
- Modificare l'intervallo di massa nell'intervallo di massa desiderato per il progetto (ad esempio, 50-2000).
- Scegli la calibrazione manuale. Scegli la modalità di risoluzione e fai clic su modifica. Selezionare il fosforo rosso come composto di riferimento.
- Fare clic sul file di dati non elaborati e scorrere fino in fondo (dovrebbe essere ciò che è stato appena acquisito).
- Vai alla cronologia in fondo, seleziona Acc mass > va bene > va bene.
- Continua a fare clic su Avanti e OK finché non viene visualizzato il pulsante di avvio e premi Start.
- Passa al segno di spunta verde e guarda quali picchi non sono stati trovati.
- Passare a Mass Lynx (avvia l'esecuzione effettiva per i campioni sperimentali).
- Premi il pulsante di riproduzione e verifica che il numero di campioni sia corretto
- Passare a Imaging HD.
- Verificare che la dimensione dei pixel sia ancora quella impostata in origine
- Passare a Synapt per controllare la polarità.
- Fai clic sul pulsante della fotocamera per assicurarti che si muova e spari il laser.
- Controlla e assicurati che stia raccogliendo dati. Lo spettro di massa acquisito dovrebbe essere visualizzato nella pagina Synapt.
- Analisi di MS
- Utilizza il software HD Imaging e Mass Lynx per l'analisi.
- Elaborare i dati di imaging utilizzando la scheda Processo in Imaging HD.
- Visualizzare le immagini di ioni molecolari, chiamate anche immagini MS, in HD Imaging nella scheda Analisi . Una mappa termica di un'immagine di uno ione molecolare rappresenta l'intensità di quello ione m/z su una regione (Figura 5). Utilizzare le sovrapposizioni di immagini MS per mostrare la distribuzione spaziale delle molecole (Figura 6 e Figura 7).
- Normalizzare i dati utilizzando TIC o la normalizzazione appropriata per l'imaging.
- Identificare i picchi m/z che si correlano spazialmente con le regioni di interesse. Esegui una correlazione spaziale (R > 0,65) su HD Imaging per identificare i picchi co-localizzati.
- Cerca l'identificazione lipidica putativa di questi picchi con LipidMaps16 e METASPACE17.
- Confermare le annotazioni lipidiche con i successivi dati LC-MS. Utilizzare Metaboanalyst per l'analisi statistica dei dati LC-MS.
Risultati
I protocolli di imaging sopra descritti sono stati utilizzati per due studi preliminari: rimodellamento dell'infarto del miocardio (IM) e invecchiamento vascolare. Per gli esperimenti cardiaci, è stato eseguito un intervento chirurgico permanente di legatura dell'arteria coronarica al fine di indurre l'infarto miocardico acuto18,19. L'ecografia 4D e la MALDI MSI sono state eseguite progressivamente sullo stesso tessuto, svelando...
Discussione
L'imaging ecografico può dipendere dall'operatore, ma l'uso di punti di riferimento anatomici e una formazione adeguata possono limitare i pregiudizi dell'utente. Gli ultrasuoni 2D sono particolarmente suscettibili alla variabilità tra gli utenti perché le viste dipendono dall'angolo, mentre il 4DUS è meno suscettibile in quanto l'acquisizione comprende l'intero volume ed è indipendente dall'angolo. È stato inoltre stabilito che la riproducibilità dell'immagine è più facile da o...
Divulgazioni
Craig J. Goergen è un consulente retribuito di FUJIFILM VisualSonics.
Riconoscimenti
Allison Jones è supportata dalla University of Tennessee, Mechanical Aerospace & Biomedical Engineering Department Graduate Fellowship. La ricerca riportata in questa pubblicazione (Conner Earl) è stata supportata dal National Heart, Lung, and Blood Institute del National Institutes of Health F30HL162452. Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente le opinioni ufficiali del National Institutes of Health.
Materiali
| Name | Company | Catalog Number | Comments |
| 2,5-dihydroxybenzoic acid (DHB) | Supelco, >99.0% (HPLC) | 85707-10MG-F | DHB matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sigma/85707?cm_sp=Insite-_-wimsShippingEmailRecs_wims EmailAPI_wimsGruCrossEntropy-_-wimsEmailAPI10-3 |
| 9-aminoacridine (9AA) | Supelco, ≥99.5% (HPLC) | 92817-1G | 9-Aminoacridine matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sial/92817?srsltid=AfmBOooiQjQ4pWv_XxITkU 4Lkm0UnHXKekGS_ dFl7V40V9QLWoPpNLoc |
| Aquasonic Ultrasound Gel | Parker Laboratories | Parker 01-02 | Ultrasound Gel; https://www.parkerlabs.com/products/aquasonic-100-ultrasound-transmission-gel/ |
| Benchtop Dewar Flasks | ThermoScientific | 4150-2000 | Container for liquid nitrogen; https://www.thermofisher.com/order/catalog/product/4150-4000?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sQVc2f-NxN04Nb5Mv F6TZ7GLHWWDEeqDYmEvtKJSQ YHDeVgZ9qylvYaAs27EALw_wcB &source=google_shopping&ISO_ CODE=us&LANG_CODE=en&ef_id =Cj0KCQjwpvK4BhDUARIsADHt9 sQVc2f-NxN04Nb5MvF6TZ7GLHWWDE eqDYmEvtKJSQYHDeVgZ9qylvYa As27EALw_wcB:G:s&s_kwcid=AL!3652 !3!716188292869!!!g!2366243726129 !!21787513085!171591181194&ev_chn =shop&cid=0se_gaw_30092024_ PBYTXL&source=google_shopping &ISO_CODE=us&LANG_CODE= en&gad_source=1 |
| Cryostat | Leica Biosystems | CM Series | https://www.leicabiosystems.com/us/histology-equipment/cryostats/ |
| Dessicator | VWR | 89054-052 | https://us.vwr.com/store/product/9104882/desiccator-plastic-ace-glass-incorporated |
| Epredia MX35 Premier Disposable Low-profile Microtome Blades | Fisher Scientific | 3052835 | Cryostat blade; https://www.fishersci.com/shop/products/mx35-premier-disposable-low-profile-microtome-blades/3052835 |
| Falcon 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | Conical Tubes; https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/1495953A?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sSBcy5n-lhShligJUOX5KKVGn0bt87 8AB2_muOD2PPTue1phpZgeqwa AqgiEALw_wcB&ef_id=Cj0KCQjw pvK4BhDUARIsADHt9sSBcy5n-lhS hligJUOX5KKVGn0bt878AB2_muO D2PPTue1phpZgeqwaAqgiEALw_ wcB:G:s&ppc_id=PLA_goog_20861 45674_81843405034_1495953A__ 386247001345_165426395473886 37329&ev_chn=shop&s_kwcid=AL!4428!3 !386247001345!!!g!856907751004!& gad_source=1 |
| Flex-Tubes Microcentrifuge Tubes | Eppendorf | EP022364120 | Centrifuge tubes; https://www.sigmaaldrich.com/US/en/product/sigma/ep022364120?utm_source=google%2Cgoogle&utm _medium=organicshopping%2Ccpc&utm _campaign=21043330280&utm_ content=&gclid=Cj0KCQjwpv K4BhDUARIsADHt9sTHRD35k CHPtfI2A41axodnMVr6a1eBKk zM4bSUYQAyfEKo3UgTAEQa Ap7wEALw_wcB |
| Gas Nitrogen | Airgas | N/A | |
| Glass microscope slides | Electron Microscopy Sciences | 71873-02 | https://www.emsdiasum.com/positive-charge-microscope-slides |
| Liquid Nitrogen | Airgas | N/A | |
| Mass Spectrometer | Waters | Synapt G2-Si | https://www.waters.com/waters/en_US/SYNAPT-G2-Si-Mass-Spectrometry/nav.htm?locale=en_mkcid=1000251Foodety%3C/a%3E&cid=134740653&bcid= 134528734 |
| Matrix Sprayer | HTX Technologies | M3+ | https://www.htximaging.com/htx-m3-sprayer |
| Methanol (HPLC), >99.9% | Fisher Chemical | A4524 | Methanol; https://www.fishersci.com/shop/products/methanol-hplc-fisher-chemical-9/A4524?crossRef=A4524#?keyword=A4524 |
| Preclinical Ultrasound System | FUJIFILM VisualSonics | Vevo 3100 | https://www.visualsonics.com/product/imaging-systems/vevo-3100; Vevo F2 has replaced the Vevo 3100 in production. System includes isoflurane vaporizer and induction box. |
| Reynolds Wrap | N/A | N/A | Aluminum foil |
| Signagel Electrode Gel | Parker Laboratories | Parker 15-60 | Electrode Conducting Gel; https://www.parkerlabs.com/products/signagel-electrode-gel/ |
| Sterile Lubricating Jelly | Medline | MDS032273Z | Lubricating Gel; https://www.medline.com/ce/product/Sterile-Lubricating-Jelly/Lubricating-Jelly/Z05-PF03664?sku=MDPMDS032273H |
| Surgical instruments: scissors, forceps/tweezers, sutures | Fine Science Tools | 11252-00, 11050-10, 14016-14, 14084-08, 15000-08 | info@finescience.com |
| Surgical Sponges 200 Pack –Gauze Pads Non sterile -First Aid Wound Care Dressing Sponge –Woven Medical Nonstick, Non Adherent Mesh Scrubbing | Medpride | B08RZGQ5GW | Gauze; https://www.amazon.com/Medpride-Surgical-Sponges-200-Pack/dp/B08RZGQ5GW/ref=asc_df_B08RZGQ5GW/?tag=hyprod-20&linkCode=df0&h vadid=693270340506&hvpos= &hvnetw=g&hvrand=960915122 2290977669&hvpone=&hvptwo= &hvqmt=&hvdev=c&hvdvcmdl=& hvlocint=&hvlocphy=9192978&hv targid=pla-1245491514869&psc= 1&mcid=33f4d647c88630c79116 888d565a63b0 |
| Tissue-Plus O.C.T. Compound | Fisher Scientific | 23-730-571 | OCT; https://www.fishersci.com/shop/products/tissue-plus-o-c-t-compound-2/23730571 |
| Wood Handled Cotton Swabs and Applicators | Fisherbrand | 22-363-160 | Cotton swab; https://www.fishersci.com/shop/products/wood-handled-cotton-swabs-applicators-8/p-7146852 |
Riferimenti
- Tsao, C. W., et al. Heart disease and stroke statistics-2022 update: A report from the American Heart Association. Circulation. 145 (8), E153-E639 (2022).
- Kassab, G. S. Biomechanics of the cardiovascular system: The aorta as an illustratory example. J R Soc Interface. 3 (11), 719-740 (2006).
- Moore, J. E., Xu, C., Glagov, S., Zarins, C. K., Ku, D. N. Fluid wall shear stress measurements in a model of the human abdominal aorta: Oscillatory behavior and relationship to atherosclerosis. Atherosclerosis. 110 (2), 225-240 (1994).
- Nerem, R. M. Vascular fluid mechanics, the arterial wall, and atherosclerosis. J Biomech Eng. 114 (3), 274 (1992).
- Cecchi, E., et al. Role of hemodynamic shear stress in cardiovascular disease. Atherosclerosis. 214 (2), 249-256 (2011).
- Good, B. The influence of blood composition and loading conditions on the behavior of embolus analogs. J Mech Behav Biomed Mater. 140, 105738 (2023).
- Bao, G., et al. Molecular biomechanics: The molecular basis of how forces regulate cellular function NIH public access. Mol Cell Biomech. 3 (2), 91-105 (2010).
- Schwamborn, K., Caprioli, R. M. Molecular imaging by mass spectrometry-looking beyond classical histology. Nat Rev Cancer. 10 (9), 639-646 (2010).
- Soepriatna, A. H., Kevin Yeh, A., Clifford, A. D., Bezci, S. E., O'Connell, G. D., Goergen, C. J. Three-dimensional myocardial strain correlates with murine left ventricular remodelling severity post-infarction. J R Soc Interface. 16 (160), 20190570 (2019).
- Damen, F. W., et al. High-frequency 4-dimensional ultrasound (4DUS): A reliable method for assessing murine cardiac function. Tomography. 3 (4), 180-187 (2017).
- McDonald, R., Poulos, D., Gutzwiller, L., Sheth, R. A., Good, B., Crouch, A. C. A MALDI mass spectrometry imaging sample preparation method for venous thrombosis with initial lipid characterization of lab made and murine clots. J Am Soc Mass Spectrom. 34 (9), 1879-1889 (2023).
- Benetos, A., et al. Influence of age, risk factors, and cardiovascular and renal disease on arterial stiffness: Clinical applications. Am J Hypertens. 15 (12), 1101-1108 (2002).
- Pistner, A., Belmonte, S., Coulthard, T., Blaxall, B. C. Murine echocardiography and ultrasound imaging. J Vis Exp. (42), v2100 (2010).
- Bellantuono, I., et al. A toolbox for the longitudinal assessment of healthspan in aging mice. Nat Protoc. 15 (2), 540-574 (2020).
- Henriques, F. C., Moritz, A. R. Studies of thermal injury: I. The conduction of heat to and through skin and the temperatures attained therein. A theoretical and an experimental investigation. Am J Pathol. 23 (4), 530 (1947).
- Conroy, M. J., et al. LIPID MAPS: Update to databases and tools for the lipidomics community. Nucleic Acids Res. 52 (D1), D1677-D1682 (2024).
- Palmer, A., et al. FDR-controlled metabolite annotation for high-resolution imaging mass spectrometry. Nat Methods. 14 (1), 57-60 (2016).
- Michael, L. H., et al. Myocardial ischemia and reperfusion: A murine model. Am J Physiol. 269 (6 Pt 2), H2147-H2154 (1995).
- Clark, J. E., Flavell, R. A., Faircloth, M. E., Davis, R. J., Heads, R. J., Marber, M. S. Post-infarction remodeling is independent of mitogen-activated protein kinase kinase 3 (MKK3). Cardiovasc Res. 75 (3), 523-530 (2007).
- Hattori, Y., Konno, H., Abe, M., Miyoshi, H., Goto, T., Makabe, H. Synthesis, determination of the absolute configuration of tonkinelin, and inhibitory action with bovine heart mitochondrial complex I. Bioorg Med Chem. 15 (8), 3026-3031 (2007).
- Golam Mostofa, M., et al. CLIP and cohibin separate rDNA from nucleolar proteins destined for degradation by nucleophagy. J Cell Biol. 217 (8), 2675-2690 (2018).
- Chan, J. N. Y., Poon, B. P. K., Salvi, J., Olsen, J. B., Emili, A., Mekhail, K. Perinuclear cohibin complexes maintain replicative life span via roles at distinct silent chromatin domains. Dev Cell. 20 (6), 867-879 (2011).
- Dann, M. M., et al. Quantification of murine myocardial infarct size using 2-D and 4-D high-frequency ultrasound. Am J Physiol Heart Circ Physiol. 322 (3), H359-H372 (2022).
- Kaya, I., Sämfors, S., Levin, M., Borén, J., Fletcher, J. S. Multimodal MALDI imaging mass spectrometry reveals spatially correlated lipid and protein changes in mouse heart with acute myocardial infarction. J Am Soc Mass Spectrom. 31 (10), 2133-2142 (2020).
- Tressler, C., et al. Factorial design to optimize matrix spraying parameters for MALDI mass spectrometry imaging. J Am Soc Mass Spectrom. 32 (12), 2728 (2021).
- Zhang, L., et al. Vascular lipidomics analysis reveals increased levels of phosphocholine and lysophosphocholine in atherosclerotic mice. Nutr Metab. 20 (1), 1-16 (2023).
- Kohno, M., et al. Induction by lysophosphatidylcholine, a major phospholipid component of atherogenic lipoproteins, of human coronary artery smooth muscle cell migration. Circulation. 98 (4), 353-359 (1998).
- Crouch, A. C., Batra, A., Greve, J. M. Hemodynamic response to thermal stress varies with sex and age: A murine MRI study. Int J Hyperthermia. 39 (1), 69-80 (2022).
- Crouch, A. C., Scheven, U. M., Greve, J. M. Cross-sectional areas of deep/core veins are smaller at lower core body temperatures. Physiol Rep. 6 (16), e13839 (2018).
- Crouch, A. C., Castle, P. E., Fitzgerald, L., Scheven, U. M., Greve, J. M. Assessing structural and functional response of murine vasculature to acute β-adrenergic stimulation in vivo during hypothermic and hyperthermic conditions. Int J Hyperthermia. 36 (1), 1136-1145 (2019).
- Crouch, A. C., Manders, A. B., Cao, A. A., Scheven, U. M., Greve, J. M. Cross-sectional area of the murine aorta linearly increases with increasing core body temperature. Int J Hyperthermia. 34 (7), 1121-1133 (2018).
- Clift, C. L., Mehta, A., Drake, R. R., Angel, P. M. Multiplexed imaging mass spectrometry of histological staining, N-glycan and extracellular matrix from one tissue section: A tool for fibrosis research. Methods Mol Biol. 2350, 313-329 (2021).
- Salehi, H. S., et al. Coregistered photoacoustic and ultrasound imaging and classification of ovarian cancer: Ex vivo and in vivo studies. J Biomed Opt. 21 (4), 046006 (2016).
- Verbeeck, N., et al. Connecting imaging mass spectrometry and magnetic resonance imaging-based anatomical atlases for automated anatomical interpretation and differential analysis. Biochim Biophys Acta. 1865 (7), 967-977 (2017).
- Yang, E. Y., et al. Real-time co-registration using novel ultrasound technology: Ex vivo validation and in vivo applications. J Am Soc Echocardiogr. 24 (7), 720 (2011).
- Maris, L., et al. Method for co-registration of high-resolution specimen PET-CT with histopathology to improve insight into radiotracer distributions. EJNMMI Phys. 11 (1), 1-20 (2024).
- Lin, B. J., et al. MSIr: Automatic registration service for mass spectrometry imaging and histology. Anal Chem. 95 (6), 3317-3324 (2023).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon