Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Étude multimodale du remodelage cardiovasculaire murin : imagerie quadridimensionnelle par ultrasons et spectrométrie de masse
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici, nous décrivons un protocole pour utiliser l’imagerie par ultrasons quadridimensionnelle in vivo et l’imagerie par spectrométrie de masse ex vivo pour évaluer les altérations biomécaniques et biomoléculaires dans le système cardiovasculaire murin. Cette technique est appliquée pour analyser le remodelage cardiaque dans l’infarctus du myocarde induit chirurgicalement et les changements vasculaires chez les animaux vieillissants.
Résumé
Les maladies cardiovasculaires (MCV) sont la principale cause de décès aux États-Unis. Les dommages au système cardiovasculaire peuvent être dus à une exposition environnementale, à un traumatisme, à la toxicité d’un médicament ou à de nombreux autres facteurs. En conséquence, le tissu cardiaque et le système vasculaire subissent des changements structurels et présentent une fonction diminuée. Les dommages et le remodelage qui en résulte peuvent être détectés et quantifiés à l’aide de l’imagerie par ultrasons (US) au niveau de l’organe et de l’imagerie par spectrométrie de masse (MSI) au niveau moléculaire. Ce manuscrit décrit une méthodologie innovante pour l’étude de la physiopathologie cardiaque murine, couplant l’imagerie et l’analyse par ultrasons quadridimensionnelles (4D) in vivo avec la désorption/ionisation laser assistée par matrice ex vivo (MADLI) MSI du cœur. L’échographie 4D peut fournir des mesures volumétriques dynamiques, y compris le déplacement radial, la déformation de surface et la déformation longitudinale tout au long d’un cycle cardiaque. Dans le système vasculaire, la MSI et les ultrasons sont utilisés pour évaluer la composition de la paroi des vaisseaux, l’hémodynamique et la dynamique de la paroi des vaisseaux. La méthodologie peut être adaptée pour étudier une myriade de maladies cardiovasculaires en ajustant les paramètres fonctionnels d’intérêt et/ou en faisant varier le protocole MALDI MSI pour cibler des molécules spécifiques. MALDI MSI peut être utilisé pour étudier les lipides, les petits métabolites, les peptides et les glycanes. Ce protocole décrit l’utilisation de MALDI MSI pour l’analyse lipidomique non ciblée et l’utilisation de l’imagerie par ultrasons pour l’hémodynamique et la biomécanique cardiovasculaires.
Introduction
Les maladies cardiovasculaires (MCV) sont l’une des principales causes de mortalité dans le monde1. La prévention et le traitement des MCV nécessitent une compréhension approfondie des adaptations moléculaires aux forces biomécaniques et des modifications des propriétés mécaniques qui en résultent. Dans l’ensemble du système cardiovasculaire, les forces biomécaniques jouent un rôle important dans la fonction et la structure du tissu2. Les propriétés mécaniques du tissu cardiovasculaire (CV) sont influencées par ces forces, ce qui en fait des indicateurs de santé et de maladie 3,4,5,6. Pour prévenir, diagnostiquer et traiter les MCV, il est crucial de développer des méthodes pour comprendre et observer les processus d’initiation et de progression de la maladie. L’imagerie biomédicale a joué un rôle clé dans la production d’informations physiologiques et mécanistes, et de nouvelles technologies d’imagerie et techniques d’analyse sont constamment développées. Ce protocole démontre une méthodologie permettant de combiner deux techniques d’imagerie et d’analyse cardiovasculaires afin de valider le potentiel de ces modalités d’imagerie dans les maladies cardiaques ischémiques et le vieillissement vasculaire.
Les chercheurs dans le domaine de la biomécanique abordent souvent l’étude de la biomécanique via une combinaison de méthodes in vivo, ex vivo et in silico . Les recherches antérieures en biomécanique moléculaire se sont principalement concentrées sur les protéines7 (en particulier les protéines de la matrice extracellulaire, le collagène et l’élastine en raison de leur impact sur les propriétés biomécaniques), et les travaux visant à combiner la biomécanique de l’imagerie in vivo avec les études moléculaires se sont limités à l’histologie et à l’immunohistochimie. Bien que ces approches puissent produire de nombreux indicateurs moléculaires et aient permis de proposer des mécanismes de remodelage de l’ECM et des cellules, elles sont généralement limitées aux colorants ou anticorps actuellement disponibles, respectivement. Ce domaine de recherche manque de grandes classes de molécules, par exemple les lipides. Bien que ces classes moléculaires puissent ou non être impliquées mécaniquement, il est important de comprendre les adaptations moléculaires qui en résultent, car ces molécules pourraient être des cibles potentielles pour les marqueurs diagnostiques et les traitements. Des techniques de chimie analytique, telles que la chromatographie liquide couplée à la spectrométrie de masse (LC-MS), peuvent être appliquées ; Cependant, l’orientation spatiale des molécules dans ces techniques est perdue. Avec l’imagerie par spectrométrie de masse (MSI), la distribution spatiale des molécules reste intacte et plusieurs types d’analytes (classes de molécules) peuvent être imagés avec des coupes en série. MSI est un outil analytique puissant pour étudier les distributions spatiales de presque tous les types de molécules dans les tissus biologiques, y compris les métabolites, les lipides, les glycanes, les peptides et les médicaments de faible poids moléculaire8. La désorption/ionisation laser assistée par matrice (MALDI) MSI est un type de MSI bien adapté à l’analyse basée sur la découverte de poids moléculaires dans la gamme de 50 à 8000 Da. MALDI-MSI est une technique d’ionisation qui applique une matrice d’absorption d’énergie laser à l’échantillon pour faciliter l’ionisation des analytes d’intérêt. Cette approche évite d’être limitée à une cible moléculaire et permet d’utiliser des outils bioinformatiques pour déterminer quelles molécules ont un impact sur les propriétés biomécaniques et le remodelage.
L’échographie quadridimensionnelle (4DUS) est une méthode in vivo non invasive utile pour la caractérisation temporelle et spatiale du cœur. 4DUS utilise une série de boucles cinéma à fréquence d’images élevée provenant de différents plans, les compilant dans un ensemble de données 3D qui comprend des informations temporelles. Cela permet de visualiser et de quantifier directement les changements de forme 3D complexes des cavités cardiaques au cours du cycle cardiaque sans s’appuyer sur des hypothèses géométriques comme l’exige l’échocardiographie 2D traditionnelle. 4DUS permet de calculer des paramètres fonctionnels in vivo à partir de la forme et du mouvement complexes du cœur 9,10, et MALDI MSI permet l’étude spatiale de molécules biologiques dans le tissu cardiaque ex vivo11. Pour bien comprendre les altérations cardiaques atteintes de MCV, il faut étudier les mécanismes mécaniques et moléculaires. Ainsi, une méthodologie combinée est proposée pour l’étude de la physiopathologie cardiaque murine, couplant l’imagerie et l’analyse 4DUS avec MALDI MSI des lipides dans le cœur. Cette méthodologie est démontrée dans un modèle murin d’infarctus du myocarde.
La biomécanique vasculaire joue également un rôle essentiel dans la régulation de la fonction cardiovasculaire2. Le raidissement vasculaire, associé au vieillissement, est un facteur de risque de MCV12. Les changements biomécaniques et hémodynamiques dans les vaisseaux peuvent être imagés à l’aide d’échographie. Les compositions moléculaires des parois des vaisseaux sont des composants importants de la biomécanique et sont également extrêmement sensibles aux forces hémodynamiques. Par exemple, la contrainte de cisaillement de la paroi oscillatoire a été impliquée dans le développement de la plaque d’athérosclérose3. Les données préliminaires de la mécanique des vaisseaux et de l’hémodynamique chez les animaux âgés seront présentées ultérieurement.
L’équipe s’intéresse à la relation entre la biomécanique et la composition moléculaire dans divers états pathologiques. L’imagerie par ultrasons précliniques et la MSI sont utilisées pour déterminer la distribution spatiale des changements moléculaires dans un tissu et les changements biomécaniques associés qui se produisent au cours de la progression de la maladie. Ce rapport décrit en détail ces méthodologies et présente des données préliminaires sur le cœur et le système vasculaire de la tête et du cou.
Access restricted. Please log in or start a trial to view this content.
Protocole
Les expériences sur les animaux décrites sont réalisées avec l’approbation du comité institutionnel de l’Université du Tennessee, Knoxville, sur le soin et l’utilisation des animaux.
1. Imagerie par ultrasons13
- Installation et préparation des animaux
- Avant de commencer l’expérience, vérifiez le niveau d’anesthésie à l’isoflurane et remplissez-le si nécessaire. Vérifiez les niveaux des réservoirs d’oxygène avant d’allumer le gaz. Pesez le charbon de bois et notez-le sur le récipient.
- Installez l’espace d’imagerie en plaçant les fournitures à proximité de la plate-forme d’imagerie. Les fournitures comprennent du ruban prédécoupé, de la crème dépilatoire, de la gaze, des cotons-tiges, de l’eau autoclave, du lubrifiant pour cornée, du gel lubrifiant, une sonde de température rectale, des fils d’électrodes, du gel à ultrasons, du gel d’électrode (le cas échéant) et une lampe chauffante (figure 1A).
- Allumez l’échographe et initialisez le moteur (le système est également capable de collecter des données d’image 4DUS). Pour configurer une nouvelle expérience sur l’échographe : sélectionnez Nouvelle étude ou Nouvelle série et remplissez les informations sur la souris. Allumez la tablette pour le suivi physiologique.
- Vérifiez le robinet d’arrêt pour vous assurer que le débit est ouvert au boîtier d’induction et fermé au cône de nez. Pré-remplissez la boîte d’induction de l’anesthésie en allumant l’oxygène à 1 L/min sur le vaporisateur et en tournant le cadran à 3 %-5 %.
- Retirez la souris de la cage du boîtier.
- Placez la souris dans la boîte d’induction jusqu’à ce que l’animal soit complètement anesthésié avec de l’isoflurane entre 3 % et 5 %.
- Basculez le robinet d’arrêt pour changer le débit vers le cône nasal sur la plaque d’imagerie à ultrasons. Tournez le cadran de l’isoflurane entre 1 % et 2 %.
- Retirez la souris de la boîte à induction et pesez l’animal. Notez le poids de l’animal.
- Placez la souris en position couchée sur la plaque d’imagerie.
- Appliquez un lubrifiant cornéen sur les yeux de l’animal.
- S’il y a de la fourrure, retirez la fourrure de la zone d’imagerie à l’aide d’une crème dépilatoire disponible dans le commerce et de cotons-tiges. Essuyez la crème de la peau avec de l’eau sur des éponges pour éliminer tout excès de crème dépilatoire de la peau et prévenir les brûlures.
- Insérez les dérivations d’électrocardiographie (ECG) par voie sous-cutanée de chaque côté du thorax et des membres postérieurs, en fonction du positionnement de l’animal.
- Une fois insérés, maintenez les dérivations ECG en place à l’aide de ruban adhésif. Si les dérivations ECG ne sont pas utilisées (c’est-à-dire que les sondes ECG ne sont pas insérées), surveillez l’ECG via la plaque d’imagerie animale.
- Pour la conduction électrique de la peau à la plaque, repérez le gel conducteur sur la plaque et maintenez les membres de l’animal en position.
- Maintenez les membres de l’animal en place à l’aide d’un ruban adhésif ou d’un élastique placé autour de la plaque et sur les membres (figure 1B).
- Utilisez du gel lubrifiant pour insérer la sonde de température rectale et fixez la sonde avec du ruban adhésif.
- Vérifiez les signaux ECG, respiratoires et de température sur la tablette. Assurez-vous que la fréquence cardiaque est de ~350-600 bpm, la respiration de ~50-100 respirations/min et la température rectale entre 35-37 °C. Ajustez les niveaux d’isoflurane au besoin.
- Au besoin, allumez une lampe chauffante (p. ex., une ampoule chauffante infrarouge de 250 W)14 et ajustez la hauteur pour maintenir la température centrale (figure 1C).
- Placez la lampe chauffante à moins de 12 pouces de l’animal. Pour les études initiales visant à déterminer la distance optimale entre l’ampoule et l’animal, ajustez la hauteur et enregistrez la température de la peau à l’aide d’un thermomètre infrarouge, qui ne doit pas dépasser 42 °C15.
- Échographie cardiaque 4D
- Placez le transducteur dans le support en position semi-verrouillée.
- Orientez le point en relief sur le transducteur avec le point bleu sur l’écran. La convention est de placer le point vers le côté droit de l’animal.
- Tournez le transducteur de manière à ce qu’il soit orienté le long du plan sagittal de la souris avec une encoche surélevée pointant dans la direction caudale.
- Utilisez le levier à la base de la plaque d’échographie pour incliner l’animal. Utilisez le levier situé à la base de la sonde pour régler l’angle de la sonde.
- Appliquez une quantité généreuse de gel à ultrasons sur la surface ventrale de la cavité thoracique pour le couplage acoustique entre la surface de la peau et le transducteur. Assurez-vous qu’il n’y a pas de fourrure restante avant l’accouplement.
- Abaissez le transducteur pour entrer en contact avec le gel à ultrasons.
- Faites des micro-ajustements avec les boutons X/Y à la base de la plaque, ou déplacez toute la plaque pour des ajustements importants.
- Assurez-vous qu’une vue parasternale grand angle comprend l’apex, la voie d’éjection ventriculaire gauche et l’aorte alignées horizontalement sur l’écran pour une image plus précise sur l’axe court (figures 2A, B).
- Sélectionnez Nom de l’image dans le coin inférieur pour enregistrer l’image dans la série actuelle.
- Tournez la sonde de 90° dans le sens des aiguilles d’une montre pour passer à une vue parasternale à court axe (Figure 2C).
REMARQUE : Pour une meilleure qualité d’image 4D, les utilisateurs doivent essayer d’aligner le moteur pas à pas le long axe parasternal du ventricule qui n’est pas toujours directement parallèle au sternum. Le cœur/VG se trouve souvent à un angle gauche vers le bas. - Assurez-vous que le ventricule gauche est visible sur le côté droit de l’écran et que le ventricule droit se trouve sur le côté gauche de l’écran (Figure 2D).
- Si la souris a un rythme respiratoire stable, appuyez sur la case ECG dans le coin supérieur gauche de l’image pour activer le déclenchement respiratoire pour l’image 4D. S’il n’est pas stable, terminez cette étape lors du post-traitement.
- Sélectionnez le cube dans le coin supérieur gauche de l’écran pour configurer une image 4D.
- Réinitialisez la sonde avant de régler les positions Start et Stop de la sonde.
- Ajustez la position de départ juste en dessous de l’apex et la position d’arrêt à l’arc aortique.
- Réglez la taille du pas sur 0,08-0,13 mm et la fréquence d’images sur 200-300 Hz.
REMARQUE : Des pas plus petits peuvent offrir une meilleure résolution d’image pour l’analyse, mais augmenteront le temps d’acquisition. Des pas plus grands sont acceptables si les données sont recueillies avec un alignement ventriculaire gauche presque parfait et qu’une réorientation minimale, voire nulle, est nécessaire pendant l’analyse. Envisagez d’augmenter la fréquence d’images lorsque vous travaillez avec des animaux ayant des fréquences cardiaques plus élevées (>450 bpm). - Assurez-vous que les signes vitaux et le signal ECG sont constamment stables (>350 bpm et respiration supérieure à 50 resp/min) avant de passer l’examen.
- Une fois le balayage et le traitement terminés, activez l’option Enregistrer les données EKV/4D pour le post-traitement et l’activation du contrôle respiratoire .
- Sélectionnez Nom de l’image en bas à droite et incluez l’ID de la souris dans le nom.
- Pour vérifier la qualité de l’image, appuyez sur Plus de commandes et sélectionnez Charger dans 4D.
- Examinez chaque vue plane du cœur, en vous assurant que le centre du cœur ne se déplace pas tout au long du cycle cardiaque. Le mouvement du centre indique une variabilité de l’ECG et/ou du déclenchement respiratoire et complique ainsi le processus d’analyse. Réajustez les moniteurs et répétez le balayage si nécessaire.
- Analyse échographique cardiaque 4D
- Accédez au navigateur de l’étude. Exportez toutes les images 2D en mode B vers Vevo Lab et exportez les données 4D comme autre type de fichier, en sélectionnant le format de données « .raw » sur un disque dur.
REMARQUE : La boîte à outils de l’interface utilisateur graphique 4D utilisée dans ce travail n’est actuellement pas accessible au public. Pour demander l’accès et des instructions détaillées, contactez le Dr Craig Goergen, de l’Université Purdue. - Réalisez une analyse alternative de données 4D à l’aide du logiciel VevoLab. Pour utiliser ce cadre, exportez les données 4D de la machine vers les fichiers Vevo Lab.
- Accédez au navigateur de l’étude. Exportez toutes les images 2D en mode B vers Vevo Lab et exportez les données 4D comme autre type de fichier, en sélectionnant le format de données « .raw » sur un disque dur.
- Imagerie vasculaire
- Pour obtenir des images en mode B des artères carotides, placez le transducteur à ultrasons parallèlement aux artères carotides, près de la ligne médiane.
- Une fois que l’artère carotide est visible, déplacez le transducteur vers le haut pour trouver la bifurcation carotidienne.
- Lorsqu’une image claire de la bifurcation carotidienne est trouvée, réglez le réglage de gain sur 35 dB et capturez l’image en mode B.
- L’inclinaison du transducteur vers la gauche ou la droite peut améliorer l’image. Ajustez l’angle de la sonde ou de la platine de sorte que le récipient d’intérêt ne soit pas directement parallèle à la sonde. Cela permet à l’angle Doppler de rester inférieur à 60°. Sinon, un décalage de +/- 15° peut être nécessaire.
- Passez en mode Doppler à ondes pulsées pour obtenir les mesures de vitesse.
- Placez le volume de l’échantillon au centre du récipient. Ajustez l’angle du curseur de manière à ce qu’il soit parallèle à l’artère carotide. Ensuite, appuyez sur play pour démarrer les mesures de vélocité et enregistrez-les pour les enregistrer.
- Pour l’image en mode B des veines jugulaires, placez le transducteur à ultrasons parallèlement aux veines jugulaires. Les veines jugulaires sont antérieures et latérales aux artères carotides.
- Une fois la veine jugulaire localisée, déplacez le transducteur à l’endroit où la jugulaire interne et externe se confondent. Lorsque vous trouvez une image claire, réglez le réglage de gain sur 35 dB et capturez l’image en mode B.
- Passez en mode Doppler à ondes pulsées pour obtenir les mesures de vitesse.
- Placez le volume de l’échantillon au centre du récipient. Ajustez l’angle du curseur de manière à ce qu’il soit parallèle à la veine jugulaire. Ensuite, appuyez sur play pour démarrer les mesures de vélocité et enregistrez-les pour les enregistrer.
- Analyse par échographie vasculaire
- Après avoir collecté les images en mode B et les mesures Doppler à ondes pulsées, mesurez les diamètres et les vitesses à l’aide du logiciel.
- Une fois les images chargées dans le logiciel, sélectionnez le package vasculaire.
- Sélectionnez chaque côté du récipient pour obtenir le diamètre complet.
- Pour l’artère carotide, effectuez des mesures de diamètre en systole et en diastole à l’aide de l’option de diamètre dans l’emballage vasculaire.
- Effectuez des mesures de vitesse à l’aide de l’option de vitesse dans l’emballage vasculaire.
- Pour l’artère carotide, sélectionnez le pic le plus élevé pour la mesure de la vitesse en systole et le pic le plus bas pour la mesure de la vitesse en diastole.
- À l’aide des valeurs des diamètres, calculer la composante circonférentielle du tenseur de déformation de Green Lagrange à l’aide de l’équation suivante,
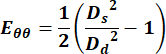 (1)
(1)
où Ds représente le diamètre pendant la systole et Dd représente le diamètre pendant la diastole. - Pour la veine jugulaire, sélectionnez n’importe quel point du cycle pour obtenir la mesure de la vitesse.
2. Euthanasie et prélèvement de tissus
- Préparez des bateaux en papier d’aluminium pour la congélation instantanée des tissus (figure 3).
- À la fin de l’étude, euthanasier l’animal par surdosage d’isoflurane à une concentration de 5 % et soit par pneumothorax bilatéral, soit par luxation cervicale (étude cardiaque seulement).
- À l’aide d’une pince pour tenter la peau, coupez la peau de la tente avec des ciseaux sur le cou pour le système vasculaire ou juste en dessous du sternum pour le cardiaque.
- Coupez la peau et les couches musculaires pour exposer le système vasculaire ou coupez l’os pour exposer le cœur.
- À l’aide d’une dissection contondante avec des cotons-tiges, isolez le cœur ou le système vasculaire des tissus environnants, y compris la graisse. Assurez-vous de séparer le vaisseau carotidien du nerf. Retirez le cœur et les vaisseaux à l’aide d’outils chirurgicaux.
REMARQUE : Des sutures peuvent être utilisées pour enlever le système vasculaire en suturant proximal et distal avant d’enlever les vaisseaux. - Placez le mouchoir sur un bateau en papier d’aluminium pré-étiqueté et placez le bateau dans de l’azote liquide (Figure 3).
- Conservez les tissus à -80 °C jusqu’à l’heure de la cryosection. Transportez les tissus avec de la glace sèche pour maintenir la température.
3. Imagerie par spectrométrie de masse
- Cryosectionnement et montage à température de coupe optimale (OCT) sur lames de verre
- Réglez la température du cryostat sur -25 °C et insérez la lame.
- Préparez un mandrin métallique en appliquant de l’OCT et en le laissant congeler dans le cryostat.
- Fixez la base du cœur au mandrin métallique préparé à l’aide de l’OCT (Figure 4A). Ne laissez pas l’OCT toucher la région d’intérêt de l’échantillon, car l’OCT contaminera les spectres de masse (contamination par le polyéthylène glycol [PEG]) et réduira les signaux lipidiques d’intérêt.
REMARQUE : Possibilité d’effectuer le montage à l’eau pour éviter la contamination de l’échantillon. Consultez le protocole supplémentaire ci-dessous pour obtenir des instructions. - Compte tenu de l’importance de l’alignement spatial dans cette méthodologie, utilisez le rendu numérique à l’échelle du cœur fourni par l’interface graphique de MATLAB pour guider le sectionnement (Figure 4B).
- Comptez chaque tour (10 μm) pendant la coupe et notez la profondeur en millimètres de chaque tranche à monter sur la lame.
REMARQUE : il est nécessaire de garder une trace de positions spécifiques dans le cœur ou le système vasculaire pour une correspondance précise avec le rendu numérique des données d’échographie fourni par MATLAB. - Décongeler des coupes de tissu (10 μm d’épaisseur) sur des lames de microscope.
REMARQUE : Le type de lames dépendra du spectromètre de masse utilisé. Chaque diapositive comprend une section de chaque région du cœur. Un minimum de trois lames est requis (mode ions positifs, mode ions négatifs, extra). - Pour n = 1/groupe, sectionnez le cœur via le grand axe pour visualiser les changements moléculaires de l’apex à la base.
- Pour la vascularisation, incorporez le tissu dans de la gélatine et congelez-le avant de sectionner11.
- Conservez les lames dans des enveloppes de lames à -80°C jusqu’aux expériences d’imagerie par spectrométrie de masse.
- Si vous effectuez une chromatographie en phase liquide couplée spectrométrie de masse (LC-MS) pour identifier et quantifier davantage les lipides ou les métabolites, sectionnez en série des coupes de tissus de ~60-100 μm et collectez-les dans des tubes de 0,5 à 2 mL avant de congeler.
- Cryosectionnement et montage de tissus à l’eau sur des lames de verre
- Remplissez un bécher avec de l’eau de chromatographie liquide à haute performance (CLHP) et réservez avec des seringues de 5 ml et 1 ml.
- Réglez la température du cryostat sur -25 °C et insérez la lame.
- Placez une paire de pinces dans le cryostat pour qu’il refroidisse avant de le monter.
- Prélevez 5 ml d’eau HPLC dans une seringue et placez-la dans le cryostat.
- Juste avant que la seringue ne soit complètement congelée, videz l’eau sur le mandrin en métal et laissez-la geler complètement.
- Prélevez 1 mL d’eau HPLC et placez la seringue dans le cryostat.
- Après environ 30 à 60 s, placez un petit point d’eau partiellement solidifiée au centre du mandrin. Saisissez immédiatement le cœur avec une pince et placez-le dans le point avant qu’il ne gèle complètement. Maintenez le cœur en place jusqu’à ce que l’eau environnante soit complètement gelée.
REMARQUE : Le cœur peut être monté par l’apex ou la base, selon la région d’intérêt. Pour cette étude, la base sera montée avec le sommet vers l’extérieur. - Effectuez le reste des étapes de cette méthode comme décrit à l’étape 3.1.
- Application matricielle
- Sortez la lame du congélateur et placez-la dans un dessiccateur jusqu’à ce qu’elle soit sèche.
- Allumez le pulvérisateur HTX M3+, ouvrez l’application HTX sur l’ordinateur portable et sélectionnez la méthode au milieu de l’écran. Les concentrations matricielles standard se trouvent dans les méthodes stockées (méthodes sur le côté gauche de l’écran > matrice OI_usermethods >).
- Pour ce travail, réglez la température de la buse à 75 °C, le débit à 100 μL/min et la pression à 10 psi.
- Notez le nom de l’échantillon, la polarité, la matrice, le solvant et la concentration dans le cahier de laboratoire. Calculer la quantité de matrice nécessaire par concentration (p. ex., 5 mL à 10 mg/mL = 50 mg de matrice).
- Fabriquer du solvant (par exemple, 70 % de MeOH).
- Pour ce travail, préparer une solution matricielle d’acide 2,5-dihydroxybenzoïque (DHB) à 40 mg/mL pour le mode positif et une solution matricielle de 9-aminoacridine (9AA) à 10 mg/mL pour le mode négatif. Fabriquez les deux matrices avec un solvant MeOH à 70 %. Pour d’autres matrices et paramètres de pulvérisation courants, suivez les étapes 3.3.2.
- Pesez la matrice et ajoutez-la dans un tube conique de 15 ml. Assurez-vous que le montant est proche du montant requis calculé à l’étape 3.3.3, mais qu’il n’est pas nécessaire qu’il soit exact. Calculez la quantité de solvant en fonction de la masse mesurée.
- Ajouter le solvant dans le tube conique à l’aide d’une pipette.
REMARQUE : Le volume est basé sur la masse mesurée à l’étape 3.3.5. - Sonicer le mélange pendant 10 min. Pendant que la matrice est sonicée, retirez la lame du dessiccateur.
- Ouvrez le plateau du pulvérisateur, placez la glissière dans le coin inférieur gauche et fixez du ruban adhésif sur les bords.
REMARQUE : Pliez une extrémité du ruban pour un retrait facile. - Sélectionnez la région de pulvérisation de l’échantillon . Fermez le plateau.
- Utilisez une seringue et un filtre et versez la matrice dans la seringue, puis filtrez la matrice à travers la seringue dans le flacon avec le couvercle noir sur le côté gauche du pulvérisateur.
- Remettez le flacon à sa place sur le pulvérisateur et insérez le tube de la ligne D dans le flacon.
REMARQUE : Assurez-vous que le tube ne touche pas le fond du flacon et qu’il est complètement immergé dans le liquide. - Allumez le gaz inerte et assurez-vous que la jauge du pulvérisateur indique 10 psi. Appuyez sur start. Une fois que le pulvérisateur est à la température souhaitée, sélectionnez le début du clignotement.
- Une fois la pulvérisation terminée, ouvrez le plateau, retirez l’échantillon et placez-le dans le support de lames MALDI ou dans le dessiccateur (Figure 4C).
- Numérisez le support de diapositives MALDI et faites-le glisser sur le scanner, ou prenez une photo avec un téléphone. Enregistrez l’image sur une clé USB pour l’utiliser avec MSI.
- Sélectionnez le lavage approprié ou la prochaine pulvérisation sur le pulvérisateur. Déplacez la ligne D du flacon matriciel dans le bécher à déchets si vous lavez.
- Vaporisez du méthanol sur le plateau et essuyez pour nettoyer. Désactivez l’azote.
- MALDI MSI
- Sur l’ordinateur, accédez à Synapt, vérifiez que la polarité est modifiée sur le mode nécessaire, et basculez si ce n’est pas le cas.
- Chargez la diapositive dans l’instrument sur la source MALDI et cliquez sur le bouton de chargement de l’ordinateur à l’aide du logiciel.
- Déplacez l’image (étape 1.2.17) dans le dossier Images du dossier Projet .
- Accédez à Imagerie HD et ouvrez le projet précédent (qui utilise la même matrice).
- Dans un cahier de laboratoire, enregistrez la plage de masse, piégez et transférez l’énergie de collision, l’énergie laser, le mode neg/pos.
- Dans l’imagerie HD, accédez à l’icône de la page blanche. Cliquez ensuite sur la flèche déroulante et sélectionnez une nouvelle plaque.
- Recherchez l’image dans le dossier du projet > le dossier des images et ouvrez-la.
- Changez le type de plaque en MALDI Standard et définissez les bords de la plaque (choisissez les 4 coins indiqués ci-dessous avec des signes plus rouges).
REMARQUE : La case en haut à gauche permet de naviguer vers chaque coin. La case en haut à droite permet de sélectionner les coins. La boîte du bas sert simplement à visualiser l’ensemble de la diapositive. - Accédez au petit onglet motif dans le coin supérieur gauche et cliquez sur le crayon ou sur d’autres options de rectangle ou de cercle pour délimiter le tissu. Décrivez le mouchoir à l’aide des clics gauche et du clic droit pour terminer.
- La taille de pixel par défaut est de 50 μm. Changez si nécessaire.
- Cliquez sur Enregistrer sous et accédez à Project Imaging AcqFolder et étiquetez-le comme nom de la diapositive (format AAAAMMJJ). Ensuite, enregistrez.
- Cliquez sur le bouton Mass Lynx en haut au milieu pour exporter vers Mass Lynx.
- Accédez au logiciel Mass Lynx.
- Cliquez sur Fichier > ouvrir le dossier du projet. Modifiez le nom du fichier et le nom du texte de la diapositive (format AAAAMMJJ).
- Faites un clic droit sur le fichier MS, parcourez et choisissez la diapositive actuelle.
- Faites un clic droit à nouveau sur le fichier MS et cliquez sur Modifier. Faites un double clic gauche sur la case bleue.
- Accédez aux conditions d’analyse. Recherchez les paramètres source et passez à défini par l’utilisateur. Accédez ensuite au dossier de projet approprié > au dossier acquis > diapositive actuelle > OK.
- Enregistrez en cliquant sur Enregistrer sous et incluez le nom de la diapositive.
- Étalonnez l’instrument à l’aide d’un composé de référence (p. ex., phosphore rouge)
- Accédez à Synapt (Calibrer). Cliquez sur le bouton de lecture pour commencer à acquérir des données. Étiquetez comme YYYYMMDD_cal1 (ex : 20230614_cal1).
- Assurez-vous qu’il a la même plage de masse 50 - 2000. L’énergie laser peut être d’environ 175.
- Appuyez sur fire laser play, puis cliquez sur le bouton suivant . Laissez-le fonctionner jusqu’à environ 15 ou 20. Cliquez sur stop rouge.
- Accédez à la console W.
- Créez l’étalonnage et cliquez sur Démarrer. Sous l’éditeur de profil d’étalonnage, cliquez sur > fichier nouveau. Tapez YYYYMMDD_cal1.
- Modifiez la plage de masse en fonction de la plage de masse souhaitée pour le projet (par exemple, 50-2000).
- Choisissez l’étalonnage manuel. Choisissez le mode de résolution et cliquez sur modifier. Choisissez le phosphore rouge comme composé de référence.
- Cliquez sur le fichier de données brutes et faites défiler vers le bas (il doit s’agir de ce qui vient d’être acquis).
- Allez à l’historique en bas, sélectionnez Acc mass > ok > okay.
- Continuez à cliquer sur suivant et d’accord jusqu’à ce que le bouton de démarrage soit vu, puis appuyez sur Démarrer.
- Naviguez jusqu’à la coche verte et regardez quels sommets n’ont pas été trouvés.
- Accédez à Mass Lynx (Démarrez l’exécution réelle des échantillons expérimentaux).
- Appuyez sur le bouton de lecture et vérifiez que le nombre d’échantillons est correct
- Accédez à l’imagerie HD.
- Assurez-vous que la taille des pixels est toujours celle définie à l’origine
- Accédez à Synapt pour vérifier la polarité.
- Cliquez sur le bouton de la caméra pour vous assurer qu’elle se déplace et tire le laser.
- Vérifiez et assurez-vous qu’il collecte des données. Le spectre de masse acquis doit être affiché sur la page Synapt.
- Analyse MS
- Utilisez le logiciel d’imagerie HD et Mass Lynx pour l’analyse.
- Données d’imagerie de processus à l’aide de l’onglet Processus de l’imagerie HD.
- Visualisez des images d’ions moléculaires, également appelées images MS, dans l’imagerie HD dans l’onglet Analyse . Une carte thermique d’une image d’ion moléculaire représente l’intensité de cet ion m/z sur une région (Figure 5). Utilisez les superpositions d’images MS pour montrer la distribution spatiale des molécules (Figure 6 et Figure 7).
- Normalisez les données à l’aide de TIC ou d’une normalisation appropriée pour l’imagerie.
- Identifiez les pics m/z qui sont en corrélation spatiale avec les régions d’intérêt. Exécutez une corrélation spatiale (R > 0,65) sur l’imagerie HD pour identifier les pics co-localisés.
- Recherche d’identification lipidique présumée de ces pics avec LipidMaps16 et METASPACE17.
- Confirmez les annotations lipidiques avec les données LC-MS ultérieures. Utilisez Metaboanalyst pour l’analyse statistique des données LC-MS.
Access restricted. Please log in or start a trial to view this content.
Résultats
Les protocoles d’imagerie décrits ci-dessus ont été utilisés pour deux études préliminaires : le remodelage de l’infarctus du myocarde (IM) et le vieillissement vasculaire. Pour les expériences cardiaques, une chirurgie permanente de ligature de l’artère coronaire a été pratiquée afin d’induire un infarctus aigu du myocarde18,19. L’échographie 4D et le MALDI MSI ont été réalisés progressivement sur le m?...
Access restricted. Please log in or start a trial to view this content.
Discussion
L’imagerie échographique peut dépendre de l’opérateur, mais l’utilisation de repères anatomiques et une formation adéquate peuvent limiter les biais de l’utilisateur. L’échographie 2D est particulièrement sensible à la variabilité entre les utilisateurs car les vues dépendent de l’angle, tandis que l’échographie 4DUS est moins sensible car l’acquisition englobe tout le volume et est indépendante de l’angle. Il a également été déterminé que la reproducti...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Craig J. Goergen est un consultant rémunéré de FUJIFILM VisualSonics.
Remerciements
Allison Jones est soutenue par la bourse d’études supérieures du département de génie mécanique, aérospatial et biomédical de l’Université du Tennessee. La recherche rapportée dans cette publication (Conner Earl) a été soutenue par le National Heart, Lung, and Blood Institute des National Institutes of Health F30HL162452. Le contenu relève de la seule responsabilité des auteurs et ne représente pas nécessairement les opinions officielles des National Institutes of Health.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 2,5-dihydroxybenzoic acid (DHB) | Supelco, >99.0% (HPLC) | 85707-10MG-F | DHB matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sigma/85707?cm_sp=Insite-_-wimsShippingEmailRecs_wims EmailAPI_wimsGruCrossEntropy-_-wimsEmailAPI10-3 |
| 9-aminoacridine (9AA) | Supelco, ≥99.5% (HPLC) | 92817-1G | 9-Aminoacridine matrix substance for MALDI-MS; https://www.sigmaaldrich.com/US/en/product/sial/92817?srsltid=AfmBOooiQjQ4pWv_XxITkU 4Lkm0UnHXKekGS_ dFl7V40V9QLWoPpNLoc |
| Aquasonic Ultrasound Gel | Parker Laboratories | Parker 01-02 | Ultrasound Gel; https://www.parkerlabs.com/products/aquasonic-100-ultrasound-transmission-gel/ |
| Benchtop Dewar Flasks | ThermoScientific | 4150-2000 | Container for liquid nitrogen; https://www.thermofisher.com/order/catalog/product/4150-4000?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sQVc2f-NxN04Nb5Mv F6TZ7GLHWWDEeqDYmEvtKJSQ YHDeVgZ9qylvYaAs27EALw_wcB &source=google_shopping&ISO_ CODE=us&LANG_CODE=en&ef_id =Cj0KCQjwpvK4BhDUARIsADHt9 sQVc2f-NxN04Nb5MvF6TZ7GLHWWDE eqDYmEvtKJSQYHDeVgZ9qylvYa As27EALw_wcB:G:s&s_kwcid=AL!3652 !3!716188292869!!!g!2366243726129 !!21787513085!171591181194&ev_chn =shop&cid=0se_gaw_30092024_ PBYTXL&source=google_shopping &ISO_CODE=us&LANG_CODE= en&gad_source=1 |
| Cryostat | Leica Biosystems | CM Series | https://www.leicabiosystems.com/us/histology-equipment/cryostats/ |
| Dessicator | VWR | 89054-052 | https://us.vwr.com/store/product/9104882/desiccator-plastic-ace-glass-incorporated |
| Epredia MX35 Premier Disposable Low-profile Microtome Blades | Fisher Scientific | 3052835 | Cryostat blade; https://www.fishersci.com/shop/products/mx35-premier-disposable-low-profile-microtome-blades/3052835 |
| Falcon 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | Conical Tubes; https://www.fishersci.com/shop/products/falcon-15ml-conical-centrifuge-tubes-5/1495953A?gclid=Cj0KCQjwpvK4BhDUARIsA DHt9sSBcy5n-lhShligJUOX5KKVGn0bt87 8AB2_muOD2PPTue1phpZgeqwa AqgiEALw_wcB&ef_id=Cj0KCQjw pvK4BhDUARIsADHt9sSBcy5n-lhS hligJUOX5KKVGn0bt878AB2_muO D2PPTue1phpZgeqwaAqgiEALw_ wcB:G:s&ppc_id=PLA_goog_20861 45674_81843405034_1495953A__ 386247001345_165426395473886 37329&ev_chn=shop&s_kwcid=AL!4428!3 !386247001345!!!g!856907751004!& gad_source=1 |
| Flex-Tubes Microcentrifuge Tubes | Eppendorf | EP022364120 | Centrifuge tubes; https://www.sigmaaldrich.com/US/en/product/sigma/ep022364120?utm_source=google%2Cgoogle&utm _medium=organicshopping%2Ccpc&utm _campaign=21043330280&utm_ content=&gclid=Cj0KCQjwpv K4BhDUARIsADHt9sTHRD35k CHPtfI2A41axodnMVr6a1eBKk zM4bSUYQAyfEKo3UgTAEQa Ap7wEALw_wcB |
| Gas Nitrogen | Airgas | N/A | |
| Glass microscope slides | Electron Microscopy Sciences | 71873-02 | https://www.emsdiasum.com/positive-charge-microscope-slides |
| Liquid Nitrogen | Airgas | N/A | |
| Mass Spectrometer | Waters | Synapt G2-Si | https://www.waters.com/waters/en_US/SYNAPT-G2-Si-Mass-Spectrometry/nav.htm?locale=en_mkcid=1000251Foodety%3C/a%3E&cid=134740653&bcid= 134528734 |
| Matrix Sprayer | HTX Technologies | M3+ | https://www.htximaging.com/htx-m3-sprayer |
| Methanol (HPLC), >99.9% | Fisher Chemical | A4524 | Methanol; https://www.fishersci.com/shop/products/methanol-hplc-fisher-chemical-9/A4524?crossRef=A4524#?keyword=A4524 |
| Preclinical Ultrasound System | FUJIFILM VisualSonics | Vevo 3100 | https://www.visualsonics.com/product/imaging-systems/vevo-3100; Vevo F2 has replaced the Vevo 3100 in production. System includes isoflurane vaporizer and induction box. |
| Reynolds Wrap | N/A | N/A | Aluminum foil |
| Signagel Electrode Gel | Parker Laboratories | Parker 15-60 | Electrode Conducting Gel; https://www.parkerlabs.com/products/signagel-electrode-gel/ |
| Sterile Lubricating Jelly | Medline | MDS032273Z | Lubricating Gel; https://www.medline.com/ce/product/Sterile-Lubricating-Jelly/Lubricating-Jelly/Z05-PF03664?sku=MDPMDS032273H |
| Surgical instruments: scissors, forceps/tweezers, sutures | Fine Science Tools | 11252-00, 11050-10, 14016-14, 14084-08, 15000-08 | info@finescience.com |
| Surgical Sponges 200 Pack –Gauze Pads Non sterile -First Aid Wound Care Dressing Sponge –Woven Medical Nonstick, Non Adherent Mesh Scrubbing | Medpride | B08RZGQ5GW | Gauze; https://www.amazon.com/Medpride-Surgical-Sponges-200-Pack/dp/B08RZGQ5GW/ref=asc_df_B08RZGQ5GW/?tag=hyprod-20&linkCode=df0&h vadid=693270340506&hvpos= &hvnetw=g&hvrand=960915122 2290977669&hvpone=&hvptwo= &hvqmt=&hvdev=c&hvdvcmdl=& hvlocint=&hvlocphy=9192978&hv targid=pla-1245491514869&psc= 1&mcid=33f4d647c88630c79116 888d565a63b0 |
| Tissue-Plus O.C.T. Compound | Fisher Scientific | 23-730-571 | OCT; https://www.fishersci.com/shop/products/tissue-plus-o-c-t-compound-2/23730571 |
| Wood Handled Cotton Swabs and Applicators | Fisherbrand | 22-363-160 | Cotton swab; https://www.fishersci.com/shop/products/wood-handled-cotton-swabs-applicators-8/p-7146852 |
Références
- Tsao, C. W., et al. Heart disease and stroke statistics-2022 update: A report from the American Heart Association. Circulation. 145 (8), E153-E639 (2022).
- Kassab, G. S. Biomechanics of the cardiovascular system: The aorta as an illustratory example. J R Soc Interface. 3 (11), 719-740 (2006).
- Moore, J. E., Xu, C., Glagov, S., Zarins, C. K., Ku, D. N. Fluid wall shear stress measurements in a model of the human abdominal aorta: Oscillatory behavior and relationship to atherosclerosis. Atherosclerosis. 110 (2), 225-240 (1994).
- Nerem, R. M. Vascular fluid mechanics, the arterial wall, and atherosclerosis. J Biomech Eng. 114 (3), 274(1992).
- Cecchi, E., et al. Role of hemodynamic shear stress in cardiovascular disease. Atherosclerosis. 214 (2), 249-256 (2011).
- Good, B. The influence of blood composition and loading conditions on the behavior of embolus analogs. J Mech Behav Biomed Mater. 140, 105738(2023).
- Bao, G., et al. Molecular biomechanics: The molecular basis of how forces regulate cellular function NIH public access. Mol Cell Biomech. 3 (2), 91-105 (2010).
- Schwamborn, K., Caprioli, R. M. Molecular imaging by mass spectrometry-looking beyond classical histology. Nat Rev Cancer. 10 (9), 639-646 (2010).
- Soepriatna, A. H., Kevin Yeh, A., Clifford, A. D., Bezci, S. E., O'Connell, G. D., Goergen, C. J. Three-dimensional myocardial strain correlates with murine left ventricular remodelling severity post-infarction. J R Soc Interface. 16 (160), 20190570(2019).
- Damen, F. W., et al. High-frequency 4-dimensional ultrasound (4DUS): A reliable method for assessing murine cardiac function. Tomography. 3 (4), 180-187 (2017).
- McDonald, R., Poulos, D., Gutzwiller, L., Sheth, R. A., Good, B., Crouch, A. C. A MALDI mass spectrometry imaging sample preparation method for venous thrombosis with initial lipid characterization of lab made and murine clots. J Am Soc Mass Spectrom. 34 (9), 1879-1889 (2023).
- Benetos, A., et al. Influence of age, risk factors, and cardiovascular and renal disease on arterial stiffness: Clinical applications. Am J Hypertens. 15 (12), 1101-1108 (2002).
- Pistner, A., Belmonte, S., Coulthard, T., Blaxall, B. C. Murine echocardiography and ultrasound imaging. J Vis Exp. (42), v2100(2010).
- Bellantuono, I., et al. A toolbox for the longitudinal assessment of healthspan in aging mice. Nat Protoc. 15 (2), 540-574 (2020).
- Henriques, F. C. Jr, Moritz, A. R. Studies of thermal injury: I. The conduction of heat to and through skin and the temperatures attained therein. A theoretical and an experimental investigation. Am J Pathol. 23 (4), 530(1947).
- Conroy, M. J., et al. LIPID MAPS: Update to databases and tools for the lipidomics community. Nucleic Acids Res. 52 (D1), D1677-D1682 (2024).
- Palmer, A., et al. FDR-controlled metabolite annotation for high-resolution imaging mass spectrometry. Nat Methods. 14 (1), 57-60 (2016).
- Michael, L. H., et al. Myocardial ischemia and reperfusion: A murine model. Am J Physiol. 269 (6 Pt 2), H2147-H2154 (1995).
- Clark, J. E., Flavell, R. A., Faircloth, M. E., Davis, R. J., Heads, R. J., Marber, M. S. Post-infarction remodeling is independent of mitogen-activated protein kinase kinase 3 (MKK3). Cardiovasc Res. 75 (3), 523-530 (2007).
- Hattori, Y., Konno, H., Abe, M., Miyoshi, H., Goto, T., Makabe, H. Synthesis, determination of the absolute configuration of tonkinelin, and inhibitory action with bovine heart mitochondrial complex I. Bioorg Med Chem. 15 (8), 3026-3031 (2007).
- Golam Mostofa, M., et al. CLIP and cohibin separate rDNA from nucleolar proteins destined for degradation by nucleophagy. J Cell Biol. 217 (8), 2675-2690 (2018).
- Chan, J. N. Y., Poon, B. P. K., Salvi, J., Olsen, J. B., Emili, A., Mekhail, K. Perinuclear cohibin complexes maintain replicative life span via roles at distinct silent chromatin domains. Dev Cell. 20 (6), 867-879 (2011).
- Dann, M. M., et al. Quantification of murine myocardial infarct size using 2-D and 4-D high-frequency ultrasound. Am J Physiol Heart Circ Physiol. 322 (3), H359-H372 (2022).
- Kaya, I., Sämfors, S., Levin, M., Borén, J., Fletcher, J. S. Multimodal MALDI imaging mass spectrometry reveals spatially correlated lipid and protein changes in mouse heart with acute myocardial infarction. J Am Soc Mass Spectrom. 31 (10), 2133-2142 (2020).
- Tressler, C., et al. Factorial design to optimize matrix spraying parameters for MALDI mass spectrometry imaging. J Am Soc Mass Spectrom. 32 (12), 2728(2021).
- Zhang, L., et al. Vascular lipidomics analysis reveals increased levels of phosphocholine and lysophosphocholine in atherosclerotic mice. Nutr Metab. 20 (1), 1-16 (2023).
- Kohno, M., et al. Induction by lysophosphatidylcholine, a major phospholipid component of atherogenic lipoproteins, of human coronary artery smooth muscle cell migration. Circulation. 98 (4), 353-359 (1998).
- Crouch, A. C., Batra, A., Greve, J. M. Hemodynamic response to thermal stress varies with sex and age: A murine MRI study. Int J Hyperthermia. 39 (1), 69-80 (2022).
- Crouch, A. C., Scheven, U. M., Greve, J. M. Cross-sectional areas of deep/core veins are smaller at lower core body temperatures. Physiol Rep. 6 (16), e13839(2018).
- Crouch, A. C., Castle, P. E., Fitzgerald, L., Scheven, U. M., Greve, J. M. Assessing structural and functional response of murine vasculature to acute β-adrenergic stimulation in vivo during hypothermic and hyperthermic conditions. Int J Hyperthermia. 36 (1), 1136-1145 (2019).
- Crouch, A. C., Manders, A. B., Cao, A. A., Scheven, U. M., Greve, J. M. Cross-sectional area of the murine aorta linearly increases with increasing core body temperature. Int J Hyperthermia. 34 (7), 1121-1133 (2018).
- Clift, C. L., Mehta, A., Drake, R. R., Angel, P. M. Multiplexed imaging mass spectrometry of histological staining, N-glycan and extracellular matrix from one tissue section: A tool for fibrosis research. Methods Mol Biol. 2350, 313-329 (2021).
- Salehi, H. S., et al. Coregistered photoacoustic and ultrasound imaging and classification of ovarian cancer: Ex vivo and in vivo studies. J Biomed Opt. 21 (4), 046006(2016).
- Verbeeck, N., et al. Connecting imaging mass spectrometry and magnetic resonance imaging-based anatomical atlases for automated anatomical interpretation and differential analysis. Biochim Biophys Acta. 1865 (7), 967-977 (2017).
- Yang, E. Y., et al. Real-time co-registration using novel ultrasound technology: Ex vivo validation and in vivo applications. J Am Soc Echocardiogr. 24 (7), 720(2011).
- Maris, L., et al. Method for co-registration of high-resolution specimen PET-CT with histopathology to improve insight into radiotracer distributions. EJNMMI Phys. 11 (1), 1-20 (2024).
- Lin, B. J., et al. MSIr: Automatic registration service for mass spectrometry imaging and histology. Anal Chem. 95 (6), 3317-3324 (2023).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon