Method Article
El aislamiento de las variantes de los virus de ARN de fidelidad y caracterización de la frecuencia de mutación de virus
En este artículo
Resumen
El presente artículo describe los pasos necesarios para aislar y caracterizar las variantes de ARN polimerasa fidelidad de los virus de ARN, y cómo utilizar los datos de frecuencia de mutación para confirmar los cambios en la fidelidad de cultivo de tejidos.
Resumen
Uso de los virus de ARN polimerasas de ARN dependiente de ARN para replicar su genoma. La tasa de error intrínsecamente alta de estas enzimas es un gran contribuyente a la generación de diversidad de la población extremo que facilita la adaptación y la evolución del virus. Cada vez más evidencia muestra que las tasas de error intrínseco, y las frecuencias de mutación resultante, de los virus de ARN puede ser modulada por sutiles cambios en los aminoácidos de la polimerasa viral. Aunque existen ensayos bioquímicos para algunos ARN polimerasa viral que permiten medición cuantitativa de la incorporación de la fidelidad, aquí se describe un método simple de medir las frecuencias de mutación de los virus de ARN que ha demostrado ser tan exactos como los enfoques bioquímicos en la identificación de mutaciones alteran la fidelidad. El enfoque utiliza técnicas convencionales virológica y la secuencia que se pueden realizar en los laboratorios de la mayoría de la biología. Basándonos en nuestra experiencia con una serie de virus diferentes, se han identificado los principales pasos que se deben optimizar para aumentar la probabilidad de aislar variantes fidelidad y la generación de datos de significación estadística. El aislamiento y caracterización de las mutaciones alteran la fidelidad puede proporcionar nuevos conocimientos sobre la estructura y la función de la polimerasa 1-3. Además, estas variantes de la fidelidad pueden ser herramientas útiles en la caracterización de los mecanismos de adaptación del virus y la evolución de 4-7.
Protocolo
1. Determinar el rango de concentraciones de mutágeno que es mínimamente tóxico para las células
El propósito de este ejercicio es determinar qué intervalo de concentraciones mutágeno se puede utilizar durante una infección sin toxicidad celular excesivo. Esencialmente, usted desea reproducir las condiciones que se requerirán para la infección por virus. Para la mayoría de los virus, las infecciones duran entre 2 y 7 días. Prepare suficientes platos a las células de la muestra en cada día. Si las células no adherentes se utilizan, modificar el protocolo de acuerdo.
- El día antes del experimento, la semilla de 7 x 10 5 células HeLa / pocillo en una placa de 6 pocillos que lograr un sub-confluentes (75%) monocapa el día del experimento. Cada pocillo de la placa será tratada con una concentración diferente de mutágeno, que permite un rango de 6 concentraciones.
- En el día del experimento, preparar diluciones mutágeno en el medio de cultivo de tejidos. Para las células HeLa, utilizan una gama de 0 a 1.000 M de análogos de base (ribavirina, 5-fluorouracilo, 5-azacitidina), de 0 a 50 mM de MgCl 2, y de 0 a 5 mM de MnCl2.
- Aspirar el medio de los pozos y reemplazar con 2 ml mutágeno-complementado medio y retorno a la incubadora.
- Cada 24 horas, utilice una placa de células para detectar la viabilidad celular. Esto se puede lograr mediante la realización de azul tripano manchas o comercializados con fluorescencia / luminiscencia ensayos (CellTiter-Glo ® luminiscentes por ejemplo, Promega ensayo de viabilidad celular).
- Para la tinción de azul de tripan exclusión, separar las células de una placa (tratados con diferentes concentraciones de mutágeno) y suavemente las células de pellets por centrifugación.
- Descartar el sobrenadante y resuspender las células en PBS (suero puede interferir con manchas).
- Mezclar 1 volumen de suspensión de células en PBS con un volumen de 0,4% con azul de tripano, se incuban durante 2 minutos a temperatura ambiente.
- En un hemocitómetro, cuente el viables (sin teñir) y no viables (teñido de azul) las células. Calcular el porcentaje de células viables para cada concentración de mutágeno, incluyendo el control sin tratar. Nos encontramos con que las condiciones que dan lugar a menos de 50% la muerte celular por el extremo de la infección (cuando los títulos máximos se alcanzan) son ideales para el aislamiento de la resistencia mutágeno.
2. Determinar la concentración óptima no mutágeno tóxico que reduce moderadamente los títulos de virus (aproximadamente 0,5 a 2 log)
Este ejercicio sirve para determinar la concentración de mutágeno que ejercerá una fuerte presión selectiva, sin exceso de mutagénesis de la población. Para el ARN mutágenos, nos encontramos con que esto se corresponde con los registros 10.5-2 reducciones en el título de virus. En estas concentraciones, cada genoma se encuentra mutado en al menos una o dos posiciones. El mutágeno puede ayudar en la generación de la mutación de resistencia, que luego serán seleccionadas en el paso. Si las mutaciones se introducen demasiados (en concentraciones mutágeno muy alta), los mutantes portadores de la mutación de resistencia serán a su vez letalmente mutadas, lo que dificulta su aislamiento.
- Placas de semillas con las células con las mismas condiciones que para el paso 1
- En el día del experimento, preparar diluciones mutágeno en el medio de cultivo de tejidos. Utilizar el mismo intervalo de concentraciones como se determinó anteriormente, pero no incluyen las concentraciones que resultó en la muerte celular del 50%. Prepare suficiente para cubrir cada medio y dos veces (4 ml por pocillo), lo que permite un tratamiento previo de las células antes de la infección, con cada concentración de mutágeno.
- Aspirar las células medianas y tratamiento previo con mutágeno incubando en mutágeno-complementado medio durante 2 horas. Para la mayoría de los tipos de células, este es el tiempo necesario para la captación de mutágeno.
- Quitar medio e infectan con el virus a baja multiplicidad de infección MOI (0,1 o 0,01) en un volumen mínimo (200 l de placas de 6 pocillos). Incubar 15-60 minutos para permitir que el virus para infectar las células. Rocas de la placa a intervalos regulares para asegurar que el inóculo cubre la monocapa celular.
- Aspirar el virus inoculado y lavar dos veces con 2 ml de PBS para eliminar la mayor cantidad de inóculo como sea posible.
- Añadir media con las concentraciones adecuadas de mutágeno a cada pocillo e incubar las células por el equivalente de 3.6 ciclos de replicación.
- Cosecha de virus de cada pozo y determinar el efecto antiviral de título de virus. Esto se puede hacer por ensayo de placa estándar o limitación de la dilución (DICT 50). Nota: La cuantificación de virus mediante técnicas que la síntesis de ARN única medida puede no ser adecuada, porque los efectos mutagénicos pueden no ser detectados. Genomas mutadas que contienen mutaciones letales aún puede ser detectada por QRT-PCR, por ejemplo, pero no se observaron en los ensayos de viabilidad del virus.
- De los títulos calculados, identificar la concentración de mutágeno, que reduce los títulos de virus (en comparación con el control de la infección no tratada) por 10.5-2 registros que tampoco es altamente tóxico para las células (lo ideal es, menos del 50% de toxicidad).
3. El aislamiento y la identificacióncación de las variantes resistentes mutágeno
Realizar grandes pasajes tamaño de la población en la concentración óptima mutágeno se definió anteriormente y comprobar los títulos de virus a través de la serie pasaje. Como control, el virus de paso en medio de cultivo sin ningún tipo de mutágeno. Como otro de control para vigilar la posible aparición de partículas defectivas interferentes (DI), realice las infecciones nuevas en ausencia de mutágeno en cada paso paso (control unpassaged).
- El día antes del 25 de infección, la semilla cm 2 frascos con 1,5 x 10 6 células HeLa (otros tamaños frasco puede ser utilizado) para obtener sub-confluente monocapas al día siguiente.
- En el día de la infección, tratamiento previo a las células durante 2 horas en un medio con la concentración óptima de cada mutágeno, determinado en el punto 2.
- Quitar medio e infectar las células con un volumen mínimo a una MOI de 1 o el mayor momento de inercia que no da lugar a defectos de formación de partículas de interferencia (DI) para que el virus se está estudiando.
- Después de 30-60 minutos de la infección, aspiración de inóculo y lavar dos veces con PBS, a continuación, añadir medio fresco suplementado con mutágeno en las concentraciones adecuadas.
- Incubar durante el período de tiempo determinado en las secciones 1 y 2 que corresponde al título máximo de virus en estas condiciones. Cosecha de la progenie del virus.
- Título del virus en cada paso y repite los pasos 3 anterior.
- Durante los primeros pasajes, los títulos de las muestras de virus mutágeno tratado debe caer en consecuencia, en comparación con el título del virus original y el control (sin tratamiento y unpassaged) los títulos de virus. Si los títulos de virus mutágeno pasajes de las muestras a alcanzar los mismos niveles que el control no tratado, la población que contiene una variante resistente a mutágeno. Hasta 20 o 30 pasos sean necesarios, aunque más aisladas de nuestro variantes fidelidad entre 5 y 15 pasajes.
- Una vez que los títulos de virus para una serie pasaje dado alcanzar la misma magnitud que los títulos de control sin tratamiento, el extracto de ARN de las muestras como los controles no tratados en el número mismo pasaje. Kits de extracción de RNA o la extracción de Trizol se puede utilizar.
- Realizar RT-PCR utilizando los cebadores que amplifican los genes de la polimerasa o replicasa del virus de interés. En un segundo paso, el genoma completo (al menos en las regiones de codificación) deben ser secuenciados para determinar si los fenotipos de resistencia mapa de los genes de otros virus también. Esto es particularmente importante para los mutágenos base analógica, como la ribavirina, que afecta a otros aspectos del virus y la función celular. En este caso, la variante puede ser resistente a una de estas actividades antivirales y no será una variante de la fidelidad.
- Purificar los productos de RT-PCR utilizando un kit de purificación de PCR y la secuencia para obtener la secuencia de la población mutágeno resistente al consenso. Incluyen controles de fondo para la secuencia de error (ver Discusión).
- El uso de un software de alineación de la secuencia y la secuencia del virus de consenso como una referencia, alinear las secuencias. Identificar las mutaciones puntuales nuevas, con especial atención a las que aparecen exclusivamente en la población mutágeno tratados en el pasaje en el que los títulos de virus alcanzan los niveles normales. Si esta mutación no está presente en los pasajes anteriores y que no están presentes en los controles no tratados en el número mismo pasaje (lo que indica la adaptación a la aprobación de cultivo celular), entonces esta mutación es probable responsable, al menos en parte, por la resistencia mutágeno. No confíe en la base de las llamadas de secuencia (la versión de texto de la secuencia) y el software de alineación solo. Comprobar los cromatogramas con picos de las minorías que pueden haberse pasado por alto por el software de alineación. Un mutante que representan el 20-30% de la población total aún se mostrará como un pico, pero demasiado pequeño para ser identificado como una 'N' por el análisis de la secuencia estándar.
4. Una vez que una mutación ha sido identificada, aislar o generar la variante y confirmar el fenotipo de resistencia a varios agentes mutágenos de ARN
A continuación, la variante de la presentación de la mutación identificada está limitado a confirmar su relación con el fenotipo de resistencia. Es esencial que la mutación se sospecha de cambiar la fidelidad es estudiado en un fondo genético limpio (es decir, que no presentan mutaciones adicionales en otras partes del genoma). En la mejor situación, un clon de cDNA infeccioso existe que permita la generación de un depósito de la variante resistente a mutágeno por mutagénesis dirigida en un fondo genético limpio. En este caso, la sección 4 no es necesario. Sin embargo, si un clon de ADNc no está disponible, el aislamiento se puede hacer mediante la purificación del virus de la placa, se describe a continuación. Más de una ronda de purificación de la placa puede ser necesario para aislar la variante sobre un fondo limpio y genética.
- El aislamiento de los mutantes resistentes mutágeno por la placa de ensayo.
- Para aislar los mutantes identificados, lleve a cabo un ensayo de placa estándar en agarosa (0,5 a 1% final wt / vol) de superposición en placas de 6 pocillos. Prepare diluciones seriadas del virus, sobre la base de los títulos valores, a dilluciones que se producen entre 10 y 50 bien separadas las placas.
- Cuando las placas son claramente visibles (por lo general 2 a 5 días después de la infección, dependiendo del virus), marcar la ubicación de las placas en las placas y el uso de una pipeta de P200 con la punta de filtro, con cuidado hundir la punta a través de la superposición de agarosa con cuidado de no desplazar y cambiar de posición de la superposición (que daría lugar a la contaminación cruzada de las placas individuales).
- Levante con cuidado la punta de la superposición y la transferencia de la clavija de agarosa contenidas en la punta de un eppendorf que contiene 250 l de medio y mezclar. No se preocupe si la punta de la que se extrae no contiene agarosa, para muchos virus de ARN, una placa de medio de muchos virus de ARN contiene 10 5 virus y una cantidad suficiente se transferirá con sólo tocar la punta de la superficie de la placa.
- Elige un máximo de 10 placas por tratamiento mutagénico. Dependiendo de los cromatogramas de secuenciación que identificó la mutación, la estimación de aproximadamente qué porcentaje de la población contiene la mutación deseada. El objetivo es aislar a tres o cuatro placas con la mutación. Algunos de estos mutantes también son portadores de mutaciones adicionales, no deseados, que posteriormente se identificaron mediante la secuenciación.
- Extraer el ARN de las muestras (pero ahorrar la mitad de la muestra para hacer un mayor stock de virus), y realizar RT-PCR, que permiten la secuenciación del genoma completo. En promedio, los virus de ARN que contienen un máximo de dos mutaciones diferencias con respecto a la secuencia de consenso, por lo tanto, la secuencia de 3 o 4 placas virus purificados en un momento de identificar la cepa que contiene la mutación deseada sin ningún tipo de mutaciones adicionales.
- Una vez identificados, hacer un mayor stock de este virus en todos los estudios posteriores, con la muestra de placa purificada obtenida anteriormente para infectar a un frasco más grande de células, por ejemplo, 8x10 6 células HeLa en un matraz T75 ..
- Confirmar la sensibilidad mutágeno / resistencia conferida por la mutación identificada.
- Utilizando el clon aislado o generado nuevos, y un control de virus de tipo salvaje en condiciones similares, repetir los experimentos en la sección 2 utilizando una amplia gama de concentraciones de mutágeno, o la concentración a la que se generó la mutación de resistencia.
- Utilizar varias condiciones diferentes de ARN mutagénico (ribavirina, 5-fluorouracilo, 5-azacitidina, el aumento de Mg 2 +, Mn 2 +). Si la variante de la polimerasa es resistente a más de un tipo de mutágeno, entonces lo más probable es que esta variante es de alta fidelidad. Alternativamente, es posible que una mutación de resistencia es específica para una enfermedad mutagénico único, en particular, ya que algunos de estos compuestos afectan los virus de ARN a través de una serie de mecanismos 8.
5. Compruebe los precios de replicación
Desde la fidelidad que alteran las mutaciones con más frecuencia mapa a la polimerasa, es posible que la mutación de la polimerasa misma alterará significativamente la cinética de replicación y es importante para determinar las similitudes y diferencias en la replicación que permita una mejor comparación de las diferencias en las frecuencias de mutación realizado a continuación. Para ello, examine la replicación por lo menos dos enfoques complementarios - que analiza la producción de virus y otro que examina la síntesis de ARN.
- Un solo paso cinética de crecimiento de virus
- El día antes del experimento, la semilla de placas de 6 pocillos, según sea necesario, una placa por cada punto de tiempo para hacerse la prueba. Considere el uso de pozos por triplicado para cada mutante y el virus de tipo salvaje.
- En el día del experimento, se elimina el medio y los pozos de infectar con el virus de cada uno a una MOI de 10 para asegurar que cada célula se infecta simultáneamente. Incubar 30-60 minutos a 37 ° C.
- Roca placas cada 10 minutos para evitar la desecación de la monocapa celular. Eliminar el virus y lavar dos veces con 2 ml de PBS. Es importante eliminar la mayor cantidad de inóculo como sea posible. Vuelva a colocar con medio de cultivo.
- Después de la infección en el tiempo = 0, recoger el virus de una placa. Volver placas a la incubadora y la cosecha de los virus a intervalos regulares que abarcan un solo ciclo de replicación (por ejemplo, 3H, 5H, 7h, 9h, 12h, 24h).
- Virus título, cosechadas en cada momento (por ejemplo, la placa de ensayo, TCID50, ensayo FFU), y el gráfico de las curvas de crecimiento de título en función del tiempo.
- Cinética de la síntesis de ARN
La cinética de la síntesis de ARN pueden ser monitoreados utilizando uno de los métodos indicados a continuación. Si es posible, las mismas muestras utilizadas para determinar un paso cinética de crecimiento se deben utilizar para medir los niveles de ARN.- QRT-PCR. Este procedimiento da muy medidas cuantitativas de la replicación a través de una amplia gama, a partir de una copia del genoma de unos pocos> 10 10, dependiendo de la sensibilidad de la prueba. Cebadores y sondas de diseño que cubren un pequeño fragmento (<200 pb) de una región altamente conservada genómica.
- Blot del Norte. Aunque menos cuantitativo que QRT-PCR, esta técnica permite la confirmación visual de que los resultados de la replicación de los genomas de larga duración y que no terminación de la cadena significativa se produce como consecuencia de la mutación de la polimerasa.
- Expresión de un gen reportero. Si un clon de cDNA expresión de un gen reportero (luciferasa, por ejemplo) está disponible, entonces este puede ser utilizado como un sustituto para examinar la capacidad replicativa. Sin embargo, el virus recombinante no debe ser utilizado para otras aplicaciones (como la determinación de la frecuencia de mutación), ya que las presiones selectivas que actúan sobre este virus no será la misma, en particular, ya que estos virus tienen una tendencia a eliminar el gen reportero insertado.
6. Medir frecuencias de mutación
Este es un paso crítico en el que confirma que la mutación de la polimerasa identificó que confieren resistencia a mutágeno replicación altera la fidelidad. Es importante tener en cuenta que las frecuencias de mutación medido aquí no son las tasas de mutación. Para determinar las tasas, una medida muy cuidadosa de la cinética de replicación (cantidad de ARN sintetizado y la duración del ciclo de replicación) deben tenerse en cuenta las frecuencias de mutación de medición in embargo, siempre y cuando el número de pases y la cinética de replicación son controlados, proporciona medidas reproducibles y cuantitativa de la replicación fidelidad. Las frecuencias de mutación se puede determinar, ya sea en la población de virus viables (clones placa o dilución límite) o en la población total de virus (virus de archivo o el sobrenadante). Para determinar las frecuencias de mutación, preparar las reservas de virus de un pasaje posterior (por ejemplo, el paso 2 o más allá). Es importante que la población de virus ha tenido tiempo para ampliar su diversidad genética más cerca de un equilibrio mutación-selección.
- Las frecuencias de mutación de la población de virus viable
Este enfoque, aunque más laborioso, da información sobre cómo las mutaciones están presentes en el genoma del promedio que tiene competencias de replicación. Cabe señalar, sin embargo, que un prejuicio para las variantes de mayor aptitud se produce y menor aptitud, las variantes viables que no son fáciles de placa, por ejemplo, no pueden ser detectados. Como tal, también permite mejorar las medidas de sinónimos (DS) y no sinónimas (dN) sustituciones de nucleótidos que se pueden utilizar para explorar si la selección positiva actúa sobre la población. Sin embargo, debido a que menos mutaciones se pueden cuantificar, un mayor número de secuencias serán necesarios para el análisis estadístico. Esta técnica se basa en el aislamiento de virus individuales mediante la purificación de placa ya sea o dilución límite, como se describió anteriormente. Como punto de partida, se recomienda el aislamiento de 48 individuales "clones" del virus tipo salvaje y el mutágeno variante resistente. Cada población clonal aislados de esta manera se espera que lleve todo lo que la mutación del genoma fundador presentado. La cantidad de ARN presente en la placa aislada o dilución limitante y es generalmente suficiente para la amplificación por RT-PCR. Si es necesario, una amplificación de corta duración (menos de un ciclo de replicación) en un número mínimo de células (por ejemplo, el formato de 24 pocillos) se puede utilizar para obtener más ARN, sin embargo, la amplificación mínimo se debe realizar para evitar la acumulación de nuevas mutaciones. Tenga en cuenta que cada clon y la población a comparar debe someterse al mismo número de ciclos de replicación.- Aislar 24 a 48 clones de virus mediante la purificación de placa o dilución limitante.
- Extraer el ARN de las poblaciones clonales aislados
- RT-PCR amplificar un fragmento que abarca hasta 3kb para cada muestra. Lo mejor es cubrir la región de la proteína estructural, que tiende a tolerar las mutaciones más viable que las regiones más conservadas de genes no estructurales.
- Purificar los productos PCR de secuencia y el análisis de mutación (artículo 7).
- Las frecuencias de mutación de la población total de virus
A pesar de la ventaja de este segundo método es que, incluso variantes de fitness baja se incluirán en la secuencia, lo que permite una visión más amplia del espectro de mutaciones. Sin embargo, no puede ser ideal para los análisis filogenéticos que asumen las poblaciones viables de virus (por ejemplo, dN / dS valores) y la identificación de las mutaciones más relevantes, ya que los cambios letales (alterado la estructura del ARN, codones de parada, los dramáticos cambios de aminoácidos) no puede ser totalmente identificados y se mantendría en el análisis. Sin embargo, esta técnica permite al investigador para obtener la información más significativa para confirmar la alteración de la fidelidad cuando hay una falta de un ensayo in vitro bioquímicos. Esta técnica se basa en la RT-PCR de amplificación de ARN del virión total, incluyendo los genomas con baja condición física o mutaciones letales que no se producen placas. Las frecuencias de mutación obtenida por este método puede ser 10 veces mayor que por la placa o limitar la clonación de dilución.- Extraer el ARN de la población total de virión
- RT-PCR amplificafy una región 800 a 1200 nucleótidos de una parte de la secuencia genómica de codificación que se conoce a tolerar las mutaciones y tienen varianza genética (por ejemplo, las proteínas estructurales). Fragmentos más grandes no se inserte rápidamente en vectores de clonación como TopoTA y se produce un número insuficiente de transformantes. A pesar de fragmentos más pequeños son aún mejores, la cobertura de la secuencia del genoma puede ser demasiado poco para obtener la significación estadística. Un fragmento de al menos 800 pb permite la cobertura de secuencia con dos primers y es un buen compromiso entre la maximización de la cobertura de secuencia y minimizar los costos de secuenciación. Nos encontramos con que entre 70 y 100 secuencias que cubren una región de 800 nucleótidos reproducible confirma la fidelidad de alteración de las variantes que hemos estudiado en el laboratorio. Tenga en cuenta que otros vector / clonación métodos se pueden utilizar con la misma eficacia.
- Purificar el producto RT-PCR utilizando un kit comercial o por la extracción de ADN estándar / precipitación.
- Si las enzimas RT-PCR que utilizan no producen un salientes, realizar una extensión de 10 minutos mediante la adición de 1 mM ATP y Taq polimerasa
- TopoTA clon siguiendo las instrucciones del fabricante
- Para cada población de virus a estudiar, seleccionar 96 colonias, identificadas con una inserción positiva en azul / blanco de análisis en Xgal placas cubiertas. Inicialmente, la prueba de la presencia de insertos para cada región del genoma diferente de ser clonada, para confirmar la validez de azul / blanco de detección por cribado el tamaño del plásmido en geles de agarosa o PCR de una sola colonia. Utilizando el tamaño de los fragmentos y las condiciones de arriba, podemos lograr resultados positivos del 90%
- Crecer cada colonia en caldo de líquido durante la noche en 1 ml de medio LB en 96 placas de cultivo de bacterias y
- Al día siguiente, preparar minipreps en formato de 96 pocillos.
- Secuencia de cada plato con los primers suficiente (los cebadores utilizados para la RT-PCR, por ejemplo, o de los cebos TopoTA m13) para obtener la cobertura máxima del segmento clonado. Realizar análisis de la mutación (artículo 7).
7. Análisis de secuencias
Realizar análisis de la secuencia utilizando una referencia o una secuencia de consenso para cada población y el software de la alineación adecuada. Recomendamos Lasergene o Sequencher que puede identificar fácilmente SNPs con respecto al consenso.
- Alinear las secuencias usando el software apropiado (por ejemplo, o Lasergene Sequencher).
- Deseche las secuencias de baja calidad (llamada base de malo, demasiados "N o demasiado corto en longitud). Identificar el rango de nucleótidos que está cubierto por todas las secuencias. Desde las diferentes regiones del genoma más o menos tolerar las mutaciones, por razones de comparación, es esencial que la misma región está totalmente cubierto por cada clon secuenciado retenidos para su análisis. Por lo tanto, si las reacciones se llevan a cabo varias secuencias por clon, y no una secuencia de un clon determinado, deseche el clon (todas las secuencias) del análisis.
- Identificar y contar los SNPs que son diferentes de la cepa de referencia.
- Calcular la frecuencia de mutación de dividir el número total de SNPs identificados por el número total de nucleótidos secuenciados (número de clones de longitud x de la región de la secuencia). La presentación de este número como el número promedio de mutaciones por 10K nt secuencia hace que el número más fácil de usar luego dejarlo como por nucleótidos. Por ejemplo, para la población silvestre en la Tabla 1, 55 mutations/121, 978 nucleótidos totales x 10.000 = 4,51 por cada 10.000 nucleótidos mutaciones secuenciado.
- Si el mismo SNP aparece en un gran número de clones, se presentan dos valores que incluyen o excluyen estas mutaciones repetidas. Generalmente, para una población de virus producido a partir de una matriz homogénea (por la purificación de placa o de un clon infeccioso) y se pasaron sólo un par de veces en cultivo celular, la selección positiva no ha ejercido sus efectos como para causar la acumulación de la misma SNP y la mutación los valores de frecuencia para medir la mejor manera de reflejar la frecuencia de errores de la polimerasa con efectos mínimos de selección positiva o purificación.
- Determinar la distribución de las mutaciones. Haga una lista clasificada de la cantidad de clones en cada población que presente 0, 1, 2, 3, etc .. mutaciones en la región secuenciada.
- Calcular la diversidad de la población viral en comparación pairwise distancia. De la limpieza y las secuencias de editar manualmente preparar una alineación que incluye la región de referencia. Hay varios programas de cotejo: ClustalW / X ( http://www.clustal.org/ ), músculo ( http://www.drive5.com/muscle/ ), EbioX para usuarios de Mac ( http://www. ebioinformatics.org / ebiox / ), etc Asegúrese de que todas las secuencias tienen la misma longitud, de los cultivos si es necesario. Se recomienda que el inicio de las secuencias es un codón de codificación para facilitar el análisis posterior. Sugerimos mantener todas las alineaciones en formato FASTA, que es fácil de leer por la mayoría del software disponible.
- Para realizar el análisis pairwise distancia, calcular todas las posibles comparaciones por parejas entre las secuencias que pertenecen a la misma población. Las mutaciones se encuentran en medio de las comparaciones se puede calcular para indicar la heterogeneidad de la población.
- Los valores de sinónimos (DS) y no sinónimas (dN) dentro de la población se puede obtener fácilmente. Nos encontramos con el software MEGA (http://www.megasoftware.net/) útil y fácil de usar para este tipo de análisis.
- Para obtener la dirección de selección le damos dos posibilidades, aunque no son las únicas posibles. 1) Obtener la relación entre dN y dS. Valores superiores a 1 significa que la selección positiva. Valores inferiores a 1 significa que la selección purificadora. 2) Utilice el servidor web Datamonkey ( http://www.datamonkey.org/ ). Sube tus alineaciones en formato fasta y analizarlos con el módulo de SLAC. Esto le dará una estimación de dN / dS.
- Realizar análisis estadísticos. Dependiendo de la cantidad de datos y el número de secuencias, una serie de pruebas se puede utilizar. Algunos estudios se han basado en pruebas de chi cuadrado del número total de mutaciones en función del número total de nucleótidos consenso en todos los clones se combinan 9. En otros estudios se han realizado pruebas exacta de Fisher, calculado sobre el número de secuencias que presentan mutaciones en comparación con el número de secuencias sin mutaciones 10. Si una cantidad suficiente de datos de mutaciones se genera, se recomienda realizar una prueba de suma clasificado como de Mann-Whitney U. Para ello, el rango del número de clones en cada población de virus por el número de mutaciones presentes en cada secuencia de ARN. El Mann Whitney se pondrá a prueba las diferencias en la distribución de la mutación de las poblaciones. Por esta razón, se recomienda la secuencia de al menos 800 pares de bases, para aumentar la probabilidad de encontrar clones con múltiples mutaciones. Esta prueba es robusta, pero requiere de mayor tamaño de la muestra. Por otro lado, que no requiere el mismo tamaño de muestra para las dos poblaciones que se comparan (por ejemplo, las muestras de la Tabla 1 son n = 1 148 y 2 n = 84).
8. Los resultados representativos:
El efecto dosis-dependiente de la concentración de mutágeno en la viabilidad celular y la viabilidad del virus se muestra en la Figura 1. En este ejemplo, se encontró que el paso del virus en 100 AZC M se redujo en el título de virus por el objetivo 10.5-2 registro, pero la viabilidad de células HeLa no un impacto negativo en los dos días necesarios para la infección por el virus. Esta experiencia piloto llevó a la elección de la concentración 100 mM AZC para el paso de serie de virus, para seleccionar animales resistentes mutágeno. La figura 2 muestra la reducción inicial en el título, seguido por la aparición de un fenotipo de resistencia mutágeno. Durante los primeros pasos en pocos mutágeno, como se acumulan las mutaciones letales, un descenso significativo en los títulos de virus se produce. Poco a poco, una variante resistente a mutágeno surge una su aparición coincide con el regreso a los títulos de virus no es diferente de los controles no tratados. En esta etapa, un gran porcentaje de la población de virus presenta la mutación de resistencia. La secuenciación del virus de esta población pone de manifiesto el cambio de aminoácido (s) responsable. Una vez identificados y aislados o generados recientemente, el virus mutágeno resistentes pueden ser menos sensibles que las cepas silvestres de diferentes mutágenos ARN (análogos de base de estructura diferente, por ejemplo). La figura 3 muestra un ARN mutágeno resistente a virus Coxsackie B3 que los títulos superiores de tipo silvestre en presencia de la ribavirina, 5-fluorouracilo y 5 azacytidine y alto MgCl2 y MnCl2. Amplia resistencia a los agentes mutágenos de ARN es un fuerte indicador de la fidelidad de aumento de la replicación. Verificar que la cinética de replicación de la variante de la fidelidad es similar al virus de tipo salvaje ayudar en la comparación de las frecuencias de mutación. Figura 4 muestra el crecimiento paso a la cinética de una variante de alta fidelidad en comparación con el tipo salvaje. Si las tasas de repetición y los títulos finales no son similares, entonces se deben tomar medidas para comparar las poblaciones de virus de tamaño similar, que han sufrido el mismo número de ciclos de replicación. La relación entre la tasa de síntesis de ARN y la fidelidad de replicación no está bien caracterizado, sobre todo en vivo. Una tasa de replicación más lenta puede resultar en una disminución de la frecuencia de mutación (alta fidelidad), aunque esto no es una regla absoluta, como se muestra en la Figura 4. Con los parámetros anteriores establecidos, las frecuencias de mutación de la variante de la fidelidad y las poblaciones de tipo salvaje se puede comparar a obtener la confirmación genética de la fidelidad de la replicación alterada. Figura 5 se muestra un alineamiento de secuencias de tipo salvaje y de alta fidelidad de la variante, con mutaciones puntuales identificadas. Las mutaciones se cuentan, según el orden de las mutaciones por clon (Tabla 1), y se representa como una frecuencia de mutación por medio de la población, por cada 10.000 nucleótidos secuenciados, la Figura 6.

Figura 1. Determinar las condiciones óptimas para seleccionar la resistencia de ARN mutágeno:. Retención de la viabilidad celular con células de alta moderada (caída de registro de 1-2 en el título de virus) HeLa fueron tratadas con concentraciones indicadas de ribavirina e infectadas con el virus de tipo salvaje Coxsackie B3 en un momento de inercia de 0,01. 48 horas después de la infección, el virus de la progenie fue cosechada y los títulos fueron determinados por DICT 50. El porcentaje de células que sobrevivan al tratamiento a las 48 horas, determinado por tinción con azul de tripano, se indica a continuación el eje-x. Los resultados muestran que las concentraciones de 100 y M 200 reducir los títulos de virus por registro 2.1, sin afectar la viabilidad celular.
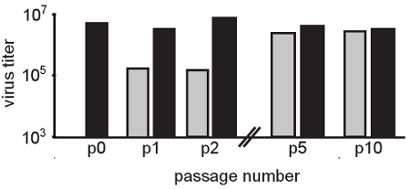
Figura 2. Pases en serie en la presencia de concentraciones moderadas de ARN mutágenos selecciona a las poblaciones resistentes mutágeno. En esta figura, virus Chikungunya se pasó en las células HeLa en presencia de M ribavirina 50 (barras grises). Pasajes de control se realizaron en ausencia de ribavirina (barras de color negro). Después de cada paso, la progenie de virus se cuantificó mediante un ensayo clásico sobre la placa de células BHK. El efecto mutagénico es evidente durante los primeros pasos (P1 y P2 en comparación con la población a partir p0) donde el virus de bajada tratado con los títulos de 2 log. Poco a poco, los títulos de volver a la normalidad (sin tratar) los niveles. No se observan diferencias significativas en el paso 5 poblaciones mutágeno tratados en comparación con el tratamiento, lo que sugiere que las variantes resistentes se han seleccionado. De hecho, la secuencia de consenso de la población identificado mutaciones únicas en la población de virus que reciben tratamiento ribavirina.
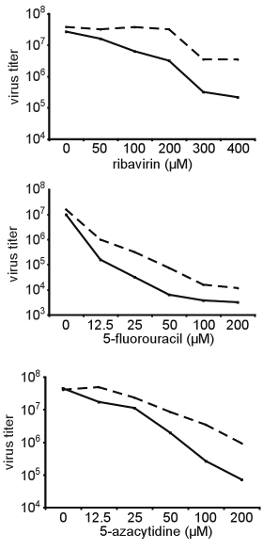
Figura 3. La confirmación de la amplia resistencia a los agentes mutágenos de ARN de estructura diferente. Se muestra aquí, la alta fidelidad variante A372V del virus Coxsackie B3 que se aisló inicialmente en la pantalla se describen en la sección 3 se generó a partir de un clon infeccioso y la prueba de su sensibilidad en relación con diferentes concentraciones de diferentes mutágenos ARN (ribavirina, 5-fluorouracilo, 5-azacitidina). Las células HeLa fueron tratadas con concentraciones indicadas de ribavirina e infectadas con el virus de tipo salvaje Coxsackie B3 a una MOI de 0,01. 48 horas después de la infección, el virus de la progenie fue cosechada y los títulos fueron determinados por DICT 50. Aquí se presentan los títulos de tipo salvaje (líneas continuas) y A372V variante (líneas discontinuas) en función de la concentración de mutágeno. A372V consistentemente más altos que los títulos de tipo salvaje en todas las condiciones de la prueba.
Figura 4. Las tasas de replicación y las variantes de la fidelidad. Para determinar la cinética de crecimiento de un solo paso de producción de virus, las células HeLa fueron infectadas a MOI = 10, ya sea con el tipo salvaje (línea continua), de alta fidelidad variante A372V (guiones largos) o variante de replicación deficiente Cx64 (corto guiones) de los virus Coxsackie B3. En intervalos de tiempo indicados, la progenie de virus se cosechó a partir de células y sobrenadantes de congelación-descongelación y la titulación de DICT 50. El aumento de la fidelidad de A372V no coincide con un defecto de replicación se observa en el cultivo de tejidos. La Cx64 variante presenta un retraso significativo en la cinética de replicación y alcanza los títulos de máximo que son 1000 veces menores que el virus de tipo salvaje.
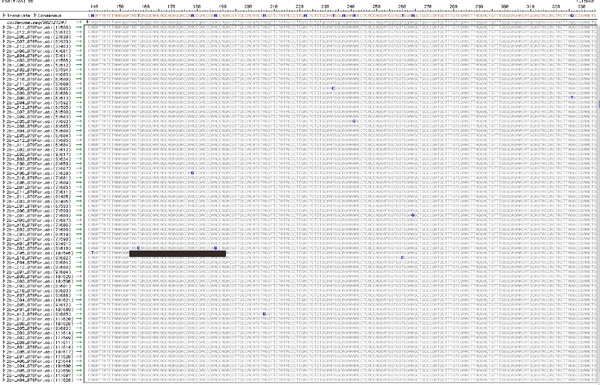
Figura 5. Alineación de las secuencias de TopoTA clonado a partir de cada población de virus. Utilizando el método descrito en la Sección 7, cada secuencia obtenida a partir de RT-PCR clonado producto presumiblemente se origina a partir de un genoma único, único en la población total de virus y por lo tanto sería, son portadores de mutaciones únicas. La figura muestra una alineación típica, después de la limpieza de las secuencias de baja calidad y la visualización de los SNPs. El total de SNPs (10 en esta figura) dentro de una población son contados, y el número de SNPs que aparecen en cada clon es anotada. Por ejemplo, el clon subrayada por un bar, contiene dos mutaciones únicas, mientras que otros ocho clones contienen una sola mutación, único. Estos datos son utilizados para elaborar la tabla 1. Para ver una versión mayor de esta cifra por favor haga clic aquí .
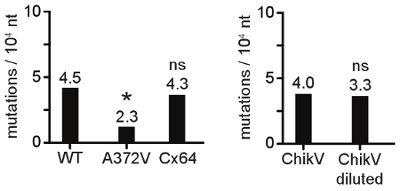
Figura 6. Representación gráfica de las frecuencias de mutación de las poblaciones de virus. Para facilitar la interpretación, los datos numéricos obtenidos de la secuencia y los análisis estadísticos se puede representar como una tabla, o un histograma (en la foto). A372V virus genera menos mutaciones de tipo salvaje y presenta una frecuencia de mutación significativamente menor (*, p <0,01). La variante de Cx64, que se replica en los títulos de 1000 veces menor que la de tipo salvaje, la presiónlos padres de la frecuencia de la misma mutación (ns, no significativo) que indica que la velocidad de la replicación y la fidelidad no están necesariamente ligadas. El mismo virus Chikungunya (CHICKV) de la población da una frecuencia similar mutación si la acción del virus, o una dilución de 10 5 veces, se utiliza para la extracción de RNA.
Mutación resumen de la distribución para el análisis estadístico.
Nota: Para cada clon, es esencial que la misma región genómica (y la longitud de la secuencia) está cubierta. En este caso, 859 nucleótidos por clon. Esto es fundamental para el análisis estadístico. Por otro lado, las pruebas de la suma de rangos para el análisis estadístico no requieren el tamaño de la muestra a ser el mismo, el investigador es libre de comparar la población de diferentes tamaño de la muestra. Por lo tanto, los 142 clones de tipo salvaje se puede comparar con los 84 clones de A372V.
| # Clones con mutaciones n | de tipo salvaje | A372V |
| 7 mutaciones | 0 | 0 |
| 6 mutaciones | 0 | 0 |
| 5 mutaciones | 0 | 0 |
| 4 mutaciones | 0 | 0 |
| 3 mutaciones | 1 | 0 |
| 2 mutaciones | 6 | 2 |
| Una mutación | 40 | 14 |
| 0 mutaciones | 95 | 68 |
| Total de mutaciones | 55 | 18 |
| Total de clones secuenciados | 142 | 84 |
| Nucleótidos totales secuenciado | 121978 | 72156 |
| Mutations/10 4 nt | 4.51 | 2.49 |
Tabla 1. . Resumen mutación de distribución para el análisis estadístico Nota: Para cada clon, es esencial que la misma región genómica (y la longitud de la secuencia) está cubierta. En este caso, 859 nucleótidos por clon. Esto es fundamental para el análisis estadístico. Por otro lado, las pruebas de la suma de rangos para el análisis estadístico no requieren el tamaño de la muestra a ser el mismo, el investigador es libre de comparar la población de diferentes tamaño de la muestra. Por lo tanto, los 142 clones de tipo salvaje se puede comparar con los 84 clones de A372V.
Discusión
Elección de la línea celular. La eficacia de los análogos de base como ARN mutágenos se correlaciona con la captación relativa de diferentes tipos de células 11. Si la línea celular que normalmente se utiliza para el paso de virus ha demostrado ser resistente a la absorción de mutágeno o demasiado sensible (alta toxicidad celular), puede ser necesario el uso de otra línea celular que cumpla con estos requisitos y sigue siendo permisiva para la replicación viral. Una vez que la variante de la resistencia mutágeno es aislado, el resto de la caracterización se puede realizar en la línea original, de celda preferida. En nuestra experiencia, las células HeLa tome fácilmente el mutágeno; células BHK requerir hasta 10 veces las concentraciones más altas y las células Vero son refractarios a la captación de mutágeno.
Elección de mutágeno. Al tratar de aislar variantes fidelidad por el tratamiento mutagénico, la probabilidad de éxito aumenta si hay más de un tipo de mutágeno se utiliza. Mutágenos analógicas de estructura diferente, que son erróneamente incorporado en el genoma durante la replicación predominantemente inducirá resultar en un subconjunto específico de mutaciones en los ciclos de replicación posterior: el tratamiento con ribavirina favorece GtoA y mutaciones CtoU de transición de 12, 5-azacitidina tiene una tendencia similar, con la Además de CtoG y transversiones GtoC 13, 5-fluorouracilo preferentemente induce transiciones Atog y UtoC 14. Por otra parte, la mayor concentración de Mg 2 + o Mn 2 + se puede complementar con el medio para aumentar la frecuencia de mutación de los virus ARN total sin el sesgo se ha descrito anteriormente 12. En función de las secuencias de codones del virus, y los cambios codón necesaria para generar una variante de la fidelidad, algunas de estas condiciones favorecen la aparición de esta variante respecto a otros. Para la mayor fidelidad G64S poliovirus y el virus Coxsackie A372V, el tratamiento con ribavirina más fácilmente seleccionados para las variantes debido a la transición Atog requerido en el sitio codón corresponde a las mutaciones sobre todo generada por este ribavirina.
MOI vs tamaño de la población. En la virología, los protocolos para la infección por el cultivo de tejidos preste especial atención a la multiplicidad de infección (MOI), para evitar la acumulación de partículas defectivas interferentes (MOI baja) o para promover la recombinación entre el virus (de alto MOI), para ejemplo. Para seleccionar los eventos aparición en serie de pases, también es importante tener en cuenta el tamaño de la población de virus. Desde el mutante resistente inicialmente existe en baja frecuencia, lo mejor es la transferencia de tan gran tamaño de la población como sea posible de un pasaje a la siguiente (10 5 -10 6 virus, por ejemplo) para evitar la pérdida de estas variantes emergentes en cada paso. Ampliar el tamaño del pozo o frasco (número de células infectadas) puede ayudar a minimizar el aumento de MOI, si esto es motivo de preocupación. Por otro lado, para los experimentos en que se la sensibilidad de un virus mutágeno a prueba, la infección MOI baja se realiza con el fin de aumentar el número de ciclos de replicación que ocurren en el experimento y para evitar rescate de los genomas de mutagénesis por los genomas mayor aptitud a través de complementación en cooperación con las células infectadas. Esto es importante porque las mutaciones generadas en los genomas de la progenie durante la primera ronda de replicación no se detectan inmediatamente. La mayoría de estos RNAs mutagenizadas todavía se empaquetan en viriones. Es en la siguiente ronda de la infección que las mutaciones letales presentes en estos genomas se traducirá en un ciclo de replicación abortados, y la reducción en el título de virus. Puede que sea necesario para permitir que varias rondas de acumulación de mutaciones antes de que un efecto significativo de la mutagénesis letal se observa. Por último, si más de la serie de pasaje en la presencia de mutágenos, los títulos de virus siguen bajando hasta la extinción, el investigador debe tratar de pases del virus en forma gradual las cantidades crecientes de mutágeno (a partir de una concentración muy baja).
El aislamiento y la generación de la copia del ARN mutágeno resistente a partir del ARN mutágeno resistente a la población. Mutágenos ARN presentar múltiples mutaciones al azar para cada genoma, pero la selección de resistencia sólo se enriquecen (y fijar la secuencia consenso) la mutación de resistencia. Para identificar esta mutación, la secuencia de la población resistente mutágeno (consenso de la población) y los virus no individual. Por lo tanto, las mutaciones individuales, aleatoria creada por el mutágeno no se detectan en la secuencia, sólo las mutaciones que dan lugar a cambios en el consenso tras la selección, se encuentran. En nuestra experiencia, sólo identificar a uno o dos cambios secuencia de consenso. Una vez que la población resistente mutágeno se obtiene y la mutación de resistencia es identificado, es necesario generar una acción más pura de esta variante. Anteriormente, se describe un procedimiento de purificación en placa. Por otra parte, si el virus de interés no produce placas fácilmente identificables, la variante deseada puede ser PurifIED por dilución límite. Este enfoque es esencialmente un TCID 50 en formato de 96 pocillos, donde se diluye el stock de virus de manera que menos del 50% de los pozos están infectadas. El uso de este dilución, el mismo enfoque que el anterior se toma, en el aislamiento de hasta 10 variantes individuales y confirmar sus secuencias. Como se ha mencionado, en el mejor de los casos, un clon infeccioso de cDNA de la cepa del virus está disponible. El aislamiento de la variante tanto, no sería necesario. En nuestra experiencia, las variantes de la fidelidad son el resultado de un solo sustituciones de aminoácidos y por lo tanto se pueden generar utilizando kits simple, mutagénesis comercializado como QuikChange (Agilent). Una opción secundaria es el uso de un clon de ADNc de una cepa estrechamente relacionados. Sin embargo, si una cepa relacionada se utiliza, le recomendamos que emplee tanto este enfoque y el aislamiento del virus (por ejemplo, purificación en placa), ya que hemos encontrado que la misma mutación altera la fidelidad de dos virus estrechamente relacionados, no necesariamente tendrá el mismo efecto.
La fidelidad y la replicación. Selección de ARN variantes resistentes mutágeno han dado como resultado el aislamiento de los mayores y menores las variantes de la fidelidad con características de crecimiento que son similares a sus homólogos de tipo salvaje 4,12,15. En la actualidad, la relación entre las tasas de actividad de la polimerasa y la fidelidad no se entiende completamente. Los estudios in vitro bioquímicos utilizando ARN polimerasa purificada han demostrado que el aumento de las variantes de la fidelidad tiene un ritmo más lento de procesamiento, mientras que la menor fidelidad variantes tienden a tener un procesamiento más rápido 1-3,12. En cultivo de tejidos, estas diferencias no son evidentes, lo que sugiere que la disponibilidad de recursos, en lugar de la cinética intrínseca de la actividad de la polimerasa, es el paso limitante. Si la variante de la fidelidad se replica con una cinética que no son significativamente diferentes de tipo salvaje, y luego una comparación de sus frecuencias de mutación puede ser hecho directamente. Si un cambio muy significativo en la cinética de replicación en vigor, los datos deben ser normalizados para dar cuenta de las diferencias cinética, por ejemplo mediante la comparación de los virus que han sufrido el mismo número de ciclos de replicación. En nuestra experiencia, aunque no hubo diferencias significativas en un solo paso cinética de crecimiento se observaron entre los de tipo natural y de alta fidelidad de las variantes, se observó que el aumento de las variantes de la fidelidad constante título más alto (a menos de 1 log) en comparación con el tipo salvaje, pero que hacen un poco menos de ARN (dentro de el mismo orden de magnitud), lo que sugiere que el genoma que producen contienen menos mutaciones y, por tanto, más infeccioso.
Preparación de la muestra y la secuencia. Para todos los pasos de estos protocolos, es imperativo que los de alta fidelidad, corrección de pruebas, las enzimas se utilizan para la PCR y RT-PCR para limitar la introducción de mutaciones adicionales, ya que no se puede distinguir de las mutaciones biológicamente relevantes. Es fundamental que las poblaciones de virus para ser comparados han sido preparados en las mismas condiciones (el número de pases, medio de cultivo de tejidos, la temperatura, el método de extracción de RNA, RT-PCR protocolos, etc) También es importante asegurarse de que suficiente material de partida fue obtenido de la extracción de RNA de tal manera que una fuerte banda es generado por RT-PCR. Una dilución 1 / 100 de la muestra de ARN también debe dar una detectable por RT-PCR banda, lo que indica que la muestra contiene un número suficiente de moléculas de ARN para evitar el sesgo de representación (amplificación del genoma del mismo en varias ocasiones). Dado que la frecuencia de mutación es una distribución, es de esperar que los mismos valores se obtendrán independientemente del tamaño de la población, siempre que el sesgo mencionado no está ocurriendo. Como muestra la Figura 6 muestra, a 10 5 veces la dilución de un stock de virus ofrece una frecuencia de mutación que no es significativamente diferente de la población reproductora.
Hasta que las condiciones óptimas para TopoTA clonación se encuentran, confirmar la presencia de inserciones después de la evaluación azul / blanco, por PCR colonia antes de la secuenciación. Como control de ruido de mutación (mutaciones introducidas por la RT-PCR y secuenciación), clonar un producto de PCR de un plásmido teniendo la misma secuencia viral y / o la clonación de la secuencia y los productos de RT-PCR in vitro de ARN transcrito correspondiente al genoma del virus (se conscientes de que diferentes enzimas de transcripción in vitro tienen diferentes tasas de error y no puede dar información útil sobre el error de fondo real en el procedimiento). Algunas secuencias de virus pueden ser tóxicos para las bacterias, por lo que es importante verificar esto antes de decidir sobre la región del genoma viral para ser secuenciados para la frecuencia de mutación. En el análisis de secuencias obtenidas por TopoTA, tenga en cuenta que cada clon debe contener un solo insertar / secuencia. Si un pico doble, lo cual sugiere una población mixta, es posible que dos colonias de bacterias vecinos fueron seleccionados. También es posible, aunque muy poco probable dada la baja las frecuencias de mutación en la replicación bacteriana, que la mutación se presentó durante la amplificación del plásmido en el cultivo de bacterias. En la placa purified poblaciones, un doble pico puede representar placas superpuestas, o un virus que está adquiriendo una nueva mutación o la reversión de una mutación durante el desarrollo de la placa. Sea consistente y decidir sobre la conveniencia de contar o no contar con estas mutaciones.
Por último, tenga en cuenta que las frecuencias de mutación utilizados aquí son valores relativos. Sólo son válidas en la comparación de las poblaciones de virus cultivados en las mismas condiciones, y la secuencia en la misma región! No deben tomarse como valores absolutos de la tasa de mutación, o la frecuencia de mutación del genoma en su conjunto. Sin embargo, cuando las condiciones son controladas, es posible hacer comparaciones reproducible y cuantitativa de las diferencias en la distribución y la frecuencia de mutación.
Divulgaciones
Agradecimientos
Este trabajo fue apoyado por fondos de la Medicina y la Investigación en Salud de subvención de la Ciudad de París, la Nacional de Francia conceder ANR-09-JCJC-0118-1, y el ERC Starting Grants del proyecto RNAvirusPopDivNVax no. 242719.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
|---|---|---|---|
| ribavirina | Sigma | R9644-10MG | |
| 5-fluorouracilo | Sigma | F6627-1G | |
| 5-azacitidina | Sigma | A2385-100MG | |
| MgCl2 | Sigma | M1028-100 ML | |
| MnCl2 | Sigma | M1787 | |
| El azul tripán | Sigma | T8154-20ML | |
| TopoTA kit de clonación | Invitrogen | 10351021 | |
| QuikChange kit de mutagénesis | Agilent | 200516 | Si un clon infeccioso de cDNA está disponible |
| 96 y kit minipreparación | Macherey-Nagel | 740625 | |
| Lasergene, Sequencher | DNAstar, Gene Codes Corporation | www.dnastar.com www.genecodes.com | O el software de alineación de otros |
Referencias
- Arias, A. Determinants of RNA-dependent RNA polymerase (in)fidelity revealed by kinetic analysis of the polymerase encoded by a foot-and-mouth disease virus mutant with reduced sensitivity to ribavirin. J Virol. 82, 12346-12355 (2008).
- Arnold, J. J., Vignuzzi, M., Stone, J. K., Andino, R., Cameron, C. E. Remote site control of an active site fidelity checkpoint in a viral RNA-dependent RNA polymerase. J Biol Chem. 280, 25706-25716 (2005).
- Korneeva, V. S., Cameron, C. E. Structure-function relationships of the viral RNA-dependent RNA polymerase: fidelity, replication speed, and initiation mechanism determined by a residue in the ribose-binding pocket. J Biol Chem. 282, 16135-16145 (2007).
- Pfeiffer, J. K., Kirkegaard, K. A single mutation in poliovirus RNA-dependent RNA polymerase confers resistance to mutagenic nucleotide analogs via increased fidelity. Proc Natl Acad Sci U S A. 100, 7289-7294 (2003).
- Pfeiffer, J. K., Kirkegaard, K. Increased fidelity reduces poliovirus fitness and virulence under selective pressure in mice. PLoS Pathog. 1, e11-e11 (2005).
- Vignuzzi, M., Stone, J. K., Arnold, J. J., Cameron, C. E., Andino, R. Quasispecies diversity determines pathogenesis through cooperative interactions in a viral population. Nature. 439, 344-348 (2006).
- Vignuzzi, M., Wendt, E., Andino, R. Engineering attenuated virus vaccines by controlling replication fidelity. Nat Med. 14, 154-161 (2008).
- Crotty, S., Cameron, C., Andino, R. Ribavirin's antiviral mechanism of action: lethal mutagenesis. J Mol Med. 80, 86-95 (2002).
- Coffey, L. L., Vignuzzi, M. Host alternation of chikungunya virus increases fitness while restricting population diversity and adaptability to novel selective pressures. J Virol. 85, 1025-1035 (2011).
- Ciota, A. T. Role of the mutant spectrum in adaptation and replication of West Nile virus. J Gen Virol. 88, 865-874 (2007).
- Ibarra, K. D., Pfeiffer, J. K. Reduced ribavirin antiviral efficacy via nucleoside transporter-mediated drug resistance. J Virol. 83, 4538-4547 (2009).
- Levi, L. I. Fidelity variants of RNA dependent RNA polymerases uncover an indirect, mutagenic activity of amiloride compounds. PLoS Pathog. 6, e1001163-e1001163 (2010).
- Sierra, S., Dávila, M., Lowenstein, P. R., Domingo, E. Response of foot-and-mouth disease virus to increased mutagenesis: influence of viral load and fitness in loss of infectivity. J Virol. 74, 8316-8323 (2000).
- Ruiz-Jarabo, C. M., Ly, C., Domingo, E., de la Torre, J. C. Lethal mutagenesis of the prototypic arenavirus lymphocytic choriomeningitis virus (LCMV). Virology. 308, 37-47 (2003).
- Sierra, M. Foot-and-mouth disease virus mutant with decreased sensitivity to ribavirin: implications for error catastrophe. J Virol. 81, 2012-2024 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados