Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Determinación de la balsa de lípidos Partición de las sondas marcadas con fluorescencia en las células vivas mediante espectroscopia de correlación de fluorescencia (FCS)
En este artículo
Resumen
Una técnica para sondear la compartimentación balsa lipídica de proteínas fluorescentes en la membrana plasmática de las células vivas se describe. Se aprovecha de la disparidad en los tiempos de difusión de proteínas localizadas dentro o fuera de las balsas lipídicas. Adquisición puede llevar a cabo dinámicamente en condiciones de control o después de la adición del fármaco.
Resumen
En los últimos quince años la idea de que las membranas celulares no son homogéneas y se basan en microdominios de ejercer sus funciones ha sido ampliamente aceptada. Las balsas de lípidos son microdominios de membrana enriquecidos en colesterol y esfingolípidos. Ellos juegan un papel importante en procesos fisiológicos celulares, tales como la señalización y el tráfico de 1,2, pero también se cree que ser los principales actores en varias enfermedades, incluyendo infecciones virales o bacterianas y las enfermedades neurodegenerativas 3.
Sin embargo, su existencia sigue siendo un tema de controversia 4,5. De hecho, el tamaño de lípidos balsa ha sido estimada en alrededor de 20 nm, 6, ahora bajo el límite de resolución de la microscopía convencional (alrededor de 200 nm), lo que impide su imagen directa. Hasta ahora, las principales técnicas utilizadas para evaluar la partición de proteínas de interés dentro de las balsas de lípidos son las membranas resistente a detergentes (DRM) de aislamiento y co-parches con anticuerpos. Aunque ampliamente utilizado becael uso de su puesta en práctica bastante fácil, estas técnicas eran propensos a objetos y, por tanto criticó 7,8. Las mejoras técnicas fueron por tanto, necesario para superar estos artefactos y ser capaz de sondear lípidos partición balsas en células vivas.
Aquí presentamos un método para el análisis de sensibilidad de los lípidos balsas partición de las proteínas marcadas con fluorescencia o lípidos en la membrana plasmática de las células vivas. Este método, denominado espectroscopía de correlación de fluorescencia (FCS), se basa en la disparidad de tiempos de difusión de sondas fluorescentes situados dentro o fuera de las balsas de lípidos. De hecho, como se evidencia en las dos membranas artificiales y cultivos de células, las sondas se difundiría mucho más rápido fuera que dentro de las balsas de lípidos densos 9,10. Para determinar los tiempos de difusión, minuto fluctuaciones de fluorescencia se mide como una función del tiempo en un volumen focal (aproximadamente 1 femtoliter), situado en la membrana plasmática de las células con un microscopio confocal (fig.1). Las curvas de correlación automática, entonces se puede extraer de estas fluctuaciones y provistos de adecuados modelos de difusión matemáticos 11.
FCS puede ser utilizado para determinar la balsa lipídica de partición de sondas diferentes, siempre y cuando se fluorescentemente marcados. Fluorescente de marcado se puede lograr mediante la expresión de proteínas de fusión fluorescentes o mediante la unión de ligandos fluorescentes. Además, FCS puede ser utilizado no sólo en membranas artificiales y líneas celulares, sino también en cultivos primarios, como se ha descrito recientemente 12. También puede ser utilizado para seguir la dinámica de la partición de lípidos balsa después de la adición de drogas o lípidos de membrana cambio composición 12.
Protocolo
1. La calibración de la instalación de FCS
- Inicie el microscopio confocal, el láser, los ordenadores, incubadora de control de temperatura y CO2.
- Asegúrese de que el SPAD (solo diodo avalancha de fotones) está activado y el filtro de fluorescencia en el interior del SPAD se adapta bien a su muestra. Compruebe que el SPAD se sincroniza en el tiempo. Tenga cuidado para que sólo arranque el software de FCS, una vez la configuración de SPAD están listos para su adquisición.
- Preparar una solución fresca de la toxina del cólera-Alexa488 diluido en PBS para alcanzar una concentración de 1 mg / ml (17,5 nM).
- Optimizar imagen confocal de la solución usando la detección interna del microscopio.
- Cambiar a señalar el modo de escaneo y elegir un punto de la muestra de solución. Asegúrese de que la duración de la iluminación láser es lo suficientemente largo para llevar a cabo sus adquisiciones FCS (más de 5 minutos).
- Cambie a la detección externa por el SPAD y el software de FCS. Controle su señal de fluorescencia y asegúrese de que está bien suiTed el SPAD (señal suficiente, pero sin la saturación, de 10 000 a 50 000 cuentas / s está muy bien). Si es necesario, modificar la posición Z, la ganancia y / o la potencia del láser para conseguir señal de fluorescencia correcta.
- Adquirir 10 series de 30 segundos las mediciones. El tiempo de adquisición deben ser optimizados para la muestra (el mejor compromiso entre el muestreo y los problemas de photobleaching).
- Analice sus datos (ver paso 4) para asegurarse de que tiene una curva de auto-correlación correspondiente a una de las especies fluorescentes difunden en la solución (ecuación en la fig. 2) y que el tiempo de difusión obtenidos tras el montaje es correcto (aproximadamente 0,2 ms) (Fig. . 3).
2. La tinción de las células vivas con el marcador de lípidos balsas
- Placas HEK293 células en 8-así Labteks recubiertas con poli-L-lisina (1mg/ml) el día antes de formación de imágenes para asegurarse de que su adhesión es correcta. Utilizar medio sin rojo fenol para asegurar que no hay perturbación de la señal de fluorescencia.
- Lave las células dos veces con HBSS (buffer de Hank solución salina).
- Añadir la toxina del cólera-Alexa488 (1 mg / ml) / BSA (0,1%) en 500 l de HBSS a las células durante 30 minutos a 37 ° C. La toxina del cólera se une a gangliósido GM1, conocido por ser preferentemente dividido en las balsas lipídicas.
- Lave las células dos veces con HBSS (buffer de Hank solución salina).
3. FCS de adquisición de datos en las células vivas
- Colocar las células teñidas en la platina del microscopio. Haga la temperatura y CO 2 que las condiciones son óptimas caso contrario, podría dar lugar a artefactos en tiempos de difusión.
- Optimizar imagen confocal de una célula de interés usando la detección interna del microscopio (fig. 4).
- Cambiar a modo de punto de escaneo y elegir un punto en la membrana plasmática de la célula de interés. Realizar 1-2 imágenes en directo de la célula para asegurarse de que la célula no se está moviendo durante la adquisición.
- Cambie a la detección externa por el SPAD y el suave FCSware. Controle su señal de fluorescencia y asegúrese de que es muy adecuado para el SPAD (señal suficiente, pero la saturación, no, 10 000 a 50 000 cuentas / s está muy bien). Si es necesario, modificar la posición Z, la ganancia y / o la potencia del láser para conseguir señal de fluorescencia correcta. Si esto no es suficiente, usted puede considerar el cambio de las condiciones de tinción.
- Adquirir 10 muestras de 30 segundos las mediciones. Como la mayoría de las células se auto-fluorescente, puede ver una disminución de la fluorescencia durante las primeras adquisiciones, debido a la auto-fluorescencia decoloración.
4. FCS Análisis de Datos
- Establezca el intervalo de la correlación y la generación de auto-correlación de curvas con el software de FCS. El intervalo de correlación variará típicamente desde el menor tiempo de retraso resoluble hasta el mayor tiempo posible (como las estadísticas de la medición se vuelve más y más insuficiente cuando el tiempo máximo de correlación se aproxima el tiempo de adquisición total, el tiempo de retardo máximo está limitado a 80% del tiempo de adquisición sobre la Picoquant de software).
- Para cada muestra, exportar archivos correspondientes a las fluctuaciones de fluorescencia y auto-correlación como una función del tiempo.
- Use un análisis de datos y software de adaptación tales como el origen Pro para importar los archivos.
- Para cada muestra, asegúrese de que no hay photobleaching es decir, durante la adquisición de la fluorescencia se mantiene estable durante los 30 segundos. Deseche cualquier medida que muestra photobleaching ya que esto puede causar artificiales más largos tiempos de difusión.
- Comprobar que la forma de la restante curvas FCS es correcto (ver Fig. 5). Determinar la media curva de FCS.
- Ajustar la curva con el modelo matemático apropiado (Fig. 2). Este ajuste le dará el tiempo de difusión (s) y la proporción de moléculas que se difunden con este tiempo de difusión (ESE) (s).
5. Los resultados representativos
Un ejemplo de una calibración de FCS con una toxina del cólera-Alexa488 solución se muestra en la Figura 3 . Después de comprobar que las medidas individuales de fluorescencia como una función del tiempo no mostró ninguna fotoblanqueo (Figura 3A), individual y medio de curvas de FCS se calcularon. Mean curvas FCS se ajustaron con las ecuaciones que corresponden a diferentes modelos de difusión (ejemplos de la Figura 2). El parámetro clásicamente considerados para determinar la calidad de un ajuste es el coeficiente de determinación R 2. Cuanto más cerca R 2 a 1, mejor será el ajuste. En este caso, el modelo más exacto para adaptarse a la curva media FCS es la que describe una una población de moléculas que se difunden libremente fluorescentes en tres dimensiones (Ecuación 1 en la Figura 2 y Figura 3B). El tiempo de difusión derivado del ajuste es de 0,32 ms. Residuos de ajuste de curvas (Figura 3C) y R 2 factores (0,99906) dar una estimación de la calidad del ajuste.
Un ejemplo de análisis de FCS de la toxina del cólera Alexa488 manchada HEK293 células se muestra en la Figura 5. La media multifásica forma de la curva de FCS pone de manifiesto la existencia de poblaciones de moléculas fluorescentes con diferentes tiempos de difusión. El mejor ajuste para esta curva corresponde a un modelo con tres poblaciones de sondas fluorescentes: dos con una difusión obstaculizado (difusión en dos dimensiones como en el plano de membrana) y un difusor libremente en tres dimensiones (ecuación 2 en la Figura 2 y Figura 5) . Esta última población corresponde a moléculas fluorescentes que se desplazan fuera del plano de la membrana, es decir, ya sea vinculante o no vinculante a sus objetivos de la membrana, alcanzando la membrana a través de la vía de secreción o reciclado, o salir de la membrana por endocitosis. Los dos tiempos de difusión en la membrana, que corresponden a la toxina del cólera obligado a GM1, eran de 2 ms (25% de las moléculas), correspondiente a la difusión fuera de las balsas de lípidos, y 75 ms (50% de las moléculas), correspondiente a la difusión en las balsas de lípidos . Tenga en cuenta que cualquier fotoblanquearción durante la adquisición dará lugar a largos tiempos de difusión artificiales por lo tanto, posiblemente, la creación de un sesgo hacia la localización de GM1 en los dominios de balsas lipídicas.

Figura 1. Representación esquemática de la FCS de configuración (foto modificada de Marquer et al. 12)

Figura 2. Ejemplo de modelos de difusión y las ecuaciones correspondientes se utilizan para ajustar las curvas de autocorrelación. El parámetro estructura S se puede escribir como S = 0 z / w 0 con z 0 el radio focal efectiva a lo largo del eje óptico en 1/2 e intensidad y la w0 efectiva lateral de coordinación en la radio de 1/2 e intensidad. Estos valores se pueden extraer de un diferencial clásica función de punto (PSF) de medición.
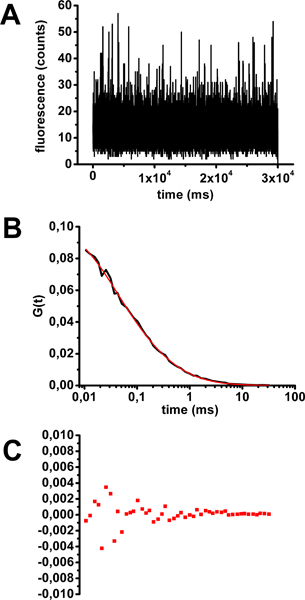
Figura 3. Evaluación de tiempo de difusión de la toxina del cólera-Alexa488 en solución para la calibración de FCS. A) fluctuación de fluorescencia como una función del tiempo para un ejemplo representativo de una adquisición de 30 segundos. B) La media de la curva de autocorrelación obtenido a partir de 10 muestras de 30 segundos de adquisición equipados con la ecuación 1 (ver Figura 2). C) Residuos de ajuste de la curva.
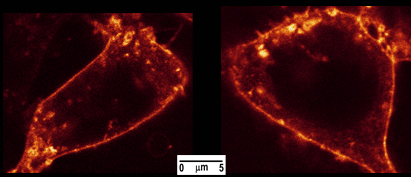
Figura 4. HEK-293 células se tiñeron con la toxina del cólera Alexa488. Las celdas eran imágenes en un microscopio confocal SP5 (Leica Microsystems, Wetzlar, Alemania) con la línea de láser de 488 nm interna. La fluorescencia se recogió con un objetivo x60 apocromático plan de inmersión en aceite entre 500y 650 nm.
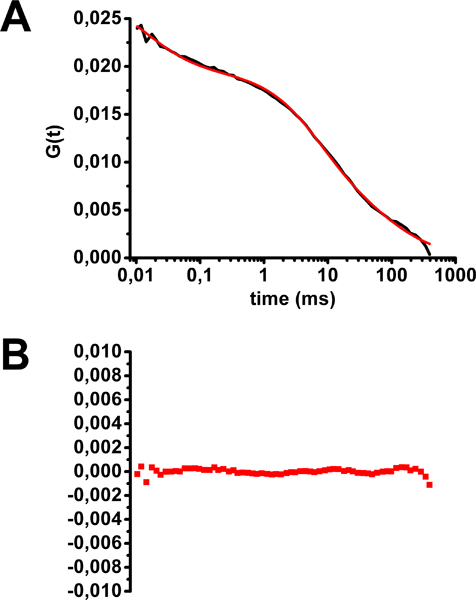
Figura 5. Evaluación de tiempo de difusión de la toxina del cólera-Alexa488 en la membrana plasmática de las células HEK293. A) La media de la curva de autocorrelación equipado con la ecuación 2 (véase Figura 2). B) Residuos de ajuste de la curva. (Modificado de Marquer et al. 12)
Discusión
El método de FCS se presenta aquí permite un análisis sensible y rápido de la compartimentación balsa lipídica de sondas fluorescentes de interés en células vivas. FCS combina la precisión de la localización de la microscopía confocal con la sensibilidad de conteo de fotones individuales. La diferencia principal entre FCS y técnicas estándar bioquímicos es que FCS permite la determinación absoluta de la partición de lípidos balsas de la diana y no la partición relativa como es el caso para el aislamien...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo fue apoyado por una beca de la Agence Nationale de la Recherche (ChoAD). También estamos agradecidos a la Fundación ICM (Instituto del Cerveau et de la Moelle) por su apoyo financiero.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| El cólera subunidad B de la toxina-Alexa 488 | Invitrogen | C-34775 | MW (pentámero) = 57 kg / mol |
| Microscopio confocal | Leica | SP5 | |
| Incubadora de la temperatura y el CO 2 de control | La vida de imágenes de servicios | El Cubo y la Caja de | |
| SPAD (solo diodo avalancha de fotones) | MPD (Micro Devices fotón) | PDM serie (100 m zona sensible) | |
| Paso alto 488 nm filtro | Semrock | 488 nm de bloqueo borde Brightline largo filtro de paso Parte #ails.aspx? id = FF01-488/LP-25 "target =" _blank "> FF01-488/LP-25 | |
| FCS unidad de detección | Picoquant | Picoharp 300 módulos | |
| Adquisición y software de auto-correlación | Picoquant | SymPhoTime | |
| Software de adaptación | OriginLab | OriginPro8 |
Referencias
- Brown, D. A., London, E. Functions of lipid rafts in biological membranes. Annu. Rev. Cell Dev. Biol. 14, 111-136 (1998).
- Simons, K., Gerl, M. J. Revitalizing membrane rafts: new tools and insights. Nat. Rev. Mol. Cell Biol. 11, 688-699 (2010).
- Simons, K., Ehehalt, R. Cholesterol, lipid rafts, and disease. J. Clin. Invest. 110, 597-603 (2002).
- Munro, S. Lipid rafts: elusive or illusive. Cell. , 115-377 (2003).
- Shaw, A. S. Lipid rafts: now you see them, now you don't. Nat. Immunol. 7, 1139-1142 (2006).
- Pralle, A., Keller, P., Florin, E. L., Simons, K., Horber, J. K. Sphingolipid-cholesterol rafts diffuse as small entities in the plasma membrane of mammalian cells. J. Cell Biol. 148, 997-1008 (2000).
- Brown, D. A., London, E. Structure and function of sphingolipid- and cholesterol-rich membrane rafts. J. Biol Chem. 275, 17221-17224 (2000).
- Sharma, P., Sabharanjak, S., Mayor, S. Endocytosis of lipid rafts: an identity crisis. Semin. Cell Dev. Biol. 13, 205-214 (2002).
- Kahya, N., Scherfeld, D., Bacia, K., Poolman, B., Schwille, P. Probing lipid mobility of raft-exhibiting model membranes by fluorescence correlation spectroscopy. J. Biol. Chem. 278, 28109-28115 (2003).
- Bacia, K., Scherfeld, D., Kahya, N., Schwille, P. Fluorescence correlation spectroscopy relates rafts in model and native membranes. Biophys J. 87, 1034-1043 (2004).
- Kim, S. A., Heinze, K. G., Schwille, P. Fluorescence correlation spectroscopy in living cells. Nat. Methods. 4, 963-973 (2007).
- Marquer, C. Local cholesterol increase triggers amyloid precursor protein-Bace1 clustering in lipid rafts and rapid endocytosis. FASEB J. 25, 1295-1305 (2011).
- Tian, Y., Martinez, M. M., Pappas, D. Fluorescence correlation spectroscopy: a review of biochemical and microfluidic applications. Appl. Spectrosc. 65, 115A-124A (2011).
- Haustein, E., Schwille, P. Fluorescence correlation spectroscopy: novel variations of an established technique. Annu. Rev. Biophys. Biomol. Struct. 36, 151-169 (2007).
- Ilien, B. Pirenzepine promotes the dimerization of muscarinic M1 receptors through a three-step binding process. J. Biol. Chem. 284, 19533-19543 (2009).
- Lieto, A. M., Cush, R. C., Thompson, N. L. Ligand-receptor kinetics measured by total internal reflection with fluorescence correlation spectroscopy. Biophys. J. 85, 3294-3302 (2003).
- Thompson, N. L., Burghardt, T. P., Axelrod, D. Measuring surface dynamics of biomolecules by total internal reflection fluorescence with photobleaching recovery or correlation spectroscopy. Biophys. J. 33, 435-454 (1981).
- Thompson, N. L., Steele, B. L. Total internal reflection with fluorescence correlation spectroscopy. Nat. Protoc. 2, 878-890 (2007).
- Eggeling, C. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457, 1159-1162 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49, 141-164 (2007).
- Shvartsman, D. E., Kotler, M., Tall, R. D., Roth, M. G., Henis, Y. I. Differently anchored influenza hemagglutinin mutants display distinct interaction dynamics with mutual rafts. J. Cell Biol. 163, 879-888 (2003).
- White, R. Holin triggering in real time. Proc. Natl. Acad. Sci. U.S.A. 108, 798-803 (2011).
- Petersen, N. O., Hoddelius, P. L., Wiseman, P. W., Seger, O., Magnusson, K. E. Quantitation of membrane receptor distributions by image correlation spectroscopy: concept and application. Biophys. J. 65, 1135-1146 (1993).
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88, 3601-3614 (2005).
- Digman, M. A. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89, 1317-1327 (2005).
- Digman, M. A. Fluctuation correlation spectroscopy with a laser-scanning microscope: exploiting the hidden time structure. Biophys. J. 88, L33-L36 (2005).
- Nohe, A., Keating, E., Fivaz, M., van der Goot, F. G., Petersen, N. O. Dynamics of GPI-anchored proteins on the surface of living cells. Nanomedicine. 2, 1-7 (2006).
- Semrau, S., Schmidt, T. Particle image correlation spectroscopy (PICS): retrieving nanometer-scale correlations from high-density single-molecule position data. Biophys. J. 92, 613-621 (2007).
- Bates, I. R. Membrane lateral diffusion and capture of CFTR within transient confinement zones. Biophys. J. 91, 1046-1058 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados