É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Determinação da Raft Lipid Particionamento de Sondas marcadas com fluorescência em células vivas por Espectroscopia de Correlação de Fluorescência (FCS)
Neste Artigo
Resumo
Uma técnica para sondar o particionamento jangada lipídico de proteínas fluorescentes na membrana plasmática de células vivas é descrito. Leva vantagem da disparidade em tempos de difusão de proteínas localizadas dentro ou fora de jangadas lipídicas. A aquisição pode ser realizada de forma dinâmica em condições de controlo ou após a adição da droga.
Resumo
Nos últimos quinze anos, a noção de que as membranas celulares não são homogêneas e dependem de microdomínios de exercer suas funções tornou-se amplamente aceita. Jangadas lipídicas são microdomínios de membrana enriquecidos em colesterol e esfingolipídios. Eles desempenham um papel em processos fisiológicos celulares, tais como sinalização, e 1,2 tráfico, mas também estão pensados para ser os principais intervenientes em várias doenças, incluindo infecções virais ou bacterianas e doenças neurodegenerativas 3.
No entanto, sua existência é ainda uma questão controversa 4,5. Na verdade, o tamanho rafts lipídicos foi estimado em cerca de 20 nm 6, muito abaixo do limite de resolução do microscópio convencional (cerca de 200 nm), impossibilitando a sua imagem direta. Até agora, as principais técnicas utilizadas para avaliar a partição de proteínas de interesse dentro de balsas lipídicas foram Membranas resistentes detergentes (DRMs) de isolamento, e co-patch com os anticorpos. Beca Embora amplamente utilizadauso de sua execução bastante fácil, estas técnicas eram propensos a artefatos e, portanto, criticou 7,8. Melhoramentos técnicos foram, portanto, necessário para ultrapassar estes artefactos e para ser capaz de detectar partição jangadas lipídica em células vivas.
Apresentamos aqui um método para a análise sensível de partição lípido jangadas de proteínas fluorescentes-etiquetados ou lípidos na membrana plasmática de células vivas. Este método, denominado Espectroscopia de Correlação de fluorescência (FCS), baseia-se na disparidade em tempos de difusão de sondas fluorescentes localizados dentro ou fora de jangadas lipídicas. De fato, como evidenciado em ambas as membranas artificiais e culturas de células, as sondas seria difundir muito mais rapidamente do lado de fora balsas lipídicas densos 9,10. Para determinar tempos de difusão, hora flutuações de fluorescência são medidos como uma função do tempo em um volume focal (aproximadamente 1 femtoliter), localizada na membrana plasmática de células com um microscópio confocal (Fig.1). As curvas de auto-correlação pode, então, ser extraída estas flutuações e equipado com modelos de difusão adequadas matemáticas 11.
FCS pode ser usado para determinar a jangada lípido particionamento de sondas diferentes, contanto que eles são fluorescentemente marcado. Fluorescente marcação pode ser conseguida através da expressão de proteínas de fusão fluorescentes ou por ligação de ligandos fluorescentes. Além disso, FCS pode ser usado não apenas em membranas artificiais e linhas de células, mas também em culturas primárias, como descrito recentemente 12. Também pode ser utilizado para seguir a dinâmica da compartimentação jangada lípido após a adição da droga ou a mudança de membrana composição lipídica 12.
Protocolo
1. Calibração do Setup FCS
- Inicie o microscópio confocal, lasers, computadores, incubadora para controle de temperatura e CO 2.
- Certifique-se que a SPAD (Diodo Avalanche de fóton único) é ligado e que o filtro de fluorescência dentro do SPAD está bem adaptado à sua amostra. Verificar que a SPAD está sincronizado com o tempo. Cuidado apenas para iniciar o seu software FCS uma vez que suas configurações SPAD estão prontos para a aquisição.
- Prepara-se uma solução fresca de toxina da cólera-Alexa488 diluído em PBS para atingir uma concentração de 1 ug / ml (17,5 nM).
- Otimizar imagem confocal da solução utilizando detecção interna do microscópio.
- Mude para o modo de digitalização apontar e escolher um ponto na amostra solução. Assegure-se a duração da iluminação laser é suficientemente longo para executar as suas aquisições de FCS (> 5 minutos).
- Alternar para detecção externa pelo SPAD e para o software de FCS. Monitore seu sinal de fluorescência e verifique se ele está bem suibuído para a SPAD (sinal suficiente, mas sem saturação, 10 000 a 50 000 contagens / s é bom). Se necessário, modificar a posição z, ganho e / ou potência do laser para atingir sinal de fluorescência correcta.
- Adquirir 10 conjuntos de 30 medições segundos. Tempo de aquisição deve ser otimizado para a sua amostra (compromisso entre a melhor amostragem e problemas fotodegradação).
- Analise seus dados (ver passo 4) para assegurar que você tem uma curva de auto-correlação correspondente a uma espécie fluorescentes difusão em solução (equação da Figura. 2) e que o tempo de difusão obtidas após a montagem está correta (aproximadamente 0,2 ms) (Fig. . 3).
2. A coloração de células vivas com marcador de balsas lipídicas
- Placa células HEK293 sobre 8-assim Labteks revestidas com poli-L-lisina (1mg/ml) um dia antes de imagem para se certificar de que a sua aderência é correcta. Use meio sem vermelho de fenol para assegurar que não há perturbação do sinal de fluorescência.
- Lave as células duas vezes com HBSS (Solução de Hank sal tamponada).
- Adicionar toxina da cólera-Alexa488 (1 ug / ml) / BSA (0,1%) em 500 uL HBSS para as células durante 30 minutos a 37 ° C. Toxina da cólera vai ligar para o gangliosídeo GM1, conhecido por ser preferencialmente particionado em balsas lipídicas.
- Lave as células duas vezes com HBSS (Solução de Hank sal tamponada).
3. FCS Aquisição de Dados em células vivas
- Colocar as células coradas sobre a platina do microscópio. Faça temperatura certa e CO 2 condições são ideais caso contrário isso pode levar a artefactos em tempos de difusão.
- Otimizar imagem confocal de uma célula de interesse usando detecção interna do microscópio (Fig. 4).
- Para apontar modo de digitalização e escolher um ponto na membrana plasmática da célula de interesse. Execute 1-2 imagens ao vivo da célula para garantir que a célula não está se movendo durante a aquisição.
- Mudar para detecção externa pela SPAD e ao macio FCSware. Monitore seu sinal de fluorescência e verifique se ele está bem adaptado à SPAD (sinal suficiente, mas sem saturação, de 10 000 para 50 000 contagens / s é bom). Se necessário, modificar a posição z, ganho e / ou potência do laser para atingir sinal de fluorescência correcta. Se isso não for suficiente, você pode considerar mudanças nas condições de coloração.
- Adquirir 10 amostras de 30 medições segundos. Como a maioria das células são auto-fluorescente, você poderá ver uma diminuição na fluorescência durante as primeiras aquisições, devido a auto-fluorescência desbotamento.
4. FCS Análise de Dados
- Defina o intervalo de correlação e gerar auto-correlação de curvas com o software FCS. O intervalo de correlação variará tipicamente de o menor tempo de retardação resolúvel até o maior tempo possível (como as estatísticas da medição torna-se cada vez mais insuficiente quando o tempo de correlação máxima se aproxima o tempo de aquisição total, o tempo de atraso máximo é limitado a 80% do tempo de aquisição sobre a Picoquant software).
- Para cada amostra, exportar arquivos correspondentes a flutuações de fluorescência e de auto-correlação como uma função do tempo.
- Use uma análise de dados e software de adaptação, tais como origem Pro para importar os arquivos.
- Para cada amostra, certifique-se não há fotobranqueamento durante a aquisição ou seja, a fluorescência permanece estável durante os 30 segundos. Descarte qualquer medida exibindo fotodegradação, pois isso pode causar artificiais vezes maior difusão.
- Verificar que a forma do restante FCS curvas é correcta (ver Fig. 5). Determinar a curva de FCS média.
- Ajustar a curva com o modelo matemático apropriado (Fig. 2). Este ajuste vai dar-lhe o tempo de difusão (s) e da proporção de moléculas de difusão com este tempo de difusão (ese) (s).
5. Os resultados representativos
Um exemplo de uma calibração de FCS, com uma solução de toxina da cólera-Alexa488 é mostrado na Figura 3 . Depois de verificar que as medidas individuais de fluorescência como uma função do tempo não mostraram qualquer fotobranqueamento (Figura 3A), individuais e médios curvas de FCS foram calculados. Curvas médias FCS foram equipados com as equações que correspondem a modelos de difusão diferentes (exemplos da Figura 2). O parâmetro classicamente considerado para determinar a qualidade de um ajuste é o coeficiente de determinação R 2. Quanto mais próximo de R 2 é de 1, melhor o ajuste. Neste caso, o modelo mais preciso para se ajustar a curva de FCS média é a uma descrevendo uma população de moléculas fluorescentes que difundem livremente em três dimensões (equação 1 na Figura 2 e Figura 3B). O tempo de difusão derivado do ajuste é 0,32 ms. Resíduos de ajuste de curva-(Figura 3C) e factor de R 2 (0,99906) fornecer uma estimativa da qualidade do ajuste.
Um exemplo de análise de FCS para a cólera manchado-toxina Alexa488 HEK293 células é mostrado na Figura 5. A forma da curva média multifásica FCS revela a existência de populações de moléculas fluorescentes com diferentes tempos de difusão. O melhor ajuste para esta curva corresponde a um modelo com três populações de sondas fluorescentes: dois com uma difusão impedida (difusão em duas dimensões como no plano da membrana) e um difusor livremente em três dimensões (equação 2 na Figura 2 e Figura 5) . Esta última corresponde à população de moléculas fluorescentes que se deslocam para fora do plano da membrana, isto é, ou ligação ou unbinding para os seus objectivos de membrana, atingindo a membrana através da via de secreção ou reciclagem, ou deixando a membrana por endocitose. Os dois tempos de difusão na membrana, correspondentes a toxina da cólera ligado a GM1, eram de 2 ms (25% de moléculas), correspondendo a difusão para fora do balsas lipídicas, e 75 ms (50% de moléculas), correspondendo a difusão em balsas lipídicas . Por favor, note que qualquer photobleachção durante a aquisição levará a difusão artificiais vezes mais, portanto, possivelmente criando um viés para a localização de GM1 em domínios rafts lipídicos.

Figura 1. Representação esquemática do conjunto de FCS até-(modificado a partir de imagem Marquer et al. 12)

Figura 2. Exemplo de modelos de difusão e equações correspondentes utilizados para o ajuste de curvas de autocorrelação. O parâmetro S estrutura pode ser escrita como S = 0 z / w 0 com 0 z o raio eficaz focal ao longo do eixo óptico a 1/2 e intensidade e w0 a la eficazraio focal parenteral a 1 / e intensidade 2. Estes valores podem ser extraídos a partir de um ponto de propagação clássica função de medição (PSF).
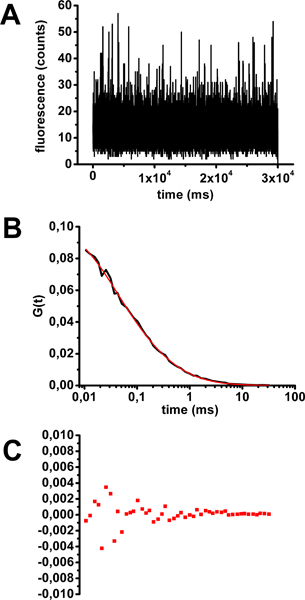
Figura 3. Avaliação do tempo de difusão de toxina da cólera-Alexa488 em solução para a calibração de FCS. A) flutuação de fluorescência como uma função do tempo para um exemplo representativo de um segundo aquisição 30. B) A média de curva de autocorrelação obtido a partir de 10 amostras de 30 aquisição segundo equipado com a equação 1 (ver Figura 2). C) resíduos de ajuste de curva.
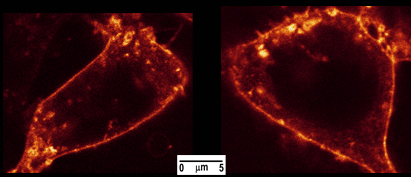
Figura 4. HEK-293 de células coradas com toxina da cólera-Alexa488. As células foram gravadas em um microscópio confocal SP5 (Leica Microsystems, Wetzlar, Alemanha) com a linha de laser interna 488nm. Fluorescência foi coletado com um plano objectivo Apochromat x60 imersão em óleo entre 500e 650 nm.
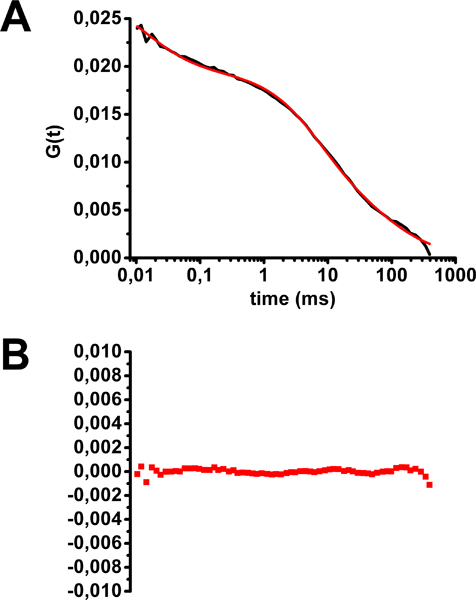
Figura 5. Avaliação do tempo de difusão de toxina da cólera-Alexa488 na membrana plasmática de células HEK293. A) média da curva de autocorrelação equipado com a equação 2 (ver Figura 2). B) Resíduos de ajuste de curva. (Modificado de Marquer et al. 12)
Discussão
O método aqui apresentado FCS permite uma análise sensível e rápido da compartimentação jangada lipídico de sondas fluorescentes de interesse em células vivas. FCS combina a precisão da localização de microscopia confocal com a sensibilidade de contagem de fotão único. A principal diferença entre FCS e técnicas padrão de bioquímicos é que permite a determinação de FCS absoluta da partição jangadas lipídica do alvo e não a partição relativa como é o caso para GDD isolamento ou de co-patching. <...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado por uma subvenção da Agence Nationale de la Recherche (ChoAD). Também somos gratos ao ICM Fondation (Institut du Cerveau et de la Moelle) pelo apoio financeiro.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| Cólera subunidade toxina B-488 Alexa | Invitrogen | C-34.775 | MW (pentâmero) = 57 kg mol / |
| Microscópio confocal | Leica | SP5 | |
| Incubadora de temperatura e CO controle 2 | Vida serviços de imagem | O Cubo ea Caixa | |
| SPAD (Diodo Avalanche de fóton único) | MPD (Micro Devices Photon) | PDM serie (100 mM área sensível) | |
| Passa-alto 488 nm filtro | Semrock | 488 nm de bloqueio borda BrightLine longo filtro passa Part #ails.aspx? id = FF01-488/LP-25 "target =" _blank "> FF01-488/LP-25 | |
| FCS unidade de detecção | Picoquant | Picoharp módulo 300 | |
| Aquisição e auto-correlação software | Picoquant | SymPhoTime | |
| Software de adaptação | OriginLab | OriginPro8 |
Referências
- Brown, D. A., London, E. Functions of lipid rafts in biological membranes. Annu. Rev. Cell Dev. Biol. 14, 111-136 (1998).
- Simons, K., Gerl, M. J. Revitalizing membrane rafts: new tools and insights. Nat. Rev. Mol. Cell Biol. 11, 688-699 (2010).
- Simons, K., Ehehalt, R. Cholesterol, lipid rafts, and disease. J. Clin. Invest. 110, 597-603 (2002).
- Munro, S. Lipid rafts: elusive or illusive. Cell. , 115-377 (2003).
- Shaw, A. S. Lipid rafts: now you see them, now you don't. Nat. Immunol. 7, 1139-1142 (2006).
- Pralle, A., Keller, P., Florin, E. L., Simons, K., Horber, J. K. Sphingolipid-cholesterol rafts diffuse as small entities in the plasma membrane of mammalian cells. J. Cell Biol. 148, 997-1008 (2000).
- Brown, D. A., London, E. Structure and function of sphingolipid- and cholesterol-rich membrane rafts. J. Biol Chem. 275, 17221-17224 (2000).
- Sharma, P., Sabharanjak, S., Mayor, S. Endocytosis of lipid rafts: an identity crisis. Semin. Cell Dev. Biol. 13, 205-214 (2002).
- Kahya, N., Scherfeld, D., Bacia, K., Poolman, B., Schwille, P. Probing lipid mobility of raft-exhibiting model membranes by fluorescence correlation spectroscopy. J. Biol. Chem. 278, 28109-28115 (2003).
- Bacia, K., Scherfeld, D., Kahya, N., Schwille, P. Fluorescence correlation spectroscopy relates rafts in model and native membranes. Biophys J. 87, 1034-1043 (2004).
- Kim, S. A., Heinze, K. G., Schwille, P. Fluorescence correlation spectroscopy in living cells. Nat. Methods. 4, 963-973 (2007).
- Marquer, C. Local cholesterol increase triggers amyloid precursor protein-Bace1 clustering in lipid rafts and rapid endocytosis. FASEB J. 25, 1295-1305 (2011).
- Tian, Y., Martinez, M. M., Pappas, D. Fluorescence correlation spectroscopy: a review of biochemical and microfluidic applications. Appl. Spectrosc. 65, 115A-124A (2011).
- Haustein, E., Schwille, P. Fluorescence correlation spectroscopy: novel variations of an established technique. Annu. Rev. Biophys. Biomol. Struct. 36, 151-169 (2007).
- Ilien, B. Pirenzepine promotes the dimerization of muscarinic M1 receptors through a three-step binding process. J. Biol. Chem. 284, 19533-19543 (2009).
- Lieto, A. M., Cush, R. C., Thompson, N. L. Ligand-receptor kinetics measured by total internal reflection with fluorescence correlation spectroscopy. Biophys. J. 85, 3294-3302 (2003).
- Thompson, N. L., Burghardt, T. P., Axelrod, D. Measuring surface dynamics of biomolecules by total internal reflection fluorescence with photobleaching recovery or correlation spectroscopy. Biophys. J. 33, 435-454 (1981).
- Thompson, N. L., Steele, B. L. Total internal reflection with fluorescence correlation spectroscopy. Nat. Protoc. 2, 878-890 (2007).
- Eggeling, C. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457, 1159-1162 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49, 141-164 (2007).
- Shvartsman, D. E., Kotler, M., Tall, R. D., Roth, M. G., Henis, Y. I. Differently anchored influenza hemagglutinin mutants display distinct interaction dynamics with mutual rafts. J. Cell Biol. 163, 879-888 (2003).
- White, R. Holin triggering in real time. Proc. Natl. Acad. Sci. U.S.A. 108, 798-803 (2011).
- Petersen, N. O., Hoddelius, P. L., Wiseman, P. W., Seger, O., Magnusson, K. E. Quantitation of membrane receptor distributions by image correlation spectroscopy: concept and application. Biophys. J. 65, 1135-1146 (1993).
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88, 3601-3614 (2005).
- Digman, M. A. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89, 1317-1327 (2005).
- Digman, M. A. Fluctuation correlation spectroscopy with a laser-scanning microscope: exploiting the hidden time structure. Biophys. J. 88, L33-L36 (2005).
- Nohe, A., Keating, E., Fivaz, M., van der Goot, F. G., Petersen, N. O. Dynamics of GPI-anchored proteins on the surface of living cells. Nanomedicine. 2, 1-7 (2006).
- Semrau, S., Schmidt, T. Particle image correlation spectroscopy (PICS): retrieving nanometer-scale correlations from high-density single-molecule position data. Biophys. J. 92, 613-621 (2007).
- Bates, I. R. Membrane lateral diffusion and capture of CFTR within transient confinement zones. Biophys. J. 91, 1046-1058 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados