このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
脂質ラフトの決定は、蛍光相関分光法(FCS)による生細胞の蛍光タグ付きプローブの分割
要約
生きた細胞の原形質膜における蛍光タンパク質の脂質ラフトのパーティショニングを調べるための手法が説明されています。これは、脂質ラフトの内側または外側に位置するタンパク質の拡散時間に格差を利用しています。買収は、コントロール条件でまたは薬物を添加した後、動的に実行することができます。
要約
過去15年間で、細胞膜が均一ではありませんし、その機能を発揮するマイクロドメインに依存している概念は広く受け入れられるようになっています。脂質ラフトは、コレステロールとスフィンゴ脂質に富んだ細胞膜マイクロドメインである。彼らはこのようなシグナル伝達などの細胞生理学的プロセスにおいて役割を果たしており、人身売買の1,2だけでなく、ウイルスや細菌感染症や神経変性疾患の3を含むいくつかの疾患の主要なプレーヤーであると考えられている。
まだ彼らの存在は依然として論争4,5の問題です。確かに、脂質ラフトの大きさは、このように彼らのダイレクトイメージングを排除し、はるかに、従来の顕微鏡の分解能の限界(200 nm付近)の下で、約20 nmの6であると推定されている。現在までに、脂質ラフト内の目的タンパク質のパーティションを評価するために使用される主な技術は、界面活性剤耐性膜(ラフト)抗体を用いた分離と共同パッチでした。でも広く使われているbeca彼らは、むしろ簡単な実装を使用して、これらの技術は工芸品に傾向があったので、7,8を批判した 。技術的な改良は、したがって、これらの工芸品を克服し、生きた細胞における脂質ラフトのパーティションを調べることができるように必要であった。
ここでは、生きた細胞の細胞膜に蛍光タグタンパク質や脂質の脂質ラフトパーティションの高感度分析手法を提案する。このメソッドは、蛍光相関分光法(FCS)と呼ばれる、脂質ラフトの内側または外側に位置する蛍光プローブの拡散時間の格差に依存しています。実際には、人工膜や細胞培養の両方で明らかなように、プローブは、はるかに高速外緻密な脂質ラフト9,10の内側よりも拡散するだろう。拡散時間を決定するために、分蛍光変動がある( 図共焦点顕微鏡で細胞の細胞膜に位置し、焦点体積(約1フェムトリットル)内の時間の関数として測定される1)。自己相関曲線は、これらの変動から引き出され、適切な数学的拡散モデル11に取り付けることができます。
FCSは、それらが蛍光標識されている限り、様々なプローブのパーティショニング脂質ラフトを決定するために使用することができます。蛍光タグは、蛍光融合蛋白質の発現によって、または蛍光リガンドの結合によって達成することができます。また、FCSは、最近12を説明したように、人工の膜と細胞株でも初代培養だけでなく使用することができます。また、薬剤の添加や膜脂質組成の変化12の後にパーティショニング脂質ラフトの動態を追跡するために使うことができます。
プロトコル
1。 FCSセットアップのキャリブレーション
- 温度とCO 2制御のための共焦点顕微鏡、レーザー、コンピューター、インキュベーターを開始します。
- SPAD(単一光子アバランシェダイオード)が上にあり、SPAD内部の蛍光フィルターはよくあなたのサンプルに適していることを確認してください。 SPADは、時間で同期されていることを確認します。あなたのSPAD設定が取得の準備ができているに一度だけあなたのFCSのソフトウェアを起動するために注意してください。
- 1μg/ mlの(17.5 nm)の濃度に到達するためにPBSで希釈したコレラ毒素-Alexa488の新鮮な溶液を調製します。
- 顕微鏡の内部検出を使用したソリューションの共焦点イメージングを最適化します。
- スキャンモードを指し、溶液試料中のポイントを選択するスイッチ。レーザー照射の時間は(> 5分)あなたのFCSの買収を実行するために十分な長さであることを確認してください。
- SPADによる外部検出およびFCSのソフトウェアに切り替えます。あなたの蛍光シグナルを監視し、それがよく隋であることを確認してくださいSPADのテッド(十分な信号がない彩度、10 000 50 000までカウント/ sで結構です)。必要であれば、z位置、利得および/または適切な蛍光シグナルを達成するためにレーザーパワーを変更します。
- 30秒測定の10セットを取得します。アクイジション時間(最高サンプリングと退色の問題の間の妥協点)をサンプル用に最適化する必要があります。
- 図 ((約0.2ミリ)が溶液中に拡散1蛍光種に対応する自己相関曲線( 図の式2)を持っていることを確認して、フィッティング後に得られる拡散時間が正しいことをする(ステップ4を参照)データを分析する3)。
2。脂質ラフトマーカーと共に生きた細胞の染色
- ポリ-L-リジン(1mg/ml)の接着が正しいことを確認するために、イメージングの前日で被覆された8ウェルLabteks上にプレートしたHEK293細胞。蛍光シグナルのない摂動がないことを確認するにはフェノールレッドを含まない培地を使用しています。
- HBSS(ハンクス緩衝塩溶液)で細胞を2回洗浄します。
- 37℃で30分間細胞に500μlのHBSSでコレラ毒素-Alexa488(1μg/ ml)を/ BSA(0.1%)を追加℃、コレラ毒素は、優先的に脂質ラフトに分割することが知られているガングリオシドGM1に結合するであろう。
- HBSS(ハンクス緩衝塩溶液)で細胞を2回洗浄します。
3。生きている細胞にFCSデータ·アクイジション
- 顕微鏡ステージ上で染色した細胞を配置します。温度とCO 2の条件は、これは拡散時間の工芸品につながる可能性がありそうでなければ最適であることを確認してください。
- 顕微鏡内部の検出( 図4)を使用して、目的の細胞の共焦点イメージングを最適化します。
- スキャンモードを指し、目的の細胞の原形質膜内のポイントを選択して切り替えます。細胞が取り込み中に移動されていないことを確認するには、セル1-2のライブ映像を実行します。
- SPADすることにより、外部検出に切り替えて、FCSソフトにウェア。あなたの蛍光シグナルを監視し、それがよく(十分な信号がない彩度、10 000から50 000カウント/ sは結構です)SPADに適していることを確認してください。必要であれば、z位置、利得および/または適切な蛍光シグナルを達成するためにレーザーパワーを変更します。これが十分でない場合は、染色条件を変えると考えるかもしれません。
- 30秒測定の10サンプルを取得します。ほとんどの細胞は自動蛍光性であるように、自家蛍光フェージングに起因する、最初の買収時の蛍光の減少が表示されることがあります。
4。 FCSデータ解析
- 相関間隔を設定し、FCSのソフトウェアとの自己相関曲線を生成します。相関間隔は一般的に最大の相関時間は合計捕捉時間に近づくと最短解決の遅れ時間から最大可能な最長時間に(測定の統計はますます不十分になると、最大遅延時間が80%に制限されている範囲になりますPicoqua上に捕捉時間NTソフトウェア)。
- それぞれのサンプルについては、時間の関数としての蛍光の変動と自己相関に対応するファイルをエクスポートします。
- データ分析と、そのようなファイルをインポートするには、Originのプロとしてフィッティングソフトウェアを使用しています。
- それぞれのサンプルについては、蛍光が30秒の間に安定したまま、すなわち全く退色は、取得時に存在しないことを確認してください。これは人工的な長い拡散時間の原因となりますので退色が表示され、任意の測定値を破棄します。
- 残りのFCSカーブの形状は( 図5参照)が正しいことを確認します。平均FCSカーブを決定します。
- 適切な数学モデル( 図2)カーブに合う。このフィットは、あなたの拡散時間(s)と、この(ESE)の拡散時間(s)と拡散分子の割合を与える。
5。代表的な結果
コレラ毒素Alexa488ソリューションとFCSのキャリブレーションの例を図3に示されている。時間の関数として蛍光の個々の対策を確認した後、任意の退色( 図3A)を示す個人やFCSカーブを算出したわけではありませんでした。 FCSカーブは様々な拡散モデル( 図2の例)に対応する方程式を装備したを意味します。古典的なフィットの質を決定すると考えパラメータは、決定係数R 2である。 R 2に近い優れた、1に適合している。このケースでは、平均FCSカーブに合わせて、最も正確なモデルが3次元( 図2および図3Bの式1)で自由に拡散する蛍光分子の集団を記述するものです。フィットから派生した拡散時間は0.32ミリ秒です。カーブフィッティング( 図3C)およびR 2は係数(0.99906)からの残差はフィットの品質の推定値を与える。
コレラ毒素Alexa488で染色HEK2のFCS解析の例93細胞は、 図5に示します。多面平均FCSカーブの形状が異なる拡散時間で蛍光分子の集団の存在を明らかにする。妨げ拡散(膜面内の2つの次元の拡散)の2次元および3次元で自由に拡散します( 図2と図5の式(2)):この曲線のベストフィットは、蛍光プローブの3つの集団を持つモデルに対応。この後者の集団は、分泌物やリサイクル経路を介して膜に到達するか、エンドサイトーシスによって細胞膜を残し、それらの膜ターゲットへのバインディングまたはアンバインドのいずれかの膜面、すなわち、外に移動する蛍光分子に対応しています。 GM1に結合したコレラ毒素に対応した膜で2つの拡散時間は、脂質ラフトの外に拡散に対応した2つのMS(分子の25%)と、脂質ラフトの拡散に対応する75ミリ秒(分子の50%)であった。すべての光退色に注意してください買収時のINGは、人工的な長い拡散時間はこうしておそらく脂質ラフトドメインのGM1の局在化に向かってバイアスを作成することにつながるでしょう。

図1。FCSセットアップ(Marquerらから変更された画像12)の模式図

図2拡散モデルと自己相関曲線をフィットするために使用される対応する式の例を次に示します。構造パラメータSはS = Z 0 / W、Z 0 0 1 / e 2の強度で光軸に沿って効果的な焦点の半径とW0効果的なアラカルトのように書くことができます1 / e 2の強度でteralフォーカル半径。これらの値は、古典的な点広がり関数(PSF)の測定から抽出することができます。
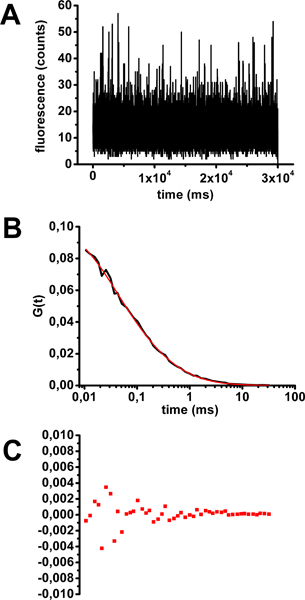
図3 FCSキャリブレーション用溶液中でのコレラ毒素Alexa488の拡散時間の評価。 30秒の買収の代表例の時間の関数としてA)蛍光変動。 B)式(1)を装備し、30秒の取得の10サンプル(図2を参照)。C)曲線の当てはめからの残差から得られる自己相関曲線を意味します。
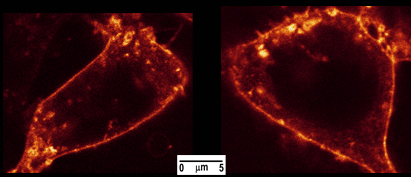
図4:HEK-293細胞は、コレラ毒素-Alexa488で染色した。細胞は内部488nmのレーザーラインでSP5共焦点顕微鏡(ライカマイクロシステムズ、ヴェッツラー、ドイツ)上に結像された。蛍光は500〜X60プランアポクロマートの油浸対物レンズで集められた650 nmである。
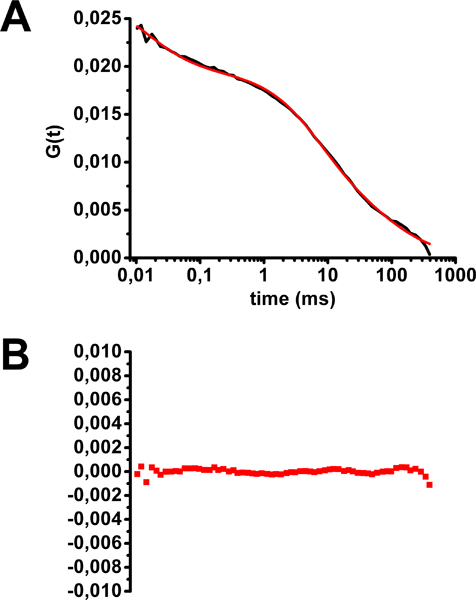
図5。HEK293細胞の細胞膜におけるコレラ毒素-Alexa488の拡散時間の評価。 A)( 図2を参照)、式(2)を装備し、自己相関曲線を意味します。 Bカーブフィッティングから)残差。 (Marquerらから変更された12)
ディスカッション
ここで紹介するFCS法は、生きた細胞への関心の蛍光プローブの分割脂質ラフトの高感度で迅速な分析を可能にします。 FCSは、単一光子計数の感度を持つ共焦点顕微鏡の局在化の精度を兼ね備えています。 FCS、標準的な生化学的手法の主な違いは、ラフトの分離又は共パッチの場合のようにFCSがターゲットの脂質ラフトのパーティションではなく、相対的なパーティションの絶対的な決定を?...
開示事項
利害の衝突が宣言されません。
謝辞
この作品は、AFP通信国立·デ·ラ·ルシェルシュ(ChoAD)からの助成金によってサポートされていました。我々はまた、財政支援のための財団ICM(研究所·デュ·CerveauらデラMoelle)に感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | コメント |
| コレラ毒素サブユニットB-アレクサ488 | インビトロジェン | C-34775 | MW(ペンタマー)= 57キロ/モル |
| 共焦点顕微鏡 | ライカ | SP5 | |
| 温度とCO 2制御のためのインキュベーター | 寿命イメージングサービス | キューブとボックス | |
| SPAD(単一光子アバランシェダイオード) | MPD(マイクロ光子デバイス) | PDMセリエA(100μmの敏感なエリア) | |
| 高域488 nmのフィルター | Semrock | エッジBrightLineロングパスフィルタをブロックする488 nmの パーツ#ails.aspx?ID = FF01-488/LP-25 "ターゲット=" _blank "> FF01-488/LP-25 | |
| FCS検出部 | Picoquant | Picoharp 300モジュール | |
| 取得と自己相関のソフトウェア | Picoquant | SymPhoTime | |
| フィッティングソフト | OriginLabの | OriginPro8 |
参考文献
- Brown, D. A., London, E. Functions of lipid rafts in biological membranes. Annu. Rev. Cell Dev. Biol. 14, 111-136 (1998).
- Simons, K., Gerl, M. J. Revitalizing membrane rafts: new tools and insights. Nat. Rev. Mol. Cell Biol. 11, 688-699 (2010).
- Simons, K., Ehehalt, R. Cholesterol, lipid rafts, and disease. J. Clin. Invest. 110, 597-603 (2002).
- Munro, S. Lipid rafts: elusive or illusive. Cell. , 115-377 (2003).
- Shaw, A. S. Lipid rafts: now you see them, now you don't. Nat. Immunol. 7, 1139-1142 (2006).
- Pralle, A., Keller, P., Florin, E. L., Simons, K., Horber, J. K. Sphingolipid-cholesterol rafts diffuse as small entities in the plasma membrane of mammalian cells. J. Cell Biol. 148, 997-1008 (2000).
- Brown, D. A., London, E. Structure and function of sphingolipid- and cholesterol-rich membrane rafts. J. Biol Chem. 275, 17221-17224 (2000).
- Sharma, P., Sabharanjak, S., Mayor, S. Endocytosis of lipid rafts: an identity crisis. Semin. Cell Dev. Biol. 13, 205-214 (2002).
- Kahya, N., Scherfeld, D., Bacia, K., Poolman, B., Schwille, P. Probing lipid mobility of raft-exhibiting model membranes by fluorescence correlation spectroscopy. J. Biol. Chem. 278, 28109-28115 (2003).
- Bacia, K., Scherfeld, D., Kahya, N., Schwille, P. Fluorescence correlation spectroscopy relates rafts in model and native membranes. Biophys J. 87, 1034-1043 (2004).
- Kim, S. A., Heinze, K. G., Schwille, P. Fluorescence correlation spectroscopy in living cells. Nat. Methods. 4, 963-973 (2007).
- Marquer, C. Local cholesterol increase triggers amyloid precursor protein-Bace1 clustering in lipid rafts and rapid endocytosis. FASEB J. 25, 1295-1305 (2011).
- Tian, Y., Martinez, M. M., Pappas, D. Fluorescence correlation spectroscopy: a review of biochemical and microfluidic applications. Appl. Spectrosc. 65, 115A-124A (2011).
- Haustein, E., Schwille, P. Fluorescence correlation spectroscopy: novel variations of an established technique. Annu. Rev. Biophys. Biomol. Struct. 36, 151-169 (2007).
- Ilien, B. Pirenzepine promotes the dimerization of muscarinic M1 receptors through a three-step binding process. J. Biol. Chem. 284, 19533-19543 (2009).
- Lieto, A. M., Cush, R. C., Thompson, N. L. Ligand-receptor kinetics measured by total internal reflection with fluorescence correlation spectroscopy. Biophys. J. 85, 3294-3302 (2003).
- Thompson, N. L., Burghardt, T. P., Axelrod, D. Measuring surface dynamics of biomolecules by total internal reflection fluorescence with photobleaching recovery or correlation spectroscopy. Biophys. J. 33, 435-454 (1981).
- Thompson, N. L., Steele, B. L. Total internal reflection with fluorescence correlation spectroscopy. Nat. Protoc. 2, 878-890 (2007).
- Eggeling, C. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457, 1159-1162 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49, 141-164 (2007).
- Shvartsman, D. E., Kotler, M., Tall, R. D., Roth, M. G., Henis, Y. I. Differently anchored influenza hemagglutinin mutants display distinct interaction dynamics with mutual rafts. J. Cell Biol. 163, 879-888 (2003).
- White, R. Holin triggering in real time. Proc. Natl. Acad. Sci. U.S.A. 108, 798-803 (2011).
- Petersen, N. O., Hoddelius, P. L., Wiseman, P. W., Seger, O., Magnusson, K. E. Quantitation of membrane receptor distributions by image correlation spectroscopy: concept and application. Biophys. J. 65, 1135-1146 (1993).
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88, 3601-3614 (2005).
- Digman, M. A. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89, 1317-1327 (2005).
- Digman, M. A. Fluctuation correlation spectroscopy with a laser-scanning microscope: exploiting the hidden time structure. Biophys. J. 88, L33-L36 (2005).
- Nohe, A., Keating, E., Fivaz, M., van der Goot, F. G., Petersen, N. O. Dynamics of GPI-anchored proteins on the surface of living cells. Nanomedicine. 2, 1-7 (2006).
- Semrau, S., Schmidt, T. Particle image correlation spectroscopy (PICS): retrieving nanometer-scale correlations from high-density single-molecule position data. Biophys. J. 92, 613-621 (2007).
- Bates, I. R. Membrane lateral diffusion and capture of CFTR within transient confinement zones. Biophys. J. 91, 1046-1058 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved