È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Determinazione del Raft Lipid partizionamento delle sonde fluorescenti-tag nelle cellule viventi da spettroscopia di fluorescenza di correlazione (FCS)
In questo articolo
Riepilogo
Una tecnica per sondare il partizionamento zattera lipidi di proteine fluorescenti a livello della membrana plasmatica delle cellule viventi è descritto. Si avvale della disparità nei tempi di diffusione delle proteine situati all'interno o all'esterno delle zattere lipidiche. Acquisizione può essere eseguita dinamicamente in condizioni di controllo o dopo l'aggiunta farmaco.
Abstract
Negli ultimi quindici anni l'idea che le membrane cellulari non sono omogenee e si basano su microdomini di esercitare le loro funzioni è stato ampiamente accettato. Raft lipidici sono microdomini di membrana arricchiti di colesterolo e sfingolipidi. Essi giocano un ruolo nella cellulari processi fisiologici quali la segnalazione, e 1,2 il traffico, ma sono anche pensato di essere protagonisti in diverse malattie tra cui infezioni virali o batteriche e malattie neurodegenerative 3.
Eppure la loro esistenza è ancora oggetto di controversia 4,5. Infatti, zattera dimensioni dei lipidi è stata stimata essere di circa 20 nm 6, ben al di sotto del limite di risoluzione della microscopia convenzionale (circa 200 nm), precludendo così la loro immagine diretta. Fino ad oggi, le principali tecniche utilizzate per valutare la partizione di proteine di interesse all'interno rafts lipidici sono membrane resistente ai detergenti (DRM) isolamento e co-patching con anticorpi. Anche se ampiamente utilizzati Becautilizzo della loro attuazione piuttosto facile, queste tecniche erano inclini a manufatti e quindi criticato 7,8. I miglioramenti tecnici sono pertanto necessarie per superare questi manufatti e di poter sondare lipidi partizione zattere in cellule viventi.
Presentiamo qui un metodo per l'analisi sensibile di partizione lipidi zattere di proteine fluorescenti-etichetta o lipidi nella membrana plasmatica di cellule viventi. Questo metodo, denominato spettroscopia di fluorescenza di correlazione (FCS), si basa sulla disparità nei tempi di diffusione di sonde fluorescenti situati all'interno o all'esterno delle zattere lipidiche. Infatti, come evidenziato in entrambe le membrane artificiali e colture cellulari, le sonde avrebbero diffondere molto più velocemente fuori che dentro lipid rafts dense 9,10. Per determinare tempi di diffusione, minute fluttuazioni fluorescenza vengono misurati in funzione del tempo in un volume focale (circa 1 femtoliter), situato a livello della membrana plasmatica di cellule con un microscopio confocale (Fig.1). Le auto-correlazione curve possono quindi trarre da queste fluttuazioni e dotate di adeguati modelli matematici di diffusione 11.
FCS può essere utilizzato per determinare la zattera lipidica partizionamento di sonde diverse, purché siano fluorescenti tag. Fluorescente etichettatura può essere ottenuto mediante l'espressione di proteine di fusione fluorescenti o dal legame di ligandi fluorescenti. Inoltre, FCS può essere utilizzato non solo nelle membrane artificiali e linee cellulari, ma anche in colture primarie, come descritto di recente 12. Può anche essere utilizzata per seguire la dinamica di partizionamento lipidi zattera dopo aggiunta farmaco o lipide cambiamento composizione della membrana 12.
Protocollo
1. Calibrazione del Setup FCS
- Avviare il microscopio confocale, laser, computer, incubatore per la temperatura e CO 2 di controllo.
- Assicurarsi che la SPAD (Single Photon Avalanche Diode) è e che il filtro a fluorescenza all'interno della SPAD ben si adatta al vostro campione. Controllare che la SPAD è sincronizzato nel tempo. Attenzione per avviare solo il software FCS una volta le impostazioni SPAD sono pronti per l'acquisizione.
- Preparare una soluzione fresca di tossina del colera-Alexa488 diluito in PBS per raggiungere una concentrazione di 1 pg / ml (17,5 nM).
- Ottimizzazione confocale di soluzione con rilevamento interna del microscopio.
- Passa a punto modalità di scansione e scegliere un punto nella soluzione di campione. Assicurarsi che la durata di illuminazione laser è abbastanza a lungo per eseguire le acquisizioni FCS (più di 5 minuti).
- Passa alla rilevazione esterna da parte della SPAD e al software FCS. Monitorare il segnale di fluorescenza e assicurarsi che sia ben suiTed al SPAD (segnale sufficiente, ma senza saturazione, 10 000 a 50 000 conteggi / s va bene). Se necessario, modificare la posizione Z, di guadagno e / o potenza del laser per ottenere corretto segnale di fluorescenza.
- Acquisire 10 serie di 30 misure secondi. Il tempo di acquisizione dovrebbe essere ottimizzato per il campione (il miglior compromesso tra campionamento e problemi photobleaching).
- Analizzare i dati (vedi punto 4) per garantire che si dispone di un auto-correlazione curva corrispondente ad una specie fluorescenti diffusione in soluzione (equazione in figura. 2) e che il tempo di diffusione ottenuti dopo il montaggio è corretto (circa 0,2 ms) (Fig . 3).
2. La colorazione delle cellule viventi con la Lipid Marker Zattere
- Piastre su cellule HEK293 8-Labteks e rivestiti con poli-L-lisina (1mg/ml) il giorno prima di imaging per assicurarsi che la loro adesione è corretta. Utilizzare mezzo senza rosso fenolo per garantire che non perturbazione del segnale di fluorescenza.
- Lavare le cellule due volte con HBSS (soluzione salina tampone di Hank).
- Aggiungere tossina del colera-Alexa488 (1 pg / ml) / BSA (0,1%) in 500 pl HBSS alle cellule per 30 minuti a 37 ° C. Tossina del colera si legherà alla ganglioside GM1, note per essere preferenzialmente partizionato in lipid rafts.
- Lavare le cellule due volte con HBSS (soluzione salina tampone di Hank).
3. FCS di acquisizione dati sulle cellule viventi
- Posizionare le cellule colorate sul palco microscopio. Assicurarsi che la temperatura e CO 2 le condizioni sono ottimali altrimenti questo può portare a manufatti in tempi di diffusione.
- Ottimizzazione confocale di una cella di interesse utilizzando rilevamento interna del microscopio (Fig. 4).
- Passare al punto modalità di scansione e scegliere un punto nella membrana plasmatica della cellula di interesse. Eseguire 1-2 immagini in diretta della cella per assicurarsi che la cella non è in movimento durante l'acquisizione.
- Passa alla rilevazione esterna da parte della SPAD e il morbido FCSware. Monitorare il segnale di fluorescenza e assicurarsi che sia adatto alla SPAD (segnale sufficiente, ma senza saturazione, 10 000 a 50 000 conteggi / s va bene). Se necessario, modificare la posizione Z, di guadagno e / o potenza del laser per ottenere corretto segnale di fluorescenza. Se questo non è sufficiente, si può pensare di cambiare le condizioni di colorazione.
- Acquisire 10 campioni di 30 misurazioni secondi. Poiché la maggior parte delle cellule sono auto-fluorescente, si può vedere una diminuzione della fluorescenza durante le prime acquisizioni, a causa di auto-fluorescenza fading.
4. FCS Analisi dei dati
- Impostare l'intervallo di correlazione e auto-generare le curve di correlazione con il software FCS. L'intervallo di correlazione variano tipicamente tra più breve tempo di ritardo fino al risolvibile il maggior tempo possibile (come le statistiche della misurazione diventa sempre più inadeguata quando tempo correlazione massima avvicina tempo totale di acquisizione, il tempo di ritardo massimo è limitato al 80% dei tempo di acquisizione sul Picoquant software).
- Per ciascun campione, esportare file corrispondenti alle fluttuazioni di fluorescenza e di auto-correlazione in funzione del tempo.
- Utilizzare un software di analisi dei dati e montaggio, come Origin Pro per importare i file.
- Per ogni campione, assicurarsi che non vi è alcuna photobleaching durante l'acquisizione ovvero la fluorescenza rimane stabile durante i 30 secondi. Eliminare la visualizzazione di qualsiasi misura photobleaching in quanto può provocare artificiali tempi di diffusione più lunghi.
- Verificare che la forma dei restanti curve FCS è corretta (vedi Fig. 5). Determinare la curva media FCS.
- Montare la curva con il modello matematico appropriato (Fig. 2). Questa misura darà il tempo di diffusione (s) e la proporzione di molecole che diffondono con questo (ESE) tempo di diffusione (s).
5. Risultati rappresentativi
Un esempio di calibrazione FCS con una tossina colera-Alexa488 soluzione è mostrata in figura 3 . Dopo aver verificato che le singole misure di fluorescenza in funzione del tempo non ha mostrato alcun fotodecolorazione (Figura 3A), individuale e media curve FCS sono stati calcolati. Significano curve FCS sono stati dotati di corrispondenti equazioni di diversi modelli di diffusione (esempi della Figura 2). Il parametro classicamente considerata per determinare la qualità di un accoppiamento è il coefficiente di determinazione R 2. La stretta R 2 è a 1, migliore è la vestibilità. In questo caso, il modello più accurato per adattarsi alla curva media FCS è quella che descrive una popolazione di molecole fluorescenti diffondono liberamente in tre dimensioni (equazione 1 in Figura 2 e Figura 3B). Il tempo di diffusione deriva dal fit è 0,32 ms. Residui di curva-montaggio (Figura 3C) e R 2 fattore (0,99,906 mila) fornire una stima della qualità della calzata.
Un esempio di analisi per la tossina colerica FCS-Alexa488 tinto HEK293 celle è mostrata in Figura 5. La forma della curva media multifasico FCS rivela l'esistenza delle popolazioni di molecole fluorescenti con tempi di diffusione diversi. La soluzione migliore per questa curva corrisponde ad un modello a tre popolazioni di sonde fluorescenti: due con una diffusione ostacolato (diffusione in due dimensioni, come nel piano di membrana) e una liberamente diffusore in tre dimensioni (equazione 2 nella figura 2 e figura 5) . Questa popolazione seconda corrisponde al molecole fluorescenti che si spostano al di fuori del piano di membrana, cioè, sia vincolante o non vincolante ai loro bersagli membrana, raggiungendo la membrana attraverso il percorso secrezione o riciclaggio, o lasciando la membrana per endocitosi. I due tempi di diffusione a livello della membrana, corrispondenti alla tossina del colera legato GM1, erano 2 ms (25% di molecole), corrispondente alla diffusione al di fuori di zattere lipidiche, e 75 ms (50% di molecole), corrispondente alla diffusione in zattere lipidiche . Si prega di notare che qualsiasi fotosbiancanteING durante l'acquisizione porterà ad artificiali più lunghi tempi di diffusione, quindi, eventualmente creando una tendenza verso la localizzazione di GM1 in domini raft lipidici.

Figura 1. Rappresentazione schematica del FCS set-up (foto modificata da Marquer et al. 12)

Figura 2. Esempio di modelli di diffusione ed equazioni corrispondenti usati per soddisfare le curve di autocorrelazione. Il parametro struttura S può essere scritta come S = 0 z / w 0 con z 0 il raggio effettivo focale lungo l'asse ottico a 1/2 e l'intensità e W0 efficace lateral raggio focale a 1/2 e intensità. Questi valori possono essere estratte da un classico spread punto funzione (PSF) misurazione.
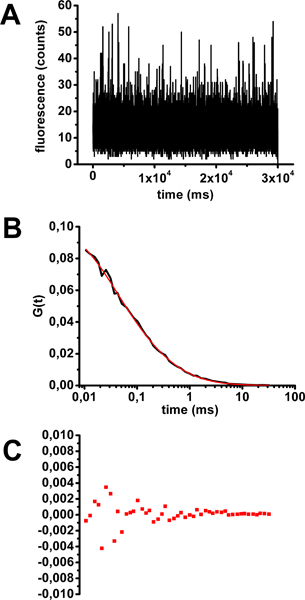
Figura 3. Valutazione del tempo di diffusione della tossina del colera-Alexa488 in soluzione per la calibrazione FCS. A) fluttuazione di fluorescenza in funzione del tempo per un esempio rappresentativo di una acquisizione 30 secondi. B) indica la curva ottenuta autocorrelazione da 10 campioni di acquisizione 30 secondi munito equazione 1 (vedi figura 2). C) Residui di adattamento della curva.
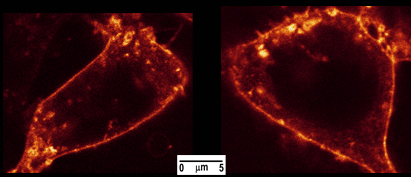
Figura 4. HEK-293 cellule colorate con tossina colerica-Alexa488. Le cellule sono state proietta sulla SP5 un microscopio confocale (Leica Microsystems, Wetzlar, Germania) con la linea interna laser 488nm. La fluorescenza è stata raccolta con un obiettivo apocromatico x60 piano di immersione in olio tra i 500e 650 nm.
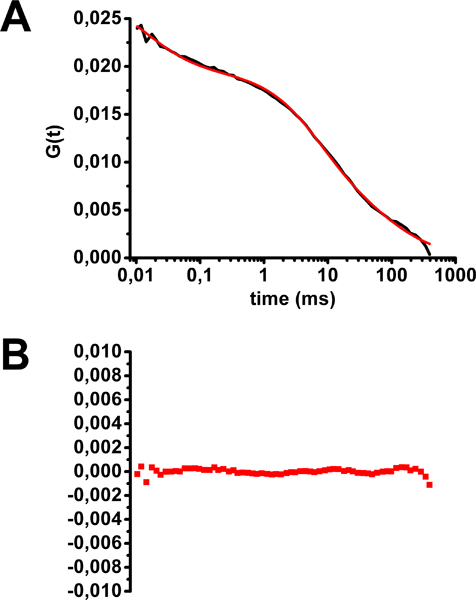
Figura 5. Valutazione del tempo di diffusione della tossina del colera-Alexa488 alla membrana plasmatica di cellule HEK293. A) media curva autocorrelazione munito equazione 2 (vedi figura 2). B) I residui di curve fitting. (Modificato da Marquer et al. 12)
Discussione
Il metodo qui presentato FCS permette una analisi sensibile e rapida la compartimentazione lipidi serie di sonde fluorescenti di interesse in cellule viventi. FCS combina l'accuratezza della localizzazione della microscopia confocale con la sensibilità di conteggio di singolo fotone. La differenza principale tra FCS e tecniche standard biochimiche che è FCS permette la determinazione assoluta della partizione lipidi zattere del bersaglio e non la partizione relativo come è il caso per l'isolamento DRM o co-pa...
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto da una borsa da Agence Nationale de la Recherche (ChoAD). Siamo anche grati alla Fondazione ICM (Institut du cerveau et de la Moelle) per il loro sostegno finanziario.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| Subunità B della tossina colerica Alexa-488 | Invitrogen | C-34775 | MW (pentamero) = 57 kg / mol |
| Microscopio confocale | Leica | SP5 | |
| Incubatore per temperatura e CO 2 di controllo | Vita di imaging servizi | Il cubo e il Box | |
| SPAD (Single Photon Avalanche Diode) | MPD (Micro Photon Devices) | PDM serie (100 micron zona sensibile) | |
| Passa alto 488 nm filtro | Semrock | 488 nm blocco bordo Brightline lungo filtro passa Part #ails.aspx? id = "target =" _blank FF01-488/LP-25 "> FF01-488/LP-25 | |
| FCS unità di rivelazione | Picoquant | Picoharp 300 Modulo | |
| Acquisizione e di auto-correlazione software | Picoquant | SymPhoTime | |
| Software di fitting | OriginLab | OriginPro8 |
Riferimenti
- Brown, D. A., London, E. Functions of lipid rafts in biological membranes. Annu. Rev. Cell Dev. Biol. 14, 111-136 (1998).
- Simons, K., Gerl, M. J. Revitalizing membrane rafts: new tools and insights. Nat. Rev. Mol. Cell Biol. 11, 688-699 (2010).
- Simons, K., Ehehalt, R. Cholesterol, lipid rafts, and disease. J. Clin. Invest. 110, 597-603 (2002).
- Munro, S. Lipid rafts: elusive or illusive. Cell. , 115-377 (2003).
- Shaw, A. S. Lipid rafts: now you see them, now you don't. Nat. Immunol. 7, 1139-1142 (2006).
- Pralle, A., Keller, P., Florin, E. L., Simons, K., Horber, J. K. Sphingolipid-cholesterol rafts diffuse as small entities in the plasma membrane of mammalian cells. J. Cell Biol. 148, 997-1008 (2000).
- Brown, D. A., London, E. Structure and function of sphingolipid- and cholesterol-rich membrane rafts. J. Biol Chem. 275, 17221-17224 (2000).
- Sharma, P., Sabharanjak, S., Mayor, S. Endocytosis of lipid rafts: an identity crisis. Semin. Cell Dev. Biol. 13, 205-214 (2002).
- Kahya, N., Scherfeld, D., Bacia, K., Poolman, B., Schwille, P. Probing lipid mobility of raft-exhibiting model membranes by fluorescence correlation spectroscopy. J. Biol. Chem. 278, 28109-28115 (2003).
- Bacia, K., Scherfeld, D., Kahya, N., Schwille, P. Fluorescence correlation spectroscopy relates rafts in model and native membranes. Biophys J. 87, 1034-1043 (2004).
- Kim, S. A., Heinze, K. G., Schwille, P. Fluorescence correlation spectroscopy in living cells. Nat. Methods. 4, 963-973 (2007).
- Marquer, C. Local cholesterol increase triggers amyloid precursor protein-Bace1 clustering in lipid rafts and rapid endocytosis. FASEB J. 25, 1295-1305 (2011).
- Tian, Y., Martinez, M. M., Pappas, D. Fluorescence correlation spectroscopy: a review of biochemical and microfluidic applications. Appl. Spectrosc. 65, 115A-124A (2011).
- Haustein, E., Schwille, P. Fluorescence correlation spectroscopy: novel variations of an established technique. Annu. Rev. Biophys. Biomol. Struct. 36, 151-169 (2007).
- Ilien, B. Pirenzepine promotes the dimerization of muscarinic M1 receptors through a three-step binding process. J. Biol. Chem. 284, 19533-19543 (2009).
- Lieto, A. M., Cush, R. C., Thompson, N. L. Ligand-receptor kinetics measured by total internal reflection with fluorescence correlation spectroscopy. Biophys. J. 85, 3294-3302 (2003).
- Thompson, N. L., Burghardt, T. P., Axelrod, D. Measuring surface dynamics of biomolecules by total internal reflection fluorescence with photobleaching recovery or correlation spectroscopy. Biophys. J. 33, 435-454 (1981).
- Thompson, N. L., Steele, B. L. Total internal reflection with fluorescence correlation spectroscopy. Nat. Protoc. 2, 878-890 (2007).
- Eggeling, C. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457, 1159-1162 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49, 141-164 (2007).
- Shvartsman, D. E., Kotler, M., Tall, R. D., Roth, M. G., Henis, Y. I. Differently anchored influenza hemagglutinin mutants display distinct interaction dynamics with mutual rafts. J. Cell Biol. 163, 879-888 (2003).
- White, R. Holin triggering in real time. Proc. Natl. Acad. Sci. U.S.A. 108, 798-803 (2011).
- Petersen, N. O., Hoddelius, P. L., Wiseman, P. W., Seger, O., Magnusson, K. E. Quantitation of membrane receptor distributions by image correlation spectroscopy: concept and application. Biophys. J. 65, 1135-1146 (1993).
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88, 3601-3614 (2005).
- Digman, M. A. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89, 1317-1327 (2005).
- Digman, M. A. Fluctuation correlation spectroscopy with a laser-scanning microscope: exploiting the hidden time structure. Biophys. J. 88, L33-L36 (2005).
- Nohe, A., Keating, E., Fivaz, M., van der Goot, F. G., Petersen, N. O. Dynamics of GPI-anchored proteins on the surface of living cells. Nanomedicine. 2, 1-7 (2006).
- Semrau, S., Schmidt, T. Particle image correlation spectroscopy (PICS): retrieving nanometer-scale correlations from high-density single-molecule position data. Biophys. J. 92, 613-621 (2007).
- Bates, I. R. Membrane lateral diffusion and capture of CFTR within transient confinement zones. Biophys. J. 91, 1046-1058 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon