A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
קביעת רפסודה ליפידים חלוקת בדיקות fluorescently-מתוייגים בתאים חיים באמצעות מתאם ספקטרוסקופיה פלואורסצנטי (FCS)
In This Article
Summary
טכניקה לחקור את הרפסודה השומנים חלוקה של חלבונים ניאון על הממברנה של התאים החיים המתואר. הוא מנצל את הפער בזמנים דיפוזיה של חלבונים המצויים בתוך או מחוץ רפסודות שומנים בדם. הרכישה יכולה להתבצע באופן דינמי בתנאי בקרה, או אחרי כן התרופה.
Abstract
בחמש עשרה השנים האחרונות את הרעיון קרום התא לא הומוגנית להסתמך על microdomains להפעיל פונקציות שלהם הפכה מקובלת מאוד. רפסודות שומנים בדם הם microdomains קרום המועשר כולסטרול sphingolipids. הם ממלאים תפקיד בתהליכים פיזיולוגיים הסלולר כגון איתות, ו 1,2 סחר אבל נחשבים גם להיות שחקני מפתח מחלות שונות, כולל זיהומים נגיפיים או חיידקים ומחלות ניווניות 3.
עם זאת, קיומם הוא עדיין עניין של מחלוקת 4,5. אכן, רפסודה בגודל שומנים בדם כבר מוערך כ 20 ננומטר 6, הרבה מתחת לגבול הרזולוציה של מיקרוסקופ רגיל (סביב 200 ננומטר), ובכך precluding הדמיה ישירה שלהם. עד עכשיו, את הטכניקות העיקריות להערכת החלוקה של חלבונים בעלי עניין בתוך רפסודות שומנים בדם היו ממברנות עמידות דטרגנט (DRMs) בידוד ושיתוף תיקון עם נוגדנים. למרות שימוש נרחב becaהשימוש ביישום די קל שלהם, טכניקות אלה היו מועדים חפצים ובכך מתח ביקורת על 7,8. שיפורים טכניים היו לכן, יש להתגבר על חפצים כדי להיות מסוגל לחקור רפסודות מחיצה שומנים בתאים חיים.
כאן אנו מציגים שיטה ניתוח רגישות של מחיצת רפסודות שומנים בדם של חלבונים או שומנים fluorescently-מתוייגים בקרום פלזמה של תאים חיים. שיטה זו, המכונה ספקטרוסקופיה מתאם פלואורסצנטי (FCS), מסתמך על הפער בזמנים דיפוזיה של בדיקות ניאון הממוקמים או מחוצה לו, רפסודות שומנים בדם. למעשה, כפי שעולה הן ממברנות מלאכותיים בתרביות תאים, בדיקות היה לפזר הרבה יותר מהר מאשר בחוץ בתוך רפסודות שומנים בדם צפופים 9,10. כדי לקבוע את הזמנים דיפוזיה, תנודות זעירות הקרינה נמדדים כפונקציה של הזמן בנפח מוקד (כ 1 femtoliter), הממוקם בקרום הפלסמה של התאים באמצעות מיקרוסקופ confocal (איור1). אוטומטי המתאם עיקולים לאחר מכן, ניתן להסיק שינויים אלו מצויד המתאימים מודלים מתמטיים דיפוזיה 11.
FCS ניתן להשתמש כדי לקבוע את הרפסודה השומנים מחיצות של בדיקות שונות, כל עוד הם מתויגים fluorescently. תיוג פלורסנט ניתן להשיג על ידי ביטוי של חלבונים היתוך ניאון או באמצעות קשירה של ligands ניאון. יתר על כן, FCS יכול לשמש לא רק ממברנות מלאכותיים שורות תאים, אלא גם בתרבויות העיקריות, כפי שתואר לאחרונה 12. זה יכול לשמש גם כדי לעקוב אחר הדינמיקה של חלוקת השומנים הרפסודה לאחר הוספת התרופה או שינוי הרכב השומנים קרום 12.
Protocol
1. כיול של תוכנית ההתקנה של FCS
- התחל מיקרוסקופ confocal, לייזרים, מחשבים, חממה טמפרטורה בקרת CO 2.
- ודא SPAD (דיודה פוטון יחיד מפולת שלגים) פועל מסנן הקרינה בתוך SPAD הוא גם מתאים המדגם שלך. בדוק SPAD מסונכרן בזמן. היזהרו רק להתחיל תוכנת FCS שלך פעם אחת ההגדרות SPAD שלך מוכנים לרכישה.
- הכן פתרון חדש של כולרה הרעלן Alexa488 מדולל PBS להגיע ריכוז של 1 מיקרוגרם / מ"ל (17.5 ננומטר).
- מטב הדמיה confocal של פתרון באמצעות זיהוי הפנים של המיקרוסקופ.
- לעבור למצב סריקה להצביע ולבחור נקודה במדגם פתרון. לוודא את משך התאורה לייזר מספיק זמן כדי לבצע רכישות FCS שלך (> 5 דקות).
- לעבור זיהוי חיצוני על ידי SPAD ו לתוכנה FCS. מעקב אחר אותות הקרינה שלך לוודא שהוא גם סויטד ל SPAD (האות מספיק אך לא רוויה, 10 000 50 000 ספירת / s זה בסדר). במידת הצורך, לשנות עמדה רווח Z, ו / או כוח לייזר על מנת להשיג אות הקרינה הנכון.
- רוכשת 10 סטים של 30 מדידות שניות. בזמן הרכישה צריך להיות מותאם במיוחד מדגם שלך (פשרה בין הדגימה הטובה ביותר ובעיות photobleaching).
- לנתח את הנתונים (ראה שלב 4) כדי לוודא שיש לך עקומת אוטומטי המתאם המתאים מין אחד ניאון מתפשטים והולכים בתמיסה (משוואה בתרשים. 2), וכי הפעם דיפוזיה המתקבל לאחר מתאים נכון (כ 0.2 ms) (איור . 3).
2. צביעת תאים החיים עם דה מרקר ליפידים רפסודות
- HEK293 צלחת התאים על 8-גם Labteks מצופה פולי-L-ליזין (1mg/ml) יום לפני הדמיה לוודא כי הידבקות שלהם נכונה. השתמש בינוני ללא פנול אדום כדי להבטיח שאין הפרעה של אותות הקרינה.
- לשטוף את התאים פעמיים עם HBSS (שנאגרו של האנק תמיסת מלח).
- הוסף כולרה הרעלן Alexa488 (1 מיקרוגרם / מ"ל) / BSA (0,1%) ב -500 HBSS μl על התאים למשך 30 דקות ב 37 ° C. רעלן הכולרה יהיה לאגד ganglioside GM1, ידוע כי למחיצות מועדף על רפסודות שומנים בדם.
- לשטוף את התאים פעמיים עם HBSS (שנאגרו של האנק תמיסת מלח).
3. FCS רכישת נתונים על תאים חיים
- מניחים את התאים צבעונית על הבמה מיקרוסקופ. הפוך את הטמפרטורה CO בטוח ו -2 התנאים הם אופטימליים אחרת זה עלול להוביל חפצים בזמנים דיפוזיה.
- מטב הדמיה confocal של תא עניין באמצעות זיהוי הפנים של המיקרוסקופ (איור 4).
- לעבור למצב סריקה להצביע ולבחור טעם קרום הפלזמה של התא של עניין. בצעו 1-2 תמונות חיות של התא כדי לוודא התא לא זז במהלך הרכישה.
- לעבור זיהוי חיצוני על ידי SPAD וכדי הרך FCSכלי. מעקב אחר אותות הקרינה שלך לוודא שהוא מתאים גם SPAD (האות מספיק אך לא רוויה, 10 000 50 000 ספירות / s זה בסדר). במידת הצורך, לשנות עמדה רווח Z, ו / או כוח לייזר על מנת להשיג אות הקרינה הנכון. אם זה לא מספיק, ניתן לשקול שינוי התנאים צביעה.
- רוכשת 10 דגימות של 30 מדידות שניות. כמו רוב התאים הם אוטומטי ניאון, אתה עשוי לראות ירידה הקרינה במהלך הרכישות הראשונות, עקב דהייה אוטומטי פלואורסצנטי.
4. FCS ניתוח נתונים
- הגדר את מרווח המתאם וליצור אוטומטית עקומות קורלציה עם תוכנות FCS. מרווח המתאם יהיה בדרך כלל נע בין הזמן הקצר ביותר לפתרון בעומר עד הזמן הארוך ביותר האפשרי (כמו נתוני המדידה הופך להיות יותר ויותר בלתי מספקת כאשר זמן הקורלציה המקסימלי מתקרב בזמן הרכישה הכולל, זמן השהיה המרבי מוגבל ל 80% רכישת זמן PicoquaNT תוכנה).
- עבור מדגם זה, לייצא קבצים המתאימים תנודות פלואורסצנציה אוטומטי מתאם כפונקציה של הזמן.
- השתמש ניתוח נתונים ותוכנה מתאימה, כגון מקור Pro לייבא את הקבצים.
- עבור מדגם זה, ודא שיש photobleaching לא במהלך הרכישה כלומר הקרינה נשארת יציבה במשך 30 שניות. לבטל את כל אמצעי להצגת photobleaching שכן הדבר עלול לגרום מלאכותיים פעמים דיפוזיה יותר.
- בדוק את צורת הנותרים FCS עקומות נכון (ראה איור 5). לקבוע את עקומת הממוצע FCS.
- להתאים את עקומת עם מודל מתמטי המתאים (איור 2). התאמה זה ייתן לך זמן דיפוזיה (ים) ו שיעור מולקולות מתפשטים והולכים עם הזמן (מזרח) זה דיפוזיה (ים).
5. נציג תוצאות
דוגמה כיול FCS פתרון כולרה הרעלן Alexa488 מוצגת באיור 3 חזק. לאחר בדיקה, כי צעדים בודדים של הקרינה כפונקציה של הזמן לא הראה שום photobleaching (איור 3 א), כלומר הפרט עקומות FCS חושבו. מתכוון עקומות FCS צוידו משוואות דיפוזיה המתאימים מודלים שונים (דוגמאות באיור 2). פרמטר נחשב קלאסי כדי לקבוע את איכות ההתאמה הוא מקדם בנחישות R 2. ככל R 2 היא 1, יותר טוב מתאים. במקרה זה, המודל המדויק ביותר כדי להתאים את עקומת הממוצע FCS הוא זה המתאר האוכלוסייה של מולקולות ניאון מתפשטים והולכים באופן חופשי בשלושה ממדים (משוואה 1 באיור 2, איור 3 ב). זמן דיפוזיה נגזר בכושר הוא 0.32 אלפיות שנייה. שאריות מן הראוי, עקומת (איור 3 ג) ו - R 2 גורם (0.99906) לתת הערכה של איכות ההתאמה.
דוגמה של ניתוח FCS עבור מוכתם הרעלן Alexa488 כולרה HEK293 תאים מוצגת באיור 5. ממוצע multiphasic FCS צורת עקומת חושף את קיומם של אוכלוסיות של מולקולות ניאון עם פעמים דיפוזיה שונים. ההתאמה הטובה ביותר עבור עקומת זו תואמת את המודל עם שלוש אוכלוסיות של בדיקות ניאון: שני עם דיפוזיה הפריע (דיפוזיה בשני ממדים כמו במישור הממברנה) ואחד לשדר באופן חופשי בשלושה ממדים (משוואה 2 באיור 2, איור 5) . אוכלוסייה זו האחרונה תואמת את מולקולות ניאון נעים מחוץ למטוס קרום, כלומר, בין אם מחייב או unbinding ליעדים הממברנה שלהם, להגיע דרך הממברנה מסלול הפרשת או מיחזור, או לעזוב את הממברנה על ידי אנדוציטוזה. שתי פעמים דיפוזיה על הממברנה, המתאימים רעלן הכולרה חייב GM1, היו 2 ms (25% מכלל מולקולות), המקביל ל דיפוזיה מחוץ רפסודות שומנים בדם, טרשת נפוצה ו -75 (50% מכלל מולקולות), המקביל ל דיפוזיה של רפסודות שומנים בדם . שים לב, שכל photobleaching במהלך הרכישה תביא מלאכותיים פעמים דיפוזיה יותר ובכך אולי ליצור הטיה כלפי לוקליזציה של GM1 בתחומים רפסודות שומנים בדם.

באיור 1. ייצוג סכמטי של FCS הגדרת (תמונה שונה מן Marquer et al. 12)

איור 2. דוגמה מודלים ומשוואות דיפוזיה המתאימים המשמשים להתאים עקומות autocorrelation. פרמטר מבנה S ניתן לכתוב כמו S = Z רדיוס 0 / ל 0 עם Z 0 מוקד אפקטיבי לאורך הציר האופטי ב 1 עוצמת / 2 E ו w0 la יעילרדיוס מיקוד teral בעוצמה 2 1 / e. ערכים אלה ניתן להפיק מדידה קלאסית התפשטות נקודה (כוחות הביטחון הפלסטינים) function.
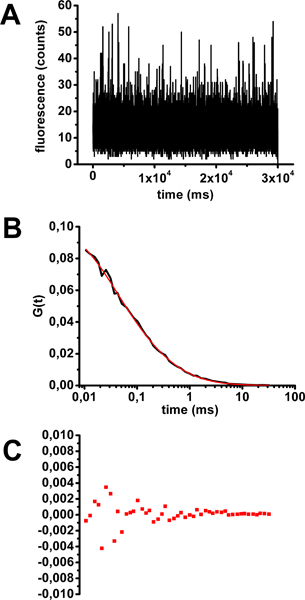
איור 3. הערכה של זמן הדיפוזיה של כולרה הרעלן Alexa488 בתמיסה לכיול FCS. א) תנודות Fluorescence כפונקציה של זמן למשל נציג של רכישת 30 שניות. ב) ממוצע עקומת autocorrelation להשיג 10 דוגמאות של רכישת 30 שניות מצויד משוואה 1 (ראה איור 2). C) שאריות של עקומת הולם.
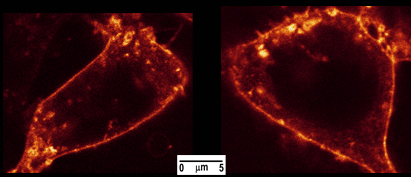
4. איור HEK-293 תא מוכתם כולרה הרעלן Alexa488. התאים היו צילמו על מיקרוסקופ SP5 confocal (Leica Microsystems, Wetzlar, גרמניה) עם קו לייזר פנימית 488nm. הקרינה נאסף עם X60 apochromat מטרת התוכנית שמן טבילה בין 500ו -650 ננומטר.
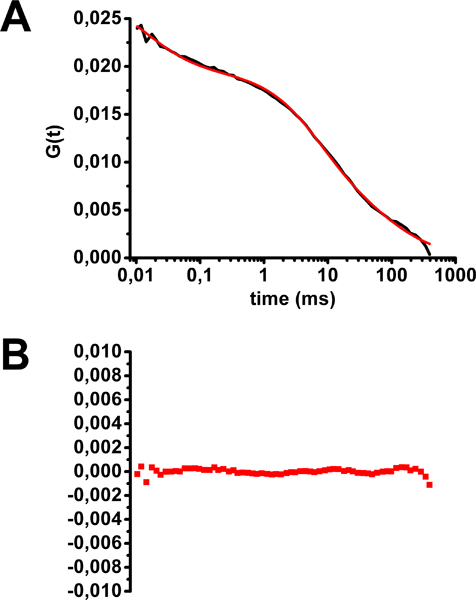
איור 5. הערכת הזמן דיפוזיה של כולרה הרעלן Alexa488 על קרום הפלזמה של HEK293 תאים. א) ממוצע עקומת autocorrelation מצויד משוואה 2 (ראה תרשים 2). ב) שאריות של עקומת הולם. (שונה מ Marquer et al. 12)
Discussion
שיטת FCS המוצג כאן מאפשר ניתוח רגיש מהירה של חלוקת השומנים הרפסודה של בדיקות ניאון של עניין תאים חיים. FCS המשלבת את הדיוק של לוקליזציה של מיקרוסקופיה confocal עם רגישות של ספירת פוטון יחיד. ההבדל העיקרי בין FCS וטכניקות ביוכימיים התקן היא זאת FCS מאפשר קביעה מוחלטת של חלוקת הש...
Disclosures
אין ניגוד עניינים הצהיר.
Acknowledgements
עבודה זו נתמכה על ידי מענק מטעם סוכנות הידיעות הלאומית דה לה משוכלל ונדיר (ChoAD). אנחנו גם מודים ICM Fondation (Institut du Cerveau et de la Moelle) עבור תמיכה כלכלית שלהם.
Materials
| Name | Company | Catalog Number | Comments |
| שם מגיב | חברה | מספר קטלוגי | תגובות |
| כולרה למקטע רעלן B-Alexa 488 | Invitrogen | C-34775 | MW (pentamer) = 57 ק"ג / mol |
| Confocal מיקרוסקופ | Leica | SP5 | |
| החממה עבור 2 טמפרטורה CO | חיים שירותי הדמיה | הקוביה Box | |
| SPAD (דיודה פוטון יחיד מפולת שלגים) | MPD (Micro Devices פוטון) | PDM Serie (100 מיקרומטר אזור רגיש) | |
| 488 nm גבוהה לעבור סינון | Semrock | 488 ננומטר חסימת קצה BrightLine ארוך לעבור סינון חלק #ails.aspx? id = FF01-488/LP-25 "target =" _blank "> FF01-488/LP-25 | |
| FCS יחידת איתור | Picoquant | Picoharp 300 מודול | |
| רכישה אוטומטית מתאם תוכנה | Picoquant | SymPhoTime | |
| מתאים תוכנה | OriginLab | OriginPro8 |
References
- Brown, D. A., London, E. Functions of lipid rafts in biological membranes. Annu. Rev. Cell Dev. Biol. 14, 111-136 (1998).
- Simons, K., Gerl, M. J. Revitalizing membrane rafts: new tools and insights. Nat. Rev. Mol. Cell Biol. 11, 688-699 (2010).
- Simons, K., Ehehalt, R. Cholesterol, lipid rafts, and disease. J. Clin. Invest. 110, 597-603 (2002).
- Munro, S. Lipid rafts: elusive or illusive. Cell. , 115-377 (2003).
- Shaw, A. S. Lipid rafts: now you see them, now you don't. Nat. Immunol. 7, 1139-1142 (2006).
- Pralle, A., Keller, P., Florin, E. L., Simons, K., Horber, J. K. Sphingolipid-cholesterol rafts diffuse as small entities in the plasma membrane of mammalian cells. J. Cell Biol. 148, 997-1008 (2000).
- Brown, D. A., London, E. Structure and function of sphingolipid- and cholesterol-rich membrane rafts. J. Biol Chem. 275, 17221-17224 (2000).
- Sharma, P., Sabharanjak, S., Mayor, S. Endocytosis of lipid rafts: an identity crisis. Semin. Cell Dev. Biol. 13, 205-214 (2002).
- Kahya, N., Scherfeld, D., Bacia, K., Poolman, B., Schwille, P. Probing lipid mobility of raft-exhibiting model membranes by fluorescence correlation spectroscopy. J. Biol. Chem. 278, 28109-28115 (2003).
- Bacia, K., Scherfeld, D., Kahya, N., Schwille, P. Fluorescence correlation spectroscopy relates rafts in model and native membranes. Biophys J. 87, 1034-1043 (2004).
- Kim, S. A., Heinze, K. G., Schwille, P. Fluorescence correlation spectroscopy in living cells. Nat. Methods. 4, 963-973 (2007).
- Marquer, C. Local cholesterol increase triggers amyloid precursor protein-Bace1 clustering in lipid rafts and rapid endocytosis. FASEB J. 25, 1295-1305 (2011).
- Tian, Y., Martinez, M. M., Pappas, D. Fluorescence correlation spectroscopy: a review of biochemical and microfluidic applications. Appl. Spectrosc. 65, 115A-124A (2011).
- Haustein, E., Schwille, P. Fluorescence correlation spectroscopy: novel variations of an established technique. Annu. Rev. Biophys. Biomol. Struct. 36, 151-169 (2007).
- Ilien, B. Pirenzepine promotes the dimerization of muscarinic M1 receptors through a three-step binding process. J. Biol. Chem. 284, 19533-19543 (2009).
- Lieto, A. M., Cush, R. C., Thompson, N. L. Ligand-receptor kinetics measured by total internal reflection with fluorescence correlation spectroscopy. Biophys. J. 85, 3294-3302 (2003).
- Thompson, N. L., Burghardt, T. P., Axelrod, D. Measuring surface dynamics of biomolecules by total internal reflection fluorescence with photobleaching recovery or correlation spectroscopy. Biophys. J. 33, 435-454 (1981).
- Thompson, N. L., Steele, B. L. Total internal reflection with fluorescence correlation spectroscopy. Nat. Protoc. 2, 878-890 (2007).
- Eggeling, C. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457, 1159-1162 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49, 141-164 (2007).
- Shvartsman, D. E., Kotler, M., Tall, R. D., Roth, M. G., Henis, Y. I. Differently anchored influenza hemagglutinin mutants display distinct interaction dynamics with mutual rafts. J. Cell Biol. 163, 879-888 (2003).
- White, R. Holin triggering in real time. Proc. Natl. Acad. Sci. U.S.A. 108, 798-803 (2011).
- Petersen, N. O., Hoddelius, P. L., Wiseman, P. W., Seger, O., Magnusson, K. E. Quantitation of membrane receptor distributions by image correlation spectroscopy: concept and application. Biophys. J. 65, 1135-1146 (1993).
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88, 3601-3614 (2005).
- Digman, M. A. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89, 1317-1327 (2005).
- Digman, M. A. Fluctuation correlation spectroscopy with a laser-scanning microscope: exploiting the hidden time structure. Biophys. J. 88, L33-L36 (2005).
- Nohe, A., Keating, E., Fivaz, M., van der Goot, F. G., Petersen, N. O. Dynamics of GPI-anchored proteins on the surface of living cells. Nanomedicine. 2, 1-7 (2006).
- Semrau, S., Schmidt, T. Particle image correlation spectroscopy (PICS): retrieving nanometer-scale correlations from high-density single-molecule position data. Biophys. J. 92, 613-621 (2007).
- Bates, I. R. Membrane lateral diffusion and capture of CFTR within transient confinement zones. Biophys. J. 91, 1046-1058 (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved