Method Article
Del genoma del ratón Ingeniería Utilizando Nucleasas Designer
En este artículo
Resumen
Nucleasas de diseñadores como nucleasas de dedos de zinc (ZFNs) y transcripción nucleasas efectoras del tipo activador (Talens) se pueden utilizar para modificar el genoma de preimplantación de embriones de ratones mediante la activación de ambos al final no homóloga (NHEJ) y recombinación homóloga (HR) itinerarios. Estos avances permiten la rápida generación de ratones con modificaciones genéticas precisas.
Resumen
Los ratones transgénicos que llevan modificaciones específicas del sitio del genoma (knockout, knock-in) son de vital importancia para la disección de los sistemas biológicos complejos, así como para la modelización de enfermedades humanas y probar estrategias terapéuticas. Los recientes avances en el uso de las nucleasas de diseño tales como nucleasas de dedos de zinc (ZFNs), transcripción nucleasas efectoras del tipo activador (Talens) y los agrupados interspaced regularmente repeticiones palindrómicas cortas (CRISPR) / sistema CRISPR-asociado (CAS) 9 para el sitio- la ingeniería del genoma específico abierta la posibilidad de realizar la modificación del genoma centrada y urgente en prácticamente cualquier especie de laboratorio sin la necesidad de depender de la tecnología de células madre embrionarias (ES). Un experimento de edición genoma normalmente comienza con la identificación de diseño de nucleasa sitios diana dentro de un gen de interés seguido por la construcción de los dominios de unión a ADN personalizadas para dirigir la actividad de nucleasa para el locus genómico investigador-definido. Plásmidos de nucleasa Designer son in vitro </ Em> transcrita para generar ARNm para la microinyección de los ovocitos de ratón fertilizados. Aquí, ofrecemos un protocolo para el logro de modificación del genoma dirigida por inyección directa de TALEN mRNA en ovocitos de ratón fertilizados.
Introducción
Los ratones son, con mucho, la plataforma más popular para la generación de modelos animales transgénicos. La caja de herramientas versátiles para la ingeniería genética del embrión de ratón 1-3 se ha extendido recientemente por los enfoques de edición del genoma basado en las nucleasas de diseñadores como nucleasas de dedos de zinc (ZFN) 4-6, nucleasas activador de transcripción efectoras (Talen) 7,8, y las repeticiones agrupadas interspaced regularmente cortas palindrómicas (CRISPR) / sistema CRISPR-asociado (CAS) 9 9. ZFN y función TALEN como pares de dos dominios de diseño personalizado a base de proteínas de unión al ADN (arrays de proteínas dedo de zinc y repetir variables di-residuos (RVDS), respectivamente) que están acoplados cada uno a la endonucleasa FokI 10-12. Por el contrario, la especificidad de la escisión de ADN mediada por Cas9 es proporcionado por transactivación de ARN de CRISPR (crRNA y tracrRNA, que también se pueden combinar en una sola molécula de ARN quimérico de ARN denominado guía) 11 que actúan en un complejo con laProteína CRISPR.
Talens una secuencia definida de RVDS se puede construir rápidamente por experimentadores individuales con una multitud de estrategias de montaje para elegir 13-17. CRISPR/Cas9 promete aún menor generación de mano de obra de las nucleasas de diseño, sin embargo, la especificidad de la guía de ARN-ADN vinculante aún no se resuelve por completo 18,19. Generación de ZFNs personalizados hasta ahora se ha limitado a laboratorios académicos especializados y proveedores comerciales tales como Sangamo Biosciences y el servicio Sigma CompoZr.
En general, el genoma edición con nucleasas de diseño tiene como objetivo la introducción de la línea se rompe dobles (OSD) en loci genómica definida, el cual posteriormente atraer fin homóloga (NHEJ) o recombinación homóloga (HR) los mecanismos de reparación del ADN 10,12. La reparación mediada por NHEJ de un OSD a menudo resulta en la introducción de inserciones y deleciones en estrecha proximidad al sitio de la reparación. Por lo tanto la reparación NHEJ cun ser explotados para la anulación de la función de un gen diana mediante la introducción de una mutación de cambio de marco en el genes de codificación de proteínas secuencia 4,7,9. Alternativamente, la adición o sustitución de la información genética definida se puede lograr proporcionando un donante de ADN junto con las nucleasas de diseño. Un donante de ADN comprende secuencias de ADN investigador-diseñado flanqueadas por regiones de homología con el locus diana, por lo que sirve como una plantilla para la reparación de DSB por HR. Ambos plásmidos 5,6,20 y oligonucleótidos monocatenarios 8,9,21 se han utilizado con éxito como donantes. Ni NHEJ-ni de edición genoma HR-mediada requieren la introducción de un marcador seleccionable en el genoma del embrión del ratón, lo que hace que estas estrategias particularmente bien adaptado para la creación de pequeñas alteraciones en la secuencia de nucleótidos sin afectar a la arquitectura genética general.
En este protocolo se describen todos los procedimientos esenciales para su edición en el genomaembrión de ratón utilizando Talens. Estos incluyen: 1) la identificación de un sitio de destino TALEN 22, 2) la construcción de Talens por clonación golden gate 13, 3) la síntesis in vitro de mRNA TALEN, 4) la microinyección de ARNm TALEN en oocitos fertilizados de ratón, 5) los procedimientos quirúrgicos para la transferencia de embriones , y 6) análisis de mutagénesis inducida por TALEN en animales fundadores. Nos centramos en TALEN mRNA microinyección y la detección de los fundadores de inducidos NHEJ inserciones / supresiones. Con este fin hemos generado construcciones bifuncionales TALEN que permiten tanto la expresión en células de mamífero cuando se transfecta como plásmidos y en la síntesis in vitro de ARNm TALEN para la microinyección en embriones de ratón. Estos constructos comprenden una cadena principal CUENTO truncada 23 fusionado a heterodimérica FokI dominios de 24,25 para la edición genoma óptima en células de mamífero. Este protocolo también puede ser adoptado para la microinyección de otras nucleasas de diseño o para preparaciones inyectables combinados de nucleasas diseñadory construcciones de donantes (diseño de los donantes de ADN ha sido descrito en publicaciones técnicas excelentes por Wefers et al. 26,27).
Declaración de Ética
Todos los experimentos con animales se realizaron de acuerdo con las directrices y reglamentos de la Oficina Veterinaria Cantonal del Cantón de Zurich.
Protocolo
1. Identificación de los Sitios Objetivo Talen
- Visite el sitio web 2.0 TAL efectoras de nucleótidos de establecimiento de objetivo (http://tale-nt.cac.cornell.edu) y seleccione "TALEN de establecimiento de objetivo".
- Introduzca la secuencia del gen diana. Si la expresión pCAG-T7-TALEN construye está siendo utilizado (Figura 1C) elija "Miller et al., 2011" en "Utilizar una arquitectura de presets" con el fin de predecir los sitios de destino que pueden ser objetivo con la longitud óptima espaciador (15-20 pb) y los números de RVDS TALE (15-20) para esta arquitectura TALEN.
- Elija "NN" bajo "G sustituto" (RVDS NH-Guanosina específicos también están disponibles, pero todavía no ampliamente probados para el montaje TALEN).
- Opcional: otras configuraciones y opciones por favor, siga las instrucciones que aparecen en el sitio web y los enlaces que aparecen allí.
- Se genera un archivo de texto con los posibles sitios diana-, que puede serimportado en un programa de hoja de cálculo para una visualización más cómoda. Seleccione el par TALEN (s) para el montaje de la puerta de oro.
- Opcional: Secuencia de la región genómica de interés en la cepa de ratón que se utilizará para la microinyección de detectar posibles polimorfismos de nucleótido único (SNP) que podrían no ser contabilizados de bases de datos públicas y podría potencialmente prevenir nucleasa vinculante.
- Opcional: Consulte la Tabla 3 para el diseñador nucleasas relacionadas con los recursos en línea adicionales.
- La mayoría de los plásmidos requeridos para ZFN y TALEN construcción se pueden obtener de Addgene:
http://www.addgene.org/special-collections/
Addgene IDs para el kit de la puerta de oro TALEN y construcciones de expresión de mamíferos Talen se proporcionan en la Tabla 2.
Por otra parte, determinados módulos funcionales, tales como dominios de dedo de cinc se puede pedir a un proveedor de servicios de síntesis de genes.
2. Golden Gate de la Asamblea TALEN
En esta sección se describe el montaje de Talens utilizando un protocolo publicado por Cermak et al. 13 Talens largos se construyen utilizando dos posteriores Golden Gate clonación pasos (Figura 1). Este enfoque permite la incorporación de cualquier número de RVDS entre 12 y 31 en las construcciones de expresión finales. El protocolo de ensamblaje ha sido adaptado a destino vectores diseñados para la generación de los ARNm (Figura 1C) que expresan talens altamente activos siguientes microinyección de ovocitos. Por favor, consulte también el protocolo de Internet del equipo Golden Gate TALEN para el establecimiento y mantenimiento de la biblioteca de plásmidos ( http://www.addgene.org/TALeffector/goldengateV2/ ).
- Día 1 - Golden Gate Respuesta n ° 1 - Montaje de RVD Arrays en pfus Vectores
- Opcional: Para cada par TALEN diseñado en Protocol 1, escriba la secuencia de ADN de los dos de los sitios de destino (incluyendo la inicial T, 5 'a 3') en la hoja de cálculo TALEN_Voytas_Pipetting para generar un esquema de pipeteo para cada reacción de la puerta de oro (también entrar en las concentraciones de todos los plásmidos utilizados para las asambleas ). La hoja de cálculo también proporciona la secuencia de ADN esperado de todos los RVDS que se pueden utilizar para la alineación de los resultados de secuenciación en la sección 2.5.2.
- Por cada única TALEN diseñado en el Protocolo 1:
Si la longitud es TALEN 12-21 (estándar), seleccione la variable de repetición di-residuos (RVDS) 1.10 y destino vector pFUS_A. Seleccione restantes RVDS y un vector destino pFUS_B, por ejemplo, para un TALEN con 15 RVDS (incluyendo la última repetición, que se sumará a la asamblea final en la sección 2.3.3, seleccione RVDS 11-14 y pFUS_B4 destino vector (Figura 1A).
Si la longitud TALEN es 22-31, uso RVDS 1-10 y destino vector pFUS_A30A. Elige RVDS 11-20 y destino vector pFUS_A30B. Seleccione restantes RVDS y un vector pFUS_Bdestination, por ejemplo, para TALEN con 24 RVDS recoger RVDS 21-23 y destino vector pFUS_B3. - Configure la puerta de oro de reacción # 1 por cada pfus + combinación RVD por separado, es decir, 1-10 + pFUS_A y restantes RVDS + respectivo vector pFUS_B o para TALEN más de 21 RVDS, 1-10 + pFUS_A30A, 11-20 pFUS_A30B y restantes RVDS + respectivo vector pFUS_B. Utilice 150 ng de cada vector de RVD, 150 ng de vector de pfus, 1 l BsaI, 1 l de T4 ADN ligasa, 2 l de tampón 10x de T4 ADN ligasa, y H 2 O a 20 l de volumen de reacción total. (Se recomienda el uso de alícuotas frescas de tampón de ligasa de T4 para cada ronda de conjuntos de Golden Gate desde repetida descongelación / congelación de tampón de ligasa de T4 puede reducir la eficiencia de las reacciones.)
- Coloque reacciones Golden Gate en un ciclador térmico.
Programa:
37 ° C 5 min
16 ° C 10 min
10 ciclos
50 ° C 5 min
80 ° C 5 min - Añadir 1 l de ATP 10 mM y 1 l-plásmido Caja de seguridad nucleasa a cada mezcla y se incuba a 37 ° C durante 1 hora. Este tratamiento elimina productos de ligación incompletas lineales que podrían ser clonados en los vectores de destino por recombinación homóloga in vivo en las bacterias transformadas.
- Transformar E. coli con reacciones individuales de ligación (o electrocompetentes de E. coli químicamente competentes que facilitan la α-complementación tales como XL1-Blue o DH5a se puede utilizar aquí y en transformaciones posteriores).
- Las bacterias de la placa sobre la espectinomicina (50 ug / ml) placas con X-Gal e IPTG (40 mg / ml cada uno) para la selección de colonias azul / blanco.
- Día 2 - Confirmación de la correcta Asamblea UFP-RVDS
- Mediante el uso de colonia PCR con cebadores pCR8_F1 y pCR8_R1 (Véase la Tabla 1 para las secuencias de primers) Pantalla 1-3 blancocolonias de cada placa. Pfus-RVDS Corregir conjuntos típicamente muestran una banda correspondiente a la longitud combinada de todos los RVDS clonados (por ejemplo, alrededor de 1,1 kb para RVDS 10) y una "escalera" de bandas menos prominentes más pequeños (Figura 1B).
Programa de PCR:
95 ° C 3 min
95 ° C 30 seg
55 ° C 30 seg
72 ° C 1 min 45 seg
30-35 ciclos
72 ° C 10 min - Uso confirmó clones para iniciar cultivo durante la noche (2-5 ml de LB con 50 mg / ml de espectinomicina).
- Mediante el uso de colonia PCR con cebadores pCR8_F1 y pCR8_R1 (Véase la Tabla 1 para las secuencias de primers) Pantalla 1-3 blancocolonias de cada placa. Pfus-RVDS Corregir conjuntos típicamente muestran una banda correspondiente a la longitud combinada de todos los RVDS clonados (por ejemplo, alrededor de 1,1 kb para RVDS 10) y una "escalera" de bandas menos prominentes más pequeños (Figura 1B).
- Día 3 - Golden Gate Respuesta n ° 2 - Matrices RVD en vectores de expresión Talen
- Realizar "minipreps" para aislar las asambleas UFP-RVD (dependiendo del número de RVDS cualquiera pFUS_A y pFUS_B o pFUS_A30A, pFUS_A30B y pFUS_B).
- Opcional: vectores pfus Secuencia individuales utilizando primers pCR8_F1, pCR8_R1 (véase la Tabla 1 para las secuencias de primers). La secuenciación puede realizarse también en final TALEN construye (sección 2.5.2); No obstante, durante más completa Talens lee de todos RVDS podría no ser posible usando secuenciación de Sanger.
- Configure la puerta de oro de reacción # 2 para cada sola TALEN. 150 ng de cada vector UFP, 150 ng de PLR-HD, PLR-NG, PLR-NI, PLR-NN (última "media-repeat") de acuerdo con el diseño de la secuencia de RVD y para el l eft TALEN utilizar 75 ng de pCAG-T7-TALEN-ELD-destino y para el derecho de utilizar TALEN 75 ng pCAG-T7-TALEN-KKR-destino (o viceversa). Añadir 1 l Esp3I, 1 l de T4 ADN ligasa, 2 l de 10x tampón T4 ADN ligasa, el volumen total de reacción H 2 O a 20 l.
- Coloque reacciones Golden Gate en un ciclador térmico.
Programa:
37 ° C 5 min
16 ° C 10 min
10 ciclos
37 ° C 15 min
80 ° C 5 min - Utilice las reacciones de la sección 2.3.4 para transformar E. coli.
- Día 4 - Confirmación de la correcta Asamblea TALEN
- Pantalla 1-3 colonias blancas de cada placa por colonia PCR con cebadores TAL_F1 y TAL_R2 (véase la Tabla 1 para las secuencias de primers). Talens correctamente ensamblados muestran un producto de PCR con una longitud correspondiente al número total de RVDS Incorporated (Figura 1D, esta banda es a veces difícil de detectar, mientras que el "efecto escalera" representa un sólido indicador de conjunto de éxito).
Programa de PCR:
95 ° C 3 min
95 ° C 30 seg
55 ° C 30 seg
72 ° C 3 min
30-35 ciclos
72 ° C 10 min - Uso confirmó clones correctos para iniciar cultivos bacterianos durante la noche (2-5 ml de LB con 100 mg / ml de ampicilina).
- Pantalla 1-3 colonias blancas de cada placa por colonia PCR con cebadores TAL_F1 y TAL_R2 (véase la Tabla 1 para las secuencias de primers). Talens correctamente ensamblados muestran un producto de PCR con una longitud correspondiente al número total de RVDS Incorporated (Figura 1D, esta banda es a veces difícil de detectar, mientras que el "efecto escalera" representa un sólido indicador de conjunto de éxito).
- Día 5 - Confirmación de la correcta Asamblea TALEN
- Lleve a cabo "minipreps" aislar plásmidos pCAG-T7 Talen-.
- Si la secuencia no se realizó en sícción 2.3.2, uso cebadores TAL_Seq_5-1 y TAL_R2 (ver Tabla 1 para las secuencias de primers) para determinar asamblea RVD correcta en integral TALEN.
3. Nucleasa ARNm Síntesis
- Generación de la plantilla de ADN
- Preparar midi de alta calidad o Maxipreps de plásmidos pCAG-T7-Talen para la síntesis de ARNm.
- Linealice 10 g del plásmido síntesis TALEN o ZFN ARNm usando una enzima de restricción que escinde preferencialmente aguas abajo y en estrecha proximidad al codón de parada de nucleasa (por vectores pCAG-T7-talen utilizan SacI). plásmidos de síntesis de ARNm incluyen típicamente un promotor de T7 o SP6 fago aguas arriba de la secuencia de codificación de nucleasa.
- Ejecutar 200-500 ng de plásmido digerido en un gel de agarosa al 0,7-1% para comprobar la digestión completa.
- Eliminar las sales del plásmido digerir por precipitación del ADN con acetato de 0,1 volumen de sodio y 3 volúmenes de etanol durante 1 hora a temperatur habitacióne. Pellet el ADN por centrifugación a 14.000 x g o más durante 10 min, lavar el precipitado con etanol 200 l 70%, centrifugado durante otros 5 min, eliminar el etanol, secar al aire el precipitado y se resuspende en un volumen apropiado de agua libre de RNasa. La purificación del plásmido linealizado también es posible el uso de un sistema basado en columna, por ejemplo, kit de purificación de PCR QIAquick.
- Determinar la concentración de la plantilla lineal y utilizar 1 g de configurar la transcripción in vitro.
- la síntesis de ARNm y la poliadenilación
- Para la transcripción in vitro de pCAG-T7-TALEN plásmidos utilizan el Ultra Kit de mMESSAGEmMACHINE T7. Configurar la reacción de transcripción para cada TALEN sobre hielo: 20 l con agua libre de nucleasa, 10 l T7 2x NTP / ARCA, 2 l de 10x T7 Reaction Buffer, 1 g de ADN molde, 2 l T7 mezcla de enzimas. Mezcle la reacción y se incuba durante 1-2 horas a 37 ° C.
- Utilice la transcripción completa 20 lmezcla de reacción ción para establecer la reacción de poliadenilación en el hielo: 36 l de agua libre de nucleasa, 20 l de tampón 5x EPAP, 10 l 25 mM MnCl2, ATP 10 l 10 mM. Mezclar y retirar 2,5 l de la mezcla de reacción como muestras de control L1 y R1 (por nucleasa izquierda y derecha, respectivamente). Añadir 4 l de enzima E-PAP e incubar la reacción durante 45-60 min a 37 ° C. Retire otros 2,5 l de mezcla de reacción como muestras de control L2 y R2.
- de purificación de ARNm
- Utilice NucAway spin columnas para la purificación de ARNm (intercambio de tampón y la eliminación de los nucleótidos no incorporados). Toque en las columnas de resolver gel seco en la parte inferior de la columna. Hidrato columna con 650 l de tampón de RNasa libre de microinyección (1 mM de Tris-Cl, 0,1 de EDTA, pH 7,5). Cap, vórtice, toque las burbujas de aire, y el hidrato durante 5-15 minutos a temperatura ambiente.
- Colocar la columna en un tubo de recogida y el giroa 750 xg, 4 ° C durante 2 min para eliminar el exceso de líquido intersticial. Deseche la columna tubo colector y colocar en un tubo de 1,5 ml de elución.
- Aplique la mezcla de reacción completa de la sección 3.2.2 de la columna y centrifugado a 750 xg, 4 º C durante 2 min. Nucleasa ARNm Ahora se disolvió en tampón de microinyección. Retirar 2,5 l de las muestras purificadas y L3 R3.
- MRNA Almacenar a -80 ° C hasta que se preparan alícuotas de microinyección.
- Gel mRNA Electroforesis
- Limpie una cámara de gel para eliminar contaminaciones RNAsa usando solución de SDS al 10% o RNaseZAP.
- Preparar un gel de agarosa al 1% en tampón 1xTBE corriendo.
- Mezcle cada L1/R1 muestra de ARN, L2/R2, L3/R3 con 3 volúmenes de NorthernMax formaldehído tinte de carga y se incuba durante 15 minutos a 65 ° C.
- Cargue las muestras y una escalera de tamaño de ARN (por ejemplo, el ARN del Milenio marcador de tamaño) sobre el gely ejecutar gel a 10 V / cm en TBE 1x hasta colorante de carga alcanza el final del gel.
- Tinción del gel con solución de verde SYBR (Invitrogen) durante 30-60 min con agitación a temperatura ambiente.
- Imagen del gel y comparar tamaños de L1/R1 con L2/R2 y L3/R3. Las muestras L2/R2 y L3/R3 deben mostrar bandas de un mayor tamaño en relación con L1/R1 indicando poliadenilación éxito en la (Figura 2).
- Determinar la concentración de ARNm utilizando un espectrofotómetro.
- Preparar alícuotas de ARNm para microinyecciones mediante la mezcla de nucleasa izquierda y derecha ARNm de nucleasa en una proporción de 1:1. Se recomienda preparar alícuotas con una concentración total de 200 ng / l (100 ng / l de cada nucleasa) diluyendo con tampón de microinyección. Alícuotas de ARNm Almacenar a -80 ° C.
4. Aislamiento de embriones y microinyección
- Aislamiento de Embriones. Este protocolo ha sido utilizado con éxito con cepa C57BL/6J y BDF1s y lo más probable se puede adaptar a otras cepas comúnmente utilizados en los experimentos de microinyección como la FVB o CBF1. Los ratones se alojaron en un ciclo de luz-oscuridad de 12 h-12 h en una instalación y la temperatura y humedad controladas con comida y agua ad libitum.
- Hembras donantes superovular para aumentar el rendimiento de los embriones. 16 mujeres (4 semanas de edad) se superovularon por vía intraperitoneal (ip) inyección de gonadotropina sérica 5 UI de yegua preñada (PMSG), seguido de la inyección ip de 5 UI de gonadotropina coriónica humana (hCG) 48 horas más tarde.
- Mate de las 16 mujeres superovuladas a 16 la edad de reproducción (2-8 meses) hombres inmediatamente después de la inyección de hCG.
- Sacrificar las mujeres utilizando un protocolo de eutanasia aprobado, como inhalación de CO2.
- Recuperar los ovocitos fertilizados. Los ovocitos se recogen de oviductos la mañana siguiente y luego liberados de cualquier resto de células del cúmulo por un tratamiento de 3-5 minutos en hialuronidasa bovina 0,1% disuelto en medio M2. Dependiendo de la capacidad de apareamiento y la cepa utilizada el rendimiento de embriones puede variar. 16 hembras C57BL/6J suelen producir 150-250, mientras que BDF1 dió 300-400 ovocitos fertilizados, respectivamente.
- Microinyección de embriones
- Inyectar el ARNm nucleasa. Ovocitos etapa pronuclear suelen inyectarse en la tarde temprano. Citoplásmica ARNm microinyección se lleva a cabo en medio M2 bajo aceite mineral usando un microscopio invertido equipado con DIC Nomarski usando objetivo 20X y con micromanipuladores embrión así como una unidad de inyección. Se recomienda comenzar las inyecciones de TALEN ARNm a una concentración de 10 ng / l (20 ng / l en total).
- Aspirar el ovocito con un capilar de retención e inyectar el ARNm nucleasa en el citoplasma del embrión evitando el contacto con los pronúcleos. La inyección debe ser poco profunda, cerca de la membrana plasmática. El volumen de inyección debe mantenerse baja y la aguja de microinyección debe ser ingeniohdrawn al primero signo distención citoplasmática. Una serie de microinyección consistirá normalmente en 100-300 ovocitos. Típicamente, al menos el 80-90% de los embriones debe sobrevivir a la inyección sin lisis inmediata.
- Tras la microinyección, colocar los embriones durante 1 hora en la M16 (Sigma) a 37 ° C y 5% de CO 2 y transferir los embriones que sobreviven en madres adoptivas pseudopreñadas.
5. Transferencia de Embriones Quirúrgica
- Preparar receptoras de embriones pseudopreñadas (madres adoptivos) un día antes de la microinyección de ARNm. Las hembras maduras (3-6 meses de edad) de una cepa no consanguínea robusto, dicho CD-1, son muy adecuadas para el papel. Inducir pseudopreñez en el apareamiento de las hembras en un día anterior con los hombres, ya sea quirúrgicamente o genéticamente vasectomizados 28. Sólo las hembras 0,5 dpc con un tapón copulatory clara se utilizan como receptores embriones. En las mujeres sin usar la pseudopreñez desaparece después de alrededor de 3 weeks permiten su uso repetido.
- Anestesiar los 0,5 dpc madre adoptiva hembras por inyección ip de ketamina, xilazina (120 mg / kg y 16 mg / kg, respectivamente). Esta formulación garantías ~ 30 min de tiempo de tolerancia quirúrgica es más que suficiente para el tiempo de funcionamiento previsto de 5-10 min.
- Coloque el animal anestesiado en el abdomen sobre una superficie caliente y desinfectar el área de la incisión con un desinfectante adecuado tal como etanol 70% o clorhexidina y mezcla de alcoholes.
- Una incisión en la piel en la parte posterior del animal y abrir la cavidad peritoneal con tijeras estériles.
- Visualizar y exteriorizar el cuerno uterino, tirando de la almohadilla de grasa unida al ovario. Mantenga los órganos húmedo utilizando cálida solución de NaCl al 0,9%. Tenga en cuenta que el lugar de la cirugía puede tener que ser cubierta con un vendaje estéril o afeitado con el fin de ajustarse a las directrices veterinarias locales.
- Inmovilizar los órganos reproductivos de la saturación de la almohadilla de grasa de ovario con una pinza bulldog.
- Agarre suavemente el oviducto justo aguas arriba de la ampolla con pinzas de relojero y utilizar una aguja hipodérmica 30 G para crear un pequeño agujero en la pared del oviducto.
- Retire la aguja y vuelva a insertar un capilar de vidrio delgado que contiene los embriones dentro del agujero que permite la colocación de los embriones en la ampolla soplando suavemente en la boquilla del soporte capilar. Retirar el capilar una vez que los embriones se depositan en la ampolla y reemplazar los órganos reproductivos de nuevo en la cavidad del cuerpo.
- Cierre la cavidad peritoneal con una serie de suturas estériles (Prolene 6-0.)
- Cierre la piel con 1-2 grapas para heridas Autoclip (9 mm), dependiendo del tamaño de la abertura.
- Después de la operación de retorno de los animales a sus jaulas y supervisan hasta que el efecto de la anestesia en desaparecer.
- Con el fin de minimizar el estrés, la casa de los ratones en grupos sociales estables (2-4 animales / jaula tipo III) siempre que sea posible.
- Aplicar analgésico postoperatorios en la forma de Dafalgan añaden al agua de bebida (paracetamol 200 mg / kg de peso corporal) durante 3 días después de la cirugía.
6. Análisis de los Fundadores por PCR y T7 endonucleasa o restricción Digestión enzimática
- Diseño cebadores para amplificar una región entre 200-700 pb alrededor del sitio de unión nucleasa. Las distancias hacia adelante región espaciadora-cebador de nucleasa y nucleasa espaciador-región de cebador inverso deberían ser lo suficientemente diferentes para permitir la detección de dos bandas separadas de los productos digeridos en un gel de agarosa (ver Figuras 3 y 4 para ejemplos).
- Ejecute PCR con condiciones optimizadas. Compruebe tamaño de amplificación de PCR en un gel de agarosa.
- Opcional: productos Purificar PCR, por ejemplo, con el Kit de Purificación de PCR QIAquick para eliminar las sales y los nucleótidos de la mezcla de PCR. Muchas enzimas de restricción son activos en muestras de PCR complementado con el tampón de enzima de restricción apropiado y purificación no se necesita. También tenemos successfuutilizado LLY T7 endonucleasa en tampón de PCR QiagenTaq complementa con NEBuffer 2.
- Ensayo de T7 endonucleasa. Mezclar 17 l de producto de PCR con 2 l de NEBuffer 2 y ejecutar la formación de heterodúplex (Figura 3b) del programa en un termociclador:
95 ° C 2 min
95 ° C a 85 ° C (-2 ° C / seg)
85 ° C a 25 ° C (-0,1 º C / seg)
4 ° C espera.
Añadir 1 l de T7 endonucleasa a cada muestra y se incuba a 37 ° C durante 20 min. (El ensayo de agrimensor (Transgenomic), que se basa en la CEL 1 nucleasa, también se puede utilizar aquí. Por favor, consulte las instrucciones del fabricante.) - Digestión de restricción de productos PCR. Mezclar 17 l de producto de PCR con 2 l de 10x tampón de enzima de restricción y 1 l de enzima de restricción. Digerir a temperatura adecuada durante 1 hora o más.
- Ensayo de T7 endonucleasa. Mezclar 17 l de producto de PCR con 2 l de NEBuffer 2 y ejecutar la formación de heterodúplex (Figura 3b) del programa en un termociclador:
- Añadir colorante de carga de ADN de muestras y correr en un gel de agarosa al 2% para detectar la digestión distintapatrones para animales de tipo salvaje y fundadores portadores de alelos mutados. Vea las Figuras 3 y 4 para los resultados esperados.
- Productos de PCR de Clon fundadores positivos para los alelos mutados para la secuenciación de Sanger (por ejemplo, por TA-clonación en pGEM-T Easyor secuenciar directamente las mezclas de los productos de PCR usando secuenciación de próxima generación.
Resultados
Hemos construido plásmidos de destino compatible con el conjunto de oro GateTALEN publicado por Cermak et al. 13 que permiten la expresión de talens en células de mamífero, así como in vitro la síntesis de ARNm a partir del promotor del fago T7 (Figura 1C). Estos plásmidos llevan dominios FokI heterodiméricas (mutaciones ELD o KKR) que han sido mostrados para reducir fuera de objetivo efectos relativos a homodímeros FokI y para mejorar la actividad de escisión en comparación con la primera generación FokI heterodímeros 25,29. Reacciones de montaje de puerta de oro # 1 y # 2 son por lo general muy eficiente y cada colonia blanco, cuando se analiza por PCR de colonias, muestra el patrón esperado para el número particular de RVDS clonados (Figuras 1B y 1D).
Análisis en gel de ARNm sintetizado in vitro (Figura 2) debe revelar una sola banda distinguibles con poco o nada de frotis para cada muestra analizada. Debe haber un cambio de tamaño claramente entre muestras L1/R1 y L2/R2, L3/R3, lo que indica poliadenilación éxito.
Los animales fundadores pueden ser examinados para alelos mutados inducidas-NHEJ utilizando PCR genotipo seguido de digestión con endonucleasas de T7 (Figura 3) o una digestión de restricción usando una enzima que escinde la secuencia de tipo salvaje dentro de la región espaciadora del par de nucleasa (Figura 4). El ensayo de T7 endonucleasa es aplicable a cualquier tipo de mutación, independientemente de la secuencia genómica específica dentro de la región espaciadora del par nucleasa inyectado; sin embargo, sólo detecta desajustes entre las hebras de ADN en los productos de PCR heterodúplex. Así, en el raro caso de que uno de los fundadores lleva dos alelos mutados de forma idéntica, los productos PCR se no mostrar ningún patrón de digestión T7. Tal distinción es, sin embargo, siempre es posible cuando un sitio de restricción específica se encuentra dentro de la región espaciadora nucleasa que se elimina por NHEJInducida inserciones / supresiones (Figura 5). Aquí, las bandas no digeridas indican la presencia de mutaciones, y la ausencia de cualquiera de los productos digeridos sugiere fuertemente un fundador llevar a mutaciones en ambos alelos del gen diana (marcadas con asteriscos en la Figura 4b).
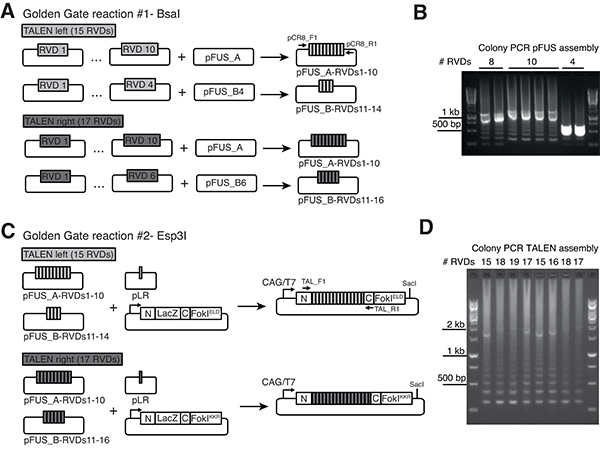
Figura 1. Golden Gate clonación de Talen RVDS en vectores pCAG-T7-Destination heterodímeras. A) Asamblea de arrays RVD en vectores UFP. Aquí se muestra un ejemplo de un par TALEN con matrices CUENTO individuales que comprende 15 RVDS y 17 RVDS, respectivamente (para matrices CUENTO más de 21 RVDS, se requieren tres UFP-RVDS asambleas, no se muestra). Las flechas indican cebadores para pfus específica reacciones de PCR colonia. B) Los productos de PCRamplificado a partir de conjuntos de pfus correctas típicamente muestran una banda correspondiente a la longitud combinada de todos los RVDS clonados (por ejemplo, alrededor de 1,1 kb para RVDS 10) y una "escalera" de pequeñas bandas menos prominentes debido a la naturaleza repetitiva de matrices de RVD. C) El montaje final de arrays pfus-RVD y un plásmido que contiene la última repetición (PLR) en vectores de expresión heterodímeras Talen con FokIELD y FokIKKR variantes, respectivamente. La columna vertebral TALEN (anotado como N y C) se asemeja a la arquitectura publicado por Miller et al. 23 El CAG (CMV elemento potenciador temprano / pollo beta-actina) promotor asegura altos niveles de expresión en células de mamífero transfectadas, mientras que el promotor del fago T7 permite en la síntesis de ARNm in vitro (SacI utilizar para la linealización del vector de aguas abajo del codón STOP nucleasa). D) PCR de colonias usando cebadores indicados por las flechas en C) permite la identificación de ensamblado correctamente Talens. Full-lengtproductos h PCR suelen ser menos prominente, mientras que el "efecto escalera" representa un sólido indicador de reunión exitosa. Haga clic aquí para ver la imagen más grande .
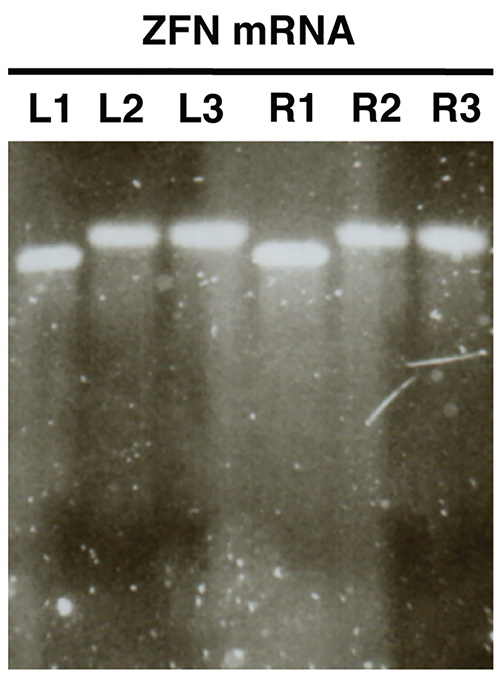
Figura 2. . Control de calidad de ARNm de nucleasa en la síntesis in vitro usando electroforesis en gel de agarosa ARNm ZFN se muestra como un ejemplo (L, izquierda ZFN, R, ZFN derecha). Muestras L1/R1 muestran ARNm antes de poliadenilación, muestras L2/R2 espectáculo polyadenylated mRNA y L3/R3 muestran mRNA poliadenilado purificado. Haga clic aquí para ver la imagen más grande .
alt = "Figura 3" fo: content-width = "6 pulgadas" fo: src = "/ files/ftp_upload/50930/50930fig3highres.jpg" src = "/ files/ftp_upload/50930/50930fig3.jpg" width = "600" />
Figura 3. Ejemplo de un ensayo de T7 endonucleasa utilizado para la identificación de animales fundadores que llevan las mutaciones inducidas por nucleasa-de el locus diana. A) Un par TALEN fue diseñado para romper dentro de la región codificante del gen de la proteína del prión del ratón (Prnp, secuencia diana TALEN pueden ser disponibles bajo petición). Un producto de la PCR se genera usando un cebador directo (F) situado 110 pb aguas arriba y un cebador inverso (R) 250 pb aguas abajo del sitio de escisión TALEN. B) El producto de PCR posteriormente se somete a la formación de heterodúplex y T7 digestión con endonucleasas. C) mutagénesis inducida por TALEN dentro de la región genómica específica de los fundadores individuales se revela por la presencia de un producto de PCR de longitud completa con productos de digestión de 250 y 110 pb.50930/50930fig3highres.jpg "target =" _blank "> Haga clic aquí para ver la imagen más grande.

Figura 4. Ejemplo de un digesto de restricción de productos de PCR utilizados para la identificación de animales fundadores que llevan las mutaciones inducidas por nucleasa-de el locus diana. ZFN específicas para el locus de ratón Rosa26 29 diana un sitio de restricción XbaI en el intrón 1.) Fundadores A fueron seleccionadas por PCR genotipo utilizando un cebador directo (F) situado 500 pb aguas arriba y un cebador inverso (R) 250 pb aguas abajo del sitio de escisión. B) La digestión de los productos de PCR con XbaI revela patrones de digestión que indican ratones con mutaciones bi-alélicas (marcados con asteriscos), mono -alélicas mutaciones (bandas digeridos y no digeridos, por ejemplo, animales 2)y los ratones wt (digestión completa, por ejemplo, los animales 21.) C) La secuenciación de los ratones con posibles modificaciones bi-alélica muestra un máximo de 3 (24 animales) distintas inserciones / supresiones. Haga clic aquí para ver la imagen más grande .
| Nombre de la cartilla | Secuencia 5 'a 3' |
| pCR8_F1 | ttgatgcctggcagttccct |
| pCR8_R1 | cgaaccgaacaggcttatgt |
| TAL_F1 | ttggcgtcggcaaacagtgg |
| TAL_R2 | ggcgacgaggtggtcgttgg |
| TAL_Seq_5-1 | catcgcgcaatgcactgac |
Tabla 1. Las secuencias de los primers utilizados para la PCR de colonias y secuenciación dentro del conjunto de la puerta de oro TALENprotocolo.
| Plásmido / Colección | Contribuyente | Addgene ID | Comentarios |
| Golden Gate TALEN y TAL Efector Kit 2.0 | Laboratorio Voytas | 1000000024 | Contiene todos los plásmidos necesarios para el montaje de la puerta de oro TALEN |
| destino vectores pCAG-T7-TALEN -KKR/ELD | Laboratorio Pelczar | 40131, 40132 | Add-on plásmidos para la expresión TALEN en células de mamíferos y en la síntesis de ARNm vitro |
Tabla 2. Plásmidos y colecciones de plásmidos requeridos para el montaje de puerta de oro TALEN se pueden obtener de Addgene ( www.addgene.org ).
| OnlineRecurso | Comentarios |
| http://tale-nt.cac.cornell.edu | Diseño de TALEN; TALEN predicción fuera del objetivo |
| http://zifit.partners.org/ZiFiT/ | Diseño de TALEN, OPEN ZFN, CoDA ZFN, CRISPR/Cas9 |
| http://www.genome-engineering.org | Diseño de TALEN, CRISPR/Cas9; CRISPR/Cas9 fuera del objetivo de predicción |
| http://baolab.bme.gatech.edu/Research/BioinformaticTools/assembleTALSequences.html | Asamblea de TALEN secuencias para la confirmación de los resultados de secuenciación |
| http://pgfe.umassmed.edu/ZFPmo dularsearchV2.html | Diseño de montaje modular ZFN |
| www.genomecenter.ucdavis.edu/s egallab / segallabsoftware | Diseño de montaje modular ZFN |
Tabla 3. Recursos en línea para diseñar ZFN, TALEN y CRISPR/Cas9.
Discusión
Genoma enfoques edición nucleasa impulsada diseño han ampliado considerablemente la gama de especies susceptibles de modificaciones específicas de sus respectivos genomas 10,12. En ratones, el gen de la orientación en células madre embrionarias ha sido una técnica estándar para más de dos décadas; sin embargo, ha demostrado ser difícil de adaptar a las células madre embrionarias de especies distintas de la del ratón, aunque ha habido algunos éxitos recientes en ratas células madre embrionarias. Incluso con la disponibilidad de "off-the-shelf" clones de células madre embrionarias de ratón con genes dirigidos proporcionadas por consorcios como EUCOMM, KOMP o NorCOMM 3 edición genoma por ZFN y TALEN proporciona mayor precisión y flexibilidad en cuanto al espectro de las modificaciones que pueden ser introducido en el genoma de ratón. Los animales fundadores que llevan mutaciones nucleasa mediada parecen ser altamente línea germinal competente 4-6,20,21, que no es siempre el caso para quimeras procedentes de inyecciones de blastocistos de células ES. Por lo tanto, en ciertos casos la microinyección de desnucleasas igner pueden dar lugar a significativamente más rápida generación de líneas novedosas de ratón con modificaciones del genoma específicas.
La generación exitosa de ratones knockout por inyección de ZFN y TALEN depende en gran medida de la actividad de la nucleasa par inyectado. Talens han demostrado tener una alta tasa de éxito en la selección de una amplia gama de genes en un número de organismos; Sin embargo, estudios recientes sugieren que TALEN unión es sensible a la metilación de citosina 30,31. Por lo tanto, los pares de nucleasa recién generadas, por ejemplo Talens clonó en vectores de pCAG-T7, pueden ser transfectadas transitoriamente en una línea celular de ratón, tales como NIH-3T3 o Neuro -2a, que imitan el estado de la cromatina del embrión de ratón en cierta medida. Aquí, la actividad nucleasa puede estimarse utilizando el ensayo de T7 endonucleasa o un compendio de restricción de productos PCR como se describe en la sección 5 antes de la síntesis de ARNm y microinyección. Recomendamos secuenciación de la región genómica de interés en el respetoive línea celular y la cepa de ratón utilizada para experimentos de microinyección.
En cigotos de ratón, diferentes pares TALEN o ZFN trabajarán de manera óptima a diferentes concentraciones de ARNm y por lo tanto la concentración de trabajo óptima del ARNm nucleasa microinyectados pueden tener que ser determinado experimentalmente. Dependiendo del par nucleasa, una concentración demasiado baja dará lugar a ninguna escisión, mientras que demasiado alto puede dar lugar a la letalidad embrionaria. Dependiendo del par nucleasa, hemos tenido éxito en el uso total de las concentraciones de ARNm tan bajas como 2 ng / l y tan alto como 200 mg / ul. Estos efectos son difíciles de predecir a partir de experimentos en cultivo de células y la concentración óptima de nucleasa tanto para la supervivencia del embrión y la velocidad de modificación del locus diana tiene que ser determinado empíricamente.
ZFN de gran actividad o TALEN pueden escindir su secuencia objetivo más allá de la fase de una célula del embrión microinyectado y por lo tanto causar patrones complejos de una mutagénesisd mosaicismo en fundadores. Nosotros y otros 4 han observado tres o más alelos mutados distintos en un único fundador (Figura 5C). Por lo tanto, al establecer una nueva línea de ratón a partir de estos fundadores, la descendencia deben ser examinadas cuidadosamente mediante la secuenciación de la presencia de la mutación favorable ya que los ensayos de digestión proporcionan evidencia de que sólo una mutación sin definir está presente.
Una de las críticas con frecuencia expresaron en contra de los sistemas de ZFN y TALEN es la posibilidad de que estas nucleasas también son capaces de escindir secuencias presentes en otro lugar en el genoma que son similares a los sitios diana. Tales fuera de objetivo efectos se han observado con reactivos de primera generación utilizando el dominio FokI homodimérica y construcciones heterodímero fueron diseñados para aliviar fuera de objetivo efectos 25. Sitios fuera de objetivo potencial se pueden predecir en cierta medida en silico 32,33 y se cribaron por PCR y secuenciación. Una ventaja obvia Of utilizando ZFNs y Talens para generar los ratones en lugar de líneas celulares es la posibilidad de eliminar de las mutaciones de destino no ligados a la modificación del genoma deseada mediante la realización de varios retrocruzamientos a una cepa de tipo salvaje de la elección. Para el análisis de un gran número de ratones fundadores, de próxima generación profunda secuenciación de los productos PCR generados a partir del locus nucleasa específica y en silico predichos desviado loci podría ofrecer una lectura cualitativa y cuantitativa alternativa a los ensayos de digestión de productos de PCR.
Las técnicas de reproducción asistida que se describen en este protocolo se optimizan para las cepas de ratón estándar utilizados para experimentos de microinyección como C57Bl/6J o B6D2F1. Los ratones de diferentes orígenes, como las cepas no consanguínea, pueden, en principio, se utilizarán para las aproximaciones de edición del genoma y podrían proporcionar una base genética más adecuado para las preguntas de investigación específicas. El rendimiento de las técnicas de reproducción asistida como la superovulación puede ser predicadocted para un número de cepas de 34-36 pero podría requerir una mayor optimización para las cepas no estándar con el fin de obtener un número suficiente de embriones para microinyección nucleasa.
Además ZFN y TALEN, nuevos nucleasas de diseñadores como el sistema CRISPR/Cas9 ARN guiada 9,37,38 ahora se han introducido para aplicaciones de edición de genoma. Todos los métodos para la microinyección y análisis de animales fundadores descritos aquí son también aplicables a CRISPR/Cas9 y futuras modos de edición genoma.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Nos gustaría dar las gracias a Monika Tarnowska, Cornelia Albrecht, y Ewa Skoczylas por su excelente asistencia técnica. Este estudio fue financiado por SNF Sinergia subvención CRSI33-125073 al PP.
Materiales
| Name | Company | Catalog Number | Comments |
| BsaI | NEB | R0535S or L | |
| Esp3I | Thermo Scientific | ER0451 | |
| T4 Ligase | NEB | M0202S or L | |
| Spectinomycin | Sigma | S0692-1ML | |
| Ampicillin | Sigma | A0166 | |
| X-Gal | Sigma | B4252 | |
| IPTG | Sigma | I6758 | |
| Plasmid-Safe nuclease | Epicentre | E3101K | |
| QIAprep Spin Miniprep Kit | Qiagen | 27106 | |
| QIAGEN Plasmid Midi Kit | Qiagen | 12143 | |
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| mMESSAGE mMACHINE T7 Ultra Kit | Invitrogen | AM1345 | |
| NucAway Spin Columns | Invitrogen | AM10070 | |
| RNaseZAP | Sigma | R2020-250ML | |
| NorthernMax Formaldyde Load Dye | Invitrogen | AM8552 | |
| RNA Millennium Markers | Invitrogen | AM7150 | |
| 10x TBE buffer | Thermo Scientific | B52 | |
| T7 endonuclease | NEB | M0302S or L | |
| pGEM-T EasyVector System I | Promega | A3600 | |
| SYBR Green I Nucleic Acid Gel Stain | Invitrogen | S-7563 | |
| pregnant mare's serum gonadotrophin (PMSG) | Sigma | G4877 | |
| human chorionic gonadotropin (hCG) | Sigma | CG5 | |
| M2 embryo culture medium | Sigma | M7167 | |
| M16 embryo culture medium | Sigma | M7292 | |
| Mineral oil, embryo tested | Sigma | M8410 | |
| Ketamine | CentraVet | Ket 201 | |
| Xylazine | Sigma Aldrich | 46995 | |
| Equipment/Tools | |||
| Inverted microscope with Nomarski DIC optics (for example Nikon Eclipse TE200) | Nikon | ||
| Micromanipulator units (for example Narishige, NT88NF) | Narishige | ||
| Embryo holding capillaries | Sutter Instruments | B100-75-10 | |
| Embryo injection capillaries | Narishige | GD-1 | |
| Capillary puller (for example Sutter P97) | Sutter Instruments | ||
| Microforge (for example Narishige MF-900) | Narishige | ||
| Walton skin scissors | FST | 14077-10 | |
| Surgical scissors | FST | 14041-10 | |
| Surgical probe | FST | 10140-03 | |
| Reflex wound clip system (9 mm) | FST | 12031-09 | |
| Reflex wound clips (9 mm) | FST | 12032-09 | |
| Dumont fine forceps 5 | FST | 11254-20 | |
| Moria curved forceps | FST | 11370-31 | |
| Moria fine forceps | FST | 11399-80 | |
| Dietrich bulldog clamp | FST | 18038-45 | |
| C57BL/6J mice | Jackson Labs | strain code 000664 | |
| CD-1 mice | Charles River | strain code 000664 | |
Referencias
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat. Rev. Genetics. 6, 507-512 (2005).
- Johansson, T., et al. Building a zoo of mice for genetic analyses: a comprehensive protocol for the rapid generation of BAC transgenic mice. Genesis. 48, 264-280 (2010).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Carbery, I. D., et al. Targeted genome modification in mice using zinc-finger nucleases. Genetics. 186, 451-459 (2010).
- Meyer, M., de Angelis, M. H., Wurst, W., Kuhn, R. Gene targeting by homologous recombination in mouse zygotes mediated by zinc-finger nucleases. Proc. Natl. Acad. Sci. U.S.A. 107, 15022-15026 (2010).
- Cui, X., et al. Targeted integration in rat and mouse embryos with zinc-finger nucleases. Nat. Biotechnol. 29, 64-67 (2011).
- Sung, Y. H., et al. Knockout mice created by TALEN-mediated gene targeting. Nat. Biotechnol. 31, 23-24 (2013).
- Wefers, B., et al. Direct production of mouse disease models by embryo microinjection of TALENs and oligodeoxynucleotides. Proc. Natl. Acad. Sci. U.S.A. 110, 3782-3787 (2013).
- Wang, H., et al. One-Step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat. Rev. Genetics. 11, 636-646 (2010).
- ZFN, T. A. L. E. N. CRISPR/Cas-based methods for genome engineering. Trends Biotechnol. 31, 397-405 (2013).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nat. Rev. Mol. Cell Biol. 14, 49-55 (2013).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39, (2011).
- Reyon, D., et al. FLASH assembly of TALENs for high-throughput genome editing. Nat. Biotechnol. 30, 460-465 (2012).
- Sanjana, N. E., et al. A transcription activator-like effector toolbox for genome engineering. Nat. Protoc. 7, 171-192 (2012).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nat. Biotechnol. 31, 251-258 (2013).
- Schmid-Burgk, J. L., Schmidt, T., Kaiser, V., Honing, K., Hornung, V. A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes. Nat. Biotechnol. 31, 76-81 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 31, 827-832 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat. Biotechnol. 31, 822-826 (2013).
- Hermann, M., et al. Evaluation of OPEN zinc finger nucleases for direct gene targeting of the ROSA26 locus in mouse embryos. PLoS ONE. 7, (2012).
- Meyer, M., Ortiz, O., Hrabe de Angelis, M., Wurst, W., Kuhn, R. Modeling disease mutations by gene targeting in one-cell mouse embryos. Proc. Natl. Acad. Sci. U.S.A. 109, 9354-9359 (2012).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat. Biotechnol. 29, 143-148 (2011).
- Guo, J., Gaj, T., Barbas, C. F. Directed evolution of an enhanced and highly efficient FokI cleavage domain for zinc finger nucleases. J. Mol. Biol. 400, 96-107 (2010).
- Doyon, Y., et al. Enhancing zinc-finger-nuclease activity with improved obligate heterodimeric architectures. Nat. Methods. 8, 74-79 (2011).
- Wefers, B., et al. . Current Protocols in Mouse Biology. , (2011).
- Wefers, B., Wurst, W., Kühn, R. . Current Protocols in Mouse Biology. , (2011).
- Haueter, S., et al. Genetic vasectomy-overexpression of Prm1-EGFP fusion protein in elongating spermatids causes dominant male sterility in mice. Genesis. 48, 151-160 (2010).
- Perez-Pinera, P., Ousterout, D. G., Brown, M. T., Gersbach, C. A. Gene targeting to the ROSA26 locus directed by engineered zinc finger nucleases. Nucleic Acids Res. 40, 3741-3752 (2012).
- Bultmann, S., et al. Targeted transcriptional activation of silent oct4 pluripotency gene by combining designer TALEs and inhibition of epigenetic modifiers. Nucleic Acids Res. 40, 5368-5377 (2012).
- Valton, J., et al. Overcoming TALE DNA binding domain sensitivity to cytosine methylation. J. Biol. Chem. 287, 38427-38432 (2012).
- Cradick, T. J., Ambrosini, G., Iseli, C., Bucher, P., McCaffrey, A. P. ZFN-Site searches genomes for zinc finger nuclease target sites and off-target sites. BMC Bioinform. 12, (2011).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Byers, S. L., Payson, S. J., Taft, R. A. Performance of ten inbred mouse strains following assisted reproductive technologies (ARTs). Theriogenology. 65, 1716-1726 (2006).
- Luo, C., et al. Superovulation strategies for 6 commonly used mouse strains. J. Am. Assoc. Lab. Animal Sci. 50, 471-478 (2011).
- Pease, S., Saunders, T. L. . International Society for Transgenic Technologies. Advanced protocols for animal transgenesis : an ISTT manual. , (2011).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339, 819-823 (2013).
- Mali, P., et al. RNA-Guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados