Method Article
Maus-Genom-Engineering mit Designer Nukleasen
In diesem Artikel
Zusammenfassung
Designer-Nukleasen wie Zink-Finger-Nukleasen (ZFNs) und Transkriptionsaktivator-Effektor wie Nukleasen (Talens) kann verwendet werden, um das Genom der Maus Präimplantationsembryonen durch Auslösen sowohl die nicht-homologe Ende (NHEJ) und die homologe Rekombination (HR) Wege ändern. Diese Fortschritte ermöglichen die schnelle Generierung von Mäusen, die mit präzisen genetischen Veränderungen.
Zusammenfassung
Transgene Mäuse, die ortsspezifische Genom-Modifikationen (KO, Knock-in) sind von entscheidender Bedeutung für die Zerlegung komplexer biologischer Systeme als auch für die Modellierung menschlichen Krankheiten und Therapiestrategien testen. Jüngste Fortschritte in der Verwendung von Designer-Nukleasen wie Zink-Finger-Nukleasen (ZFNs), Transkriptions-Aktivator-Effektor wie Nukleasen (Talens) und den Cluster regelmäßig beabstandet kurze Palindrom-Wiederholungen (CRISPR) / CRISPR-assoziierten (Cas) 9-System für Website- spezifische Genomtechnik die Möglichkeit offen, um eine schnelle gezielte Modifikation Genom in praktisch allen Labortieren durchführen, ohne die Notwendigkeit, an embryonalen Stammzellen (ES)-Technologie verlassen. Ein Genom Bearbeitung Experiment beginnt in der Regel mit der Identifizierung von Designer-Nuklease Zielorten innerhalb eines Gens von Interesse, gefolgt von Bau von kundenspezifischen DNA-bindenden Domänen Nuklease-Aktivität direkt an die Ermittler definierten genomischen Locus. Designer Nuklease Plasmide sind in vitro </ Em> transkribiert, um mRNA für die Mikroinjektion von befruchteten Eizellen der Maus zu erzeugen. Hier bieten wir ein Protokoll für das zielgerichtete Modifikation Genom durch direkte Injektion TALEN mRNA in befruchtete Eizellen der Maus.
Einleitung
Mäuse sind mit Abstand die beliebteste Plattform zur Erzeugung transgener Tiermodelle. Die vielseitige Werkzeugkasten für die Gentechnik von Mausembryo 1-3 wurde kürzlich von Genom Bearbeitung Ansätzen für Designer Nukleasen wie Zink-Finger-Nukleasen (ZFN) verlängert 4-6, Transkriptionsaktivator-Effektor wie Nukleasen (TALEN) 7,8, und die Cluster regelmäßig beabstandet kurze Palindrom-Wiederholungen (CRISPR) / CRISPR-assoziierten (Cas) 9 System 9. ZFN und TALEN Funktion als Paare von zwei maßgeschneiderten Protein-basierten DNA-bindende Domänen (Arrays von Zinkfinger-Proteine und wiederholen Variablen di-Rückstände (RVDS), respectively), die jeweils mit der FokI Endonuklease 10-12 gekoppelt sind. Umgekehrt wird die Spezifität der Cas9-vermittelten DNA-Spaltung durch zu trans CRISPR RNAs (crRNA und tracrRNA, die auch in einem einzigen chimären RNA-Molekül bezeichnet Führungs RNA kombiniert werden können) zur Verfügung gestellt, die 11 in einem Komplex mit dem GesetzCRISPR Protein.
TALENS mit einer definierten Sequenz des RVDS schnell einzelner Experimentatoren mit einer Vielzahl von Strategien zur Montage 13-17 wählen konstruiert werden. CRISPR/Cas9 verspricht noch weniger arbeitsintensiv Generation von Designer-Nukleasen, aber die Spezifität von Führungs RNA-DNA-Bindung ist immer noch nicht vollständig gelöst 18,19. Erstellung von benutzerdefinierten ZFNs bisher an spezialisierte akademische Labors und kommerzielle Anbieter wie Sangamo Biosciences und Sigma CompoZr Dienst beschränkt.
In der Regel soll Genom Bearbeitung mit Designer-Nukleasen auf die Einführung Doppelstrangbrüche (DSB) an definierten genomischen Loci, die anschließend zu gewinnen nicht-homologe Ende (NHEJ) oder homologe Rekombination (HR) DNA-Reparatur-Maschinen 10,12. NHEJ-vermittelte Reparatur eines DSB oft zur Einführung von Insertionen und Deletionen in der Nähe der Stelle der Reparatur. So NHEJ Reparatur czum Ausschlagen der Funktion eines Zielgens durch die Einführung einer Rahmenverschiebungsmutation in der Gene-Protein-kodierende Sequenz ein 4,7,9 ausgenutzt werden. Alternativ kann definiert Hinzufügen oder Austauschen von genetischer Information durch die Bereitstellung einer DNA-Donor zusammen mit den Designer Nukleasen erreicht werden. Ein DNA-Donor umfasst Forschern entwickelt DNA-Sequenzen, die durch Bereiche der Homologie mit dem Ziel-Locus flankiert, so als Vorlage für DSB-Reparatur dien von HR. Beide Plasmide 5,6,20 und einzelsträngige Oligonukleotide 8,9,21 wurden erfolgreich als Donoren verwendet. Weder NHEJ-oder HR-vermittelte Genom Bearbeitung erfordern die Einführung eines selektierbaren Markers in das Genom der Maus-Embryo, der diese Strategien besonders gut für die Erstellung von kleinen Veränderungen in der Nukleotidsequenz, ohne die Gesamtarchitektur genetische Störung geeignet macht.
In diesem Protokoll beschreiben wir alle wesentlichen Verfahren für die Genom-Bearbeitung in derMaus-Embryo mit Talens. Dazu gehören 1) Identifizierung eines TALEN Zielstelle 22, 2) Bau von Talens von golden gate Klonen 13, 3) in vitro Synthese von mRNA TALEN, 4), die Mikroinjektion von mRNA TALEN in befruchtete Eizellen der Maus, 5) chirurgische Verfahren zur Embryo-Transfer und 6) Analyse der TALEN-induzierten Mutagenese in Gründertiere. Wir konzentrieren uns auf TALEN mRNA-Mikroinjektion und Screening von Gründern für NHEJ-induzierte Insertionen / Deletionen. Zu diesem Zweck haben wir bifunktionellen TALEN Konstrukte, die sowohl die Expression in Säugerzellen, wenn sie als Plasmide und in vitro Synthese von mRNA TALEN für die Mikroinjektion in transfizierten Mäuse-Embryonen ermöglichen erzeugt. Diese Konstrukte umfassen einen Kegel GESCHICHTE Rückgrat 23 bis heterodimeren FokI-Domänen fusioniert 24,25 optimale Genom Bearbeitung in Säugerzellen. Dieses Protokoll kann auch für die Mikroinjektion von anderen Designer-Nukleasen oder zur kombinierten Injektionen von Designer-Nukleasen angenommen werdenund Geber Konstrukte (Design von DNA-Geber ist in der ausgezeichneten technischen Publikationen von Wefers et al. 26,27) beschrieben worden.
Ethische Erklärung
Alle Tierversuche wurden in Übereinstimmung mit den Richtlinien und Bestimmungen der kantonalen Veterinäramt des Kantons Zürich durchgeführt.
Protokoll
1. Identifizierung von Ziel TALEN Seiten
- Besuchen Sie die TAL-Effektor Nucleotide Targeter 2.0-Website (http://tale-nt.cac.cornell.edu) und wählen Sie "TALEN Targeter".
- Geben die Sequenz des Zielgens. Wenn pCAG-T7-TALEN Expressionskonstrukte verwendet wird (Abbildung 1C) wählen Sie "Miller et al., 2011" unter "Verwenden Sie eine Preset-Architektur", um Ziel Websites, die mit der optimalen Distanzlänge richtet werden kann vorhersagen (15-20 bp) und die Zahl der TALE RVDS (15-20) für diesen TALEN Architektur.
- Wählen Sie "NN" unter "G Substitute" (Guanosin-spezifische NH RVDS sind auch vorhanden, aber noch nicht ausgiebig für TALEN Montage getestet).
- Optional: für andere Einstellungen und Optionen, folgen Sie bitte den Anweisungen auf der Website und die dort angegebenen Links.
- Eine Textdatei mit potenziellen Zielseiten erzeugt, die sein können,in ein Tabellenkalkulationsprogramm für bequemere Betrachtung importiert. Wählen Sie das TALEN Paar (e) für Golden Gate Montage.
- Optional: Sequenz der genomischen Region von Interesse in dem Mausstamm, die für die Mikroinjektion verwendet, um mögliche Einzel-Nukleotid-Polymorphismen (SNPs), die nicht für die in öffentlichen Datenbanken berücksichtigt werden könnten, und erkennen werden könnte verhindern Nuklease verbindlich.
- Optional: Siehe Tabelle 3 für weitere Designer-Nukleasen bezogenen Online-Ressourcen.
- Die meisten Plasmide für die ZFN und TALEN Bau erforderlich ist, kann von Addgene erhalten:
http://www.addgene.org/special-collections/
Addgene-IDs für den Golden Gate TALEN Kit und Säugetier TALEN Expressionskonstrukte sind in Tabelle 2 angegeben.
Alternativ können bestimmte Funktionsmodule wie Zink-Finger-Domänen von einem Gen-Synthese Dienstleister bestellt werden.
2. Golden Gate TALEN Versammlung
Dieser Abschnitt beschreibt die Montage von Talens über ein von Cermak et al. 13. In voller Länge Talens werden mit zwei anschließenden Golden Gate Klonierungsschritten (Abbildung 1) konstruiert veröffentlichten Protokoll. Dieser Ansatz ermöglicht den Einbau einer beliebigen Anzahl von RVDS zwischen 12 und 31 in die endgültige Expressionskonstrukte. Die Montage-Protokoll hat zum Ziel Vektoren für die Erzeugung von mRNAs (1C), die sehr aktiv Talens folgenden Eizelle Mikroinjektion auszudrücken entworfen angepasst. Sie in der Online-Protokoll der Golden Gate TALEN Kit für den Aufbau und die Aufrechterhaltung der Plasmid-Bibliothek (siehe auch http://www.addgene.org/TALeffector/goldengateV2/ ).
- Tag 1 - Golden Gate Reaktion # 1 - Versammlung der RVD-Arrays in PFUS Vektoren
- Optional: Für jedes Paar in TALEN P entwickeltrotocol ein, geben Sie die DNA-Sequenz der beiden Zielstellen (einschließlich der Anfangs T, 5 'nach 3') in die TALEN_Voytas_Pipetting Tabellenkalkulation, ein Pipettierschema für jeden Golden Gate Reaktion erzeugen (auch geben Konzentration aller für die Baugruppen verwendeten Plasmide ). Die Tabellenkalkulation bietet auch die erwartete DNA-Sequenz aller RVDS, die zur Ausrichtung der Sequenzierungsergebnisse in Abschnitt 2.5.2 verwendet werden können.
- Für jeden einzigartigen TALEN entworfen in Protokoll Nr. 1:
Wenn TALEN Länge beträgt 12-21 (Standard), wählen Sie Wiederholungsvariable di-Rückstände (RVDS) 1-10 und Zielvektor pFUS_A. Wählen restlichen RVDS und Ziel pFUS_B einen Vektor, zB für eine TALEN mit 15 RVDS (einschließlich der letzten Wiederholung, die bis zur Endmontage in Abschnitt 2.3.3 hinzugefügt werden, wählen RVDS 11-14 und Zielvektor pFUS_B4 (Abbildung 1A).
Wenn TALEN Länge beträgt 22-31, Verwendung RVDS 1-10 und Ziel vector pFUS_A30A. Wählen RVDS 11-20 und Zielvektor pFUS_A30B. Wählen restlichen RVDS und pFUS_Bdestination Vektor, z. B. für TALEN mit 24 RVDS holen RVDS 21-23 und Zielvektor pFUS_B3. - Richten Sie Golden Gate Reaktion Nr. 1 für jeweils PFUS + RVD Kombination separat, dh 1-10 + pFUS_A und Rest RVDS + jeweiligen pFUS_B Vektor oder für länger als 21 TALEN RVDS, 1-10 + pFUS_A30A, 11-20 pFUS_A30B und Rest RVDS + jeweiligen pFUS_B Vektor. Verwenden 150 ng jedes RVD Vektor, 150 ng Vektor PFUS, 1 ul BsaI, 1 ul T4-DNA-Ligase, 2 ul 10fach T4 DNA-Ligase-Puffer und H 2 O zu 20 &mgr; l Reaktionsvolumen. (Mit frischen Aliquots von T4-Ligase-Puffer für jede Runde des Golden Gate Baugruppen wird empfohlen, da mehrmaliges Auftauen / Einfrieren von T4-Ligase-Puffer kann die Effizienz der Reaktionen zu reduzieren.)
- Zeigen Golden Gate Reaktionen in einem Thermocycler.
Programm:
37 ° C 5 min
16 ° C 10 min
10 Zyklen
50 ° C 5 min
80 ° C 5 min - 1 ul 10 mM ATP und 1 &mgr; l Plasmid-Safe-Nuklease zu jeder Mischung und Inkubation bei 37 ° C für 1 Stunde. Diese Behandlung wird linear unvollständige Ligationsprodukte, die durch in vivo homologe Rekombination in transformierten Bakterien in die Zielvektoren kloniert werden könnten, zu entfernen.
- Trans E. coli mit den einzelnen Ligationsreaktionen (elektro oder chemisch kompetente E. coli, die α-Komplementierung zu ermöglichen, wie XL1-Blue oder DH5 &agr; verwendet werden hier und in den nachfolgenden Transformationen).
- Teller Bakterien auf Spectinomycin (50 ug / ml)-Platten mit X-Gal und IPTG (40 ug / ml) für blau / weiß Kolonie Auswahl.
- Tag 2 - Bestätigung der Richtige PFUS-RVDS Versammlung
- Durch die Verwendung von Kolonie-PCR mit Primern pCR8_F1 und pCR8_R1 (siehe Tabelle 1 für die Primer-Sequenzen) 1-3 Bildschirm weißKolonien von jeder Platte. Richtig PFUS-RVDS Baugruppen zeigen typischerweise eine Bande, die dem Gesamtlänge aller RVDS kloniert (zB rund 1,1 kb für 10 RVDS) und eine "Leiter" der kleineren, weniger prominenten Bands (Abbildung 1B).
PCR-Programm:
95 ° C 3 min
95 ° C 30 s
55 ° C 30 s
72 ° C 1 min 45 sec
30-35 Zyklen
72 ° C 10 min - Verwenden bestätigt Klone über Nacht-Kultur (2-5 ml LB mit 50 ug / ml Spectinomycin) starten.
- Durch die Verwendung von Kolonie-PCR mit Primern pCR8_F1 und pCR8_R1 (siehe Tabelle 1 für die Primer-Sequenzen) 1-3 Bildschirm weißKolonien von jeder Platte. Richtig PFUS-RVDS Baugruppen zeigen typischerweise eine Bande, die dem Gesamtlänge aller RVDS kloniert (zB rund 1,1 kb für 10 RVDS) und eine "Leiter" der kleineren, weniger prominenten Bands (Abbildung 1B).
- Tag 3 - Golden Gate Reaktion # 2 - RVD Arrays in TALEN Expressionsvektoren
- Perform "Minipreps", um PFUS RVD-Baugruppen zu isolieren (je nach Anzahl der RVDS entweder pFUS_A und pFUS_B oder pFUS_A30A, pFUS_A30B und pFUS_B).
- Optional: Sequence einzelnen PFUS Vektoren unter Verwendung der Primer pCR8_F1, pCR8_R1 (siehe Tabelle 1 für die Primer-Sequenzen). Sequenzierung können auch auf f durchgeführt werdeninal TALEN Konstrukte (Abschnitt 2.5.2); jedoch für längere Talens komplette liest aller RVDS vielleicht nicht möglich sein, mit Sanger-Sequenzierung.
- Richten Sie Golden Gate Reaktion Nr. 2 für jeden einzelnen TALEN. 150 ng von jedem PFUS Vektor, 150 ng der PLR-HD, PLR-NG, PLR-NI, PLR-NN (letzte "halbe-repeat") nach dem Entwurf des RVD-Sequenz und für die l inks TALEN verwenden 75 ng von pCAG-T7-TALEN-ELD-Destination und für das Recht TALEN verwenden 75 ng pCAG-T7-TALEN-KKR-Ziel (oder umgekehrt). 1 ul Esp3I, 1 ul T4-DNA-Ligase, 2 ul 10fach T4 DNA-Ligase-Puffer H 2 O auf 20 &mgr; l Reaktionsvolumen.
- Zeigen Golden Gate Reaktionen in einem Thermocycler.
Programm:
37 ° C 5 min
16 ° C 10 min
10 Zyklen
37 ° C 15 min
80 ° C 5 min - Verwenden Sie Reaktionen von Abschnitt 2.3.4 zu verwandeln E. coli.
- Tag 4 - Bestätigung der Richtige TALEN Versammlung
- Bildschirm 1-3 weiße Kolonien von jeder Platte durch Kolonie-PCR mit Primern TAL_F1 und TAL_R2 (siehe Tabelle 1 für die Primer-Sequenzen). Richtig zusammengesetzt Talens zeigen ein PCR-Produkt mit einer Länge auf die Gesamtzahl der einge RVDS (1D, diese Band ist manchmal schwer zu erkennen, während die "Leitereffekt" stellt eine robuste Indikator für die erfolgreiche Montage) entspricht.
PCR-Programm:
95 ° C 3 min
95 ° C 30 s
55 ° C 30 s
72 ° C 3 min
30-35 Zyklen
72 ° C 10 min - Verwenden bestätigt korrekte Klone über Nacht Bakterienkulturen (2-5 ml LB mit 100 ug / ml Ampicillin) starten.
- Bildschirm 1-3 weiße Kolonien von jeder Platte durch Kolonie-PCR mit Primern TAL_F1 und TAL_R2 (siehe Tabelle 1 für die Primer-Sequenzen). Richtig zusammengesetzt Talens zeigen ein PCR-Produkt mit einer Länge auf die Gesamtzahl der einge RVDS (1D, diese Band ist manchmal schwer zu erkennen, während die "Leitereffekt" stellt eine robuste Indikator für die erfolgreiche Montage) entspricht.
- Tag 5 - Bestätigung der Richtige TALEN Versammlung
- Perform "Minipreps", um pCAG-T7-TALEN Plasmide zu isolieren.
- Wenn Sequenzierung wurde nicht durchgeführt section 2.3.2 Verwendung TAL_Seq_5 Primer-1 und TAL_R2 (siehe Tabelle 1 für die Primer-Sequenzen), um die korrekte Montage RVD in voller Länge TALEN bestimmen.
3. Nuclease-mRNA-Synthese
- Erzeugung von DNA-Matrize
- Bereiten hochwertigen midi oder Maxipreps von pCAG-T7-TALEN Plasmide für die mRNA-Synthese.
- Linearisieren 10 ug des TALEN oder ZFN mRNA-Synthese Plasmid mit einem Restriktionsenzym, vorzugsweise spaltet stromabwärts und in enger Nachbarschaft zu der Nuklease STOP-Codon (zum pCAG-T7-TALEN Vektoren verwenden SacI). mRNA-Synthese Plasmide umfassen typischerweise einen T7-oder SP6-Phagen-Promotor stromaufwärts der Nuklease-codierenden Sequenz.
- Führen 200-500 ng des verdauten Plasmid auf 0,7-1% Agarose-Gel für eine vollständige Verdauung zu überprüfen.
- Entfernung von Salzen aus dem Plasmid durch Verdauung Ausfällen der DNA mit 0,1 Volumen Natriumacetat und 3 Volumina Ethanol für 1 h bei RaumtemperaturE. Pellet die DNA durch Zentrifugation bei 14.000 × g oder mehr für 10 Minuten, waschen Sie die Pellets mit 200 ul 70% Ethanol, Spin weitere 5 Minuten, entfernen Sie das Ethanol, das Pellet an der Luft trocknen und resuspendieren in einem geeigneten Volumen RNase-freies Wasser. Reinigung des linearisierten Plasmids ist auch möglich, unter Verwendung einer Säule basierten System, zB QIAquick PCR Purification Kit.
- Bestimmen Sie die Konzentration des linearen Vorlage verwenden und 1 ug in-vitro-Transkription zu setzen.
- mRNA-Synthese und Polyadenylierung
- Für die in vitro Transkription pCAG-T7-TALEN Plasmide verwenden mMessagemMachine T7 Ultra-Kit. 20 ul mit Nuklease-freies Wasser, 10 ul T7 2x NTP / ARCA, 2 ul 10x T7 Reaktionspuffer, 1 ug DNA-Vorlage, 2 ul T7 Enzym-Mix: die Transkriptionsreaktion für jede TALEN auf Eis gesetzt. Mischen Sie die Reaktion und Inkubation für 1-2 Stunden bei 37 ° C
- Verwenden Sie die komplette 20 ul Transkriptionstion Reaktionsmischung zum Einrichten der Polyadenylierung Reaktion auf Eis: 36 ul Nuklease-freies Wasser, 20 ul 5x EPAP-Puffer, 10 ul 25 mM MnCl2, 10 ul 10 mM ATP. Mischen Sie und entfernen 2,5 ul des Reaktionsgemisches als Kontrollproben L1 und R1 (für links und rechts Nuklease, beziehungsweise). Hinzufügen von 4 ul E-PAP Enzym und Inkubation der Reaktion für 45-60 min bei 37 ° C. Entfernen weitere 2,5 ul Reaktionsmischung als Kontrollproben L2 und R2.
- mRNA Purification
- Verwenden NucAway Spin-Säulen für die mRNA-Reinigung (Puffer Austausch und Entfernung der eingebauten Nukleotide). Tippen Sie auf Spalten, um trockene Gel in der Spalte unten absetzen. Hydrat Säule mit 650 &mgr; l RNase-freies Mikroinjektionspuffer (1 mM Tris-Cl, 0,1 EDTA, pH 7,5). Cap-, Vortex-, klopfen Sie Luftblasen, und Hydrat für 5-15 Minuten bei Raumtemperatur.
- Legen Sie die Spalte in einem Sammelrohr und Spinbei 750 × g, 4 ° C für 2 min, um überschüssiges interstitiellen Flüssigkeit zu entfernen. Entsorgen Sammelrohr und Ort Spalte in einem 1,5 ml Elution Röhre.
- Bewerben komplette Reaktionsgemisch aus dem Abschnitt 3.2.2 zu Säule und Spin bei 750 g, 4 ° C für 2 min. Nuklease-mRNA wird nun in Mikroinjektion Puffer gelöst werden. Entfernen 2,5 ul gereinigten Proben L3 und R3.
- Speicher mRNA bei -80 ° C bis Mikroinjektion Aliquots hergestellt.
- mRNA-Gel-Elektrophorese
- Reinigen ein Gel Kammer zu RNase-Kontaminationen zu entfernen mit 10% SDS-Lösung oder RNaseZAP.
- Vorbereitung einer 1% igen Agarosegel in 1xTBE Laufpuffer.
- Mischen Sie jede RNA-Probe L1/R1, L2/R2, L3/R3 mit 3 Volumina NorthernMax Formaldehyd laden Dye und Inkubation für 15 min bei 65 ° C
- Laden Sie die Proben und ein RNA-Größenleiter (z. B. Millennium RNA-Größenmarker) auf dem Gelund führen Gel bei 10 V / cm in 1x TBE bis Ladefarbstoff das Ende des Gels erreicht hat.
- Fleck das Gel mit SYBR Grün-Lösung (Invitrogen) für 30-60 min unter Rühren bei Raumtemperatur.
- Bild das Gel und vergleichen Größen mit L1/R1 L2/R2 und L3/R3. Proben L2/R2 und L3/R3 die Bänder eines erhöhten Größe relativ zu zeigen, was eine erfolgreiche L1/R1 Polyadenylierung in (Abbildung 2).
- Bestimmen Sie mRNA-Konzentration mit einem Spektralphotometer.
- Aliquote herzustellen mRNA für Mikroinjektionen durch Mischen linken und rechten Nuklease Nuklease-mRNA im Verhältnis 1:1. Wir empfehlen Herstellung Aliquots mit einer Gesamtkonzentration von 200 ng / &mgr; l (100 ng / &mgr; l jeder Nuklease) durch Verdünnen mit Mikroinjektionspuffer. Speicher mRNA Aliquots bei -80 ° C.
4. Embryo Isolierung und Mikroinjektion
- Embryo Isolation. Dieses Protokoll wurde erfolgreich mit C57BL/6J und BDF1 verwendete Stamms und kann am ehesten zu anderen Stämmen häufig in Mikroinjektionsexperimente wie FVB oder CBF1 verwendet angepasst werden. Die Mäuse werden unter einem 12 h-12 h Licht-Dunkel-Zyklus in einer temperatur-und feuchtigkeitsgesteuerten Anlage mit Futter und Wasser ad libitum untergebracht.
- Superovulation Spenderweibchen, die Ausbeute an Embryonen zu erhöhen. 16 Frauen (4 Wochen alt) werden durch intraperitoneale (ip) Injektion von Serum Gonadotropin 5 IU schwangeren Stuten (PMSG), gefolgt von der ip-Injektion von 5 IU menschliches Choriongonadotropin (hCG) 48 Std. später superovuliert.
- Kamerad die 16 superovulierten Weibchen zu 16 Jahren Zucht (2-8 Monate) Männchen unmittelbar nach der hCG-Injektion.
- Opfere die Weibchen mit einem zugelassenen Euthanasie-Protokoll wie CO 2-Inhalation.
- Recover die befruchteten Eizellen. Die Eizellen werden von Eileitern am nächsten Morgen gesammelt und dann durch eine Behandlung 3-5 min in 0,1% Rinderhyaluronidase in M2 Medium gelöst von allen übrigen Cumuluszellen befreit. Abhängig von der Paarungsverhalten und der Dehnung verwendet der Embryo Ausbeute variieren. 16 C57BL/6J Weibchen produzieren in der Regel 150 bis 250, während BDF1 ergeben 300-400 befruchtete Eizellen auf.
- Embryo-Mikroinjektion
- Spritzen Sie die Nuklease mRNA. Vorkernstadium Oozyten werden in der Regel am frühen Nachmittag injiziert. Zytoplasmatische mRNA Mikroinjektion wird in M2-Medium unter Mineralöl mit einem inversen Mikroskop mit Nomarski DIC mit 20x-Objektiv und Embryo-Mikromanipulatoren und einer Einspritzeinheit ausgestattet ist. Wir empfehlen Beginn der Injektionen TALEN mRNA in einer Konzentration von 10 ng / &mgr; l (20 ng / &mgr; l insgesamt).
- Saugt den Oozyten mit einer Halte Kapillare und spritzen die Nuklease-mRNA im Cytoplasma der Embryo der Kontakt mit der Vorkerne. Die Injektion sollte flache, in der Nähe der Plasmamembran sein. Das Injektionsvolumen sollte niedrig gehalten werden und die Mikroinjektionsnadel sollte Witz seinhdrawn bei den ersten Anzeichen von zytoplasmatischen Spannungsgefühl. Eine Mikroinjektion Serie besteht normalerweise aus 100 bis 300 Eizellen. Typischerweise sollten mindestens 80-90% der Embryonen die Injektion ohne sofortige Lyse überleben.
- Nach Mikroinjektion, legen Sie die Embryonen für 1 Stunde in M16 (Sigma)-Medium bei 37 ° C und 5% CO 2 und übertragen Sie die überlebenden Embryonen in pseudoPflegeMütter.
5. Chirurgische Embryo Transfer
- Bereiten schein Embryo Empfänger (Pflegemütter) einen Tag vor der mRNA-Mikroinjektion. Ältere Weibchen (3-6 Monate alt) aus einem robusten outbred Belastung, wie CD-1, sind gut für die Rolle geeignet. Induzieren Scheinträchtigkeit durch Paarung die Weibchen auf einem früheren Tag mit entweder chirurgisch oder genetisch vasektomierten Männer 28. Nur 0,5 dpc Frauen mit einem klaren copulatory Stecker werden als Embryonen-Empfänger verwendet. In ungenutzten Frauen verschwindet die Scheinträchtigkeit nach ca. 3 weeks so dass ihre mehrfache Verwendung.
- Betäuben die 0,5 dpc Pflegemutter Weibchen durch ip Injektion von Ketamin, Xylazin (120 mg / kg und 16 mg / kg). Diese Formulierung Garantien ~ 30 min chirurgischen Toleranzzeit ist mehr als ausreichend für die geplante Betriebszeit von 5-10 min.
- Positionieren Sie den narkotisierten Tier auf seinen Bauch auf einer warmen Oberfläche und desinfizieren den Bereich der Schnitt mit einem geeigneten Desinfektionsmittel wie Ethanol oder 70% Chlorhexidin und Alkohol-Gemisch.
- Inzision der Haut auf dem Rücken des Tieres und öffnen Sie die Bauchhöhle mit einer sterilen Schere.
- Visualisieren und externalisieren die Uterushorn durch Ziehen an der Fettpolster an den Eierstöcken befestigt. Halten Sie die Organe feucht mit warmen 0,9% NaCl-Lösung. Beachten Sie, dass die Operationsstelle, kann mit sterilen Verband oder drapiert, um den lokalen Veterinär Richtlinien entsprechen rasiert werden.
- Immobilisieren, die Fortpflanzungsorgane von Clipping den Eierstock Fettpolster mit einer Bulldogge Klemme.
- Fassen Sie den Eileiter kurz vor der Ampulle mit Uhrmacher Pinzette und verwenden Sie eine 30 G Injektionsnadel, ein kleines Loch in der Wand des Eileiters erstellen.
- Ziehen Sie die Nadel und setzen Sie eine dünne Glaskapillare, die die Embryonen in das Loch so dass die Platzierung der Embryonen in der Ampulle durch leichtes Blasen in das Mundstück des Kapillarenhalter. Ziehen Sie die Kapillare einmal die Embryonen in der Ampulle abgeschieden und ersetzen die Fortpflanzungsorgane wieder in der Körperhöhle.
- Schließen der Bauchhöhle mit einer Reihe von sterilen Nahtmaterialien (Prolene 6-0).
- Schließen der Haut mit 1-2 Wundklammern (Autoclip 9 mm) in Abhängigkeit von der Größe der Öffnung.
- Nach dem Betrieb zurück, die Tiere in ihre Heimat Käfig und betreuen sie, bis die Wirkung der Narkose nachlässt.
- Um Stress zu minimieren, Haus der Mäuse in stabilen sozialen Gruppen (2-4 Tiere / Typ-III-Käfig), wenn möglich.
- Bewerben postoperative analgetisches in Form Dafalgan hinzugefügt Trinkwasser (Paracetamol 200 mg / kg Körpergewicht) für 3 Tage nach der Operation.
6. Analyse der Gründer von PCR-und T7-Endonuklease oder Restriktionsenzymverdau
- Design-Primer, um einen Bereich zwischen 200 bis 700 bp um die Nuklease Bindungsstelle zu verstärken. Die Abstände Vorwärtsprimer-Nuklease Spacerregion und Nuklease-Spacer-Bereich-Reverse-Primer müssen ausreichend verschieden, um den Nachweis von zwei separaten Bändern für verdauten Produkte auf einem Agarose-Gel (siehe Fig. 3 und 4 für Beispiele) sein.
- Führen PCR mit optimierten Bedingungen. Überprüfen PCR Amplikon Größe auf einem Agarose-Gel.
- Optional: Entschlacken PCR-Produkte, z. B. mit QIAquick PCR Purification Kit, um Salze und Nukleotide aus dem PCR-Mix zu entfernen. Viele Restriktionsenzyme sind in PCR-Proben mit dem geeigneten Restriktionsenzym-Puffer und Reinigung ergänzt aktiv ist nicht erforderlich. Wir haben auch successfully verwendet T7-Endonuklease in QiagenTaq PCR-Puffer mit NEBuffer 2 ergänzt.
- T7-Endonuklease-Assays. Mischen Sie 17 ul PCR-Produkt mit 2 ul NEBuffer 2 und laufen Heteroduplexbildung (3b)-Programm in einem Thermocycler:
95 ° C 2 min
95 ° C bis 85 ° C (2 ° C / sec)
85 ° C bis 25 ° C (-0,1 ° C / sec)
4 ° C halten.
1 &mgr; l T7-Endonuklease zu jeder Probe und inkubiere bei 37 ° C für 20 min. (Der Vermesser-Assay (Transgenomics), die auf ein CEL Nuklease beruht, können auch hier benutzt werden. Bitte beachten Sie die Anweisungen des Herstellers beziehen.) - Restriktionsverdau des PCR-Produkts. Mischungs 17 ul PCR-Produkt mit 2 ul 10x Restriktionsenzym-Puffer und 1 ul Restriktionsenzym. Digest unter geeigneten Temperatur für 1 h oder mehr.
- T7-Endonuklease-Assays. Mischen Sie 17 ul PCR-Produkt mit 2 ul NEBuffer 2 und laufen Heteroduplexbildung (3b)-Programm in einem Thermocycler:
- In DNA-Ladefarbstoff Proben und auf einem 2% Agarose-Gel laufen, um unterschiedliche Verdauung erkennenMuster für Wildtyp-Tiere und Gründer tragen mutierte Allele. Siehe Abbildungen 3 und 4 für den erwarteten Ergebnissen.
- Klon-PCR-Produkte von Gründern positiv für mutierte Allele für die Sanger-Sequenzierung (zB durch TA-Klonierung in pGEM-T Easyor direkt zu sequenzieren Mischungen von PCR-Produkten mit der nächsten Generation Sequenzierung.
Ergebnisse
Wir konstruierten Ziel Plasmide mit dem Goldenen GateTALEN Anordnung nach Cermak et al. 13, der die Expression in Säugetierzellen TALENS als auch in vitro-mRNA-Synthese aus dem T7-Phagen-Promotor (Fig. 1C) zu ermöglichen veröffentlicht kompatibel. Diese Plasmide tragen heterodimeren Fokl Domains (ELD oder KKR Mutationen), die gezeigt haben, um Off-Target-Effekte in Bezug auf Fokl Homodimere zu reduzieren und die Spaltungsaktivität verbessern im Vergleich zu der ersten Generation Fokl Heterodimere 25,29. Golden Gate Anordnung Reaktionen Nr. 1 und Nr. 2 sind in der Regel sehr effizient, und jedes weiße Kolonie, wenn durch Kolonie-PCR analysiert, zeigt das erwartete Muster für die bestimmte Anzahl von RVDS kloniert (Fig. 1B und 1D).
Gel-Analyse der in vitro synthetisierten mRNA (Abbildung 2) sollte eine einzelne unterscheidbar Band mit wenig oder ohne Abstrich für jede Probe analysiert offenbaren. Es sollte eine klare Größe Verschiebung zwischen Proben und L1/R1 L2/R2, L3/R3, die erfolgreiche Polyadenylierung zeigt sein.
Gründer-Tiere können beispiels NHEJ-induzierten mutierten Allele Genotypisierung mit PCR, gefolgt von entweder T7 Endonuklease-Verdauung (Fig. 3) oder einen Restriktionsverdau unter Verwendung eines Enzyms untersucht werden, dass spaltet die Wildtyp-Sequenz in der Spacer-Region der Nuklease Paar (Abbildung 4). Die T7-Endonuklease Assay ist auf jede Art von Mutation unabhängig von der speziellen genomische Sequenz innerhalb der Spacer-Region der Nuklease Paar injiziert wird; jedoch erkennt nur Fehlanpassungen zwischen DNA-Strängen in Heteroduplex-PCR-Produkte. So wird in dem seltenen Fall, dass einer der Gründer trägt zwei identisch mutierten Allele, PCR-Produkte würden keine T7 Verdauungsmuster zeigen. Solche Unterscheidung ist jedoch immer möglich, wenn eine spezifische Restriktionsstelle innerhalb der Nuklease Spacerregion, die von NHEJ eliminiert werden entferntInduzierten Insertionen / Deletionen (Abbildung 5). Hier unverdaute Banden weisen auf das Vorhandensein von Mutationen und das Fehlen von Abbauprodukte stark darauf eine gegründet, die Mutationen in beiden Allelen des Zielgens (mit Sternchen in Fig. 4b dargestellt).
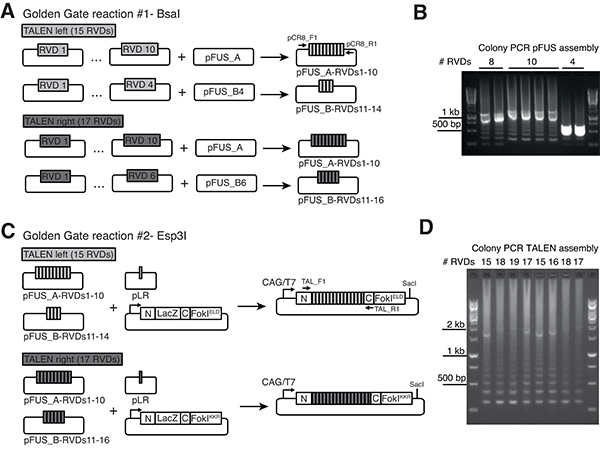
Fig. 1 ist. Golden Gate Klonen von TALEN RVDS in heterodimeren pCAG-T7-Destination-Vektoren. A) Versammlung der RVD Arrays in PFUS Vektoren. Hier ein Beispiel für ein Paar mit individuellen TALEN TALE-Arrays mit 15 und 17 RVDS RVDS jeweils dargestellt (für TALE Arrays länger als 21 RVDS werden drei PFUS-RVDS Baugruppen erforderlich ist, nicht gezeigt). Die Pfeile zeigen die Primer für PFUS spezifischen Kolonie-PCR-Reaktionen. B) PCR-Produktevon der richtigen PFUS Baugruppen verstärkt zeigen typischerweise eine Band, die der Gesamtlänge aller RVDS kloniert (zB rund 1,1 kb für 10 RVDS) und eine "Leiter" der kleineren, weniger prominenten Bands aufgrund der repetitiven Natur der RVD-Arrays. C) Endmontage von PFUS-RVD-Arrays und einem Plasmid, das die letzte Wiederholung (PLR) in heterodimeren TALEN Expressionsvektoren mit FokIELD und FokIKKR Varianten auf. Die TALEN Rückgrat (wie N-und C-kommentierten) ähnelt dem von Miller et al. 23 veröffentlicht Architektur Die CAG (frühen CMV-Enhancer-Element / Huhn-beta-Aktin)-Promotor für hohe Expressionsniveaus in transfizierten Säugerzellen, während der T7-Phagen-Promotor kann in vitro-mRNA-Synthese (Verwendung SacI für die Linearisierung des Vektors stromabwärts von der Nuklease STOP-Codon). D) Kolonie-PCR unter Verwendung von Primern durch Pfeile in C angegeben) ermöglicht die Identifizierung korrekt montiert TALENS. Voll length PCR-Produkte sind oft weniger prominent, während die "Leitereffekt" stellt eine robuste Indikator für die erfolgreiche Montage. Klicken Sie hier für eine größere Ansicht .
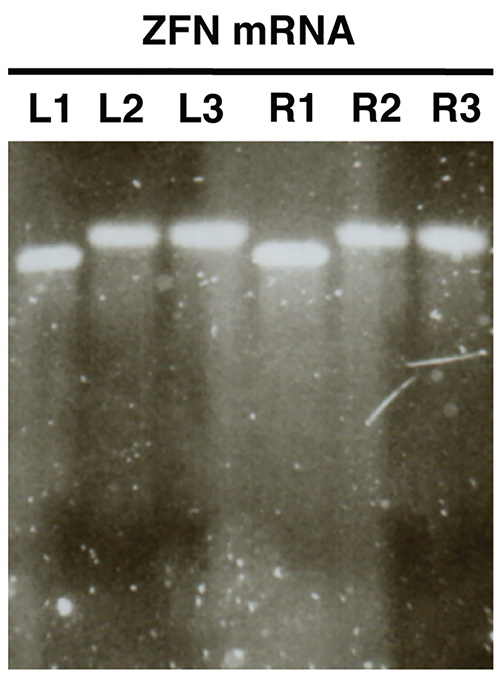
2. . Qualitätskontrolle von Nuklease-mRNA in vitro-Synthese unter Verwendung von Agarose-Gelelektrophorese ZFN mRNAs werden als Beispiel gezeigt (L, linke ZFN, R rechten ZFN). Proben L1/R1 mRNA vor Polyadenylierung zeigen, Proben L2/R2 Show polyadenyliert mRNA und L3/R3 zeigen gereinigte polyadenylierten mRNA. Klicken Sie hier für eine größere Ansicht .
alt = "3" fo: content-width = "6in" fo: src = "/ files/ftp_upload/50930/50930fig3highres.jpg" src = "/ files/ftp_upload/50930/50930fig3.jpg" width = "600" />
3. Beispiel für einen T7-Endonuklease-Assay zur Identifizierung von Gründertieren Durchführung Nuklease-induzierten Mutationen des Ziel-Locus verwendet werden. A) Eine TALEN Paar wurde entwickelt, um innerhalb der kodierenden Region des Maus-Prionprotein-Gen (Prnp spalten kann TALEN Zielsequenz auf Anfrage zur Verfügung gestellt werden). Ein PCR-Produkt wird mit einem Vorwärtsprimer (F) liegt 110 bp stromaufwärts und ein Rückwärts-Primer (R) 250 bp stromabwärts der Spaltstelle TALEN. B) Das PCR-Produkt wird anschließend Bildung ausgesetzt und T7-Endonuklease-Verdauung erzeugten Heteroduplex. C) TALEN-induzierte Mutagenese innerhalb der genomischen Region des Zieleinzel Gründer wird durch die Anwesenheit von einer Volllängen-PCR-Produkts mit Produkten des Verdaus von 250 und 110 bp ergab.50930/50930fig3highres.jpg "target =" _blank "> Klicken Sie hier für eine größere Ansicht.

4. Beispiel für einen Restriktionsverdau zur Identifizierung der Tiere, die Gründer Nuklease-induzierte Mutationen des Zielorts. ZFN spezifisch für die Maus Rosa26 Locus 29 Ziel eine XbaI-Schnittstelle in Intron 1 verwendeten PCR-Produkte. A) Gründer wurden durch PCR Genotypisierung geschirmten ein Vorwärtsprimer (F) 500 bp stromaufwärts und ein Rückwärts-Primer (R) 250 bp stromabwärts der Spaltstelle. B) Verdau der PCR-Produkte mit XbaI Verdau zeigt Muster, die Mäuse, die mit bi-allelische Mutationen (markiert mit Sternchen), mono -Allel-Mutationen (verdaut und unverdaute Bands, z. B. Tier 2)und wt-Mäuse (vollständige Verdauung, zB Tier 21). C) Sequenzierung von Mäusen mit Potential bi-allelische Modifikationen zeigt bis zu 3 (Tier 24) verschiedene Insertionen / Deletionen. Klicken Sie hier für eine größere Ansicht .
| Name der Primer | Sequenz 5 'zu 3' |
| pCR8_F1 | ttgatgcctggcagttccct |
| pCR8_R1 | cgaaccgaacaggcttatgt |
| TAL_F1 | ttggcgtcggcaaacagtgg |
| TAL_R2 | ggcgacgaggtggtcgttgg |
| TAL_Seq_5-1 | catcgcgcaatgcactgac |
Tabelle 1. Sequenzen von Primern für die Kolonie PCR und Sequenzierung innerhalb der Golden Gate TALEN Montage verwendetProtokoll.
| Plasmid / Sammlung | Anbieter | Addgene ID | Kommentare |
| Golden Gate TALEN und TAL-Effektor Kit 2.0 | Voytas Labor | 1000000024 | Enthält alle Plasmide für die Golden Gate TALEN Montage notwendig |
| pCAG-T7-TALEN -KKR/ELD Zielvektoren | Pelczar Labor | 40131, 40132 | Add-on-Plasmide für TALEN Expression in Säugerzellen und in vitro-mRNA-Synthese |
Tabelle 2. Plasmide und Plasmid-Kollektionen für Golden Gate TALEN Montage erforderlich kann von Addgene (erhältlich www.addgene.org ).
| OnlineRessource | Kommentare |
| http://tale-nt.cac.cornell.edu | Design TALEN; TALEN Off-Target-Vorhersage |
| http://zifit.partners.org/ZiFiT/ | Design TALEN, OPEN ZFN, CoDA ZFN, CRISPR/Cas9 |
| http://www.genome-engineering.org | Design TALEN, CRISPR/Cas9; CRISPR/Cas9 Off-Target-Vorhersage |
| http://baolab.bme.gatech.edu/Research/BioinformaticTools/assembleTALSequences.html | Versammlung der TALEN Sequenzen für die Bestätigung der Sequenzierungsergebnisse |
| http://pgfe.umassmed.edu/ZFPmo dularsearchV2.html | Gestaltung von Baukasten ZFN |
| www.genomecenter.ucdavis.edu/s egallab / segallabsoftware | Gestaltung von Baukasten ZFN |
Tabelle 3. Online-Ressourcen für die Gestaltung von ZFN, TALEN und CRISPR/Cas9.
Diskussion
Designer Nuklease-driven Genom Bearbeitung Ansätze haben das Spektrum der Arten zugänglich gezielte Modifikationen der jeweiligen Genome 10,12 deutlich erweitert. Bei Mäusen, Gen-Targeting in ES-Zellen hat eine Standard-Technik seit über zwei Jahrzehnten; jedoch ist es schwierig, an ES-Zellen von anderen Spezies als der Maus anpassen bewährt, obwohl es gab einige jüngsten Erfolg in ES-Zellen der Ratte. Auch mit der Verfügbarkeit von "off-the-shelf"-Gen-Targeting Maus-ES-Zellklone von Konsortien wie EUCOMM, KOMP oder NorCOMM 3 Genom Bearbeitung von ZFN TALEN und bietet eine höhere Präzision und Flexibilität in Bezug auf das Spektrum der Veränderungen, die sein können, sofern in das Mausgenom eingeführt. Gründer Tiere, die Nuclease-vermittelten Mutationen scheinen sehr Keimbahn zuständigen 4-6,20,21, was nicht immer der Fall bei Chimären aus Blastozysteninjektion von ES-Zellen Ursprung sein. Somit wird in bestimmten Fällen die Mikroinjektion designer Nukleasen kann deutlich schneller Generation von neuen Mauslinien mit gezielten Genom-Modifikationen führen.
Die erfolgreiche Generierung von Knockout-Mäusen durch Injektion von ZFN und TALEN hängt zu einem großen Teil auf die Aktivität des injizierten Nuklease-Paar. TALENS wurde gezeigt, dass eine hohe Erfolgsrate bei der Ausrichtung einer Vielzahl von Genen in verschiedenen Organismen; Jedoch weisen neuere Studien, dass TALEN Bindung empfindlich Cytosin-Methylierung 30,31. Somit neu erzeugten Nuklease Paare, beispielsweise TALENS kloniert in pCAG T7-Vektoren, können vorübergehend in eine Maus-Zelllinie, wie NIH-3T3-oder Neuro transfizierenden -2a, die die Chromatinstruktur Zustand des Mausembryo zu einem gewissen Grad zu imitieren. Hier können Nuklease-Aktivität unter Verwendung des T7-Endonuklease-Assay oder einen Restriktionsverdau von PCR-Produkt, wie in Abschnitt 5 vor der mRNA-Synthese und die Mikroinjektion beschrieben geschätzt werden. Wir empfehlen Sequenzierung der genomischen Region von Interesse in der Hinsicht,ive Zelllinie und die Maus-Stamm für die Mikroinjektion Experimente verwendet.
In Maus-Zygoten werden verschiedene TALEN oder ZFN Paare optimal auf verschiedenen mRNA-Konzentrationen zu arbeiten und damit die optimale Arbeitskonzentration der Mikroinjektion Nuklease mRNA kann müssen experimentell ermittelt werden. Abhängig von der Nuklease Paar wird eine zu niedrige Konzentration in keiner Spaltung führen, während eine zu hohe Letalität in Embryo führen. Abhängig von der Nuklease Paar haben wir Erfolg mit Gesamt-mRNA-Konzentrationen so niedrig wie 2 ng / ul und so hoch wie 200 ug / ul. Diese Effekte sind nur schwer von Versuchen in der Zellkultur und die Nuklease eine optimale Konzentration für sowohl Embryoüberlebensrate und Veränderung des Zielorts muß empirisch bestimmt werden, vorherzusagen.
Hochaktive ZFN oder TALEN können ihre Zielsequenz über die Ein-Zell-Stadium des Embryos mikroinjizierten spalten und damit zu komplexen Mustern der Mutagenese eind Mosaik in Gründer. Wir und andere 4 haben drei oder mehr verschiedene mutierte Allele in einer einzigen Gründer (5C) beobachtet. Wenn also zur Schaffung einer neuen Mauslinie aus diesen Gründern, Nachwuchs sollte sorgfältig durch Sequenzierung auf die Anwesenheit des günstigen Mutation, da die Verdauung Tests belegen, dass nur eine undefinierte Mutation vorhanden ist abgeschirmt werden.
Einer der Kritik häufig geäußerte gegen die ZFN und TALEN Systemen ist die Möglichkeit, dass diese Nukleasen sind auch zur Spaltung irgendwo anders im Genom vorhanden, die ähnlich zu den Zielstellen-Sequenzen sind. Solche Off-Target-Effekte wurden mit frühen Generation Reagenzien mit dem homodimeres Fokl Domäne beobachtet und Heterodimer Konstrukte wurden entwickelt, um Off-Target-Effekte 25 zu lindern. Potential off-Zielstellen in einem gewissen Ausmaß in silico vorhergesagten 32,33 und durch PCR und Sequenzierung gescreent werden. Ein offensichtlicher Vorteil of mit ZFNs Talens und zur Erzeugung von Mäusen und nicht-Zelllinien ist die Möglichkeit der Entfernung vom Ziel Mutationen nicht verknüpfte, um die gewünschten Änderungen durch Genom Durchführung mehrerer Rückkreuzungen zu einem Wildtyp-Stamm der Wahl. Für die Analyse einer großen Anzahl von Gründermäuse nächsten Generation tiefen Sequenzierung von PCR-Produkten aus der Nuklease geplanten Ortskurve erzeugt und in silico vorhergesagten Off-Target-Loci könnte eine alternative qualitative und quantitative Anzeige für die Verdauung des PCR-Assays Produkte.
Die in diesem Protokoll beschriebenen Techniken der assistierten Reproduktion sind für Standard-Mausstämme für die Mikroinjektion Experimente wie C57Bl/6J oder B6D2F1 verwendet optimiert. Mäuse unterschiedlicher Herkunft, wie outbred Stämme, kann im Prinzip für die Genom Bearbeitung Ansätze verwendet werden und vielleicht einen besser geeigneten genetischen Hintergrund für bestimmte Forschungsfragen bieten. Die Leistung der assistierten Reproduktionstechniken wie Superovulation kann Prädikat seinfür eine Reihe von Stämmen 34-36 cted könnte aber eine weitere Optimierung für Nicht-Standard-Stämme erfordern, um eine ausreichende Anzahl von Embryonen für die Nuklease Mikroinjektion erhalten.
Neben ZFN und TALEN haben neue Designer-Nukleasen wie die RNA-guided CRISPR/Cas9 System 9,37,38 jetzt für Genom-Editing-Anwendungen eingeführt. Alle Verfahren für die Mikroinjektion und Analyse der hier beschriebenen Gründertiere sind auch auf CRISPR/Cas9 und zukünftige Arten der Genom-Bearbeitung.
Offenlegungen
Die Autoren erklären, keine Interessenskonflikte.
Danksagungen
Wir möchten Monika Tarnowska, Cornelia Albrecht, und Ewa Skoczylas für hervorragende technische Unterstützung danken. Diese Studie wurde vom SNF Sinergia Zuschuss CRSI33-125073 PP finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| BsaI | NEB | R0535S or L | |
| Esp3I | Thermo Scientific | ER0451 | |
| T4 Ligase | NEB | M0202S or L | |
| Spectinomycin | Sigma | S0692-1ML | |
| Ampicillin | Sigma | A0166 | |
| X-Gal | Sigma | B4252 | |
| IPTG | Sigma | I6758 | |
| Plasmid-Safe nuclease | Epicentre | E3101K | |
| QIAprep Spin Miniprep Kit | Qiagen | 27106 | |
| QIAGEN Plasmid Midi Kit | Qiagen | 12143 | |
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| mMESSAGE mMACHINE T7 Ultra Kit | Invitrogen | AM1345 | |
| NucAway Spin Columns | Invitrogen | AM10070 | |
| RNaseZAP | Sigma | R2020-250ML | |
| NorthernMax Formaldyde Load Dye | Invitrogen | AM8552 | |
| RNA Millennium Markers | Invitrogen | AM7150 | |
| 10x TBE buffer | Thermo Scientific | B52 | |
| T7 endonuclease | NEB | M0302S or L | |
| pGEM-T EasyVector System I | Promega | A3600 | |
| SYBR Green I Nucleic Acid Gel Stain | Invitrogen | S-7563 | |
| pregnant mare's serum gonadotrophin (PMSG) | Sigma | G4877 | |
| human chorionic gonadotropin (hCG) | Sigma | CG5 | |
| M2 embryo culture medium | Sigma | M7167 | |
| M16 embryo culture medium | Sigma | M7292 | |
| Mineral oil, embryo tested | Sigma | M8410 | |
| Ketamine | CentraVet | Ket 201 | |
| Xylazine | Sigma Aldrich | 46995 | |
| Equipment/Tools | |||
| Inverted microscope with Nomarski DIC optics (for example Nikon Eclipse TE200) | Nikon | ||
| Micromanipulator units (for example Narishige, NT88NF) | Narishige | ||
| Embryo holding capillaries | Sutter Instruments | B100-75-10 | |
| Embryo injection capillaries | Narishige | GD-1 | |
| Capillary puller (for example Sutter P97) | Sutter Instruments | ||
| Microforge (for example Narishige MF-900) | Narishige | ||
| Walton skin scissors | FST | 14077-10 | |
| Surgical scissors | FST | 14041-10 | |
| Surgical probe | FST | 10140-03 | |
| Reflex wound clip system (9 mm) | FST | 12031-09 | |
| Reflex wound clips (9 mm) | FST | 12032-09 | |
| Dumont fine forceps 5 | FST | 11254-20 | |
| Moria curved forceps | FST | 11370-31 | |
| Moria fine forceps | FST | 11399-80 | |
| Dietrich bulldog clamp | FST | 18038-45 | |
| C57BL/6J mice | Jackson Labs | strain code 000664 | |
| CD-1 mice | Charles River | strain code 000664 | |
Referenzen
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat. Rev. Genetics. 6, 507-512 (2005).
- Johansson, T., et al. Building a zoo of mice for genetic analyses: a comprehensive protocol for the rapid generation of BAC transgenic mice. Genesis. 48, 264-280 (2010).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Carbery, I. D., et al. Targeted genome modification in mice using zinc-finger nucleases. Genetics. 186, 451-459 (2010).
- Meyer, M., de Angelis, M. H., Wurst, W., Kuhn, R. Gene targeting by homologous recombination in mouse zygotes mediated by zinc-finger nucleases. Proc. Natl. Acad. Sci. U.S.A. 107, 15022-15026 (2010).
- Cui, X., et al. Targeted integration in rat and mouse embryos with zinc-finger nucleases. Nat. Biotechnol. 29, 64-67 (2011).
- Sung, Y. H., et al. Knockout mice created by TALEN-mediated gene targeting. Nat. Biotechnol. 31, 23-24 (2013).
- Wefers, B., et al. Direct production of mouse disease models by embryo microinjection of TALENs and oligodeoxynucleotides. Proc. Natl. Acad. Sci. U.S.A. 110, 3782-3787 (2013).
- Wang, H., et al. One-Step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat. Rev. Genetics. 11, 636-646 (2010).
- ZFN, T. A. L. E. N. CRISPR/Cas-based methods for genome engineering. Trends Biotechnol. 31, 397-405 (2013).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nat. Rev. Mol. Cell Biol. 14, 49-55 (2013).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39, (2011).
- Reyon, D., et al. FLASH assembly of TALENs for high-throughput genome editing. Nat. Biotechnol. 30, 460-465 (2012).
- Sanjana, N. E., et al. A transcription activator-like effector toolbox for genome engineering. Nat. Protoc. 7, 171-192 (2012).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nat. Biotechnol. 31, 251-258 (2013).
- Schmid-Burgk, J. L., Schmidt, T., Kaiser, V., Honing, K., Hornung, V. A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes. Nat. Biotechnol. 31, 76-81 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 31, 827-832 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat. Biotechnol. 31, 822-826 (2013).
- Hermann, M., et al. Evaluation of OPEN zinc finger nucleases for direct gene targeting of the ROSA26 locus in mouse embryos. PLoS ONE. 7, (2012).
- Meyer, M., Ortiz, O., Hrabe de Angelis, M., Wurst, W., Kuhn, R. Modeling disease mutations by gene targeting in one-cell mouse embryos. Proc. Natl. Acad. Sci. U.S.A. 109, 9354-9359 (2012).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat. Biotechnol. 29, 143-148 (2011).
- Guo, J., Gaj, T., Barbas, C. F. Directed evolution of an enhanced and highly efficient FokI cleavage domain for zinc finger nucleases. J. Mol. Biol. 400, 96-107 (2010).
- Doyon, Y., et al. Enhancing zinc-finger-nuclease activity with improved obligate heterodimeric architectures. Nat. Methods. 8, 74-79 (2011).
- Wefers, B., et al. . Current Protocols in Mouse Biology. , (2011).
- Wefers, B., Wurst, W., Kühn, R. . Current Protocols in Mouse Biology. , (2011).
- Haueter, S., et al. Genetic vasectomy-overexpression of Prm1-EGFP fusion protein in elongating spermatids causes dominant male sterility in mice. Genesis. 48, 151-160 (2010).
- Perez-Pinera, P., Ousterout, D. G., Brown, M. T., Gersbach, C. A. Gene targeting to the ROSA26 locus directed by engineered zinc finger nucleases. Nucleic Acids Res. 40, 3741-3752 (2012).
- Bultmann, S., et al. Targeted transcriptional activation of silent oct4 pluripotency gene by combining designer TALEs and inhibition of epigenetic modifiers. Nucleic Acids Res. 40, 5368-5377 (2012).
- Valton, J., et al. Overcoming TALE DNA binding domain sensitivity to cytosine methylation. J. Biol. Chem. 287, 38427-38432 (2012).
- Cradick, T. J., Ambrosini, G., Iseli, C., Bucher, P., McCaffrey, A. P. ZFN-Site searches genomes for zinc finger nuclease target sites and off-target sites. BMC Bioinform. 12, (2011).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Byers, S. L., Payson, S. J., Taft, R. A. Performance of ten inbred mouse strains following assisted reproductive technologies (ARTs). Theriogenology. 65, 1716-1726 (2006).
- Luo, C., et al. Superovulation strategies for 6 commonly used mouse strains. J. Am. Assoc. Lab. Animal Sci. 50, 471-478 (2011).
- Pease, S., Saunders, T. L. . International Society for Transgenic Technologies. Advanced protocols for animal transgenesis : an ISTT manual. , (2011).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339, 819-823 (2013).
- Mali, P., et al. RNA-Guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten