Method Article
Souris ingénierie des génomes Utilisation nucléases de créateurs
Dans cet article
Résumé
nucléases de créateurs tels que nucléases à doigts de zinc (ZFN) et transcription nucléases effectrices activateur comme (Talens) peuvent être utilisés pour modifier le génome d'embryons de pré-implantation de souris en déclenchant à la fois la fin non homologue rejoindre (NHEJ) et la recombinaison homologue (RH) des voies. Ces progrès permettent la génération rapide de la souris avec des modifications génétiques précises.
Résumé
Les souris transgéniques portant des modifications spécifiques au site génome (knock-out, knock-in) sont d'une importance vitale pour disséquer les systèmes biologiques complexes ainsi que pour la modélisation des maladies humaines et tester des stratégies thérapeutiques. Les avancées récentes dans l'utilisation des nucléases de créateurs tels que nucléases à doigts de zinc (de ZFNs), transcription nucléases effectrices activateur comme (Talens), et les répétitions palindromiques courts régulièrement espacées cluster (CRISPR) / CRISPR-associé (Cas) 9 système pour le site- l'ingénierie des génomes spécifique ouvrir la possibilité d'effectuer la modification ciblée du génome rapide dans pratiquement toutes les espèces de laboratoire sans la nécessité de s'appuyer sur cellules souches embryonnaires (ES) de la technologie cellulaire. Une expérience d'édition de génome commence typiquement avec identification de concepteur nucléase sites cibles au sein d'un gène d'intérêt, suivi par la construction de domaines de liaison à l'ADN mesure de diriger l'activité nucléase de l'investigateur locus génomique définie. Plasmides designer de nucléase sont in vitro </ Em> transcrit pour produire l'ARNm pour la microinjection d'ovocytes fécondés de souris. Ici, nous fournissons un protocole pour la réalisation de la modification génomique ciblée par injection directe de TALEN ARNm dans des ovocytes de souris fécondés.
Introduction
Les souris sont de loin la plateforme la plus populaire pour générer des modèles animaux transgéniques. La boîte à outils polyvalente pour le génie génétique de l'embryon de souris 1-3 a récemment été étendu par des approches d'édition du génome sur la base de nucléases de créateurs tels que nucléases à doigts de zinc (ZFN) 4-6, transcription nucléases effectrices activateur comme (Talen) 7,8, et les répétitions groupées régulièrement espacées courts palindromiques (CRISPR) / système CRISPR-associé (Cas) 9 9. ZFN et la fonction TALEN par paires de deux domaines sur mesure à base de protéines liant l'ADN (arrays de protéines à doigts de zinc et répéter variable di-résidus (DRV), respectivement) qui sont couplés chacun à l'endonucléase Fokl 10-12. En revanche, la spécificité du clivage médié Cas9 d'ADN est fourni par transactiver ARN CRISPR (crRNA et tracrRNA, qui peuvent également être combinées en une seule molécule d'ARN chimérique d'ARN de guidage appelé) 11 qui agissent dans un complexe avec l'protéine de CRISPR.
TALENs avec une séquence définie de DRV peut être construit rapidement par les expérimentateurs individuels avec une multitude de stratégies de montage au choix de 13 à 17. CRISPR/Cas9 promet encore moins génération de main-d'œuvre des nucléases de créateurs, mais la spécificité de guidage ARN-ADN de liaison n'est pas encore complètement résolu 18,19. Génération de mesure ZFNs a jusqu'ici été limitée à des laboratoires universitaires spécialisés et de fournisseurs commerciaux tels que Sangamo Biosciences et le service Sigma CompoZr.
En général, génome édition avec des nucléases de créateurs vise à introduire des cassures double brin (DSB) à des loci génomiques défini, qui attirent ensuite fin non homologue rejoindre (NHEJ) ou recombinaison homologue (RH) des mécanismes de réparation d'ADN 10,12. Réparation NHEJ médiée par une DSB se traduit souvent par l'introduction d'insertions et de suppressions à proximité immédiate de l'emplacement de la réparation. Ainsi NHEJ réparation cun être exploitées pour assommer la fonction d'un gène cible par introduction d'une mutation décalage de cadre à l'intérieur de la gènes codant pour une protéine séquence 4,7,9. En variante, l'addition ou le remplacement de l'information génétique définie peuvent être atteints par la fourniture d'un donneur d'ADN avec les nucleases de luxe. Un donneur d'ADN comprend des séquences d'ADN enquêteur conçu flanquées de régions d'homologie avec le locus cible, servant ainsi de modèle pour la réparation des CDB par les RH. Les deux plasmides 5,6,20 et oligonucléotides simple brin 8,9,21 ont été utilisés avec succès en tant que donateurs. Ni NHEJ-nor édition de génome HR médiée nécessitent l'introduction d'un marqueur sélectionnable dans le génome de l'embryon de souris, ce qui rend ces stratégies particulièrement bien adapté pour la création de petites modifications dans la séquence de nucleotides sans perturber l'architecture génétique globale.
Dans ce protocole, nous décrivons toutes les procédures essentielles pour l'édition du génome dans leembryon de souris à l'aide TALENs. Il s'agit notamment 1) l'identification d'un site TALEN cible 22, 2) la construction de TALENs par golden gate clonage 13, 3) la synthèse in vitro de TALEN ARNm, 4) la micro-injection de TALEN ARNm dans des ovocytes fécondés de souris, 5) les procédures chirurgicales pour le transfert d'embryons et 6) l'analyse de la mutagenèse induite dans TALEN animaux fondateurs. Nous nous concentrons sur TALEN ARNm micro-injection et le dépistage des fondateurs de NHEJ induites insertions / suppressions. A cet effet, nous avons généré des constructions bifonctionnelles Talen qui permettent à la fois l'expression dans des cellules de mammifères transfectées lorsque que des plasmides et dans la synthèse in vitro d'ARNm TALEN pour micro-injection dans des embryons de souris. Ces constructions comprennent un squelette de CONTE tronqué 23 fusionnée à hétérodimère Fokl Domaines 24,25 pour l'édition du génome optimale dans les cellules de mammifères. Ce protocole peut également être adopté pour la microinjection d'autres nucléases de luxe ou pour préparations injectables combinés des nucléases de créateurset les constructions de donateurs (conception des bailleurs de fonds de l'ADN a été décrit dans d'excellentes publications techniques par Wefers et al. 26,27).
Déclaration éthique
Toutes les expériences sur les animaux ont été effectuées en conformité avec les directives et règlements de l'Office vétérinaire cantonal du canton de Zurich.
Protocole
Une. Identification de Talen sites cibles
- Visitez le site Web 2.0 TAL effecteur nucléotides Cibleur (http://tale-nt.cac.cornell.edu) et choisissez "TALEN Cibleur".
- Entrer la séquence du gène cible. Si expression pCAG-T7-TALEN construit est utilisé (figure 1C) choisir "Miller et al., 2011" sous "Utiliser un préréglage Architecture" afin de prévoir des sites cibles qui peuvent être ciblées avec la longueur d'écartement optimal (15-20 pb) et le nombre de DRV TALE (15-20) pour cette architecture TALEN.
- Choisissez "NN" sous "G de remplacement" (NH DRV spécifique de la guanosine sont également disponibles mais non encore testés intensivement pour le montage TALEN).
- Facultatif: pour les autres paramètres et les options s'il vous plaît suivez les instructions sur le site et les liens qui y sont dispensés.
- Un fichier texte avec cibles des sites potentiels est généré, qui peut êtreimporté dans un tableur pour un visionnement plus commode. Sélectionnez la paire (s) TALEN pour le montage Golden Gate.
- Facultatif: Séquence de la région génomique d'intérêt pour la souche de souris qui sera utilisé pour la micro-injection de détecter d'éventuels polymorphismes nucléotidiques simples (SNP) qui pourraient ne pas être pris en compte dans les bases de données publiques et pourrait empêcher la nucléase de liaison.
- Facultatif: Reportez-vous au tableau 3 pour liés nucléases-ressources en ligne de créateurs.
- La plupart des plasmides requis pour ZFN et TALEN construction peuvent être obtenus auprès Addgene:
http://www.addgene.org/special-collections/
Addgene ID pour le kit Golden Gate TALEN et mammifères constructions d'expression Talen sont fournis dans le tableau 2.
Alternativement, notamment des modules fonctionnels tels que les domaines à doigts de zinc peuvent être commandés à partir d'un fournisseur de services de synthèse de gènes.
2. Golden Gate Assemblée TALEN
Cette section décrit l'ensemble de TALENs utilisant un protocole publié par Cermak et al. 13 TALENs longs sont construits en utilisant deux étapes de clonage Golden Gate suivantes (figure 1). Cette approche permet l'incorporation d'un certain nombre de DRV entre 12 et 31 dans les constructions d'expression finales. Le protocole de montage a été adapté à des vecteurs de destination conçus pour générer des ARNm (figure 1C) qui expriment TALENs hautement actives suivantes ovocytes micro-injection. S'il vous plaît se référer également au protocole en ligne du kit Golden Gate TALEN pour établir et maintenir la bibliothèque de plasmide ( http://www.addgene.org/TALeffector/goldengateV2/ ).
- Jour 1 - Golden Gate Réaction n ° 1 - Assemblée des DRV tableaux en PFUS Vecteurs
- Facultatif: Pour chaque paire TALEN conçu en Protocol 1, entrez la séquence d'ADN des deux sites cibles (y compris l'initiale T, 5 'à 3') dans la feuille de calcul de TALEN_Voytas_Pipetting pour générer un schéma de pipetage pour chaque réaction Golden Gate (entrer également des concentrations de tous les plasmides utilisés pour les assemblages ). La feuille de calcul fournit également la séquence d'ADN attendue de toutes les DRV qui peuvent être utilisés pour l'alignement des résultats de séquençage dans la section 2.5.2.
- Pour chaque TALEN unique conçu dans le protocole 1:
Si la longueur est TALEN 12-21 (standard), sélectionnez variables de répétition di-résidus (DRV) 1-10 et vecteur de destination pFUS_A. Sélectionnez DRV restants et un vecteur de destination pFUS_B, par exemple pour un TALEN avec 15 DRV (y compris la dernière répétition, qui sera ajouté à l'assemblage final dans la section 2.3.3, sélectionnez DRV 11-14 et vecteur de destination pFUS_B4 (figure 1A).
Si la longueur est TALEN 22-31, 1-10 utilisation DRV et de destination vecteur pFUS_A30A. Choisissez DRV 11-20 et vecteur de destination pFUS_A30B. Sélectionnez DRV restants et un vecteur pFUS_Bdestination, par exemple pour TALEN avec 24 DRV choisir DRV 21-23 et vecteur de destination pFUS_B3. - Mettre en place Golden Gate réaction # 1 pour chaque PFU + combinaison DRV séparément, soit 1-10 + pFUS_A et DRV + vecteur pFUS_B respective restants ou pour TALEN plus de 21 DRV, 1-10 + pFUS_A30A, 11-20 pFUS_A30B et DRV restants + vecteur pFUS_B respectif. Utiliser 150 ng de chaque vecteur DRV, 150 ng de vecteur PFU, une BsaI ul, 1 ul d'ADN ligase T4, 2 ul de tampon 10x T4 ADN ligase, et H 2 O à 20 pi de volume de réaction total. (Utilisation aliquotes frais de tampon de ligase T4 pour chaque tour des assemblées Golden Gate est recommandé depuis réitéré décongélation / congélation de tampon de ligase T4 peut réduire l'efficacité des réactions.)
- Placez réactions Golden Gate dans un cycleur thermique.
Programme:
37 ° C 5 min
16 ° C 10 min
10 cycles
50 ° C 5 min
80 ° C 5 min - Ajouter 1 pi de ATP 10 mM et 1 ul de plasmide-Safe nuclease à chaque mélange et incuber à 37 ° C pendant 1 heure. Ce traitement élimine linéaires produits de ligature incomplètes qui pourraient être clonées dans les vecteurs de destination par recombinaison homologue in vivo dans des bactéries transformées.
- Transformer E. coli avec des réactions individuelles de ligature (électrocompétent ou chimiquement compétentes E. coli qui facilitent α-complémentation tels que XL1-Blue ou DH5ct peut être utilisé ici et dans les transformations ultérieures).
- les bactéries sur des plaques de spectinomycine (50 pg / ml) avec des plaques de X-Gal et de l'IPTG (40 ng / ml chacun) pour la sélection de colonies bleu / blanc.
- Jour 2 - Confirmation de Corriger Assemblée UFP-DRV
- En utilisant colonie PCR avec des amorces pCR8_F1 et pCR8_R1 (Voir le tableau 1 pour les séquences d'amorces) 1-3 écran blanccolonies de chaque plaque. Corriger les assemblées UFP-DRV montrent généralement une bande correspondant à la longueur combinée de toutes les DRV clonés (par exemple environ 1,1 ko pour 10 DRV) et une «échelle» de petits groupes moins importants (figure 1B).
Programme de PCR:
95 ° C 3 min
95 ° C 30 sec
55 ° C 30 sec
72 ° C 1 min 45 sec
30-35 cycles
72 ° C 10 min - Utilisation confirmé clones pour démarrer la culture pendant une nuit (2-5 ml de LB avec 50 ug / ml de spectinomycine).
- En utilisant colonie PCR avec des amorces pCR8_F1 et pCR8_R1 (Voir le tableau 1 pour les séquences d'amorces) 1-3 écran blanccolonies de chaque plaque. Corriger les assemblées UFP-DRV montrent généralement une bande correspondant à la longueur combinée de toutes les DRV clonés (par exemple environ 1,1 ko pour 10 DRV) et une «échelle» de petits groupes moins importants (figure 1B).
- Jour 3 - Golden Gate Réaction n ° 2 - Tableaux DRV dans Talen vecteurs d'expression
- Effectuer "minipreps" d'isoler des ensembles UFP-DRV (selon le nombre de DRV soit pFUS_A et pFUS_B ou pFUS_A30A, pFUS_A30B, et pFUS_B).
- Facultatif: Séquence individuels vecteurs PFUS utilisant des amorces pCR8_F1, pCR8_R1 (voir le tableau 1 pour les séquences d'amorces). Le séquençage peut également être effectuée sur final TALEN construit (section 2.5.2); Toutefois, pour plus TALENs complète lit de toutes les DRV pourrait ne pas être possible en utilisant le séquençage Sanger.
- Mettre en place Golden Gate réaction # 2 pour chaque unique TALEN. 150 ng de chaque vecteur UFP, 150 ng de DPP-HD, PLR-NG, PLR-NI, PLR-NN (dernier "demi-repeat") en fonction de la conception de la séquence DRV et pour la l auche TALEN utiliser 75 ng de pCAG-T7-TALEN-ELD-Destination et pour le droit TALEN utiliser 75 ng pCAG-T7-TALEN-KKR-Destination (ou vice versa). Ajouter 1 pi Esp3I, 1 ul de ligase d'ADN T4, 2 ul de tampon de ligase d'ADN T4 10x, H 2 O à 20 pi de volume réactionnel total.
- Placez réactions Golden Gate dans un cycleur thermique.
Programme:
37 ° C 5 min
16 ° C 10 min
10 cycles
37 ° C 15 min
80 ° C 5 min - Utilisez des réactions de la section 2.3.4 pour transformer E. coli.
- Jour 4 - Confirmation de Corriger Assemblée TALEN
- Écran 1-3 colonies blanches de chaque plaque par colonie PCR avec des amorces TAL_F1 et TAL_R2 (voir le tableau 1 pour les séquences d'amorces). TALENs correctement assemblés montrent un produit PCR avec une longueur correspondant au nombre total de DRV incorporé (figure 1D, ce groupe est parfois difficile de détecter lorsque l '«effet d'échelle» représente un indicateur robuste de réunion réussie).
Programme de PCR:
95 ° C 3 min
95 ° C 30 sec
55 ° C 30 sec
72 ° C 3 min
30-35 cycles
72 ° C 10 min - Utilisez confirmé clones corrects pour démarrer des cultures bactériennes durant la nuit (2-5 ml de LB avec 100 pg / ml d'ampicilline).
- Écran 1-3 colonies blanches de chaque plaque par colonie PCR avec des amorces TAL_F1 et TAL_R2 (voir le tableau 1 pour les séquences d'amorces). TALENs correctement assemblés montrent un produit PCR avec une longueur correspondant au nombre total de DRV incorporé (figure 1D, ce groupe est parfois difficile de détecter lorsque l '«effet d'échelle» représente un indicateur robuste de réunion réussie).
- Jour 5 - Confirmation de Corriger Assemblée TALEN
- Effectuer "minipreps" pour isoler les plasmides pCAG-T7-Talen.
- Si le séquençage n'a pas été effectuée dans soiction 2.3.2, l'utilisation des amorces TAL_Seq_5-1 et TAL_R2 (voir le tableau 1 pour les séquences d'amorces) pour déterminer ensemble de DRV correct en pleine longueur TALEN.
3. Nucléase ARNm de synthèse
- Génération de modèle de l'ADN
- Préparer midi de haute qualité ou Maxipreps de plasmides pCAG-T7-Talen pour la synthèse de l'ARNm.
- 10 pg de linéariser le plasmide TALEN synthèse ou ZFN ARNm en utilisant une enzyme de restriction qui clive préférentiellement en aval et à proximité immédiate de la nucléase de codon STOP (pour les vecteurs pCAG-T7-Talen utilisent SacI). des plasmides de synthèse de l'ARNm comprennent typiquement un promoteur T7 ou SP6 de phage en amont de la séquence codante de la nucléase.
- Exécutez 200-500 ng de plasmide digéré sur un gel d'agarose à 0,7-1% de vérifier pour la digestion complète.
- Éliminer les sels à partir du plasmide digérer par précipitation de l'ADN avec 0,1 volume d'acétate de sodium et de 3 volumes d'éthanol pendant 1 heure à températur ambiantee. Pellet l'ADN par centrifugation à 14 000 xg ou plus pendant 10 minutes, laver le culot avec 200 ul 70% d'éthanol, rotation pendant 5 min, retirer de l'éthanol, sécher à l'air le culot et remettre en suspension dans un volume approprié d'eau RNAse free. La purification du plasmide linéarisé est également possible en utilisant un système basé sur colonne, par exemple un kit de purification de PCR QIAquick.
- Déterminer la concentration du modèle linéaire et utiliser 1 pg de mettre en place la transcription in vitro.
- La polyadénylation de l'ARNm et la synthèse
- Pour la transcription in vitro de pCAG-T7-TALEN plasmides utilisent le kit Ultra mMESSAGEmMACHINE T7. Mettre en place la réaction de transcription pour chaque TALEN sur glace: de 20 pi avec de l'eau sans nucléase, 10 pl T7 2x NTP / ARCA, 2 pl 10x T7 Reaction Buffer, 1 pg de matrice d'ADN, 2 pl T7 mélange d'enzymes. Mélanger la réaction et incuber pendant 1-2 heures à 37 ° C.
- Utilisez la transcription complète de 20 pition mélange réactionnel pour mettre en place la réaction de polyadénylation sur glace: 36 ul d'eau sans nucléase, tampon 5x EPAP de 20 ul, 10 ul 25 mM MnCl2, ATP 10 mM pi 10. Mélanger et retirer 2,5 pi du mélange de réaction que des échantillons de contrôle L1 et R1 (pour nucléase gauche et à droite, respectivement). Ajouter 4 ul de l'enzyme E-PAP et incuber le mélange réactionnel pendant 45 à 60 min à 37 ° C. Retirer un autre 2,5 pi de mélange réactionnel que des échantillons de contrôle L2 et R2.
- La purification de l'ARNm
- Utilisez NucAway colonnes de centrifugation pour la purification de l'ARNm (d'échange de tampon et l'élimination des nucléotides non incorporés). Appuyez sur les colonnes pour régler gel sec dans le bas de la colonne. Colonne hydrate avec 650 pi de tampon RNase micro-injection (1 mM Tris-Cl, 0,1 EDTA, pH 7,5). Cap, vortex, appuyez sur les bulles d'air, et hydrate pour 5-15 min à température ambiante.
- Placer la colonne dans un tube de collecte et de spinà 750 xg, 4 ° C pendant 2 minutes pour éliminer l'excès de liquide interstitiel. Jeter colonne de tube de collecte et placer dans un tube de 1,5 ml d'élution.
- Appliquer complet mélange de réaction de la section 3.2.2 de la colonne et centrifuger à 750 xg, 4 ° C pendant 2 min. Nucléase ARNm va maintenant être dissous dans un tampon de micro-injection. Retirer 2,5 pi d'échantillons purifiés L3 et R3.
- ARNm de magasin à -80 ° C jusqu'à ce que des parties aliquotes de micro-injection sont préparées.
- ARNm électrophorèse sur gel de
- Nettoyer une chambre de gel pour éliminer les contaminations RNAse en utilisant une solution de SDS 10% ou RNaseZAP.
- Préparation d'un gel d'agarose à 1% dans du tampon de migration 1xTBE.
- Mélanger chaque échantillon L1/R1 d'ARN, L2/R2, L3/R3 avec 3 volumes de charge NorthernMax formaldéhyde Dye et incuber pendant 15 min à 65 ° C.
- Charger les échantillons et une échelle de taille d'ARN (par exemple, l'ARN du Millénaire pour le marqueur de taille) sur le gelet exécuter gel à 10 V / cm dans 1x TBE jusqu'à colorant de charge atteint la fin du gel.
- Colorer le gel en utilisant une solution de vert de SYBR (Invitrogen) pour 30 à 60 minutes sous agitation à température ambiante.
- L'image du gel et de comparer les tailles de L1/R1 avec L2/R2 et L3/R3. Échantillons L2/R2 et L3/R3 devraient montrer bandes d'une augmentation de la taille par rapport à L1/R1 indiquant polyadénylation succès (Figure 2).
- Déterminer la concentration de l'ARNm en utilisant un spectrophotomètre.
- Préparer des aliquots d'ARNm pour micro-injections en mélangeant la nucléase de gauche et de droite de l'ARNm nucléase dans un rapport 1:1. Il est recommandé de préparer des aliquotes à une concentration totale de 200 ng / pl (100 ng / ul de chaque nucléase) par dilution avec du tampon de microinjection. aliquotes d'ARNm de magasin à -80 ° C.
4. Embryon isolement et microinjection
- Embryon isolement. Ce protocole a été utilisé avec succès C57BL/6J et souche BDF1s et peut probablement être adapté à d'autres souches couramment utilisées dans les expériences de micro-injection telles que FVB ou CBF1. Les souris sont logées sous un cycle de lumière-obscurité de 12 heures-12 heures à une température et installation à humidité contrôlée avec de la nourriture et de l'eau ad libitum.
- Femelles donneuses Superovulate pour augmenter le rendement en embryons. 16 femmes (âgés de 4 semaines) sont-ovulation par voie intrapéritonéale (ip) injection de gonadotrophine de sérum de jument gravide 5 UI (PMSG), suivie d'une injection ip de 5 UI de gonadotrophine chorionique humaine (hCG) 48 heures plus tard.
- Compagnon les 16 femmes superovulées 16 ans d'élevage (2-8 mois) mâles immédiatement après l'injection d'hCG.
- Sacrifier les femmes utilisant un protocole d'euthanasie approuvé comme inhalation de CO 2.
- Récupérer les ovocytes fécondés. Les ovocytes sont prélevés à partir des oviductes et le lendemain matin, puis libérés de toutes les cellules du cumulus restants par un traitement de 3 à 5 min dans 0,1% de hyaluronidase bovine dissous dans le milieu M2. En fonction de la capacité d'accouplement et de la souche utilisée, le rendement de l'embryon peut varier. 16 femelles C57BL/6J produisent généralement 150-250, tandis que BDF1 donné 300-400 ovocytes fécondés, respectivement.
- Embryon microinjection
- Injecter la nucléase ARNm. Ovocytes de stade pronucléaire sont généralement injectés dans l'après-midi. La micro-injection cytoplasmique de l'ARNm est réalisée dans le milieu M2 sous de l'huile minérale à l'aide d'un microscope inversé équipé d'DIC Nomarski objectif 20X et à l'aide de micromanipulateurs d'embryon, ainsi que une unité d'injection. Il est recommandé de commencer les injections de TALEN ARNm à une concentration de 10 ng / pl (20 ng / pl au total).
- Aspirer l'ovocyte avec un capillaire de maintien et d'injecter l'ARNm de nucléase dans le cytoplasme de l'embryon en évitant le contact avec les pronuclei. L'injection doit être peu profonde, à proximité de la membrane plasmique. Le volume d'injection doit rester faible et l'aiguille de microinjection devrait être l'esprithdrawn au premier signe de distension cytoplasmique. Une série de micro-injection se compose normalement de 100-300 ovocytes. Typiquement, au moins 80-90% des embryons devrait survivre à l'injection sans lyse immédiate.
- Après microinjection, placer les embryons pendant 1 heure dans M16 (Sigma) milieu à 37 ° C et 5% de CO 2 et de transférer les embryons survivants dans des mères porteuses pseudo-.
5. Chirurgicale de transfert d'embryons
- Préparer les bénéficiaires de l'embryon pseudo-(mères d'accueil) un jour avant l'ARNm microinjection. Les femelles matures (3-6 mois) d'une souche consanguine robuste, tel CD-1, sont bien adaptés pour le rôle. Provoquer pseudogestation en accouplant les femelles sur une veille avec les hommes soit chirurgicalement ou génétiquement vasectomisés 28. Seuls 0,5 femelles ced avec un bouchon de copulation clair sont utilisés comme des embryons bénéficiaires. Chez les femelles inutilisés le pseudogestation disparaît après environ 3 weeks permettant leur utilisation répétée.
- Anesthésier les 0,5 dpc mère nourricière femelles par injection ip de la kétamine, xylazine (120 mg / kg et 16 mg / kg, respectivement). Cette formulation garanties ~ 30 min chirurgicale temps de tolérance est plus que suffisante pour la durée d'utilisation prévue de 5 à 10 min.
- Placez l'animal anesthésié sur son abdomen sur une surface chaude et désinfecter la zone d'incision avec un désinfectant approprié tel que 70% d'éthanol ou de chlorhexidine et le mélange de l'alcool.
- Inciser la peau sur le dos de l'animal et d'ouvrir la cavité peritoneale avec des ciseaux stériles.
- Visualisez et externaliser la corne utérine en tirant sur le coussinet adipeux attaché à l'ovaire. Gardez les organes humide à l'aide chaleureuse solution de NaCl 0,9%. Gardez à l'esprit que le site chirurgical peut être drapée avec un pansement stérile ou rasée afin de se conformer aux directives vétérinaires locaux.
- Immobiliser les organes reproducteurs par clipsage le coussinet adipeux ovaire avec un collier de bouledogue.
- Saisir doucement l'oviducte juste en amont de l'ampoule avec une pince horloger et utiliser une aiguille hypodermique 30 G pour créer un petit trou dans le mur de l'oviducte.
- Retirer l'aiguille et insérez un capillaire de verre mince contenant les embryons dans le trou permettant le placement des embryons dans l'ampoule en soufflant doucement dans l'embouchure du porte-capillaire. Retirer le capillaire une fois que les embryons sont déposés dans l'ampoule et remplacer les organes reproducteurs de retour dans la cavité du corps.
- Fermez la cavité péritonéale avec une série de sutures stériles (Prolene 6-0).
- Fermer la peau à l'aide des agrafes 1-2 Autoclip (9 mm) en fonction de la taille de l'ouverture.
- À la suite de l'opération de retour les animaux à leur cage et les surveiller jusqu'à ce que l'effet de l'anesthésie se dissipent.
- Afin de minimiser le stress, abritera la souris dans les groupes sociaux stables (2-4 animaux / cage de type III) chaque fois que possible.
- Appliquer analgésique post-opératoires sous forme de Dafalgan ajoutés à l'eau potable (paracétamol 200 mg / kg de poids corporel) pendant 3 jours après la chirurgie.
6. Analyse des Fondateurs par PCR et T7 endonucléase ou digestion enzymatique
- Conception des amorces pour amplifier une région située entre 200 à 700 pb dans le site de liaison à la nucléase. Les distances région d'espaceur de l'avant-nucléase amorce et la nucléase région espaceur-amorce inverse doivent être suffisamment différentes pour permettre la détection de deux bandes distinctes pour les produits digérés sur un gel d'agarose (voir figures 3 et 4 pour des exemples).
- Exécutez PCR avec des conditions optimisées. Vérifier la taille de l'amplicon de PCR sur un gel d'agarose.
- Facultatif: produits Purify PCR, par exemple avec un kit de purification QIAquick PCR pour éliminer les sels et les nucléotides du mélange PCR. De nombreuses enzymes de restriction sont actives dans des échantillons de PCR complémentées avec le tampon et la purification d'enzyme de restriction approprié n'est pas nécessaire. Nous avons également successfully utilisé endonucléase de T7 dans un tampon PCR QiagenTaq complété par NEBuffer 2.
- T7 endonucléase de dosage. Mélanger 17 pi de produit de PCR avec 2 pi de NEBuffer 2 et exécutez formation hétéroduplexe (Figure 3b) programme dans un thermocycleur:
95 ° C 2 min
95 ° C à 85 ° C (-2 ° C / sec)
85 ° C à 25 ° C (-0,1 ° C / sec)
4 ° C en attente.
Ajouter 1 pi de T7 endonucléase à chaque échantillon et incuber à 37 ° C pendant 20 min. (Le test de géomètre (Transgenomic), qui s'appuie sur CEL 1 nucléase, peut également être utilisé ici. S'il vous plaît se référer aux instructions du fabricant.) - Digestion de restriction de produit de PCR. Mélanger 17 ul de produit de PCR avec 2 ul de 10 x tampon d'enzyme de restriction et 1 ul d'enzyme de restriction. Digérer à la température appropriée pendant 1 heure ou plus.
- T7 endonucléase de dosage. Mélanger 17 pi de produit de PCR avec 2 pi de NEBuffer 2 et exécutez formation hétéroduplexe (Figure 3b) programme dans un thermocycleur:
- Ajouter chargement colorant de l'ADN des échantillons et sur un gel d'agarose à 2% pour détecter la digestion distinctemodèles pour les animaux de type sauvage et les fondateurs portant allèles mutés. Voir les figures 3 et 4 pour les résultats attendus.
- Produits de PCR clone de fondateurs positifs pour allèles mutés pour le séquençage Sanger (par exemple par TA-clonage dans pGEM-T Easyor séquence directement des mélanges de produits de PCR utilisant le séquençage de prochaine génération.
Résultats
Nous avons construit des plasmides de destination compatibles avec l'ensemble d'Or GateTALEN publié par Cermak et al. Qui 13 permettent l'expression de TALENs dans des cellules de mammifères, ainsi que la synthèse d'ARNm in vitro à partir du promoteur du phage T7 (figure 1C). Ces plasmides portent les domaines de FokI hétérodimères (ELD ou KKR mutations) qui ont été montrés pour réduire les effets hors-cible par rapport à des homodimères FokI et pour améliorer l'activité de clivage par rapport à la première génération Fokl hétérodimères 25,29. Réactions Golden Gate de montage n ° 1 et n ° 2 sont généralement très efficace et chaque colonie blanche, lorsqu'on l'analyse par PCR sur colonie, montre l'évolution attendue pour le nombre particulier de DRV clonés (figures 1B et 1D).
L'analyse sur gel de synthèse in vitro de l'ARNm (figure 2) doit faire apparaître une seule bande distinguable avec peu ou pas de frottis pour chaque échantillon analysé. Il devrait y avoir un changement de taille claire entre les échantillons L1/R1 et L2/R2, L3/R3, qui indique polyadénylation succès.
animaux fondateurs peuvent être criblés pour des allèles mutés NHEJ-induites en utilisant le génotypage par PCR suivie soit digestion T7 endonucléase (Figure 3) ou une digestion de restriction en utilisant une enzyme qui clive la séquence de type sauvage au sein de la région d'espacement de la paire de nuclease (figure 4). L'analyse par endonucléase de T7 est applicable à n'importe quel type de mutation, indépendamment de la séquence génomique spécifique à l'intérieur de la région d'espacement de la paire de nucléases injecté; Cependant, il ne détecte que les inadéquations entre les brins d'ADN dans les produits de PCR heteroduplexes. Ainsi, dans les rares cas où l'un des fondateurs porteur de deux allèles mutés de manière identique, les produits de PCR ne montrer aucun modèle T7 de digestion. Une telle distinction est cependant toujours possible quand un site de restriction spécifique est situé à l'intérieur de la région d'espaceur de nucléase qui sera éliminé par NHEJInduite par des insertions / délétions (figure 5). Ici, les bandes non digérés indiquent la présence de mutations, et l'absence de produits digérés suggère fortement l'un des fondateurs portant des mutations dans les deux allèles du gène ciblé (marquées par des astérisques dans la figure 4b).
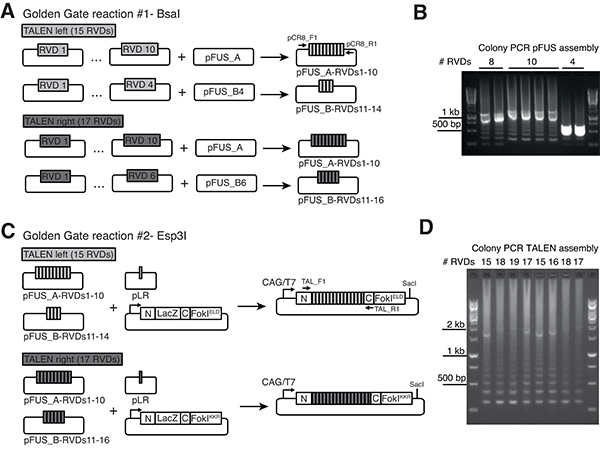
Figure 1. Golden Gate clonage de Talen DRV en hétérodimères vecteurs pCAG-T7-Destination. A) Assemblée des tableaux DRV dans des vecteurs UFP. Voici un exemple est montré pour une paire TALEN avec des tableaux de fées individuelles comprenant 15 DRV et 17 DRV, respectivement (pour les tableaux de CONTE plus de 21 DRV, trois ensembles UFP-DRV sont nécessaires, non représenté). Les flèches indiquent les amorces pour B) produits PFUS spécifique réactions de PCR de colonie de PCR.amplifié à partir des ensembles de PFU correctes montrent généralement une bande correspondant à la longueur combinée de toutes les DRV clonés (par exemple environ 1,1 ko pour 10 DRV) et une «échelle» de petits groupes moins importants en raison de la nature répétitive des tableaux DRV. C) L'assemblage final de tableaux UFP-DRV et un plasmide contenant la dernière répétition (PLR) dans hétérodimères vecteurs d'expression Talen avec des variantes, respectivement FokIELD et FokIKKR. Le squelette TALEN (annoté comme N et C) ressemble à l'architecture publié par Miller et al. 23 Le promoteur CAG (CMV élément activateur précoce / chicken beta-actine) assure des niveaux d'expression élevés dans des cellules de mammifère transfectées, tandis que le promoteur du phage T7 permet en synthèse in vitro d'ARNm (utiliser Sacl pour la linéarisation du vecteur en aval de la nucléase codon STOP). D) Colony PCR utilisant des amorces indiquées par les flèches dans C) permet d'identifier correctement assemblé TALENs. Full-lengtproduits h PCR sont souvent moins important tandis que l '«effet d'échelle» représente un indicateur robuste de réunion réussie. Cliquez ici pour agrandir l'image .
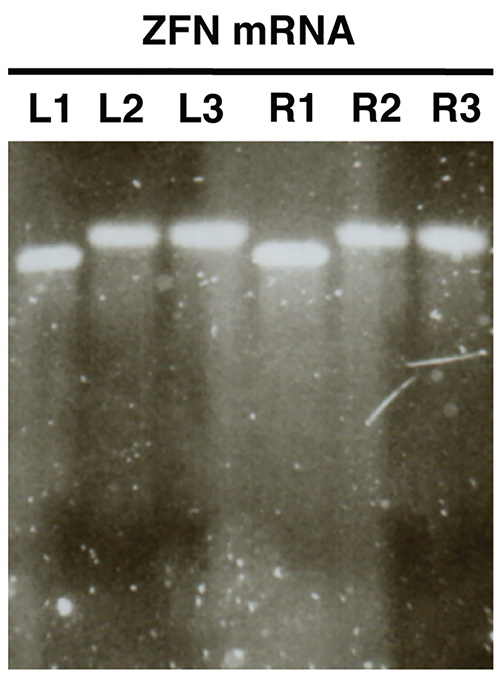
Figure 2. . contrôle de la qualité de la nucléase ARNm dans la synthèse in vitro en utilisant une électrophorèse sur gel d'agarose ARNm ZFN sont présentées à titre d'exemple (L, gauche ZFN, R, droit ZFN). Échantillons L1/R1 montrent ARNm avant polyadénylation, des échantillons L2/R2 spectacle ARNm polyadénylé et L3/R3 montrer ARNm polyadénylé purifié. Cliquez ici pour agrandir l'image .
alt = "Figure 3" fo: contenu width = "6 po" fo: src = "/ files/ftp_upload/50930/50930fig3highres.jpg" src = "/ files/ftp_upload/50930/50930fig3.jpg" width = "600" />
Figure 3. Exemple de test d'endonucléase de T7 utilisé pour l'identification des animaux fondateurs portant des mutations induites par la nuclease du locus cible. A) Une paire TALEN a été conçu pour cliver l'intérieur de la région codante du gène de la protéine prion de souris (Prnp, TALEN séquence cible peut être fourni sur demande). Un produit de PCR est généré en utilisant une amorce sens (F) situé à 110 pb en amont et une amorce antisens (R) de 250 pb en aval du site de clivage TALEN. B) Le produit de PCR est ensuite soumis à heteroduplex formation et T7 digestion par une endonucléase. C) TALEN mutagenèse induite à l'intérieur de la région génomique ciblée des fondateurs simples est révélée par la présence d'un produit de PCR de pleine longueur avec des produits de digestion de 250 et 110 pb.50930/50930fig3highres.jpg "target =" _blank "> Cliquez ici pour agrandir l'image.

Figure 4. Exemple d'une digestion de restriction des produits de PCR utilisées pour l'identification des animaux fondateurs portant des mutations induites par la nuclease du locus cible. Spécifiques ZFN pour la souris Rosa26 locus 29 cible un site de restriction XbaI au sein de l'intron 1. A) Les fondateurs ont été criblés par génotypage par PCR en utilisant une amorce avant (F) situé à 500 pb en amont et une amorce antisens (R) de 250 pb en aval du site de clivage. B) La digestion des produits de PCR avec Xbal révèle schémas de digestion indiquant les souris avec des mutations bi-alléliques (repérées par des astérisques), mono alléliques mutations (bandes digérés et non digérés, par exemple animale 2)et souris en poids (digestion complète, par exemple, un animal 21). C) Ordonnancement des souris avec des modifications bi-allélique potentiels montre jusqu'à 3 (24) animaux distincts insertions / suppressions. Cliquez ici pour agrandir l'image .
| Nom de Primer | Séquence 5 'à 3' |
| pCR8_F1 | ttgatgcctggcagttccct |
| pCR8_R1 | cgaaccgaacaggcttatgt |
| TAL_F1 | ttggcgtcggcaaacagtgg |
| TAL_R2 | ggcgacgaggtggtcgttgg |
| TAL_Seq_5-1 | catcgcgcaatgcactgac |
Tableau 1. Séquences d'amorces utilisées pour PCR et séquençage des colonies dans l'ensemble du Golden Gate TALENprotocole.
| Plasmide / Collection | Collaborateur | Addgene ID | Commentaires |
| Golden Gate TALEN et TAL effecteur Kit 2.0 | Voytas laboratoire | 1000000024 | Contient tous les plasmides nécessaires à l'assemblage Golden Gate TALEN |
| pCAG-T7-TALEN -KKR/ELD vecteurs de destination | Pelczar laboratoire | 40131, 40132 | Add-on plasmides pour l'expression TALEN dans les cellules de mammifères et de la synthèse de l'ARNm in vitro |
Tableau 2. Les plasmides et les collections de plasmides nécessaires à l'assemblage Golden Gate TALEN peuvent être obtenus auprès Addgene ( www.addgene.org ).
| En ligneRessource | Commentaires |
| http://tale-nt.cac.cornell.edu | Conception de TALEN; TALEN prévision hors cible |
| http://zifit.partners.org/ZiFiT/ | Conception de TALEN, OPEN ZFN, CoDA ZFN, CRISPR/Cas9 |
| http://www.genome-engineering.org | Conception de TALEN, CRISPR/Cas9; CRISPR/Cas9 prévision hors cible |
| http://baolab.bme.gatech.edu/Research/BioinformaticTools/assembleTALSequences.html | Assemblée de TALEN séquences pour la confirmation des résultats de séquençage |
| http://pgfe.umassmed.edu/ZFPmo dularsearchV2.html | Conception d'assemblage modulaire ZFN |
| www.genomecenter.ucdavis.edu/s egallab / segallabsoftware | Conception d'assemblage modulaire ZFN |
Tableau 3. Ressources en ligne pour la conception ZFN, TALEN, et CRISPR/Cas9.
Discussion
Designer génome approches d'édition axée sur la nucléase ont considérablement élargi la gamme des espèces qui se prêtent à des modifications ciblées de leurs génomes respectifs 10,12. Chez la souris, le gène de ciblage dans des cellules ES a été une technique standard pour plus de deux décennies; Cependant, il s'est avéré difficile d'adapter à des cellules ES provenant d'espèces autres que la souris, bien qu'il y ait eu un certain succès récent dans les cellules ES de rat. Même avec la disponibilité de la souris ES clones cellulaires de gènes ciblés "off-the-shelf» fournis par des consortiums tels que EUCOMM, KOMP, ou NorCOMM 3 génome édition par ZFN et TALEN offre une plus grande précision et de flexibilité en ce qui concerne le spectre de modifications qui peuvent être introduit dans le génome de la souris. animaux fondateurs portant des mutations de la nucléase médiation semblent être très germinale compétente 4-6,20,21, ce qui n'est pas toujours le cas pour des chimères provenant des injections de blastocyste de cellules ES. Ainsi, dans certains cas, la micro-injection de desnucléases igner peuvent engendrer une beaucoup plus rapidement de nouvelles lignées de souris avec des modifications génomiques ciblées.
La génération réussie de souris knock-out par injection de ZFN et TALEN dépend dans une large mesure sur l'activité de la paire de nucléase injecté. TALENs se sont révélés avoir un taux élevé de succès pour cibler un large éventail de gènes dans un certain nombre d'organismes; Toutefois, des études récentes suggèrent que la liaison TALEN est sensible à la méthylation de la cytosine 30,31. Ainsi, les paires de nuclease nouvellement générés, par exemple TALENs cloné dans des vecteurs pCAG-T7, peuvent être transfectées de manière transitoire dans une lignée cellulaire de souris telles que les NIH-3T3 ou Neuro -2a, qui imite l'état de l'embryon de souris de la chromatine dans une certaine mesure. Ici, l'activité nucléase peut être évaluée en utilisant l'analyse par endonucléase de T7 ou une digestion de restriction des produits de PCR comme décrit dans la section 5 avant la synthèse de l'ARNm et la micro-injection. Nous vous recommandons le séquençage de la région génomique d'intérêt dans le respective lignée cellulaire et la souche de souris utilisée pour des expériences de micro-injection.
En zygotes de souris, différentes paires Talen ou ZFN vont travailler de manière optimale à différentes concentrations d'ARNm et donc la concentration optimale de travail de la nucléase ARNm microinjecté peuvent être déterminées expérimentalement. Selon la paire de nucléase, une concentration trop faible se traduira par aucun clivage alors trop élevé peut entraîner une létalité embryonnaire. En fonction de la paire de nucléase, nous avons eu du succès en utilisant des concentrations d'ARNm totaux aussi faibles que 2 ng / pl et aussi élevées que 200 ng / ul. Ces effets sont difficiles à prédire, à partir des expériences en culture cellulaire et la concentration optimale de la nuclease à la fois pour la survie embryonnaire et la vitesse du locus cible de modification doit être déterminé de manière empirique.
ZFN très actif ou TALEN peuvent cliver leur séquence cible au-delà du stade d'une cellule de l'embryon microinjecté et provoquer ainsi des motifs complexes de mutagenèse uned mosaïque dans fondateurs. Nous et d'autres avons observé quatre trois ou plusieurs allèles mutés distincts en un seul fondateur (figure 5C). Ainsi, lors de l'établissement d'une nouvelle lignée de souris à partir de ces fondateurs, progéniture doit être soigneusement projeté par le séquençage de la présence de la mutation favorable depuis les essais de digestion fournissent des preuves seulement qu'une mutation undefined est présent.
Une des critiques souvent exprimé contre les systèmes ZFN et Talen est la possibilité que ces nucléases sont également capables de cliver des séquences présentes ailleurs dans le génome qui sont similaires à des sites cibles. Ces effets hors-cible ont été observées avec des réactifs de première génération utilisant le domaine homodimérique de Fokl, et des constructions hétérodimères ont été conçus pour atténuer les effets hors-cible 25. Réduction des sites cibles potentiels peuvent être prédits dans une certaine mesure in silico 32,33 et criblés par PCR et séquençage. Un avantage évident of utilisant ZFNs TALENs et pour générer des souris plutôt que des lignées cellulaires est la possibilité de retirer hors mutations cibles non liées à la modification du génome désiré en effectuant plusieurs croisements en retour à une souche de type sauvage de choix. Pour l'analyse d'un grand nombre de souris fondatrices de la prochaine génération de profondeur séquençage des produits de PCR générés à partir du locus de nucléase ciblée et in silico prédites hors cible loci pourrait offrir une lecture qualitative et quantitative alternative aux essais de digestion des produits de PCR.
Les techniques de procréation assistée décrites dans ce protocole sont optimisés pour les souches de souris standard utilisés pour des expériences de micro-injection telles que C57BL/6J ou B6D2F1. Souris d'origines différentes, telles que les souches non consanguins, peuvent en principe être utilisés pour les approches d'édition du génome et pourraient fournir une base génétique plus approprié pour des questions de recherche spécifiques. La performance des techniques de procréation assistée, comme la superovulation peut être prédicatDECT pour un certain nombre de souches 34-36 mais peut nécessiter une optimisation plus poussée des souches atypiques afin d'obtenir un nombre suffisant d'embryons pour la nucléase microinjection.
Outre ZFN et TALEN, nouvelles nucléases de créateurs tels que le système CRISPR/Cas9 ARN-guidée 9,37,38 ont été mis en place pour les applications d'édition du génome. Tous les procédés de la micro-injection et l'analyse des animaux fondateurs décrits ici sont également applicables à CRISPR/Cas9 et futurs modes d'édition de génome.
Déclarations de divulgation
Les auteurs déclarent aucun conflit d'intérêts.
Remerciements
Nous tenons à remercier Monika Tarnowska, Cornelia Albrecht, et Ewa Skoczylas pour une excellente assistance technique. Cette étude a été financée par la société SNF Sinergia subvention CRSI33-125073 à PP.
matériels
| Name | Company | Catalog Number | Comments |
| BsaI | NEB | R0535S or L | |
| Esp3I | Thermo Scientific | ER0451 | |
| T4 Ligase | NEB | M0202S or L | |
| Spectinomycin | Sigma | S0692-1ML | |
| Ampicillin | Sigma | A0166 | |
| X-Gal | Sigma | B4252 | |
| IPTG | Sigma | I6758 | |
| Plasmid-Safe nuclease | Epicentre | E3101K | |
| QIAprep Spin Miniprep Kit | Qiagen | 27106 | |
| QIAGEN Plasmid Midi Kit | Qiagen | 12143 | |
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| mMESSAGE mMACHINE T7 Ultra Kit | Invitrogen | AM1345 | |
| NucAway Spin Columns | Invitrogen | AM10070 | |
| RNaseZAP | Sigma | R2020-250ML | |
| NorthernMax Formaldyde Load Dye | Invitrogen | AM8552 | |
| RNA Millennium Markers | Invitrogen | AM7150 | |
| 10x TBE buffer | Thermo Scientific | B52 | |
| T7 endonuclease | NEB | M0302S or L | |
| pGEM-T EasyVector System I | Promega | A3600 | |
| SYBR Green I Nucleic Acid Gel Stain | Invitrogen | S-7563 | |
| pregnant mare's serum gonadotrophin (PMSG) | Sigma | G4877 | |
| human chorionic gonadotropin (hCG) | Sigma | CG5 | |
| M2 embryo culture medium | Sigma | M7167 | |
| M16 embryo culture medium | Sigma | M7292 | |
| Mineral oil, embryo tested | Sigma | M8410 | |
| Ketamine | CentraVet | Ket 201 | |
| Xylazine | Sigma Aldrich | 46995 | |
| Equipment/Tools | |||
| Inverted microscope with Nomarski DIC optics (for example Nikon Eclipse TE200) | Nikon | ||
| Micromanipulator units (for example Narishige, NT88NF) | Narishige | ||
| Embryo holding capillaries | Sutter Instruments | B100-75-10 | |
| Embryo injection capillaries | Narishige | GD-1 | |
| Capillary puller (for example Sutter P97) | Sutter Instruments | ||
| Microforge (for example Narishige MF-900) | Narishige | ||
| Walton skin scissors | FST | 14077-10 | |
| Surgical scissors | FST | 14041-10 | |
| Surgical probe | FST | 10140-03 | |
| Reflex wound clip system (9 mm) | FST | 12031-09 | |
| Reflex wound clips (9 mm) | FST | 12032-09 | |
| Dumont fine forceps 5 | FST | 11254-20 | |
| Moria curved forceps | FST | 11370-31 | |
| Moria fine forceps | FST | 11399-80 | |
| Dietrich bulldog clamp | FST | 18038-45 | |
| C57BL/6J mice | Jackson Labs | strain code 000664 | |
| CD-1 mice | Charles River | strain code 000664 | |
Références
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat. Rev. Genetics. 6, 507-512 (2005).
- Johansson, T., et al. Building a zoo of mice for genetic analyses: a comprehensive protocol for the rapid generation of BAC transgenic mice. Genesis. 48, 264-280 (2010).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Carbery, I. D., et al. Targeted genome modification in mice using zinc-finger nucleases. Genetics. 186, 451-459 (2010).
- Meyer, M., de Angelis, M. H., Wurst, W., Kuhn, R. Gene targeting by homologous recombination in mouse zygotes mediated by zinc-finger nucleases. Proc. Natl. Acad. Sci. U.S.A. 107, 15022-15026 (2010).
- Cui, X., et al. Targeted integration in rat and mouse embryos with zinc-finger nucleases. Nat. Biotechnol. 29, 64-67 (2011).
- Sung, Y. H., et al. Knockout mice created by TALEN-mediated gene targeting. Nat. Biotechnol. 31, 23-24 (2013).
- Wefers, B., et al. Direct production of mouse disease models by embryo microinjection of TALENs and oligodeoxynucleotides. Proc. Natl. Acad. Sci. U.S.A. 110, 3782-3787 (2013).
- Wang, H., et al. One-Step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat. Rev. Genetics. 11, 636-646 (2010).
- ZFN, T. A. L. E. N. CRISPR/Cas-based methods for genome engineering. Trends Biotechnol. 31, 397-405 (2013).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nat. Rev. Mol. Cell Biol. 14, 49-55 (2013).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39, (2011).
- Reyon, D., et al. FLASH assembly of TALENs for high-throughput genome editing. Nat. Biotechnol. 30, 460-465 (2012).
- Sanjana, N. E., et al. A transcription activator-like effector toolbox for genome engineering. Nat. Protoc. 7, 171-192 (2012).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nat. Biotechnol. 31, 251-258 (2013).
- Schmid-Burgk, J. L., Schmidt, T., Kaiser, V., Honing, K., Hornung, V. A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes. Nat. Biotechnol. 31, 76-81 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 31, 827-832 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat. Biotechnol. 31, 822-826 (2013).
- Hermann, M., et al. Evaluation of OPEN zinc finger nucleases for direct gene targeting of the ROSA26 locus in mouse embryos. PLoS ONE. 7, (2012).
- Meyer, M., Ortiz, O., Hrabe de Angelis, M., Wurst, W., Kuhn, R. Modeling disease mutations by gene targeting in one-cell mouse embryos. Proc. Natl. Acad. Sci. U.S.A. 109, 9354-9359 (2012).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat. Biotechnol. 29, 143-148 (2011).
- Guo, J., Gaj, T., Barbas, C. F. Directed evolution of an enhanced and highly efficient FokI cleavage domain for zinc finger nucleases. J. Mol. Biol. 400, 96-107 (2010).
- Doyon, Y., et al. Enhancing zinc-finger-nuclease activity with improved obligate heterodimeric architectures. Nat. Methods. 8, 74-79 (2011).
- Wefers, B., et al. . Current Protocols in Mouse Biology. , (2011).
- Wefers, B., Wurst, W., Kühn, R. . Current Protocols in Mouse Biology. , (2011).
- Haueter, S., et al. Genetic vasectomy-overexpression of Prm1-EGFP fusion protein in elongating spermatids causes dominant male sterility in mice. Genesis. 48, 151-160 (2010).
- Perez-Pinera, P., Ousterout, D. G., Brown, M. T., Gersbach, C. A. Gene targeting to the ROSA26 locus directed by engineered zinc finger nucleases. Nucleic Acids Res. 40, 3741-3752 (2012).
- Bultmann, S., et al. Targeted transcriptional activation of silent oct4 pluripotency gene by combining designer TALEs and inhibition of epigenetic modifiers. Nucleic Acids Res. 40, 5368-5377 (2012).
- Valton, J., et al. Overcoming TALE DNA binding domain sensitivity to cytosine methylation. J. Biol. Chem. 287, 38427-38432 (2012).
- Cradick, T. J., Ambrosini, G., Iseli, C., Bucher, P., McCaffrey, A. P. ZFN-Site searches genomes for zinc finger nuclease target sites and off-target sites. BMC Bioinform. 12, (2011).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Byers, S. L., Payson, S. J., Taft, R. A. Performance of ten inbred mouse strains following assisted reproductive technologies (ARTs). Theriogenology. 65, 1716-1726 (2006).
- Luo, C., et al. Superovulation strategies for 6 commonly used mouse strains. J. Am. Assoc. Lab. Animal Sci. 50, 471-478 (2011).
- Pease, S., Saunders, T. L. . International Society for Transgenic Technologies. Advanced protocols for animal transgenesis : an ISTT manual. , (2011).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339, 819-823 (2013).
- Mali, P., et al. RNA-Guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon