Method Article
Rato Genoma Engenharia Usando Nucleases Designer
Neste Artigo
Resumo
Nucleases designers, tais como as nucleases de dedos de zinco (ZFNs) e activadores de transcrição, tal como nucleases efectoras (TALENS) pode ser utilizado para modificar o genoma de embriões préimplantação de ratinho por desencadear tanto a extremidade não homóloga de união (NHEJ) e recombinação homóloga (HR) vias. Estes avanços permitir a rápida geração de ratinhos com modificações genéticas precisas.
Resumo
Camundongos transgênicos portadores modificações específicas do local do genoma (ko, batida-in) são de vital importância para dissecar sistemas biológicos complexos, bem como para a modelagem de doenças humanas e testar estratégias terapêuticas. Os recentes avanços no uso de nucleases de designer, tais como nucleases dedo de zinco (ZFNs), ativador de transcrição-como nucleases efetoras (Talens), e os aglomerados se repete regularmente palindrômicas curtas intercaladas (CRISPR) / (CAS) associada à CRISPR 9 sistema para o local- engenharia genoma específico em aberto a possibilidade de realizar rápida modificação do genoma segmentado em praticamente qualquer espécie de laboratório, sem a necessidade de contar com caule embrionárias (ES) tecnologia celular. Uma edição experimento genoma tipicamente inicia-se com a identificação de locais alvo de nuclease designer dentro de um gene de interesse, seguido por construção de domínios de ligação de ADN personalizados para dirigir a actividade de nuclease para o locus genómico definido pelo investigador. Plasmídeos nuclease Designer são in vitro </ Em> transcrito para gerar mRNA para microinjeção de oócitos fertilizados de camundongo. Aqui, nós fornecemos um protocolo para alcançar a modificação do genoma alvo de injeção direta de TALEN mRNA em oócitos fertilizados de camundongo.
Introdução
Os ratos são, de longe, a plataforma mais popular para a geração de modelos de animais transgênicos. A caixa de ferramentas versáteis para a engenharia genética do embrião de rato 1-3 foi recentemente prorrogado por abordagens edição do genoma com base em nucleases designer, tais como nucleases dedo de zinco (ZFN) 4-6, transcrição nucleases efetoras ativador-like (Talen) 7,8, e os aglomerados regularmente intercaladas repete palindrômicas curtas (CRISPR) / (Cas) 9-associados CRISPR sistema 9. ZFN e função TALEN como pares de dois domínios de design personalizado à base de proteína de ligação a DNA (arrays de proteínas dedo de zinco e repetem-variable di-resíduos (RVDS), respectivamente), que são todos acoplados ao endonuclease FokI 10-12. Por outro lado, a especificidade de clivagem de ADN mediada por Cas9 é fornecido pela transactivação RNAs CRISPR (crRNA e tracrRNA, que também podem ser combinadas numa única molécula de ARN quimérico guia de ARN denominado 11) que age em complexo com oProteína CRISPR.
Talens com uma seqüência definida de RVDS podem ser rapidamente construído por pesquisadores individuais, com uma multiplicidade de estratégias de montagem para escolher 13-17. CRISPR/Cas9 promete ainda menor geração de trabalho intensivo de nucleases grife, no entanto, a especificidade da guia de RNA-DNA de ligação ainda não está completamente resolvido 18,19. Geração de ZFNs personalizados até agora tem sido limitado a laboratórios acadêmicos especializados e fornecedores comerciais, como Sangamo Biosciences eo serviço Sigma CompoZr.
Em geral, o genoma edição com nucleases grife visa introduzir vertente quebras duplas (DSB) em loci genômico definido, que, posteriormente, atrair final não homóloga juntar (NHEJ) ou recombinação homóloga (HR) máquinas de reparo do DNA 10,12. Reparação NHEJ-mediada de um DSB muitas vezes resulta na introdução de inserções e deleções, em estreita proximidade ao local de reparação. Assim NHEJ reparação cum ser explorada para bater para fora a função de um gene-alvo através da introdução de uma mutação frame-turno dentro do genes codificadores de proteínas seqüência 4,7,9. Alternativamente, a adição ou substituição de informação genética definida podem ser conseguidos proporcionando um doador de ADN em conjunto com as nucleases de design. Um doador de ADN compreende sequências de ADN concebidas para o investigador flanqueadas por regiões de homologia com o locus alvo, servindo assim como um molde para a reparação de DSB por HR. Ambos os plasmídeos 5,6,20 e de fita simples oligonucleotídeos 8,9,21 têm sido utilizados com sucesso como doadores. Nem NHEJ-nor genoma edição mediada por AR exigir a introdução de um marcador seleccionável para o genoma do embrião de rato, o que faz com que essas estratégias particularmente bem adequado para a criação de pequenas alterações na sequência de nucleótidos, sem perturbar a arquitectura genética global.
Neste protocolo, descrevemos todos os procedimentos essenciais para a edição de genoma naembrião de rato usando Talens. Estes incluem: 1) identificação de um site de destino TALEN 22, 2) construção de Talens por clonagem Golden Gate 13, 3) síntese in vitro de TALEN mRNA, 4) microinjeção de TALEN mRNA em oócitos de camundongos fertilizados, 5) procedimentos cirúrgicos para transferência de embriões e 6) a análise de mutagénese induzida por TALEN em animais fundadores. Nós nos concentramos em microinjeção TALEN mRNA e triagem dos fundadores para induzidas NHEJ inserções / exclusões. Para este fim, geraram construções talen bifuncionais que permitem tanto a expressão em células de mamíferos quando transfectado como plasmídeos e na síntese in vitro do ARNm TALEN para microinjecção em embriões de ratinho. Estas construções compreendem um backbone CONTO truncado 23 fundido a heterodimeric FokI domínios 24,25 para a edição genoma ideal em células de mamíferos. Este protocolo também pode ser adotado para microinjeção de outras nucleases grife ou por injeções combinadas de nucleases grifee construções doadores (projeto de doadores de DNA tem sido descrita em excelentes publicações técnicas por Wefers et al. 26,27).
Declaração de ética
Todos os experimentos com animais foram realizados de acordo com as diretrizes e regulamentos do Instituto Cantonal de Veterinária da Cantão de Zurique.
Protocolo
1. Identificação de Talen alvo Sites
- Visite o site da TAL 2.0 Effector Nucleotide Targeter (http://tale-nt.cac.cornell.edu) e escolha "TALEN Targeter".
- Introduza a sequência do gene alvo. Se a expressão pCAG-T7-TALEN constrói está sendo usado (Figura 1C), escolha "Miller et al., 2011" em "Usar uma arquitetura Preset", a fim de prever locais-alvo que podem ser direcionados com o comprimento espaçador ideal (15-20 pb) e número de RVDS Tale (15-20) para esta arquitetura TALEN.
- Escolha "NN" em "L Substitute" (NH RVDS específicas de guanosina, também estão disponíveis, mas ainda não testado extensivamente para a montagem TALEN).
- Opcional: para outras configurações e opções, por favor siga as instruções no site e os links fornecidos lá.
- Um ficheiro de texto com potenciais locais-alvo é gerado, o qual pode serimportado para um programa de planilha para visualização mais conveniente. Selecione o par TALEN (s) para a montagem Golden Gate.
- Opcional: Sequência da região genômica de interesse na cepa do mouse que será usado para microinjeção para detectar possíveis polimorfismos de nucleotídeo único (SNPs) que não podem ser contabilizados em bases de dados públicas e poderia evitar nuclease vinculativo.
- Opcional: Consulte a Tabela 3 para nucleases relacionados com recursos adicionais de design on-line.
- A maior parte dos plasmídeos necessários para ZFN e TALEN construção pode ser obtido a partir de Addgene:
http://www.addgene.org/special-collections/
Addgene IDs para o kit de Golden Gate TALEN e mamíferos expressão construções Talen são fornecidos na Tabela 2.
Alternativamente, em particular módulos funcionais, tais como domínios dedo de zinco pode ser encomendado a partir de um provedor de serviço síntese de genes.
2. Assembléia TALEN Golden Gate
Esta seção descreve a montagem de Talens usando um protocolo publicado pela Cermak et al. 13 TALENS Full-length são construídos usando dois passos de clonagem Golden Gate subseqüentes (Figura 1). Esta abordagem permite a incorporação de um número qualquer de RVDS entre 12 e 31 para as construções de expressão finais. O protocolo de montagem foi adaptada para vetores de destino projetado para gerar mRNAs (Figura 1C) que expressam TALENS altamente ativos seguintes microinjeção oócito. Por favor, consulte também o protocolo online do kit Golden Gate TALEN por estabelecer e manter a biblioteca plasmídeo ( http://www.addgene.org/TALeffector/goldengateV2/ ).
- Dia 1 - Golden Gate Reaction # 1 - Assembleia da RVD Arrays em PFUs Vetores
- Opcional: Para cada par TALEN projetado em Protocolo 1, introduzir a sequência de ADN de um dos dois locais do alvo (incluindo o T inicial, de 5 'para 3') para a folha de cálculo TALEN_Voytas_Pipetting para gerar um esquema de pipetagem para cada reacção Golden Gate (também introduzir as concentrações de todos os plasmídeos utilizados para os conjuntos ). A folha de cálculo, também proporciona a sequência de ADN esperada de todos RVDS que podem ser utilizados para o alinhamento dos resultados de sequenciação da secção 2.5.2.
- Para cada TALEN único concebido no Protocolo 1:
Se o comprimento TALEN é 12-21 (padrão), selecione variáveis repetição di-resíduos (RVDS) 1-10 e destino vetor pFUS_A. Selecione restantes RVDS e um vetor de destino pFUS_B, por exemplo, para um TALEN com 15 RVDS (incluindo a última repetição, que será adicionada à montagem final no ponto 2.3.3, selecione RVDS 11-14 e pFUS_B4 destino vetor (Figura 1A).
Se o comprimento TALEN é 22-31, 1-10 e uso RVDS destino vector pFUS_A30A. Escolha RVDS 11-20 e destino vetor pFUS_A30B. Selecione restantes RVDS e um vetor pFUS_Bdestination, por exemplo, para TALEN com 24 RVDS escolher RVDS 21-23 e destino vetor pFUS_B3. - Configure Golden Gate reação º 1 para cada PFUs + combinação RVD separadamente, ou seja, 1-10 + pFUS_A e restantes RVDS + respectivo vetor pFUS_B ou para TALEN mais de 21 RVDS, 1-10 + pFUS_A30A, 11-20 pFUS_A30B e RVDS restantes + respectivo vetor pFUS_B. Utilizar 150 ng de cada um dos vectores RVD, 150 ng de vector PFUs, 1 ul BSAI, 1 ul de DNA ligase de T4, 2 ul de tampão 10x DNA ligase de T4, e H 2 O para 20 l de volume total de reacção. (Usando alíquotas frescas de tampão ligase T4 para cada rodada de assembléias Golden Gate é recomendável, pois repetiu o descongelamento / congelamento de tampão ligase T4 pode reduzir a eficiência das reações.)
- Coloque reações Golden Gate em um termociclador.
Programa:
37 ° C 5 min
16 ° C 10 min
10 ciclos
50 ° C 5 min
80 ° C 5 min - Adicionar 1 mL de 10 mM de ATP e 1 ul-Safe plasmídeo nuclease a cada mistura e incubar a 37 ° C durante 1 hora. Este tratamento vai remover produtos de ligação incompleta lineares que podem ser clonadas nos vectores de destino por recombinação homóloga in vivo, em bactérias transformadas.
- Transforme E. coli com reações individuais ligadura (electrocompetentes ou E. coli quimicamente competente que facilitam a α-complementação, como XL1-Blue ou DH5ct pode ser usado aqui e em transformações posteriores).
- Bactérias placa em spectinomicina (50 mg / ml) placas com X-Gal e IPTG (40 mg / ml cada) para o azul seleção / colônia branco.
- Dia 2 - A confirmação da correta Assembléia PFUs-RVDS
- Usando colônia PCR com primers pCR8_F1 e pCR8_R1 (Ver Tabela 1 para seqüências de primers) tela branca 1-3colónias de cada placa. Corrigir PFUs-RVDS conjuntos tipicamente mostram uma banda correspondente ao comprimento combinado de todos os RVDS clonadas (por exemplo, cerca de 1,1 kb para 10 RVDS) e uma "escada" de menores bandas menos proeminente (Figura 1B).
Programa de PCR:
95 ° C 3 min
95 ° C 30 seg
55 ° C 30 seg
72 ° C 1 min 45 seg
30-35 ciclos
72 ° C 10 min - Utilização confirmou clones para iniciar a cultura durante a noite (2-5 ml de LB com 50 ug / ml de espectinomicina).
- Usando colônia PCR com primers pCR8_F1 e pCR8_R1 (Ver Tabela 1 para seqüências de primers) tela branca 1-3colónias de cada placa. Corrigir PFUs-RVDS conjuntos tipicamente mostram uma banda correspondente ao comprimento combinado de todos os RVDS clonadas (por exemplo, cerca de 1,1 kb para 10 RVDS) e uma "escada" de menores bandas menos proeminente (Figura 1B).
- Dia 3 - Golden Gate Reaction # 2 - Matrizes RVD em Talen Expressão Vetores
- Execute "minipreps" para isolar assembléias PFUs-RVD (dependendo do número de RVDS ou pFUS_A e pFUS_B ou pFUS_A30A, pFUS_A30B e pFUS_B).
- Opcional: Seqüência individuais vetores PFUs usando primers pCR8_F1, pCR8_R1 (ver Tabela 1 para seqüências de primers). A sequenciação pode também ser realizada em final TALEN constrói (seção 2.5.2); no entanto, por mais completo TALENS lê de todos RVDS pode não ser possível usando seqüenciamento Sanger.
- Configure Golden Gate reação # 2 para cada única TALEN. 150 ng de cada vetor PFUs, 150 ng de PLR-HD, PLR-NG, PLR-NI, PLR-NN (último "meio-repeat") de acordo com o projeto da seqüência RVD e para o l eft TALEN usar 75 ng de pCAG-T7-TALEN-ELD-Destino e para a direita TALEN usar 75 ng pCAG-T7-TALEN-KKR-Destino (ou vice-versa). Adicionar 1 mL Esp3I, 1 uL de T4 DNA ligase, 2 ul de tampão ligase do DNA de T4 de 10x, H 2 O e 20 mL de volume total de reacção.
- Coloque reações Golden Gate em um termociclador.
Programa:
37 ° C 5 min
16 ° C 10 min
10 ciclos
37 ° C 15 min
80 ° C 5 min - Use reações da seção 2.3.4 para transformar E. coli.
- Dia 4 - A confirmação da correta Assembléia TALEN
- Tela de 1-3 colónias brancas a partir de cada uma das placas de colónias por PCR com iniciadores TAL_F1 e TAL_R2 (ver Tabela 1 para as sequências dos iniciadores). TALENS correctamente montados mostram um produto de PCR com um comprimento correspondente ao número total de RVDS incorporados (Figura 1D, esta banda é por vezes difícil de detectar, enquanto o "efeito de escada" representa um indicador robusto de montagem bem sucedida).
Programa de PCR:
95 ° C 3 min
95 ° C 30 seg
55 ° C 30 seg
72 ° C 3 min
30-35 ciclos
72 ° C 10 min - Use confirmou clones corretos para iniciar culturas bacterianas durante a noite (2-5 ml LB com 100 mg / ml de ampicilina).
- Tela de 1-3 colónias brancas a partir de cada uma das placas de colónias por PCR com iniciadores TAL_F1 e TAL_R2 (ver Tabela 1 para as sequências dos iniciadores). TALENS correctamente montados mostram um produto de PCR com um comprimento correspondente ao número total de RVDS incorporados (Figura 1D, esta banda é por vezes difícil de detectar, enquanto o "efeito de escada" representa um indicador robusto de montagem bem sucedida).
- Dia 5 - A confirmação da correta Assembléia TALEN
- Execute "minipreps" para isolar plasmídeos pCAG-T7-Talen.
- Se seqüenciamento não foi realizada em sicção 2.3.2, uso primers TAL_Seq_5-1 e TAL_R2 (ver Tabela 1 para seqüências de primers) para determinar a montagem RVD correto em full-length TALEN.
3. Nuclease mRNA Síntese
- Geração do modelo de ADN
- Prepare midi de alta qualidade ou maxipreps de plasmídeos pCAG-T7-talen para síntese de ARNm.
- Linearizar 10 ug do TALEN ou ZFN ARNm síntese plasmídeo utilizando uma enzima de restrição que cliva a jusante e preferencialmente em estreita proximidade com o codão de paragem da nuclease (por vectores pCAG-T7-talen usar Saci). plasmídeos de síntese de ARNm incluem, tipicamente, uma de T7 ou SP6 fago promotor a montante da sequência de codificação de nuclease.
- Executar 200-500 ng de plasmídeo digerido num gel de agarose a 0,7-1% para verificar a existência de uma digestão completa.
- Remover os sais a partir do plasmídeo por digestão precipitar o ADN com 0,1 volumes de acetato de sódio e de 3 volumes de etanol durante 1 hora à temperatur ambientee. Sedimentar o ADN por centrifugação a 14000 x g ou mais, durante 10 minutos, lava-se a pelete com 200 ul de etanol a 70%, de rotação durante mais 5 min, remover o etanol, o ar seco do sedimento e ressuspender em um volume apropriado de água isenta de RNase. A purificação do plasmídeo linearizado é também possível utilizar um sistema baseado em coluna, por exemplo, Kit QIAquick PCR Purification.
- Determinar a concentração do modelo linear e usar um ug de configurar na transcrição in vitro.
- Síntese e poliadenilação de mRNA
- Para transcrição in vitro de pCAG-T7-TALEN plasmídeos usar o mMESSAGEmMACHINE T7 Ultra Kit. Defina-se a reacção de transcrição para cada TALEN em gelo: a 20 ul com água isenta de nuclease, 10 uL de T7 2x NTP / ARCA, 2 ul de Tampão 10 x Reacção de T7, 1 ug de molde de ADN, 2 ul de mistura de enzimas de T7. Misture a reacção e incubar durante 1-2 horas a 37 ° C.
- Use a completa transcrição 20 lmistura de reacção ção para configurar a reacção de poliadenilação em gelo: 36 ul de água isenta de nuclease, 20 ul de tampão 5x EPAP, 10 uL de MnCl2 25 mM, 10 ul de ATP 10 mM. Misture e remover 2,5 ul da mistura de reacção como as amostras de controlo L1 e R1 (por nuclease esquerdo e direito, respectivamente). Adiciona-se 4 ul de enzima E-PAP e incubar a reacção durante 45-60 minutos a 37 ° C. Retirar mais 2,5 ul de mistura de reacção como as amostras de controlo L2 e R2.
- mRNA Purification
- Use NucAway Colunas rotação para a purificação do mRNA (troca de tampão e remoção de nucleótidos não incorporados). Toque colunas para resolver gel seca na parte inferior da coluna. Hidrato de coluna com 650 mL de tampão de ARNase livre de microinjecção (1 mM Tris-Cl, 0,1 de EDTA, pH 7,5). Cap, vortex, bater para fora bolhas de ar, e hidrato de 5-15 min à temperatura ambiente.
- Colocar a coluna no tubo de recolha e de spina 750 xg, a 4 ° C durante 2 minutos para remover o excesso de fluido intersticial. Descarte tubo de coleta e local de coluna em um tubo de 1,5 ml de eluição.
- Aplique mistura de reacção completa da seção 3.2.2 para coluna e rotação a 750 xg, 4 ° C por 2 min. Nuclease ARNm irá agora ser dissolvido em tampão de microinjecção. Retirar 2,5 mL de amostras purificadas L3 e R3.
- ARNm armazenar a -80 ° C, até se alíquotas de microinjecção são preparados.
- mRNA Gel Eletroforese
- Limpe a câmara de gel para remover contaminações RNAse usando solução de SDS 10% ou RNaseZAP.
- Preparar um gel de agarose a 1% em tampão de corrida 1xTBE.
- Misturar cada L1/R1 amostra de RNA, L2/R2, L3/R3 com 3 volumes de carga de corante NorthernMax formaldeído e incubar durante 15 min a 65 ° C.
- Carregar as amostras e uma escada de tamanho de ARN (por exemplo, marcador de tamanho de ARN Millennium) no gele executar o gel a 10 V / cm em 1 x TBE até corante de carga atinge a extremidade do gel.
- Corar o gel utilizando uma solução de SYBR green (Invitrogen) durante 30-60 minutos com agitação à temperatura ambiente.
- Imagem do gel e comparar tamanhos de L1/R1 com L2/R2 e L3/R3. Amostras L2/R2 e L3/R3 deverá mostrar bandas de um tamanho aumentado em relação à L1/R1 indicando poliadenilação bem sucedido na (Figura 2).
- Determinar a concentração de ARNm usando um espectrofotômetro.
- Preparar alíquotas de mRNA para microinjeções misturando nuclease esquerda e mRNA nuclease direito na proporção de 1:1. Nós recomendamos preparar aliquotas com uma concentração total de 200 ng / mL (100 ng / mL de cada um de nuclease), diluindo com tampão de microinjecção. Alíquotas de mRNA armazenar a -80 ° C.
4. O isolamento de embriões e microinjeção
- O isolamento de embriões. Este protocolo tem sido usado com sucesso com C57BL/6J e BDF1 tensãos e pode muito provavelmente ser adaptado para outras estirpes vulgarmente utilizadas em experiências de micro-injecção, tais como FVB ou CBF1. Os ratinhos são alojados sob um ciclo de luz-escuro de 12 h-12 h em um e-temperatura facilidade com humidade controlada com alimento e água ad libitum.
- Superovulate fêmeas dadoras para aumentar o rendimento de embriões. 16 do sexo feminino (4 semanas de idade) são superovuladas por intraperitoneal (ip) de injeção de gonadotrofina sérica de 5 UI égua prenhe (PMSG), seguida pela injeção ip de 5 UI de gonadotrofina coriônica humana (hCG) 48 horas mais tarde.
- Companheiro das 16 fêmeas superovuladas aos 16 anos de criação (2-8 meses) machos imediatamente após a injeção de hCG.
- Sacrificar as fêmeas usando um protocolo de eutanásia aprovada como inalação de CO 2.
- Recuperar os oócitos fertilizados. Os oócitos são coletados de trompas na manhã seguinte e, em seguida, libertado de todas as células cumulus restantes por um tratamento de 3-5 min em 0,1% hialuronidase bovino dissolvido em meio M2. Dependendo do desempenho do acasalamento e a estirpe utilizada na produção de embriões pode variar. 16 fêmeas C57BL/6J costumam produzir 150-250, enquanto BDF1 produzir 300-400 oócitos fertilizados, respectivamente.
- Embrião microinjeção
- Injetar o mRNA nuclease. Oócitos palco pronuclear normalmente são injetados no início da tarde. Microinjecção de ARNm citoplásmico é realizada em meio M2 sob óleo mineral utilizando um microscópio invertido equipado com Nomarski DIC usando objectivo de 20X e com micromanipuladores de embriões, assim como uma unidade de injecção. Recomendamos início das injecções de TALEN ARNm a uma concentração de 10 ng / mL (20 ng / mL no total).
- Aspirar o ovócito com um capilar de exploração e injectar o ARNm da nuclease no citoplasma do embrião evitando o contacto com o pró-núcleos. A injecção deve ser rasa, perto da membrana plasmática. O volume de injecção deve ser mantido baixo ea agulha microinjeção deve ser sagacidadehdrawn ao primeiro sinal de distensão citoplasmática. Uma série microinjecção consistirá normalmente 100-300 oócitos. Tipicamente, pelo menos, 80-90% dos embriões devem sobreviver a injecção, sem lise imediato.
- Seguindo microinjecção, colocar os embriões durante 1 hora em M16 (Sigma), meio a 37 ° C e 5% de CO 2 e transferir os embriões sobreviventes em mães adoptivas pseudo-.
5. Transferência de Embriões Cirúrgica
- Prepare receptoras de embrião pseudográvidas (mães adotivas) um dia antes da microinjeção mRNA. As fêmeas maduras (3-6 meses de idade) de uma estirpe outbred robusto, como CD-1, são adequados para o papel. Induzir pseudociese por acasalamento as fêmeas em um dia anterior com machos ou cirurgicamente ou geneticamente vasectomizados 28. Somente 0,5 fêmeas DPC com um plug copulatory claro são usados como recipientes embriões. Nas fêmeas não utilizados a pseudociese desaparece após cerca de 3 weeks permitindo seu uso repetido.
- Anestesiar os 0,5 dpc adoptivos fêmeas mãe por injecção ip de cetamina, xilazina (120 mg / kg e 16 mg / kg, respectivamente). Esta formulação garante ~ 30 min de tempo de tolerância cirúrgico é mais do que suficiente para que o tempo de operação projectada de 5-10 min.
- Posicione o animal anestesiado em seu abdômen em uma superfície quente e desinfetar a área de incisão com um desinfectante adequado, como álcool 70% ou clorexidina e mistura álcool.
- Incisar a pele no dorso do animal e abrir a cavidade peritoneal com uma tesoura esterilizada.
- Visualizar e externalizar o corno uterino, puxando a almofada de gordura ligada ao ovário. Mantenha os órgãos úmido quente usando solução de NaCl 0,9%. Tenha em mente que o local da cirurgia pode ter que ser coberto com curativo estéril ou raspada, a fim de estar em conformidade com as orientações veterinárias locais.
- Imobilizar os órgãos reprodutivos grampeando a almofada de gordura ovário com uma braçadeira bulldog.
- Gentilmente segure o oviduto logo acima da ampola com uma pinça relojoeiro e usar uma agulha hipodérmica 30 G para criar um pequeno buraco na parede do oviduto.
- Retire a agulha e insira novamente um capilar de vidro fino, contendo os embriões para o buraco permitindo a colocação dos embriões na ampola, soprando suavemente no bocal do suporte do capilar. Retirar o tubo capilar, uma vez que os embriões são depositados na ampola e substituir os órgãos reprodutivos de volta na cavidade do corpo.
- Feche a cavidade peritoneal com uma série de suturas estéreis (Prolene 6-0).
- Fechar a pele com agrafos de 1-2 (Autoclip 9 mm), dependendo do tamanho da abertura.
- Após a operação retornar os animais para sua gaiola casa e supervisioná-los até que o efeito da anestesia desgastar.
- A fim de minimizar o stress, abrigar os ratos em grupos sociais estáveis (2-4 animais / gaiola do tipo III) sempre que possível.
- Aplicar analgésico pós-operatórios, sob a forma de Dafalgan adicionado à água de beber (Paracetamol 200 mg / kg de peso corporal) durante 3 dias após a cirurgia.
6. Análise de Fundadores por PCR e T7 Endonuclease ou Restrição Enzimática Digestão
- Conceber iniciadores para amplificar uma região entre 200-700 pb em torno do local de ligação da nuclease. As distâncias da região espaçadora iniciador-nuclease para a frente e nuclease espaçador iniciador região inversa deve ser suficientemente diferente para permitir a detecção de duas bandas separadas de produtos digeridos de um gel de agarose (ver Figuras 3 e 4, para exemplos).
- Execute PCR com condições otimizadas. Verifique PCR tamanho amplicon em gel de agarose.
- Opcional: Purificar produtos de PCR, por exemplo, com o Kit QIAquick PCR Purification para remover os sais e os nucleótidos a partir da mistura de PCR. Muitos enzimas de restrição são activos em amostras de PCR complementadas com o tampão de enzima de restrição adequado e purificação não é necessária. Temos também successfuutilizado lly endonuclease de T7 em tampão de PCR QiagenTaq complementado com NEBuffer 2.
- Ensaio endonuclease T7. Misturar 17 ul de produto de PCR com 2 pi de NEBuffer 2 e executar a formação de heteroduplex (Figura 3b) programa no termociclador:
95 ° C 2 min
95 ° C a 85 ° C (-2 ° C / seg)
85 ° C a 25 ° C (-0,1 ° C / seg)
4 ° C preensão.
Adicionar 1 ml de T7 endonuclease para cada amostra e incubar a 37 ° C durante 20 min. (O ensaio de agrimensor (Transgenomic), que se baseia na CEL uma nuclease, também podem ser usados aqui. Favor consulte as instruções do fabricante.) - Restrição resumo de produto da PCR. Misturar 17 ul de produto de PCR com 2 pi de tampão de enzima de restrição de 10x e 1 ul da enzima de restrição. Digerir à temperatura apropriada, durante 1 hora ou mais.
- Ensaio endonuclease T7. Misturar 17 ul de produto de PCR com 2 pi de NEBuffer 2 e executar a formação de heteroduplex (Figura 3b) programa no termociclador:
- Adicionar corante de carga de ADN de amostras e corrido num gel de agarose a 2% para detectar digestão distintapadrões para animais do tipo selvagem e fundadores com alelos mutantes. Veja as Figuras 3 e 4 para os resultados esperados.
- Produtos Clone PCR dos fundadores positivos para alelos mutantes para seqüenciamento Sanger (por exemplo, por TA-clonagem em pGEM-T Easyor sequenciar diretamente misturas de produtos de PCR utilizando sequenciamento de última geração.
Resultados
Foram construídos plasmídeos de destino compatível com o conjunto de Ouro GateTALEN publicado por 13 Cermak et al. Que permitem a expressão de TALENS em células de mamíferos, bem como a síntese in vitro de ARNm a partir do promotor do fago T7 (Figura 1C). Estes plasmídeos carregam domínios FokI heterodiméricos (ELD ou KKR mutações) que foram mostrados para reduzir fora do alvo efeitos relativos ao homodímeros FokI e aumentar a atividade clivagem em relação a primeira geração FokI heterodímeros 25,29. As reacções de montagem Golden Gate # 1 e # 2 são geralmente muito eficiente e cada colónia branca, quando analisados por PCR de colónias, mostra o padrão esperado para o número particular de RVDS clonados (Figuras 1B e 1D).
A análise em gel de ARNm sintetizada in vitro (Figura 2) deverá revelar uma única banda distinguíveis com pouca ou nenhuma mancha para cada amostra analisada. Deve haver uma mudança clara de tamanho entre amostras L1/R1 e L2/R2, L3/R3, que indica poliadenilação sucesso.
Animais fundadores podem ser rastreados para alelos mutantes induzidos por NHEJ genotipagem utilizando PCR seguido por digestão com endonuclease de T7 (Figura 3) ou de uma digestão de restrição com uma enzima que cliva a sequência de tipo selvagem na região espaçadora do par de nuclease (Figura 4). O ensaio de T7 endonuclease é aplicável a qualquer tipo de mutação, independentemente da sequência genómica específica dentro da região espaçadora do par nuclease injectado; no entanto, ele detecta apenas desemparelhamentos entre cadeias de ADN de produtos de PCR de heteroduplex. Assim, no caso raro que um fundador carrega dois alelos mutantes de forma idêntica, os produtos de PCR que não mostram qualquer padrão de digestão T7. Essa distinção é, no entanto, sempre possível quando um local específico de restrição está localizado no interior da região espaçadora da nuclease que irá ser eliminado por NHEJInduzida por inserções / eliminações (Figura 5). Aqui, as bandas não digeridos indicam a presença de mutações e a ausência de quaisquer produtos digeridos sugere fortemente um fundador transportando mutações em ambos os alelos do gene alvo (marcado com um asterisco na Figura 4b).
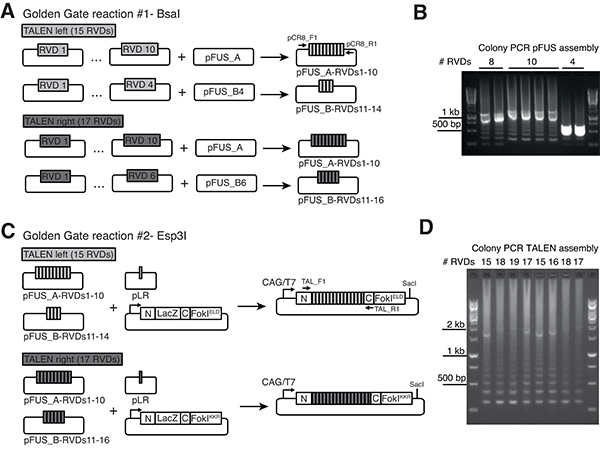
Figura 1. Golden Gate clonagem de Talen RVDS em heterodiméricos vetores pCAG-T7-Destino. A) Assembléia de matrizes RVD em vetores PFUs. Aqui é mostrado um exemplo de um par TALEN com matrizes CONTO individuais com 15 e 17 RVDS RVDS, respectivamente (para arrays CONTO mais de 21 RVDS, são necessárias três PFUs-RVDS assembléias, não apresentado). As setas indicam primers para reações de PCR colônia PFUs específicas do B). Produtos de PCRamplificados a partir de assembléias PFUs corretas normalmente mostram uma banda correspondente ao comprimento total de todos os RVDS clonados (por exemplo, cerca de 1,1 kb para 10 RVDS) e uma "escada" de menores bandas menos proeminentes devido à natureza repetitiva de matrizes RVD. C) A montagem final de matrizes PFUs-RVD e um plasmídeo contendo a última repetição (PLR) em vetores de expressão heterodiméricos Talen com FokIELD e FokIKKR variantes, respectivamente. A espinha dorsal TALEN (anotada como N e C) assemelha-se a arquitectura publicada por Miller et al. CAG 23 A (CMV elemento potenciador precoce / beta-actina de frango), o promotor permite níveis elevados de expressão em células de mamífero transfectadas, ao passo que o promotor do fago T7 permite em a síntese in vitro de mRNA (usar Saci para linearização do vector a jusante do codão de paragem da nuclease). D) Colónia de PCR utilizando os iniciadores indicados por setas em C) permite a identificação de montado correctamente TALENS. -Lengt completaprodutos h PCR são menos proeminentes, enquanto o "efeito escada" representa um indicador robusto de montagem bem-sucedida. Clique aqui para ver a imagem ampliada .
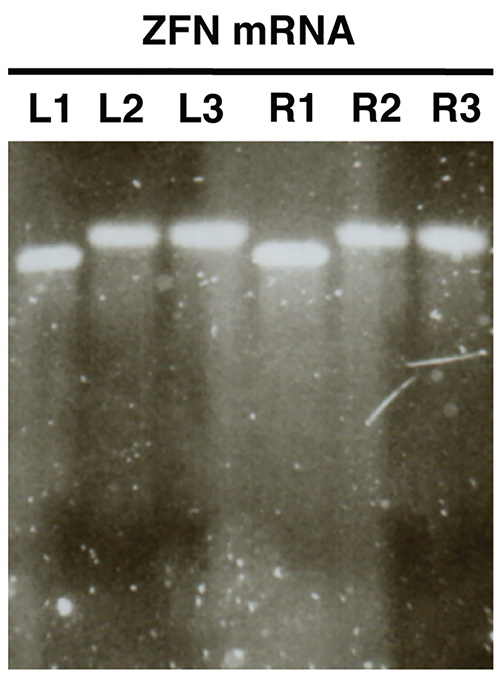
Figura 2. . Controle de qualidade de nuclease de ARNm na síntese in vitro utilizando electroforese em gel de agarose mRNAs ZFN são mostradas como um exemplo (L, deixou ZFN; R, ZFN direita). Amostras L1/R1 mostrar mRNA antes de poliadenilação, amostras L2/R2 espetáculo polyadenylated mRNA e L3/R3 mostrar mRNA poliadenilado purificada. Clique aqui para ver a imagem ampliada .
alt = "Figura 3" fo: content-width = "6 pol" fo: src = "/ files/ftp_upload/50930/50930fig3highres.jpg" src = "/ files/ftp_upload/50930/50930fig3.jpg" width = "600" />
Figura 3. Exemplo de uma digestão com endonucleases de T7 utilizados para a identificação de animais fundadores portadores de mutações induzidas por nuclease do locus alvo. A) Um par TALEN foi projetado para unir dentro da região codificadora do gene da proteína prion rato (Prnp, TALEN seqüência alvo pode ser fornecido mediante solicitação). Um produto de PCR é gerado usando um iniciador para a frente (F) localizado 110 pb a montante e um iniciador inverso (R) de 250 pb a jusante do local de clivagem TALEN. B) O produto de PCR é, subsequentemente, sujeito a formação de heteroduplex e digestão com endonuclease de T7. C) mutagénese induzida por TALEN dentro da região genómica alvo de fundadores individuais é revelada pela presença de um produto de comprimento completo por PCR com os produtos da digestão de 250 e 110 pb.50930/50930fig3highres.jpg "target =" _blank "> Clique aqui para ver imagem ampliada.

Figura 4. Exemplo de uma digestão de restrição dos produtos de PCR utilizados para a identificação de animais fundadores portadores de mutações induzidas por nuclease do locus alvo específicos. ZFN para o locus alvo rato Rosa26 29 um local de restrição Xbal dentro do intrão 1. Fundadores A) foram rastreados por PCR utilizando a genotipagem um iniciador para a frente (F), localizado a 500 bp a jusante e um iniciador inverso (R) de 250 pb a jusante do local de clivagem. B) A digestão dos produtos de PCR com Xba I revela padrões de digestão, indicando ratinhos com mutações bi-alélicos (marcados com asteriscos), mono alélicas mutações (bandas digeridas e não digeridas, por exemplo, animais 2)e camundongos em peso (digestão completa, por exemplo, animais 21). C) Seqüenciamento de camundongos com potenciais modificações bi-alélicas mostra até 3 (animais 24) distintas inserções / exclusões. Clique aqui para ver a imagem ampliada .
| Nome do Primer | Sequência de 5 'para 3' |
| pCR8_F1 | ttgatgcctggcagttccct |
| pCR8_R1 | cgaaccgaacaggcttatgt |
| TAL_F1 | ttggcgtcggcaaacagtgg |
| TAL_R2 | ggcgacgaggtggtcgttgg |
| TAL_Seq_5-1 | catcgcgcaatgcactgac |
Tabela 1. Seqüências de primers utilizados para a PCR de colônia e sequenciamento dentro do conjunto Golden Gate TALENprotocolo.
| Plasmídeo / Colecção | Contribuinte | Addgene ID | Comentários |
| Golden Gate TALEN e TAL Effector Kit 2.0 | Voytas laboratório | 1000000024 | Contém todos os plasmídeos necessários para a montagem Golden Gate TALEN |
| pCAG-T7-TALEN -KKR/ELD vetores destino | Pelczar laboratório | 40131, 40132 | Add-on plasmídeos para a expressão em células de mamíferos TALEN e na síntese de ARNm in vitro |
Tabela 2. Plasmídeos e coleções de plasmídeos necessários para a montagem Golden Gate TALEN podem ser obtidas Addgene ( www.addgene.org ).
| On-lineRecurso | Comentários |
| http://tale-nt.cac.cornell.edu | Projeto de TALEN; TALEN previsão off-alvo |
| http://zifit.partners.org/ZiFiT/ | Projeto de TALEN, ABERTO ZFN, CoDA ZFN, CRISPR/Cas9 |
| http://www.genome-engineering.org | Projeto de TALEN, CRISPR/Cas9; CRISPR/Cas9 previsão off-alvo |
| http://baolab.bme.gatech.edu/Research/BioinformaticTools/assembleTALSequences.html | Assembleia da TALEN seqüências para confirmação de resultados de sequenciamento |
| http://pgfe.umassmed.edu/ZFPmo dularsearchV2.html | Projeto de montagem modular ZFN |
| www.genomecenter.ucdavis.edu/s egallab / segallabsoftware | Projeto de montagem modular ZFN |
Tabela 3. Recursos on-line para a concepção de ZFN, TALEN e CRISPR/Cas9.
Discussão
Orientada por nuclease Designer genoma abordagens edição estenderam significativamente a gama de espécies passíveis de alterações específicas dos respectivos genomas 10,12. Em ratinhos, em células ES de direccionamento de genes tem sido uma técnica padrão para mais de duas décadas; no entanto, tem sido difícil para se adaptar às células-tronco embrionárias de outros do que o mouse espécies, embora tenha havido algum sucesso recente em ratos células-tronco embrionárias. Mesmo com a disponibilidade de "off-the-shelf" rato ES clones de células alvo de genes fornecidos por consórcios como EUCOMM, KOMP ou NorCOMM 3 genoma edição por ZFN e TALEN proporciona maior precisão e flexibilidade em relação ao espectro de modificações que podem ser introduzida no genoma do rato. Animais fundadores portadores de mutações mediada por nuclease parece ser altamente germe linha competente 4-6,20,21, o que nem sempre é o caso para as quimeras provenientes injecções blastocisto de células ES. Assim, em certos casos, a microinjecção de desnucleases Igner pode resultar em geração significativamente mais rápido de novas linhas de mouse com modificações do genoma direcionados.
A geração bem sucedida de camundongos knockout por injeção de ZFN e TALEN depende, em grande medida, da atividade do par nuclease injetado. TALENS tenham demonstrado ter uma elevada taxa de sucesso em atingir uma grande variedade de genes em vários organismos; No entanto, estudos recentes sugerem que a ligação é sensível ao TALEN metilação da citosina 30,31. Assim, os pares de nuclease recém-gerados, por exemplo TALENS clonado em vectores pCAG-T7, pode ser transfectada de forma transiente numa linha de células de rato tais como NIH-3T3 ou Neuro -2a, que imitam o estado da cromatina do embrião de rato, em certa medida. Aqui, a atividade nuclease pode ser estimada utilizando o ensaio endonuclease T7 ou uma restrição resumo de produto de PCR, como descrito na seção 5 antes da síntese de mRNA e microinjeção. Recomendamos seqüenciamento da região genômica de interesse no que diz respeitoive linha celular ea tensão do rato usado para experimentos de microinjeção.
Em zigotos murinos, diferentes pares talen ou ZFN funcionará optimamente a diferentes concentrações de mRNA e, por conseguinte, a concentração de trabalho óptima do ARNm de nuclease microinjectado podem ter que ser determinadas experimentalmente. Dependendo do par de nuclease, uma concentração demasiado baixa irá resultar em nenhuma clivagem enquanto demasiado elevada pode resultar na letalidade de embrião. Dependendo do par de nuclease, tivemos sucesso utilizando as concentrações de mRNA total de tão baixas quanto 2 ng / mL e tão alta quanto 200 ug / ul. Estes efeitos são difíceis de prever a partir de experiências em cultura de células e a concentração óptima de nuclease para a sobrevivência do embrião e taxa de alteração do locus alvo precisa de ser determinada empiricamente.
ZFN altamente ativa ou TALEN pode unir sua seqüência alvo além do estágio de uma célula do embrião microinjeção e, assim, causar padrões complexos de mutagênese umd mosaicismo em fundadores. Nós e os outros quatro têm observado três ou mais alelos mutantes distintos em um único fundador (Figura 5C). Assim, ao estabelecer uma nova linha de rato a partir desses fundadores, filhos devem ser cuidadosamente selecionados por sequenciamento para a presença da mutação favorável desde que os ensaios de digestão fornecem evidências de que apenas uma mutação indefinido está presente.
Uma das críticas frequentemente manifestadas contra os sistemas de ZFN e talen é a possibilidade de que estas nucleases são também capazes de clivagem de sequências presentes em outro lugar no genoma, que são semelhantes aos sítios alvo. Tais efeitos fora do alvo têm sido observados com reagentes primeira geração usando o domínio homodimérico FokI e construções heterodímero foram projetados para aliviar os efeitos fora do alvo 25. Locais fora do alvo potencial podem ser previstos, em certa medida in silico 32,33 e rastreados por PCR e sequenciação. Uma vantagem óbvia of usando ZFNs TALENS e para a geração de ratinhos em vez de linhas de células é a possibilidade de remoção de fora mutações alvo não ligados a modificação do genoma desejado realizando retrocruzamentos a uma estirpe de tipo selvagem de escolha. Para a análise de um grande número de ratinhos progenitores, a próxima geração de profundidade sequenciação dos produtos de PCR gerados a partir do locus alvo de nuclease e in silico previsto fora do alvo loci pode oferecer uma leitura qualitativa e quantitativa alternativa aos ensaios de digestão dos produtos de PCR.
As técnicas de reprodução assistida descritos neste protocolo são otimizados para cepas padrão de mouse usados para experimentos de microinjeção, como C57Bl/6J ou B6D2F1. Ratos de diferentes origens, como cepas outbred, pode, em princípio, ser utilizada para abordagens edição do genoma e pode fornecer um fundo genético mais adequado para questões de pesquisa específicas. O desempenho de técnicas de reprodução assistida, como a superovulação pode ser predicted para um número de estirpes de 34-36, mas pode requerer mais de optimização para estirpes diferentes do padrão, a fim de se obter um número suficiente de embriões para nuclease microinjecção.
Para além ZFN e TALEN, novas nucleases grife como o sistema CRISPR/Cas9 guiado RNA-9,37,38 foram agora introduzidas para aplicações de edição de genoma. Todos os métodos para a microinjecção e análise de animais fundadores aqui descritas são também aplicáveis aos CRISPR/Cas9 e modos futuros de edição genoma.
Divulgações
Os autores declaram não haver interesses conflitantes.
Agradecimentos
Gostaríamos de agradecer a Monika Tarnowska, Cornelia Albrecht, e Ewa Skoczylas pela excelente assistência técnica. Este estudo foi financiado pela SNF Sinergia concessão CRSI33-125073 para PP.
Materiais
| Name | Company | Catalog Number | Comments |
| BsaI | NEB | R0535S or L | |
| Esp3I | Thermo Scientific | ER0451 | |
| T4 Ligase | NEB | M0202S or L | |
| Spectinomycin | Sigma | S0692-1ML | |
| Ampicillin | Sigma | A0166 | |
| X-Gal | Sigma | B4252 | |
| IPTG | Sigma | I6758 | |
| Plasmid-Safe nuclease | Epicentre | E3101K | |
| QIAprep Spin Miniprep Kit | Qiagen | 27106 | |
| QIAGEN Plasmid Midi Kit | Qiagen | 12143 | |
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| mMESSAGE mMACHINE T7 Ultra Kit | Invitrogen | AM1345 | |
| NucAway Spin Columns | Invitrogen | AM10070 | |
| RNaseZAP | Sigma | R2020-250ML | |
| NorthernMax Formaldyde Load Dye | Invitrogen | AM8552 | |
| RNA Millennium Markers | Invitrogen | AM7150 | |
| 10x TBE buffer | Thermo Scientific | B52 | |
| T7 endonuclease | NEB | M0302S or L | |
| pGEM-T EasyVector System I | Promega | A3600 | |
| SYBR Green I Nucleic Acid Gel Stain | Invitrogen | S-7563 | |
| pregnant mare's serum gonadotrophin (PMSG) | Sigma | G4877 | |
| human chorionic gonadotropin (hCG) | Sigma | CG5 | |
| M2 embryo culture medium | Sigma | M7167 | |
| M16 embryo culture medium | Sigma | M7292 | |
| Mineral oil, embryo tested | Sigma | M8410 | |
| Ketamine | CentraVet | Ket 201 | |
| Xylazine | Sigma Aldrich | 46995 | |
| Equipment/Tools | |||
| Inverted microscope with Nomarski DIC optics (for example Nikon Eclipse TE200) | Nikon | ||
| Micromanipulator units (for example Narishige, NT88NF) | Narishige | ||
| Embryo holding capillaries | Sutter Instruments | B100-75-10 | |
| Embryo injection capillaries | Narishige | GD-1 | |
| Capillary puller (for example Sutter P97) | Sutter Instruments | ||
| Microforge (for example Narishige MF-900) | Narishige | ||
| Walton skin scissors | FST | 14077-10 | |
| Surgical scissors | FST | 14041-10 | |
| Surgical probe | FST | 10140-03 | |
| Reflex wound clip system (9 mm) | FST | 12031-09 | |
| Reflex wound clips (9 mm) | FST | 12032-09 | |
| Dumont fine forceps 5 | FST | 11254-20 | |
| Moria curved forceps | FST | 11370-31 | |
| Moria fine forceps | FST | 11399-80 | |
| Dietrich bulldog clamp | FST | 18038-45 | |
| C57BL/6J mice | Jackson Labs | strain code 000664 | |
| CD-1 mice | Charles River | strain code 000664 | |
Referências
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat. Rev. Genetics. 6, 507-512 (2005).
- Johansson, T., et al. Building a zoo of mice for genetic analyses: a comprehensive protocol for the rapid generation of BAC transgenic mice. Genesis. 48, 264-280 (2010).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Carbery, I. D., et al. Targeted genome modification in mice using zinc-finger nucleases. Genetics. 186, 451-459 (2010).
- Meyer, M., de Angelis, M. H., Wurst, W., Kuhn, R. Gene targeting by homologous recombination in mouse zygotes mediated by zinc-finger nucleases. Proc. Natl. Acad. Sci. U.S.A. 107, 15022-15026 (2010).
- Cui, X., et al. Targeted integration in rat and mouse embryos with zinc-finger nucleases. Nat. Biotechnol. 29, 64-67 (2011).
- Sung, Y. H., et al. Knockout mice created by TALEN-mediated gene targeting. Nat. Biotechnol. 31, 23-24 (2013).
- Wefers, B., et al. Direct production of mouse disease models by embryo microinjection of TALENs and oligodeoxynucleotides. Proc. Natl. Acad. Sci. U.S.A. 110, 3782-3787 (2013).
- Wang, H., et al. One-Step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat. Rev. Genetics. 11, 636-646 (2010).
- ZFN, T. A. L. E. N. CRISPR/Cas-based methods for genome engineering. Trends Biotechnol. 31, 397-405 (2013).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nat. Rev. Mol. Cell Biol. 14, 49-55 (2013).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39, (2011).
- Reyon, D., et al. FLASH assembly of TALENs for high-throughput genome editing. Nat. Biotechnol. 30, 460-465 (2012).
- Sanjana, N. E., et al. A transcription activator-like effector toolbox for genome engineering. Nat. Protoc. 7, 171-192 (2012).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nat. Biotechnol. 31, 251-258 (2013).
- Schmid-Burgk, J. L., Schmidt, T., Kaiser, V., Honing, K., Hornung, V. A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes. Nat. Biotechnol. 31, 76-81 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 31, 827-832 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat. Biotechnol. 31, 822-826 (2013).
- Hermann, M., et al. Evaluation of OPEN zinc finger nucleases for direct gene targeting of the ROSA26 locus in mouse embryos. PLoS ONE. 7, (2012).
- Meyer, M., Ortiz, O., Hrabe de Angelis, M., Wurst, W., Kuhn, R. Modeling disease mutations by gene targeting in one-cell mouse embryos. Proc. Natl. Acad. Sci. U.S.A. 109, 9354-9359 (2012).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat. Biotechnol. 29, 143-148 (2011).
- Guo, J., Gaj, T., Barbas, C. F. Directed evolution of an enhanced and highly efficient FokI cleavage domain for zinc finger nucleases. J. Mol. Biol. 400, 96-107 (2010).
- Doyon, Y., et al. Enhancing zinc-finger-nuclease activity with improved obligate heterodimeric architectures. Nat. Methods. 8, 74-79 (2011).
- Wefers, B., et al. . Current Protocols in Mouse Biology. , (2011).
- Wefers, B., Wurst, W., Kühn, R. . Current Protocols in Mouse Biology. , (2011).
- Haueter, S., et al. Genetic vasectomy-overexpression of Prm1-EGFP fusion protein in elongating spermatids causes dominant male sterility in mice. Genesis. 48, 151-160 (2010).
- Perez-Pinera, P., Ousterout, D. G., Brown, M. T., Gersbach, C. A. Gene targeting to the ROSA26 locus directed by engineered zinc finger nucleases. Nucleic Acids Res. 40, 3741-3752 (2012).
- Bultmann, S., et al. Targeted transcriptional activation of silent oct4 pluripotency gene by combining designer TALEs and inhibition of epigenetic modifiers. Nucleic Acids Res. 40, 5368-5377 (2012).
- Valton, J., et al. Overcoming TALE DNA binding domain sensitivity to cytosine methylation. J. Biol. Chem. 287, 38427-38432 (2012).
- Cradick, T. J., Ambrosini, G., Iseli, C., Bucher, P., McCaffrey, A. P. ZFN-Site searches genomes for zinc finger nuclease target sites and off-target sites. BMC Bioinform. 12, (2011).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Byers, S. L., Payson, S. J., Taft, R. A. Performance of ten inbred mouse strains following assisted reproductive technologies (ARTs). Theriogenology. 65, 1716-1726 (2006).
- Luo, C., et al. Superovulation strategies for 6 commonly used mouse strains. J. Am. Assoc. Lab. Animal Sci. 50, 471-478 (2011).
- Pease, S., Saunders, T. L. . International Society for Transgenic Technologies. Advanced protocols for animal transgenesis : an ISTT manual. , (2011).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339, 819-823 (2013).
- Mali, P., et al. RNA-Guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados