Method Article
デザイナーヌクレアーゼを用いてマウスゲノムエンジニアリング
要約
このようなジンクフィンガーヌクレアーゼ(ZFNを)および転写活性化因子のようなエフェクターヌクレアーゼ(TALENS)などのデザイナーヌクレアーゼは非相同エンド入社(NHEJ)と相同組換え(HR)経路の両方をトリガすることによって、マウスの着床前胚のゲノムを変更するために使用することができます。これらの進歩は、正確な遺伝子改変マウスの迅速な生成を可能にする。
要約
サイト固有のゲノム修飾(ノックアウト、ノックイン)を有するトランスジェニックマウスは、複雑な生物学的システムを解剖するためにだけでなく、ヒト疾患をモデル化し、治療戦略をテストするために極めて重要である。サイトのためのそのようなジンクフィンガーヌクレアーゼ(ZFNは)、デザイナーヌクレアーゼ、転写活性化因子のようなエフェクターヌクレアーゼ(TALENS)、およびクラスタ化され、定期的に互いに間隔を短くパリンドローム反復(CRISPR)/ CRISPR関連(CAS)9システムの使用の最近の進歩特定のゲノム工学は、胚性幹(ES)細胞技術に依存することなく、実質的に任意の実験室種において急速標的ゲノム改変を実行する可能性を開く。ゲノム編集の実験は、一般的に研究者が定義したゲノム遺伝子座にヌクレアーゼ活性を指示するために、カスタムDNA結合ドメインの構造が続き、目的の遺伝子の中にデザイナーのヌクレアーゼ標的部位の同定で始まる。デザイナーヌクレアーゼプラスミドは、in vitroである</ em>は受精したマウス卵母細胞のマイクロインジェクションのためのmRNAを生成するために転写した。ここでは、受精したマウス卵母細胞にTALENのmRNAの直接注入の対象となるゲノム改変を実現するためのプロトコルを提供する。
概要
マウスは、トランスジェニック動物モデルを生成するためのこれまでで最も人気のあるプラットフォームである。マウス胚1-3の遺伝子工学のための多目的なツールボックスは、最近では、ジンクフィンガーヌクレアーゼ(ZFN)、デザイナーヌクレアーゼに基づくゲノム編集アプローチによって4-6、転写活性化因子のようなエフェクターヌクレアーゼ(TALEN)を7,8に拡張されましたおよびクラスタ化され、定期的に互いに間隔を短くパリンドローム反復(CRISPR)/ CRISPR関連(CAS)9システム9。各FokIエンドヌクレアーゼ10月12日に連結されている2カスタム設計のタンパク質に基づくDNA結合ドメイン(ジンクフィンガータンパク質の配列をそれぞれ繰り返し変数をジ-残基(RVDS))のペアとしてZFNとTALEN機能。逆に、Cas9媒介DNA開裂の特異性は、CRISPR RNAをトランス活性によって提供される(また、単一のキメラRNA分子に組み合わせることができるcrRNAとtracrRNAは、ガイドRNAとも呼ばれる)11との複合体に作用するCRISPRタンパク質。
RVDSの定義された順序でTALENSは急速に13から17を選択するアセンブリ戦略の多数の個々の実験者によって構築することができる。 CRISPR/Cas9しかしガイドRNA-DNA結合の特異性は、まだ完全に18,19を解決しない、デザイナーヌクレアーゼのさらに少ない労働集約的な世代をお約束します。カスタムのZFNの生成は、これまで専門的な学術の研究室、およびこのようなサンガモBiosciences社とシグマCompoZrサービスとして、商業的な供給に限定されていた。
一般的には、デザイナーのヌクレアーゼで編集ゲノムは続いて非相同末端結合(NHEJ)または相同組換え(HR)DNA修復機械10,12を引き付ける定義されたゲノム遺伝子座、で二本鎖切断(DSB)を導入することを目指しています。 DSBのNHEJ媒介修理は、多くの場合、修理の現場に近接して挿入および欠失の導入になる。このようにNHEJリペアC遺伝子タンパク質コード配列の4,7,9内のフレームシフト変異を導入することにより標的遺伝子の機能をノックアウトするために利用される。代替的に、定義された追加又は遺伝情報の交換は、設計者ヌクレアーゼと共にDNA供与体を提供することによって達成することができる。 DNA供与体は、このようにHRによってDSB修復のための鋳型としての標的遺伝子座と相同な領域によって隣接治験設計のDNA配列を含む。両プラスミド5,6,20及び一本鎖オリゴヌクレオチド8,9,21正常ドナーとして使用されてきた。 NHEJ - ノルHR媒介ゲノム編集のいずれも、全体的な遺伝的構造を乱すことなく、塩基配列のわずかな変更を作成するためのこれらの戦略に特に適していますマウス胚のゲノム中に、選択マーカーの導入を必要とする。
このプロトコルでは、ゲノム編集のためのすべての基本的な手順を説明しますTALENSを使用して、マウスの胚。これらは、胚移植のための5)の外科的処置、受精したマウス卵母細胞へのTALEN mRNAの4)マイクロインジェクション、3)TALEN mRNAのインビトロ合成 、2)ゴールデンゲートによるTALENSの建設は13のクローンを作成、TALEN標的部位22の1)識別を含む創始動物におけるTALEN誘発される突然変異誘発、および6)分析。我々はTALEN mRNAのマイクロインジェクションとNHEJ誘発の挿入/削除の創設者のスクリーニングに焦点を当てています。この目的のために我々はプラスミドとして、マウス胚へのマイクロインジェクションのためのTALEN mRNAのin vitro合成にトランスフェクトされた場合、哺乳動物細胞の両方の発現を可能にする二官能TALEN構成物を生成した。これらの構築物は、ヘテロ二量体のFokIに融合し切り詰められた物語のバックボーン23は、哺乳動物細胞における最適なゲノム編集のために24,25のドメインからなる。このプロトコルは、他のデザイナーのヌクレアーゼのマイクロインジェクションのために、またはデザイナーのヌクレアーゼの組み合わせ注射のために採用することができるドナー構築物(DNA供与体の設計はWefers らにより優れた技術刊行物に記載されている。26,27)。
倫理的な声明
全ての動物実験は、カントンチューリッヒのカントン獣医局のガイドラインと規則に従って行った。
プロトコル
1。 TALEN標的部位の同定
- TALエフェクターヌクレオチドターゲッター2.0のWebサイト(http://tale-nt.cac.cornell.edu)を訪問し、「TALENターゲッター」を選択します。
- 標的遺伝子の配列を入力してください。のpCAG-T7-TALEN式が使用されている構築物と( 図1C)を選択し、「Miller ら 、2011、「下(最適スペーサー長を有する標的化され得る標的部位を予測するために、「プリセット·アーキテクチャを使用する」15-20 BP)、このTALENアーキテクチャの物語RVDS(15から20)の数。
- 「G代替」の「NN」を選択します(グアノシン固有のNH RVDSもご用意していますが、まだ広くTALENアセンブリをテスト)。
- オプション:他の設定やオプションについては、Webサイトの指示とあって、リンクに従ってください。
- 潜在的なターゲット部位とテキストファイルがあることができる、生成されより便利に鑑賞するための表計算プログラムにインポート。ゴールデンゲートアセンブリ用TALENペアを選択します。
- オプション:公共のデータベースでは考慮されない可能性があり、潜在的にヌクレアーゼが結合を妨げる可能性の可能な一塩基多型(SNP)を検出するためにマイクロインジェクションのために使用されるマウス系統における配列の関心のゲノム領域。
- オプション:追加のデザイナーヌクレアーゼ関連のオンラインリソースについては、表3を参照してください。
- ZFNとTALEN構築に必要なほとんどのプラスミドはAddgeneから取得できます。
http://www.addgene.org/special-collections/
ゴールデンゲートTALENキットおよび哺乳動物TALEN発現構築物のためのAddgene IDは、 表2に示す 。
あるいは、そのようなジンクフィンガードメインのような特定の機能モジュールは、遺伝子合成サービスプロバイダに注文することができる。
2。ゴールデンゲートTALEN総会
このセクションでは、サーマクらによって公開プロトコルを使用してTALENSのアセンブリを記載している。13全長TALENS二つ後続のゴールデンゲートクローニング工程( 図1)を用いて構築される。このアプローチは、最終的な発現構築物に12〜31のRVDS任意の数の組み込みを可能にする。アセンブリプロトコルは、卵母細胞マイクロインジェクション以下TALENS高活性を発現するmRNA( 図1C)を生成するために設計されたデスティネーションベクターに適合されている。また、プラスミドライブラリー(確立し、維持するためのゴールデンゲートTALENキットのオンラインプロトコルを参照してくださいhttp://www.addgene.org/TALeffector/goldengateV2/を )。
- 1日目-ゴールデンゲートの反応#1 - PFUのベクターにRVD配列の組み立て
- オプション:Pに設計された各TALENペアについてrotocol 1、各ゴールデンゲート反応のためのピペッティングスキームを生成するためにTALEN_Voytas_Pipettingスプレッドシートに( 'から3'への初期T、5を含む)2つの標的部位のDNA配列を入力する(これもアセンブリに使用される全てのプラスミドの濃度を入力する)。スプレッドシートはまた、セクション2.5.2に配列決定結果の位置合わせのために使用することができるすべてのRVDSの予想DNA配列を提供する。
- :プロトコル1で設計し、すべてのユニークなTALEN用
TALEN長は12月21日(標準)の場合は 、1から10までの繰り返し変数ジ-残基(RVDS)を選択し、デスティネーションベクターpFUS_A。残りのRVDSおよび2.3.3項では、最終的なアセンブリに追加されます、最後の繰り返し、選択RVDS 11月14日とデス ティネーションベクターpFUS_B4( 図1A)を含む15 RVDS(とTALEN 用などpFUS_Bデスティネーションベクターを、選択します。
TALEN長が22〜31の場合は 、使用してRVDS 1月10日、宛先はVEのctorのpFUS_A30A。 RVDS 11月20日とデスティネーションベクターpFUS_A30Bを選ぶ。 24 RVDSにTALEN 用など、残りのRVDSおよびpFUS_Bdestinationベクトルを、選択RVDS 21〜23とデス ティネーションベクターpFUS_B3を選択します。 - 各PFUの+ RVDの組み合わせのゴールデンゲートの反応#1を設定して、別々に、 すなわち 1から10 + pFUS_A、残りのRVDS +それぞれのpFUS_Bベクトル以上21 RVDS、1から10 + pFUS_A30A、11月20日pFUS_A30B、残りのRVDSよりTALEN用+それぞれのpFUS_Bベクトル。 20μlの全反応容量を各RVDベクター150 ngの、PFUのベクター150 ngの、1μLBsaIの、1μlのT4 DNAリガーゼ、2μlの10倍のT4 DNAリガーゼバッファー、及びH 2 Oを使用してください。 (T4リガーゼ緩衝液を繰り返し解凍/凍結が反応の効率を低下させることができるので、ゴールデンゲート·アセンブリの各ラウンドのためのT4リガーゼ緩衝液の新鮮なアリコートを使用することをお勧めします。)
- thermoサイクラーゴールデンゲート反応をPlace。
プログラム:
37℃、5分
16℃で10分
10サイクル
50℃、5分
80℃、5分 - 各ミックスに1μlの10 mMのATP及び1μlのプラスミド - セーフヌクレアーゼを加え、1時間37℃でインキュベートする。この処理は、形質転換された細菌でのインビボ相同組換えによりデスティネーションベクターにクローン化することができたリニア不完全ライゲーション生成物を除去します。
- Eを変換個々の連結反応と大腸菌 (例えばXL1-ブルーまたはDH5αなどのα-相補性を容易にエレクトロまたは化学的にコンピテント大腸菌は、ここで、後続の変換で使用することができます)。
- スペクチノ上のプレートの細菌(50μg/ ml)を青/白コロニー選択のためのX-galおよびIPTG(40μg/ mlの各)でプレート。
- 2日目-正しいのPFU-RVDS国会の確認
- プライマーpCR8_F1とpCR8_R1でコロニーPCRを使用して(プライマー配列については表1を参照してください)画面1-3ホワイト各プレートからのコロニー。正しいPFUの-RVDSアセンブリは、通常、クローン化されたすべてのRVDS(10 RVDS用などの周りの1.1キロバイト)の合計の長さに相当するバンドと小さく目立たバンド( 図1B)の「はしご」を示しています。
PCRプログラム:
95℃、3分
95℃30秒
55℃30秒
72℃で1分45秒
30〜35サイクル
72℃で10分 - 使用は(50μg/ mlのスペクチノと2〜5ミリリットルのLB)の一晩培養を開始するためにクローンを確認した。
- プライマーpCR8_F1とpCR8_R1でコロニーPCRを使用して(プライマー配列については表1を参照してください)画面1-3ホワイト各プレートからのコロニー。正しいPFUの-RVDSアセンブリは、通常、クローン化されたすべてのRVDS(10 RVDS用などの周りの1.1キロバイト)の合計の長さに相当するバンドと小さく目立たバンド( 図1B)の「はしご」を示しています。
- 3日目-ゴールデンゲートの反応#2 - TALEN発現ベクターにRVD配列
- (RVDS pFUS_AとpFUS_BまたはpFUS_A30A、pFUS_A30B、およびpFUS_Bいずれかの数に応じて)のPFU-RVDアセンブリを隔離する「ミニプレップ」を行う。
- オプション:プライマーpCR8_F1、pCR8_R1を(プライマー配列については表1を参照) を用いて配列の個々のPFUベクトル。シーケンシングにもFに行うことができますinal TALEN(セクション2.5.2)を構築。しかし、長いTALENSの完全なためサンガー配列決定を使用できない場合があり、すべてのRVDSの読み取り。
- 各単一TALENためのゴールデンゲートの反応#2を設定します。各PFUのベクター150 ngの、RVDシーケンスの設計に従ってPLR-HD、PLR-NG、PLR-NI、PLR-NN(最後の「半リピート」)の150 ngのとL EFT TALENは 75 ngのを使用するためにのpCAG-T7-TALEN-ELD-出向と右TALENは 75 NGのpCAG-T7-TALEN-KKR-宛先(またはその逆)を使用するため。 20μlの全反応容量を1μLEsp3I、1μlのT4 DNAリガーゼ、2μlの10倍のT4 DNAリガーゼ緩衝液、H 2 Oを追加します。
- サーモサイクラーにゴールデンゲート反応を置きます。
プログラム:
37℃、5分
16℃で10分
10サイクル
37°Cで15分間
80℃、5分 - Eを変換するためにセクション2.3.4からの反応を使用大腸菌 。
- <強い> 4日目 - 正しいTALEN国会の確認
- 画面1-3プライマーTAL_F1とTAL_R2でコロニーPCRによる各プレートから白色コロニーを(プライマー配列については表1を参照)。正確に組み立てTALENSは、長さが( 図1D、このバンドは「ラダー効果」が成功したアセンブリのロバストな指標を表して検出することが困難な場合がある)に組み込まRVDSの総数に対応するPCR産物を示す。
PCRプログラム:
95℃、3分
95℃30秒
55℃30秒
72℃、3分
30〜35サイクル
72℃で10分 - 使用は一晩細菌培養(2-5 100mlのμg/ mlのアンピシリンを含むLB)を開始するために、正しいクローンを確認した。
- 画面1-3プライマーTAL_F1とTAL_R2でコロニーPCRによる各プレートから白色コロニーを(プライマー配列については表1を参照)。正確に組み立てTALENSは、長さが( 図1D、このバンドは「ラダー効果」が成功したアセンブリのロバストな指標を表して検出することが困難な場合がある)に組み込まRVDSの総数に対応するPCR産物を示す。
- 5日目-正しいTALEN国会の確認
- のpCAG-T7-TALENプラスミドを分離する「ミニプレップ」を行う。
- 配列決定は、SEで実行されなかった場合ction 2.3.2は、使用プライマーTAL_Seq_5-1とTAL_R2完全長TALEN正しいRVDアセンブリを決定するために、(プライマー配列については表1を参照)。
3。ヌクレアーゼのmRNAの合成
- DNAを鋳型の生成
- 高品質のMIDIやmRNA合成のためのpCAG-T7-TALENプラスミドの大量調製(Maxiprep)を準備します。
- それを優先的に切断下流およびヌクレアーゼ終止コドン(のpCAG-T7-TALENベクトルはSacIでの使用のため)に近接した制限酵素を用いてTALENまたはZFN mRNAの合成プラスミド10μgの線形化。 mRNA合成プラスミドは、一般的に、ヌクレアーゼをコードする配列の上流にT7またはSP6ファージプロモーターなどが挙げられる。
- 完全な消化をチェックするために百分の0.7から1アガロースゲルで消化したプラスミドの200〜500 ngのを実行します。
- 室温TEMPERATURで1時間、0.1体積の酢酸ナトリウムと3容量のエタノールでDNAを沈殿させることにより消化プラスミドから塩を除去するE。 10分間14,000 XG以上での遠心分離によってDNAをペレット化、200μlの70%エタノールでペレットを洗浄、さらに5分間スピン、エタノールを除去し、空気RNaseフリー水の適切な量のペレットを再懸濁を乾燥させます。線状化プラスミドの精製は、カラムベースのシステム、 例えば、QIAクイックPCR精製キットを用いても可能である。
- リニアテンプレートの濃度を測定し、in vitro転写にセットアップするために1μgのを使用しています。
- mRNAの合成およびポリアデニル
- のpCAG-T7-TALENプラスミドのインビトロ転写のためにT7 mMESSAGEmMACHINEウルトラキットを使用しています。ヌクレアーゼフリー水で20μL、10μLT7 2X NTP / ARCA、2μlの10倍のT7反応バッファー、DNAテンプレートを1μg、2μlのT7酵素ミックスへ:氷上の各TALENための転写反応をセットアップします。反応を混合し、37℃で1〜2時間インキュベート
- 完全な20μlの書き起こしを使用ヌクレアーゼフリー水36μL、20μL5X EPAPバッファー、10μlの25 mMののMnCl、10μlの10 mMのATP:氷上のポリアデニル化反応をセットアップするためのTiON反応ミックス。混合し、(それぞれ、左右のヌクレアーゼのための)コントロールサンプルL1とR1と反応混合物の2.5μLを削除します。 E-PAP酵素の4μLを加え、37℃で45〜60分間反応をインキュベートコントロールサンプルL2とR2との反応混合物をさらに2.5μLを削除します。
- mRNAの精製
- mRNA精製(バッファー交換および取り込まれなかったヌクレオチドの除去)のためNucAwayスピンカラムを使用してください。カラムの底に乾燥ゲルを解決する列をタップします。 RNA分解酵素フリーのマイクロインジェクションバッファー(1 mMのトリス-Cl、0.1 EDTA、pH7.5)を650μLと水和物の列。キャップ、渦気泡をタップし、室温で5〜15分間水和。
- コレクションチューブとスピンカラムを配置過剰な間質液を除去するために2分間750×gで、4℃。コレクションチューブを捨て、1.5ミリリットルの溶出チューブに列を配置します。
- 750 XG、2分間、4℃でカラムとスピンにセクション3.2.2からの完全な反応ミックスを適用します。ヌクレアーゼmRNAは、現在のマイクロインジェクション緩衝液中に溶解する。精製サンプルL3とR3の2.5μLを削除します。
- -80℃で保存されたmRNAのマイクロインジェクションのアリコートが準備されるまでは。
- mRNAをゲル電気泳動
- 10%SDS溶液またはRNaseZAPを使用してRNA分解酵素汚染を除去するためのゲル室を清掃してください。
- 1xTBEランニングバッファー中の1%アガロースゲルを調製する。
- NorthernMaxホルムアルデヒド負荷色素の3倍量の各RNAサンプルL1/R1、L2/R2、L3/R3を混合し、65℃で15分間インキュベート
- 負荷サンプルとゲル上のRNAサイズラダー( 例えばミレニアムRNAサイズマーカー)ローディング色素がゲルの終わりに到達するまで1×TBE中10 V / cmのでゲルを実行します。
- 室温で攪拌しながら30〜60分間SYBRグリーン溶液(Invitrogen)を用いてゲルを染色する。
- 画像のゲルとL2/R2やL3/R3でL1/R1のサイズを比較。サンプルL2/R2とL3/R3( 図2)に成功したポリアデニルを示すL1/R1と比較して増加した大きさのバンドを示す必要があります。
- 分光光度計を用いてmRNAの濃度を決定する。
- 1:1の比率で、左と右のヌクレアーゼ、ヌクレアーゼmRNAを混合することにより、マイクロインジェクションのためのmRNAのアリコートを準備します。我々はマイクロインジェクション緩衝液で希釈して200 ngの/μL(各ヌクレアーゼ100 ngの/μL)の合計濃度で一定分量の準備をお勧めします。 -80℃で保存するmRNAのアリコート
4。胚の単離およびマイクロインジェクション
- 胚分離 。このプロトコルは、成功したC57BL/6JとBDF1株で使用されているsおよびおそらく一般に、FVB又はCBF1などのマイクロインジェクション実験で使用される他の株に適合させることができる。マウスは、食物と水を自由に温度と湿度が管理施設において12時間〜12時間の明暗サイクル下に収容されている。
- 胚の収率を増加させる過剰排卵ドナー雌。 (4週齢)16人の女性が48時間後に5 IUのヒト絨毛性ゴナドトロピン(hCG)の腹腔内注射に続く5 IUの妊馬血清ゴナドトロピン(PMSG)を腹腔内(IP)注射によって過剰排卵される。
- 16繁殖年齢16過排卵メスを交尾(2-8ヶ月)すぐにhCG注射後の男性が。
- このようなCO 2吸入として承認され、安楽死のプロトコルを使用して、女性を生け贄に捧げる。
- 受精卵を回復。卵母細胞を、翌朝卵管から採取し、M2培地中に溶解し、0.1%ウシヒアルロニダーゼで3〜5分間処理することにより、残りの卵丘細胞から解放さ。嵌合性能と使用した株に応じて、胚の収率が異なる場合があります。 BDF1はそれぞれ、300〜400受精卵が得られるのに対し、16のC57BL/6J雌は通常、150〜250を生成する。
- 胚のマイクロインジェクション
- ヌクレアーゼmRNAを注入します。前核期の卵母細胞は通常、午後の早い時間に注入される。細胞質のmRNAマイクロインジェクションは、20X対物レンズを用いノマルスキーDICと共に、胚のマニピュレーター並びに射出装置を備えた倒立顕微鏡を用いて鉱物油の下M2培地中で行われる。私たちは、10 ngの/μLの濃度(合計20 NG /μL)でTALEN mRNAの注射を開始をお勧めします。
- 保持キャピラリーを卵母細胞を吸引し、前核に接触しないように、胚の細胞質にヌクレアーゼmRNAを注入する。注入は、細胞膜の近くに、浅くする必要があります。注入量は低く保たれるべきであり、マイクロインジェクション針ウィットであるべきである細胞質膨満の最初の兆候でhdrawn。マイクロインジェクションシリーズは、通常、100〜300の卵母細胞で構成されます。一般的には、胚の少なくとも八十から九十%は即時溶解せずに注入を存続する必要があります。
- マイクロインジェクションに続いて、37℃でM16(シグマ)培地中で1時間の胚を配置し、5%CO 2および偽妊娠養母に生き残った胚を移す。
5。外科胚移植
- mRNAのマイクロインジェクションの前に偽妊娠胚の受信者(代理母)1日を準備します。堅牢な近交系株では、CD-1の(3-6ヶ月)成熟した女性は、役割に適しています。外科的または遺伝的に精管切除した雄28のいずれかで、前日にメスを交配させて偽妊娠を誘発する。明確な交尾プラグを持つ唯一の0.5 DPC雌を胚のレシピエントとして使用されています。未使用の女性では偽妊娠は約3おしっこの後に消えるその繰り返しの使用を可能にするksを。
- (それぞれ、120 mg / kgで、および16 mg / kg)をケタミン、キシラジンの腹腔内注射により0.5のDPC養母女性を麻酔し。この製剤を保証〜30分の手術の許容時間は5〜10分の投影動作時間のために十分ではありません。
- 温かみのある表面にその腹に麻酔した動物を置き、そのようの70%エタノールまたはクロルヘキシジンやアルコールの混合物などの適切消毒で、切開の面積を駆除。
- 動物の背中の皮膚を切開し、滅菌ハサミで腹腔を開きます。
- 卵巣に添付脂肪パッドを引っ張ることによって子宮角を視覚化し、外部化。温かい0.9%NaCl溶液を使用して湿った臓器を保管してください。手術部位は滅菌包帯で覆わまたはローカル獣医のガイドラインに準拠するためには剃らなければならない場合があることを覚えておいてください。
- ブルドッグクランプで卵巣脂肪パッドをクリッピングすることによって生殖器官を固定する。
- 優しく時計メーカーの鉗子ですぐ上流に膨大部の卵管を把握し、卵管の壁に小さな穴を作成するために30のG皮下注射針を使用しています。
- 針を撤回し、優しくキャピラリーホルダーのマウスピースに吹き込むことによって膨大部における胚の配置を可能にする穴に胚を含む薄いガラスキャピラリーを挿入します。胚は膨大部に堆積されると、毛細管を撤回し、バック体腔内の生殖器官を交換してください。
- 無菌縫合糸(プロレン6-0)一連の腹腔を閉じます。
- 開口部の大きさに応じて、1〜2創傷クリップ(オートクリップ9ミリメートル)を使用して皮膚を閉じます。
- 操作後にそれぞれのホームケージに動物を返し、脱ぎ履き麻酔の効果が得られるまで、それらを監督する。
- ストレス、家の安定した社会のグループのマウス(2-4動物/ III型ケージ)可能な限りを最小にするために。
- 術後の鎮痛剤を適用Dafalganの形でSは、手術後3日間、飲料水(パラセタモール200 mg / kg体重)に追加。
6。 PCRおよびT7エンドヌクレアーゼまたは制限酵素消化によって創設者の分析
- デザインは、プライマーヌクレアーゼ結合部位の周囲に200〜700塩基の領域を増幅した。距離順方向プライマー-ヌクレアーゼスペーサー領域およびヌクレアーゼスペーサー領域リバースプライマーは、(例については図3および図4を参照)アガロースゲル上で消化産物のための2つの別々のバンドの検出を可能にするのに十分に異なるべきである。
- 最適化した条件を用いてPCRを実行します。アガロースゲル上でPCRアンプリコンのサイズを確認してください。
- オプション:PCRミックスから塩およびヌクレオチドを除去するためにキアクイックPCR精製キットで例えば PurifyのPCR産物を、。多くの制限酵素が必要とされない適切な制限酵素緩衝液および精製により補完PCR試料において活性である。またsuccessfu持ってLLY NEバッファー2で補完QiagenTaq PCRバッファーに、T7エンドヌクレアーゼを使用していました。
- T7エンドヌクレアーゼアッセイ。 NEバッファー2の2μlのPCR産物17μLを混合し、サーマルサイクラー中でヘテロ二本鎖形成( 図3b)プログラムを実行します。
95°Cの2分
95℃〜85℃(-2℃/秒)
85℃〜25℃(-0.1℃/秒)
4℃で保持。
各サンプルに、T7エンドヌクレアーゼの1μLを加え、20分間37℃でインキュベートする。 (CEL 1ヌクレアーゼに依存している測量アッセイ(TRANSGENOMIC)も、メーカーの説明書を参照してください。ここで使用することができる。) - PCR産物の制限消化。 10X制限酵素バッファー2μlの制限酵素の1μlのPCR産物17μLを混合します。 1時間以上の適切な温度で消化する。
- T7エンドヌクレアーゼアッセイ。 NEバッファー2の2μlのPCR産物17μLを混合し、サーマルサイクラー中でヘテロ二本鎖形成( 図3b)プログラムを実行します。
- サンプルにDNAローディング色素を追加し、別個の消化を検出するために、2%アガロースゲル上で泳動変異した対立遺伝子を運ぶ野生型動物と創業者のパターン。期待される結果については、図3と図4を参照してください。
- をpGEM-T EasyorにTA-クローニングすることにより、 例えばサンガー配列決定のために変異した対立遺伝子のための肯定的な創設者のクローンPCR産物を(直接、次世代シーケンシングを使用して、PCR産物の混合物をシーケンス。
結果
私たちは、哺乳動物細胞におけるTALENSの発現ならびにT7ファージプロモーター( 図1C)からのin vitro mRNA合成を可能にするサーマクら 13によって公開ゴールデンGateTALENアセンブリと互換性のあるデスティネーションプラスミドを構築した。これらのプラスミドは、FokIエンドホモ二量体と比較してオフターゲット効果を減少させ、第一世代のFokIヘテロ25,29と比較して、切断活性を増強することが示されているヘテロ二量体のFokIドメイン(ELDやKKRの変異)を運ぶ。ゴールデンゲートアセンブリ反応#1と#2は、通常、非常に効率的であり、すべての白いコロニーは、コロニーPCRで分析したとき、クローン化されたRVDS( 図1Bおよび1D)の特定の数の予想パターンを示している。
in vitroで合成されたmRNAのゲル分析( 図2)は、分析され、すべてのサンプルはほとんどまたは全く汚れを持つ単一の識別可能なバンドを明らかにしなければならない。成功したポリアデニル化を示したサンプルL1/R1とL2/R2、L3/R3、間に明確なサイズシフトがあるはずです。
創始動物は、酵素を使用するかT7エンドヌクレアーゼ消化、続いて遺伝子型決定PCR( 図3)又は制限消化を用いNHEJ誘発突然変異した対立遺伝子についてスクリーニングすることができるヌクレアーゼ対のスペーサー領域内で切断する野生型配列( 図4)こと。 T7エンドヌクレアーゼアッセイは、注入されたヌクレアーゼ対のスペーサー領域内にかかわらず、特定のゲノム配列の変異の任意の種類に適用可能である。しかし、ヘテロ二本鎖PCR産物におけるDNA鎖間のミスマッチのみを検出する。このように、創業者2同じように変異した対立遺伝子を運ぶ稀な事象で、PCR産物は、任意のT7消化パターンを示しませんでした。特定の制限部位は、NHEJにより除去されるヌクレアーゼスペーサ領域内に位置しているとき、そのような区別は常に、しかしながら、可能である誘発の挿入/欠失( 図5)。ここで、未消化のバンドは、突然変異の存在を示し、任意の消化物の非存在下で強く( 図4bにおけるアスタリスクでマークされた)標的遺伝子の両方の対立遺伝子における変異を有する創始者を示唆している。
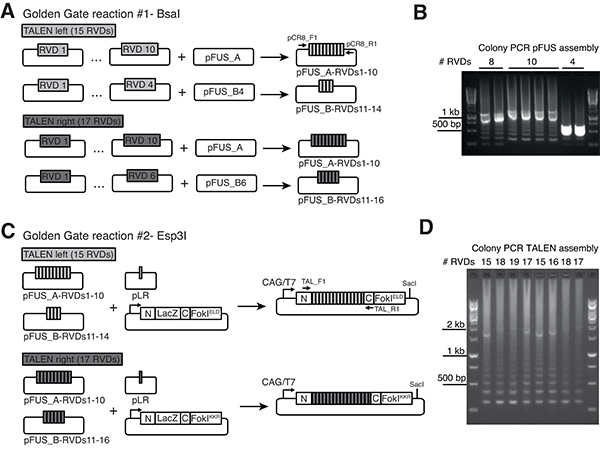
図1。 PFUのベクターへのRVDアレイのヘテロ二量体のpCAG-T7-デスティネーションベクターへTALEN RVDSのゴールデンゲートクローニング。A)アセンブリ。ここでの例は、(図示されていない、長い21 RVDSよりも物語のアレイに対して、3 PFUの-RVDSアセンブリが必要とされる)は、それぞれ、15 RVDSおよび17 RVDSを構成する個々の物語アレイとTALENペアのために示されている。矢印は、PFUの特異的コロニーPCR反応のためのプライマーを示す。B)PCR産物正しいPFUの集合体から増幅された、典型的RVDアレイの反復的な性質のためにクローン化されたすべてのRVDS(10 RVDS用などの周りの1.1キロバイト)の合計長さと小さく目立たバンドの「はしご」に対応するバンドを示す。C)最終組立PFUの-RVD·アレイと、それぞれFokIELDとFokIKKR亜種とのヘテロTALEN発現ベクターに、最後の繰り返し(PLR)を含むプラスミドの。 (NおよびCとして注釈)TALEN骨格は、T7ファージプロモーターで可能にしながら、CAG(CMV初期エンハンサーエレメント/ニワトリβ-アクチン)プロモーターが、トランスフェクトされた哺乳動物細胞中で高い発現レベルを保証するMiller ら 23によって発行アーキテクチャに類似しているインビトロ mRNA合成(ヌクレアーゼSTOPコドンの下流のベクターの線形化のためのSacIを用いる)。D)Cの矢印に示すプライマーを用いてコロニーPCR)は正確に組み立てTALENSの同定を可能にする。フルlengt「ラダー効果は「成功アセンブリの堅牢な指標を表しているH PCR産物は、多くの場合、あまり顕著である。 拡大画像を表示するには、ここをクリックしてください 。
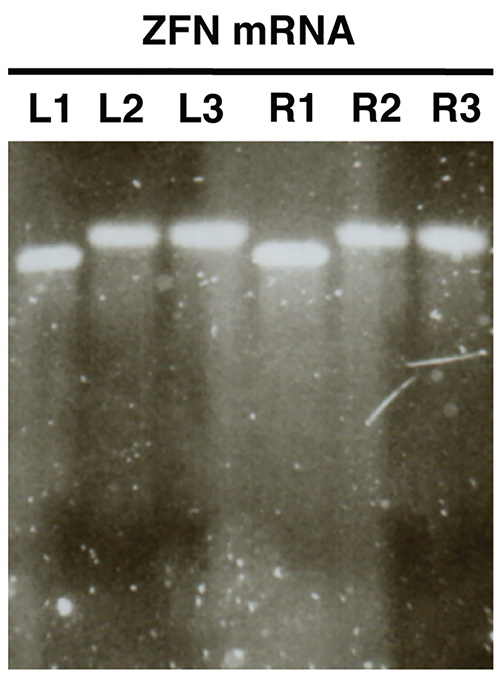
図2。アガロースゲル電気泳動を用いてインビトロ合成におけるヌクレアーゼmRNAの品質管理のZFN mRNAが例として示されている(Lは、ZFNを残し、R、右ZFN)。サンプルは前ポリアデニルにmRNAを示しL1/R1、mRNA及びL3/R3ポリアデニルサンプルL2/R2のショーは、精製されたポリアデニル化mRNAを示しています。 拡大画像を表示するには、ここをクリックしてください 。
ALT FO =「図3」:SRC = "/ files/ftp_upload/50930/50930fig3highres.jpg" SRC = "/ files/ftp_upload/50930/50930fig3.jpg"幅= "600":FOコンテンツ幅= "6インチ" />
図3。標的遺伝子座のヌクレアーゼ誘発変異を有する創始動物の同定のために使用されるT7エンドヌクレアーゼアッセイの例。 A)TALEN対は、マウスプリオンタンパク質遺伝子(PRNPのコード領域内で切断するように設計された、TALEN標的配列)は、要求に応じて提供することができる。 PCR産物を、110上流bpおよびリバースプライマー(R)TALEN切断部位の下流の250塩基対に位置するフォワードプライマー(F)を用いて生成される。B)PCR産物は、その後、ヘテロ二本鎖形成およびT7エンドヌクレアーゼ消化に供される。C)シングル創設者の目標とするゲノム領域内TALEN誘発される突然変異誘発は、250と110 BPSの消化産物との完全長PCR産物の存在によって明らかにされている。50930/50930fig3highres.jpg "ターゲット=" _blank ">拡大画像を表示するにはここをクリックしてください。

図4。標的遺伝子座のヌクレアーゼ誘発変異を有する創始動物の同定のために使用されるPCR産物の制限消化の例は 、マウスRosa26遺伝子座のためのZFNの特異的29標的XbaI制限部位、イントロン1以内A)創設者は、使用してPCR遺伝子型決定によりスクリーニングしたフォワードプライマー(F)は500上流bpおよびリバースプライマー(R)切断部位。Bの下流250 bp)をXbaIでPCR産物の消化がアスタリスクでマークされた2対立遺伝子変異を持つマウスを()を示す消化パターンを明らかにし、モノを置か対立遺伝子変異(消化し 、未消化のバンド、 例えば動物2)および潜在的な2対立遺伝子の改変によるマウスのwtマウス(完全な消化、 例えば動物21)。C)配列決定により、3(動物24)明確な挿入/欠失に表示されます。 拡大画像を表示するには、ここをクリックしてください 。
| プライマーの名前 | 'から3'配列5 |
| pCR8_F1 | ttgatgcctggcagttccct |
| pCR8_R1 | cgaaccgaacaggcttatgt |
| TAL_F1 | ttggcgtcggcaaacagtgg |
| TAL_R2 | ggcgacgaggtggtcgttgg |
| TAL_Seq_5-1 | catcgcgcaatgcactgac |
表1。ゴールデンゲートTALENアセンブリ内のコロニーのPCRおよび配列決定のために使用したプライマーの配列プロトコル。
| プラスミド/コレクション | 貢献者 | Addgene ID | 注釈 |
| ゴールデンゲートTALENと、TALエフェクターキット2.0 | Voytasラボ | 1000000024 | ゴールデンゲートTALEN組み立てに必要な全てのプラスミドが含まれています |
| のpCAG-T7-TALEN -KKR/ELDのデスティネーションベクター | Pelczarラボ | 40131、40132 | アドオン、哺乳動物細胞およびin vitro mRNA合成における TALEN発現用プラスミド |
表2。ゴールデンゲートTALEN組立に必要なプラスミド及びプラスミドのコレクションはAddgene(から入手でき www.addgene.org )。
| オンラインリソース | 注釈 |
| http://tale-nt.cac.cornell.edu | TALENの設計; TALENオフターゲット予測 |
| http://zifit.partners.org/ZiFiT/ | TALEN、OPEN ZFN、CODA ZFN、CRISPR/Cas9のデザイン |
| http://www.genome-engineering.org | TALEN、CRISPR/Cas9の設計; CRISPR/Cas9オフターゲット予測 |
| http://baolab.bme.gatech.edu/Research/BioinformaticTools/assembleTALSequences.html | TALENのアセンブリは、配列決定の結果を確認するための配列 |
| http://pgfe.umassmed.edu/ZFPmo dularsearchV2.html | モジュールアセンブリZFNのデザイン |
| www.genomecenter.ucdavis.edu/s egallab / segallabsoftware | モジュールアセンブリZFNのデザイン |
表3。 ZFN、TALEN、およびCRISPR/Cas9を設計するためのオンラインリソース。
ディスカッション
デザイナーヌクレアーゼ主導のゲノム編集アプローチが大幅にそれぞれのゲノム10,12のターゲット変更の影響を受けやすい種の範囲を拡大した。マウスでは、遺伝子標的ES細胞では、20年以上のための標準的な技術である;ラットES細胞におけるいくつかの最近の成功があったが、しかしながら、それは、マウス以外の種由来のES細胞に適応することが困難であることが証明されている。でも、このようなEUCOMM、KOMPとしてコンソーシアムが提供する「既製」遺伝子標的マウスES細胞クローンの利用可能性を持つ、またはNorCOMM 3ゲノム編集ZFNとTALENによることができる修正のスペクトルに関して、より高い精度と柔軟性を提供しますマウスゲノムに導入した。ヌクレアーゼ媒介変異を有する創始動物は、常に、ES細胞の胚盤胞の注入に起因するキメラの場合ではない、高度生殖系列有能4-6,20,21、ように見える。このように、DESの特定の場合に微量注入中ignerヌクレアーゼは、ターゲットを絞ったゲノム修飾を有する新たなマウス系統の大幅に高速生成をもたらすことができます。
ZFNとTALENの注入によるノックアウトマウスの正常な発生は、注入されたヌクレアーゼペアの活性に大きく依存。 TALENSは、生物の数の遺伝子の広い範囲を標的に高い成功率を有することが示されている;しかしながら、最近の研究は、TALEN結合はシトシンメチル30,31に感受性であることを示唆しているので、新たに生成されたヌクレアーゼ対は、例えばTALENSでのpCAG-T7ベクターにクローン化し、例えば、NIH-3T3または神経などのマウス細胞株に一過性にトランスフェクトすることができるある程度のマウス胚のクロマチン状態を模倣-2aは、。ここで、ヌクレアーゼ活性が従来mRNA合成およびマイクロインジェクションのセクション5で説明されるようにT7エンドヌクレアーゼアッセイまたはPCR産物の制限消化を用いて推定することができる。我々は尊敬に興味のあるゲノム領域の塩基配列決定を推奨アイブ細胞株およびマイクロインジェクション実験に使用したマウス株。
マウスの受精卵では、異なるTALENまたはZFNペアが異なるmRNA濃度で最適に動作するため、マイクロインジェクションし、ヌクレアーゼmRNAの最適な作用濃度は、実験的に決定する必要があります。高すぎる胚致死性をもたらすことができるのに対して、ヌクレアーゼペアに応じて、濃度が低すぎるのない切断が発生します。ヌクレアーゼの対に応じて、我々は、2 ngの/μlの200μgの/ ulのと同じ高程度の低い全mRNA濃度を使用して成功を収めている。これらの効果は、細胞培養および胚の生存および標的遺伝子座の変形速度の両方を経験的に決定する必要があるため、ヌクレアーゼの濃度で最適な実験から予測することは困難である。
高活性ZFNまたはTALENは顕微注入胚の1細胞期を超えて、その標的配列を切断し、このように変異誘発ANの複雑なパターンを引き起こす可能性があります創設者におけるDモザイク。我々は、他の4つのファウンダー( 図5C)3つ以上の異なる突然変異対立遺伝子を観察した。これらの創立者からの新しいマウス系統を確立するときにこのように、子孫は慎重に消化アッセイは、未定義の変異が存在するのみであるという証拠を提供するので、有利な変異の存在のために配列決定することによりスクリーニングされるべきである。
批判の一つは、頻繁にZFNとTALENシステムに対して有声音これらのヌクレアーゼはまた、標的部位に類似しているゲノム中にどこかに存在する配列を切断することができるという可能性である。このようなオフターゲット効果は、ホモ二量体のFokIドメインを使用して初期の世代の試薬 を用いて観察されており、ヘテロ二量体構築物は、オフターゲット効果25を軽減するために設計されていた。潜在的なオフターゲット部位は、 インシリコ 32,33 にある程度予測PCRおよび配列決定によりスクリーニングすることができる。明らかな利点のOfはマウスよりもむしろ細胞株を生成するためのZFNとTALENSを使用して、選択した野生型株にいくつかの戻し交配を行うことにより、所望のゲノム改変にリンクされていない標的変異をオフに除去する可能性がある。創始マウスの多数の分析のために、ヌクレアーゼ標的遺伝子座から、およびインシリコで生成されたPCR産物の次世代ディープシークエンシングは、オフターゲット遺伝子座がPCR産物の消化アッセイに別の定性的および定量的な読み出しを提供するかもしれないと予測した。
このプロトコルに記載の生殖補助技術は、C57BL/6J又はB6D2F1としてマイクロインジェクション実験のために使用される標準的なマウス系統のために最適化される。このような近交系株のような異なる起源のマウスは、原則的にゲノム編集アプローチを使用することができ、特定の研究課題に適し遺伝的背景を提供することがあります。このような過剰排卵などの生殖補助技術の性能はprediすることができます株34〜36の数CTEDが、ヌクレアーゼマイクロインジェクションのための胚の十分な数を得るために、非標準の株についてさらなる最適化が必要になる場合があります。
ZFNとTALENのほかに、そのようなRNA誘導CRISPR/Cas9システム9,37,38などの新しいデザイナーヌクレアーゼは現在、ゲノム編集アプリケーションのために導入されている。ここで説明する創始動物のマイクロインジェクションおよび分析のためのすべてのメソッドもCRISPR/Cas9ゲノム編集の将来のモードにも適用可能である。
開示事項
著者らは、競合する利害を宣言していません。
謝辞
私たちは、優れた技術支援のためのモニカTarnowska、コーネリアアルブレヒトとエワSkoczylasに感謝したいと思います。この研究は、PPへのSNF SinergiaグラントCRSI33-125073によって資金を供給された。
資料
| Name | Company | Catalog Number | Comments |
| BsaI | NEB | R0535S or L | |
| Esp3I | Thermo Scientific | ER0451 | |
| T4 Ligase | NEB | M0202S or L | |
| Spectinomycin | Sigma | S0692-1ML | |
| Ampicillin | Sigma | A0166 | |
| X-Gal | Sigma | B4252 | |
| IPTG | Sigma | I6758 | |
| Plasmid-Safe nuclease | Epicentre | E3101K | |
| QIAprep Spin Miniprep Kit | Qiagen | 27106 | |
| QIAGEN Plasmid Midi Kit | Qiagen | 12143 | |
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| mMESSAGE mMACHINE T7 Ultra Kit | Invitrogen | AM1345 | |
| NucAway Spin Columns | Invitrogen | AM10070 | |
| RNaseZAP | Sigma | R2020-250ML | |
| NorthernMax Formaldyde Load Dye | Invitrogen | AM8552 | |
| RNA Millennium Markers | Invitrogen | AM7150 | |
| 10x TBE buffer | Thermo Scientific | B52 | |
| T7 endonuclease | NEB | M0302S or L | |
| pGEM-T EasyVector System I | Promega | A3600 | |
| SYBR Green I Nucleic Acid Gel Stain | Invitrogen | S-7563 | |
| pregnant mare's serum gonadotrophin (PMSG) | Sigma | G4877 | |
| human chorionic gonadotropin (hCG) | Sigma | CG5 | |
| M2 embryo culture medium | Sigma | M7167 | |
| M16 embryo culture medium | Sigma | M7292 | |
| Mineral oil, embryo tested | Sigma | M8410 | |
| Ketamine | CentraVet | Ket 201 | |
| Xylazine | Sigma Aldrich | 46995 | |
| Equipment/Tools | |||
| Inverted microscope with Nomarski DIC optics (for example Nikon Eclipse TE200) | Nikon | ||
| Micromanipulator units (for example Narishige, NT88NF) | Narishige | ||
| Embryo holding capillaries | Sutter Instruments | B100-75-10 | |
| Embryo injection capillaries | Narishige | GD-1 | |
| Capillary puller (for example Sutter P97) | Sutter Instruments | ||
| Microforge (for example Narishige MF-900) | Narishige | ||
| Walton skin scissors | FST | 14077-10 | |
| Surgical scissors | FST | 14041-10 | |
| Surgical probe | FST | 10140-03 | |
| Reflex wound clip system (9 mm) | FST | 12031-09 | |
| Reflex wound clips (9 mm) | FST | 12032-09 | |
| Dumont fine forceps 5 | FST | 11254-20 | |
| Moria curved forceps | FST | 11370-31 | |
| Moria fine forceps | FST | 11399-80 | |
| Dietrich bulldog clamp | FST | 18038-45 | |
| C57BL/6J mice | Jackson Labs | strain code 000664 | |
| CD-1 mice | Charles River | strain code 000664 | |
参考文献
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat. Rev. Genetics. 6, 507-512 (2005).
- Johansson, T., et al. Building a zoo of mice for genetic analyses: a comprehensive protocol for the rapid generation of BAC transgenic mice. Genesis. 48, 264-280 (2010).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Carbery, I. D., et al. Targeted genome modification in mice using zinc-finger nucleases. Genetics. 186, 451-459 (2010).
- Meyer, M., de Angelis, M. H., Wurst, W., Kuhn, R. Gene targeting by homologous recombination in mouse zygotes mediated by zinc-finger nucleases. Proc. Natl. Acad. Sci. U.S.A. 107, 15022-15026 (2010).
- Cui, X., et al. Targeted integration in rat and mouse embryos with zinc-finger nucleases. Nat. Biotechnol. 29, 64-67 (2011).
- Sung, Y. H., et al. Knockout mice created by TALEN-mediated gene targeting. Nat. Biotechnol. 31, 23-24 (2013).
- Wefers, B., et al. Direct production of mouse disease models by embryo microinjection of TALENs and oligodeoxynucleotides. Proc. Natl. Acad. Sci. U.S.A. 110, 3782-3787 (2013).
- Wang, H., et al. One-Step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat. Rev. Genetics. 11, 636-646 (2010).
- ZFN, T. A. L. E. N. CRISPR/Cas-based methods for genome engineering. Trends Biotechnol. 31, 397-405 (2013).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nat. Rev. Mol. Cell Biol. 14, 49-55 (2013).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39, (2011).
- Reyon, D., et al. FLASH assembly of TALENs for high-throughput genome editing. Nat. Biotechnol. 30, 460-465 (2012).
- Sanjana, N. E., et al. A transcription activator-like effector toolbox for genome engineering. Nat. Protoc. 7, 171-192 (2012).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nat. Biotechnol. 31, 251-258 (2013).
- Schmid-Burgk, J. L., Schmidt, T., Kaiser, V., Honing, K., Hornung, V. A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes. Nat. Biotechnol. 31, 76-81 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 31, 827-832 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat. Biotechnol. 31, 822-826 (2013).
- Hermann, M., et al. Evaluation of OPEN zinc finger nucleases for direct gene targeting of the ROSA26 locus in mouse embryos. PLoS ONE. 7, (2012).
- Meyer, M., Ortiz, O., Hrabe de Angelis, M., Wurst, W., Kuhn, R. Modeling disease mutations by gene targeting in one-cell mouse embryos. Proc. Natl. Acad. Sci. U.S.A. 109, 9354-9359 (2012).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat. Biotechnol. 29, 143-148 (2011).
- Guo, J., Gaj, T., Barbas, C. F. Directed evolution of an enhanced and highly efficient FokI cleavage domain for zinc finger nucleases. J. Mol. Biol. 400, 96-107 (2010).
- Doyon, Y., et al. Enhancing zinc-finger-nuclease activity with improved obligate heterodimeric architectures. Nat. Methods. 8, 74-79 (2011).
- Wefers, B., et al. . Current Protocols in Mouse Biology. , (2011).
- Wefers, B., Wurst, W., Kühn, R. . Current Protocols in Mouse Biology. , (2011).
- Haueter, S., et al. Genetic vasectomy-overexpression of Prm1-EGFP fusion protein in elongating spermatids causes dominant male sterility in mice. Genesis. 48, 151-160 (2010).
- Perez-Pinera, P., Ousterout, D. G., Brown, M. T., Gersbach, C. A. Gene targeting to the ROSA26 locus directed by engineered zinc finger nucleases. Nucleic Acids Res. 40, 3741-3752 (2012).
- Bultmann, S., et al. Targeted transcriptional activation of silent oct4 pluripotency gene by combining designer TALEs and inhibition of epigenetic modifiers. Nucleic Acids Res. 40, 5368-5377 (2012).
- Valton, J., et al. Overcoming TALE DNA binding domain sensitivity to cytosine methylation. J. Biol. Chem. 287, 38427-38432 (2012).
- Cradick, T. J., Ambrosini, G., Iseli, C., Bucher, P., McCaffrey, A. P. ZFN-Site searches genomes for zinc finger nuclease target sites and off-target sites. BMC Bioinform. 12, (2011).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Byers, S. L., Payson, S. J., Taft, R. A. Performance of ten inbred mouse strains following assisted reproductive technologies (ARTs). Theriogenology. 65, 1716-1726 (2006).
- Luo, C., et al. Superovulation strategies for 6 commonly used mouse strains. J. Am. Assoc. Lab. Animal Sci. 50, 471-478 (2011).
- Pease, S., Saunders, T. L. . International Society for Transgenic Technologies. Advanced protocols for animal transgenesis : an ISTT manual. , (2011).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339, 819-823 (2013).
- Mali, P., et al. RNA-Guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved