Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Caracterización de Sistemas Complejos Uso del Diseño de Experimentos Enfoque: Expresión de Proteínas transitoria en tabaco como un estudio de caso
En este artículo
Resumen
Se describe un diseño de enfoque experimentos que se puede utilizar para determinar y modelar la influencia de los elementos reguladores de transgenes, los parámetros de crecimiento y desarrollo de la planta, y las condiciones de incubación en la expresión transitoria de los anticuerpos monoclonales y proteínas reportero en plantas.
Resumen
Las plantas proporcionan múltiples beneficios para la producción de productos biofarmacéuticos incluyendo bajos costos, escalabilidad y seguridad. La expresión transitoria ofrece la ventaja adicional de desarrollo a corto y tiempos de producción, pero los niveles de expresión puede variar significativamente entre los lotes, dando así lugar a preocupaciones de reglamentación en el contexto de las buenas prácticas de fabricación. Se utilizó un diseño de enfoque experimentos (DoE) para determinar el impacto de los factores más importantes, como elementos reguladores en la construcción de expresión, crecimiento de las plantas y de los parámetros de desarrollo y las condiciones de incubación durante la expresión, en la variabilidad de la expresión entre lotes. Hemos probado plantas que expresan un anticuerpo monoclonal anti-VIH modelo (2G12) y una proteína marcadora fluorescente (DsRed). Se discute la justificación de la selección de ciertas propiedades del modelo e identificar sus posibles limitaciones. El enfoque general puede ser fácilmente transferido a otros problemas debido a que los principios del modelo de unre ampliamente aplicable: selección de parámetros basada en el conocimiento, la reducción de la complejidad mediante la división del problema inicial en módulos más pequeños, la instalación del software guiado de las combinaciones óptimas de experimentos y el aumento de diseño por etapas. Por lo tanto, la metodología no sólo es útil para la caracterización de la expresión de proteínas en las plantas, sino también para la investigación de otros sistemas complejos que carecen de una descripción mecanicista. Las ecuaciones de predicción que describen la interconectividad entre los parámetros pueden ser utilizados para establecer modelos mecánicos para otros sistemas complejos.
Introducción
La producción de proteínas biofarmacéuticas en las plantas es ventajoso porque las plantas son de bajo costo para crecer, la plataforma puede ser ampliado con sólo crece más plantas, y los patógenos humanos son incapaces de replicarse 1,2. Las estrategias de expresión transitoria basados por ejemplo en la infiltración de las hojas con Agrobacterium tumefaciens proporciona beneficios adicionales ya que el tiempo entre el punto de entrega de ADN y la entrega de un producto purificado se reduce de año a menos de 2 meses 3. La expresión transitoria también se utiliza para el análisis funcional, por ejemplo, para probar los genes por su capacidad para complementar mutantes de pérdida de función o para investigar las interacciones de proteínas 4-6. Sin embargo, los niveles de expresión transitoria tienden a mostrar una mayor variación de lote a lote de los niveles de expresión en plantas transgénicas 7-9. Esto reduce la probabilidad de que los procesos de fabricación biofarmacéutica basan en wi expresión transitoriall ser aprobado en el marco de las buenas prácticas de fabricación (GMP), ya que la reproducibilidad es un atributo crítico calidad y está sujeta a la evaluación de riesgos 10. Esta variación también puede enmascarar cualquier interacción que los investigadores tienen la intención de investigar. Por lo tanto, nos propusimos identificar los principales factores que afectan los niveles de expresión transitoria en plantas y construir un modelo de predicción cuantitativa de alta calidad.
El enfoque de un factor-en-un-tiempo (OFAT) se utiliza a menudo para caracterizar el impacto (efecto) de ciertos parámetros (factores) sobre el resultado (respuesta) de un experimento 11. Pero esto no es óptima debido a que las pruebas individuales (corre) durante una investigación (experimento) estarán alineados como perlas en una cuerda a través del área potencial abarcado por los factores que se evalúan (espacio de diseño). La cobertura del espacio de diseño y por lo tanto el grado de información derivada de la experimento esbajo, como se muestra en la Figura 1A 12. Además, las interdependencias entre los diferentes factores (interacciones de factores) pueden permanecer ocultos que resulta en modelos pobres y / o la predicción de falsa Optima, como se muestra en la Figura 1B 13.
Los inconvenientes descritos anteriormente se pueden evitar mediante el uso de un diseño de experimentos de aproximación (DoE) en la que se ejecuta de un experimento se dispersan de manera más uniforme en todo el espacio de diseño, lo que significa que más de un factor se varía entre dos carreras 14. Hay diseños especializados para las mezclas, los factores (diseños factoriales) y la cuantificación de los impactos de los factores sobre las respuestas (métodos de superficie de respuesta, RSM s) 15 de detección. Además, RSM se pueden realizar como diseños-compuesto central, pero también se puede lograr de manera efectiva mediante el uso de software especializado que puede aplicar diferentes criterios para la selección de carreras. Por ejemplo, el llamado D-optimalitcriterio y seleccionará carreras a fin de minimizar el error en los coeficientes del modelo resultante, mientras que el criterio IV-optimalidad selecciona carreras que logran la varianza de predicción más bajo en todo el espacio de diseño 15,16. El RSM como se describe aquí permite la cuantificación precisa de la expresión de proteína transitoria en plantas, pero que puede ser fácilmente transferido a cualquier sistema que implica varios (~ 5-8) factores numéricos (por ejemplo, temperatura, tiempo, concentración) y unos pocos (~ 2 - 4) factores categóricas (por ejemplo, promotor, color) en el que una descripción mecanicista no está disponible o demasiado complejo para modelar.
El enfoque DoE se originó en las ciencias agrícolas, pero se ha extendido a otras áreas, ya que es transferible a cualquier situación en la que es útil para reducir el número de ciclos necesarios para obtener datos fiables y generar modelos descriptivos de procesos complejos. Esto a su vez ha dado lugar a la inclusión del DoE en la "Guía para laIndustria, Q8 (R2) Desarrollo Farmacéutico ", publicado por la Conferencia Internacional sobre Armonización de los Requisitos Técnicos para el Registro de Productos Farmacéuticos para Uso Humano (ICH) 17. DoE ahora se utiliza ampliamente en la investigación científica y la industria 18. Sin embargo, se debe tener cuidado durante la planificación y ejecución del experimento, porque la selección de un grado del polinomio impropio para el modelo de regresión lineal múltiple (modelo base) pueden presentar una necesidad de tiradas adicionales para modelar los efectos de los factores correctamente. Además, dañados o datos faltantes generan modelos incorrectos y defectuoso predicciones, y puede incluso prevenir cualquier intento de construcción de modelos, como se describe en el protocolo y de discusión de los artículos 18. En la sección de protocolo, que inicialmente se establecerán los pasos de planificación más importantes para un experimento basado en la RSM y luego explicar el diseño basado en el DoE software v8.1 DesignExpert. Pero diseños similares se pueden construir con otros includi softwareng JMP, Modde y STATISTICA. Los procedimientos experimentales son seguidos por instrucciones para el análisis y evaluación de datos.
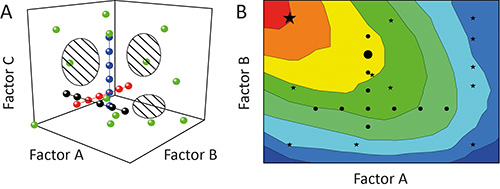
Figura 1. Comparativa de. OFAT y DoE A. Variación secuencial de un factor a la vez (OFAT) en un experimento (círculos negros, rojos y azules) logra una baja cobertura del diseño de espacio (regiones sombreadas). Por el contrario, la variación de más de un factor a la vez usando el diseño de experimentos de estrategia (DoE) (círculos verdes) mejora la cobertura y por lo tanto la precisión de los modelos resultantes. B. La cobertura de espacio de diseño sesgada significa que los experimentos OFAT (círculos negros) también pueden fallar para identificar regiones óptimas de operación (rojo) y predecir soluciones sub-óptimas (gran círculo negro), mientras que el DoE strategies (estrellas negras) son más propensos a identificar condiciones preferentes (gran estrella negro).
Protocolo
1. Planificación de una estrategia DoE
- Identificar los factores y las respuestas pertinentes para su inclusión en el diseño.
- Definir una o varias respuestas para la medición. Aquí, se utilizaron niveles de expresión 2G12 y DsRed (g / ml), incluyendo la diferencia mínima detectable considerado como relevantes (10 y 20 mg / ml, respectivamente) y un valor aproximado para la desviación estándar estimada del sistema (4 y 8 mg / ml, respectivamente) sobre la base de los experimentos anteriores.
- Utilizar la literatura disponible, los datos de los experimentos previos o diseños especializadas de selección (por ejemplo, un diseño factorial, ver la introducción) para seleccionar factores importantes cuyo impacto en las respuestas que se cuantificarán (Tabla 1) 7,8,19,20.
- Asignar los tipos de factores (numéricas o categóricas) y seleccionar rangos en los que se variaron los factores numéricos durante la investigación del Departamento de Energía (Tabla 1).
- Identificar numeric factores para los que la variación continua es difícil de implementar.
- Evitar el uso de variación continua de factores que no se pueden ajustar de manera precisa a un cierto nivel. Por ejemplo, temperatura de incubación típicamente se puede controlar sólo dentro de ± 2 ° C, por lo tanto, la variación continua utilizando los valores tales como 27,2 ° C, 25,9 ° C, y 29,3 ° C debe ser evitado.
- En su lugar, seleccione un número de niveles discretos de estos factores (Tabla 1). El número de niveles debe coincidir con el modelo base anticipada (paso 1.2). Esto sólo es importante para los diseños óptimos, porque los diseños centrales compuestos siempre tienen niveles de factor discretos.
- Asignar si un factor categórico es nominal, es decir, sin ningún orden implícito (por ejemplo, diferentes fabricantes), o ordinal y por lo tanto un parámetro numérico discreto (por ejemplo hojas diferentes en una planta).
- Seleccione los niveles de ambos tipos de factores categóricos.
- Seleccione un modelo de base útil.
- Sobre la base de los experimentos y la literatura preliminares, anticipar la relación entre cada factor y la respuesta, así como las interacciones de los factores y la respuesta. Por ejemplo, lineal (cuanto más mejor), cuadrática (single aumento / disminución óptima o no lineal) o cúbica (sesgado óptima).
- Asegúrese de que el número de niveles de factores numéricos discretos es n + 1, siendo n el grado del polinomio de la relación entre el factor y la respuesta. Por ejemplo, se espera que si la temperatura para tener un efecto cuadrático en la respuesta, n = 2 y por lo tanto al menos tres niveles de temperatura debe ser investigado para conseguir un ajuste para el efecto cuadrático.
- Definir la fracción de espacio de diseño (FDS) para los que la varianza de predicción debe estar por debajo de un cierto umbral (nivel alfa). El FDS debe ser> 0,95 (cubrir más del 95% del espacio de diseño) para lograr modelos que producen predicciones sólidas a lo largotodo el 21-23 de espacio de diseño.
Nota: El umbral de 0,05 corresponde a un nivel de significación del 5% (error tipo I) y es un valor típico. Al aumentar este valor se reducirá el número de experimentos necesarios para una estrategia DoE pero aumentará simultáneamente la probabilidad de que los efectos significativos se puede perder y que las estimaciones de su impacto en la respuesta será incorrecta.

Tabla 1. Factores que afectan a la expresión de proteína transitoria en tabaco, incluidos la variación oscila durante el DoE. Factores en negrita sólo se incluyeron en el diseño de los experimentos descritos en "Un modelo descriptivo para la acumulación de DsRed durante la expresión transitoria utilizando diferentes promotores / 5'UTRs" mientras que los factores en cursiva Sólo se incluyeron en el diseño para "Optimización incubation condiciones y regímenes de cosecha para la producción de anticuerpos monoclonales en las plantas utilizando la expresión transitoria ".
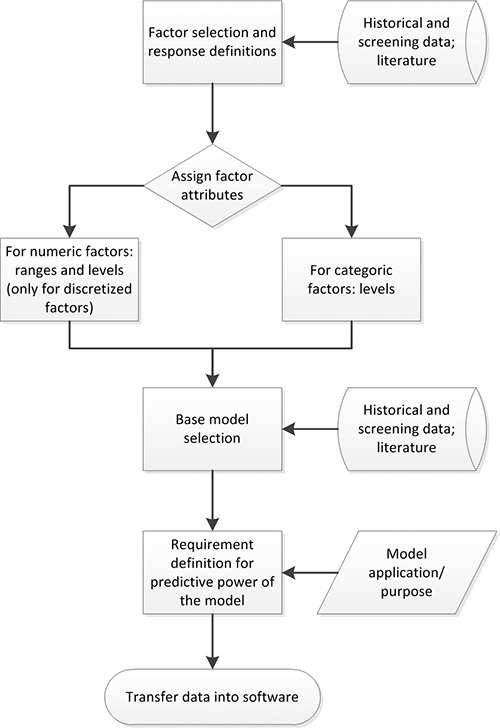
Figura 2. Proceso de planificación del Departamento de Energía. Factores con un impacto significativo en la respuesta bajo investigación son seleccionados sobre la base de los datos disponibles. A continuación se les asigna atributos de factores (por ejemplo, numéricos), rangos y niveles. Conocimientos y experimentos anterior se utilizan para definir un modelo de base adecuada. Los requisitos de potencia de predicción se definen sobre la base de la aplicación / propósito del modelo final. Los datos recopilados pueden ser transferidos al software DoE apropiado.
2. La creación de un RSM en DesignExpert
- Comience DesignExpert y selecciona "Nuevo Diseño". En la ventana de "Respuesta de la superficie", seleccione y# 34; Optimal "e introduzca el número de factores numéricos y categóricos seleccionados en la sección 1.1).
- Introduzca los nombres de los factores, las unidades, los tipos, subtipos, el número de niveles / fronteras y valores de los niveles de nivel para todos los factores en los campos correspondientes.
- Continuar a la página siguiente y en "Buscar" Opciones seleccione "mejor", así como el criterio de la "optimalidad" deseada, aquí "-D óptimo".
- En el menú "Editar modelo de ...", seleccione el modelo que contiene los factores y las interacciones previstas en la sección 1.2). Si no está seguro, seleccione un modelo completo que cubre todos los factores e interacciones para un cierto grado del polinomio, aquí "cuadráticas". Tenga en cuenta que la selección de un modelo completo (que contiene todas las interacciones) puede aumentar el número de experimentos necesarios para el Departamento de Energía.
- Seleccione el número de "Bloques". Aquí, se utilizó un solo bloque, porque todas las plantas fueron del mismo lote, todas las bacterias habían sido cultivadas simultáneamente yTodas las inyecciones se llevaron a cabo en el mismo día por el mismo operador. Utilice más de un bloque, si más de un lote de plantas se utiliza o varios operadores manejar las inyecciones.
- Equilibrar el diseño
- Activar "Forzar el equilibrio categórico" con el fin de distribuir las ejecuciones del experimento de manera uniforme entre los niveles de los factores categóricos, por ejemplo, diferentes promotores que se prueban.
Nota: esto puede reducir la optimalidad del diseño a un grado que DesignExpert informará como un valor de% dependiendo de los puntos seleccionados por el algoritmo de optimalidad. - Recalcular el diseño varias veces utilizando el algoritmo de inicialización aleatorio implementado para minimizar la reducción de optimalidad.
Nota: las pérdidas en optimalidad pueden variar desde ~ 3-40% utilizando los mismos datos de entrada, pero cambiando el número de repeticiones puede ser utilizado para mantener el equilibrio categórica.
- Activar "Forzar el equilibrio categórico" con el fin de distribuir las ejecuciones del experimento de manera uniforme entre los niveles de los factores categóricos, por ejemplo, diferentes promotores que se prueban.
- El software le sugerirá un valor para el número de "puntos de modelo" basado en el modelo base, Aquí 70 carreras. Ajuste el número de carreras de "réplicas" y "Para la estimación de la falta de ajuste" para asegurar que es> 5% de la cantidad de "puntos" para cada modelo, aquí 10 carreras cada uno.
- Continuar a la siguiente página, seleccione el número de respuestas y escriba sus nombres y unidades. Respuestas adicionales también se pueden añadir en cualquier momento durante la evaluación de datos sin ningún impacto negativo en el diseño.
- Continuar para iniciar el algoritmo, el cálculo de los niveles de factores para los recorridos del DoE. Si el modelo de base seleccionada no coincide con el número de niveles de un determinado factor, una notificación aparecerá en pantalla y el cálculo no se iniciará. En este caso, ya sea:
- Aumentar el número de niveles de este factor, o
- Anule la selección de los términos en el modelo base que no puede ser calculado con base en el número actual de los niveles.
- Por ejemplo, si hay dos niveles para el factor T "tiempo de incubación" (es decir, 2 d y 5 d) en tque el diseño actual y un modelo de base cuadrática que incluye el término t 2 fue seleccionada, ya sea agregar un tercer nivel para t (es decir, 8 d), o eliminar el factor t 2 del modelo.
- Guarde la hoja de cálculo que contiene las combinaciones de factores óptimos que se muestra una vez finalizado el cálculo.
- En el nodo de "Diseño", seleccione el sub-nodo "Evaluación" y vaya a la pestaña "Gráficos".
- En la "herramienta de gráficos", seleccione "FDS" y en el cuadro "FDS Graph" elegir "Pred" como el tipo de error. A continuación, introduzca la diferencia mínima detectable, así como el valor aproximado de la desviación estándar estimada del sistema definido en la sección 1.1.1) como valores de "d" y "s", respectivamente (en este caso 20 y 8 mg / ml para DsRed). Introduzca también el nivel alfa (aceptables% faltante un efecto significativo) adecuado para la aplicación, por lo general 0,05 (5%).
- Asegúrese de que la FDS calculado coincide con el porcentaje definido en la sección 1.3) (normalmente> 0.95 para los modelos de predicción). Una curva plana en la parcela FDS es preferible, lo que indica la exactitud de predicción uniforme en todo el espacio de diseño.
- Si este no es el caso (como se encuentra aquí, FDS fue del 1%, como se muestra en la Figura 3A), vaya al nodo "Diseño" y en la barra de tareas, seleccione "Herramientas de Diseño", luego "Diseño Augment ..." y elegir la opción " Aumentar ".
- Seleccione las mismas "Buscar" y "criterios de optimalidad" como antes. También eligió el mismo "modelo Editar" y "fuerza de equilibrio categórico" ajustes. Si el aumento de diseño se lleva a cabo antes de cualquier experimento (como se hace aquí), a continuación, cambie la opción "Poner carreras en el bloque" a "Bloque1".
- En la sección "Carreras" introduzca el número de "puntos" Modelo adicionales, manteniendo al menos el 5% "réplicas" y "Para la estimación de la faltade ajuste "en el diseño total. Aquí, se añadieron 100 carreras adicionales y no" réplicas "y" Para la estimación de la falta de ajuste "fueron incluidos.
- Una vez que el cálculo se ha terminado, reexaminar la gráfica FDS como se describió anteriormente. Si el FDS todavía no es satisfactoria, repetir el aumento de diseño como se describe anteriormente. Aquí el FDS fue 100% después del aumento y por lo tanto no más lejos de aumento fue necesario (Figura 3B).

Figura 3. Comparación de parcelas FDS. Una. Una gama que consta de 90 carreras produce una FDS insuficiente de sólo 1% para el error estándar de predicción, utilizando un modelo de base cuadrática en combinación con los valores para la diferencia mínima detectable (20 g / ml) y estimaciónacoplado desviación estándar del sistema (8 g / ml). B. Aumento del DoE a un total de 210 carreras logró un FDS 100% y una curva plana que indica la precisión del modelo uniforme en todo el espacio de diseño.
3. Clonación y Análisis de Expresión Casetes
- Cultivar Escherichia coli en 5 ml de medio LB (10 g / L de triptona, extracto de levadura 5 g / l, NaCl 170 mM, 50 mg / l de ampicilina; para placas de LB-agar que incluyen 15 g / l de agar) a 37 ° C durante 6 -8 horas o toda la noche en un agitador orbital a 160 rpm. PRECAUCIÓN: ampicilina es una sustancia dañina. Inocular el medio LB, ya sea con 50 l de cultivo líquido o células de transferencia de colonias cultivadas en placas utilizando una punta de pipeta.
- Para la purificación de ADN plásmido PSO, y otros derivados de ppam (GenBank AY027531), a partir de E. coli cepa K12 DH5a, siga las instrucciones en el manual del kit de purificación de ADN 24. PRECAUCIÓN: la purificación kque contiene sustancias químicas nocivas (véase más arriba el manual para más detalles). La secuencia del plásmido se describe en la Figura 4 y por el comprador et al. 20.
- Determinar la concentración de ADN en el eluato purificado por medición de la absorbancia de una muestra de 2 l a 260 nm en un dispositivo NanoDrop.
- Confirmar la identidad del plásmido de ADN purificado por digestión con endonucleasas de restricción (RES).
- Seleccionar las FER de acuerdo con la secuencia del plásmido de manera que se generan patrones de fragmentos únicos y distinguibles. Siga las recomendaciones del fabricante para las condiciones de digestión, como el volumen de reacción, el tiempo, la temperatura y la concentración de la albúmina sérica bovina estabilizador. Dependiendo de la sensibilidad del dispositivo de análisis, utilizar 50-500 ng de ADN plásmido por la digestión.
- Preparar 0,8-2,0% en geles para la separación de fragmentos de ADN por ebullición de agarosa en tampón Tris-borato-EDTA (TBE) tampón (90 mM de Tris, borato 90 mM, EDTA 2 mM, pH 8,0). Tque más grandes los fragmentos esperados, menos de agarosa se debe utilizar en el gel.
- Añadir 5 l de tampón de muestra de cinco veces (5x SABU, 0,1% (w / v) de azul de bromofenol, 0,1% (w / v) de xileno cianol, 10% (w / v) de glicerol disuelto en TBE) a 50 l de la tratados con RE muestra de ADN y separar los fragmentos por electroforesis en gel de agarosa a 100 V durante ~ 40 min o hasta la separación clara de los fragmentos se logra. En cada gel, incluirá un carril que contiene marcadores de tamaño escalera del ADN para la comparación, por ejemplo, 2-3 l escalera de 1 kb.
- Vuelva a colocar la 5'UTR omega en PSO con uno de los otros tres 5'UTRs.
- Suelte la secuencia 5'UTR omega de ~ 4 ADN purificado PSO mg por tratamiento con 20 unidades de Eco RI-HF y suboficiales ER I-HF en NEBuffer 4 a 37 ° C durante aproximadamente 60 min. Luego separar los fragmentos como se describe en las secciones 3.4.2 y 3.4.3.
- Aislar el fragmento más grande (pS, "columna vertebral") a partir del gel de agarosa utilizando un gel adicionalkit cción de acuerdo con el manual del fabricante 25 y determinar la concentración del ADN purificado como se describe en la sección 3.3). PRECAUCIÓN: el kit de extracción de gel contiene productos químicos nocivos, consulte el manual para más detalles.
- Aislar la CHS, CHS-LPH y TL 5'UTRs de vectores de donantes adecuados mediante el tratamiento de ~ 10 mg de cada vector con 20 unidades de Eco RI-HF y cosa Nco I-HF en NEBuffer 4 a 37 º C durante aproximadamente 60 min. Luego separar los fragmentos como se describe en los apartados 3.4.2 y 3.4.3 y se purifica el fragmento 5'UTR contiene más pequeña como se describe en la sección 3.5.2.
- Se liga el 5'UTRs purificadas aisladas en alícuotas separadas para el aislado purificado vector de pS lineal a 25 ° C durante 5 min de acuerdo con las recomendaciones del fabricante 26. Utilice ~ 50 ng de ADN de vector y un exceso molar de tres veces de ADN 5'UTR en un volumen total de 20 l.
- Transformación E coli células con ªe plásmidos recombinantes 26,27.
- Añadir ~ 10 ng (~ 4.5 l) de la mezcla de unión (véase la sección 3.5.4) a 50 l RbCl E. competente coli y mezclar suavemente, y luego se incuba durante 30 a 60 minutos en hielo. El choque térmico durante 1,5 minutos a 42 ° C y enfriar en hielo durante 5 a 30 min.
- Añadir 950 l de medio LB sin antibiótico y se incuba durante 1 hora a 37 ° C y 160 rpm. Para la selección de transformantes, se extendió 50 y 100 l de la cultura en placas de agar LB que contenían ampicilina y se incuba durante ~ 16-20 horas a 37 ° C.
- Inocular 5-10 colonias separadas que representan a cada ligadura promotor-5'UTR se describe en la sección 3.1 en alícuotas de 5 ml de medio LB que contenían ampicilina. Luego de purificar el ADN plásmido y confirmar su identidad tal como se describe en las secciones 3.2 a 3.4.
- Vuelva a colocar el promotor 35SS con el promotor nos en cada uno de los cuatro plásmidos 5'UTR utilizando ER Asc I y Eco RI en NEBuffer 4 a 37 ° C durante 1 hora como se describe en las secciones 3.5 y 3.6.
- Dar a conocer a cada uno de los ocho plásmidos resultantes en A. tumefaciens cepa GV3101: pMP90RK por electroporación 28.
- Añadir ~ 500 ng de ADN plásmido purificado a 50 A. l competente tumefaciens células en hielo. Mezclar suavemente y transferir en una cubeta de 0,2 cm preenfriado electroporación. Asegúrese de que la mezcla es en la parte inferior de la cubeta y no contiene burbujas.
- Células pulso en 2,5 kV durante 5 ms y confirmar la intensidad y la duración del pulso.
- Evite elevadas concentraciones de sal en la muestra de ADN o la preparación incorrecta de la A. competente tumefaciens células, ya que pueden resultar en corrientes altas de iones causantes de la vaporización instantánea de la suspensión de células, reduciendo en gran medida la eficacia de transformación.
- Añadir 950 l de medio YEB sin antibióticos (extracto de 5 g / L de carne, extracto de levadura 1 g / l, 5 g / L de peptonaE, 5 g / L de sacarosa, 2 mM de MgSO4, pH 7,0), mezclar suavemente y transferir inmediatamente en un tubo de reacción de 1,5 ml estéril. Se incuba durante 2-4 horas a 26-28 ° C y 160 rpm.
- Corre 1-2 l en placas de agar YEB contengan antibióticos (50 mg / L carbenicilina, 25 mg / l de kanamicina, 25 mg / L de rifampicina) para la selección de transformantes. PRECAUCIÓN: rifampicina es una sustancia tóxica.
- Inocular tres alícuotas de 5 ml de medio YEB que contienen antibióticos con células procedentes de colonias separadas que representan a cada ligadura de promotor-5'UTR y se incuba durante 48-72 horas a 26-28 ° C y 160 rpm.
- Confirmar el éxito de A. tumefaciens transformación.
- Transferencia de 2 l de cada alícuota de la sección 3.8.6 para separar 48 ml de alícuotas de mezcla maestra de PCR (2 l de cada cebador 10 mM de stock (400 nM concentración final de cada cebador),1 l de mezcla de dNTP 10 mM (200 mM concentración final de cada trifosfato de desoxinucleósido), 5 l Expand 10 veces de tampón de alta fidelidad con 15 mM de MgCl 2, 0,75 l mezcla de enzima Expand High Fidelity, y 39,25 l de agua destilada estéril).
- Amplificar el casete de expresión de cada plásmido utilizando cebadores apropiados (FWD: 5'-CCT CAG GAA GAG CAA TAC-3 ', la unión 1026 nucleótidos aguas arriba del promotor; REV: 5'-CCA AAG CGA GTA AAC CAC-3', la unión dentro del sitio de poliadenilación 35S) bajo condiciones de PCR apropiados. Aquí, 94 ° C se utilizó para la desnaturalización inicial, seguido de 30 ciclos de 94 ° C de desnaturalización durante 15 s, 51 ° C recocido durante 30 seg y 72 ° C para la elongación 120 seg, y un paso de elongación final a 72 ° C durante 8 min.
- Determine el tamaño de los productos de PCR mediante electroforesis en gel de agarosa como se describe en el apartado 3.4.3.
- Preparar A. tumefaciens stocks de glicerol porla mezcla de 500 l 50% (v / v) de glicerol estéril con 500 l de A. tumefaciens culturas de la sección 3.8.6 para los que la transformación que se ha confirmado (sección 3.9.3). Guarde los stocks de glicerol a -80 º C hasta su uso posterior.
- Calcular las energías de plegado de los diferentes ARNm utilizando el servidor web RNAfold 29 e incluir la secuencia de nucleótidos desde el sitio de inicio de la transcripción a través de la primera 50 pb de la región codificante.
- En la sección "Doblar algoritmos y opciones básicas", seleccione el "mínimo de energía libre (MFE) y la función de partición" y "evitar pares de bases aisladas" opciones.
- En las "opciones de plegado avanzadas" eligen "colgando energías en ambos lados de una hélice en cualquier caso", "Parámetros de ARN (modelo de Turner, 2004)" y cambiar "reescalar los parámetros de energía a determinada temperatura (C)" a 25 ° C.
- En la sección "Opciones de salida", seleccione todas las opciones.
- A partir de los "Resultados para termodinámico de predicción por conjuntos" sección del archivo de salida, el extracto de la "energía libre del conjunto termodinámico" y la "frecuencia de la estructura MFE en el conjunto" Los valores de comparación.
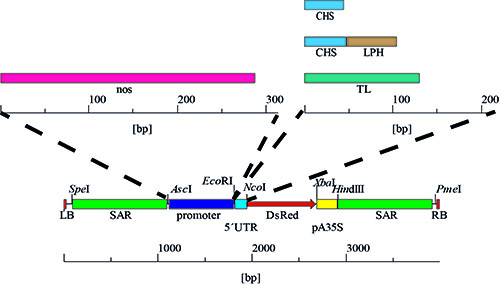
Figura 4. Promotor y 5 'UTR de variantes. Los casetes de expresión se generaron por el intercambio paso a paso de la 5'UTR, resultando en cuatro combinaciones con el promotor CaMV 35SS, seguido por la sustitución de este promotor con la secuencia nn produciendo cuatro variantes adicionales y un total de ocho combinaciones de promotor / 5'UTR diferentes.
4. Cultivo de Plantas
- Preparar una solución de 0,1% del fertilizante Ferty2 Mega en agua desionizada a pH 5,9.
- Preparar 10 bloques x 10 x 8 cm de lana de roca por una extensa lavado con agua desionizada para eliminar los residuos químicos y finalmente equilibre con fertilizante.
- Semilla de plantas de tabaco mediante la colocación 1-2 semillas de tabaco en cada bloque de lana de roca, seguido por una corta al ras de fertilizantes teniendo cuidado de evitar el lavado de las semillas de distancia.
- Germinar y cultivar las plantas de tabaco durante 42 días en un invernadero a temperatura 25/22 ° C día / noche, la humedad relativa del 70% y un fotoperíodo de 16 h (180 mmol m -2 s -1; λ = 400-700 nm) . Durante este fotoperíodo, regar con fertilizantes durante 15 min cada hora en cultivo hidropónico.
5. Protein Expression transitoria
- Preparar A. tumefaciens para la inyección en las hojas.
- Inocular 5-50 ml de medio YEB contengan antibióticos con 1% de A. tumefaciens crio-cultivo madre y se incuba a 27 ° C hasta que la DO 600 nm alcanza 5,0 (~ 48 a 72 horas, dependiendo del volumen y tipo de buque).
- Diluir la A. cultura Agrobacterium con agua y medio de la infiltración de 2 veces (sales de 4,3 g / l de Murashige y Skoog (pH 5,6), 5 g / L de sacarosa, 1,8 g / L de glucosa, acetosiringona 100 mM) para que coincida con la OD 600 nm requerido para la inyección. Confirme la OD600nm justo antes de la inyección. Nota: Una 600nm OD de 1,0 corresponde a ~ 1,43 ± 0,12 x 10 9 unidades formadoras de colonias por ml de conformación.
- Inyectar la A. tumefaciens suspensión en hojas.
- Seleccionar y etiquetar las hojas noncotyledon y posiciones al respecto a tratar (por ejemplo, de acuerdo con una estrategia DoE).
- No se inyecte diferente A. tumefaciens soluciones en el mismo campo intercostal. En lugar de ello, utilizar las secciones opuestas de cada lado del eje medio de la vena. Si más de dos soluciones diferentes necesitan ser inyectado en la misma posición de uso una additional planta.
- Agite A. solución de Agrobacterium para volver a suspender las células que se hayan depositado desde la preparación de la dilución y aspirado en una jeringuilla de 1 ml.
- Arañar suavemente la epidermis en la posición prevista para la inyección con una punta de pipeta o similar para facilitar la afluencia de la A. tumefaciens solución. Evitar la ruptura de la lámina de la hoja mientras lo hace.
- Mantenga la jeringa perpendicular a la lámina de la hoja tocar el cañón contra el campo intercostal a tratar y empuje suavemente hacia la salida (sin aguja acoplada) sobre el lado inferior de la hoja. Pulse el lado superior de la hoja con cuidado, al mismo tiempo para evitar que la lámina de la hoja se desplace o se rompa.
- Presione suavemente el émbolo de la jeringa. El A. solución de Agrobacterium entrará en los espacios intercelulares dentro de la lámina de la hoja como se indica por las zonas tratadas que aparecen verde más oscuro y húmedo. Repita este procedimiento en varias posiciones hasta que todo el intercostal campo se infiltró con A. tumefaciens. Luego continúe con el siguiente campo intercostal.
- Asegúrese de que la jeringa se mantiene perpendicular a la hoja. La inclinación de la jeringa hará que la suspensión bacteriana salga a borbotones a alta presión.
- Si es diferente a A. se utilizan soluciones tumefaciens (por ejemplo, para probar diferentes promotores), eliminar cualquier exceso de solución que queda en la parte inferior de la hoja de la primera inyección usando una toalla de papel o similar antes de aplicar la siguiente solución.
- Incubación post-infiltración de plantas y muestreo.
- Preparar un fitotrón para las plantas tratadas ~ 24 horas antes de la inyección para permitir que la temperatura y la humedad de equilibrio en los niveles requeridos para el Departamento de Energía.
- Después de la inyección, la transferencia de las plantas en el fitotrón y colocarlos en bandejas lo suficientemente grandes como para el riego con agua hasta el final del periodo de incubación determinado por el Departamento de Energía.
- Establecer un phot de 16 horasoperiod usando seis Osram blanco 36 tubos fluorescentes W cool por 0,7 m 2 (75 mmol s -1 m -2; λ = 400-700 nm). Evitar que las plantas haciéndose mutuamente sombra por limitar el número de plantas a seis por 0,7 m 2.
- Antes del muestreo, asegúrese de que el campo intercostal correcta fue seleccionado mediante la comparación de la etiqueta agregada en la sección 5.2.1 y el plan del Departamento de Energía.
- Utilice un sacabocados para extraer 4-5 discos de hojas de los campos intercostales tratados en las posiciones y los tiempos indicados por el Departamento de Energía. No retire la hoja entera de la planta durante el muestreo. Estabilizar la hoja con una toalla de papel de mano mientras se quita los discos para evitar la ruptura.
- Determinar la masa de cada muestra y colocarla en un tubo de reacción de plástico de 1,5 ml etiquetado con el nombre de la muestra y de masas. Almacenar las muestras a -20 ° C o -80 ° C antes de la cuantificación de proteínas. El proceso se puede pausar en esta etapa durante varios meses, dependiendo de la estabilidad de la muestray la temperatura de almacenamiento.
6. Cuantificación de proteínas
- Extraer las proteínas de las muestras de disco de hoja.
- Añadir 3 ml de tampón de extracción (fosfato de sodio 50 mM, cloruro sódico 500 mM, pH 8,0) por mg de masa de la muestra y moler discos de hojas en el tubo de reacción utilizando un mortero eléctrica hasta que no hay grandes fragmentos permanecen. Evitar el sobrecalentamiento de la muestra.
- Eliminar los sólidos dispersos mediante la centrifugación de la muestra dos veces a 16.000 xg durante 20 min a 4 ° C. Transferir el sobrenadante a un tubo de reacción de 1,5 ml limpio después de cada paso sin perturbar el sedimento.
- Después de la centrifugación, el proceso se puede pausar mediante la congelación de la planta extrae a -20 ° C o -80 ° C durante varios meses, dependiendo de la estabilidad de la muestra y la temperatura de almacenamiento. Confirmar que un ciclo de congelación-descongelación no afecta a la concentración de la proteína (s) objetivo.
- Medir la fluorescencia DsRed.
- Preparar tres repeticiones técnica de cada muestra en placas de 96 pocillos de media área negra (50 l de extracto por pocillo). Evitar la formación de burbujas durante el pipeteado.
- Utilizar un conjunto de seis diluciones (0, 25, 75, 125, 175 y 225 mg / ml) de un estándar de DsRed por placa de 96 pocillos para generar una curva de referencia revestimiento. Preparar las diluciones en PBS y almacenar a 4 ° C para su uso dentro de los 3 meses.
- Medir la fluorescencia dos veces, de forma secuencial, en un lector de placas de 96 pocillos equipada con 530/25 nm de excitación y 590/35 nm filtros de emisión.
- Para cada muestra, el promedio de la fluorescencia en los dos lecturas y los tres técnicos repeticiones y restar el valor registrado para el ensayo en blanco, que contiene 0 g / ml DsRed. También restar este valor de la lee de las diluciones estándar y usar estos valores en blanco-corregido para una regresión lineal produciendo una curva de referencia (por el origen de coordenadas).
- Utilice la pendiente de la curva de referencia para convertir el Fluorescence medido para las muestras en concentraciones de DsRed. Si es necesario, diluir las muestras de manera que la lectura está comprendida en el intervalo de concentración de los estándares y considerar este factor de dilución en los cálculos posteriores.
- Determinar la concentración de 2G12.
- Preparar un dispositivo de resonancia de plasmón superficial (SPR) para la medición de la concentración de anticuerpo mediante el acoplamiento de la proteína A a la superficie activada de una celda de flujo. Utilice otra celda de flujo como referencia al inactivar la superficie sin el acoplamiento de la proteína A 30,31.
- Diluir 1:20 en extractos de plantas de SPR funcionamiento de amortiguación (10 mM de HEPES, pH 7,4, EDTA 3 mM, NaCl 150 mM, 0,05% v / v de Tween-20) y medir las unidades de respuesta (RU) de unión del anticuerpo a la proteína A en tres técnicos repeticiones de cada muestra al final de una inyección de 90 l (180 segundos a 30 l / min). Reste el RU medido en la celda de flujo de referencia (sin proteína A) del RU medida en el fl experimentalOW celular (proteína A de la superficie).
- Medir un estándar de 585 ng / ml de 2G12 después de cada 10-15 muestras y restar el valor medido en las células de flujo de referencia, como se describe más arriba. Utilice el RU promedio de estas normas para el cálculo de una curva de referencia lineal por el origen de coordenadas.
- Cálculo de las concentraciones 2G12 en las muestras basándose en la dilución 1:20 y la pendiente de la curva de referencia.
- Compruebe si hay una fuerte unión no específica a la célula de referencia, que puede corromper la medición. Compruebe también si las normas 2G12 mantienen valores aproximadamente constantes (<5% de variación) en todo el análisis, como una mayor variación refleja el envejecimiento de la proteína A de la superficie.
7. Análisis de datos y evaluación
- Analizar manualmente las respuestas observadas en los experimentos de expresión para (i) valores extremos errores (altas o bajas), que indica la medición, (ii) los resultados inusuales, por ejemplo, las combinaciones de factores que geneevaluar las respuestas inesperadas que indican el intercambio de datos, y (iii) los valores perdidos.
Observación: utilice estas acciones para evitar la construcción de modelos basados en datos erróneos. - La transferencia de los datos de respuesta analizadas (en este caso las concentraciones de proteína) en el "diseño" nodo de DesignExpert. Asegúrese de que los datos de respuesta se asignan correctamente a la configuración de factores correspondientes. Hay una extensa sección de ayuda disponibles en el software DesignExpert que cubre los aspectos abordados a continuación.
- En el nodo "Analysis", elija la respuesta que analizar e inicialmente seleccionar "ninguno" en la pestaña de transformación.
Nota: Se recomienda una transformación de min / max coeficientes de la respuesta mayor que 10. La transformación más útil puede ser obtenida de la Box-Cox-parcela en el registro "Diagnóstico" en la sección "Diagnóstico" de la "herramienta de diagnóstico" que se describe más adelante (sección 7.8). Una transformación logarítmica 10 es a menudo apropiado. - Continuar a la pestaña "Resumen Fit", que proporciona información general acerca de los factores que son importantes para el sistema objeto de la investigación (por ejemplo, interacciones de dos factores, los efectos de segundo grado). El software le sugerirá un modelo inicial basado en su importancia.
- En la pestaña de "Modelo", un modelo inicial está preseleccionada en base a los resultados "Resumen Fit". Utilice el modo automatizado para editar este modelo:
- Seleccione un "fin del proceso" que es un orden superior al modelo sugerido, por ejemplo, si el modelo propuesto es "2FI" (dos interacción de los factores) a continuación, seleccione "cuadrática".
- Elija "hacia atrás" en el campo "Selección", que eliminará de forma iterativa términos no significativos desde el modelo tras proceder a la pestaña "ANOVA", basada en el valor "Alfa a cabo", que inicialmente deberían ser 0.100.
- Si se le pregunta por el software, siempre que corrija automáticamente la jerarquía del modelo porque se trata necesariamentey para generar modelos confiables 32,33.
- En la pestaña "ANOVA", investigar el modelo propuesto y los factores incluidos. Si es necesario, eliminar manualmente los factores con valores de p por encima de un umbral predefinido (en este caso 0,05, correspondiente a un nivel de significación del 5%) o aquellas que se basan en la consideración mecanicista poco probable por cambiar de nuevo a la pestaña de "Modelo", el cambio de "Selección" "Manual" y eliminar los factores apropiados de la modelo.
- De vuelta en la pestaña "ANOVA", evaluar el modelo renovado en términos del valor de p de la "Modelo" (un valor bajo es deseable porque esto indica significación) y la "falta de ajuste" (un valor alto es deseable porque esto indica falta de significado), así como los valores de "R-cuadrado", "R-cuadrado ajustado" y "R-cuadrado prevista" (valores> 0,80 son deseables para los tres valores).
- Compare estos valores for diferentes modelos incluyendo / excluyendo factores en varios niveles de significación.
Nota: puede ser útil para duplicar la columna de la respuesta en el nodo "Diseño" y realizar cada análisis individual, o para exportar toda la tabla "ANOVA" en otro programa, como una hoja de cálculo.
- Compare estos valores for diferentes modelos incluyendo / excluyendo factores en varios niveles de significación.
- Continuar a la ficha "Diagnóstico" para confirmar la calidad del modelo y detectar posibles valores atípicos en el conjunto de datos que tiene una fuerte influencia en el modelo mediante el examen de todas las fichas de la "Herramienta de diagnóstico" (sección "Influencia" y "Diagnóstico") .
- En la sección de "Diagnóstico" de la "herramienta de diagnóstico", lleve a cabo las siguientes acciones:
- Asegúrese de que los puntos se encuentran dispersos al azar dentro de los límites de la "Residuos vs auguró" gráfico. Aquí, una distribución en forma de V indica la necesidad de transformación de datos.
- Asegurar los puntos están dispersos al azar dentro de los límites de la"Residuos vs predicha" gráfico. Aquí, una distribución en forma de V indica la necesidad de transformación de datos.
- Investigar si al azar los puntos del gráfico "Residuos vs run" de dispersión dentro de los límites. Un patrón (por ejemplo, "escalera") indica una tendencia en el edificio de datos y el bloque puede ser utilizado para contrarrestar su influencia en el modelo.
- Busque una línea recta diagonal que indica el modelo ideal de la "Predicción vs Real" trama. Cuanto mayor es la dispersión de los datos alrededor de la diagonal, la menos precisa el modelo.
- Compruebe si hay una superposición de la nada (el mejor) y verde (reales) líneas verticales en el Box-Cox-trama que indica la transformación de datos. Si la línea verde no coincide con el azul (fuera del intervalo indicado por las líneas verticales de color rojo) se remontan a la sección 7.3 y mejorar la ronda de construcción de modelos mediante la selección de la transformación de los datos sugiere el Box-Cox-parcela. Una vez más la duplicación de la recolumna de respuesta puede ser útil para comparar diferentes modelos.
- Asegúrese de que los puntos en los "Residuos vs Factor" cartas (hay un gráfico para cada factor de modelo) se dispersan al azar dentro de los límites. La curvatura en la distribución de datos puede indicar un factor que falta en el modelo.
- En la sección de "influencia" de la "herramienta de diagnóstico", asegúrese de que hay dispersión aleatoria de los datos dentro de los límites de los "residuos Externamente estudentizado", "Leverage", "DFFITS" y parcelas "DfBeta" y uniformemente bajo la distribución sin valores extremos en la trama "de Cook Distancia".
- En la sección de "Diagnóstico" de la "herramienta de diagnóstico", lleve a cabo las siguientes acciones:
- En la pestaña "gráficos Modelo", visualiza el modelo evaluado. Para un número limitado de factores numéricos (por ejemplo, 3) la superficie de respuesta ("superficie 3D") la representación es útil para evaluar optima / características manualmente.
Nota: Las superficies de respuesta sólo ilustran el impacto de dos factoresdores sobre la respuesta bajo investigación. El efecto de cualquier factor adicional en la respuesta se revela al cambiar su valor (factores numéricos) o nivel (factores categóricos) en la ventana de "herramienta de factores". Por otra parte, los factores que pueden ser asignados al eje parcela haciendo clic derecho en la ventana de "herramienta de Factores" y seleccionando el eje variable independiente deseada.- Manipular los niveles de los factores y asignarlos a las ordenadas del gráfico utilizando la herramienta "Factores".
- Exportar los gráficos utilizando el "gráfico Exportar a archivo ..." comando en la ficha "Archivo".
- Utilice la opción "numérica" sub-nodo en el nodo "Optimización" para optimizar la respuesta numérica (minimizar, maximizar, en el rango) en función de los factores de modelo, para que a su vez ciertas restricciones pueden ser aplicadas (por ejemplo, límites y pesos) a través de la pestaña "Criterios".
- Calcular y analizar soluciones numéricas en las "soluciones"ficha basado en las aportaciones de la pestaña "Criterios".
- Exportación de estas soluciones a otros programas (por ejemplo, hojas de cálculo) para su posterior análisis (por ejemplo, histogramas) que revela la configuración de factores asociados con valores altos o bajos de respuesta.
Nota: esta opción es útil si se investigan más de tres factores numéricos y representación 3D es difícil.
- Utilice la opción "predicción Point" sub-nodo para predecir la respuesta a la configuración específica de los factores.
- Utilice esta predicción para todas las configuraciones a ser evaluados (aquí, todo promotor y combinaciones 5'UTR, así como todas las hojas y los tiempos de incubación o posiciones de la hoja y temperaturas de incubación).
- Exportar los valores predichos y compilar a una matriz de datos que se puede utilizar para describir la expresión transitoria en plantas de tabaco, por ejemplo, mediante el cálculo de la expresión para una combinación específica de promotor-5'UTR en un momento determinado de media sobre todas las posiciones de la hoja o across todas las hojas de una planta.
- Normalizar los datos de expresión sobre la base de las masas de las diferentes hojas para producir el nivel de expresión específica (mg g -1) o la tasa de expresión específica (mg g -1 h -1).
- Alternativamente, exportar los coeficientes de la ecuación "final" de la parte inferior de la pestaña "ANOVA" en una hoja de cálculo y multiplicarlas con una serie de ajustes para el factor para producir la misma matriz de datos.
Nota: este array sólo puede contener valores de los factores de dentro del espacio de diseño inicial, porque la ecuación del modelo ajustado no es adecuado para la extrapolación.
- Llevar a cabo un experimento de confirmación para los puntos de modelo que son de gran interés.
- Lleve a cabo la expresión de proteína transitoria en condiciones seleccionadas para la "predicción Point" (punto 7.11). Eg utilizar las mismas temperaturas y las hojas, etc
- Determinar las concentraciones de proteínas de esta expresión transitoria eXperiment como se describe más arriba (sección 6) y compararlos con los valores pronosticados por el modelo.
- Compruebe si la concentración promedio de proteína en el experimento de confirmación cae dentro del intervalo de predicción del modelo de superficie de respuesta obtenida en el punto de predicción. Un partido afirma el poder de predicción del modelo. La falta de concordancia indica un modelo de baja calidad y se ejecuta adicionales pueden ser necesarios.
- Nota: Planta de la variabilidad de lote a lote amplía el intervalo de predicción y puede devaluar experimentos de confirmación realizadas en otro lote de plantas. Sin embargo, las muestras que no se han utilizado para generar el modelo, sino que se originó en el mismo lote de plantas como las muestras contenidas en el modelo pueden servir como controles eficaces.
Resultados
Un modelo descriptivo para la acumulación de DsRed durante la expresión transitoria utilizando diferentes promotores y 5'UTRs
DsRed de fluorescencia en los extractos de hoja se utiliza para indicar el nivel de expresión de la proteína recombinante y de este modo se utilizó como la respuesta en la estrategia DoE. La diferencia mínima detectable consideramos relevante fue de 20 mg / ml y la desviación estándar estimada del sistema fue de 8 mg / ml en base a los exp...
Discusión
Cada experimento requiere una planificación cuidadosa, porque los recursos suelen ser escasos y caros. Esto es particularmente cierto para las estrategias del Departamento de energía debido a errores durante la fase de planificación (por ejemplo, la selección de un modelo base que no cubra todas las interacciones significativas factores) pueden disminuir sustancialmente la capacidad predictiva de los modelos resultantes y así devaluar todo el experimento. Sin embargo, estos errores se pueden evitar fácilm...
Divulgaciones
La tasa de publicación fue parcialmente patrocinado por las empresas Statease, Inc. (EE.UU.) y STATCON (Alemania), que no participaron en los involucrados en la preparación del manuscrito o responsable de cualquiera de sus contenidos.
Agradecimientos
Los autores agradecen al Dr. Thomas Rademacher para proporcionar el vector de expresión de planta ppam e Ibrahim Al Amedi para el cultivo de las plantas de tabaco utilizadas en este estudio. Nos gustaría dar las gracias al doctor Richard M. Twyman por su ayuda con la edición del manuscrito. Este trabajo ha sido parcialmente financiado por el Advanced Grant "Future-Pharma" Consejo Europeo de Investigación, la propuesta número 269.110 y la Fraunhofer Zukunftsstiftung (Fundación Fraunhofer Futuro).
Materiales
| Name | Company | Catalog Number | Comments |
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life Technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10 x10 cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life Technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
Referencias
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ - The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5'UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados