É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Caracterização de Sistemas Complexos Usando o Projeto de Experimentos Abordagem: Transient Protein Expression em tabaco como um Estudo de Caso
Neste Artigo
Resumo
Descreve-se um desenho de experiências abordagem que pode ser utilizada para determinar e modelar a influência dos elementos de regulação de transgenes, de crescimento e de desenvolvimento de parâmetros de planta, e as condições de incubação sobre a expressão transiente de anticorpos monoclonais e proteínas repórter em plantas.
Resumo
As plantas fornecem benefícios múltiplos para a produção de biofármacos incluindo os custos baixos, escalabilidade e segurança. A expressão transiente oferece a vantagem adicional de curto desenvolvimento e tempos de produção, mas os níveis de expressão podem variar significativamente entre os lotes, dando assim origem a preocupações regulatórias no contexto das boas práticas de fabrico. Foi utilizado um planejamento de experimentos (DoE) abordagem para determinar o impacto dos principais fatores, como elementos reguladores na construção de expressão, o crescimento das plantas e os parâmetros de desenvolvimento e as condições de incubação durante a expressão, sobre a variabilidade de expressão entre os lotes. Foram testadas as plantas que expressam um anticorpo modelo anti-HIV anticorpo monoclonal (2G12) e uma proteína de marcador fluorescente (DsRed). Discutimos a justificativa para a seleção de certas propriedades do modelo e identificar suas possíveis limitações. A abordagem geral pode ser facilmente transferido para outros problemas, porque os princípios do modelo de umre amplamente aplicável: seleção de parâmetros baseada no conhecimento, a redução da complexidade, dividindo o problema inicial em módulos menores, de configuração guiada por software de combinações experimentais óptimas e aumento do projeto passo a passo. Portanto, a metodologia é útil não só para caracterizar a expressão da proteína em plantas, mas também para a investigação de outros sistemas complexos que faltam uma descrição mecanicista. As equações de previsão que descrevem a interligação entre os parâmetros podem ser utilizados para estabelecer modelos mecanísticos para outros sistemas complexos.
Introdução
A produção de proteínas biofarmacêutica em plantas é vantajoso porque as plantas são baratos a crescer, a plataforma pode ser aumentado apenas por crescimento mais plantas, e os agentes patogénicos humanos são incapazes de se replicar de 1,2. Estratégias de expressão transitória por exemplo com base na infiltração das folhas com Agrobacterium tumefaciens proporciona benefícios adicionais, porque o tempo entre o ponto de libertação de ADN e a entrega de um produto purificado é reduzida de anos a menos do que 2 meses 3. A expressão transiente é também utilizado para a análise funcional, por exemplo, para testar os genes para a sua capacidade para complementar os mutantes de perda de função ou para investigar interacções proteína 4-6. No entanto, os níveis de expressão transiente tendem a mostrar maior variação de lote para lote do que os níveis de expressão em plantas transgénicas 7-9. Isto reduz a probabilidade de que os processos de fabricação de produtos biofarmacêuticos com base na expressão transitória wivai ser aprovada no contexto de boas práticas de fabricação (BPF), pois a reprodutibilidade é um atributo essencial de qualidade e está sujeita a avaliação de risco 10. Essa variação também pode mascarar qualquer interação que os pesquisadores pretendem investigar. Por isso, nos propusemos a identificar os principais fatores que afetam os níveis de expressão transiente em plantas e para a construção de um modelo preditivo quantitativa de alta qualidade.
O (OFAT) abordagem de um fator-em-um-tempo é muitas vezes usado para caracterizar o impacto (efeito) de certos parâmetros (fatores) sobre o resultado (resposta) de um experimento 11. Mas isso é sub-ótima, pois os testes individuais (executado) no decurso do inquérito (experimento) serão alinhados como pérolas em um colar com a área potencial gerado por fatores que são testados (espaço de projeto). A cobertura do espaço de projeto e, portanto, o grau de informações derivadas da experiência ébaixo, como mostrado na Figura 1A 12. Além disso, as interdependências entre as diferentes factores (interacções do factor) pode permanecer oculto, resultando em modelos pobres e / ou a previsão de falsa óptimos, como mostrado na Figura 1B 13.
As desvantagens acima descritas podem ser evitados através da utilização de uma concepção de experiências (DOE) de aproximação na qual as pistas de uma experiência são dispersas mais uniformemente por todo o espaço de criação, o que significa que mais do que um factor é variada entre duas pistas 14. Há projetos especializados para misturas, fatores (fatoriais) ea quantificação dos impactos do fator sobre as respostas (métodos de superfície de resposta, RSM s) 15 de triagem. Além disso, RSMs pode ser realizado como projetos centro-composto, mas também pode ser conseguido, usando software especializado que pode aplicar diferentes critérios para a seleção de pistas. Por exemplo, o assim chamado D-optimalitcritério y irá selecionar corridas de modo a minimizar o erro nos coeficientes do modelo resultante, enquanto que o critério IV-optimality seleciona corridas que atingem o menor previsão de variação ao longo do espaço de design 15,16. O MSE descrito aqui permite a quantificação precisa da expressão da proteína transiente em plantas, mas que pode ser facilmente transferido para qualquer sistema envolvendo vários (~ 5-8) factores numéricos (por exemplo, temperatura, tempo, de concentração) e alguns (~ 2 - 4) fatores categóricas (por exemplo, promotor, cor), em que uma descrição mecanicista não está disponível ou demasiado complexas para modelar.
A abordagem DoE origem nas ciências agrícolas, mas se espalhou para outras áreas porque é transferível para qualquer situação em que é útil para reduzir o número de execuções necessárias para a obtenção de dados confiáveis e gerar modelos descritivos de processos complexos. Isto, por sua vez, levou-se à inclusão de corça no "Orientação paraIndústria, Q8 (R2) Pharmaceutical Development ", publicado pela Conferência Internacional de Harmonização dos Requisitos Técnicos para Registro de Produtos Farmacêuticos para Uso Humano (ICH) 17. DoE é agora amplamente utilizado na investigação científica e na indústria 18. Contudo, cuidados devem ser tomados durante o planejamento e execução do experimento, pois a seleção de um polinômio de grau impróprio para o modelo de regressão linear múltiplo (modelo básico) pode apresentar uma necessidade de corridas adicionais para modelar todos os efeitos do fator corretamente. Além disso, dados corrompidos ou perdidos gerar modelos incorretos e falho previsões, e pode até mesmo impedir qualquer tentativa de construção do modelo, conforme descrito nas seções de protocolo e discussão 18. Na seção de protocolo, que inicialmente irá definir as etapas de planejamento mais importantes para uma experiência baseada em RSM e, em seguida, explicar o design baseado no DoE DesignExpert v8.1 software. Mas projetos similares podem ser construídas com outros includi softwareng JMP, Modde e STATISTICA. Os procedimentos experimentais são seguidas por instruções para a análise de dados e avaliação.
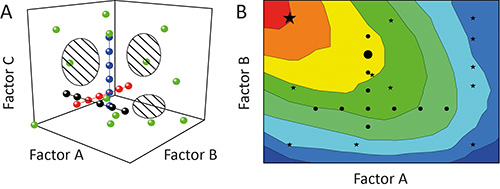
Figura 1. Comparação de OFAT e DoE. Uma. Variação sequencial de um fator de cada vez (OFAT) em um experimento (círculos pretos, vermelhos e azuis) atinge uma baixa cobertura do espaço de projeto (regiões nascidos). Em contraste, a variação de mais do que um factor de cada vez utilizando o desenho de experiências de estratégia (DOE) (círculos verdes) aumenta a cobertura e, assim, a precisão dos modelos resultantes. B. A cobertura do espaço de projeto tendencioso significa que as experiências OFAT (círculos pretos) também pode deixar de identificar as regiões operacionais ótimas (vermelho) e prever soluções sub-ótimas (grande círculo preto), enquanto DoE estrategicamentees (estrelas negras) são mais propensos a identificar as condições preferíveis (grande estrela preta).
Protocolo
1. Planejando uma Estratégia de DoE
- Identificar os fatores e as respostas relevantes para a inclusão no projeto.
- Defina uma ou várias respostas para a medição. Aqui, foram usadas 2G12 e DsRed níveis de expressão (ug / ml), incluindo a diferença mínima detectável considerados relevantes (10 e 20 ug / ml, respectivamente) e um valor aproximado para o desvio padrão estimado do sistema (4 e 8 ug / ml, respectivamente), com base em experiências anteriores.
- Use a literatura disponível, os dados de experiências anteriores ou projetos de triagem especializados (por exemplo, um planejamento fatorial, ver a introdução) para selecionar fatores significativos cujo impacto sobre as respostas serão quantificados (Tabela 1) 7,8,19,20.
- Alocar os tipos de fatores (numéricas ou categóricas) e selecionar intervalos dentro dos quais os fatores numéricos serão variaram durante a investigação DoE (Tabela 1).
- Identificar numeric fatores de variação contínua que é difícil de implementar.
- Evitar o uso de variação contínua para factores que não podem ser ajustados com precisão a um certo nível. Por exemplo, temperatura de incubação pode tipicamente ser controlado apenas dentro de ± 2 ° C, por conseguinte, a variação contínua usando valores, tais como 27,2 ° C, 25,9 ° C e 29,3 ° C mosto ser evitada.
- Em vez disso, selecione um número de níveis discretos para estes fatores (Tabela 1). O número de níveis deve corresponder ao modelo básico antecipado (passo 1.2). Isso só é importante para os projetos ideais, porque os projetos centrais compostas sempre têm níveis de fator discretos.
- Atribuir se um fator categórico é nominal, ou seja, nenhuma ordem implícita (por exemplo, fabricantes diferentes), ou ordinal e, portanto, um parâmetro numérico discreto (por exemplo, folhas diferentes em uma planta).
- Selecione os níveis para ambos os tipos de fatores categóricas.
- Selecione um modelo de base útil.
- Com base nas experiências e literatura preliminares, antecipar a relação entre cada fator ea resposta, bem como as interações ea resposta. Por exemplo, linear (quanto mais, melhor), quadrática (single aumento / diminuição ideal ou não-linear) ou cúbico (enviesada ideal).
- Certifique-se que o número de níveis de fatores numéricos discretos é n + 1, com n sendo o grau polinomial da relação entre o fator ea resposta. Por exemplo, se a temperatura se espera que tenha um efeito sobre a resposta quadrática, n = 2 e, portanto, pelo menos três níveis de temperatura devem ser investigados para obter um encaixe para o efeito quadrático.
- Definir a fração de espaço de projeto (FDS), para o qual a variância de previsão deve ser abaixo de um determinado limiar (nível alfa). O FDS devem ser> 0,95 (cobrir mais de 95% do espaço de desenho) para obter os modelos que produzem previsões robustas ao longotodo o espaço 21-23 design.
Nota: Um limiar de 0,05 corresponde a um nível de significância de 5% (erro tipo I) e é um valor típico. O aumento deste valor irá reduzir o número de experimentos necessários para uma estratégia DoE mas, simultaneamente, aumentar a probabilidade de que efeitos significativos serão atendidas e que as estimativas de seu impacto sobre a resposta será incorreto.

Tabela 1. Fatores que afetam a expressão da proteína transiente em tabaco, incluindo a variação varia durante Doe. Fatores em negrito só foram incluídos no projeto para os experimentos descritos em "Um modelo descritivo para acumulação durante DsRed expressão transitória utilizando diferentes promotores / 5'UTRs", enquanto fatores em itálico só foram incluídos no projeto para "Otimizando incubation condições e esquemas de colheita para a produção de anticorpos monoclonais em plantas usando expressão transitória ".
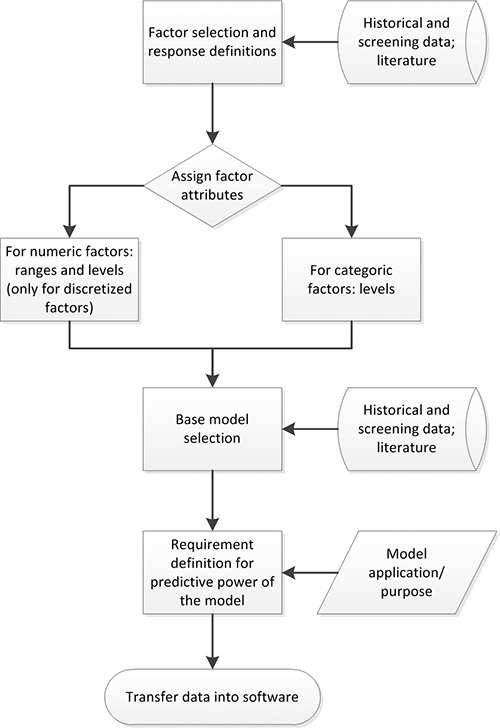
Figura 2. Processo de planejamento DoE. Fatores com um impacto significativo sobre a resposta sob investigação são selecionados com base nos dados disponíveis. Em seguida, os atributos do fator (por exemplo, numéricos), faixas e níveis são atribuídos. O conhecimento prévio e as experiências sejam usados para definir um modelo de base adequada. Os requisitos de energia de previsão são definidos com base na aplicação / propósito do modelo final. Os dados compilados pode então ser transferida para o software apropriado DOE.
2. Configurando um RSM em DesignExpert
- Comece DesignExpert e selecione "New Design". Na janela "Response Surface", escolha eº 34; Optimal "e digite o número de fatores numéricos e categóricas selecionados na seção 1.1).
- Digite o nome dos fatores, unidades, tipos, sub-tipos, número de níveis / fronteiras e valores para os níveis nível para todos os fatores nos campos correspondentes.
- Continue para a próxima página e em "Procurar" opções de escolha "Best", bem como o "Optimality" critério desejado, aqui "D-ótimo".
- No menu "Editar modelo ...", selecione o modelo que contém os fatores e interações previstas na seção 1.2). Se não tiver certeza, selecione um modelo completo que abrange todos os fatores e interações para um certo grau polinomial, aqui "quadrática". Note-se que a seleção de um modelo completo (contendo todas as interações) pode aumentar o número de experimentos necessários para o DoE.
- Selecione o número de "Blocos". Aqui, um bloco único foi utilizada porque todas as plantas foram do mesmo lote, todas as bactérias foram cultivadas e simultaneamentetodas as injecções foram realizadas no mesmo dia pelo mesmo operador. Use mais de um bloco, se mais de um lote de plantas é usado ou vários operadores lidar com as injeções.
- Equilibre o projeto
- Ativar "Forçar equilíbrio categórico", a fim de distribuir as corridas do experimento igualmente entre os níveis de fator categóricas, por exemplo, promotores diferentes que são testados.
Nota: isso pode reduzir a otimização do projeto para um grau que DesignExpert irá relatar como um valor%, dependendo dos pontos selecionados pelo algoritmo de otimização. - Recalcular o projeto várias vezes usando o algoritmo de semente aleatória implementadas para minimizar a redução da otimização.
Nota: as perdas em optimality pode variar de ~ 3-40% usando os mesmos dados de entrada, mas mudando o número de repetições pode ser usado para manter o equilíbrio categórico.
- Ativar "Forçar equilíbrio categórico", a fim de distribuir as corridas do experimento igualmente entre os níveis de fator categóricas, por exemplo, promotores diferentes que são testados.
- O software irá sugerir um valor para o número de "pontos de modelo" baseado no modelo básico, Aqui 70 corridas. Ajuste o número de execuções de "réplicas" e "Para estimar a falta de ajuste" para garantir que ele é> 5% do número de "pontos" para cada modelo, aqui 10 corridas cada.
- Continue para a próxima página, selecione o número de respostas e digitar seus nomes e unidades. Respostas adicionais podem também ser adicionados a qualquer momento, durante a avaliação de dados, sem qualquer impacto negativo sobre o cartão.
- Continuar para iniciar o algoritmo, o cálculo dos níveis de fator para as execuções do DoE. Se o modelo de base selecionado não corresponde ao número de níveis de um determinado fator, uma notificação será exibida eo cálculo não será iniciado. Neste caso, quer:
- Aumentar o número de níveis para este fator, ou
- Desmarque os termos no modelo de base que não pode ser calculado com base na quantidade actual de níveis.
- Por exemplo, se houver dois níveis para o factor de t "O tempo de incubação" (ou seja, 2 d e 5 d) em tele design atual e um modelo de base quadrática incluindo o termo t 2 foi selecionado, adicione um terceiro nível de t (ou seja, 8 d) ou remover o fator t 2 do modelo.
- Salve a planilha contendo as combinações de fatores ideais que são exibidos uma vez que o cálculo está completo.
- No nó "Design" selecione a opção "Avaliação" sub-nó e vá para a aba "Gráficos".
- Na ferramenta "Gráficos", selecione "FDS" e na caixa "FDS Graph" escolha "Pred" como o tipo de erro. Em seguida, entra a diferença mínima detectável, bem como o valor aproximado para o desvio padrão estimado do sistema definida na secção 1.1.1) como valores de "d" e "s", respectivamente (aqui 20 e 8 ng / ml para DsRed). Também entram no nível alfa (aceitável% faltando um efeito significativo) adequado para a aplicação, tipicamente 0,05 (5%).
- Certifique-se de que o FDS calculado corresponde ao percentual definido no ponto 1.3) (tipicamente> 0,95 para os modelos de previsão). Uma curva plana na trama FDS é preferível, indicando a precisão da previsão uniforme em todo o espaço de design.
- Se este não é o caso (como o encontrado aqui, FDS foi de 1%, conforme mostrado na Figura 3A), volte para o nó "design" e na barra de tarefas, selecione "Ferramentas de Design", em seguida "Aumentar projeto ..." e escolha " Aumentar ".
- Selecione o mesmo "Pesquisar" e "critérios de otimalidade" como antes. Também escolheu o mesmo "modelo Editar" e as configurações de "força equilíbrio categórico". Se o aumento do projeto é realizada antes de qualquer experiência (como foi feito aqui), então mudar o "Coloque executado em bloco" para "Block1".
- Na seção "Corre", digite o número de pontos adicionais "modelo", a manutenção de pelo menos 5% "réplicas" e "Para estimar faltade encaixar "no projeto total. Aqui, 100 corridas foram adicionados e não" réplicas "e" Para estimar falta de ajuste "foram incluídos.
- Uma vez que o cálculo tenha terminado, reexaminar o gráfico FDS, como descrito acima. Se o FDS ainda não é satisfatória, repete o aumento de criação, tal como descrito acima. Aqui, o FDS foi de 100% após o aumento e, portanto, não era necessário mais aumento (Figura 3B).

Figura 3. Comparação de parcelas FDS. Uma. Uma EOD consistindo de 90 corridas produz uma FDS insuficiente de apenas 1% para o erro padrão de previsão, utilizando um modelo de base quadrática em combinação com os valores para a diferença mínima detectável (20 ug / ml) e estiacasalado desvio padrão do sistema (8 ug / ml). B. Aumento do DOE para um total de 210 tiragens alcançado um FDS 100% e uma curva plana uniforme indicando a precisão do modelo de todo o espaço de design.
3. Clonagem e análise de cassetes de expressão
- Cultivar de Escherichia coli em 5 ml de meio LB (10 g / L de triptona, 5 g / L de extracto de levedura, NaCl 170 mM, 50 mg / L de ampicilina, para placas de LB-agar incluem 15 g / L de agar) a 37 ° C durante 6 -8 horas ou durante a noite num agitador orbital a 160 rpm. CUIDADO: ampicilina é uma substância nociva. Inocular o meio LB com 50 mL de cultura líquido ou células de transferência de colônias cultivadas em placas utilizando uma ponteira.
- Para a purificação de DNA de plasmídeo PSO, e outros derivados de ppam (GenBank AY027531), a partir de E. coli K12 tensão DH5a, siga as instruções do manual do kit de purificação de DNA 24. CUIDADO: a purificação kele contém substâncias químicas nocivas (veja acima o manual para mais detalhes). A sequência de plasmídeo está descrito na Figura 4 e por Comprador et al. 20.
- Determinar a concentração de ADN no eluato purificado através da medição da absorvância de uma amostra de 2 uL a 260 nm num dispositivo NanoDrop.
- Confirmar a identidade do ADN do plasmídeo purificado por digestão com endonucleases de restrição (FER).
- Selecione as REs de acordo com a seqüência de plasmídeo para que padrões de fragmentos únicos e distintos são gerados. Siga as recomendações do fabricante para as condições de digestão, como o volume de reação, tempo, temperatura e concentração de albumina sérica bovina estabilizador. Dependendo da sensibilidade do dispositivo de análise, utilizam 50-500 ng de DNA de plasmídeo por digestão.
- Prepare a 0,8-2,0% de gel para a separação de fragmentos de DNA por fervura de agarose em Tris-borato-EDTA-tampão (TBE) (Tris 90 mM, borato 90 mM, EDTA 2 mM, pH 8,0). Tele maiores os fragmentos esperados, a menos de agarose deve ser usado no gel.
- Adicionar 5 uL de tampão de amostra de cinco vezes (5x SABU, 0,1% (w / v) de azul de bromofenol a 0,1% (w / v), xileno cianol, 10% (w / v) de glicerol dissolvidos em TBE) a 50 ul da RE-tratada amostra de ADN e separar os fragmentos através de electroforese em gel de agarose, a 100 V durante ~ 40 min, ou até uma separação nítida dos fragmentos é conseguida. Em cada gel, incluem uma pista contendo marcadores de tamanho de escada de ADN para efeitos de comparação, por exemplo, 2-3 ul escada de 1 Kb.
- Substitua a 5'UTR omega em PSO com um dos outros três 5'UTRs.
- Solte a seqüência 5'UTR omega de ~ 4 mg DNA PSO purificada por tratamento com 20 unidades de Eco RI-HF e Nco REs I-HF em NEBuffer 4 a 37 ° C para ~ 60 min. Em seguida, separar os fragmentos, como descrito nas seções 3.4.2 e 3.4.3.
- Isolar o fragmento maior (pS, "espinha dorsal") a partir do gel de agarose, utilizando um gel adicionalkit cção de acordo com o manual do fabricante do 25 e determinar a concentração do DNA purificado como descrito na seção 3.3). ATENÇÃO: o kit de extração de gel contém substâncias químicas nocivas, consulte o manual para mais detalhes.
- Isolar o CHS, CHS-LPH e TL 5'UTRs de vetores doadores adequados, tratando ~ 10 mg de cada vetor com 20 unidades de Eco RI-HF e REs Wcol-HF em NEBuffer 4 a 37 ° C para ~ 60 min. Em seguida, separar os fragmentos, como descrito nas seções 3.4.2 e 3.4.3 e purificar o menor 5'UTR contendo fragmento, conforme descrito no ponto 3.5.2.
- Ligadura os 5'UTRs purificadas isoladas em alíquotas separadas para o vector pS linear purificado isolado a 25 ° C durante 5 minutos de acordo com as recomendações do fabricante 26. Use ~ 50 ng de ADN de vector e um excesso molar de três vezes de ADN 5'UTR em um volume total de 20 ul.
- Células coli Transformação E com the plasmídeos recombinantes 26,27.
- Adicionar ~ 10 ng (~ 4-5 mL) de mistura de ligação (ver secção 3.5.4) para 50 ul RbCl E. competente coli e misturar suavemente, e então incubar durante 30-60 minutos em gelo. O choque térmico durante 1,5 min a 42 ° C e frio em gelo durante 5-30 min.
- Adicionar 950 ul de meio LB sem antibiótico e incubar durante 1 hora a 37 ° C e 160 rpm. Para a selecção dos transformantes, espalhar 50 e 100 ul da cultura em placas de agar LB contendo ampicilina e incubar durante ~ 16-20 horas a 37 ° C.
- Inocular 5-10 colônias separadas representando cada ligadura promotor-5'UTR descrito na secção 3.1 em alíquotas de 5 ml de meio LB contendo ampicilina. Em seguida, purificar o DNA plasmídeo e confirmar a sua identidade, tal como descrito nas seções 3.2 a 3.4.
- Substituir o promotor 35SS com o promotor NOS em cada um dos quatro plasmídeos 5'UTR utilizando REs Asc I e Eco RI em NEBuffer 4 a 37 ° C durante 1 hora, tal como descrito nas secções 3.5 e 3.6.
- Introduzir cada um dos oito plasmídeos resultantes em A. tumefaciens GV3101 tensão: pMP90RK por eletroporação 28.
- Adicionar ~ 500 ng de DNA de plasmídeo purificado a 50 A. ul competente tumefaciens células no gelo. Misture delicadamente e transfira para uma prechilled tina 0,2 centímetros eletroporação. Certifique-se de que a mistura está na parte inferior da tina e não contém bolhas.
- Células pulso em 2,5 kV para 5 ms e confirmar a intensidade e duração do pulso.
- Evite elevadas concentrações de sal na amostra de DNA ou preparação incorreta do A. competente tumefaciens células porque estas podem resultar em correntes elevadas de iões causando a vaporização instantânea de suspensão de células, reduzindo a eficácia de transformação.
- Adicionar 950 ul de meio YEB sem antibióticos (extrato de 5 g / L de carne, 1 g / L de extracto de levedura, 5 g / L de peptonae, 5 g / L de sacarose, 2 mM de MgSO4, pH 7,0), misturar cuidadosamente e transferir imediatamente para um tubo de reacção de 1,5 ml estéril. Incubar durante 2-4 horas a 26-28 ° C e 160 rpm.
- Espalhe 1-2 ul em placas de agar YEB contendo antibióticos (50 mg / L carbenicilina, 25 mg / L de canamicina, 25 mg / L, rifampicina), para a selecção dos transformantes. CUIDADO: rifampicina é uma substância tóxica.
- Inocular três alíquotas de 5 ml de meio de YEB contendo antibióticos com células de colónias independentes, representando cada ligação promotor-5'UTR e incubar durante 48-72 horas a 26-28 ° C e 160 rpm.
- Confirme o sucesso de A. tumefaciens transformação.
- Transferência de 2 mL de cada alíquota da seção 3.8.6 para separar 48 mL alíquotas de PCR mastermix (2 mL de cada 10 mM estoque cartilha (400 nM concentração final de cada primer),1 ul de 10 mM de mistura de dNTP (200 uM concentração final de cada trifosfato de desoxinucleótido), 5 ul de 10 vezes de tampão Expand High Fidelity com MgCl2 15 mM, 0,75 ul mistura de enzima Expand High Fidelity, e 39,25 mL de água destilada estéril).
- Amplificar a cassete de expressão de cada plasmídeo utilizando os iniciadores apropriados (FWD: 5'-CCT CAG GAA GAG AAG TAC-3 ', a ligação 1026 nucleótidos a montante do promotor; REV: 5'-CCA AAG CGA GTA CAC AAC-3', a ligação dentro de sítio de poliadenilação do 35S) em condições de PCR adequados. Aqui, 94 ° C foi utilizado para a desnaturação inicial, seguido por 30 ciclos de desnaturação 94 ° C durante 15 seg, 51 ° C, emparelhamento durante 30 seg e 72 ° C, alongamento durante 120 segundos, e uma etapa de alongamento final a 72 ° C durante 8 min.
- Determinar o tamanho dos produtos de PCR por electroforese em gel de agarose como descrito na secção 3.4.3.
- Prepare A. tumefaciens estoques de glicerol pormistura de 500 ul de 50% (v / v) de glicerol estéril com 500 ul de A. tumefaciens culturas da seção 3.8.6 para o qual a transformação foi (seção 3.9.3) confirmados. Armazenar os estoques de glicerol a -80 ° C até à sua utilização.
- Calcular as energias de dobragem das diferentes ARNm utilizando o servidor web RNAfold 29 e incluem a sequência de nucleótidos a partir do local de início da transcrição, através do primeiro 50 pb da região codificadora.
- Na seção "Dobre algoritmos e opções básicas", selecione o "mínimo de energia livre (MFE) e função de partição" e "evitar isolados pares de bases" de opções.
- Nas "opções de dobra avançadas" escolha "pendurados energias em ambos os lados de uma hélice em qualquer caso", "parâmetros de RNA (modelo de Turner, 2004)" e mudar "redimensionar os parâmetros de energia para determinada temperatura (C)" a 25 ° C.
- Na seção "Opções de saída", selecione todas as opções.
- A partir dos "Resultados para termodinâmica previsão conjunto" do arquivo de saída, extrair a "energia livre do conjunto termodinâmico" ea "frequência da estrutura MFE no conjunto de" valores para comparação.
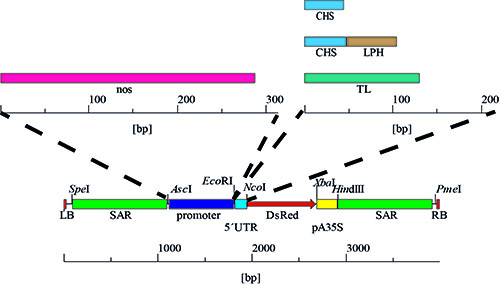
Figura 4. Promotor e 5 'UTR variantes. As cassetes de expressão foram gerados pela troca gradual do 5'UTR, resultando em quatro combinações com CaMV 35SS promotor, seguido pela substituição deste promotor com a sequência nsa rendendo quatro variantes adicionais e um total de oito combinações diferentes de promotor / 5'UTR.
4. Cultivo de Plantas
- Prepara-se uma solução a 0,1% do fertilizante Ferty2 mega em água deionizada em pH 5.9.
- Prepare 10 blocos x 10 x 8 cm de lã de rocha por extensa lavagem com água deionizada para remover resíduos químicos e, finalmente, equilibrar com fertilizante.
- Semente plantas de tabaco, colocando 1-2 sementes de tabaco em cada bloco rockwool seguido por um curto nivelada com fertilizantes tendo o cuidado de evitar lavar as sementes de distância.
- Germinar e cultivar as plantas de tabaco durante 42 dias em uma estufa à temperatura 25/22 ° C dia / noite, 70% de umidade relativa e fotoperíodo de 16 horas (180 mmol m -2 s -1; λ = 400-700 nm) . Durante este fotoperíodo, irrigar com fertilizante por 15 min a cada hora em cultivo hidropônico.
5. Transient Protein Expression
- Prepare A. tumefaciens para injeção em folhas.
- Inocular 5-50 ml de meio de YEB contendo antibióticos com 1% A. tumefaciens cultura crio-estoque e incubar a 27 ° C até que o OD 600nm atinge 5.0 (~ 48-72 horas, dependendo do volume e tipo de embarcação).
- Dilui-se a A. cultura Agrobacterium com água e em meio de infiltração de 2 vezes (4,3 g / L de Murashige e Skoog (pH 5,6), 5 g / L de sacarose, 1,8 g / L de glucose, 100 mM de acetoseringona) para coincidir com a 600nm OD é necessária para a injecção. Confirme a 600nm OD pouco antes da injeção. Nota: Um 600nm OD de 1,0 corresponde a ~ 1,43 ± 0,12 x 10 9 unidades formadoras de colônias por ml.
- Injectar a A. tumefaciens suspensão em folhas.
- Escolha e rotular as folhas e as posições noncotyledon nela a ser tratado (por exemplo, de acordo com uma estratégia de DOE).
- Não injectar diferente A. tumefaciens soluções para o mesmo campo intercostal. Em vez disso, usar secções opostas de cada lado do eixo médio venosa. Se houver mais de duas soluções diferentes têm de ser injectado na mesma posição de utilizar um additional planta.
- Agite A. solução de Agrobacterium para ressuspender as células que podem se instalaram desde a preparação da diluição e aspirada para uma seringa de 1 ml.
- Suavemente arranhar a epiderme na posição prevista para a injecção com uma ponta de pipeta ou semelhante para facilitar o fluxo do A. tumefaciens solução. Evite ruptura da lâmina foliar ao fazê-lo.
- Segurar a seringa perpendicular à lâmina de folha de tocar o cano contra o campo intercostal a ser tratada e empurre a tomada (sem agulha) para o lado inferior da folha. Pressionar o lado superior da folha suavemente, ao mesmo tempo para evitar que a lâmina de folha de deslocamento ou rotura.
- Com cuidado, empurre para baixo o pistão da seringa. A A. solução de Agrobacterium entra os espaços intercelulares dentro do limbo, como indicado pelas áreas tratadas aparecem verde mais escuro e húmido. Repita este procedimento em várias posições até que toda a intercostal campo é infiltrada com A. Agrobacterium. Em seguida, continue com o próximo campo intercostal.
- Certifique-se a seringa permanece perpendicular à folha. Inclinando a seringa causará a suspensão bacteriana a jorrar sob alta pressão.
- Se for diferente A. soluções de Agrobacterium são utilizados (por exemplo, para testar diferentes promotores), remover qualquer excesso de solução que permanece no lado de baixo da folha desde a primeira injecção, utilizando uma toalha de papel ou similar, antes de aplicar a solução seguinte.
- Pós-incubação de infiltração de plantas e de amostragem.
- Preparar um fitotron para as plantas tratadas ~ 24 horas antes da injecção para permitir o equilíbrio de temperatura e humidade nos níveis requeridos para o Departamento de Energia.
- Após a injecção, transferir as plantas para a fitotron e colocá-los em tabuleiros com dimensões suficientes para irrigação com água até ao final do período de incubação determinada pelo Departamento de Energia.
- Estabelecer um phot 16 horasoperiod usando seis Osram branco frio 36 W por lâmpadas fluorescentes de 0,7 m 2 (75 mmol sec -1 m -2; λ = 400-700 nm). Impedir as plantas a partir de sombreamento entre si através da limitação do número de plantas de seis por 0,7 m 2.
- Antes de amostragem, verifique se o campo intercostal correto foi selecionado, comparando o rótulo adicionado na seção 5.2.1 eo plano DoE.
- Use uma sonda para remover 4-5 discos de folhas dos campos intercostais tratados nas posições e horas indicadas pelo DoE. Não retire toda a folha da planta durante a amostragem. Estabilize a folha com um papel toalha de mão ao remover os discos para evitar a ruptura.
- Determine a massa de cada amostra e colocá-lo em um tubo de plástico reação 1,5 ml rotulado com o nome e massa da amostra. Armazenar as amostras a -20 ° C ou -80 ° C antes da quantificação da proteína. O processo pode ser interrompido neste estágio durante vários meses, dependendo da estabilidade da amostrae a temperatura de armazenamento.
6. Quantificação de proteínas
- Extrair proteínas a partir de amostras de discos de folha.
- Adicionar 3 ml de tampão de extracção (fosfato de sódio 50 mM, cloreto de sódio 500 mM, pH 8,0) por mg de massa de amostra e moer discos de folhas do tubo de reacção utilizando um pilão eléctrico até que não permanecem grandes fragmentos. Evitar o sobreaquecimento da amostra.
- Remover os sólidos dispersos por centrifugação da amostra duas vezes a 16.000 xg durante 20 min a 4 ° C. Transferir o sobrenadante para um tubo de reacção de 1,5 ml limpo depois de cada passo, sem perturbar o sedimento.
- Após centrifugação, o processo pode ser interrompida por congelação da planta extrai-se a -20 ° C ou -80 ° C durante vários meses, dependendo da estabilidade da amostra e da temperatura de armazenamento. Confirmar que um ciclo de congelamento-descongelamento não afecta a concentração da proteína (s) alvo.
- Medir a fluorescência DsRed.
- Preparar três repetições técnicos de cada amostra em placas de 96 poços de metade da área de preto (50 ul de extracto por poço). Evitar a formação de bolhas durante a pipetagem.
- Usar um conjunto de seis diluições (0, 25, 75, 125, 175 e 225 ug / ml) de um padrão DsRed por placa de 96 poços para construir uma curva de referência do forro. Preparar as diluições em PBS, e armazená-los, a 4 ° C para utilização no prazo de 3 meses.
- Medir a fluorescência duas vezes, sequencialmente, num leitor de placas de 96 poços equipada com 530/25 nm de excitação e 590/35 nm filtros de emissão.
- Para cada amostra, a média da fluorescência ao longo das duas leituras e as três repetições técnicas e subtrair o valor registado para o ensaio em branco, contendo 0 ug / ml DsRed. Também subtrair este valor a partir das leituras das diluições padrão e utilizar estes valores em branco corrigida para uma regressão linear produzindo uma curva de referência (através da origem das ordenadas).
- Utilizar o declive da curva de referência para converter o fluorescence medidos para as amostras em concentrações DsRed. Se necessário, diluir as amostras de modo que a leitura está dentro do intervalo de concentração dos padrões e considerar este fator de diluição nos cálculos posteriores.
- Determinar a concentração de 2G12.
- Preparar um dispositivo de ressonância de plasmon de superfície (SPR) para a medição da concentração do anticorpo por acoplamento de Proteína A para a superfície activada uma célula de fluxo. Utilize outra célula de fluxo como referência pela inativação da superfície sem acoplamento de Proteína A 30,31.
- Diluir 1:20 em extractos de plantas SPR tampão de corrida (10 mM de HEPES pH 7,4, EDTA 3 mM, NaCl 150 mM, 0,05% v / v de Tween-20) e medir as unidades de resposta (RU) de anticorpo de ligação à Proteína A em três repetições técnicos de cada amostra no final de uma injecção de 90 uL (180 seg a 30 mL / min). Subtrair o RU medida na célula de fluxo de referência (sem proteína A) a partir do RU medido no fl experimentalow celular (proteína A de superfície).
- Medir um padrão de 585 ng / ml 2G12 após cada 10-15 amostras e subtrair o valor medido nas células de fluxo de referência, tal como descrito acima. Utilizar o RU média destes padrões para calcular uma curva linear de referência através da origem das ordenadas.
- Calcular as concentrações 2G12 nas amostras com base na diluição de 1:20 e o declive da curva de referência.
- Verifique a existência de forte ligação não específica para a célula de referência, que pode corromper a medição. Além disso, verifique se as normas 2G12 manter valores aproximadamente constantes (<5% de variação) em toda a análise, como uma maior variação reflete o envelhecimento da Proteína A superfície.
7. Análise e Avaliação de Dados
- Analisar manualmente as respostas observadas nos experimentos de expressão para (i) valores extremos erros (altas ou baixas), indicando medição, (ii) resultados incomuns, por exemplo, combinações de fatores que genesavaliar as respostas inesperadas que indicam a troca de dados, e (iii) valores perdidos.
Nota: usar essas ações para impedir a construção de modelos com base em dados defeituosos. - Transfira os dados de resposta analisadas (aqui as concentrações de proteína) para o nó "Design" de DesignExpert. Certifique-se de que os dados de resposta são atribuídos corretamente com as configurações fator correspondente. Há uma extensa secção de ajuda disponíveis no software DesignExpert que abrange os aspectos abordados abaixo.
- No nó "Análise", escolha a resposta a ser analisada e, inicialmente, selecione "none" na aba de transformação.
Nota: a transformação é recomendado para min / max rácios de resposta maior do que 10. A transformação mais úteis podem ser obtidas a partir da Cox-enredo Box na aba "Diagnóstico" na seção "Diagnóstico" da "ferramenta de diagnóstico", descrito a seguir (seção 7.8). A Log 10 transformação é muitas vezes o caso. - Continuar para a aba "Resumo do Fit", que fornece informações gerais sobre os fatores que são importantes para o sistema sob investigação (por exemplo, as interações de dois fatores, quadrático). O software sugerem um modelo inicial com base no seu significado.
- Na guia "Model", um modelo inicial é pré-selecionada com base nos resultados "Resumo do Fit". Use o modo automático para editar este modelo:
- Selecione uma "ordem de Processos", que é uma ordem acima o modelo sugerido, por exemplo, se o modelo sugerido é "2FI" (dois interação fator), em seguida, selecione "quadrática".
- Escolha "para trás" no campo "Selection", que vai iterativamente remover termos não significativos do modelo depois de passar para a aba "ANOVA" com base no valor "Alpha fora", que deve ser inicialmente 0.100.
- Se solicitado pelo software, sempre corrigir automaticamente a hierarquia do modelo, porque esta é necessariamentey para gerar modelos fiáveis 32,33.
- Na guia "ANOVA", investigar o modelo sugerido e os fatores incluídos. Se necessário, remover manualmente todos os fatores com p-valores acima de um limite pré-definido (aqui 0,05, o que corresponde a um nível de significância de 5%) ou aqueles que são improváveis com base na análise mecanicista mudando de volta para a aba "Model", mudando "Seleção" para "Manual" e eliminar os factores apropriados a partir do modelo.
- De volta à aba "ANOVA", avaliar o modelo atualizado em termos de valor-p do "Model" (um valor baixo é desejável, pois isso indica significância) eo "Falta-of-fit" (um valor elevado é desejável porque isso indica ausência de significância), bem como os valores de "R-quadrado", "R-quadrado ajustado" e "Previsto R-quadrado" (valores> 0,80 é desejável para todos os três valores).
- Compare estes valores for diferentes modelos, incluindo / excluindo fatores em vários níveis de significância.
Nota: pode ser útil para duplicar a coluna de resposta no nó "Design" e realizar cada análise individual, ou para exportar toda a tabela "ANOVA" em outro programa como uma planilha.
- Compare estes valores for diferentes modelos, incluindo / excluindo fatores em vários níveis de significância.
- Continue até a aba "Diagnóstico" para confirmar a qualidade do modelo e detectar potenciais de outliers no conjunto de dados que tem uma forte influência sobre o modelo, examinando todas as guias na "Ferramenta de Diagnóstico" (seção "Influence" e "Diagnóstico") .
- Na seção "Diagnóstico" da "ferramenta de diagnóstico", execute as seguintes ações:
- Verifique se os pontos estão espalhados aleatoriamente dentro dos limites do "versus Resíduos previsto" gráfico. Aqui, uma distribuição em forma de chevron indica a necessidade de transformação de dados.
- Assegurar os pontos estão espalhados aleatoriamente dentro dos limites do"Resíduos vs previsto" gráfico. Aqui, uma distribuição em forma de chevron indica a necessidade de transformação de dados.
- Investigar se os pontos no "Resíduos vs run" gráfico de dispersão aleatoriamente dentro dos limites. Um padrão (por exemplo, "escada") indica uma tendência na construção de dados e bloco pode ser usado para neutralizar a sua influência sobre o modelo.
- Procure por uma linha diagonal reta, indicando o modelo ideal no "Previsto vs real" trama. Quanto maior o espalhamento de dados em torno da diagonal, a menos preciso do modelo.
- Verifique se há uma sobreposição de azul (melhor) e verde linhas verticais (reais) no-Cox-enredo Box indicando a transformação de dados. Se a linha verde não combina com o azul (fora do intervalo indicado pelas linhas verticais vermelhas) voltar para a seção 7.3 e melhorar a construção de modelos rodada marcando a transformação de dados sugerido pelo-Cox-enredo Caixa. Novamente a duplicação da recoluna posta pode ser útil para comparar diferentes modelos.
- Certifique-se de que os pontos nos "Resíduos vs Factor" charts (há um gráfico para cada fator de modelo) dispersam aleatoriamente dentro dos limites. Curvatura na distribuição de dados pode indicar um fator ausente no modelo.
- Na seção "Influence" da "ferramenta de diagnóstico", verifique se não há dispersão aleatória dos dados dentro dos limites dos "resíduos Externamente studentized", "Leverage", "DFFITS" e parcelas "DFBETAS" e uniformemente baixo distribuição sem valores extremos na trama "de Cook Distance".
- Na seção "Diagnóstico" da "ferramenta de diagnóstico", execute as seguintes ações:
- Na aba "gráficos modelo", visualizar o modelo avaliado. Para um número limitado de fatores numéricos (por exemplo, 3) a superfície de resposta ("superfície 3D") a representação é útil para avaliar optima / características manualmente.
Nota: superfícies de resposta apenas ilustrar o impacto de dois facres sobre a resposta sob investigação. O efeito de qualquer fator adicional sobre a resposta é revelada, alterando o seu valor (fatores numéricos) ou nível (fatores categóricas) na janela "ferramenta Fatores". Alternativamente, os fatores podem ser atribuídos ao eixo enredo clicando-los na janela "ferramenta Fatores" e selecionar o eixo variável independente desejado.- Manipular os níveis de fator e atribuir-lhes as ordenadas do gráfico usando a ferramenta "Fatores".
- Gráficos de exportação utilizando o "gráfico Exportar para o arquivo ..." comando na guia "File".
- Use o "Numérica" sub-nó no nó "Otimização" para optimizar a resposta numérica (minimizar, maximizar, no intervalo), dependendo dos fatores do modelo, para o qual, por sua vez algumas restrições podem ser aplicadas (por exemplo, limites e pesos) por meio do guia "Critérios".
- Calcular e analisar soluções numéricas nas "soluções"guia com base na entrada fornecido na aba "Critérios".
- Exportar estas soluções para outros programas (por exemplo, folhas de cálculo) para análise posterior (por exemplo, histogramas) revelando as definições do factor de associados com valores de resposta altas ou baixas.
Nota: isto é útil se mais de três fatores numéricos são investigados e representação 3D é difícil.
- Use a "previsão Point" sub-nó para prever a resposta para as configurações de fator específico.
- Use esta previsão para todas as configurações a serem avaliadas (aqui, todo promotor e combinações 5'UTR, bem como todas as folhas e os tempos de incubação ou posições da folha e temperaturas de incubação).
- Exportar os valores previstos e compilar para uma matriz de dados, que pode ser usado para descrever a expressão transiente em plantas de tabaco, por exemplo, pelo cálculo da expressão de uma combinação específica de promotor 5'UTR em um determinado momento em média, em todas as posições da folha ou across todas as folhas de uma planta.
- Normalizar os dados de expressão com base nas massas das diferentes folhas para se obter o nível de expressão específica (mg g-1) ou a taxa de expressão específica (mg g -1 h -1).
- Alternativamente, exportar os coeficientes da "equação Final" da parte inferior da guia "ANOVA" em uma planilha e multiplicá-los com uma variedade de configurações de fatores para produzir a mesma matriz de dados.
Nota: esta disposição só pode conter valores do fator de dentro do espaço de projeto inicial, porque a equação modelo ajustado não é adequado para extrapolação.
- Realizar um experimento de confirmação para os pontos de modelo que são de grande interesse.
- Realize a expressão da proteína transitória em condições selecionadas para "previsão Point" (seção 7.11). Ex usar as mesmas temperaturas e folhas etc
- Determinar as concentrações de proteína para esta expressão transitória eXperiment como descrito acima (secção 6) e compará-los com os valores previstos pelo modelo.
- Verifique se a concentração média de proteína no experimento de confirmação cai dentro do intervalo de previsão do modelo de superfície de resposta obtida durante a previsão ponto. Uma partida afirma o poder preditivo do modelo. Uma incompatibilidade indica uma baixa qualidade do modelo e corre adicionais podem ser necessárias.
- Nota: planta variabilidade de lote para lote amplia o intervalo de previsão e pode desvalorizar experimentos de confirmação realizadas com um lote diferente de plantas. No entanto, as amostras que não foram utilizados para gerar o modelo, mas que se originou a partir do mesmo lote de plantas como as amostras contidas no modelo pode servir como controles eficazes.
Resultados
Um modelo descritivo para acumulação durante DsRed expressão transitória utilizando diferentes promotores e 5'UTRs
DsRed fluorescência nos extractos de folhas foi utilizado para indicar o nível de expressão da proteína recombinante e, assim, foi utilizada como a resposta na estratégia DOE. A diferença mínima detectável foram consideradas relevantes foi de 20 ug / ml e o desvio padrão estimado do sistema foi de 8 ug / ml com base em experiências iniciais. Fa...
Discussão
Cada experimento requer um planejamento cuidadoso, porque os recursos são muitas vezes escasso e caro. Isto é particularmente verdadeiro para as estratégias DoE porque erros durante a fase de planejamento (por exemplo, a seleção de um modelo de base que não cobre todas as interações fator significativo) pode diminuir substancialmente a capacidade de previsão dos modelos resultantes e, assim, desvalorizar todo o experimento. No entanto, esses erros podem ser facilmente evitado, seguindo procedimentos b?...
Divulgações
A taxa de publicação foi parcialmente patrocinado pelas empresas Statease, Inc. (EUA) e Statcon (Alemanha), que não estavam envolvidos no envolvidos na preparação do manuscrito ou responsáveis por qualquer dos seus conteúdos.
Agradecimentos
Os autores agradecem ao Dr. Thomas Rademacher por fornecer o ppam planta vetor de expressão e Ibrahim Al Amedi para cultivar as plantas de tabaco utilizados neste estudo. Gostaríamos de agradecer ao Dr. Richard M. Twyman por sua ajuda com a edição do manuscrito. Este trabalho foi em parte financiado pelo Conselho Europeu de Investigação Avançada Grant "Future-Pharma", número da proposta 269110 eo Fraunhofer Zukunftsstiftung (Fraunhofer Fundação Futuro).
Materiais
| Name | Company | Catalog Number | Comments |
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life Technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10 x10 cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life Technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
Referências
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ - The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5'UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados