È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Caratterizzazione dei sistemi complessi Utilizzando il Design of Experiments Approccio: Transient Protein Expression in tabacco come un caso di studio
In questo articolo
Riepilogo
Descriviamo un disegno di esperimenti approccio che può essere utilizzato per determinare e modellare l'influenza del transgene elementi regolatori, parametri di crescita e sviluppo delle piante, e condizioni di incubazione sulla espressione transiente di anticorpi monoclonali e proteine reporter nelle piante.
Abstract
Le piante offrono molteplici vantaggi per la produzione di biofarmaci tra cui bassi costi, scalabilità e sicurezza. Espressione transiente offre il vantaggio supplementare di brevi tempi di sviluppo e di produzione, ma i livelli di espressione può variare notevolmente tra i lotti dando così luogo a preoccupazioni di regolamentazione nel contesto delle buone prassi di fabbricazione. Abbiamo usato un disegno degli esperimenti (DoE) approccio per determinare l'impatto dei principali fattori, quali elementi di regolamentazione del costrutto di espressione, la crescita delle piante e parametri di sviluppo, e le condizioni di incubazione durante espressione, sulla variabilità di espressione tra i lotti. Abbiamo testato piante che esprimono un modello anti-HIV anticorpo monoclonale (2G12) e una proteina marcatore fluorescente (DsRed). Discutiamo il razionale per la selezione di alcune proprietà del modello e identificare i suoi potenziali limiti. L'approccio generale può essere facilmente trasferito ad altri problemi perché i principi del modello unre ampiamente applicabile: la selezione dei parametri basata sulla conoscenza, la riduzione della complessità suddividendo il problema iniziale in moduli più piccoli, l'installazione guidata dal software di combinazioni ottimali di esperimento e graduale aumento del design. Pertanto, il metodo non è solo utile per caratterizzare l'espressione della proteina nelle piante, ma anche per la ricerca di altri sistemi complessi privi descrizione meccanicistica. Le equazioni predittive che descrivono l'interconnettività tra i parametri possono essere utilizzati per stabilire modelli meccanicistici per altri sistemi complessi.
Introduzione
La produzione di proteine biofarmaceutiche nelle piante è vantaggiosa perché le piante sono economici a crescere, la piattaforma può essere scalata solo crescendo più piante e patogeni umani sono in grado di replicare 1,2. Strategie espressione transiente basate per esempio sulla infiltrazione di foglie con Agrobacterium tumefaciens offre ulteriori vantaggi perché il tempo tra il punto di consegna del DNA e la consegna di un prodotto purificato è ridotto da anni a meno di 2 mesi 3. Espressione transiente è utilizzato anche per l'analisi funzionale, ad esempio per testare geni per la loro capacità di complementare perdita di funzione mutanti o per indagare le interazioni proteina 4-6. Tuttavia, i livelli di espressione transiente tendono a mostrare una maggiore variazione da lotto a lotto di livelli di espressione in piante transgeniche 7-9. Ciò riduce la probabilità che i processi di produzione biofarmaceutica basati su espressione transiente will essere approvato nel contesto delle buone prassi di fabbricazione (GMP), perché la riproducibilità è un attributo di qualità critico ed è soggetta a valutazione del rischio 10. Tale variazione può anche mascherare tutte le interazioni che i ricercatori intendono studiare. Pertanto, abbiamo deciso di identificare i principali fattori che influenzano i livelli di espressione transitori nelle piante e per costruire un modello predittivo quantitativo di alta qualità.
L'approccio a un fattore-at-a-tempo (OFAT) è spesso usato per caratterizzare l'impatto (effetto) di alcuni parametri (fattori) sull'esito (risposta) di un esperimento 11. Ma questo è ottimale perché le prove individuali (va) durante l'indagine (esperimento) saranno allineati come perle di una collana attraverso l'area potenziale attraversato dai fattori che vengono testati (spazio di progettazione). La copertura dello spazio di progettazione e quindi il grado di informazioni derivate da dell'esperimento èbasso, come mostrato nella Figura 1A 12. Inoltre, interdipendenze tra diversi fattori (interazioni factor) possono rimanere nascosti conseguente modelli poveri e / o la previsione di falsa Optima, come mostrato nella Figura 1B 13.
Gli inconvenienti sopra descritti possono essere evitati utilizzando un disegno di avvicinamento esperimenti (DOE) in cui viene eseguito su un esperimento sono sparsi in modo più uniforme in tutto lo spazio di progettazione, il che significa che più di un fattore è variata tra due piste 14. Ci sono disegni specializzati per le miscele, screening fattori (disegni fattoriali) e la quantificazione degli impatti dei fattori sulle risposte (metodi di superficie di risposta, RSM s) 15. Inoltre, RSMS possono essere realizzati come disegni centrali-composito ma possono anche essere realizzati efficacemente con software specializzato che può applicare diversi criteri per la selezione di piste. Ad esempio, il cosiddetto D-optimality criterio selezionerà corre in modo da minimizzare l'errore nei coefficienti del modello risultante, mentre il criterio IV-ottimalità seleziona piste che raggiungono la varianza previsione basso tutto lo spazio di progettazione 15,16. La RSM descriviamo qui permette la quantificazione precisa espressione transiente della proteina nelle piante, ma può essere facilmente trasferito su qualsiasi sistema che coinvolge vari (~ 5-8) fattori numerici (ad esempio temperatura, tempo, concentrazione) e alcuni (~ 2 - 4) fattori categorici (es. promoter, colore) in cui una descrizione meccanicistica non è disponibile o troppo complesso per modellare.
L'approccio DoE origine nelle scienze agrarie, ma si è diffuso ad altre zone, perché è trasferibile a qualsiasi situazione in cui è utile per ridurre il numero di corse necessarie per ottenere dati affidabili e generare modelli descrittivi per processi complessi. Questo a sua volta ha portato all'inserimento del DoE nella "Guida perIndustria, Q8 (R2) Pharmaceutical Development ", pubblicato dalla Conferenza internazionale sull'armonizzazione dei requisiti tecnici per la registrazione dei prodotti farmaceutici per uso umano (ICH) 17. DoE è ormai ampiamente utilizzato nella ricerca scientifica e l'industria 18. Tuttavia, la cura deve essere presa durante la pianificazione e l'esecuzione dell'esperimento, perché la scelta di un grado del polinomio improprio per il modello multi-lineare di regressione (modello base) possono introdurre la necessità di corse aggiuntive per modellare correttamente tutti gli effetti dei fattori. Inoltre, danneggiati o dati mancanti generano modelli errati e viziata previsioni, e può anche impedire qualsiasi tentativo di costruzione del modello, come descritto nel protocollo e discussione sezioni 18. Nella sezione Protocollo, ci sarà inizialmente stabilito le fasi di pianificazione più importanti per un esperimento a base di RSM e poi spiegare il progetto sulla base del DoE software DesignExpert v8.1., ma disegni simili possono essere costruiti con altri includi softwareng JMP, Modde, e STATISTICA. Le procedure sperimentali sono seguite da istruzioni per l'analisi e la valutazione dei dati.
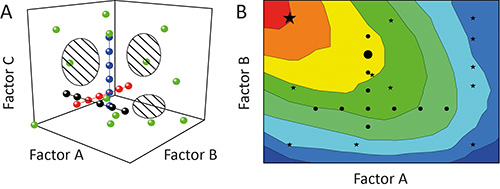
Figura 1. Confronto di OFAT e DoE. A. Variazione sequenziale di un fattore alla volta (OFAT) in un esperimento (nero, rosso e blu cerchi) raggiunge una bassa copertura dello spazio di progettazione (regioni tratteggiate). Al contrario, la variazione di più di un fattore alla volta utilizzando la progettazione di esperimenti (DoE) strategia (cerchi verdi) migliora la copertura e quindi la precisione dei modelli risultanti. B. La copertura dello spazio di progettazione di parte significa che gli esperimenti OFAT (cerchi neri) può anche non riuscire a identificare le regioni operative ottimali (rosso) e prevedere soluzioni sub-ottimali (grande cerchio nero), mentre DoE strategies (stelle nere) sono più propensi a identificare le condizioni preferibili (grande black star).
Protocollo
1. Pianificazione di una strategia DoE
- Identificare i fattori e le risposte pertinenti per l'inclusione nel progetto.
- Definire una o più risposte per la misura. Qui, sono stati usati i livelli di espressione 2G12 e DsRed (mcg / ml), compresa la differenza minima rilevabile considerate pertinenti (10 e 20 mg / ml, rispettivamente) e un valore approssimativo per la deviazione standard stimata del sistema (4 e 8 mg / ml, rispettivamente) sulla base di esperimenti precedenti.
- Utilizzare la letteratura disponibile, i dati provenienti da esperimenti precedenti o motivi di screening specializzati (ad esempio, un disegno fattoriale, vedi l'introduzione) per selezionare i fattori significativi il cui impatto sulle risposte saranno quantificati (Tabella 1) 7,8,19,20.
- Assegnare i tipi di fattori (numerici o categoriche) e selezionare gli intervalli entro cui sarà variato i fattori numerici durante l'inchiesta DoE (Tabella 1).
- Identificare i Numeric fattori per i quali la variazione continua è di difficile attuazione.
- Evitare di utilizzare variazione continua per fattori che non possono essere regolati con precisione ad un certo livello. Esempio temperatura di incubazione in genere può essere controllato solo entro ± 2 ° C, quindi variazione continua utilizzando valori quali 27,2 ° C, 25,9 ° C e 29,3 ° C must essere evitato.
- Invece, selezionare un numero di livelli discreti per questi fattori (Tabella 1). Il numero di livelli deve corrispondere al modello base previsto (punto 1.2). Questo è importante solo per i disegni ottimali, perché i disegni compositi centrali hanno sempre livelli dei fattori discreti.
- Assegnare se un fattore categorico è nominale, cioè senza un ordine implicito (ad esempio, diversi produttori), o ordinale e quindi un parametro numerico discreto (ad esempio diverse foglie su una pianta).
- Selezionare i livelli per entrambi i tipi di fattori categorici.
- Selezionare un modello base utile.
- Sulla base di esperimenti preliminari e letteratura, anticipare la relazione tra ciascun fattore e la risposta, nonché le interazioni tra fattori e la risposta. Ad esempio, lineare (più sono e meglio), quadratico (singolo aumento / diminuzione ottimale o non lineare) o cubica (distorto ottimale).
- Assicurarsi che il numero di livelli per i fattori numerici discreti è n + 1, con n è il grado del polinomio della relazione tra il fattore e la risposta. Ad esempio, se la temperatura dovrebbe avere un effetto sulla risposta quadratica, n = 2 e quindi almeno tre livelli di temperatura deve essere studiato per ottenere un adattamento all'effetto quadratico.
- Definire la frazione di spazio di progettazione (FDS) per cui la varianza previsione dovrebbe essere inferiore a una certa soglia (livello alfa). La FDS deve essere> 0,95 (coprire più del 95% dello spazio di progettazione) per ottenere modelli che generano previsioni affidabili in tuttal'intero 21-23 spazio di progettazione.
Nota: Una soglia di 0.05 corrisponde ad un livello di significatività del 5% (errore di tipo I) ed è un valore tipico. Aumentando questo valore si ridurrà il numero di esperimenti necessari per una strategia DoE ma allo stesso tempo aumentare la probabilità che effetti significativi saranno perse e che le stime di impatto sulla risposta sarà corretta.

Tabella 1. Fattori che influenzano l'espressione della proteina transitoria tabacco tra cui la variazione intervalli durante DoE. Fattori in grassetto sono stati inclusi solo nel design per gli esperimenti descritti in "Un modello descrittivo per l'accumulo DsRed durante espressione transiente utilizzando diversi promoter / 5'UTRs" considerando che i fattori in corsivo sono stati inclusi solo nel design per "Ottimizzazione incubatiocondizioni n e schemi di raccolta per la produzione di anticorpi monoclonali in impianti che utilizzano espressione transiente ".
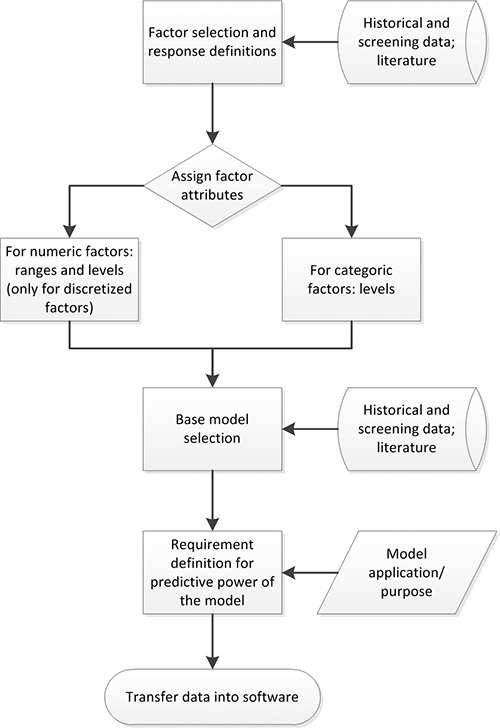
Figura 2. Processo di pianificazione DoE. Fattori con un impatto significativo sulla risposta in esame sono selezionati sulla base dei dati disponibili. Poi vengono assegnati gli attributi fattori (ad esempio numerico), gamme e livelli. Conoscenze e gli esperimenti precedenti sono usati per definire un modello base adatta. I requisiti di alimentazione predittivi sono definite in base all'applicazione / scopo del modello finale. I dati raccolti possono poi essere trasferiti in caso di software DoE.
2. Impostazione di una RSM in DesignExpert
- Avviare DesignExpert e selezionare "New Design". Nella finestra "Response Surface", scegliere &# 34; ottimale "e immettere il numero di fattori numerici e categoriche selezionati nella sezione 1.1).
- Immettere i nomi dei fattori, unità, tipi, sottotipi, il numero di livelli / frontiere e valori per i livelli di livello per tutti i fattori nei campi corrispondenti.
- Continua alla pagina seguente e sotto "Cerca" Opzioni selezionare "Best", così come criterio desiderato "ottimalità", qui "D-ottimale".
- Nel menu "Modifica modello ...", selezionare il modello che contiene i fattori e le interazioni previste al punto 1.2). In caso di dubbi, selezionare un modello completo, comprendente tutti i fattori e le interazioni per un determinato grado del polinomio, qui "quadratiche". Si noti che la selezione di un modello completo (contenente tutte le interazioni) può aumentare il numero di esperimenti necessari per il DoE.
- Selezionare il numero di "Blocchi". Qui, un unico blocco è stato utilizzato perché tutte le piante sono state dallo stesso lotto, tutti i batteri erano state coltivate simultaneamente etutte le iniezioni sono state effettuate nello stesso giorno dallo stesso operatore. Utilizzare più di un blocco se più di una partita di vegetali viene utilizzato o più operatori gestire le iniezioni.
- Bilanciare il disegno
- Abilita "Forza equilibrio categorico", al fine di distribuire i percorsi di sperimentazione in modo uniforme tra i livelli di fattore categoriche, ad esempio, diversi promotori che vengono testati.
Nota: questo può ridurre l'ottimalità della progettazione di un grado che DesignExpert riferirà come valore% a seconda dei punti selezionati dall'algoritmo ottimalità. - Ricalcolare il disegno più volte usando l'algoritmo implementato seme casuale per minimizzare la riduzione di ottimalità.
Nota: le perdite di ottimalità possono variare da ~ 3-40% utilizzando gli stessi dati di input, ma cambiando il numero di repliche può essere utilizzato per mantenere l'equilibrio categorico.
- Abilita "Forza equilibrio categorico", al fine di distribuire i percorsi di sperimentazione in modo uniforme tra i livelli di fattore categoriche, ad esempio, diversi promotori che vengono testati.
- Il software suggerisce un valore per il numero di "punti" modello basato sul modello base, Qui 70 corse. Regolare il numero di corse per "replicati" e "Per stimare la mancanza di adattamento" per assicurarsi che sia> 5% del numero di "punti" per ogni modello, qui 10 piste ciascuno.
- Continua alla pagina successiva, selezionare il numero di risposte e inserire i loro nomi e le unità. Risposte addizionali possono essere aggiunti in qualsiasi momento durante la valutazione dei dati senza alcun impatto negativo sul disegno.
- Continua per avviare l'algoritmo, calcolo dei livelli di fattore per le piste del DoE. Se il modello di base scelto non corrisponde al numero di livelli per un certo fattore, verrà visualizzata una notifica e il calcolo non si avvia. Anche in questo caso:
- Aumentare il numero di livelli di questo fattore, o
- Deselezionare i termini nel modello di base che non può essere calcolata in base al numero attuale di livelli.
- Ad esempio, se vi sono due livelli per il fattore t "tempo di incubazione" (cioè 2 d e 5 d) in tegli design attuale e un modello di base quadratico compresa la t termine è stato selezionato 2, aggiungere un terzo livello per t (cioè 8 d) o rimuovere il fattore t 2 dal modello.
- Salvare il foglio di calcolo contenente le combinazioni ottimali dei fattori che viene visualizzato una volta che il calcolo è completo.
- Nel nodo "Design" selezionare "Evaluation" sub-nodo e passare alla scheda "Grafici".
- Nella "strumento Grafici" selezionare "FDS" e nella casella "FDS Graph" scegliere "Pred", come il tipo di errore. Poi inserire la differenza rilevabile minimo e il valore approssimativo per la deviazione standard stimata del sistema definito nella sezione 1.1.1) come valori di "d" e "s" rispettivamente (qui 20 e 8 mg / ml per DsRed). Inserire anche il livello alfa (accettabile% manca un effetto significativo) adatto per l'applicazione, tipicamente 0,05 (5%).
- Assicurarsi che la FDS calcolato corrisponde alla percentuale definita al punto 1.3) (in genere> 0,95 per i modelli predittivi). Una curva piatta nella trama FDS è preferibile, indicando uniforme accuratezza della stima per tutto lo spazio di progettazione.
- Se questo non è il caso (come si trova qui, FDS è stato 1% come mostrato in Figura 3A), torna al nodo "Design" e nella barra dei task selezionare "Strumenti di progettazione", poi "Potenziati Design ..." e scegliere " Aumentare ".
- Selezionare gli stessi criteri "Ricerca" e "ottimalità" come prima. Ha scelto anche lo stesso delle impostazioni "forza equilibrio categorico" "modello Edit" e. Se l'aumento di progettazione viene eseguita prima di ogni esperimento (come fatto qui), quindi modificare l'opzione "Metti corre nel blocco" su "Block1".
- Nella sezione "corre" inserire il numero di ulteriori "punti Girl", mantenendo almeno il 5% "replicati" e "Per stimare la mancanzadi misura "nel disegno totale. Qui, 100 corse aggiuntive sono state aggiunte e senza" replicati "e" Per stimare la mancanza di adattamento "sono stati inclusi.
- Terminato il calcolo finito, riesaminare il grafico FDS come descritto sopra. Se il FDS non è ancora soddisfacente, ripetere l'aumento progettazione come descritto sopra. Qui il FDS è stata del 100% dopo l'aumento e quindi nessun ulteriore aumento era necessario (Figura 3B).

Figura 3. Confronto tra trame FDS. Un. DOE costituito da 90 piste produce un FDS insufficiente di solo 1% per l'errore standard della previsione, utilizzando il modello base quadratica in combinazione con i valori per la differenza minima rilevabile (20 mcg / ml) e stimaaccoppiato deviazione standard del sistema (8 mg / ml). B. Aumento del doe per un totale di 210 piste raggiunto un FDS 100% e una curva piana indica precisione uniforme del modello in tutto lo spazio di progettazione.
3. Clonazione e analisi di espressione cassette
- Coltivare Escherichia coli in 5 ml di terreno LB (10 g / L di triptone, estratto di lievito 5 g / L, NaCl 170 mM, 50 mg / L ampicillina; per piastre LB-agar includono 15 g / L di agar) a 37 ° C per 6 -8 ore o durante la notte su un agitatore orbitale a 160 rpm. ATTENZIONE: ampicillina è una sostanza nociva. Seminare il terreno LB sia con 50 ml di liquido di coltura o di cellule di trasferimento di colonie cresciute su piastre con una punta di pipetta.
- Per la purificazione di DNA plasmidico PSO, ed altri derivati PPAM (GenBank AY027531), da E. coli K12 DH5a, seguire le istruzioni contenute nel kit di purificazione del DNA manuale 24. ATTENZIONE: la purificazione kcontiene sostanze chimiche nocive (vedi sopra il manuale per i dettagli). La sequenza plasmide è descritto nella Figura 4 e dall'Acquirente et al. 20.
- Determinare la concentrazione di DNA nell'eluato purificata misurando l'assorbanza di un campione di 2 ml a 260 nm in un dispositivo NanoDrop.
- Confermare l'identità del DNA plasmidico purificato mediante digestione con endonucleasi di restrizione (RES).
- Selezionare ORI secondo la sequenza plasmide così che vengono generati modelli di frammento unici e distinguibili. Seguire le raccomandazioni del costruttore per le condizioni di digestione come il volume di reazione, tempo, temperatura e concentrazione dello stabilizzatore albumina sierica bovina. A seconda della sensibilità del dispositivo di analisi, utilizzare 50-500 ng di DNA plasmidico per la digestione.
- Preparare 0.8-2.0% gel per la separazione di frammenti di DNA mediante ebollizione agarosio in Tris-borato-EDTA (TBE) tampone (Tris 90 mM, borato 90 mM, EDTA 2 mM, pH 8,0). Tha grandi frammenti attesi, meno agarosio deve essere utilizzato nel gel.
- Aggiungere 5 ml di tampone campione di cinque volte (5x SABU, 0.1% (w / v) di blu di bromofenolo, 0.1% (w / v) xilene cyanol, 10% (w / v) di glicerolo disciolto in TBE) a 50 ml di RE-trattati campione di DNA e separare i frammenti mediante elettroforesi su gel di agarosio a 100 V per ~ 40 minuti o fino netta separazione dei frammenti si ottiene. Su ogni gel, includere una corsia contenente marcatori di dimensioni scaletta del DNA per il confronto, ad esempio 2-3 microlitri scala 1-kb.
- Sostituire l'omega 5'UTR in PSO con una delle altre tre 5'UTRs.
- Rilasciare la sequenza 5'UTR omega da ~ 4 microgrammi di DNA purificato PSO mediante trattamento con 20 unità di Eco RI-HF e NCO ER I-HF in NEBuffer 4 a 37 ° C per ~ 60 min. Poi separare i frammenti come descritto nelle sezioni 3.4.2 e 3.4.3.
- Isolare il frammento più grande (pS, "spina dorsale") dal gel di agarosio utilizzando un gel supplementarekit ction secondo il produttore del manuale 25 e determinare la concentrazione del DNA purificato come descritto nella sezione 3.3). ATTENZIONE: il kit di estrazione gel contiene sostanze chimiche nocive, consultare il manuale per i dettagli.
- Isolare il CHS, CHS-LPH e TL 5'UTRs da vettori donatori idonei trattando ~ 10 mcg di ogni vettore con 20 unità di Eco RI-HF e ER Nco I-HF in NEBuffer 4 a 37 ° C per ~ 60 min. Poi separare i frammenti come descritto nelle sezioni 3.4.2 e 3.4.3 e purificare il più piccolo frammento 5'UTR contenenti come descritto nella sezione 3.5.2.
- Legare isolate 5'UTRs purificati in aliquote separati per l'isolato purificato vettore pS lineare a 25 ° C per 5 minuti secondo le raccomandazioni del costruttore 26. Utilizzare ~ 50 ng di DNA del vettore e un eccesso molare triplice DNA 5'UTR in un volume totale di 20 microlitri.
- Trasformazione E coli con the plasmidi ricombinanti 26,27.
- Aggiungi ~ 10 ng (~ 4-5 ml) di miscela di legatura (vedi sezione 3.5.4) per 50 microlitri RbCl competente E. coli e mescolare delicatamente, e poi incubare per 30-60 min in ghiaccio. Shock termico per 1,5 minuti a 42 ° C e raffreddare in ghiaccio per 5-30 min.
- Aggiungere 950 microlitri di libera-antibiotico terreno LB e incubare per 1 ora a 37 ° C e 160 rpm. Per la selezione dei trasformanti, diffondere 50 e 100 microlitri della coltura su piastre di agar LB contenenti ampicillina e incubare per ~ 16-20 ore a 37 ° C.
- Inoculare 5-10 colonie separate rappresentano ogni legatura promotore-5'UTR descritto nel capitolo 3.1 in aliquote di 5 ml di terreno LB contenente ampicillina. Poi purificare il DNA plasmide e confermare la sua identità come descritto nelle sezioni 3,2-3,4.
- Sostituire il promotore 35SS con il promotore nos in ciascuna delle quattro plasmidi 5'UTR utilizzando ER Asc I e Eco RI in NEBuffer 4 a 37 ° C per 1 ora come descritto nelle sezioni 3.5 e 3.6.
- Introdurre ciascuno degli otto plasmidi risultanti in A. tumefaciens ceppo GV3101: pMP90RK mediante elettroporazione 28.
- Aggiungere ~ 500 ng di DNA plasmide purificato al 50 microlitri competente A. tumefaciens cellule su ghiaccio. Mescolare delicatamente e trasferire in un prechilled 0,2 centimetri elettroporazione cuvetta. Assicurarsi che la miscela è al fondo della cuvetta e non contiene bolle.
- Cellule Pulse a 2,5 kV per 5 msec e confermare l'intensità e la durata dell'impulso.
- Evitare concentrazioni saline elevate nel campione di DNA o scorretta preparazione del A. competente tumefaciens cellule perché questi possono causare elevate correnti ioniche causando vaporizzazione istantanea di sospensione cellulare, riducendo notevolmente trasformazione efficacia.
- Aggiungere 950 pl di mezzo YEB senza antibiotici (estratto 5 g / L di manzo, estratto di lievito 1 g / L, 5 g / L Peptone, 5 g / L di saccarosio, 2 mM MgSO 4, pH 7.0), mescolare delicatamente e trasferire immediatamente in un tubo di reazione da 1,5 ml sterile. Incubare per 2-4 ore a 26-28 ° C e 160 rpm.
- Stendere 1-2 ml su piastre di agar YEB contenenti antibiotici (50 mg / L carbenicillina, 25 mg / L di kanamicina, 25 mg / L rifampicina) per la selezione dei trasformanti. ATTENZIONE: rifampicina è una sostanza tossica.
- Inoculare tre aliquote di 5 mL di terreno YEB contenenti antibiotici con cellule provenienti da diverse colonie rappresentano ogni legatura promotore-5'UTR e incubare per 48-72 ore a 26-28 ° C e 160 rpm.
- Confermare il successo di A. tumefaciens trasformazione.
- Trasferimento di 2 ml di ciascuna aliquota da sezione 3.8.6 per separare i 48 aliquote microlitri di PCR mastermix (2 microlitri di ogni 10 micron di primer magazzino (400 Nm concentrazione finale di ciascun primer),1 microlitri 10 mM dNTP mix (200 micron concentrazione finale di ogni trifosfato deossinucleoside), 5 microlitri di 10 volte Expand buffer di alta fedeltà con 15 mM MgCl 2, 0,75 ml Espandi High Fidelity mix di enzimi, e 39,25 ml di acqua distillata sterile).
- Amplifica la cassetta di espressione di ogni plasmide utilizzando primer appropriati (FWD: 5'-CCT CAG GAA GAG CAA TAC-3 ', vincolante 1.026 nucleotidi a monte del promotore; REV: 5'-CCA AAG CGA GTA CAC AAC-3', vincolante all'interno del sito di poliadenilazione 35S) in adeguate condizioni di PCR. Qui, 94 ° C è stato utilizzato per la denaturazione iniziale seguita da 30 cicli di 94 ° C denaturazione per 15 sec, 51 ° C ricottura per 30 sec e 72 ° C per 120 sec allungamento, ed una fase di allungamento finale a 72 ° C per 8 min.
- Determinare la dimensione dei prodotti di PCR mediante elettroforesi su gel di agarosio come descritto nella sezione 3.4.3.
- Preparare A. tumefaciens scorte glicerolo damiscelando 500 microlitri 50% (v / v) di glicerolo sterile con 500 microlitri di A. tumefaciens culture sezione 3.8.6 per i quali la trasformazione è stata confermata (punto 3.9.3). Conservare le scorte di glicerolo a -80 ° C fino all'utilizzo.
- Calcolare le energie di piegatura dei diversi mRNA utilizzando il server web RNAfold 29 e comprende la sequenza nucleotidica dal sito di inizio della trascrizione attraverso il primo 50 bp della regione codificante.
- Nella sezione "Fold algoritmi e le opzioni di base" selezionare la voce "energia libera minima (MFE) e la funzione di partizione" e "evitare isolate coppie di basi" opzioni.
- In "Opzioni di piegatura avanzate" scegliere "penzoloni energie su entrambi i lati di un elica in ogni caso", "parametri di RNA (modello Turner, 2004)" e modificare "riscalare parametri energetici di data temperatura (C)" a 25 ° C.
- Nella sezione "Opzioni di uscita", selezionare tutte le opzioni.
- Dalla "Risultati per termodinamica ensemble di previsione" del file di output, estrarre i "free energy dell'ensemble termodinamico" e la "frequenza della struttura MFE nel complesso" valori per il confronto.
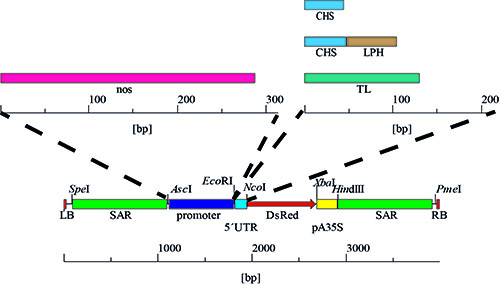
Figura 4. Promotore e 5 'UTR varianti. Le cassette di espressione sono stati generati dalla borsa graduale del 5'UTR, risultante in quattro combinazioni con promotore CaMV 35SS, seguita dalla sostituzione di questa con la sequenza promotore nos cedevole quattro ulteriori varianti e un totale di otto diverse combinazioni promotore / 5'UTR.
4. La coltivazione delle piante
- Preparare una soluzione allo 0,1% del fertilizzante Ferty2 Mega in acqua deionizzata a pH 5.9.
- Preparare 10 blocchi x 10 x 8 cm di lana di roccia da un ampio risciacquo con acqua deionizzata per rimuovere le sostanze chimiche residue e infine equilibrare con fertilizzante.
- Seed piante di tabacco, ponendo 1-2 semi di tabacco su ogni blocco lana di roccia, seguito da un breve filo con fertilizzante avendo cura di evitare di lavare i semi di distanza.
- Germinare e coltivare le piante di tabacco per 42 giorni in una serra a temperatura 25/22 ° C giorno / notte, 70% di umidità relativa e di un fotoperiodo di 16 ore (180 mmol sec -1 m -2; λ = 400-700 nm) . Durante questo fotoperiodo, irrigare con fertilizzante per 15 minuti ogni ora in coltura idroponica.
5. Transient Protein Expression
- Preparare A. tumefaciens per l'iniezione in foglie.
- Inoculare 5-50 ml di terreno YEB con antibiotici contenenti 1% A. tumefaciens crio-magazzino cultura e incubare a 27 ° C fino alla OD 600nm raggiunge 5.0 (~ 48-72 ore a seconda del volume e il tipo di nave).
- Diluire la A. cultura Agrobacterium con acqua e 2 volte medio infiltrazione (4,3 g / L Murashige e Skoog sali (pH 5,6), 5 g / L di saccarosio, 1,8 g / L di glucosio, acetosyringone 100 mM) adatta al OD 600nm richiesto per iniezione. Confermare il 600nm OD appena prima dell'iniezione. Nota: Un 600nm OD di 1.0 corrisponde a ~ 1,43 ± 0,12 x 10 9 unità formanti colonia per ml.
- Iniettare la A. tumefaciens sospensione in foglie.
- Selezionare ed etichettare le foglie e le posizioni noncotyledon al riguardo di essere trattati (ad esempio, secondo una strategia DoE).
- Non iniettare diverso A. tumefaciens soluzioni nello stesso campo intercostale. Invece, utilizzare le sezioni opposte di ciascun lato dell'asse medio-venosa. Se devono essere iniettato nella stessa posizione più di due diverse soluzioni di utilizzare un additional impianto.
- Agitare accuratamente A. soluzione tumefaciens per sospendere tutte le cellule che possono essere stabiliti dal preparare la diluizione e aspirato in una siringa da 1 ml.
- Graffiare delicatamente l'epidermide nella posizione prevista per l'iniezione con un puntale o simile per agevolare l'afflusso del A. tumefaciens soluzione. Evitare la rottura della lamina fogliare, mentre farlo.
- Tenere la siringa perpendicolare alla lama foglia toccare la canna contro il campo intercostale da trattare e spingere delicatamente l'uscita (senza ago inserito) sul lato inferiore della foglia. Premere il lato superiore della foglia delicatamente allo stesso tempo per impedire che la lama foglia di spostamento o rottura.
- Premere delicatamente il pistone della siringa. Il A. soluzione tumefaciens possa penetrare negli spazi intercellulari all'interno della lamina fogliare come indicato dalle zone trattate appaiono verde scuro e umido. Ripetere questa procedura in diverse posizioni fino a quando l'intero intercostal campo è infiltrato con A. tumefaciens. Quindi continuare con il campo intercostale successivo.
- Assicurarsi che la siringa rimane perpendicolare alla foglia. Inclinando la siringa causerà la sospensione batterica per schizzare sotto alta pressione.
- Se diverso A. soluzioni tumefaciens sono utilizzati (ad esempio per testare differenti promotori), rimuovere la soluzione in eccesso rimanente sul lato inferiore della foglia dalla prima iniezione con un tovagliolo di carta o simili prima di applicare la soluzione successiva.
- Post-infiltrazione incubazione di piante e campionamento.
- Preparare una fitotrone per le piante trattate ~ 24 ore prima dell'iniezione in modo temperatura e umidità equilibrazione ai livelli richiesti per il DoE.
- Dopo l'iniezione, trasferire piante nel fitotrone e metterli in vassoi abbastanza grandi per l'irrigazione con acqua fino al termine del periodo di incubazione determinata dal DOE.
- Stabilire un phot 16 oreoperiod utilizzando sei bianco 36 tubi fluorescenti Osram freschi W per 0,7 m 2 (75 mmol sec -1 m -2; λ = 400-700 nm). Evitare che le piante ombreggiatura reciprocamente limitando il numero di piante a sei per 0,7 m 2.
- Prima del campionamento, assicurarsi che il campo intercostale corretta è stata selezionata confrontando l'etichetta aggiunto nella sezione 5.2.1 e il piano DoE.
- Utilizzare una sonda di carotaggio per rimuovere 4-5 dischi fogliari dai campi intercostali trattati nelle posizioni ed i tempi indicati dal DoE. Non rimuovere l'intera foglia dalla pianta durante il campionamento. Stabilizzare la foglia con un tovagliolo di carta a mano durante la rimozione dei dischi per evitare la rottura.
- Determinare la massa di ciascun campione e collocarlo in un tubo di plastica di reazione da 1,5 ml etichettate con il nome del campione e di massa. Conservare i campioni a -20 ° C o -80 ° C prima proteina quantificazione. Il processo può essere messo in pausa in questa fase per diversi mesi a seconda della stabilità del campionee la temperatura di conservazione.
6. Proteine quantificazione
- Estrarre proteine dai campioni disco di foglia.
- Aggiungere 3 ml di tampone di estrazione (fosfato di sodio 50 mm, 500 mm di cloruro di sodio, pH 8,0) per mg di massa del campione e macinare dischi fogliari nel tubo di reazione con un pestello elettrico fino a quando non rimangono grandi frammenti. Evitare il surriscaldamento del campione.
- Rimuovere i solidi dispersi mediante centrifugazione del campione due volte a 16.000 xg per 20 min a 4 ° C. Trasferire il surnatante in una provetta pulita reazione 1,5 ml dopo ogni passaggio senza disturbare il pellet.
- Dopo centrifugazione, il processo può essere messo in pausa il congelamento estratti vegetali a -20 ° C o -80 ° C per diversi mesi a seconda della stabilità del campione e la temperatura di conservazione. Verificare che un ciclo di congelamento-scongelamento non influenza la concentrazione della proteina bersaglio (s).
- Misurare la fluorescenza DsRed.
- Preparare tre repliche tecniche di ogni campione in 96 pozzetti piastre mezza zona nera (50 estratto microlitri per pozzetto). Evitare la formazione di bolle durante il pipettaggio.
- Utilizzare una serie di sei diluizioni (0, 25, 75, 125, 175 e 225 mcg / ml) di uno standard DsRed per piastra a 96 pozzetti per generare una curva di riferimento liner. Preparare le diluizioni in PBS e conservare a 4 ° C per l'uso entro 3 mesi.
- Misurare la fluorescenza due volte, in sequenza, in un lettore di piastre a 96 pozzetti muniti 530/25 nm di eccitazione e 590/35 nm filtri di emissione.
- Per ogni campione, la media la fluorescenza sul due letture e le tre repliche tecniche e sottrarre il valore registrato per il controllo in bianco contenente 0 mg / ml DsRed. Sottrarre Anche questo valore dalla legge delle diluizioni standard e utilizzare questi valori vuoti corretti di una regressione lineare ottenendo una curva di riferimento (per l'origine delle ordinate).
- Utilizzare la pendenza della curva di riferimento per convertire il fluorescence misurata per i campioni in concentrazioni DsRed. Se necessario, diluire i campioni in modo che la lettura rientra nell'intervallo concentrazione degli standard e considerare questo fattore di diluizione nei calcoli successivi.
- Determinare la concentrazione di 2G12.
- Preparare un dispositivo di risonanza plasmonica di superficie (SPR) per la misura della concentrazione di anticorpi accoppiando Proteina A alla superficie attiva di una cella di flusso. Utilizzare un'altra cella di flusso come riferimento inattivando la superficie senza accoppiamento di Proteina A 30,31.
- Diluire estratti vegetali 1:20 in SPR tampone di corsa (10 mM HEPES pH 7,4, 3 mM EDTA, 150 mM NaCl, 0,05% v / v Tween-20) e misurare le unità di risposta (RU) di legame dell'anticorpo alla proteina A in tre replicati tecnici di ciascun campione al termine di una iniezione di 90 ml (180 sec a 30 ml / min). Sottrarre l'UR misurata alla cella di flusso di riferimento (non Protein A) dal RU misurata nel fl sperimentaleow cella (Protein Una superficie).
- Misura standard 585 ng / ml 2G12 dopo ogni 10-15 campioni e sottrarre il valore misurato nelle celle di flusso di riferimento come descritto sopra. Utilizzare il RU media di queste norme per calcolare una curva di riferimento lineare per l'origine delle ordinate.
- Calcolare le concentrazioni 2G12 nei campioni in base alla diluizione 1:20 e la pendenza della curva di riferimento.
- Controllare forte legame non specifico per la cella di riferimento, che può danneggiare la misura. Inoltre, verificare se le norme 2G12 mantengono valori di circa costanti (<5% di variazione) per tutta l'analisi, come una maggiore variazione riflette l'invecchiamento della proteina Una superficie.
7. Analisi dei dati e valutazione
- Analizzare manualmente le risposte osservate negli esperimenti di espressione per (i) valori estremi errori (alto o basso) che indica di misura, (ii) risultati insoliti, per esempio combinazioni di fattori che genivalutare le risposte impreviste che indicano scambio di dati, e (iii) valori mancanti.
Nota: utilizzare queste azioni per prevenire modello di edificio basato su dati errati. - Trasferire i dati di risposta analizzati (qui le concentrazioni di proteine) nel nodo "Design" di DesignExpert. Assicurarsi che i dati di risposta vengono assegnati correttamente le impostazioni dei fattori corrispondenti. Vi è una vasta sezione di aiuto disponibile nel software DesignExpert che copre gli aspetti discussi di seguito.
- Nel nodo "Analisi", scegliere la risposta da analizzare e inizialmente selezionare "none" nella scheda di trasformazione.
Nota: una trasformazione è consigliato per min / max rapporti della risposta maggiore di 10. La trasformazione più utile può essere ottenuto dal Box-Cox-plot nella scheda "Diagnostica" nella sezione "Diagnostica" della "strumento di diagnostica" descritta di seguito (paragrafo 7.8). A Log 10 trasformazione è spesso opportuno. - Continua alla scheda "Riepilogo Fit", che fornisce informazioni di carattere generale sui fattori che sono importanti per il sistema in esame (ad esempio interazioni a due fattori, effetti di secondo grado). Il software suggerisce un modello iniziale basata sul suo significato.
- Nella scheda "Modello", un modello iniziale è preselezionata in base ai risultati "riepilogo Fit". Utilizzare la modalità automatica per modificare questo modello:
- Selezionare un "ordine di processo" che è un ordine superiore al modello proposto, ad esempio, se il modello proposto è "2FI" (due interazione fattore), quindi selezionare "quadratica".
- Scegliere "indietro" nel campo "Selezione", che in modo iterativo rimuovere i parametri non significative dal modello dopo procedere alla scheda "varianza" in base al valore "Alpha out", che dovrebbe essere inizialmente 0.100.
- Se richiesto dal software, sempre correggere automaticamente la gerarchia del modello, perché questo è necessary per generare modelli affidabili 32,33.
- Nella scheda "ANOVA", studiare il modello proposto e dei fattori inclusi. Se necessario, rimuovere manualmente tutti i fattori con i valori p di sopra di una soglia predefinita (qui 0,05, corrispondente ad un livello di significatività del 5%), o quelli che si basano improbabile in considerazione meccanicistica passando torna alla scheda "Modello", cambiando "Selezione" "Manuale" ed eliminando i fattori appropriati dal modello.
- Torna nella scheda "ANOVA", valutare il modello aggiornato in termini di p-value del "Modello" (un valore basso è auspicabile, perché questo indica un significato) e la (un valore elevato "La mancanza di adattamento" è auspicabile perché questo indica mancanza di significatività) nonché i valori di "R-squared", "R-squared rettificato" e "Predicted R-squared" (valori> 0,80 sono desiderabili per tutti e tre i valori).
- Confrontare questi valori for diversi modelli con / senza fattori a vari livelli di significatività.
Nota: può essere utile per duplicare la colonna risposta nel nodo "Design" ed effettuare ogni analisi singolarmente, o per esportare l'intera tabella "varianza" in un altro programma, ad esempio un foglio di calcolo.
- Confrontare questi valori for diversi modelli con / senza fattori a vari livelli di significatività.
- Proseguire fino alla scheda "Diagnostica" per confermare la qualità del modello e rilevare potenziali valori anomali nel dataset che hanno una forte influenza sul modello esaminando tutte le schede della "Diagnostics Tool" (sezione "Influenza" e "Diagnostica") .
- Nella sezione "Diagnostica" della "strumento Diagnostica", eseguire le seguenti operazioni:
- Assicurarsi che i punti sono sparsi in modo casuale entro i limiti del "Residui vs Predicted" grafico. Qui, una distribuzione a forma di chevron indica la necessità di trasformazione dei dati.
- Assicurarsi che i punti sono sparsi in modo casuale entro i limiti del"Residui vs predetto" grafico. Qui, una distribuzione a forma di chevron indica la necessità di trasformazione dei dati.
- Indagare se i punti della "Residui vs run" grafico a dispersione nel caso i limiti. Un modello (ad esempio "scala") indica una tendenza nell'edificio dati e blocco può essere utilizzato per contrastare la sua influenza sul modello.
- Cercare una linea diagonale diritta che indica il modello ideale nel "Previsto vs Actual" complotto. Maggiore è la dispersione dei dati intorno alla diagonale, meno accurata del modello.
- Verificare la presenza di una sovrapposizione di blu (migliore) e verde (reali) le linee verticali del Box-Cox-plot che indica la trasformazione dei dati. Se la linea verde non corrisponde a quella blu (fuori dell'intervallo indicato dalle linee verticali rosse) torna alla sezione 7.3 e migliorare il giro dell'edificio modello selezionando la trasformazione dei dati suggerito dal Box-Cox-plot. Anche in questo caso la duplicazione del recolonna di risposta accettabili può essere utile per confrontare i diversi modelli.
- Assicurarsi che i punti delle "Residui vs Factor" carte (c'è un grafico per ciascun fattore del modello) si disperdono in modo casuale entro i limiti. Curvatura nella distribuzione dei dati può indicare un fattore mancante nel modello.
- Nella sezione "Influenza" della "strumento Diagnostica", assicurarsi che non vi sia dispersione casuale dei dati entro i limiti dei "residui Esternamente studentizzati", "Leverage", "DFFITS" e trame "DiffBeta" e uniformemente bassa della distribuzione senza valori estremi nella trama "di Cook Distance".
- Nella sezione "Diagnostica" della "strumento Diagnostica", eseguire le seguenti operazioni:
- Nella scheda "grafici modello", visualizzare il modello di valutazione. Per un numero limitato di fattori numerici (ad esempio 3) la superficie di risposta ("superficie 3D") rappresentazione è utile per valutare manualmente Optima / caratteristiche.
Nota: superfici di risposta mostrano solo l'impatto di due fattoritori sulla risposta sotto inchiesta. L'effetto di alcun elemento aggiuntivo sulla risposta si rivela cambiando il suo valore (fattori numerici) o livello (fattori categoriche) nella finestra "strumento Fattori". In alternativa, i fattori possono essere assegnati all'asse complotto di destra-clic su di essi nella finestra "strumento Fattori" e selezionando l'asse variabile indipendente desideri.- Manipolare i livelli di fattore e di assegnarli ai ordinate grafico usando lo "strumento Fattori".
- Grafici esportazione utilizzando il "grafico Esporta file ..." il comando nella scheda "File".
- Utilizzare il "numerica" sub-nodo del nodo "Optimization" Per ottimizzare la risposta numericamente (minimizzare, massimizzare, in range) in funzione dei fattori modello, al quale a sua volta può essere applicato determinati vincoli (ad esempio valori limite e pesi) tramite l' scheda "Criteri".
- Calcolo ed esaminare soluzioni numeriche nelle "soluzioni"scheda basata sul contributo fornito nella scheda "Criteri".
- Esportare queste soluzioni per altri software (ad esempio fogli di calcolo) per ulteriori analisi (ad esempio istogrammi) rivelando le impostazioni dei fattori associati a valori alti o bassi di risposta.
Nota: questo è utile se più di tre fattori numerici sono indagati e rappresentazione 3D è difficile.
- Utilizzare la "predizione Point" sub-nodo per predire la risposta per le impostazioni dei fattori specifici.
- Utilizzare questa previsione per tutte le impostazioni da valutare (qui, tutti promotore e combinazioni 5'UTR, così come tutte le foglie ei tempi di incubazione o posizioni di foglie e temperature di incubazione).
- Esportare i valori previsti e compilati in una matrice di dati che può essere utilizzato per descrivere l'espressione transiente in piante di tabacco, ad esempio calcolando l'espressione di una specifica combinazione promotore-5'UTR in un certo tempo mediato su tutte le posizioni foglia o across tutte le foglie di una pianta.
- Normalizzare i dati di espressione in base alle masse delle diverse foglie per produrre il livello di espressione specifico (mg g -1) o il tasso specifica espressione (mg g -1 h -1).
- In alternativa, esportare i coefficienti della "equazione finale" dalla parte inferiore della scheda "ANOVA" in un foglio e le moltiplica con una serie di impostazioni dei fattori per produrre la stessa matrice di dati.
Nota: questa matrice può contenere solo valori del fattore dall'interno lo spazio di progettazione iniziale, perché l'equazione modello adattato non è adatto per estrapolazione.
- Condurre un esperimento di conferma per i punti del modello che sono di maggiore interesse.
- Eseguire l'espressione della proteina transitoria in condizioni selezionati per (sezione 7.11) "previsione Point". Es. utilizzare le stesse temperature e le foglie, ecc
- Determinare le concentrazioni di proteine di questa transitoria espressione eXperiment come descritto in precedenza (sezione 6) e li confronta con i valori previsti dal modello.
- Controllare se la concentrazione media di proteine nella sperimentazione conferma rientra intervallo di previsione del modello superficie di risposta ottenuto durante punto previsione. Una partita afferma il potere predittivo del modello. Una mancata corrispondenza indica una scarsa qualità del modello e corse aggiuntive possono essere richieste.
- Nota: impianto di variabilità da lotto a lotto amplia l'intervallo di previsione e può svalutare esperimenti di conferma eseguite con un diverso lotto di piante. Tuttavia, i campioni che non sono stati utilizzati per generare il modello, ma che ha avuto origine dalla stessa partita pianta come i campioni contenuti nel modello possono servire come controlli efficaci.
Risultati
Un modello descrittivo per l'accumulo DsRed durante espressione transiente utilizzando diversi promotori e 5'UTRs
Fluorescenza DsRED in estratti di foglie è stato usato per indicare il livello di espressione della proteina ricombinante e quindi è stato usato come la risposta nella strategia DoE. La differenza minima rilevabile abbiamo considerato rilevante era di 20 mcg / ml e la deviazione standard stimata del sistema era di 8 mg / ml in base a esperimenti inizial...
Discussione
Ogni esperimento richiede un'attenta pianificazione, perché le risorse sono spesso scarse e costose. Ciò è particolarmente vero per le strategie DoE perché errori durante la fase di pianificazione (ad esempio la selezione di un modello di base che non copre tutte le interazioni significative dei fattori) possono diminuire sostanzialmente il potere predittivo dei modelli risultanti e quindi svalutare l'intero esperimento. Tuttavia, questi errori possono essere facilmente evitati seguendo procedure di...
Divulgazioni
La tassa di pubblicazione è stato in parte sponsorizzato da aziende Statease, Inc. (USA) e Statcon (Germania), che non sono stati coinvolti nella coinvolti nella preparazione del manoscritto o responsabile per qualsiasi dei suoi contenuti.
Riconoscimenti
Gli autori sono grati al Dott. Thomas Rademacher per fornire il PPAM pianta vettore di espressione e Ibrahim Al Amedi per la coltivazione delle piante di tabacco utilizzate in questo studio. Vorremmo ringraziare il Dott. Richard M. Twyman per la sua assistenza con la modifica del manoscritto. Questo lavoro è stato in parte finanziato dalla Advanced Grant "Future-Pharma" Consiglio europeo della ricerca, il numero di proposta 269.110 e il Fraunhofer Zukunftsstiftung (Fraunhofer Future Foundation).
Materiali
| Name | Company | Catalog Number | Comments |
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life Technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10 x10 cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life Technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
Riferimenti
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ - The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5'UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon