Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Характеристика сложных систем по постановке эксперимента подхода: Переходный Белок Expression табачными в качестве примера
В этой статье
Резюме
Опишем планирование эксперимента подхода, который может использоваться, чтобы определить и смоделировать влияние регуляторных элементов трансгенных, рост и развитие растений, параметров и условий инкубации на временной экспрессии моноклональных антител и репортерных белков в растениях.
Аннотация
Растения обеспечивают множество преимуществ для производства биофармацевтических препаратов, включая низкие затраты, масштабируемость и безопасность. Кратковременная экспрессия предлагает дополнительное преимущество короткого развития и производства раз, но уровни экспрессии могут существенно различаться между партиями таким образом, послуживших основанием для регуляторных проблем в контексте надлежащей производственной практики. Мы использовали дизайн эксперименты (DOE) подхода для определения влияния основных факторов, таких как регуляторных элементов в конструкции экспрессии, рост растений и параметров развития, а также условия инкубации в течение выражения, по изменчивости экспрессии между партиями. Мы протестировали растения, выражающие модель анти-ВИЧ моноклональное антитело (2G12) и флуоресцентный маркер белок (DsRed). Обсуждается обоснование выбора определенных свойств модели и выявить его потенциальные ограничения. Общий подход может быть легко переведены в другие проблемы, поскольку принципы модели Авновь широко применимы: основанной на знаниях выбор параметров, снижение сложности путем разделения исходную задачу на более мелкие модули, программное обеспечение наведением установки оптимальных комбинаций эксперимента и поэтапного проектирования увеличения. Таким образом, методология не только полезно для характеристики экспрессии белка в растениях, но и для исследования других сложных систем, не имеющих механистический описание. Прогнозные уравнения, описывающие взаимосвязь между параметрами можно использовать для установления механистические модели для других сложных систем.
Введение
Производство биофармацевтических белков в растениях является выгодным, поскольку растения являются недорогими расти, платформа может быть расширен только путем выращивания больше растений, и патогены человека не в состоянии воспроизвести 1,2. Переходные стратегии экспрессии на основе, например, на инфильтрацией листьев с Agrobacterium tumefaciens обеспечивает дополнительные преимущества, так как время между точкой доставки ДНК и доставки очищенного продукта снижается с нескольких лет до менее чем за 2 месяца 3. Переходный выражение используется также для функционального анализа, например, для тестирования генов для их способность к комплементации с потерей функции мутантов или для исследования белковых взаимодействий 4-6. Тем не менее, переходные уровни экспрессии как правило, показывают больший разброс от партии к партии, чем уровни экспрессии в трансгенных растениях 7-9. Это снижает вероятность того, что биофармацевтических производственных процессов на основе временной экспрессии Wiбудете утверждены в контексте надлежащей производственной практики (GMP), потому что воспроизводимость является атрибутом критическим качество и подлежит оценке риска 10. Такое изменение может также маскировать любые взаимодействия, что исследователи намерены провести расследование. Таким образом, мы намерены выявить основные факторы, которые влияют переходные уровни экспрессии в растениях и построить качественную количественную модель прогнозирования.
(OFAT) подход один фактор-на-а-время часто используется для характеристики воздействия (эффект) определенных параметров (факторов) на исход (ответ) эксперимента 11. Но это не является оптимальным, так как отдельные тесты (работает) в ходе расследования (эксперимент) будет приведена в соответствие, как жемчужины на нитке через потенциальный области, натянутого на факторы, которые проверены (дизайн помещения). Охват дизайн помещения и, следовательно, от степени информации, полученной из экспериментанизкий, как показано на рисунке 1A 12. Кроме того, взаимозависимости между различными факторами (коэффициент взаимодействия) может оставаться скрытым что приводит к плохим моделей и / или прогнозирования ложной оптимума, как показано на фиг.1В 13.
Недостатки, описанные выше, можно избежать с помощью планирование экспериментов (DOE) подход, в котором работает эксперимента разбросаны более равномерно в течение дизайна помещения, а это означает, что более чем один фактор варьируется между двумя отрезками 14. Существуют специализированные проекты для смесей, скрининг факторов (факторные планы) и количественная оценка воздействия фактора на ответах (методы поверхности отклика, RSM S) 15. Кроме того, RSMS может быть реализована как центральных композиционных конструкций, но также может быть достигнуто эффективно с помощью специализированного программного обеспечения, которое можно применить различные критерии для отбора трасс. Например, так называемый D-optimalitКритерий у выберет работает так, чтобы минимизировать ошибку в коэффициентах полученной модели, в то время как критерий IV-оптимальность выбирает работает, что достижения минимально дисперсию предсказания на протяжении всего дизайна помещения 15,16. РСМ мы описываем здесь позволяет точную количественную оценку временной экспрессии белка в растениях, но он может быть легко переведены в любой системе с участием нескольких (~ 5-8) числовые коэффициенты (например, температура, время, концентрация) и несколько (~ 2 - 4) категориальные факторы (например, промоутер, цвет), в котором механистический описание отсутствует или слишком сложны для моделирования.
Подход DoE возникла в сельскохозяйственных наук, но распространился на другие области, потому что это может быть передана любой ситуации, где это полезно уменьшить количество трасс, необходимых для получения достоверных данных и генерировать описательные модели для сложных процессов. Это в свою очередь привело к включению DoE в «Руководства дляПромышленность, Q8 (R2) Фармацевтическая разработка ", опубликованном Международной конференции по гармонизации технических требований к регистрации лекарственных препаратов для человека (ICH) 17. Министерство энергетики теперь широко используется в научных исследованиях и промышленности 18. Тем не менее, следует соблюдать осторожность во время планирование и проведение эксперимента, потому что выбора неправильного степень полинома для модели множественного линейного регрессионного (базовая модель) можно ввести необходимость в дополнительных трасс правильно моделировать все эффекты фактора. Кроме того, повреждены или отсутствуют данные генерировать неправильные модели и недостатки прогнозы, и даже может предотвратить любую модель здания попытку, как описано в секциях протокола и дискуссионных 18. В разделе протокола, мы первоначально установлен наиболее важные этапы планирования для эксперимента РСМ-основе, а затем объяснить дизайн, основанный на DoE Программное обеспечение DesignExpert v8.1. Однако подобные конструкции могут быть построены с другим программным обеспечением includiнг JMP, Modde и STATISTICA. Экспериментальные процедуры следуют инструкции для анализа и оценки данных.
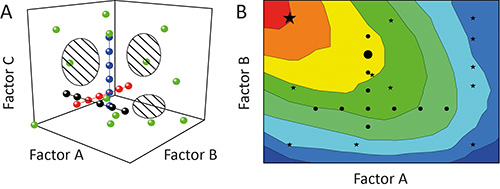
Рисунок 1. Сравнение OFAT и НОО. А. Последовательное изменение одного фактора, в то время (OFAT) в эксперименте (черный, красный и синий кружки) достигает малый охват дизайн помещения (заштрихованные области). В противоположность этому, изменение более чем один фактор за раз, используя конструкцию экспериментов (DOE) Стратегии (зеленые кружки) усиливает охват и, следовательно, точность результирующих моделей. B. Предвзятым дизайн пространства охват означает, что OFAT эксперименты (черные кружки) могут и не выявить оптимальные эксплуатационные регионы (красный) и предсказать суб-оптимальные решения (большой черный круг), в то время как Министерство энергетики стратегическиэс (черные звезды), скорее всего, определить предпочтительные условия (большой черный Star).
протокол
1. Планирование стратегии DoE
- Определите соответствующие факторы и ответы для включения в проект.
- Определите один или несколько ответов для измерения. Здесь были использованы уровни экспрессии 2G12 и DsRed (мкг / мл), в том числе минимальном допустимом разницы рассматривается как соответствующие (10 и 20 мкг / мл, соответственно) и приблизительное значение для предполагаемого стандартного отклонения системы (4 и 8 мкг / мл, соответственно) на основе предыдущих экспериментов.
- Используйте имеющуюся литературу, данные из предыдущих экспериментов или специализированных конструкций скрининга (например, факторного плана, см. введение), чтобы выбрать значимые факторы, воздействие которых на ответах будет количественно (табл. 1) 7,8,19,20.
- Выделяют типы фактор (цифровые или категориального) и выбрать диапазон, в рамках которой цифровые факторы будут изменяться в ходе расследования DoE (табл. 1).
- Определить численныйс факторами, для которых непрерывное изменение трудно реализовать.
- Не следует использовать непрерывное изменение факторы, которые не могут быть отрегулированы точно определенном уровне. Например, температура инкубации обычно можно контролировать только в пределах ± 2 ° C, поэтому непрерывное изменение, используя значения, такие как 27,2 ° С, 25,9 ° С, и 29,3 ° С должна следует избегать.
- Вместо этого, выберите ряд дискретных уровней этих факторов (табл. 1). Число уровней должно соответствовать ожидаемой базовой модели (шаг 1.2). Это важно только для оптимальных конструкций, потому центральных композиционных всегда иметь дискретные уровни факторов.
- Связать ли категорическое фактором является номинальной, то есть никакой подразумеваемый порядок (например, различные производители), или порядковый и, таким образом дискретную числовой параметр (например, различные листья на заводе).
- Выберите уровни для обоих типов категориальных факторов.
- Выберите полезную базовую модель.
- На основании предварительных экспериментов и литературы, предвидеть связь между каждого фактора и реакции, а также фактор взаимодействия и реакции. Например, линейный (чем больше, тем лучше), квадратичной (один оптимальный или нелинейное увеличение / уменьшение) или кубический (перекос оптимальный).
- Убедитесь, что количество уровней для дискретных числовых факторов п + 1, с п будучи многочлен степень связи между фактором и ответ. Например, если температура, как ожидается, имеют квадратичную действие на ответ, п = 2 и, следовательно, по крайней мере, три уровня температуры должны быть исследованы, чтобы достичь подгонку к квадратичного эффекта.
- Определить долю пространства дизайна (FDS), для которых дисперсия предсказание должно быть ниже определенного порога (альфа уровень). FDS должна быть> 0,95 (охватывают более 95% от дизайна помещения) для достижения модели, которые дают надежные прогнозы на протяжениивесь дизайн помещения 21-23.
Примечание: порог 0,05 соответствует 5% уровне значимости (тип я ошибка) и является типичным значение. Увеличение этого значения приведет к сокращению числа экспериментов, необходимых для стратегии DoE но одновременно повысит вероятность того, что значительные последствия будут пропустили и что оценки их влияния на ответ будет неправильным.

Таблица 1. Факторы, влияющие на временную экспрессию белка в табака, включая вариации колеблется во Доу. Факторы выделены жирным шрифтом были только включены в дизайн для экспериментов, описанных в разделе "A описательной модели для накопления DsRed во временной экспрессии с использованием различных промотор / 5'UTRs", тогда как факторы, выделенные курсивом были включены только в дизайне для "Оптимизация incubatioN условия и схемы урожай для производства моноклональных антител в растениях с использованием временной экспрессии ".
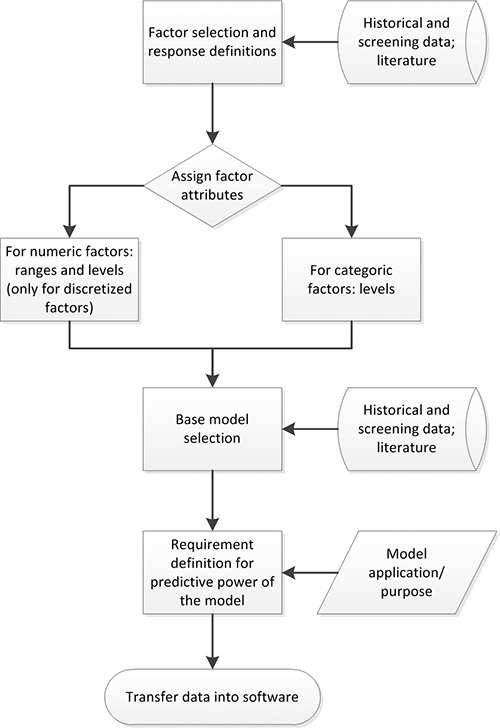
Рисунок 2. Министерство энергетики процесс планирования. Факторы со значительным воздействием на ответ исследуемого выбираются на основе имеющихся данных. Тогда атрибуты фактор (например, цифровые), диапазоны и уровни присваиваются. Предыдущий знания и эксперименты используются для определения подходящего базовой модели. Требования предсказательной силой определяются на основе применения / целью окончательной модели. Собранные данные затем могут быть переданы в соответствующее программное обеспечение Доу.
2. Настройка RSM в DesignExpert
- Начните DesignExpert и выберите пункт "New Design". В окне "поверхности отклика", выберите &# 34; Оптимальное "и введите количество числовых и категориальных факторов, выбранных в разделе 1.1).
- Введите имена фактор, блоки, типы, подтипы, количество уровней / границ уровня и значения для уровней для всех факторов в соответствующих полях.
- Продолжить на следующую страницу и под "Поиск" Варианты выбора "Бест", а также желаемый "Об оптимальности" критерий, здесь "D-оптимальный".
- В меню "Правка модель ...", выбрать модель, которая содержит факторы и взаимодействия ожидаемые в разделе 1.2). Если вы не уверены, выберите полную модель, которая охватывает все факторы и взаимодействия для определенной степени полинома, здесь "Квадратичные". Обратите внимание, что выбирая полную модель (содержащий все взаимодействия) может увеличить количество экспериментов, необходимых для DoE.
- Выберите число "блоков". Здесь, один блок был использован, так как все растения из той же партии, все бактерии культивировали одновременно иВсе инъекции были проведены в тот же день одним и тем же оператором. Используйте более одного блока, если более одного партия растений используется или несколько операторов обработки инъекции.
- Баланс дизайн
- Включить "Force категорический баланс" для того, чтобы распределить работает эксперимента поровну между категориальных уровней факторов, например, различные промоутеры, которые проверяются.
Примечание: это может уменьшить оптимальность проектирования до такой степени, что DesignExpert сообщит как% значения в зависимости от точек, выбранных по алгоритму оптимальности. - Пересчитать дизайн несколько раз с помощью реализован случайный алгоритм семян, чтобы минимизировать снижение оптимальности.
Примечание: потери в оптимальности может быть в диапазоне от ~ 3-40% с использованием тех же входных данных, но изменение количества повторов может быть использован для поддержания баланса категориальный.
- Включить "Force категорический баланс" для того, чтобы распределить работает эксперимента поровну между категориальных уровней факторов, например, различные промоутеры, которые проверяются.
- Программное обеспечение будет предложить значение для числа «точек типовых" на основе базовой модели, Здесь 70 трасс. Отрегулируйте количество показов для "повторах" и "Для оценки отсутствие подгонки", чтобы убедиться, что он> 5% из числа «точек типовых" для каждого, здесь 10 трасс каждый.
- Перейдите к следующей странице выберите количество ответов и введите их имена и единиц. Дополнительные ответы также могут быть добавлены в любое время при оценке данных без негативного воздействия на конструкции.
- Продолжить, чтобы начать алгоритм, расчета уровней фактором для прогонов DoE. Если выбранный базовая модель не соответствует количество уровней в течение определенного фактора, выводится уведомление и расчет не начнется. В этом случае либо:
- Увеличение количества уровней для этого фактора, или
- Снимите условия в базовой модели, что не может быть рассчитана исходя из текущего количества уровней.
- Например, если есть два уровня для фактора «Время инкубации" т (т.е. 2 г и 5 г) в тон в настоящее время конструкция и квадратичной базовая модель в том числе термина т 2 был выбран, либо добавить третий уровень для т (т.е. 8 г) или удалите фактор T 2 из модели.
- Сохраните таблицу, содержащую оптимальные сочетания факторов, которые отображаются сразу завершения расчета.
- В "Дизайн" узла выберите "оценка" суб-узел и перейдите на вкладку "Графики".
- В "инструмент Графики" выбрать "FDS" и в поле "FDS Graph" выберите "Pred" в качестве типа ошибки. Затем введите минимального обнаруживаемого разницу, а также приближенное значение для предполагаемого стандартного отклонения системы, определенной в разделе 1.1.1) в качестве значений "г" и "е" соответственно (здесь 20 и 8 мкг / мл для DsRed). Также введите уровень альфа (приемлемым% отсутствует существенное влияние), подходящую для применения, как правило, 0,05 (5%).
- Убедитесь в том, что расчетная FDS матчей процент, определенный в разделе 1.3) (обычно> 0,95 для прогнозных моделей). Плоская кривая на графике FDS предпочтительнее, указывая равномерное точность предсказания по всему дизайна помещения.
- Если это не так (как найти здесь, FDS был 1%, как показано на рисунке 3А), вернуться к «Дизайн» узла и на панели задач, выберите "Средства разработки", затем "Augment дизайн ..." и выберите " Дополните ".
- Выберите те же "Поиск" и "Об оптимальности" критерии, как раньше. Также выбрал тот же "Изменить модель" и "форс-категориальный баланс" Настройки. Если дизайн увеличение выполняется до любого эксперимента (как это было сделано здесь), а затем изменить "Положите работает в блок" в положение "Блок 1".
- В разделе "прогонов" введите количество дополнительных «точек модели", сохраняя, по крайней мере 5% "повторах" и "Для оценки отсутствиеиз вписаться "в общем дизайне. Здесь добавлены 100 дополнительных Опыты и никаких" Реплицирует "и" Для оценки отсутствие подгонки "не были включены.
- После расчета завершена, пересмотреть график FDS, как описано выше. Если FDS все еще неудовлетворительное, повторите дизайн увеличение как описано выше. Здесь FDS был на 100% после увеличения и таким образом не далее увеличение не требовалось (рис. 3В).

Рисунок 3. Сравнение FDS участков. . Министерство энергетики, состоящий из 90 серий производит недостаточное FDS всего в 1% для стандартной ошибки прогноза, используя квадратное базовой модели в сочетании со значениями для минимального обнаруживаемого разницы (20 мкг / мл) и оцеповязана стандартное отклонение системы (8 мкг / мл). B. Увеличение НОО в общей сложности 210 трасс достигается 100% FDS и плоскую кривую с указанием равномерное точность модели во всем дизайн помещения.
3. Клонирование и анализ экспрессии Кассеты
- Выращивают кишечной палочки в 5 мл LB среды (10 г / л триптона, 5 г / л дрожжевого экстракта, 170 мМ NaCl, 50 мг / л ампициллина; для LB-агаром включают 15 г / л агара) при 37 ° С в течение 6 -8 час или на ночь на орбитальном шейкере при 160 оборотах в минуту. ВНИМАНИЕ: ампициллин является вредным веществом. Привить среду LB с или 50 мкл жидкой культуры или передаче клеток из колоний, выросших на чашках с использованием пипетки.
- Для очистки плазмидной ДНК ПСО и других производных PPAM (GenBank AY027531), из Е. Штамм E.coli К12 DH5a, следуйте инструкциям в комплекте руководства очистки ДНК 24. ВНИМАНИЕ: очищение кон содержит вредных химических веществ (см. выше пособия для деталей). Последовательность плазмиду описано на фиг.4 и Покупатель и соавт. 20.
- Определить концентрацию ДНК в очищенном элюата путем измерения оптической плотности образца 2 мкл при 260 нм в NanoDrop устройства.
- Подтверждение личности очищенной ДНК плазмиды расщеплением рестриктаз (ВИЭ).
- Выберите УЭ в соответствии с последовательностью плазмиды, так что уникальные и различимые узоры фрагмент генерируются. Следуйте рекомендациям производителя по условиям пищеварения, таких как реакционного объема, времени, температуры и концентрации стабилизатора бычьего сывороточного альбумина. В зависимости от чувствительности устройство анализа, используют 50-500 нг плазмидной ДНК в пищеварении.
- Подготовка 0,8-2,0% гели для разделения фрагментов ДНК в агарозном кипячением в Трис-борат-ЭДТА (КЭ) буфера (90 мМ Трис, 90 мМ бората, 2 мМ ЭДТА, рН 8,0). Тон больше ожидаемых фрагментов, тем меньше агарозы должны быть использованы в геле.
- Добавьте 5 мкл пять-кратного буфера для образцов (5x SABU, 0,1% (вес / объем) бромфеноловый синий, 0.1% (вес / объем) ксиленцианол, 10% (вес / объем) глицерина в КЭ) в 50 мкл RE-обработанный образец ДНК и отделить фрагменты электрофорезом в агарозном геле при 100 В в течение ~ 40 мин или до четкого разделения фрагментов достигается. На каждый гель, включают в себя полосу, содержащую размер ДНК лестница маркеры для сравнения, например, 2-3 мкл 1-кб лестнице.
- Замените омега 5'UTR в PSO с одним из трех других 5'UTRs.
- Отпустите омега 5'UTR последовательность из ~ 4 мкг очищенной ДНК PSO обработкой 20 единиц Eco RI-HF и сержантского Я-HF ВИЭ в NEBuffer 4 при 37 ° С в течение ~ 60 мин. Тогда отделить фрагменты, как описано в разделах 3.4.2 и 3.4.3.
- Изолировать больший фрагмент (PS, "становым хребтом") из агарозного геля с помощью гель дополнительныйводств Набор по изготовитель Руководство 25 и определения концентрации очищенной ДНК, как описано в разделе 3.3). ВНИМАНИЕ: Комплект для слива гель содержит вредных химических веществ, см. в руководстве по эксплуатации.
- Изолировать CHS, CHS-ЛПХ и TL 5'UTRs из подходящих векторов доноров обработкой ~ 10 мкг каждого вектора с 20 единиц Eco RI-HF и Nco Я-HF ВИЭ в NEBuffer 4 при 37 ° С в течение ~ 60 мин. Тогда отделить фрагменты, как описано в разделах 3.4.2 и 3.4.3 и очистить меньшую 5'UTR-содержащий фрагмент, как описано в разделе 3.5.2.
- Лигируют изолированные очищенные 5'UTRs в отдельных аликвотах в изолированной очищенной линейная пСм вектора при 25 ° С в течение 5 мин в соответствии с рекомендациями 26 изготовителя. Использование ~ 50 нг векторной ДНК и три-кратный молярный избыток 5'UTR ДНК в общем объеме 20 мкл.
- Трансформация E палочка клетки с гое рекомбинантные плазмиды 26,27.
- Добавить ~ 10 нг (~ 4-5 мкл) лигирования смеси (см. раздел 3.5.4) до 50 мкл RbCl компетентным Е. палочка и аккуратно перемешать, а затем инкубировать в течение 30-60 мин на льду. Тепловой удар в течение 1,5 мин при 42 ° С и охладить на льду в течение 5-30 мин.
- Добавить 950 мкл антибиотической свободной LB среды и инкубируют в течение 1 ч при 37 ° С и 160 оборотах в минуту. Для отбора трансформантов, распространение 50 и 100 мкл культуры на чашках с агаром LB, содержащих ампициллин и инкубировать в течение ~ 16-20 часов при 37 ° С.
- Привить 5-10 отдельных колоний, представляющих каждый промоутер-5'UTR перевязки, описанный в разделе 3.1 в 5-мл аликвотами LB среде, содержащей ампициллин. Тогда очистить ДНК плазмиды и его идентификации, как описано в разделах 3.2 до 3,4.
- Замените 35SS промотор с промоутером н.у.к в каждой из четырех 5'UTR плазмид использованием ВИЭ возрастанию I и Eco RI в NEBuffER 4 при 37 ° С в течение 1 ч, как описано в разделах 3.5 и 3.6.
- Представьте каждой из восьми полученных плазмид в А. tumefaciens штамм GV3101: pMP90RK путем электропорации 28.
- Добавить ~ 500 нг очищенной плазмидной ДНК в 50 мкл компетентного А. tumefaciens клетки на льду. Аккуратно перемешайте и передать в prechilled 0,2 см кювету для электропорации. Убедитесь, что смесь на дне кюветы и не содержит пузырей.
- Импульсные клетки на 2,5 кВ в течение 5 мс и подтверждающие интенсивность и продолжительность импульса.
- Не допускать повышения концентрации соли в образце ДНК или неправильной подготовки компетентного А. tumefaciens клетки, поскольку это может привести к высоких ионных токов, вызывающих мгновенное испарение клеточной суспензии, что значительно снижает эффективность трансформации.
- Добавить 950 мкл YEB среде без антибиотиков (выдержка 5 г / л говядина, 1 г / л дрожжевого экстракта, 5 г / л пептоне, 5 г / л сахарозы, 2 мМ MgSO 4, рН 7,0), осторожно перемешать и передавать непосредственно в стерильный 1,5 мл реакционной трубки. Инкубировать в течение 2-4 ч при 26-28 ° С и 160 оборотах в минуту.
- Распространение 1-2 мкл на YEB агаровых чашках, содержащих антибиотики (50 мг / л карбенициллина, 25 мг / л канамицина, 25 мг / л рифампицина) для отбора трансформантов. ВНИМАНИЕ: рифампицин является токсичным веществом.
- Инокулировать три 5 мл аликвоты YEB среде, содержащей антибиотики с клетками из отдельных колоний, представляющих каждый промотор-5'-UTR лигирование и инкубировать в течение 48-72 ч при 26-28 ° С и 160 оборотах в минуту.
- Подтвердите успех А. tumefaciens трансформации.
- Передача 2 мкл аликвоты каждого из раздела 3.8.6 отделить 48 мкл аликвоты ПЦР Mastermix (2 мкл каждого из 10 мкМ праймера складе (400 нМ конечная концентрация каждого праймера),1 мкл 10 мМ смеси дНТФ (200 мкМ конечную концентрацию каждого дезоксинуклеозид-трифосфата), 5 мкл 10-кратного Expand High Fidelity буфера с 15 мМ MgCl 2, 0,75 мкл Expand High Fidelity смеси ферментов, и 39,25 мкл стерильной дистиллированной воды).
- Amplify кассете экспрессии каждой плазмиды с использованием соответствующих праймеров (Fwd: 5'-CCT CAG GAA GAG CAA TAC-3 ', связывающий 1026 нуклеотидов в обратном направлении от промотора; REV: 5'-AAG CCA CGA GTA CAC AAC-3', связывание в полиаденилирования сайте 35S) при соответствующих условиях ПЦР. Здесь, 94 ° C была использована для первоначальной денатурации с последующими 30 циклами 94 ° С денатурации в течение 15 сек, 51 ° C отжиг в течение 30 сек и 72 ° С для удлинения 120 сек, а конечная стадия удлинения при 72 ° С в течение 8 минимум
- Определите размер продуктов ПЦР с помощью электрофореза в агарозном геле, как описано в разделе 3.4.3.
- Подготовьте А. tumefaciens запасы глицерина посмешивание 500 мкл 50% (об / об) стерильный глицерин с 500 мкл А. tumefaciens культур из раздела 3.8.6, для которых преобразование было подтверждено (раздел 3.9.3). не хранить запасы глицерине при -80 ° С до дальнейшего использования.
- Вычислить энергию складные различных мРНК с использованием RNAfold веб-сервер 29 и включают в себя нуклеотидную последовательность от сайтом начала транскрипции до первого 50 п.н. кодирующей области.
- В разделе "Fold алгоритмы и основные параметры" выберите "минимум свободной энергии (МКЭ) и функцию секционирования" и "избежать изолированных пар оснований" варианты.
- В разделе "Дополнительные параметры складные" выберите "болтались энергии с обеих сторон спирали в любом случае", "параметры РНК (Тернер модели, 2004)" и изменить "масштабировать энергетических параметров для данной температуре (C)" до 25 ° С.
- В разделе "Опции вывода" выбрать все варианты. Из "Результаты для термодинамического ансамблевого прогнозирования" части выходного файла, извлекать "свободной энергии термодинамической ансамбля» и «частоты конструкции MFE в ансамбле" ценностей для сравнения.
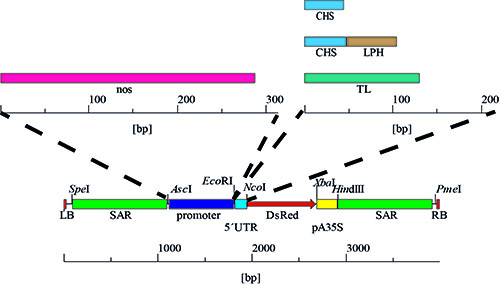
Рисунок 4. Промотором и 5 'UTR вариантов. Кассеты экспрессии были получены путем ступенчатой обмена 5'-UTR, в результате чего четыре комбинации с промотором CaMV 35SS с последующим замещением этого промотора с последовательностью NOS получают четыре дополнительные варианты и в общей сложности восемь различных комбинаций промотор / 5'UTR.
4. Растениеводство
- Подготовьте 0,1%-ный раствор удобрения Ferty2 Мега в деионизированной воде при рН 5,9.
- Подготовка 10 х 10 х 8 см Rockwool блоков обширной промывки деионизованной водой для удаления остаточных химических веществ и, наконец, равновесие с удобрением.
- Семя растения табака путем размещения 1-2 семена табака на каждого блока минеральной ваты последующим кратким заподлицо с удобрением будьте осторожны, избегайте мытья семена далеко.
- Прорастают и культивировать растения табака в течение 42 дней в теплице при 25/22 ° C температура днем / ночью, относительной влажности 70% и 16-часовой фотопериода (180 ммоль с -1 м -2; λ = 400-700 нм) . В течение этого фотопериода, орошения с удобрением в течение 15 мин каждый час в гидропоники культуры.
5. Переходные Экспрессия белка
- Подготовьте А. tumefaciens для инъекций в листьях.
- Инокулировать 5-50 мл YEB среде, содержащей антибиотики с 1% А. tumefaciens крио-исходной культуры и инкубировать при 27 ° С, пока ОП <суб> 600 нм достигает 5,0 (~ 48-72 ч в зависимости от объема и типа судна).
- Развести А. tumefaciens культура водой и 2 раза инфильтрации среды (4,3 г / л Мурасиге и Скуга солей (рН 5,6), 5 г / л сахарозы, 1,8 г / л глюкозы, 100 мМ ацетосирингона) в соответствии с ОП 600 нм, необходимое для инъекций. Подтвердите OD 600 нм непосредственно перед инъекцией. Примечание: OD 600 нм 1.0 соответствует ~ 1,43 ± 0,12 х 10 9 колониеобразующих единиц на мл.
- Введите А. tumefaciens суспензии в листьях.
- Выберите и маркировать noncotyledon листья и позиции по этому вопросу рассматриваться (например, в соответствии со стратегией DOE).
- Не вводят различные A. tumefaciens решения в том же межреберной области. Вместо этого используйте противоположные разделы по каждой стороне среднего вены оси. Если более чем два различных решения должны быть введены в том же положении использовать additionaл завод.
- Тщательно встряхните А. tumefaciens решение ресуспендируйте любые клетки, которые, возможно, собралась с подготовки разбавление и аспирации в 1-мл шприц.
- Аккуратно поцарапать эпидермис на место, предназначенное для инъекций с кончика пипетки или подобного содействия приток в А. tumefaciens решение. Избегайте разрыва листовой пластинки, делая так.
- Удерживая шприц перпендикулярно к листовой пластинки касаясь барр к межреберной области, подлежащей обработке, и осторожно нажмите на выход (без иглой) на нижней стороне листьев. Нажмите верхнюю сторону листа осторожно в то же время предотвратить листовой пластинки от сдвига или разрыва.
- Осторожно надавите на поршень шприца. А. Раствор tumefaciens войдет в межклеточные пространства внутри листовой пластинки, как показано на обработанных участках, возникающих темно-зеленый и влажной. Повторяйте эту процедуру в нескольких местах, пока вся intercostaл поле проникли с А. tumefaciens. Тогда переходите к следующему межреберной области.
- Убедитесь, что шприц остается перпендикулярно к листу. Наклон шприц может привести к бактериальной суспензии на бить струей под высоким давлением.
- Если разные А. tumefaciens используют растворы (например, для тестирования различных промоторов), удалить любой избыток раствора, остающегося на нижней стороне листа от первой инъекции с использованием бумажное полотенце или подобный перед нанесением следующего раствора.
- После инфильтрации инкубации растений и отбора проб.
- Подготовьте фитотроне для обработанных растений ~ 24 часа в сутки перед инъекцией, чтобы позволить температуры и влажности уравновешивание на уровнях, необходимых для DoE.
- После инъекции, передавать растения в фитотроне и разместить их в лотки, достаточно больших для орошения водой до конца инкубационного периода, определяемой Министерством энергетики.
- Создание 16-часовом фотoperiod с использованием шести Osram холодные белые 36 Вт Люминесцентные лампы на 0,7 м 2 (75 ммоль с -1 м -2; λ = 400-700 нм). Предотвратить растения от затенения друг друга, ограничивая количество растений до шести за 0,7 м 2.
- Перед выборки, убедитесь, что правильно межреберная поле была выбрана путем сравнения этикетку добавленную в п. 5.2.1 и план Доу.
- Используйте пробки буром для удаления 4-5 дисков листьев из обработанных межреберных полей на должности и времени, указанные в DoE. Не снимайте весь лист с завода во время отбора проб. Стабилизировать лист с ручным бумажным полотенцем, удаляя диски, чтобы предотвратить разрыв.
- Определяют массу каждого образца и поместить его в 1,5 мл пластиковую пробирку реакции с именем образца и массы. Хранить образцы при -20 ° С или -80 ° С до белка количественного определения. Этот процесс может быть приостановлен на этой стадии в течение нескольких месяцев в зависимости от стабильности образцаи температура хранения.
6. Белок Количественное
- Извлечение белков из образцов листовых дисков.
- Добавить 3 мл экстракционного буфера (50 мМ фосфата натрия, 500 мМ хлорида натрия, рН 8,0) на мг массы образца и измельчить дисков листьев в реакционной трубке с помощью электрического пестиком до тех пор, никаких больших фрагментов остаются. Избегайте перегрева образца.
- Удалить диспергированных твердых частиц центрифугированием образца в два раза при 16000 х г в течение 20 мин при 4 ° С. Передача супернатант в чистую 1,5 мл реакционной трубки после каждого шага, не нарушая гранул.
- После центрифугирования, процесс может быть приостановлена путем замораживания растительных экстрактов при температуре -20 ° С или -80 ° С в течение нескольких месяцев в зависимости от стабильности образца и температуры хранения. Убедитесь, что цикл при замораживании-оттаивании не влияет на концентрацию белка-мишени (ей).
- Измерьте флуоресценции DsRed.
- Подготовьте три технических повторов каждого образца в черные 96-луночные планшеты половине площади (50 мкл экстракта на лунку). Избегайте образования пузырьков во время пипетки.
- Использование набор из шести разведений (0, 25, 75, 125, 175 и 225 мкг / мл) стандартного DsRed в 96-луночный планшет для генерации эталонной гильзы кривой. Подготовка разведений в PBS и хранить их при 4 ° С для использования в течение 3 месяцев.
- Измерьте флуоресценции дважды, последовательно, в ридере 96-а, снабженной возбуждения 530/25 нм и 590/35 фильтров выбросов нм.
- Для каждого образца, в среднем флуоресценцию над ними гласит и три технических повторов и вычтите значение записанного для пустой управления, содержащий 0 мкг / мл DsRed. Кроме того вычесть эту величину из считывает из стандартных разведений и использовать эти пустые коррекцией значения для линейной регрессии получая калибровочную кривую (через начало координат ординат).
- Использование наклон эталонной кривой для преобразования Fluorescence, измеренна в образцах в DsRed концентрациях. При необходимости разбавить образцы так, что значение находится в интервале концентраций стандартов и учитывать этот фактор разбавления в последующих расчетах.
- Определите концентрацию 2G12.
- Подготовка поверхностного плазмонного резонанса (SPR) устройство для измерения концентрации антител путем связывания белок А к активированной поверхности одной клетки потока. Используйте другую ячейку потока в качестве эталона путем инактивации поверхность без сцепления белка 30,31.
- Развести растительные экстракты 1:20 в SPR рабочем буфере (10 мМ HEPES рН 7,4, 3 мМ ЭДТА, 150 мМ NaCl, 0,05% об / об Tween-20) и измерить единиц отклика (RU) связывания антитела с белком А в три технические повтора каждого образца в конце инъекции 90 мкл (180 сек при 30 мкл / мин). Вычтите RU оцениваются по клетке ссылки потока (без белка A) от РУ измеренного в экспериментальной этой ячейки (Белок поверхность).
- Измерение стандарт 585 нг / мл через каждые 2G12 10-15 образцов и вычесть величину, измеренную в клетках ссылки потока, как описано выше. Использование среднюю RU этих стандартов для вычисления линейной калибровочной кривой через начало координат ординат.
- Рассчитать концентрацию 2G12 в образцах на основе разведения 1:20 и наклон эталонной кривой.
- Проверьте сильный неспецифическое связывание с опорной ячейки, которые могут привести к повреждению измерение. Также проверьте стандарты 2G12 сохранить приблизительно постоянные значения (<5% вариации) на протяжении анализа, как более сильному изменению отражает старение белка поверхности.
7. Анализ и оценка данных
- Вручную анализировать ответы, наблюдаемые в экспериментах экспрессии для (I) экстремальных значений (высоких или низких), указывающий на ошибки измерения; (II) необычных результатов, например комбинаций факторов, которые генныхоценить неожиданную реакцию, указывающие обмена данными, и (III) отсутствующих значений.
Примечание: использовать эти меры для предотвращения в построение моделей на основе ошибочных данных. - Передача данных анализируемые реагирования (здесь Концентрации белка) в "Дизайн" узла DesignExpert. Убедитесь, что ответные данные правильно назначены для соответствующих установок факторов. Существует обширный раздел помощь доступна в DesignExpert программного обеспечения, которая охватывает аспекты рассматриваются ниже.
- В «Анализ» узла, выберите ответ анализируемый и первоначально выберите "нет" на вкладке трансформации.
Примечание: преобразование рекомендуется для мин / макс соотношениях ответа больше, чем 10. Самое полезное преобразование может быть получено из Box-Cox-участка на вкладке "Диагностика" в разделе "Диагностика" в "инструмент Диагностика", описанной ниже (раздел 7.8). Вход 10 преобразование часто необходимо. Перейдите к закладке «Fit резюме", которая обеспечивает общую информацию о факторах, которые важны для исследуемой системы (например взаимодействий двухфакторных, квадратичных эффектов). Программное обеспечение будет предложить начальную модель, основанную на его значении. - На вкладке "типового", начальная модель выбирается заранее на основе «Установить резюме" результатов. Используйте автоматизированном режиме редактировать эту модель:
- Выберите "порядок процесс", который является одним заказа выше предложенной модели, например, если предложенная модель является "2FI" (два фактором взаимодействия) выберите "квадратичной".
- Выберите «назад» в поле «Выбор», которая будет многократно удалить незначительное термины из модели после перейти к вкладке "ANOVA» на основе значения «Альфа из», который изначально должен быть 0,100.
- На вопрос, с помощью программного обеспечения, всегда автоматически корректировать модель иерархии, потому что это necessarу генерировать надежные модели 32,33.
- На вкладке "ANOVA", исследовать предложенную модель и включенные факторы. При необходимости вручную удалить любые факторы с р-значениями выше установленного порога (здесь 0,05, что соответствует 5% уровне значимости) или те, которые вряд ли на основе механистического рассмотрения перейти обратно на вкладке "Модель", изменяя "Выбор" "Ручной" и устранения соответствующих коэффициентов из модели.
- Вернуться во вкладку "ANOVA", оценить обновленный модели в терминах р-значения "Модели" (низкое значение является желательным, поскольку это свидетельствует о значимости) и "Отсутствие подгонки" (высокое значение желательно потому что это указывает на отсутствие значения), а также значения для "R-квадрат", "Скорректированный R-квадрат" и "Прогнозируемая R-квадрат" (значения> 0,80 желательны для всех трех значений).
- Сравните эти значения FOг различных моделей, включая / исключая факторов на нескольких уровнях значимости.
Примечание: это может быть полезно, чтобы дублировать столбец реагирования в «Дизайн» узла и выполнять каждый анализ индивидуально или экспортировать всю таблицу "ANOVA" в другую программу, например, электронную таблицу.
- Сравните эти значения FOг различных моделей, включая / исключая факторов на нескольких уровнях значимости.
- Перейдите к закладке "Диагностика", чтобы подтвердить качество модели и выявления потенциальных выбросов в наборе данных, которые имеют сильное влияние на модели путем изучения все вкладки в «Диагностика Tool" ("Диагностика" и раздел "Влияние") .
- В разделе "Диагностика" в "инструмент Диагностика", выполните следующие действия:
- Убедитесь, что точки беспорядочно в пределах в "Разности и предсказанных" диаграммы. Здесь шеврона образное распределение указывает на необходимость преобразования данных.
- Обеспечить точки беспорядочно внутри границ"Остатки против Прогнозируемая" диаграммы. Здесь шеврона образное распределение указывает на необходимость преобразования данных.
- Исследовать ли точки в "Остатки против перспективе" схемы разброса случайно в пределах. Образец (например, "лестница") указывает на тенденцию в здании данных и блок может быть использован, чтобы противодействовать его влияние на модели.
- Ищите прямой диагональной линии, указывающей идеальную модель в "Прогнозируемая и фактическое" заговора. Чем больше рассеяние данных вокруг диагонали, тем меньше точность модели.
- Проверьте перекрытием на ровном месте (лучший) и зеленый (фактические) вертикальные линии в Box-Cox-участка с указанием преобразование данных. Если зеленая линия не соответствует синюю (вне указанном промежутке красными вертикальными линиями) вернуться к разделу 7.3 и улучшить модели здания круглый выбрав преобразование данных, предложенную Box-Cox-участка. Опять дублирование реколонка Отклик может быть полезно для сравнения различных моделей.
- Убедитесь в том, что точки в «Остатки по сравнению фактор" диаграмм (есть один график для каждого фактора модели) рассеивают случайно в пределах. Кривизна в распределении данных может указывать на коэффициент отсутствующий в модели.
- В разделе "Влияние" из "инструмента диагностики", убедитесь, что есть случайные рассеяние данных в рамках в "Внешне стьюдентизированной остатков", "плечо", "DFFITS" и "DFBETAS" участки и равномерно мало распространен без экстремальные значения в "Кука Расстояние" участка.
- В разделе "Диагностика" в "инструмент Диагностика", выполните следующие действия:
- На вкладке "Модель графиков", визуализировать оценочную модель. Для ограниченного числа числовых факторов (например, 3) поверхность отклика ("3D поверхность") представление полезно для оценки Optima / характеристики вручную.
Примечание: поверхности реагирования только проиллюстрировать влияние два факТЗ на ответ под следствием. Эффект дополнительного фактора на ответ раскрывается путем изменения его значения (числовые факторы) или уровень (категориальные факторы) в окне «Факторы инструмента". Кроме того, факторы могут быть отнесены к оси участка щелкнув правой кнопкой мыши их в окне "Факторы инструмента" и выбрав нужный независимой переменной ось.- Манипулировать уровни факторов и назначить их на графике ординат, используя "Факторы функции".
- Экспорт графики с помощью "Экспорт график в файл ..." команду на вкладке "Файл".
- Используйте "Численное" суб-узел в "оптимизации" узла Для оптимизации чувствительности численно (свернуть, развернуть, в диапазоне) в зависимости от модели факторов, к которым, в свою очередь определенные ограничения могут быть применены (например, ограничения и вес) через Вкладка "Критерии".
- Рассчитать и изучить численные решения в "Solutions"Вкладка на основе материалов, представленных на вкладке «критерии».
- Экспорт этих решений с другим программным обеспечением (например, электронные таблицы) для дальнейшего анализа (например, гистограмм) выявление установки факторов, связанных с высокой или низкой значений отклика.
Примечание: это полезно, если более трех числовые факторы исследуются и 3D представление трудно.
- Используйте "Точка предсказания" суб-узел, чтобы предсказать ответ для конкретных установок факторов.
- Используйте этот прогноз для всех настроек должны быть оценены (здесь, все промотор и 5'UTR комбинации, а также все листья и времени инкубации или листьев позиции и температуру инкубации).
- Экспорт предсказанных значений и собрать их в массиве данных, которые могут использоваться для описания временной экспрессии в растениях табака, например, путем вычисления выражения для конкретной комбинации промотор-5'UTR в определенное время, усредненное по всем позиции лист или ACROсс все листья растения.
- Нормализовать данные экспрессии на основе масс различных листьев, получая определенный уровень экспрессии (мкг г -1) или удельную скорость экспрессии (мкг г -1 ч -1).
- С другой стороны, экспорт коэффициенты "Финал уравнения" из нижней части вкладки "ANOVA" в электронную таблицу и умножить их с массивом установок факторов, получая тот же массив данных.
Примечание: этот массив может содержать только значения факторов изнутри начальной дизайна помещения, так как оборудована модель уравнение не подходит для экстраполяции.
- Провести подтверждение эксперимент для модели точек, которые представляют большой интерес.
- Выполните выражение переходный белка в условиях, выбранных для "предсказания Пойнт" (раздел 7.11). Например использовать одинаковые температуры и листья и т.д.
- Определить концентрации белка для этого переходного выражения еXperiment, как описано выше (раздел 6) и сравнить их с значений, предсказываемых моделью.
- Проверьте, попадает ли средняя концентрация белка в эксперименте подтверждения в модели поверхности отклика в интервале прогнозирования, полученной во время предсказания точки. Матч подтверждает предсказательной силой модели. Несоответствие указывает, может потребоваться низкое качество модели и дополнительные трассы.
- Примечание: завод партии к партии изменчивость расширяет интервал прогнозирования и может девальвировать подтверждения экспериментов, выполненных с другой партией растений. Однако образцы, которые не были использованы для создания модели, но которые возникли из той же партии растений как образцы содержали в модели может служить в качестве эффективного контроля.
Результаты
Описательная модель для накопления DsRed во временной экспрессии с использованием различных промоутеров и 5'UTRs
DsRed флуоресценции в экстрактах листьев используется для указания уровня экспрессии рекомбинантного белка и, таким образом, был использован в качест?...
Обсуждение
Каждый эксперимент требует тщательного планирования, поскольку ресурсы часто дефицитным и дорогим. Это особенно верно для стратегий Доу, так как ошибки на этапе планирования (например, выбора базовой модели, которая не покрывает все значимые взаимодействия факторов) может сущест...
Раскрытие информации
Пошлина за публикацию частично спонсируется компаниями Statease, Inc (США) и Statcon (Германия), которые не были вовлечены в участие в подготовке рукописи или ответственность за его содержание.
Благодарности
Авторы благодарны д-р Томас Радемахер для обеспечения растений вектор экспрессии PPAM и Ибрахима аль Amedi для выращивания табачные растения, используемые в данном исследовании. Мы хотели бы поблагодарить д-ра Ричарда М. Twyman за его помощь в редактировании рукописи. Эта работа была частично финансируется Европейским исследовательским советом Расширенный Грант "Future-Фарма", число предложений 269110 и Фраунгофера Zukunftsstiftung (Fraunhofer Будущее Фонд).
Материалы
| Name | Company | Catalog Number | Comments |
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life Technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10 x10 cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life Technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
Ссылки
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ - The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5'UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены