Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Caractérisation des systèmes complexes à l'aide de la conception des expériences approche: l'expression transitoire de protéines dans le tabac comme une étude de cas
Dans cet article
Résumé
Nous décrivons une conception de l'approche des expériences qui peuvent être utilisés pour déterminer et modéliser l'influence des éléments de régulation du transgène, les paramètres de croissance et de développement des plantes, et des conditions d'incubation sur l'expression transitoire des anticorps monoclonaux et des protéines rapporteurs dans les plantes.
Résumé
Les plantes apportent de multiples avantages pour la production de produits biopharmaceutiques, y compris de faibles coûts, l'évolutivité et la sécurité. L'expression transitoire offre l'avantage supplémentaire de développement à court et temps de production, mais les niveaux d'expression peut varier considérablement entre les lots donnant ainsi naissance à des préoccupations d'ordre réglementaire dans le cadre de bonnes pratiques de fabrication. Nous avons utilisé un plan d'expériences (DoE) approche pour déterminer l'impact des principaux facteurs tels que des éléments de régulation dans la construction d'expression, la croissance des plantes et les paramètres de développement, et les conditions d'incubation pendant expression, sur la variabilité d'expression entre les lots. Nous avons testé des plantes exprimant un anticorps anti-VIH modèle monoclonal (2G12) et une protéine marqueur fluorescent (DsRed). Nous discutons de la justification du choix des propriétés du modèle et identifier ses limites éventuelles. L'approche générale peut facilement être transférée à d'autres problèmes parce que les principes du modèle dere largement applicable: le choix des paramètres de la connaissance, la réduction de la complexité en divisant le problème initial en petits modules, configuration logicielle guidée de combinaisons optimales d'expérimentation et par étapes de conception augmentation. Par conséquent, la méthode n'est pas seulement utile pour caractériser l'expression des protéines dans les plantes mais aussi pour l'étude d'autres systèmes complexes qui n'ont pas une description mécaniste. Les équations de prédiction décrivant l'interconnectivité entre les paramètres peuvent être utilisés pour établir des modèles mécanistes pour d'autres systèmes complexes.
Introduction
La production de protéines biopharmaceutiques dans les plantes est avantageux parce que les plantes sont peu coûteux à développer, la plate-forme peut être étendu simplement en cultivant plus de plantes, et les agents pathogènes humains sont incapables de se répliquer 1,2. Stratégies d'expression transitoire sur la base par exemple sur l'infiltration des feuilles avec Agrobacterium tumefaciens offre des avantages supplémentaires parce que le temps entre le point de livraison de l'ADN et la livraison d'un produit purifié est réduite de quelques années à moins de 2 mois 3. L'expression transitoire est également utilisée pour l'analyse fonctionnelle, par exemple pour tester des gènes pour leur capacité à compléter les mutants perte de fonction ou pour étudier les interactions entre protéines 6.4. Cependant, les niveaux d'expression transitoire ont tendance à montrer une plus grande variation de lot à lot que les niveaux d'expression dans des plantes transgéniques 9.7. Ceci réduit la probabilité que les processus de fabrication de produits biopharmaceutiques basés sur l'expression transitoire will être approuvé dans le cadre des bonnes pratiques de fabrication (BPF), parce que la reproductibilité est un attribut de qualité critique et est soumis à une évaluation des risques 10. Cette variation peut également masquer toutes les interactions que les chercheurs ont l'intention d'enquêter. Par conséquent, nous avons décidé d'identifier les principaux facteurs qui influent sur les niveaux d'expression transitoire dans les usines et de construire un modèle prédictif quantitatif de haute qualité.
Le (OFAT) approche à un facteur-à-un-temps est souvent utilisé pour caractériser l'impact (effet) de certains paramètres (facteurs) sur le résultat (réponse) d'une expérience 11. Mais ce n'est pas optimal parce que les tests individuels (courses) pendant une enquête (expérience) seront alignés comme des perles sur une ficelle à travers la zone potentiel engendré par les facteurs qui sont testés (espace de conception). La couverture de l'espace de conception et, partant, le degré de l'information dérivée de l'expérience estfaible, comme le montre la Figure 1A 12. En outre, les interdépendances entre les différents facteurs (interactions de facteurs) peuvent rester cachées dans les modèles résultant pauvres et / ou la prédiction de la fausse optima, comme le montre la Figure 1B 13.
Les inconvénients décrits ci-dessus peuvent être évités en utilisant une conception d'expériences (DoE) approche dans laquelle les pistes d'une expérience sont dispersées plus uniformément dans l'espace de conception, ce qui signifie que plus d'un facteur varie entre deux pistes 14. Il ya des dessins spécialisées pour les mélanges, les facteurs (dessins factoriels) et la quantification des impacts de facteurs sur les réponses (méthodes de surface de réponse, RSM s) 15 de dépistage. En outre, SMR peuvent être réalisées que des dessins centraux-composite mais peuvent également être atteints de manière efficace en utilisant un logiciel spécialisé qui peut appliquer des critères différents pour la sélection des pistes. Par exemple, ce qu'on appelle le D-OptimalITcritère d'y sélectionnera pistes afin de minimiser l'erreur dans les coefficients du modèle qui en résulte, tandis que le critère d'optimalité sélectionne IV-séries qui permettent d'atteindre le plus faible variance de prédiction dans tout l'espace de conception 15,16. Le RSM nous décrivons ici permet la quantification précise de l'expression de la protéine transitoire chez les plantes, mais il peut facilement être transférée à tout système impliquant plusieurs (~ 5-8) facteurs numériques (tels que la température, le temps, la concentration) et quelques-uns (~ 2 - 4) facteurs catégoriques (par exemple promoteur, couleur) dans lequel une description mécaniste n'est pas disponible ou trop complexes à modéliser.
L'approche DoE origine dans les sciences agricoles, mais s'est étendu à d'autres domaines, car il est transférable à toute situation où il est utile de réduire le nombre de cycles nécessaires pour obtenir des données fiables et générer des modèles descriptifs des processus complexes. Cela a conduit à l'inclusion de DoE dans les "Directives pourIndustrie, Q8 (R2) Développement pharmaceutique », publié par la Conférence internationale sur l'harmonisation des exigences techniques pour l'enregistrement des produits pharmaceutiques à usage humain (ICH) 17. DoE est maintenant largement utilisé dans la recherche scientifique et de l'industrie 18. Cependant, il faut prendre soin lors de la planification et l'exécution de l'expérience, car le choix d'un polynôme de degré mauvaise pour le modèle linéaire multiple régression (modèle de base) peuvent introduire une nécessité pour des courses supplémentaires pour modéliser correctement les effets des facteurs. Par ailleurs, corrompus ou des données manquantes génèrent des modèles erronés et défectueux prédictions, et peuvent même empêcher tout modèle tentative de construction comme décrit dans les sections de protocole et de discussion 18. Dans la section de protocole, nous allons d'abord définir les étapes de planification les plus importants pour une expérience à base de RSM et ensuite expliquer la conception basée sur le DoE logiciel DesignExpert v8.1. Mais les conceptions semblables peuvent être construites avec d'autres includi logicielng JMP, Modde, et STATISTICA. Les procédures expérimentales sont suivies par des instructions pour l'analyse et l'évaluation des données.
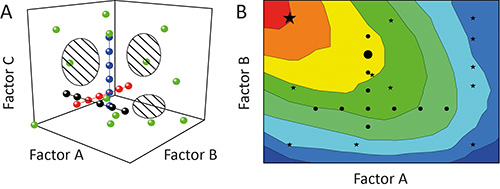
Figure 1. Comparaison des OFAT et DoE. A. Variation séquentielle d'un facteur à la fois (OFAT) dans une expérience (cercles noirs, rouges et bleues) réalise une faible couverture de l'espace de conception (des zones hachurées). En revanche, la variation de plus d'un facteur à la fois en utilisant la conception des expériences de stratégie (DoE) (cercles verts) améliore la couverture et donc la précision des modèles issus. B. La couverture de l'espace de conception biaisée signifie que les expériences OFAT (cercles noirs) peuvent aussi échouer à identifier les régions d'exploitation optimales (en rouge) et de prévoir des solutions sous-optimales (grand cercle noir), alors que DoE stratégiquementes (étoiles noires) sont plus susceptibles d'identifier les conditions préférables (grande étoile noire).
Protocole
Une. Planification d'une stratégie DoE
- Identifier les facteurs et les réponses pertinentes à inclure dans la conception.
- Définir une ou plusieurs réponses pour la mesure. Ici, les taux d'expression 2G12 et DsRed ont été utilisés (pg / ml), notamment la différence minimale détectable considéré comme (10 et 20 pg / ml, respectivement) concernés et une valeur approchée de l'écart type estimé du système (4 et 8 pg / ml, respectivement) sur la base des expériences précédentes.
- Utilisez la littérature disponible, les données provenant d'expériences antérieures ou conceptions spécialisées de sélection (par exemple, un plan factoriel, voir l'introduction) pour sélectionner des facteurs importants dont l'impact sur les réponses seront quantifiés (tableau 1) 7,8,19,20.
- Affecter les types de facteurs (numériques ou catégoriques) et sélectionner des plages dans lesquelles les facteurs numériques seront varient au cours de l'enquête DoE (tableau 1).
- Identifiez numeric facteurs pour lesquels une variation continue est difficile à mettre en œuvre.
- Évitez d'utiliser la variation continue des facteurs qui ne peuvent être précisément ajustée à un certain niveau. Par exemple la température d'incubation peut généralement être contrôlé uniquement à ± 2 ° C, variation donc continue en utilisant des valeurs telles que 27,2 ° C, 25,9 ° C, et 29,3 ° C doit être évitée.
- Au lieu de cela, sélectionner un certain nombre de niveaux discrets de ces facteurs (tableau 1). Le nombre de niveaux doit correspondre au modèle de base prévue (étape 1.2). C'est seulement important pour les plans optimaux, parce que les dessins composites centraux ont toujours des taux de facteur discrètes.
- Attribuer si un facteur est catégorique nominale, c'est à dire sans ordre implicite (par exemple, différents fabricants), ou ordinal et donc un paramètre numérique discrète (par exemple différentes feuilles sur une plante).
- Sélectionnez les niveaux pour les deux types de facteurs catégoriques.
- Sélectionnez un modèle de base utile.
- Sur la base des expériences et de la littérature préliminaires, d'anticiper la relation entre chaque facteur et la réponse ainsi que les interactions de facteurs et de la réponse. Par exemple, linéaire (le plus sera le mieux), quadratique (simple augmentation / diminution optimale ou non linéaire) ou cubique (faussé optimale).
- Assurez-vous que le nombre de niveaux de facteurs numériques discrètes est n + 1, n étant le polynôme de degré de la relation entre le facteur et la réponse. Par exemple, si la température est prévu d'avoir un effet sur la réponse quadratique, n = 2 et donc au moins trois niveaux de température devrait être étudié à obtenir un ajustement à l'effet quadratique.
- Définir la fraction de l'espace de conception (FDS) pour lequel la variance de prédiction doit être inférieure à un certain seuil (niveau d'alpha). La FDS doit être> 0,95 (couvrir plus de 95% de l'espace de conception) pour réaliser des modèles qui donnent des prévisions solides tout au long del'ensemble 21-23 de l'espace de conception.
Remarque: un seuil de 0,05 correspond à un niveau de signification de 5% (type d'erreur I) et est une valeur typique. L'augmentation de cette valeur sera de réduire le nombre d'expériences nécessaires pour une stratégie DoE mais en même temps augmenter la probabilité que des effets significatifs ne seront pas atteints et que les estimations de leur impact sur la réponse seront incorrectes.

Tableau 1. Facteurs influant sur l'expression des protéines transitoire dans le tabac, y compris la variation des gammes pendant DoE. Facteurs en gras n'ont été incluses dans la conception pour les expériences décrites sous la rubrique «Un modèle descriptif de l'accumulation DsRed lors de l'expression transitoire en utilisant différents promoteurs / 5'UTR" tandis que les facteurs en italique n'ont été inclus dans la conception de «Optimisation incubation conditions et des systèmes de récolte pour la production d'anticorps monoclonaux dans les usines utilisant l'expression transitoire ".
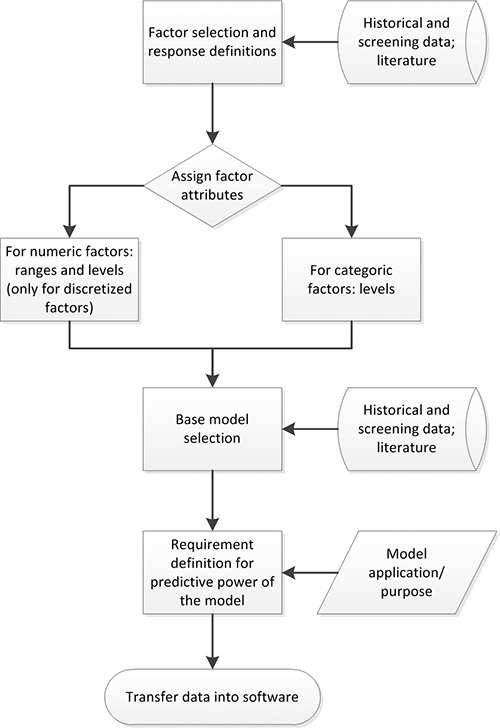
Figure 2. Processus de planification DoE. Facteurs ayant un impact significatif sur la réponse à l'étude sont choisis en fonction des données disponibles. Puis attributs de facteurs (par exemple numériques), les cuisinières et les niveaux sont affectés. Connaissances et expériences précédente sont utilisées pour définir un modèle de base appropriée. Les besoins en énergie prédictifs sont définis sur la base de l'application / le but du modèle final. Les données compilées peuvent ensuite être transférées dans le logiciel DoE approprié.
2. Mise en place d'un RSM dans DesignExpert
- Lancer DesignExpert et sélectionnez "Nouveau Design". Dans la fenêtre "Surface de réponse", choisissez &# 34; Optimal "et entrez le nombre de facteurs numériques et catégoriques sélectionnés dans la section 1.1).
- Entrez les noms des facteurs, des unités, types, sous-types, le nombre de niveaux / frontières et les valeurs pour les niveaux niveau pour tous les facteurs dans les champs correspondants.
- Continuer à la page suivante et sous la rubrique «Recherche» des options, sélectionnez "Best" ainsi que la "optimalité" critère souhaité, ici "D-optimal».
- Dans le menu "Edition modèle ...", sélectionnez le modèle qui contient les facteurs et les interactions prévues à la section 1.2). En cas de doute, sélectionnez un modèle complet qui couvre tous les facteurs et les interactions d'un certain degré de ce polynôme, ici "second degré". Notez que la sélection d'un modèle complet (contenant toutes les interactions) peut augmenter le nombre d'expériences requises pour le DoE.
- Sélectionnez le nombre de "Blocs". Ici, un seul bloc a été utilisé parce que toutes les plantes ont été du même lot, toutes les bactéries ont été cultivées simultanément ettoutes les injections ont été effectuées le même jour par le même opérateur. Utilisez plus d'un bloc si plus d'un lot de plantes est utilisé ou plusieurs opérateurs gérer les injections.
- Équilibrer la conception
- Activer "Forcer l'équilibre catégorique» afin de répartir les pistes de l'expérience de façon égale entre les niveaux de facteurs catégoriques, par exemple des promoteurs différents qui sont testés.
Remarque: cela peut réduire l'optimalité de la conception à un degré qui DesignExpert rendra compte comme valeur de% en fonction des points sélectionnés par l'algorithme d'optimalité. - Recalculer la conception plusieurs fois en utilisant l'algorithme de départ aléatoire mis en œuvre pour minimiser la réduction de l'optimalité.
Remarque: les pertes dans l'optimalité peuvent varier de ~ 3 à 40% en utilisant les mêmes données d'entrée, mais en changeant le nombre de répétitions peut être utilisé pour maintenir l'équilibre catégorique.
- Activer "Forcer l'équilibre catégorique» afin de répartir les pistes de l'expérience de façon égale entre les niveaux de facteurs catégoriques, par exemple des promoteurs différents qui sont testés.
- Le logiciel vous proposera une valeur pour le nombre de "points de modèle" basé sur le modèle de base, Ici 70 pistes. Réglez le nombre de tirages pour "répétitions" et "Pour estimer le manque d'ajustement" pour s'assurer qu'il est> 5% du nombre de "points de modèle" pour chaque, ici 10 points chacun.
- Passez à la page suivante, sélectionnez le nombre de réponses et entrer leurs noms et leurs unités. Réponses supplémentaires peuvent également être ajoutées à tout moment au cours de l'évaluation des données sans impact négatif sur la conception.
- Continuer pour démarrer l'algorithme, le calcul des taux de facteur pour les pistes de l'DoE. Si le modèle de base choisi ne correspond pas au nombre de niveaux pour un certain facteur, une notification sera affichée et le calcul ne démarre pas. Dans ce cas, soit:
- Augmenter le nombre de niveaux de ce facteur, ou
- Désélectionner les termes dans le modèle de base qui ne peut être calculée sur la base du nombre actuel de niveaux.
- Par exemple, si il existe deux niveaux pour le facteur de t "temps d'incubation" (c.-à-d 2 et 5 d) en til conception actuelle et un modèle de base quadratique dont le terme t 2 a été sélectionné, soit ajouter un troisième niveau pour t (soit 8 d) ou supprimer le facteur t 2 à partir du modèle.
- Enregistrer la feuille de calcul contenant les combinaisons de facteurs optimaux qui s'affiche une fois que le calcul est terminé.
- Dans le "Design" noeud sélectionnez l'option "évaluation" sous-nœud et cliquez sur l'onglet "Graphiques".
- Dans l'outil "Graphiques", sélectionnez "FDS" et dans la case "FDS graphique" choisir "Pred" comme type d'erreur. Entrez ensuite la différence minimale détectable ainsi que la valeur approximative de l'écart type estimé du système défini dans le paragraphe 1.1.1) en tant que valeurs de «d» et «s» respectivement (ici 20 et 8 pg / ml pour DsRed). Entrez également le niveau alpha (acceptable% manquant un effet significatif) adapté à l'application, généralement 0,05 (5%).
- Assurez-vous que la FDS calculée correspond au pourcentage défini à la section 1.3) (typiquement> 0,95 pour les modèles de prévision). Une courbe plate dans la parcelle FDS est préférable, indiquant précision de la prédiction uniforme dans tout l'espace de conception.
- Si ce n'est pas le cas (que l'on trouve ici, FDS était de 1%, comme indiqué sur la figure 3A), revenir à la "conception" noeud et dans la barre des tâches, sélectionnez «Outils de conception", puis "Augmentation de conception ..." et choisissez " augmenter ".
- Sélectionnez les mêmes "Recherche" et "critères d'optimalité" comme avant. A également choisi la même "Modifier le modèle" et les paramètres "force équilibre catégorique". Si l'augmentation de la conception est réalisée avant toute expérience (comme cela se fait ici), puis changez le "Mettez pistes en bloc» sur «Bloc1".
- Dans la section "Fonctionne", entrez le nombre de "points de modèle" supplémentaires, le maintien d'au moins 5% "répétitions" et "Pour estimer le manquede s'intégrer "dans la conception globale. Ici, 100 courses ont été ajoutés et pas de« répétitions »et« Pour estimer le manque d'ajustement »ont été incluses.
- Lorsque le calcul est terminé, réexaminer le graphe FDS comme décrit ci-dessus. Si la FDS n'est toujours pas satisfaisante, répétez l'augmentation de conception tel que décrit ci-dessus. Ici, le SDF a été de 100% après l'augmentation et donc aucune augmentation supplémentaire était nécessaire (figure 3B).

Figure 3. Comparaison des parcelles FDS. Une. Une biche constitué de 90 pistes produit une FDS insuffisant de seulement 1% pour l'erreur standard de prédiction, en utilisant un modèle de base quadratique en combinaison avec les valeurs de la différence minimale détectable (20 pg / ml) et estimationaccouplées l'écart type du système (8 ug / ml). B. Augmentation du DoE pour un total de 210 courses a réalisé un 100% FDS et une courbe plate indique précision uniforme du modèle tout au long de l'espace de conception.
3. Clonage et analyse des cassettes d'expression
- Cultiver Escherichia coli dans 5 ml de milieu LB (10 g / L de tryptone, 5 g / L d'extrait de levure, NaCl 170, 50 mg / L d'ampicilline, pour des plaques d'agar-LB comprennent 15 g / L d'agar) à 37 ° C pendant 6 -8 h ou toute la nuit sur un agitateur orbital à 160 tours par minute. ATTENTION: ampicilline est une substance nocive. Inoculer le milieu LB soit avec 50 pl de la culture liquide ou de cellules de transfert de colonies cultivées sur des plaques en utilisant un embout de pipette.
- Pour la purification d'ADN plasmidique PSO, et d'autres dérivés de ppam (GenBank AY027531), à partir de E. souche de E. coli K12 DH5a, suivez les instructions de la purification de l'ADN kit manuel 24. ATTENTION: la purification kil contient des produits chimiques nocifs (voir ci-dessus manuel pour plus de détails). La séquence du plasmide est décrite dans la figure 4 et par l'acheteur et al. 20.
- Déterminer la concentration d'ADN dans l'éluat purifié par mesure de l'absorbance d'un échantillon de 2 pl à 260 nm dans un dispositif de NanoDrop.
- Confirmer l'identité de l'ADN de plasmide purifié par digestion avec des endonucléases de restriction (RES).
- Sélectionner les ER conformément à la séquence plasmidique de sorte que des modèles de fragments uniques et distinctes sont générées. Suivez les recommandations du fabricant pour les conditions de digestion comme le volume de réaction, le temps, la température et la concentration du sérum albumine bovine stabilisateur. En fonction de la sensibilité du dispositif d'analyse, l'utilisation de 50 à 500 ng d'ADN de plasmide par digestion.
- Préparer des gels de 0,8 à 2,0% pour la séparation de fragments d'ADN par ébullition agarose dans Tris-borate et d'EDTA (TBE) (Tris 90 mM, borate 90 mM, EDTA 2 mM, pH 8,0). Til les plus grands fragments attendus, l'agarose moins doit être utilisé dans le gel.
- Ajouter 5 ul de tampon d'échantillon de cinq fois (5x SÄBU, 0,1% (p / v) de bleu de bromophénol, 0,1% (p / v) de xylene cyanol, 10% (p / v) de glycerol dissous dans du TBE) à 50 ul de l' RE-traitée échantillon d'ADN et de séparer les fragments par électrophorèse sur gel d'agarose à 100 V pour ~ 40 min ou jusqu'à ce que la séparation claire des fragments est atteint. Sur chaque gel, comprendre une voie contenant des marqueurs de taille d'échelle d'ADN à titre de comparaison, par exemple 2 à 3 pi de 1 kb ladder.
- Remplacez le 5'UTR oméga dans PSO avec l'un des trois autres 5'UTR.
- Relâchez la séquence 5'UTR oméga de ~ 4 pg d'ADN purifié PSO par traitement avec 20 unités de Eco RI-HF et Nco I-RE HF dans NEBuffer 4 à 37 ° C pendant environ 60 min. Puis séparer les fragments comme décrit dans les sections 3.4.2 et 3.4.3.
- Isoler le plus grand fragment (pS, «épine dorsale») à partir du gel d'agarose en utilisant une gel supplémentairekit ction selon le fabricant manuel 25 et déterminer la concentration de l'ADN purifié comme décrit dans la section 3.3). ATTENTION: le kit d'extraction de gel contient des produits chimiques nocifs, voir le manuel pour plus de détails.
- Isoler la SCH, CHS-LPH et TL 5'UTR de vecteurs donneurs appropriés en traitant ~ 10 ug de chaque vecteur avec 20 unités de Eco RI-HF et ER Neo I-HF dans NEBuffer 4 à 37 ° C pendant environ 60 min. Puis séparer les fragments comme décrit dans les sections 3.4.2 et 3.4.3 et purifier le plus petit fragment 5'UTR contenant tel que décrit à la section 3.5.2.
- Ligaturer les 5'UTR purifiés isolés dans des portions séparées au vecteur linéaire pS isolée et purifiée à 25 ° C pendant 5 min selon les recommandations du fabricant 26. Utilisation ~ 50 ng d'ADN vecteur et d'un excès molaire triple du 5'UTR ADN dans un volume total de 20 ul.
- La transformation des cellules de E. coli avec the plasmides recombinants 26,27.
- Ajouter ~ 10 ng (~ 4-5 pi) de mélange de ligature (voir section 3.5.4) à 50 pi RbCl E. compétente coli et mélangez délicatement, puis incuber pendant 30-60 min sur la glace. Le choc thermique pendant 1,5 minutes à 42 ° C et laisser refroidir sur glace pendant 5-30 min.
- Ajouter 950 ul de milieu LB sans antibiotique et incuber pendant 1 heure à 37 ° C et 160 tours par minute. Pour la sélection des transformants, la propagation 50 et 100 ul de la culture sur des plaques de gélose LB contenant de l'ampicilline et incuber pendant ~ 16 à 20 heures à 37 ° C.
- Inoculer 5-10 colonies distinctes représentant chacune ligature promoteur 5'UTR décrit à la section 3.1 en aliquotes de 5 ml de milieu LB contenant de l'ampicilline. Puis purifier l'ADN plasmidique et confirmer son identité, comme décrit dans les sections 3.2 à 3.4.
- Remplacer le promoteur de 35SS avec le promoteur nos dans chacun des quatre plasmides 5'UTR utilisant ER Asc I et Eco RI dans NEBuffer 4 à 37 ° C pendant 1 heure comme décrit dans les sections 3.5 et 3.6.
- Introduire chacun des huit plasmides résultants dans A. tumefaciens souche GV3101: pMP90RK par électroporation 28.
- Ajouter ~ 500 ng d'ADN plasmidique purifié à 50 A. ul compétente tumefaciens cellules sur la glace. Mélanger délicatement et transférer dans un prérefroidi 0,2 cm cuvette d'électroporation. Assurez-vous que le mélange est au fond de la cuve et ne contient pas de bulles.
- cellules d'impulsions à 2,5 kV pendant 5 ms et confirmer l'intensité et la durée de l'impulsion.
- Éviter les concentrations de sel élevées dans l'échantillon d'ADN ou d'une préparation incorrecte de la R. compétente tumefaciens cellules parce que celles-ci peuvent entraîner des courants ioniques élevées provoquant la vaporisation instantanée de la suspension de cellules, ce qui réduit considérablement l'efficacité de transformation.
- Ajouter 950 ul de milieu sans antibiotiques YEB (extrait de 5 g / L de boeuf, 1 g / L d'extrait de levure, 5 g / L de peptonee, 5 g / L de saccharose, 2 mM de MgSO 4, pH 7,0), et mélanger doucement transférer immédiatement dans un tube de réaction de 1,5 ml stérile. Incuber pendant 2-4 heures à 26-28 ° C et 160 tours par minute.
- Étaler 1-2 pi sur des plaques d'agar YEB contenant des antibiotiques (50 mg / L carbénicilline, 25 mg / L de kanamycine, 25 mg / L de rifampicine) pour la sélection des transformants. ATTENTION: la rifampicine est une substance toxique.
- Inoculer trois aliquotes de 5 ml de milieu YEB contenant les antibiotiques avec des cellules provenant de colonies distinctes représentant chacune ligature promoteur-5'UTR et incuber pendant 48-72 heures à 26-28 ° C et 160 tours par minute.
- Confirmer le succès de A. tumefaciens.
- Transfert 2 pi de chaque aliquote de la section 3.8.6 pour séparer 48 aliquotes de mélange maître PCR (2 ul de chaque 10 uM primaire actions (400 nM de concentration finale de chaque amorce),1 ul 10 mM dNTP mix (200 uM de concentration finale de chaque désoxynucléoside triphosphate), 5 pi 10 fois Expand High Fidelity tampon avec 15 mM de MgCl2, 0,75 Développer ul High Fidelity mélange d'enzymes, et 39,25 pi d'eau distillée stérile).
- Amplifier la cassette d'expression de chaque plasmide en utilisant des amorces appropriées (FWD: 5'-CCT CAG GAA GAG CAA TAC-3 ', 1026 nucléotides de liaison en amont du promoteur; REV: 5'-CCA AAG CGA GTA CAC AAC-3', la liaison au sein du site de polyadénylation 35S) dans des conditions de PCR appropriées. Ici, 94 ° C a été utilisé pour la dénaturation initiale, suivie par 30 cycles de 94 ° C dénaturation pendant 15 s, 51 ° C, une hybridation pendant 30 s et 72 ° C un allongement de 120 sec, et une étape d'élongation finale à 72 ° C pendant 8 min.
- Déterminer la taille des produits de PCR par électrophorèse sur gel d'agarose comme décrit à la section 3.4.3.
- Préparer A. tumefaciens stocks de glycérol par500 ul de mélange de 50% (v / v) de glycérol stérile avec 500 ul de A. tumefaciens cultures de la section 3.8.6 pour lequel la transformation a été confirmée (section 3.9.3). Stocker les solutions mères de glycerol à -80 ° C jusqu'à utilisation ultérieure.
- Calculer les énergies de pliage des différents ARNm en utilisant le serveur Web 29 et RNAfold inclure la séquence de nucléotides à partir du site d'initiation de la transcription par le biais de la 50 premières pb de la région codante.
- Dans la section "Pliez les algorithmes et les options de base" sélectionnez l'option "minimum d'énergie libre (MFE) et la fonction de partition» et «éviter isolés paires de bases" options.
- Dans les "Options de pliage« choisir »qui pendent énergies des deux côtés d'une hélice en tout cas", "paramètres d'ARN (modèle Turner, 2004)" et changer "redimensionner paramètres d'énergie à température donnée (C)" à 25 ° C.
- Dans la section "Options de sortie", sélectionnez toutes les options.
- De la «Résultats pour thermodynamique ensemble prédiction" section du fichier de sortie, l'extrait «l'énergie libre de l'ensemble thermodynamique" et la "fréquence de la structure MFE dans l'ensemble" des valeurs de comparaison.
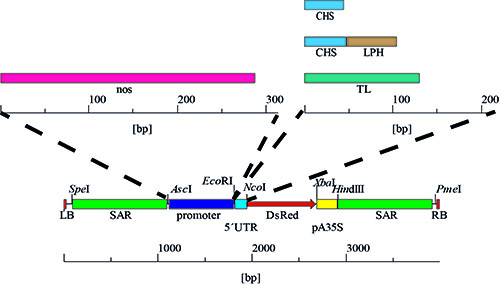
Figure 4. Promoteur et 5 'UTR variantes. Les cassettes d'expression ont été générées par l'échange par étapes de la 5'UTR, résultant en quatre combinaisons avec le promoteur CaMV 35SS, suivi par le remplacement de ce promoteur à la séquence n ° donnant quatre variantes supplémentaires et un total de huit combinaisons différentes promoteur / 5'UTR.
4. Culture des plantes
- Préparer une solution à 0,1% de l'engrais Ferty2 Mega dans de l'eau déminéralisée à pH 5,9.
- Préparer 10 blocs x 10 x 8 cm de laine de roche par une vaste chasse à l'eau déminéralisée pour éliminer les produits chimiques résiduels et finalement s'équilibrer avec engrais.
- Graines de plantes de tabac en plaçant 1-2 graines de tabac sur chaque bloc de laine de roche suivie d'une courte chasse avec de l'engrais en faisant attention à éviter de laver les graines loin.
- Germer et cultiver les plants de tabac pendant 42 jours dans une serre à température 25/22 ° C jour / nuit, 70% d'humidité relative et une photopériode de 16 h (180 mmol m -2 s -1; λ = 400-700 nm) . Au cours de cette photopériode, irriguer avec de l'engrais pendant 15 min toutes les heures dans la culture hydroponique.
5. L'expression transitoire de protéines
- Préparer A. tumefaciens pour l'injection dans les feuilles.
- Inoculer 5 à 50 ml de milieu YEB contenant des antibiotiques à 1% A. tumefaciens cryo stock culture et incuber à 27 ° C jusqu'à ce que la DO 600 nm atteint 5.0 (~ 48-72 heures selon le volume et le type de navire).
- Diluer le A. culture tumefaciens avec de l'eau et le milieu de l'infiltration de 2 fois (4,3 g / L de Murashige et Skoog sels (pH 5,6), 5 g / L de saccharose, 1,8 g / L de glucose, de l'acétosyringone 100 mM) en fonction de la DO 600nm requise pour l'injection. Confirmer la DO 600nm juste avant l'injection. Remarque: Une DO 600nm de 1,0 correspond à ~ 1,43 ± 0,12 x 10 9 unités formatrices de colonies par ml de formation.
- Injecter A. tumefaciens suspension dans les feuilles.
- Sélectionner et étiqueter les feuilles noncotyledon et positions à ce sujet à traiter (par exemple, selon une stratégie DoE).
- Ne pas injecter différente A. tumefaciens solutions dans le même domaine intercostal. Au lieu de cela, utiliser des sections opposées de chaque côté de l'axe mi-veineuse. Si plus de deux solutions différentes doivent être injectés à la même position en utilisant un additional usine.
- Secouez A. solution tumefaciens pour remettre en suspension les cellules qui peuvent se sont installés depuis la préparation de la dilution et aspirer dans une seringue de 1 ml.
- Gratter délicatement l'épiderme à l'emplacement prévu pour l'injection avec une pointe de pipette ou similaire afin de faciliter l'afflux de l'A. tumefaciens solution. Éviter une rupture du limbe de la feuille en le faisant.
- En tenant la seringue perpendiculaire à la lame de la lame de toucher le cylindre contre le domaine intercostal à traiter et pousser doucement l'orifice de sortie (sans aiguille attachée) sur la face inférieure de la feuille. Appuyer sur la face supérieure de la feuille doucement en même temps pour empêcher la lame de feuille de se déplacer ou se rompre.
- Appuyez doucement sur le piston de la seringue. A. solution tumefaciens entrera dans les espaces intercellulaires dans le limbe de la feuille comme indiqué par les zones traitées apparaissant vert plus foncé et humide. Répétez cette procédure à plusieurs positions jusqu'à ce que l'ensemble intercostadomaine de l est infiltrée par A. tumefaciens. Puis, passez à la prochaine champ intercostal.
- Assurez-vous que la seringue reste perpendiculaire à la feuille. Inclinant la seringue entraînera la suspension bactérienne à jaillir sous forte pression.
- Si différente A. tumefaciens solutions sont utilisées (par exemple, pour tester différents promoteurs), enlever tout excès de solution restant sur la face inférieure de la feuille à partir de la première injection à l'aide d'une serviette en papier ou similaire avant d'appliquer la solution suivante.
- Post-infiltration incubation de plantes et d'échantillonnage.
- Préparer un phytotron pour les plantes traitées ~ 24 heures avant l'injection afin température et l'humidité équilibre aux niveaux requis pour le DoE.
- Après l'injection, transférer les plantes dans le phytotron et les placer dans des bacs assez grands pour l'irrigation avec de l'eau jusqu'à la fin de la période d'incubation déterminée par le DoE.
- Établir une phot de 16 hoperiod utilisant six Osram blanc froid 36 W tubes fluorescents par 0,7 m 2 (75 mmol s -1 m -2; λ = 400-700 nm). Empêcher les plantes à partir de l'ombrage de l'autre en limitant le nombre de plantes à six par 0,7 m 2.
- Avant l'échantillonnage, vérifiez que le champ intercostal correct a été sélectionné en comparant l'étiquette ajoutée à la section 5.2.1 et le plan DoE.
- Utilisez un perce-bouchon pour enlever 4-5 disques foliaires dans les champs intercostaux traitées aux positions et aux heures indiquées par le DoE. Ne retirez pas la feuille entière de la plante lors de l'échantillonnage. Stabiliser la feuille avec une serviette en papier à main tout en retirant les disques pour éviter la rupture.
- Déterminer la masse de chaque échantillon et le placer dans un tube en plastique de réaction de 1,5 ml étiqueté avec le nom de l'échantillon et de la masse. Conserver les échantillons à -20 ° C ou -80 ° C avant la quantification des protéines. Le processus peut être interrompu à ce stade pour plusieurs mois en fonction de la stabilité de l'échantillonet la température de stockage.
6. La quantification de protéine
- Extraire des protéines à partir des échantillons de disques de feuille.
- Ajouter 3 ml de tampon d'extraction (phosphate de sodium 50 mM, chlorure de sodium 500 mM, pH 8,0) par mg de la masse de l'échantillon et broyer des disques de feuilles du tube de réaction en utilisant un pilon électrique jusqu'à ce que pas de grands fragments subsistent. Éviter une surchauffe de l'échantillon.
- Éliminer les matières solides dispersées par centrifugation de l'échantillon deux fois à 16000 g pendant 20 min à 4 ° C. Transférer le surnageant dans un tube de 1,5 ml propre réaction après chaque étape, sans perturber le culot.
- Après centrifugation, le processus peut être interrompu par des extraits de plantes la congélation à -20 ° C ou -80 ° C pendant plusieurs mois en fonction de la stabilité de l'échantillon et la température de stockage. Confirmer que le cycle de congélation-décongélation n'affecte pas la concentration de la protéine (s) cible.
- Mesurer la fluorescence DsRed.
- Préparer trois répétitions techniques de chaque échantillon en plaques à 96 puits demi-zone noire (50 extrait de pi par puits). Éviter la formation de bulles pendant le pipetage.
- Utilisez un ensemble de six dilutions (0, 25, 75, 125, 175 et 225 pg / ml) d'une norme DsRed par plaque de 96 puits pour générer une courbe de référence de ligne. Préparer les dilutions dans du PBS et les stocker à 4 ° C pour une utilisation dans les 3 mois.
- Mesurer la fluorescence à deux reprises, successivement, dans un lecteur de plaque de 96 puits équipés de 530/25 nm excitation et 590/35 nm filtres d'émission.
- Pour chaque échantillon, la moyenne de la fluorescence au cours des deux lectures et les trois répétitions techniques et soustraire la valeur enregistrée pour le contrôle à blanc contenant 0 ng / ml DsRed. Aussi soustraire cette valeur du lit des dilutions standard et utiliser ces valeurs vides corrigée pour une régression linéaire donnant une courbe de référence (par l'origine des ordonnées).
- Utiliser la pente de la courbe de référence pour convertir la fluorescence mesurée pour les échantillons en concentrations dsRED. Si nécessaire, diluer les échantillons de telle sorte que la lecture se situe dans l'intervalle des normes de concentration et de considérer ce facteur de dilution dans les calculs suivants.
- Déterminer la concentration de 2G12.
- Préparer un dispositif de résonance des plasmons de surface (SPR) pour la mesure de la concentration de l'anticorps par couplage de la protéine A à la surface activée d'une cellule d'écoulement. Utiliser dans une autre cellule d'écoulement en tant que référence par l'inactivation de la surface sans accouplement de la protéine A 30,31.
- Diluer extraits de plantes à 01:20 SPR courant de tampon (HEPES 10 mM pH 7,4, EDTA 3 mM, NaCl 150 mM, 0,05% v / v de Tween-20) et de mesurer les unités de réponse (RU) de liaison de l'anticorps à la protéine A dans trois réplicats techniques de chaque échantillon à la fin d'une injection de 90 ul (180 s à 30 pl / min). Soustraire le RU mesurée à la cellule d'écoulement de référence (pas la protéine A) du RU mesurée dans le fl expérimentalow cellulaire (protéine A de surface).
- Mesurer un niveau de 585 ng / ml après 2G12 chacun des échantillons 10 à 15 et de soustraire la valeur mesurée dans les cellules de flux de référence tels que décrits ci-dessus. Utilisez le RU moyenne de ces normes pour calculer une courbe de référence linéaire par l'origine des ordonnées.
- Calculer les concentrations 2G12 dans les échantillons en fonction de la dilution 1:20 et la pente de la courbe de référence.
- Vérifiez forte liaison non spécifique à la cellule de référence, qui peut corrompre la mesure. Vérifiez également si les normes 2G12 maintenir des valeurs approximativement constants (<5% de variation) tout au long de l'analyse, comme une plus grande variation reflète le vieillissement de la protéine A surface.
7. Analyse des données et de l'évaluation
- Analyser manuellement les réponses observées dans les expériences d'expression pour (i) les valeurs extrêmes des erreurs (haute ou basse) indiquant mesure, (ii) des résultats inhabituels, par exemple les combinaisons de facteurs qui gèneévaluer les réponses imprévues indiquant l'échange de données, et (iii) les valeurs manquantes.
Remarque: utilisez ces actions pour empêcher la construction du modèle basé sur des données erronées. - Transférer les données de réponse analysés (ici les concentrations de protéines) dans le noeud "Design" de DesignExpert. Assurez-vous que les données de réponse sont correctement assignés aux paramètres de facteur correspondant. Il ya une section d'aide détaillé disponible dans le logiciel de DesignExpert qui couvre les aspects abordés ci-dessous.
- Dans le noeud "Analyse", choisir la réponse à analyser et d'abord sélectionner "Non" dans l'onglet de transformation.
Remarque: une transformation est recommandé pour les min / max ratios de la réponse de plus de 10. La transformation la plus utile peut être obtenue à partir de la Box-Cox-terrain dans l'onglet "Diagnostic" dans la section "Diagnostic" de l'outil "Diagnostics" décrit ci-dessous (section 7.8). Un journal de 10 transformation est souvent approprié. - Continuer à l'onglet "résumé Fit", qui fournit des informations générales sur les facteurs qui sont importants pour le système étudié (par exemple interactions à deux facteurs, les effets du second degré). Le logiciel vous proposera un premier modèle en fonction de son importance.
- Dans l'onglet "Modèle", un modèle initial est présélectionné en fonction des résultats "sommaires" Fit. Utilisez le mode automatisé pour modifier ce modèle:
- Sélectionnez un "ordre de process" qui est un ordre au-dessus du modèle proposé, par exemple, si le modèle proposé est "2FI" (deux d'interaction des facteurs), puis sélectionnez "quadratique".
- Choisissez "arrière" dans le champ "Sélection", qui itérative effacer un terme non significatifs du modèle après avoir procédé à l'onglet «variance» sur la base de la valeur "Alpha out" qui devrait d'abord être 0.100.
- Si demandé par le logiciel, toujours corriger automatiquement la hiérarchie du modèle parce que c'est nécessairementy pour générer des modèles fiables 32,33.
- Dans l'onglet "variance", enquêter sur le modèle proposé et les facteurs pris en compte. Si nécessaire, supprimer manuellement tous les facteurs avec des valeurs p-dessus d'un seuil prédéfini (ici 0,05, correspondant à un niveau de signification de 5%) ou ceux qui sont peu probable fondée sur un examen mécanique en revenant à l'onglet "Modèle", changer "Sélection" "Manuel" et en éliminant les facteurs appropriés du modèle.
- Retour dans l'onglet "variance", d'évaluer le modèle actualisé en fonction de la p-valeur du «modèle» (une valeur faible est souhaitable, car cela indique l'importance) et la (une valeur élevée "Manque d'ajustement" est souhaitable parce que cela signifie l'absence de signification), ainsi que les valeurs de "R-squared", "R-carré ajusté» et «prédite R-squared" (valeurs> 0,80 sont souhaitables pour les trois valeurs).
- Comparez ces valeurs for différents modèles, y compris / hors facteurs à plusieurs niveaux de signification.
Remarque: il peut être utile de reproduire la colonne de réponse dans le "Design" noeud et effectuer chaque analyse individuelle, ou d'exporter la totalité de la table "variance" dans un autre programme comme un tableur.
- Comparez ces valeurs for différents modèles, y compris / hors facteurs à plusieurs niveaux de signification.
- Continuer à l'onglet "Diagnostic" pour confirmer la qualité du modèle et de détecter les valeurs aberrantes dans l'ensemble de données qui ont une forte influence sur le modèle en examinant tous les onglets de l'outil "Diagnostics" (section "Influence" "Diagnostic" et) .
- Dans la section "Diagnostic" de l'outil "Diagnostics", effectuer les actions suivantes:
- S'assurer que les points sont dispersés au hasard dans les limites de la "Résidus vs prédites" graphique. Ici, une distribution en forme de chevron indique un besoin de transformation de données.
- S'assurer que les points sont dispersés au hasard dans les limites de la"Résidus vs prédite" graphique. Ici, une distribution en forme de chevron indique un besoin de transformation de données.
- Vérifier si les points de la "Résidus vs terme" tableau dispersion au hasard dans les limites. Un modèle (par exemple, "l'escalier") indique une tendance dans le bâtiment de données et le bloc peut être utilisé pour contrecarrer son influence sur le modèle.
- Recherchez une ligne droite en diagonale indiquant le modèle idéal de la "Prévision et réel« complot. Plus la dispersion des données autour de la diagonale, la moins précise du modèle.
- Consultez un chevauchement de bleu (meilleur) et vert (réels) des lignes verticales dans le Box-Cox-terrain indiquant la transformation de données. Si la ligne verte ne correspond pas à la bleue (en dehors de l'intervalle indiqué par les lignes verticales rouges) revenir à la section 7.3 et d'améliorer le modèle rond de construction en sélectionnant la transformation de données proposé par la Box-Cox-terrain. Encore une fois la duplication de la recolonne de réponse peut être utile de comparer les différents modèles.
- Assurez-vous que les points dans les charts "Résidus vs Factor" (il ya un tableau pour chaque facteur du modèle) se dispersent aléatoirement dans les limites. Courbure de la distribution de données peut indiquer un facteur manquant dans le modèle.
- Dans la section «influence» de l'outil "Diagnostics", assurez-vous qu'il est une diffusion aléatoire des données dans les limites des «résidus Extérieurement studentisés", "effet de levier", "DFFITS" et les parcelles "DFBETAS" et uniformément bas de distribution sans valeurs extrêmes de la parcelle "de Cook Distance".
- Dans la section "Diagnostic" de l'outil "Diagnostics", effectuer les actions suivantes:
- Dans l'onglet "graphiques du modèle", de visualiser le modèle évalué. Pour un nombre limité de facteurs numériques (ex. 3) la surface de réponse ("surface 3D") la représentation est utile pour évaluer optima / caractéristiques manuellement.
Remarque: surfaces de réponse ne illustrent l'impact de deux facteursteurs sur la réponse à l'étude. L'effet d'un facteur supplémentaire de la réponse se révèle en changeant sa valeur (facteurs numériques) ou niveau (facteurs catégoriques) dans la fenêtre «Facteurs de fonction". Alternativement, les facteurs peuvent être affectés à l'axe du terrain par les droit-cliquant dessus dans la fenêtre "Facteurs de fonction" et en sélectionnant l'axe variable indépendante souhaitée.- Manipuler les niveaux de facteurs et de les affecter aux ordonnées du graphique à l'aide de l'outil "Facteurs".
- graphes dans le "graphe Exporter vers un fichier ..." dans le menu "Fichier".
- Utilisez le "numérique" sous-nœud dans le "optimisation" noeud pour optimiser la réponse numérique (minimiser, maximiser, dans la gamme) en fonction des facteurs de modèle, à qui à son tour certaines contraintes peuvent être appliquées (par exemple, les limites et les poids) par l'intermédiaire du onglet «critères».
- Calculer et examiner les solutions numériques dans les «Solutions»onglet sur la base des éléments fournis dans l'onglet "Critères".
- Export de ces solutions à d'autres logiciels (par exemple des feuilles de calcul) pour une analyse plus approfondie (par exemple des histogrammes) révélant les paramètres de facteurs associés à des valeurs élevées ou basses réponse.
Note: ceci est utile si plus de trois facteurs numériques sont étudiés et la représentation 3D est difficile.
- Utiliser la "prédiction Point" sous-noeud pour prédire la réponse à des paramètres spécifiques de facteurs.
- Utiliser cette prédiction pour tous les paramètres à évaluer (ici, tout promoteur et des combinaisons 5'UTR ainsi que toutes les feuilles et les temps d'incubation ou de positions des feuilles et les températures d'incubation).
- Exporter les valeurs prédites et les compiler un tableau de données qui peut être utilisé pour décrire l'expression transitoire dans des plants de tabac, par exemple par le calcul de l'expression pour une combinaison spécifique de promoteur-5'UTR à une certaine heure en moyenne sur l'ensemble des positions des feuilles ou des across toutes les feuilles d'une plante.
- Normaliser les données d'expression sur la base de la masse des différentes feuilles pour donner le niveau d'expression spécifique (ug g -1) ou le taux d'expression spécifique (ug g -1 h -1).
- Sinon, exporter les coefficients de «l'équation finale" du bas de l'onglet "ANOVA" dans une feuille de calcul et les multiplier avec un éventail de paramètres de facteurs pour obtenir le même ensemble de données.
Note: ce tableau ne peut contenir que des valeurs de facteur de l'intérieur de l'espace de conception initiale, car l'équation de modèle ajusté n'est pas adapté pour l'extrapolation.
- Mener une expérience de confirmation pour les points de modèle qui sont d'un intérêt majeur.
- Effectuer expression transitoire de protéines dans des conditions choisies pour la «prédiction Point" (section 7.11). Par exemple, utiliser les mêmes températures et les feuilles, etc
- Déterminer les concentrations de protéines pour cette expression transitoire experiment comme décrit ci-dessus (article 6) et les comparer avec les valeurs prédites par le modèle.
- Vérifiez si la concentration moyenne en protéines dans l'expérience de confirmation relève de l'intervalle de prédiction du modèle de surface de réponse obtenue lors de point de prédiction. Un match confirme le pouvoir prédictif du modèle. Une discordance indique une faible qualité du modèle et essais supplémentaires peuvent être nécessaires.
- Remarque: plante variabilité de lot à lot élargit l'intervalle de prédiction et peut dévaluer expériences de confirmation effectués avec un autre lot de plantes. Cependant, les échantillons qui n'ont pas été utilisées pour générer le modèle mais qui proviennent du même lot de plantes que les échantillons contenus dans le modèle peuvent servir de témoins efficaces.
Résultats
Un modèle descriptif de l'accumulation DsRed lors de l'expression transitoire en utilisant différents promoteurs et 5'UTR
DsRed fluorescence dans des extraits de feuilles a été utilisé pour indiquer le niveau de la protéine recombinante d'expression et, par conséquent a été utilisé en tant que réponse à la stratégie DoE. La différence minimale détectable nous avons considéré pertinent était de 20 pg / ml et l'écart type estimé du syst?...
Discussion
Chaque expérience nécessite une planification minutieuse, car les ressources sont souvent rares et chers. Cela est particulièrement vrai pour les stratégies DoE car les erreurs au cours de la phase de planification (par exemple, la sélection d'un modèle de base qui ne couvre pas toutes les interactions de facteurs importants) peuvent sensiblement diminuer le pouvoir prédictif des modèles issus et donc dévaluer toute l'expérience. Cependant, ces erreurs peuvent être facilement évitées en sui...
Déclarations de divulgation
La taxe de publication a été en partie financé par les entreprises Statease, Inc. (Etats-Unis) et Statcon (Allemagne), qui n'ont pas été impliqués dans la impliqué dans la préparation du manuscrit ou responsable de son contenu.
Remerciements
Les auteurs tiennent à remercier le Dr Thomas Rademacher pour fournir le vecteur d'expression PPAM végétale et Ibrahim Al Amedi pour cultiver les plants de tabac utilisées dans cette étude. Nous tenons à remercier le Dr Richard M. Twyman pour son aide dans l'édition du manuscrit. Ce travail a été en partie financé par le Conseil européen de la recherche avancée Grant "Future-Pharma", la proposition n ° 269110 et le Fraunhofer Zukunftsstiftung (Fondation Fraunhofer avenir).
matériels
| Name | Company | Catalog Number | Comments |
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life Technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10 x10 cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life Technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
Références
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ - The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5'UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon