Method Article
En Vitro Reconstitución de complejos captadores de luz de Plantas y Algas
En este artículo
Resumen
This protocol details the reconstitution of light-harvesting complexes in vitro. These integral membrane proteins coordinate chlorophylls and carotenoids and are responsible for harvesting light in higher plants and green algae.
Resumen
En las plantas y las algas verdes, la luz es capturada por los complejos captadores de luz (cachés de host local), una familia de proteínas integrales de membrana que coordinan las clorofilas y carotenoides. In vivo, estas proteínas se pliegan con pigmentos para formar complejos que se insertan en la membrana tilacoide del cloroplasto. La alta similitud en las propiedades químicas y físicas de los miembros de la familia, junto con el hecho de que pueden perder fácilmente pigmentos durante el aislamiento, hace que su purificación en un estado nativo desafiante. Un enfoque alternativo para obtener preparaciones homogéneas de cachés de host local fue desarrollado por Plumley y Schmidt en 1987 1, que mostró que era posible reconstituir estos complejos in vitro a partir de pigmentos purificados y apoproteínas desplegadas, dando como resultado complejos con propiedades muy similares a la de la nativa complejos. Esto abrió el camino para el uso de proteínas recombinantes expresadas por bacterias in vitro (por ejemplo, sitios de unión de pigmento) o proteína de dominio (por ejemplo, la interacción proteína-proteína, plegable). Este método ha sido optimizado en varios laboratorios y se aplica a la mayoría de los complejos captadores de luz. El protocolo descrito aquí se detalla el método de reconstituir los complejos captadores de luz in vitro utilizados actualmente en nuestro laboratorio, y ejemplos que describen aplicaciones del método se proporcionan.
Introducción
El aparato fotosintético de las plantas y las algas incluyen proteínas integrales de membrana que se unen clorofila a (Chl a), b (CHL b) y carotenoides (coche). Estos complejos pigmento-proteína están activos en energía de la luz solar y transferir esa energía de excitación a los centros de reacción, donde se utiliza para promover la separación de la carga 2. Ellos también están involucrados en los mecanismos de retroalimentación reguladoras que protegen el aparato fotosintético de daños luz alta 3,4. Los complejos de aprovechamiento de luz (cachés de host local) se componen de una gran familia de proteínas relacionadas en plantas y algas 5.
La purificación a homogeneidad de cada miembro de la familia se ha visto complicada por las propiedades químicas y físicas muy similares de los complejos. Además, los procedimientos de purificación a menudo resultan en la pérdida de pigmentos u otros cofactores potenciales, tales como lípidos. Reconstitución in vitro represents un poderoso método para superar estos problemas. El LHC asociado con el Fotosistema II (LHC-II) se reconstituyó primero in vitro por Plumley y Schmidt en 1987 1. Los investigadores extrajeron proteínas sin lípidos y pigmentos por separado de los cloroplastos de la planta, y luego combinar la proteína desnaturalizada por calor con pigmentos en presencia de litio dodecil sulfato (LDS), seguido de tres ciclos de congelación y descongelación 1. Ellos mostraron que las propiedades espectrales de los complejos LHC reconstituidos fueron muy similares a los complejos purificados a partir de plantas. La facilidad de reconstitución de los complejos de pigmento-proteína LHC, probablemente debido a alguna característica de auto-ensamblaje inherente, junto con la dificultad de aislar complejos purificados a partir de organismos, condujo a la rápida adopción del método por otros investigadores. La reconstitución de proteínas fotosintéticas sobreexpresados en Escherichia coli (E. coli) se logró mediante Paulsen y sus colegas en 1990 6. En E.coli, proteínas de membrana sobreexpresados son típicamente contenidos en cuerpos de inclusión, que las instalaciones de su purificación. La reconstitución se logra a través de desnaturalización por calor de los cuerpos de inclusión que contienen la proteína recombinante en presencia de LDS, seguido por la adición de pigmentos que inicia el plegado de proteínas. Plegable del complejo LHCII es un proceso de dos pasos: primero, la clorofila a se une en menos de 1 min; segundo, la clorofila b se une y se estabilizó durante varios minutos 7.
Además de proporcionar una idea de la dinámica de plegado, en la reconstitución in vitro combinado con mutagénesis dirigida al sitio ha permitido la identificación de aminoácidos específicos importantes para la estabilidad (por ejemplo, 8,9) o la coordinación de pigmento (por ejemplo, 10). La manipulación de replegamiento condiciones mediante el ajuste de parámetros como la composición del pigmento o detergentes también han identificado elementos critical para plegamiento apropiado, tales como el requisito de xantofilas para el complejo LHCII (por ejemplo, 1,11). Además, la investigación de las propiedades de los pigmentos individuales unidos a los complejos ha sido posible usando complejos reconstituidos in vivo (por ejemplo, 10).
El método descrito aquí comienza con el aislamiento de pigmentos (clorofilas y carotenoides) de la espinaca y la alga verde Chlamydomonas reinhardtii. La expresión y purificación de una proteína a partir de E. LHC coli en forma de cuerpos de inclusión se detalla a continuación, seguido de la reconstitución de LHC y posterior purificación por columna de afinidad de Ni. En el paso final, los complejos reconstituidos se purificaron adicionalmente por centrifugación en gradiente de sacarosa para eliminar pigmentos libres y apoproteína desplegada. Este protocolo representa un procedimiento optimizado que incorpora varias modificaciones que se han introducido por diferentes laboratorios mástiempo 1,6,10,12 -14.
Protocolo
1. total Pigmento Extracción de hojas de espinaca
- Homogeneizar un puñado de hojas de espinaca (~ 20 g) en 100 ml de tampón de molienda en frío (ver Tabla 1) con una batidora durante 20 segundos.
- Filtrar la solución a través de una de dos capas de tela de nylon con un diámetro de poro de 20 micras y se centrifuga el filtrado a 1.500 xg durante 10 min a 4 ° C.
- Resuspender el sedimento que contiene los cloroplastos con un artista suaves pintar pincel en 1 ml de tampón de lavado refrigerado (ver Tabla 1). Una vez que el sedimento se volvió a suspender, añadir 50 ml de tampón de lavado y centrifugar la solución a 10.000 xg durante 10 min a 4 ° C.
- Eliminar el sobrenadante y suavemente resuspender el precipitado (tilacoides) en 50 ml de tampón de lavado (ver la Tabla 1).
- Centrifugar la solución a 10.000 xg durante 10 min a 4 ° C y eliminar el sobrenadante por completo. En este punto, llevar a cabo los siguientes pasos en la oscuridad, para evitar la oxidación del pigmento.
- Añadir ~ 20 ml de acetona al 80% tamponada con Na 2 CO 3 (ver Tabla 1) para extraer los pigmentos. Dejar la solución en hielo durante 10 min, vórtice de vez en cuando.
- Se precipitan los componentes celulares por centrifugación a 12.000 xg durante 15 min a 4 ° C.
NOTA: Si los pigmentos no están totalmente extraídos, el pellet tendrá un color verde y el paso 1.6 se debe repetir. - Recoger el sobrenadante en un embudo de decantación. Añadir 0,4 volúmenes de éter dietílico, agitar vigorosamente y abra la válvula para ventilar el gas.
- Añadir 0,8 volúmenes de NaCl 0,33 y mezclar enérgicamente. Permitir ~ 10 min para las capas se separen. La fase de éter en la parte superior contiene los pigmentos extraídos. Retire la fase inferior clara.
NOTA: Si la separación no es clara, congelar y descongelar la solución para mejorar la separación de fases. - Eliminar el éter mediante el vertido desde la parte superior del embudo de decantación en un recipiente de vidrio adecuado. Dry añadiendo una cucharada de gransulfato de sodio anhidro ular. Agitar la solución y permitir ~ 5 min para el desecante para absorber agua del éter.
NOTA: Repita este paso si el sulfato de sodio aparece completamente agrupó; debe haber algunos cristales de libre flotación cuando el éter se seca suficientemente. Si se forma una capa de agua, suprimir este con una pipeta Pasteur antes de añadir sulfato de sodio anhidro adicional. - Decantar el éter a un nuevo recipiente de vidrio, dejando el sulfato de sodio sólido atrás.
- Se evapora el éter en un SpeedVac giratorio o bajo una corriente de N 2.
- Disolver los pigmentos completamente en 10 ml de acetona al 100%.
- Diluir una pequeña cantidad (~ 3 l) en 1 ml de 80% de acetona y medir los espectros de absorción y determinar una relación de la Chl / b y la concentración de Chl con el método descrito por Porra et al. (1989) 15.
- Alícuota y seque los pigmentos en un Speedvac rotatorio o bajo corriente de N2 hasta que la acetona esevaporado completamente. Guarde los pigmentos secos a -80 ° C.
2. Extracción de carotenoides de la espinaca
- Siga los pasos 1.1 a 1.5. En este punto, llevar a cabo los siguientes pasos en la oscuridad, para evitar la oxidación del pigmento.
- Resuspender el precipitado tilacoide en ~ 50 ml de etanol 96% tamponado con Na 2 CO 3 (ver Tabla 1) para extraer los pigmentos. Dejar la solución en hielo durante 5 min.
- Se precipitan los componentes celulares por centrifugación a 12.000 xg durante 15 min a 4 ° C.
NOTA: Si los pigmentos no están totalmente extraídos, el pellet tendrá un color verde y el paso 2.2 se debe repetir. - Recoger el sobrenadante y añadir 0,1 volumen de 80% de KOH (w / v) para iniciar la saponificación.
- Deje la solución a 4 ° CO / N, bien tapado y protegido de la luz.
- Recoger la solución en un embudo de separación. Añadir 1 volumen de éter dietílico y mezclar suavemente.
- Añadir 0,8 volumes de M NaCl 0,33 y mezclar suavemente. Permitir ~ 10 min para las capas se separen. La fase de éter de naranja en la parte superior contiene los carotenoides saponificados. Eliminar la fase inferior verde mediante el drenaje a través de la llave de paso del embudo.
- Añadir 3 volúmenes de agua y mezclar suavemente para eliminar el hidróxido de potasio. Deje que las capas se separen. NOTA: Si la fase superior aparece turbia, agregue una pequeña cantidad de NaCl (por ejemplo, 3 g de NaCl en 200 ml de solución) y girar suavemente para que se disuelvan.
- Eliminar la fase inferior mediante el drenaje a través de la llave de paso del embudo.
- Siga los pasos 01.10 a 01.13.
- Diluir una pequeña cantidad (~ 3 l) en 1 ml de 80% de acetona y medir los espectros de absorción a 440 nm en 80% de acetona. Para determinar la concentración, utilice la extinción media de coeficiente para los carotenoides (ε 440 = 255) 16 en la siguiente fórmula: coches [/ ml mg] = (Abs 440 nm / 225) x 11 (camino óptico) = 1 cm.
- Alícuota y seque el carotenoids en un Speedvac o bajo corriente de N2 hasta que todo dietiléter se ha evaporado. Guarde los pigmentos secos a -80 ° C.
3. total de pigmentos carotenoides y Extracción de Chlamydomonas reinhardtii
- Crecer C. reinhardtii en medio TAP sólido 17 en una placa de Petri mediante la difusión de una pequeña cantidad de cultivo líquido sobre la superficie. Crecer bajo flujo de iluminación continua de 20 mol fotos PSA m -2 s -1 hasta una capa verde de las células es visible.
- Con un asa de inoculación estéril, cosecha una pequeña cantidad de C. reinhardtii del medio TAP sólida y poner las células en 500 ml de medio TAP 17 en un matraz de 1 L. Mantener el cultivo a 25 ° C con 170 rpm de agitación bajo un flujo de iluminación continua de 20 mol fotos PSA m -2 s -1.
- Después de 5-6 días, la cultura debe llegar hasta el final de la fase logarítmica (6 x 10 6 células / mlo 2-2,5 densidad óptica a 750 nm). Se centrifuga el cultivo a 4.000 xg durante 15 min a 4 ° C.
- Para la extracción total de pigmentos, siga los pasos 1.6 a 1.15.
- El rendimiento de extracto total de pigmento a partir de 500 ml de cultivo de crecimiento completo de C. reinhardtii es de alrededor de 5 ml de solución con una concentración de 0,5 mg chl a + b / ml.
- Para la extracción de carotenoides, siga los pasos 2.2 a 2.12.
4. Purificación de Cuerpos de Inclusión
- Clonar la secuencia de codificación de la proteína LHC de interés en un vector de expresión que da como resultado una C-terminal de His fusionada usando procedimientos estándar de biología molecular. Transformar este constructo en E. coli cepa huésped tal como BL21 (DE3).
- Preparar tampón de lisis, tampón detergente, tampón Tritón, TE (Tabla 1), 1 M isopropílico β-D-1-tiogalactopiranósido (IPTG) y medio LB 18 con los antibióticos apropiados.
- Elija una single E. colonia coli que contiene el clon de expresión de una placa recién estriada en ~ 5 ml de medio LB con los antibióticos apropiados utilizando procedimientos estándar 6. Crecer a 37 ° C con agitación 220 rpm durante al menos 16 h.
- Añadir 2,5 ml de la O / N de cultivo en un matraz Erlenmeyer de 1 L con 250 ml de LB suplementado con el antibiótico apropiado.
- Cultivar las células durante 2-3 horas (o hasta que la DO 600 es ~ 0,6) a 37 ° C a 220 rpm.
- Añadir IPTG a una concentración final de 1 mM. Continuar creciendo las células a 37 ° C con 220 rpm 04.03 h.
- Centrifugar la cultura durante 10 minutos a 5000 xga 4 ° C en un tubo de centrífuga previamente pesada. Descartar el sobrenadante a fondo y determinar el peso del pellet pesando de nuevo y restando el peso del tubo de centrífuga.
- Resuspender el E. sedimento celular coli en 0,8 ml / g de tampón de lisis por agitación vigorosa.
NOTA: Alternativamente, el sedimento de células se puede congelar a -80C para su uso posterior. Si se comienza con una bolita de helado, dejar descongelar completamente antes de añadir el tampón de lisis. - Añadir 2 mg de lisozima por gramo de células, e incubar en hielo húmedo con agitación en vórtex ocasional durante 30 min.
- Añadir 20 mg / ml de DNAsa, 10 mM de MgCl 2, NaCl 1 mM, 20 mg / ml de ARNasa. Vortex y poner en hielo durante 30 min.
- Añadir 2 ml de tampón de detergente frío por gramo de células. Mezclar bien y mantener la temperatura ambiente durante 5 min.
- Transferir a tubos de centrífuga de 2 ml (dividida en dos tubos si es necesario). Centrifugar durante 10 min a 12.000 xg a 4 ° C para sedimentar los cuerpos de inclusión.
- Añadir 1 ml de tampón frío Triton y resuspender completamente el sedimento mediante sonicación (3 pulsos x 5 seg x 50% de potencia con intervalos de 20 seg). NOTA: Tenga el tubo en un vaso de precipitados pequeño rodeado de agua con hielo para mantenerlo frío durante el tratamiento con ultrasonidos. En el caso de múltiples tubos, combinar los cuerpos de inclusión se volvieron a suspender en un tubo después de la resuspensión.
- Centrifugar durante 10 min a 12.000xga 4 ° C para que sedimenten los cuerpos de inclusión.
- Repita el paso 4.13 y 4.14 dos veces.
- Resuspender los cuerpos de inclusión en 1 ml de TE frío con sonificación para un lavado final para eliminar el tampón Triton. Centrifugar durante 10 min a 12.000 xg a 4 ° C para sedimentar los cuerpos de inclusión.
- Resuspender el precipitado en 1 ml de TE frío por sonificación.
- Evaluar la concentración de proteínas por métodos estándar tales como el ensayo de Bradford 19. Tienda alícuotas de los cuerpos de inclusión a -20 ° C.
5. Reconstitución
Este protocolo produce típicamente 1-2 ml de la proteína reconstituida con un diámetro exterior de 4 cuando se mide la absorbancia en la región Qy (600-750 nm). La cantidad puede ajustarse según se desee, aunque se debe tener cuidado para mantener las proporciones adecuadas durante el procedimiento.
- Preparar las siguientes soluciones que se describen en la Tabla 1: 2x tampón de reconstitución, el 20% OG, 2 M KCl, TE. Realice el siguiente steps con poca luz.
- Resuspender 800 g de cuerpos de inclusión del LHC en un total de 400 l TE en un tubo de microcentrífuga de 2 ml. Añadir 400 l de la 2x tampón de reconstitución y agitar brevemente.
- Añadir 0,6 l de β-mercaptoetanol (14,8 M de stock) para tener una concentración final de 10 mM. Calentar la proteína durante 1 min a 98 ° C. Brevemente Vortex y lugar a TA durante 3 min.
- Resuspender 500 mg del total de pigmentos de clorofila secos más 80 mg pigmentos carotenoides en 30 l 100% EtOH por agitación vigorosa durante 1 min o lugar en un baño de ultrasonido durante 1-2 minutos.
- Gira la mezcla de pigmento y 30 segundos a 15.800 xga 4 ° C y confirmar que no hay pellet. Si hay un pellet, repetir vórtex y / o sonicación. IMPORTANTE: Después de la resuspensión y centrifugado, añadir inmediatamente pigmento a la proteína, o puede agregar y tendrá que volver a suspenderse de nuevo.
- Poco a poco agregue la mezcla de pigmentos a la proteína enfriado mientras vortex. Continúe vórtice 5-10 sec y colocar el tubo en hielo húmedo. Tenga cuidado de no agite con demasiada fuerza ya que la proteína puede desbordar la parte superior del tubo.
- Añadir 94 l de 20% de octilo β-D-glucopiranósido (OG) (concentración final 2%), vórtice brevemente y mantener en hielo 10 min.
- Añadir 90 l de KCl 2 M (concentración final 150 a 200 mM), agitar brevemente y mantener en hielo 20 min. NOTA: preparación de la columna (Sección 6) se pueden iniciar en este momento.
- Girar durante 10 minutos a 15.800 xga 4 ° C. Eliminar el sobrenadante sin perturbar el sedimento (precipitado LDS) a un tubo de 10 ml. Mantener frío y protegido de la luz.
6. columna de purificación de níquel
- Preparar las siguientes soluciones según se describe en la Tabla 1: Tampón de OG, tampón de enjuague OG, tampón de elución.
- Conectar una columna de Ni-Sepharose (1 ml) o equivalente a una bomba peristáltica asegurar que el aire no se mete dentro de la columna durante este paso y los pasos siguientes.
- Ajuste la velocidad de tque la bomba de 1 ml / min y enjuagar la columna con 5-10 ml de agua para eliminar la solución de almacenamiento.
- Equilibrar la columna con 3-4 ml de tampón de OG.
- Añadir 3-4 ml de tampón de OG a la muestra de proteína y la carga a la columna. NOTA: si la proteína se ha sentado en el hielo durante más de 10 minutos después de la eliminación de LDS, girar de nuevo a 15.800 xga 4 ° C durante 1 min para eliminar cualquier precipitación LDS adicional.
- Lavar la columna con 5 ml de tampón de OG.
- Lavar la columna con 2 ml de tampón de enjuague OG.
- Se eluye la proteína unida con 3 ml de tampón de elución. Recoger el eluido verde que contiene la proteína reconstituida. NOTA: Esto es generalmente cerca de 1 ml en total.
7. centrifugación en gradiente de sacarosa
- Preparar las siguientes soluciones según se describe en la Tabla 1: Solución de sacarosa, 0,06% β-DM, HEPES 0,01 M, pH 7,6.
- Llenar los tubos de ultracentrífuga con la solución de sacarosa y la congelación a -20 ° CO / N or -80 ° C durante al menos 1 hora.
- Retirar el tubo del congelador y dejar descongelar imperturbable a 4 ° C. NOTA: El proceso de congelación / descongelación crea un gradiente de 0,1 a 1 M de sacarosa. Un tubo de 15 ml se descongela típicamente en aproximadamente 3 h.
- Retire cuidadosamente de la parte superior el mismo volumen que la fracción verde se eluyó de la columna de Sepharose de níquel en el paso 6.8. A continuación, cargue la muestra reconstituida lentamente en la parte superior para evitar molestar a la pendiente.
- Se equilibran los tubos y centrifugado a 200.000 xg a 4 ° C en una ultracentrífuga utilizando un rotor de cubeta SW-41 o SW-60 oscilante durante 18 horas, ajuste para frenar la aceleración y detención sin frenos.
- Retire cuidadosamente el gradiente desde el soporte del tubo con unas pinzas. Use una jeringa con una aguja larga que tiene una abertura romo para recoger la fracción de la parte superior. NOTA: Alternativamente, recoger las fracciones de la parte inferior perforando el tubo con una aguja y la recolección de gotas.
Resultados
Este protocolo detalla un método para reconstituir clorofila a / b proteínas de unión in vitro. Esta técnica permite el plegado de estos complejos pigmento-proteína in vitro a partir de la apoproteína, que se puede obtener por la sobreexpresión en un sistema heterólogo, y pigmentos extraídos de plantas o algas. Después de la reconstitución, el complejo proteína-pigmento replegada se purifica del exceso de pigmentos y la apoproteína desplegada en dos pasos. El primer paso (Figura 1 AB) se basa en la presencia de His-tag en el extremo C-terminal de la proteína, que permite la eliminación de gran parte de los pigmentos no unidos. La segunda etapa de purificación utiliza la centrifugación de gradiente de densidad de sacarosa, (Figura 2) cuando la proteína desplegada generalmente migra más lento que la banda verde que contiene la proteína reconstituida. El objetivo de la reconstitución in vitro es la obtención de complejos con el mismo adecuadalazos como los nativos. Para ilustrar este resultado, las propiedades espectroscópicas de un in vivo complejos captadores de luz se compara con el mismo complejo LHC reconstituido in vitro 13,20,21. El espectro de absorción de la caché de host local en el rango visible (350 nm y 750 nm) depende de la composición de pigmento del complejo, así como en el entorno del pigmento (que incluye la proteína) y es por lo tanto una herramienta sensible para comprobar la calidad de la reconstitución. En la Figura 3, el espectro de absorción de CP24, la clorofila a / b de proteína de Arabidopsis thaliana, reconstituido in vitro de unión, se compara con el espectro de la misma complejo purificado de Arabidopsis tilacoides 21. En los espectros, es posible reconocer el Qy y la transición Soret de Chl a (picos a 671/439 nm) y Chl b (picos a 649/466 nm). Los complejos nativos y reconstituidas muestran abso idénticarption espectros, lo que indica una composición de pigmento prácticamente idénticos y organización. La espectroscopia de fluorescencia se puede utilizar para evaluar la calidad del complejo reconstituido. Los espectros de emisión de fluorescencia se mide después de la excitación a diferentes longitudes de onda, que excitan preferentemente diferentes pigmentos: Chl a a 440 nm, b Chl a 475 nm, y xantofilas a 500 nm. En un complejo de proteína-pigmento plegada apropiadamente, Chl b y xantofilas transfieren su energía de excitación principalmente a Chl a dentro de unos pocos picosegundos, y la fluorescencia se origina a partir de un sistema térmicamente equilibrado resulta en un único pico con la misma forma y en los tres máximos de excitación longitudes de onda (Figura 4A-B). La presencia de Chl b no coordinado a la proteína puede ser reconocido por un pico adicional o el hombro alrededor de 650 nm a 475 nm de excitación (Figura 4C). La presencia de la libre Chl a en vez conducea la emisión adicional alrededor de 675 nm, que está presente principalmente en 440 nm de excitación. Los espectros de emisión de fluorescencia a 475 nm de excitación de ambos reconstituido y los complejos CP24 nativas (Figura 4D) muestra un solo pico a 681 nm, lo que indica que complejo reconstituido se pliega correctamente. Una confirmación adicional de que el complejo proteína-pigmento se reconstituyó correctamente viene de dicroísmo circular (CD) mediciones. La señal de CD en la región visible depende de las interacciones entre excitónicos pigmentos y es por lo tanto muy sensibles a incluso pequeños cambios en la organización de los cromóforos 22. Figura 5 muestra los espectros de CD de reconstituido y nativa CP24, con los picos de huellas dactilares típicas en 681 nm, 650 nm y 481 nm. En conclusión, la gran similitud entre las propiedades espectroscópicas de nativos y el CP24 reconstituido confirma que los complejos de los rendimientos del procedimiento de reconstitución nativo-como suita ble para el estudio in vitro de proteínas captadores de luz.
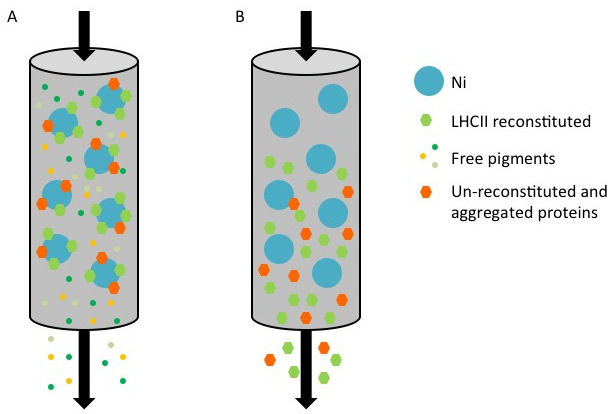
Figura 1 Representación de la purificación de proteínas recombinantes del LHC con una etiqueta His usando una columna de níquel. (A) Durante la purificación, Su-etiquetados proteína, compuesta de ambos complejos reconstituidas (hexágono) y proteína-un reconstituido / agregados (naranja hexágono) se une a la superficie de la Ni-Sepharose (punto azul), mientras que los pigmentos no consolidados (puntos de colores) fluyen a través. (B) Cuando la columna se lava con el tampón de elución que contiene imidazol, las proteínas reconstituidos y un-reconstituida se recogen en el flujo a través.
hres.jpg "width =" 500 "/>
Figura 2. gradiente de sacarosa de LHCII reconstituido después de la purificación por columna de níquel. Los complejos reconstituidos se separan del pigmento libre por el gradiente de densidad. La banda verde oscuro representa LHCII reconstituida y el fondo de color verde pálido se compone de pigmentos libres.
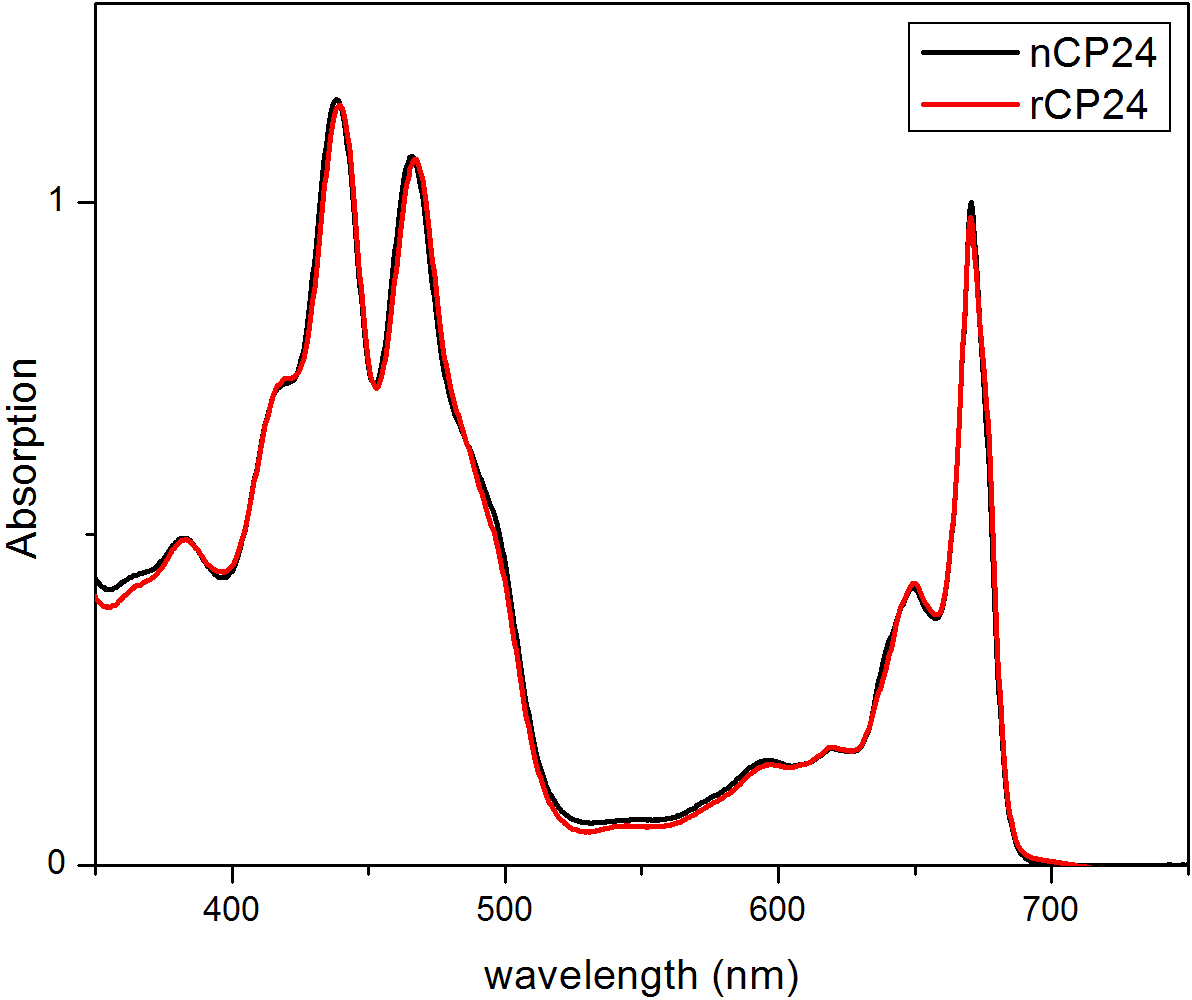
Figura 3. espectros de absorción de la proteína reconstituida CP24 (rCP24, línea roja) y la nativa (nCP24, línea de color negro) aislado de Arabidopsis thaliana. En ambos espectros, es posible reconocer el Qy y la transición Soret de Chl a (picos a 671/439 nm) y Chl b (picos a 649/466 nm). Esta cifra ha sido modificado desde Passarini et al. 2014 21.
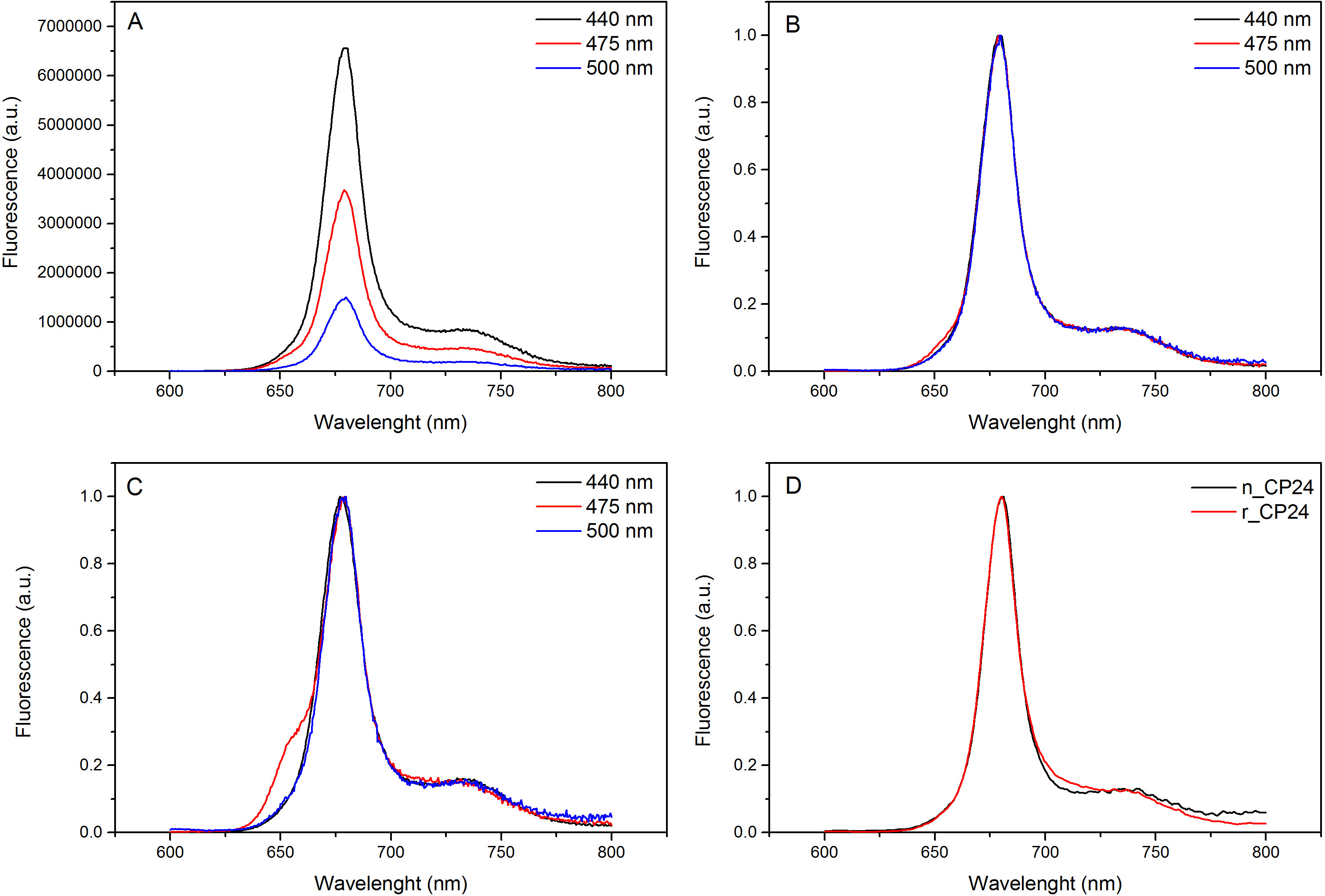
Figura 4. espectros de emisión de fluorescencia. Los espectros de emisión de fluorescencia del complejo reconstituido CP24 de tipo salvaje (A) y se normalizaron al máximo (B) muestra la transferencia de energía eficiente desde Chl b y Xanthophyls a Chl a. (C) Fluorescencia espectros de emisión de CP24 reconstituido (rCP24) y el complejo nativo (nCP24) aislado de Arabidopsis thaliana. Los espectros están normalizados al máximo del pico (D). Por favor, haga clic aquí para ver una versión más grande de esta figura.
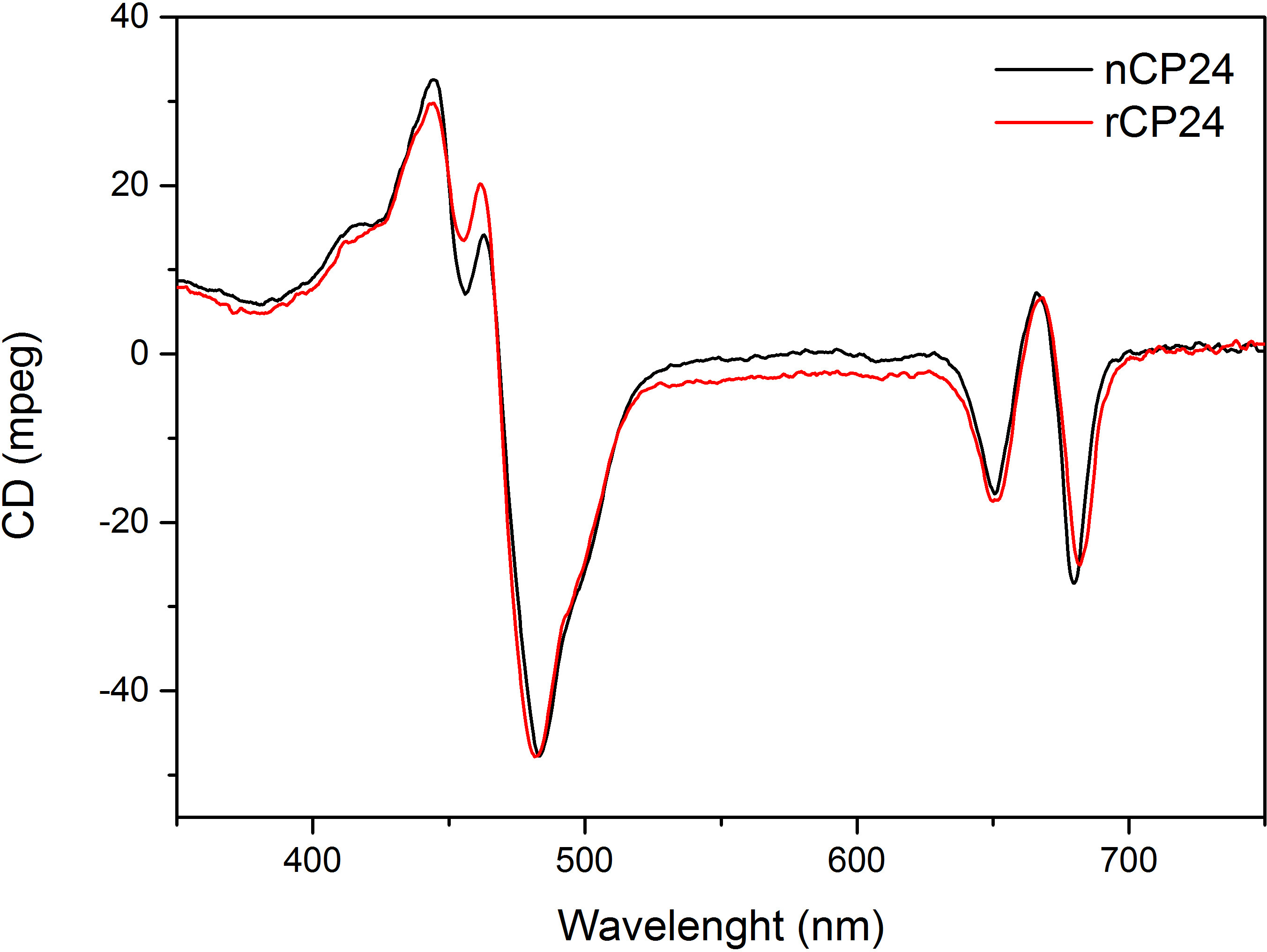
Figura 5. dicroísmo circular Spectra. Reconstituido CP24 (rCP24, línea roja) y el nativo de compleja (nCP24, línea de negro) aisladas de Arabidopsis thaliana muestra espectros muy similares.

Figura 6. Espectros de absorción CP29 tipo salvaje (CP29_WT) y mutado CP29 (CP29_A2). La línea verde muestra las diferencias entre las dos parcelas.
| Todos los tampones se pueden almacenar a 4 ° C. | |||
| Componentes | Concentración final | Notas adicionales | |
| Tampón de molienda | Sorbitol | 0,4 M | |
| Tricine | 0,1 M | pH 7,8 | |
| NaCl | 10 mM | ||
| MgCl2 | 5 mM | ||
| Leche en Polvo | 0,5% w / v | ||
| Tampón de lavado | Sorbitol | 50 mM | |
| Tricine | 5 mM | pH 7,8 | |
| EDTA | 10 mM | pH 8 | |
| Lysis Buffer | Tris | 50 mM | pH 8 |
| La sacarosa | 2,5% w / v | ||
| EDTA | 1 mM | pH 8 | |
| Tampón detergente | NaCl | NaCl 200 mM | |
| Ácido desoxicólico | 1% w / v | ||
| NONIDET P-40 | 1% w / v | ||
| Tris | 20 mM | pH 7,5 | |
| EDTA | 2 mM | pH 8 | |
| beta-mercaptoetanol | 10 mM | ||
| Triton Buffer | Triton X-100 | 0,5% w / v | |
| Tris | 20 mM | pH 7,5 | |
| beta-mercaptoetanol | 1 mM | ||
| Tampón TE | Tris | 50 mM | pH 8 |
| EDTA | 1 mM | pH 8 | |
| Tampón de reconstitución | HEPES | 200 mM | |
| La sacarosa | 5% w / v | ||
| Lithiumdodecylsulfate (LDS) | 4% w / v | ||
| Benzamidina | 2 mM | ||
| Ácido aminocaproico | 10 mM | ||
| OG Buffer | Octilglucósido | 1% w / v | |
| 12.5% w / v | |||
| NaCl | 0,2 M | ||
| HEPES | 20 mM | ||
| Imidazole | 10 mM | ||
| OG Rinse Buffer | n-dodecil-beta-D-Maltósido (β-DM) | 0.06% w / v | |
| HEPES | 40 mM | pH 7.5-9 | |
| NaCl | 0,2 M | ||
| Tampón de elución | Imidazole | 0,5 M | |
| n-dodecil-beta-D-Maltósido (β-DM) | 0.06% w / v | ||
| HEPES | 40 mM | pH 8 | |
| NaCl | 0,2 M | ||
| Solución de sucrosa | La sacarosa | 20% w / v | |
| n-dodecil-beta-D-Maltósido (β-DM) | 0.06% w / v | ||
| HEPES | 0,01 M | pH 7,6 | |
| Acetona 80% tamponado con carbonato de sodio | Acetona | 80% v / v | |
| Carbonato de Sodio | 1 M | ||
| Etanol al 96% tamponado con carbonato de sodio | Etanol | 96% v / v | |
| Carbonato de Sodio | 1 M | ||
Cuadro 1 Lista de tampones y soluciones utilizadas en este protocolo.
rCP26| Cla una mezcla b / | Cla a / b | Chl a | Chl b | Neo | Viola | Lute | Chl tot | Chl / Car | |
| nCP26 | - | 2,2 ± 0,05 | 6.2 | 2.8 | 0.61 | 0.38 | 1.02 | 9 | 4,5 ± 0,1 |
| rCP26 | 8 | 2,71 ± 0,05 | 6.57 | 2.43 | 0,72 | 0.32 | 0.97 | 3,9 ± 0,04 | |
| rCP26 | 5.5 | 2,25 ± 0,05 | 6.23 | 2.77 | 0,77 | 0.3 | 0.96 | 9 | 4,0 ± 0,1 |
| rCP26 | 3 | 2,08 ± 0,04 | 6.08 | 2.92 | 0,76 | 0.3 | 1.04 | 9 | 4,1 ± 0,1 |
| rCP26 | 1 | 1,7 ± 0,05 | 5,7 | 3.3 | 0,7 | 0.3 | 0.9 | 9 | 4,3 ± 0,05 |
| rCP26 | 0.3 | 1,11 ± 0,04 | 4.7 | 4.28 | 0,7 | 0.3 | 0.9 | 9 | 4,2 ± 0,2 |
| 0.05 | 0,23 ± 0,01 | 1.4 | 5.6 | 0.58 | 0.24 | 1.11 | 7 | 3,1 ± 0,06 | |
| rCP26 | <0,01 | 0,11 ± 0,01 | 0,7 | 6,3 | 0.64 | 0.3 | 1.08 | 7 | 3,06 ± 0,06 |
Tabla 2. contenido de pigmento de CP26 nativa complejo en comparación con los complejos de proteína-pigmento reconstituidas con diferentes Chl a / b Ratios 39.
Discusión
Las proteínas de membrana no son tan fáciles de estudiar. Aislamiento de proteínas de membrana nativas se complica por la necesidad de solubilizar la bicapa lipídica con detergentes, que puede dañar la proteína y eliminar cofactores esenciales. Estas proteínas también pueden estar presentes en niveles bajos en las membranas biológicas, o ser mezclado con proteínas estrechamente relacionadas, como en el caso de los complejos de recolección de luz, que hace que la purificación de complejos individuales difíciles. La expresión de proteínas heterólogas en E. coli y en la reconstitución in vitro ofrece la posibilidad de evitar estos problemas. In vitro reconstitución y purificación de proteínas plegadas resultados en los complejos que poseen características muy similares a las de los complejos nativos 20,21,23 y por lo tanto puede ser utilizado para estudiar complejos que no puede ser purificado hasta homogeneidad 24 - 27.
Este método utiliza la espinaca, que es fácilmente attainabLe año, como una fuente para las preparaciones de pigmento total y carotenoides. Para algunas reconstituciones de proteínas nativas a las algas, se prefiere el uso de pigmentos purificados a partir de algas debido a las diferentes composiciones de pigmentos. La proporción Chl / b y la relación / Chl coche sigue siendo el mismo, independientemente de la fuente de pigmento.
Es importante darse cuenta de que la eficiencia de la reconstitución es por lo general alrededor del 35% 28. Por lo tanto, es necesario eliminar los pigmentos no unido y la apoproteína desplegada de la solución después de la reconstitución. Un protocolo de purificación de dos etapas se presenta en este protocolo (ver también los resultados). Sin embargo, debe observarse que la etapa de gradiente de sacarosa no permite la separación completa de apo y holo-proteína. Para la mayoría de los análisis esto no es un problema, ya que la apoproteína no contiene pigmentos y por lo tanto no interfiere con las mediciones funcionales. Sin embargo, en caso de que sea necesario para eliminar completamente la apoproteína de la fracción que contiene el complejo reconstituido (por ejemplo, para calcular el pigmento a la estequiometría de proteína), una columna de intercambio aniónico se puede utilizar (ver Passarini et al. 2.009 29 para más detalles).
La capacidad se repliegue proteínas recombinantes de recolección de luz con pigmentos aislados in vitro ofrece una oportunidad de "manipular" los complejos mediante la modificación de la reconstitución "medio ambiente" de varias maneras, cambiando así las características del complejo resultante. Por ejemplo, el cambio de la composición de pigmento durante la reconstitución puede resultar en un complejo con composición de pigmento alterada. Esta característica puede ser utilizada para estudiar la influencia diversos pigmentos tienen en la estructura y la estabilidad del complejo. Por lo general, la preparación de pigmento obtenido a partir de la espinaca tiene una relación a / b Chl de 3: 1 y una relación de / coche Chl de 2,9: 1. Esta proporción típicamente produce una proteína reconstituida con las mismas propiedades que el nAtive uno. Sin embargo, el ajuste de la relación a / b Chl por la adición de Chl purificado A o B puede influir en la unión de diferentes pigmentos debido a la variación de selectividad de los sitios de unión 30 a 33. Esto es posible porque la mayoría de los sitios de unión de pigmentos no son completamente selectivo para Chl una o Chl b, pero puede acomodar tanto, aunque con diferente afinidad 10,30,34. De manera similar, los sitios de unión de carotenoides también mostraron ser capaces de acomodar más de una especie de xantofila 8,35 - 38. Diferentes reconstituciones de CP26, otro complejo proteína-pigmento de las plantas superiores, utilizando diversas composiciones de pigmento se muestran en la Tabla 2 39. Se utilizaron estas reconstituciones para evaluar la afinidad de los sitios de unión particulares pigmentos 39. Es interesante observar que con el fin de obtener un complejo con el mismo pigmento cOMPOSICIÓN como el nativo, la Chl relación a / b de la mezcla de pigmento debe ser 3: 1. Este parece ser el caso para todos los complejos LHC de plantas superiores 20,40.
La combinación de la biología molecular con la técnica de la reconstitución permite que las propiedades de un complejo Chl-unión a ser estudiados en más detalle. La importancia de los diferentes dominios de la proteína en la estabilidad y el plegamiento de los complejos, o su participación en las interacciones proteína-proteína, se han determinado truncando la apoproteína o realizar mutagénesis aleatoria 8,41 - 44. Residuos de aminoácidos Individual importantes para la coordinación de los diferentes pigmentos se pueden modificar a través de mutagénesis dirigida al sitio con el fin de analizar las propiedades de los pigmentos individuales o evaluar su contribución a la función y la estabilidad del complejo 10,28,29,45 - 52. La figura 6 muestra reconstituyeron Lhcb4 (CP29) conuna mutación de la histidina en la posición 216 53. Una comparación de la composición de pigmento de tipo salvaje y mutantes complejos muestra que la mutación induce la pérdida de una molécula de Chl a, indicando que el sitio diana acomoda una un Chl en el complejo WT. Las diferencias de los espectros de absorción de WT y mutante, en la normalización de el contenido de pigmento, también muestra las propiedades de absorción del pigmento perdido. En este caso, la diferencia se puede ver en el pico principal a 680 nm, lo que indica que la Chl a coordinado por His216 absorbe a esta longitud de onda (para más detalles sobre este mutante y las propiedades espectroscópicas ver Mozzo et al. 2008 53). Análisis de la mutación también se puede utilizar para determinar el efecto del medio ambiente sobre las propiedades espectroscópicas de los pigmentos 54.
En conclusión, las proteínas de recolección de luz fácilmente se pueden reconstituir in vitro resulta en pigmento protein complejos con propiedades muy similares a los complejos nativas. De esta manera, se eliminan las dificultades de aislar proteínas nativas, mientras que también la entrega de preparación de proteína con alto rendimiento y pureza en estudio. La importancia de una proporción de 3: a / b 1 Chl en la producción de un complejo auténtico se acentúa, y los ejemplos de tipo salvaje reconstituido y cachés de host local mutantes se proporcionan para ilustrar aplicaciones de la técnica.
Divulgaciones
The authors declare no conflict of interest
Agradecimientos
This work was supported by the European research council by a ERC starting/consolidator grant to RC and by the Dutch Foundation for research on matter (FOM) via a FOM program (10TM01).
Materiales
| Name | Company | Catalog Number | Comments |
| HisTrap HP | GE Healthcare | 17-5247-01 | |
| Nylon cloth | 20 μm pores | ||
| Soft artists paint brush | |||
| NONIDET P-40 | Sigma | 74385 | |
| Beta-DM | Sigma | D4641 | |
| DNAase | ThermoScientific | EN0525 | |
| Milk Powders | |||
| RNAase | ThermoScientific | EN0531 | |
| Sonicator | |||
| Octyl β-D-glucopyranoside | Sigma | O8001 | |
| Ultracentrifuge XL | Beckman-Coulter | ||
| TAP medium | see reference 17 | ||
| LB medium | see reference 19 |
Referencias
- Plumley, F. G., Schmidt, G. W. Reconstitution of chlorophyll a/b light-harvesting complexes: Xanthophyll-dependent assembly and energy transfer. Proceedings of the National Academy of Sciences of the United States of America. 84, 146-150 (1987).
- Croce, R., van Amerongen, H. Light-harvesting and structural organization of Photosystem II from individual complexes to thylakoid membrane. Journal of photochemistry and photobiology B Biology. 104 (1-2), 142-153 (2011).
- Li, Z., Wakao, S., Fischer, B. B., Niyogi, K. K. Sensing and responding to excess light. Annual review of plant biology. 60, 239-260 (2009).
- De Bianchi, S., Ballottari, M., Dall’osto, L., Bassi, R. Regulation of plant light harvesting by thermal dissipation of excess energy. Biochemical Society transactions. 38 (2), 651-660 (2010).
- Neilson, J. A. D., Durnford, D. G. Structural and functional diversification of the light-harvesting complexes in photosynthetic eukaryotes. Photosynthesis research. 106 (1-2), 57-71 (2010).
- Paulsen, H., Rümler, U., Rüdiger, W. Reconstitution of pigment-containing complexes from light-harvesting chlorophyll a/b-binding protein overexpressed in Escherichia coli. Planta. 181 (2), 204-211 (1990).
- Horn, R., Grundmann, G., Paulsen, H. Consecutive binding of chlorophylls a and b during the assembly in vitro of light-harvesting chlorophyll-a/b protein (LHCIIb). Journal of molecular biology. 366 (3), 1045-1054 (2007).
- Cammarata, K. V., Schmidt, G. W. In vitro reconstitution of a light-harvesting gene product: deletion mutagenesis and analyses of pigment binding. Biochemistry. 31 (10), 2779-2789 (1992).
- Paulsen, H., Hobe, S. Pigment-binding properties of mutant light-harvesting chlorophyll-a/b-binding protein. European journal of biochemistry / FEBS. 205 (1), 71-76 (1992).
- Bassi, R., Croce, R., Cugini, D., Sandonà, D. Mutational analysis of a higher plant antenna protein provides identification of chromophores bound into multiple sites. Proceedings of the National Academy of Sciences of the United States of America. 96 (18), 10056-10061 (1999).
- Paulsen, H., Finkenzeller, B., Kühlein, N. Pigments induce folding of light-harvesting chlorophyll a/b-binding protein. European journal of biochemistry / FEBS. 215 (3), 809-816 (1993).
- Caffarri, S., Croce, R., Cattivelli, L., Bassi, R. A look within LHCII differential analysis of the Lhcb1-3 complexes building the major trimeric antenna complex of higher-plant photosynthesis. Biochemistry. 43 (29), 9467-9476 (2004).
- Giuffra, E., Cugini, D., Croce, R., Bassi, R. Reconstitution and pigment-binding properties of recombinant CP29. European journal of biochemistry / FEBS. 238 (1), 112-120 (1996).
- Rogl, H., Kosemund, K., Kühlbrandt, W., Collinson, I. Refolding of Escherichia coli produced membrane protein inclusion bodies immobilised by nickel chelating chromatography. FEBS letters. (1-2), 21-26 (1998).
- Porra, R. J., Thompson, W. A., Kriedemann, P. E. Determination of accurate extinction coefficients and simultaneous equations for assaying chlorophylls a and b extracted with four different solvents: verification of the concentration of chlorophyll standards by atomic absorption spectroscopy. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 975 (3), 384-394 (1989).
- Davies, B. H. Identification of carotenoids by their absorption characteristics. Biochem J. 103 (2), (1967).
- Gorman, D. S., Levine, R. P. Cytochrome f and plastocyanin their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. Proceedings of the National Academy of Sciences of the United States of America. 54 (6), 1665-1669 (1965).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of bacteriology. 62 (3), 293-300 (1951).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical biochemistry. 72, 248-254 (1976).
- Wientjes, E., Croce, R. The light-harvesting complexes of higher-plant Photosystem I Lhca1/4 and Lhca2/3 form two red-emitting heterodimers. The Biochemical journal. 433 (3), 477-485 (2011).
- Passarini, F., Xu, P., Caffarri, S., Hille, J., Croce, R. Towards in vivo mutation analysis knock-out of specific chlorophylls bound to the light-harvesting complexes of Arabidopsis thaliana - the case of CP24 (Lhcb6). Biochimica et biophysica acta. , (2014).
- Georgakopoulou, S., van der Zwan, G., Bassi, R., van Grondelle, R., van Amerongen, H., Croce, R. Understanding the changes in the circular dichroism of light harvesting complex II upon varying its pigment composition and organization. Biochemistry. 46 (16), 4745-4754 (2007).
- Croce, R., Müller, M. G., Caffarri, S., Bassi, R., Holzwarth, A. R. Energy transfer pathways in the minor antenna complex CP29 of photosystem II a femtosecond study of carotenoid to chlorophyll transfer on mutant and WT complexes. Biophysical journal. 84 (4), 2517-2532 (2003).
- Schmid, V. H., Cammarata, K. V., Bruns, B. U., Schmidt, G. W. In vitro reconstitution of the photosystem I light-harvesting complex LHCI-730 heterodimerization is required for antenna pigment organization. Proceedings of the National Academy of Sciences of the United States of America. 94 (14), 7667-7672 (1997).
- Castelletti, S., Morosinotto, T., Robert, B., Caffarri, S., Bassi, R., Croce, R. Recombinant Lhca2 and Lhca3 subunits of the photosystem I antenna system. Biochemistry. 42 (14), 4226-4234 (2003).
- Storf, S., Jansson, S., Schmid, V. H. R. Pigment binding, fluorescence properties, and oligomerization behavior of Lhca5, a novel light-harvesting protein. The Journal of biological chemistry. 280 (7), 5163-5168 (2005).
- Mozzo, M., Mantelli, M., Passarini, F., Caffarri, S., Croce, R., Bassi, R. Functional analysis of photosystem I light-harvesting complexes (Lhca) gene products of Chlamydomonas reinhardtii. Biochimica et biophysica acta. 1797 (2), 212-221 (2010).
- Remelli, R., Varotto, C., Sandonà, D., Croce, R., Bassi, R. Chlorophyll binding to monomeric light-harvesting complex. A mutation analysis of chromophore-binding residues. The Journal of biological chemistry. 274 (47), 33510-33521 (1999).
- Passarini, F., Wientjes, E., Hienerwadel, R., Croce, R. Molecular basis of light harvesting and photoprotection in CP24 unique features of the most recent antenna complex. The Journal of biological chemistry. 284 (43), 29536-29546 (2009).
- Giuffra, E., et al. Analysis of some optical properties of a native and reconstituted photosystem II antenna complex, CP29 pigment binding sites can be occupied by chlorophyll a or chlorophyll b and determine spectral forms. Biochemistry. 36 (42), 12984-12993 (1997).
- Pagano, A., Cinque, G., Bassi, R. In vitro reconstitution of the recombinant photosystem II light-harvesting complex CP24 and its spectroscopic characterization. The Journal of biological chemistry. 273 (27), 17154-17165 (1998).
- Kleima, F. J., et al. Decreasing the chlorophyll a/b ratio in reconstituted LHCII structural and functional consequences. Biochemistry. 38 (20), 6587-6596 (1999).
- Croce, R., Morosinotto, T., Castelletti, S., Breton, J., Bassi, R. The Lhca antenna complexes of higher plants photosystem I. Biochimica et biophysica acta. 1556 (1), 29-40 (2002).
- Hobe, S., Trostmann, I., Raunser, S., Paulsen, H. Assembly of the major light-harvesting chlorophyll-a/b complex Thermodynamics and kinetics of neoxanthin binding. The Journal of biological chemistry. 281 (35), 25156-25166 (2006).
- Croce, R., Weiss, S., Bassi, R. Carotenoid-binding sites of the major light-harvesting complex II of higher plants. The Journal of biological chemistry. 274 (42), 29613-29623 (1999).
- Hobe, S., Niemeier, H., Bender, A., Paulsen, H. Carotenoid binding sites in LHCIIb. Relative affinities towards major xanthophylls of higher plants. European journal of biochemistry / FEBS. 267 (2), 616-624 (2000).
- Jahns, P., Depka, B., Trebst, A. Xanthophyll cycle mutants from Chlamydomonas reinhardtii indicate a role for zeaxanthin in the D1 protein turnover. Plant Physiology and Biochemistry. 38 (5), 371-376 (2000).
- Wehner, A., Grasses, T., Jahns, P. De-epoxidation of violaxanthin in the minor antenna proteins of photosystemII, LHCB4, LHCB5, and LHCB6. The Journal of biological chemistry. 281 (31), 21924-21933 (2006).
- Croce, R., Canino, G., Ros, F., Bassi, R. Chromophore organization in the higher-plant photosystem II antenna protein CP26. Biochemistry. 41 (23), 7334-7343 (2002).
- Caffarri, S., Passarini, F., Bassi, R., Croce, R. A specific binding site for neoxanthin in the monomeric antenna proteins CP26 and CP29 of Photosystem II. FEBS letters. 581 (24), 4704-4710 (2007).
- Hobe, S., Förster, R., Klingler, J., Paulsen, H. N-proximal sequence motif in light-harvesting chlorophyll a/b-binding protein is essential for the trimerization of light-harvesting chlorophyll a/b complex. Biochemistry. 34 (32), 10224-10228 (1995).
- Kuttkat, A., Hartmann, A., Hobe, S., Paulsen, H. The C-terminal domain of light-harvesting chlorophyll-a/b-binding protein is involved in the stabilisation of trimeric light-harvesting complex. European journal of biochemistry / FEBS. 242 (2), 288-292 (1996).
- Rupprecht, J., Paulsen, H., Schmid, V. H. Protein domains required for formation of stable monomeric Lhca1- and Lhca4-complexes. Photosynthesis research. 63 (3), 217-224 (2000).
- Yang, C., et al. The negatively charged amino acids in the lumenal loop influence the pigment binding and conformation of the major light-harvesting chlorophyll a/b complex of photosystem II. Biochimica et biophysica acta. 1777 (11), 1463-1470 (2008).
- Rogl, H., Kühlbrandt, W. Mutant trimers of light-harvesting complex II exhibit altered pigment content and spectroscopic features. Biochemistry. 38 (49), 16214-16222 (1999).
- Yang, C., Kosemund, K., Cornet, C., Paulsen, H. Exchange of pigment-binding amino acids in light-harvesting chlorophyll a/b protein. Biochemistry. 38 (49), 16205-16213 (1999).
- Morosinotto, T., Castelletti, S., Breton, J., Bassi, R., Croce, R. Mutation analysis of Lhca1 antenna complex. Low energy absorption forms originate from pigment-pigment interactions. The Journal of biological chemistry. 277 (39), 36253-36261 (2002).
- Morosinotto, T., Breton, J., Bassi, R., Croce, R. The nature of a chlorophyll ligand in Lhca proteins determines the far red fluorescence emission typical of photosystem I. The Journal of biological chemistry. 278 (49), 49223-49229 (2003).
- Ballottari, M., Mozzo, M., Croce, R., Morosinotto, T., Bassi, R. Occupancy and functional architecture of the pigment binding sites of photosystem II antenna complex Lhcb5. The Journal of biological chemistry. 284 (12), 8103-8113 (2009).
- Croce, R., et al. Origin of the 701-nm fluorescence emission of the Lhca2 subunit of higher plant photosystem I. The Journal of biological chemistry. 279 (47), 48543-48549 (2004).
- Morosinotto, T., Mozzo, M., Bassi, R., Croce, R. Pigment-pigment interactions in Lhca4 antenna complex of higher plants photosystem I. The Journal of biological chemistry. 280 (21), 20612-20619 (2005).
- Mozzo, M., Morosinotto, T., Bassi, R., Croce, R. Probing the structure of Lhca3 by mutation analysis. Biochimica et biophysica acta. 1757 (12), 1607-1613 (2006).
- Mozzo, M., Passarini, F., Bassi, R., van Amerongen, H., Croce, R. Photoprotection in higher plants the putative quenching site is conserved in all outer light-harvesting complexes of photosystem II. Biochimica et biophysica acta. 1777 (10), 1263-1267 (2008).
- Wientjes, E., Roest, G., Croce, R. From red to blue to far-red in Lhca4 how does the protein modulate the spectral properties of the pigments. Biochimica et biophysica acta. 1817 (5), 711-717 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados