Method Article
In Vitro Reconstituição de Complexos de Light-colheita de plantas e algas verdes
Neste Artigo
Resumo
This protocol details the reconstitution of light-harvesting complexes in vitro. These integral membrane proteins coordinate chlorophylls and carotenoids and are responsible for harvesting light in higher plants and green algae.
Resumo
Nas plantas e algas verdes, a luz é capturado pelos complexos absorvedores da luz (CLS), uma família de proteínas de membrana integrais que coordenam clorofilas e carotenóides. In vivo, estas proteínas são dobradas com pigmentos para formar complexos que são inseridas na membrana de tilacoide do cloroplasto. A alta similaridade nas propriedades químicas e físicas dos membros da família, junto com o fato de que eles podem facilmente perder pigmentos durante o isolamento, faz sua purificação em estado nativo desafiador. Uma abordagem alternativa para a obtenção de preparações homogéneas de CLS foi desenvolvido por Plumley e Schmidt em 1987 1, que demonstraram que era possível reconstituir estes complexos in vitro, a partir de pigmentos de apoproteínas purificadas e desdobradas, resultando em complexos com propriedades muito semelhantes às do nativa complexos. Isto abriu o caminho para a utilização de proteínas recombinantes expressas bacterianas in vitro (por exemplo, locais de ligação de pigmento) ou domínio da proteína (por exemplo, a interacção proteína-proteína, dobragem). Este método foi optimizado em vários laboratórios e aplicada à maior parte dos complexos de luz-colheita. O protocolo descrito aqui detalha o método de reconstituição de complexos de luz-colheita in vitro atualmente utilizados em nosso laboratório, e exemplos que descrevem aplicações do método são fornecidos.
Introdução
O aparato fotossintético das plantas e algas incluem proteínas integrais de membrana que se ligam clorofila a (chl a), b (chl b) e carotenóides (carro). Estes complexos proteína-pigmento são ativos na captação de energia luz e transferir essa energia de excitação para os centros de reação, onde é usado para promover a separação de carga 2. Eles também estão envolvidos em mecanismos de feedback reguladoras que protegem o aparato fotossintético contra danos luz alta 3,4. Os complexos de colheita de luz (CLS) são constituídos por uma grande família de proteínas relacionadas de plantas e algas 5.
A purificação homogênea de cada membro da família tem sido complicada pelas propriedades químicas e físicas muito semelhantes dos complexos. Além disso, os procedimentos de purificação, muitas vezes resultam em perda de pigmentos ou de outros co-factores potenciais, tais como lípidos. In vitro represen reconstituiçãots um poderoso método para ultrapassar estes problemas. O LHC associada com fotossistema II (LHC-II) foi reconstituído primeiro in vitro por Plumley e Schmidt em 1987 1. Os investigadores proteína extraída delipidado e pigmentos separadamente a partir de cloroplastos de plantas e, em seguida, combinada a proteína desnaturada com calor pigmentos na presença de lítio dodecilsulfato (LDS), seguido por três ciclos de congelamento e descongelamento 1. Eles demonstraram que as propriedades espectrais dos complexos reconstituídos LHC foram muito semelhantes para os complexos purificados a partir de plantas. A facilidade de reconstituição de complexos proteína-pigmento LHC, provavelmente devido a alguma característica de auto-montagem inerente, juntamente com a dificuldade de isolar complexos purificados a partir de organismos, levou à rápida adoção do método por outros pesquisadores. A reconstituição de proteínas fotossintéticas expresso em Escherichia coli (E. coli) foi obtida por Paulsen e colegas em 1990 6. Em E.coli, as proteínas da membrana sobreexpressos são normalmente contidos em corpos de inclusão, que as respectivas instalações de purificação. A reconstituição é conseguido através de desnaturação térmica dos corpos de inclusão contendo a proteína recombinante na presença de LDS, seguido pela adição de pigmentos, que inicia o enrolamento de proteínas. Dobragem do complexo LHCII é um processo de dois passos: primeiro, a clorofila é ligado em menos de 1 min; segundo, a clorofila b é obrigado e estabilizado ao longo de vários minutos 7.
Além de proporcionar uma visão da dinâmica de dobragem, na reconstituição in vitro combinada com mutagénese dirigida para o local permitiu a identificação de aminoácidos específicos importantes para a estabilidade (por exemplo, 8,9) ou a coordenação de pigmento (por exemplo, 10). Manipulação de redobramento condições de ajustar os parâmetros tais como a composição pigmento ou detergentes também identificaram elementos critical para a dobragem adequada, tais como a exigência de Xantofilas para o complexo LHCII (por exemplo, 1,11). Além disso, a investigação das propriedades de pigmentos individuais ligados aos complexos foi possível utilizando complexos reconstituídos in vivo (por exemplo, 10).
O método descrito aqui começa com o isolamento de pigmentos (clorofilas e carotenóides) a partir de espinafres e a alga verde Chlamydomonas reinhardtii. A expressão e purificação de uma proteína a partir de E. LHC coli na forma de corpos de inclusão é então descrito, seguindo-se a reconstituição de LHC e subsequente purificação por coluna de afinidade com Ni. No último passo, os complexos reconstituídos são ainda purificado por centrifugação em gradiente de sacarose para remover pigmentos livres e apoproteína desdobrado. Este protocolo representa um procedimento otimizado incorporando várias modificações que foram introduzidas por diferentes laboratórios maistempo 1,6,10,12 -14.
Protocolo
1. Total Pigment Extração de espinafre folhas
- Homogeneizar um punhado de folhas de espinafre (~ 20 g) em 100 ml de tampão de frio Moagem (ver Tabela 1), utilizando um misturador de 20 seg.
- Filtrar a solução através de duas camadas de tecido de nylon com um diâmetro de poro de 20 ^ m e centrifuga-se o filtrado a 1500 x g durante 10 min a 4 ° C.
- Ressuspender o pellet contendo os cloroplastos com um artistas moles paint brush em 1 ml de tampão de lavagem frio (ver Tabela 1). Uma vez que o sedimento é ressuspendido, adicionar 50 ml de tampão de lavagem e centrifugar a solução a 10000 xg durante 10 min a 4 ° C.
- Remover o sobrenadante e ressuspender cuidadosamente o sedimento (tilacóides) em 50 ml de tampão de lavagem (ver Tabela 1).
- Centrifuga-se a solução a 10000 xg durante 10 min a 4 ° C e remove-se o sobrenadante completamente. Neste ponto, realizar os seguintes passos no escuro, para evitar a oxidação do pigmento.
- Adicionar ~ 20 ml de acetona a 80% tamponada com Na 2 CO 3 (ver Tabela 1) para extrair os pigmentos. Deixar a solução em gelo durante 10 min, vortex ocasional.
- Sedimentar os componentes celulares por centrifugação a 12.000 xg durante 15 min a 4 ° C.
NOTA: Se os pigmentos não são totalmente extraído, o sedimento terá uma cor verde e passo 1.6 deve ser repetido. - Recolhe-se o sobrenadante para um funil de separação. Adicionar 0,4 volumes de éter dietílico, agitar vigorosamente e abrir a válvula para ventilar o gás.
- Adicionar 0,8 volumes de 0,33 M de NaCl e misturar vigorosamente. Permitir ~ 10 min para separar as duas camadas. A fase de éter no topo contém os pigmentos extraídos. Remover a fase inferior transparente.
NOTA: Se a separação não é clara, congelar e descongelar a solução para melhorar a separação de fases. - Remover o éter vertendo-a da parte superior do funil de separação para um recipiente de vidro adequado. Seco, adicionando uma colher de gransulfato de sódio anidro ular. Agitar a solução e permitir a ~ 5 min para o dessecante para absorver a água a partir do éter.
NOTA: Repita este passo se o sulfato de sódio aparece completamente aglutinados; deve haver alguns cristais de livre flutuação quando o éter é suficientemente seco. Se uma camada de água de formas, remover esta com uma pipeta de Pasteur, antes de se adicionar sulfato de sódio anidro adicional. - Decantar o éter a um novo recipiente de vidro, deixando o sulfato de sódio sólido para trás.
- Evapora-se o éter num speedvac rotativo ou sob uma corrente de N 2.
- Dissolve-se os pigmentos completamente em 10 ml de acetona a 100%.
- Dilui-se uma pequena quantidade (~ 3 mL) em 1 ml de 80% de acetona e medir o espectro de absorção e determinar a / b clorofila e o rácio de concentração Chl com o método descrito por Porra et al. (1989) 15.
- Alíquotas e secar os pigmentos em um speedvac rotativo ou sob N 2 córrego até a acetona éevaporado completamente. Armazenar os pigmentos se a -80 ° C.
2. extração de carotenóides Espinafre
- Siga os passos 1.1 a 1.5. Neste ponto, realizar os seguintes passos no escuro, para evitar a oxidação do pigmento.
- Ressuspender o sedimento em tilacoide ~ 50 ml de etanol a 96% tamponado com Na 2 CO 3 (ver Tabela 1) para extrair os pigmentos. Deixar a solução em gelo durante 5 min.
- Sedimentar os componentes celulares por centrifugação a 12.000 xg durante 15 min a 4 ° C.
NOTA: Se os pigmentos não são totalmente extraído, o sedimento terá uma cor verde e passo 2.2 deve ser repetido. - Recolhe-se o sobrenadante e adiciona-se 0,1 volume de 80% KOH (w / v), para iniciar a saponificação.
- Deixar a solução a 4 ° CO / N, bem tapado e protegida da luz.
- Recolher a solução para um funil de separação. Adicionar 1 volume de éter dietílico e agitar suavemente.
- Adicionar 0,8 volumes de 0,33 M NaCl e misture delicadamente. Permitir ~ 10 min para separar as duas camadas. A fase de éter de laranja em cima contém carotenóides saponificados. Remover a fase inferior verde por drenagem através da torneira de passagem do funil.
- Adicionar 3 volumes de água e agitar suavemente para remover o hidróxido de potássio. Permitir que as camadas se separem. NOTA: Se a fase superior aspecto turvo, adicionar uma pequena quantidade de cloreto de sódio (por exemplo, 3 g de NaCl em 200 ml de solução) e agitar suavemente para dissolver.
- Remover a fase inferior por drenagem através da torneira de passagem do funil.
- Siga os passos de 1,10-1,13.
- Dilui-se uma pequena quantidade (~ 3 mL) em 1 ml de 80% de acetona e medir o espectro de absorção a 440 nm de 80% de acetona. Para determinar a concentração de, utilizar o coeficiente médio de extinção para os carotenóides (ε 440 = 255) 16 na seguinte fórmula: carro [mg / ml] = (Abs 440 nm / 225) x 11 (caminho óptico) = 1 cm.
- Alíquotas e secar o carotenoids em um speedvac ou sob N 2 fluxo até que todos dietílico foi evaporado. Armazenar os pigmentos se a -80 ° C.
3. pigmento total e carotenóides Extração de Chlamydomonas reinhardtii
- Crescer C. reinhardtii em meio TAP sólido 17 em uma placa de Petri por espalhar uma pequena quantidade de líquido de cultura sobre a superfície. Crescer sob fluxo iluminação contínua de 20 mmol fotos PSA m -2 s -1 até uma camada verde de células é visível.
- Utilizando uma ansa de inoculação estéril, colher uma pequena quantidade de C. reinhardtii, a partir do meio TAP sólido e colocou as células em 500 ml de meio de TAP 17, num balão de 1 L. Crescer a cultura a 25 ° C com 170 rpm de agitação sob um fluxo contínuo de iluminação 20 umol fotos PSA m -2 s -1.
- Após 5-6 dias, a cultura deve chegar ao fim da fase logarítmica (6 x 10 6 células / mlou 2-2,5 densidade óptica a 750 nm). Centrifuga-se a cultura a 4.000 x g durante 15 min a 4 ° C.
- Para a extração de pigmento total, siga os passos 1,6-1,15.
- O rendimento do extracto total de pigmento a partir de 500 mL de cultura de crescimento de C. completo reinhardtii é de cerca de 5 ml de solução com uma concentração de 0,5 mg chl a + b / ml.
- Para a extração de carotenóides, siga os passos 2,2-2,12.
4. purificação de corpos de inclusão
- O clone da sequência de codificação da proteína de interesse LHC num vector de expressão que resulta em um C-terminal His fundidos usando procedimentos padrão de biologia molecular. Transformar este construto em E. estirpe hospedeira de E. coli, tal como BL21 (DE3).
- Preparar tampão de lise, tampão detergente, tampão Triton, TE (Tabela 1), 1 M isopropílico β-D-1-tiogalactopiranósido (IPTG) e 18 de meio LB com os antibióticos apropriados.
- Escolha um single E. colónia coli contendo o clone de expressão a partir de uma placa recentemente riscada em ~ 5 ml de meio LB com os antibióticos apropriados utilizando procedimentos padrão 6. Crescer a 37 ° C com 220 rpm de agitação durante pelo menos 16 horas.
- Adicionar 2,5 ml de S / N de cultura num balão de Erlenmeyer de 1 L com 250 ml de LB suplementado com o antibiótico apropriado.
- Crescer as células durante 2-3 horas (ou até que a OD 600 é ~ 0,6) a 37 ° C a 220 rpm.
- Adicionar IPTG para uma concentração final de 1 mM. Continuar a crescer as células a 37 ° C com 220 rpm de 3-4 horas.
- Centrifuga-se a cultura durante 10 min a 5000 xg, a 4 ° C em um tubo de centrífuga previamente pesado. Descartar o sobrenadante cuidadosamente e determinar o peso do sedimento de pesagem novo e subtraindo o peso do tubo de centrifugação.
- Ressuspender o E. coli sedimento de células em 0,8 ml / g de tampão de lise por vórtice vigoroso.
NOTA: Como alternativa, o sedimento de células podem ser congeladas a -80C para uso posterior. Se começando com um sedimento congelado, deixar descongelar completamente antes de adicionar o tampão de lise. - Adicionar 2 mg de lisozima por grama de células, e incuba-se em gelo molhado com vórtice ocasional durante 30 min.
- Adiciona-se 20 ug / ml de DNAse, 10 mM de MgCl2, NaCl a 1 mM, 20 ug / ml de RNAse. Vortex e colocar em gelo durante 30 min.
- Adicionar 2 ml de tampão frio de detergente por grama de células. Misturar bem e manter a temperatura ambiente durante 5 min.
- Transferência para tubos de centrífuga de 2 ml (dividida em dois tubos, se necessário). Centrifugar durante 10 minutos a 12.000 xg, a 4 ° C para sedimentar os corpos de inclusão.
- Adicionar 1 ml de tampão frio Triton e voltar a suspender as pelotas por sons (3 pulsos x 5 seg poder x 50%, com intervalos de 20 segundos). NOTA: Tenha o tubo em um copo pequeno cercado por água gelada para manter a frio durante a sonorização. No caso de múltiplos tubos, combinar os corpos de inclusão novamente suspensas num tubo após a ressuspensão.
- Centrifugar durante 10 minutos a 12.000xg, a 4 ° C para sedimentar os corpos de inclusão.
- Repita o passo 4.13 e 4.14 duas vezes.
- Ressuspender os corpos de inclusão em 1 ml de TE frio com sonificação para uma lavagem final para remover o tampão de Triton. Centrifugar durante 10 minutos a 12.000 xg, a 4 ° C para sedimentar os corpos de inclusão.
- Ressuspender o sedimento em 1 ml de frio TE por sonificação.
- Avaliar a concentração de proteínas por meio de métodos padrão tais como o ensaio de Bradford 19. Armazenar alíquotas dos corpos de inclusão a -20 ° C.
5. Reconstituição
Este protocolo resulta tipicamente 1-2 ml de proteína reconstituída com uma DO de 4, quando a absorvância é medida na região do Qy (600-750 nm). Quantidade pode ser ajustado como desejado, embora cuidados devem ser tomados para manter as proporções adequadas durante o procedimento.
- Prepare as seguintes soluções, conforme descrito na Tabela 1: 2x Reconstituição buffer, 20% OG, 2 M de KCl, TE. Execute o seguinte sPET na penumbra.
- Ressuspender 800 ug de Corpos de Inclusão LHC em um total de 400 ul de TE, em um tubo de microcentrifugação de 2 ml. Adicionar 400 mL da 2x Tampão de Reconstituição e vortex brevemente.
- Adicionar 0,6 mL de β-mercaptoetanol (estoque de 14,8 M) para ter uma concentração final de 10 mM. Aquece-se a proteína por 1 min a 98 ° C. Vortex e brevemente lugar à temperatura ambiente durante 3 min.
- Ressuspender 500 mg do total de pigmentos de clorofila secas mais 80 mg pigmentos carotenóides em 30 mL 100% EtOH por vórtex vigorosamente durante 1 minuto ou coloque em um banho de ultra-sons para 1-2 min.
- Girar a mistura de pigmento ~ 30 seg a 15800 xg, a 4 ° C e confirmar que não há nenhum sedimento. Se houver um sedimento, repetir vórtex e / ou sonificação. Importante: Após a ressuspensão e centrifugação, adicionar imediatamente pigmento para a proteína, ou pode agregar e terá de ser novamente suspensas novamente.
- Lentamente adicione a mistura de pigmentos para a proteína, enquanto arrefecida vórtex. Continue a vortex 5-10 sCE e coloque o tubo no gelo molhado. Tenha cuidado para não vortex com muita força, como a proteína pode transbordar o topo do tubo.
- Adicionar 94 ul de 20% de octilo β-D-glucopiranósido (OG) (concentração final 2%), vortex brevemente e manter em gelo 10 min.
- Adicionar 90 mL de KCl 2 M (concentração final de 150-200 mM), vortex brevemente e manter em gelo 20 min. NOTA: preparação da coluna (Secção 6) pode ser iniciado nesta altura.
- Rotação durante 10 minutos a 15.800 xg, a 4 ° C. Retirar o sobrenadante sem perturbar o pelete (precipitado LDS) para um tubo de 10 ml. Manter frio e protegida da luz.
6. níquel Coluna Purificação
- Preparar as seguintes soluções, tal como descrito na Tabela 1: tampão de OG, tampão de lavagem de OG, tampão de eluição.
- Conectar uma coluna de Ni-Sepharose (1 ml), ou equivalente a uma bomba peristáltica, assegurando que nenhum ar fica dentro da coluna durante este passo e os seguintes passos.
- Defina a velocidade de tele bombear a 1 ml / min e lavar a coluna com 5-10 ml de água para remover a solução de armazenamento.
- Equilibrar a coluna com 3-4 ml de tampão de OG.
- Adicionar 3-4 ml de tampão de OG para a amostra de proteína e carga para a coluna. NOTA: se a proteína foi sentado no gelo por mais de 10 minutos após a remoção da LDS, girar novamente a 15.800 xg a 4 ° C durante 1 min para remover qualquer precipitação LDS adicional.
- Lavar a coluna com 5 ml de tampão de OG.
- Lavar a coluna com 2 ml de tampão de lavagem com OG.
- Eluir a proteína ligada com tampão de eluição de 3 ml. Recolhe-se o eluato verde que contém a proteína reconstituída. NOTA: Este é geralmente de cerca de 1 ml no total.
7 Sacarose Centrifugação Gradiente
- Preparar as seguintes soluções, tal como descrito na Tabela 1: solução de sacarose, 0,06% β-MS, HEPES 0,01 M, pH 7,6.
- Preencha tubos de ultracentrífuga com a solução de sacarose e congelamento a -20 ° CO / N or -80 ° C durante pelo menos 1 hr.
- Remover o tubo do congelador e deixar descongelar imperturbado a 4 ° C. NOTA: O processo de congelação / descongelação cria um gradiente de 0,1 a 1 M de sacarose. Um tubo de 15 ml, tipicamente cerca de degelo em 3 h.
- Cuidadosamente remover a partir do topo do mesmo volume que a fracção verde eluída a partir da coluna de Sepharose de níquel no passo 6.8. Em seguida, coloque a amostra reconstituída em cima lentamente para evitar a perturbação do gradiente.
- Tubos de equilíbrio e rotação em 200.000 xg a 4 ° C em uma ultracentrífuga usando um SW-41 ou SW-60 rotor basculante para 18 horas, a abrandar aceleração e parada, sem freios.
- Retire cuidadosamente o gradiente do suporte do tubo com uma pinça. Usando uma seringa com uma agulha longa que tem uma abertura romba para recolher a fracção do topo. NOTA: Como alternativa, recolher fracções da parte inferior do tubo de perfuração com uma agulha e recolher gotas.
Resultados
Este protocolo detalha um método para reconstituir as proteínas de ligação chorophyll a / b, in vitro. Esta técnica permite a dobragem dos complexos proteína-pigmento in vitro a partir da apoproteína, que pode ser obtido por sobreexpressão num sistema heterólogo, e pigmentos extraídos de plantas ou algas. Após a reconstituição, o complexo pigmento-proteína redobrada é purificada a partir do excesso de pigmentos e a apoproteína desdobrado em duas etapas. A primeira etapa (Figura 1A-B) baseia-se na presença de His no C-terminal da proteína, o que permite a remoção da maior parte dos pigmentos não ligados. O segundo passo de purificação utiliza centrifugação em gradiente de densidade de sacarose, (Figura 2), onde a proteína desdobrada geralmente migra mais lentamente do que a banda verde contendo a proteína reconstituída. O objectivo da reconstituição in vitro é a obtenção de complexos com o mesmo bomlaços como as nativas. Para ilustrar este resultado, as propriedades espectroscópicas de um complexo in vivo decolheita é comparado com o mesmo complexo LHC reconstituída in vitro, 13,20,21. O espectro de absorção do CLS na gama do visível (350 nm e 750 nm) depende da composição de pigmento do complexo, assim como sobre o meio ambiente do pigmento (que inclui a proteína) e que é, portanto, um instrumento sensível para verificar a qualidade da reconstituição. Na Figura 3, o espectro de absorção de CP24, a clorofila a / b da proteína a partir de Arabidopsis thaliana, reconstituído de ligação in vitro, é comparado com o espectro do mesmo complexo purificado de Arabidopsis tilacóides 21. Nos espectros, é possível reconhecer a Qy ea transição Soret de Chl (picos a 671/439 nm) e Cl (b picos a 649/466 nm). Os complexos nativos e reconstituídos mostram abso idênticoespectros rption, indicando uma composição de pigmento praticamente idêntica e de organização. Espectroscopia de fluorescência pode ser utilizado para avaliar a qualidade de o complexo reconstituído. Os espectros de emissão de fluorescência é medido por excitação a comprimentos de onda diferentes, os quais excitam preferencialmente diferentes pigmentos: Chl a 440 nm, Cl b a 475 nm, e Xantofilas a 500 nm. Em um complexo proteína de pigmento correctamente dobrados, clorofila b e Xantofilas transferir a sua energia de excitação, principalmente para clorofila a poucos picossegundos, e a fluorescência se origina a partir de um sistema termicamente equilibrada resultando num pico único com a mesma forma e máxima a todos os três excitação comprimentos de onda (Figura 4A-B). A presença de clorofila b não coordenado para a proteína pode ser reconhecido por um pico ou ressalto adicional em torno de 650 nm sobre 475 nm de excitação (Figura 4C). A presença de livre Chl uma vez levaa emissão adicional ao redor de 675 nm, que está presente principalmente em cima de 440 nm de excitação. Os espectros de emissão de fluorescência 475 nm após a excitação de ambos os complexos e reconstituída CP24 nativas (Figura 4D) mostram um único pico a 681 nm, indicando que o complexo é reconstituída correctamente dobrado. Uma confirmação adicional de que o complexo pigmento-proteína é correctamente reconstituído vem a partir de medições de dicroísmo circular (CD). O sinal de CD na região visível depende das interacções entre excitônicas pigmentos e é, portanto, muito sensíveis até mesmo pequenas mudanças na organização dos cromóforos 22. Figura 5 mostra os espectros de CD de reconstituído e CP24 nativa, com os picos típicos da impressão digital em 681 nm, 650 nm e 481 nm. Em conclusão, a alta semelhança entre as propriedades espectroscópicas de nativo e o CP24 reconstituído confirma que os complexos de rendimentos do procedimento de reconstituição nativa como suita ble para estudo in vitro de proteínas light-colheita.
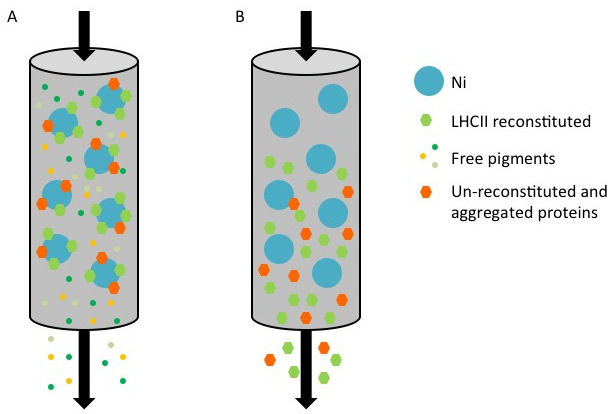
Figura 1 Representação da purificação de proteínas recombinantes do LHC com a tag Seu usando uma coluna de níquel. (A) Durante a purificação, His-marcado proteína, composta de ambos os complexos reconstituídos (hexágono) e proteína não-reconstituída / agregado (laranja hexágono) está ligado à superfície do Ni-Sepharose (mancha azul), enquanto os pigmentos não ligados (pequenos pontos coloridos) fluir. (B) Quando a coluna é lavada com o tampão de eluição contendo imidazol, as proteínas reconstituídos e un-reconstituída são recolhidos no fluxo através.
"width =" hres.jpg 500 "/>
Figura 2. gradiente de sacarose LHCII reconstituído após purificação por coluna de níquel. Os complexos reconstituídos são separados a partir do pigmento livre por o gradiente de densidade. A faixa verde escura representa LHCII reconstituída eo fundo verde pálido é composta por pigmentos livres.
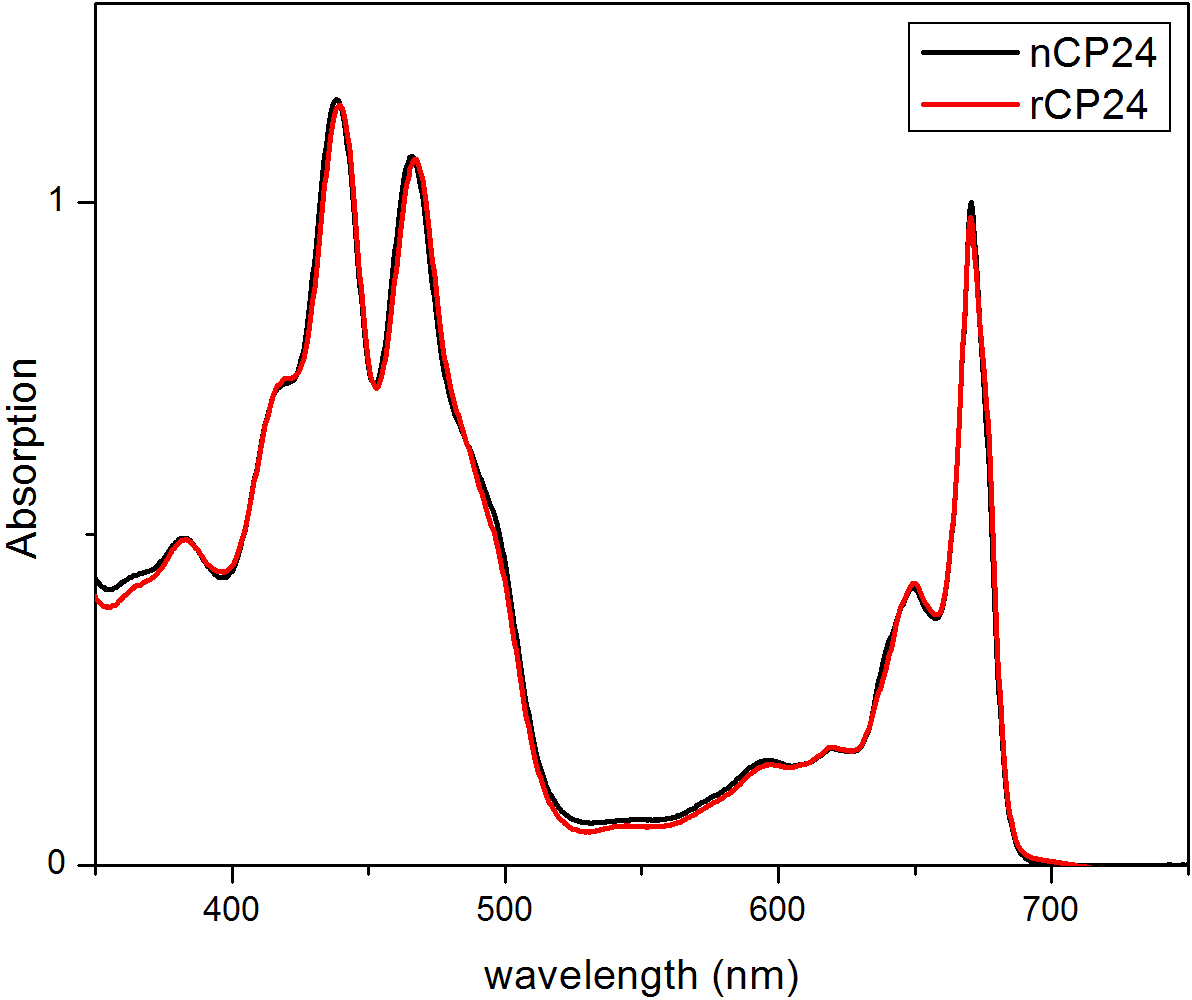
Figura 3 espectro de absorção da proteína reconstituída CP24 (rCP24, linha vermelha) e a uma nativa (nCP24, linha preta) isolado a partir de Arabidopsis thaliana. Em ambos os espectros, é possível reconhecer a Qy ea transição Soret da clorofila a (picos a 671/439 nm) e Chl b (picos em 649/466 nm). Este valor foi modificado a partir Passarini et al. 2014 21.
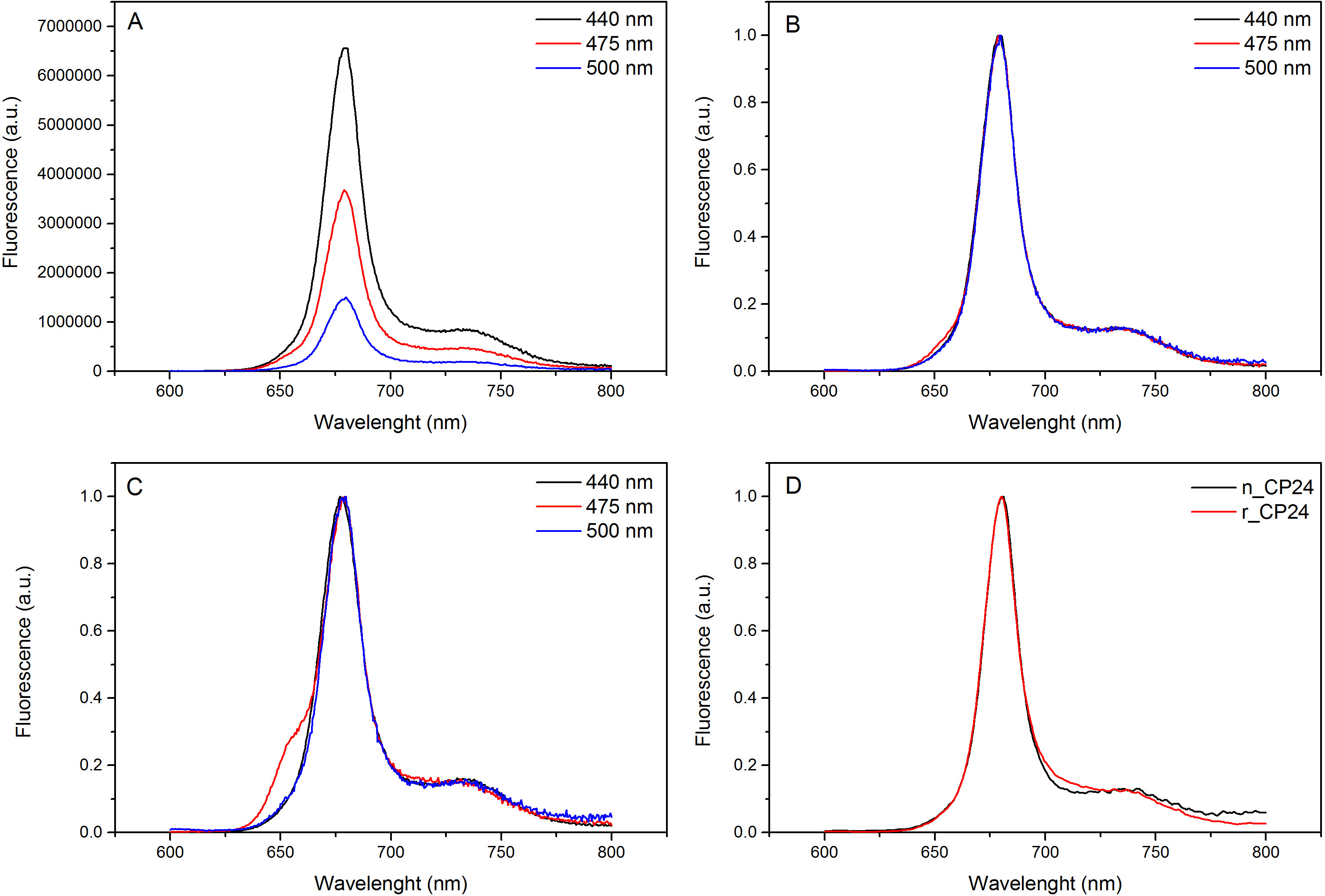
A Figura 4 o espectro de emissão de fluorescência. Os espectros de emissão de fluorescência do complexo reconstituído CP24 tipo selvagem (A) e normalizadas para o valor máximo (B) que mostra a transferência de energia eficaz a partir de Chl e b Xanthophyls CHL para um. (C) Fluorescência espectros de emissão de CP24 reconstituído (rCP24) eo complexo nativo (nCP24) isolado de Arabidopsis thaliana. Os espectros são normalizados para o máximo do pico (D). Clique aqui para ver uma versão maior desta figura.
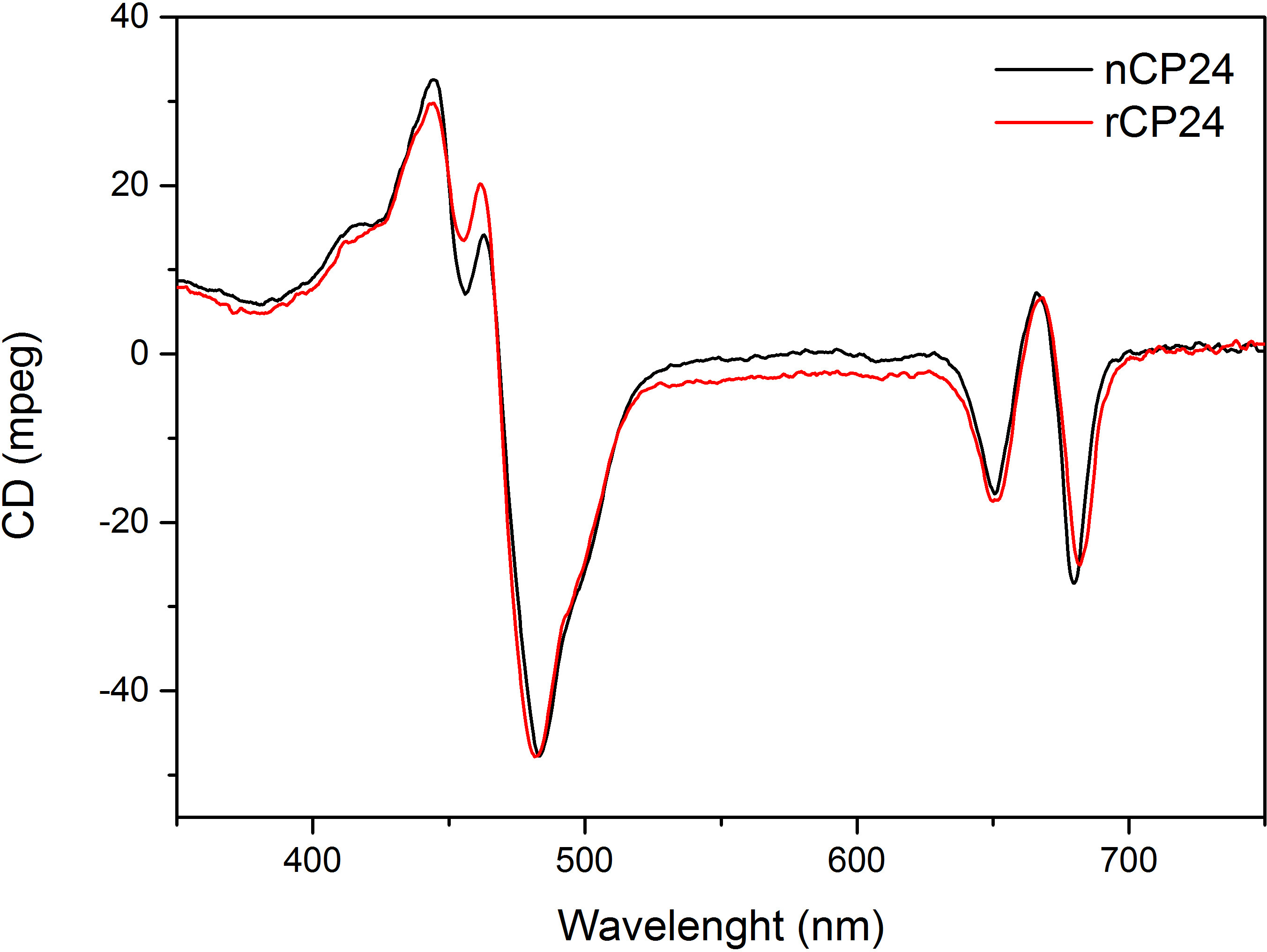
Figura 5 Dicroísmo Circular Spectra. Reconstituído CP24 (rCP24, linha vermelha) e o complexo nativo (nCP24, linha preta) isolado a partir de Arabidopsis thaliana mostra espectros muito semelhantes.

Figura 6 Espectros de absorção de CP29 tipo selvagem (CP29_WT) e mutante CP29 (CP29_A2). A linha verde mostra as diferenças entre as duas parcelas.
| Todos os tampões podem ser armazenadas a 4 ° C. | |||
| Componentes | Concentração final | Notas adicionais | |
| Moagem de buffer | Sorbitol | 0,4 M | |
| Tricine | 0,1 M | pH 7,8 | |
| NaCl | 10 mM | ||
| MgCl2 | 5 mM | ||
| Leite em Pó | 0,5% w / v | ||
| Tampão de Lavagem | Sorbitol | 50 mM | |
| Tricine | 5 mM | pH 7,8 | |
| EDTA | 10 mM | pH 8 | |
| Tampão de Lise | Tris | 50 mM | pH 8 |
| Sacarose | 2,5% w / v | ||
| EDTA | 1 mM | pH 8 | |
| Tampão detergente | NaCl | 200 mM de NaCl | |
| Ácido deoxicólico | 1% w / v | ||
| NONIDET P-40 | 1% w / v | ||
| Tris | 20 mM | pH 7,5 | |
| EDTA | 2 mM | pH 8 | |
| beta-mercaptoetanol | 10 mM | ||
| Triton Tampão | Triton X-100 | 0,5% w / v | |
| Tris | 20 mM | pH 7,5 | |
| beta-mercaptoetanol | 1 mM | ||
| Tampão TE | Tris | 50 mM | pH 8 |
| EDTA | 1 mM | pH 8 | |
| Tampão de Reconstituição | HEPES | 200 mM | |
| Sacarose | 5% w / v | ||
| Lithiumdodecylsulfate (LDS) | 4% w / v | ||
| Benzamidina | 2 mM | ||
| Aminocapróico | 10 mM | ||
| OG Tampão | Octilglucosido | 1% w / v | |
| 12.5% w / v | |||
| NaCl | 0,2 M | ||
| HEPES | 20 mM | ||
| Imidazole | 10 mM | ||
| OG Rinse Tampão | n-dodecil-beta-D-maltósido (β-MS) | 0,06% w / v | |
| HEPES | 40 mM | pH 7,5-9 | |
| NaCl | 0,2 M | ||
| Tampão de eluição | Imidazole | 0,5 M | |
| n-dodecil-beta-D-maltósido (β-MS) | 0,06% w / v | ||
| HEPES | 40 mM | pH 8 | |
| NaCl | 0,2 M | ||
| Solução de sacarose | Sacarose | 20% w / v | |
| n-dodecil-beta-D-maltósido (β-MS) | 0,06% w / v | ||
| HEPES | 0,01 M | pH 7,6 | |
| Acetona 80% tamponada com carbonato de sódio | Acetona | 80% v / v | |
| Carbonato de Sódio | 1 M | ||
| Etanol a 96% tamponada com carbonato de sódio | Etanol | 96% v / v | |
| Carbonato de Sódio | 1 M | ||
Tabela 1 Lista de buffers e soluções utilizadas neste protocolo.
rCP26| Chla a / b mix | Chla a / b | Chl a | Chl b | Neo | Viola | Lute | Chl tot | Chl / Car | |
| nCP26 | - | 2,2 ± 0,05 | 6.2 | 2.8 | 0,61 | 0,38 | 1.02 | 9 | 4,5 ± 0,1 |
| rCP26 | 8 | 2,71 ± 0,05 | 6,57 | 2.43 | 0,72 | 0,32 | 0,97 | 3,9 ± 0,04 | |
| rCP26 | 5.5 | 2,25 ± 0,05 | 6.23 | 2.77 | 0,77 | 0,3 | 0,96 | 9 | 4,0 ± 0,1 |
| rCP26 | 3 | 2,08 ± 0,04 | 6,08 | 2.92 | 0,76 | 0,3 | 1.04 | 9 | 4,1 ± 0,1 |
| rCP26 | 1 | 1,7 ± 0,05 | 5,7 | 3.3 | 0,7 | 0,3 | 0.9 | 9 | 4,3 ± 0,05 |
| rCP26 | 0,3 | 1,11 ± 0,04 | 4.7 | 4.28 | 0,7 | 0,3 | 0.9 | 9 | 4,2 ± 0,2 |
| 0,05 | 0,23 ± 0,01 | 1.4 | 5.6 | 0.58 | 0,24 | 1.11 | 7 | 3,1 ± 0,06 | |
| rCP26 | <0,01 | 0,11 ± 0,01 | 0,7 | 6,3 | 0,64 | 0,3 | 1.08 | 7 | 3,06 ± 0,06 |
Tabela 2 Teor de pigmentos de CP26 nativa complexo em comparação com reconstituídos complexos proteína-pigmento com diferentes Chl a / b Rácios 39.
Discussão
As proteínas da membrana não são tão fáceis de estudar. O isolamento de proteínas de membrana nativa é complicado pela necessidade de solubilizar a bicamada lipídica com detergentes, o que pode danificar a proteína e remover cofactores essenciais. Estas proteínas também pode estar presente em níveis baixos em membranas biológicas, ou ser misturado com as proteínas intimamente relacionadas, como no caso dos complexos de colheita de luz, que faz com que a purificação dos complexos individuais difíceis. Expressão de proteínas heterólogas em E. coli e na reconstituição in vitro oferece a possibilidade de evitar estes problemas. in vitro reconstituição e purificação de proteínas dobradas resultados em complexos que possuem características muito semelhantes às dos complexos nativos 20,21,23 e, portanto, pode ser usado para estudar os complexos que não podem ser purificados até à homogeneidade 24 - 27.
Este método utiliza o espinafre, a qual é facilmente attainable durante todo o ano, como uma fonte para o pigmento e carotenóides totais preparativos. Para algumas reconstituições de proteínas nativas em algas, a utilização de pigmentos purificados a partir de algas é preferido devido a diferentes composições de pigmento. O a / b razão Chl e relação / carro Chl permanece a mesma, independentemente da fonte de pigmento.
É importante perceber que a eficiência da reconstituição é geralmente cerca de 35% 28. Assim, é necessário remover os pigmentos não-ligados e a apoproteína desdobrado a partir da solução depois da reconstituição. Um protocolo de purificação de dois passos é apresentado neste protocolo (ver também os resultados). No entanto, deve notar-se que o passo do gradiente de sacarose não permite a separação completa de apo e holo-proteína. Para a maioria das análises isto não é um problema, a apoproteína, como não contêm pigmentos e, portanto, não interferem com as medições funcionais. No entanto, no caso de ser necessário remover completamente a apoproteína do fração contendo o complexo reconstituído (por exemplo, para calcular o pigmento a estequiometria de proteína), uma coluna de permuta aniónica podem ser utilizados (ver Passarini et al. 2009 29 para detalhes).
A capacidade para redobrar proteínas recombinantes de colheita de luz, com pigmentos isoladas in vitro, oferece a oportunidade de "manipular" os complexos modificando a reconstituição "ambiente" de vários modos, alterando assim as características do complexo resultante. Por exemplo, alterando a composição do pigmento durante a reconstituição pode resultar em um complexo com a composição de pigmento alterada. Esta característica pode ser utilizada para estudar a influência vários pigmentos têm sobre a estrutura e a estabilidade do complexo. Normalmente, a preparação do pigmento obtido de espinafre tem um a / b clorofila proporção de 3: 1 e uma proporção / chl carro de 2,9: 1. Este rácio tipicamente produz uma proteína reconstituída com as mesmas propriedades que o nAtive um. No entanto, o ajuste de um a / b clorofila proporção pela adição de uma purificado Chl ou b podem influenciar a ligação de diferentes pigmentos, devido à variação de selectividade dos locais de ligação de 30-33. Isto é possível porque a maior parte dos locais de ligação de pigmentos não são completamente selectivas para clorofila a ou clorofila b, mas pode acomodar ambos, embora com diferente afinidade 10,30,34. De um modo semelhante, os locais de ligação de carotenóides, também foram exibidas para ser capaz de acomodar mais do que uma espécie de xantofila 8,35 - 38. Diferentes reconstituições de CP26, outro complexo pigmento-proteína de plantas superiores, usando várias composições de pigmento estão apresentados na Tabela 2 39. Estas reconstituições foram usadas para avaliar a afinidade dos sítios de ligação específicos de pigmentos 39. É interessante notar que a fim de obter um complexo com o mesmo pigmento composição como uma nativa, a clorofila a relação a / b da mistura de pigmentos deve ser de 3: 1. Este parece ser o caso para todos os complexos do LHC de plantas superiores 20,40.
A combinação de biologia molecular, com a técnica de reconstituição permite que as propriedades de um complexo de ligação a clorofila a ser estudado em mais detalhe. A importância dos diferentes domínios de proteínas na estabilidade e dobramento dos complexos, ou o seu envolvimento nas interações proteína-proteína, foram determinados por truncar a apoproteína ou realizar mutagênese aleatória 8,41 - 44. Resíduos de aminoácidos standard importantes para a coordenação das diferentes pigmentos pode ser alterado através de mutagénese dirigida ao local, de modo a analisar as propriedades de pigmentos individuais ou avaliar a sua contribuição para a função e estabilidade do complexo de 10,28,29,45 - 52. Figura 6 mostra reconstituído Lhcb4 (CP29) comuma mutação da histidina na posição 216 53. Uma comparação da composição de pigmento de tipo selvagem e mutantes de complexos indica que a mutação induz a perda de uma molécula de clorofila a, indicando que o sítio alvo acomoda um um Chl no complexo WT. As diferenças do espectro de absorção de WT e mutantes, mediante a normalização para o teor de pigmentos, também mostra as propriedades de absorção do pigmento perdido. Neste caso, a diferença pode ser visto no pico principal a 680 nm, o que indica que a clorofila a coordenada por His216 absorve a este comprimento de onda (para mais detalhes sobre este mutante e as propriedades espectroscópicas ver Mozzo et al. 2,008 53). Análise de mutação também pode ser utilizado para determinar o efeito do ambiente nas propriedades espectroscópicas dos pigmentos 54.
Em conclusão, as proteínas de colheita de luz pode facilmente ser reconstituída in vitro, resultando em pigmento-protein complexos com propriedades muito semelhantes aos complexos nativos. Deste modo, as dificuldades de isolamento de proteínas nativas são eliminados, ao mesmo tempo, proporcionando a preparação de proteínas com elevado rendimento e grau de pureza para mais estudos. A importância de uma proporção 3: a / b clorofila 1 na produção de um complexo autêntico é enfatizado, e exemplos de tipo selvagem reconstituído e CLS mutantes são fornecidos para ilustrar as aplicações da técnica.
Divulgações
The authors declare no conflict of interest
Agradecimentos
This work was supported by the European research council by a ERC starting/consolidator grant to RC and by the Dutch Foundation for research on matter (FOM) via a FOM program (10TM01).
Materiais
| Name | Company | Catalog Number | Comments |
| HisTrap HP | GE Healthcare | 17-5247-01 | |
| Nylon cloth | 20 μm pores | ||
| Soft artists paint brush | |||
| NONIDET P-40 | Sigma | 74385 | |
| Beta-DM | Sigma | D4641 | |
| DNAase | ThermoScientific | EN0525 | |
| Milk Powders | |||
| RNAase | ThermoScientific | EN0531 | |
| Sonicator | |||
| Octyl β-D-glucopyranoside | Sigma | O8001 | |
| Ultracentrifuge XL | Beckman-Coulter | ||
| TAP medium | see reference 17 | ||
| LB medium | see reference 19 |
Referências
- Plumley, F. G., Schmidt, G. W. Reconstitution of chlorophyll a/b light-harvesting complexes: Xanthophyll-dependent assembly and energy transfer. Proceedings of the National Academy of Sciences of the United States of America. 84, 146-150 (1987).
- Croce, R., van Amerongen, H. Light-harvesting and structural organization of Photosystem II from individual complexes to thylakoid membrane. Journal of photochemistry and photobiology B Biology. 104 (1-2), 142-153 (2011).
- Li, Z., Wakao, S., Fischer, B. B., Niyogi, K. K. Sensing and responding to excess light. Annual review of plant biology. 60, 239-260 (2009).
- De Bianchi, S., Ballottari, M., Dall’osto, L., Bassi, R. Regulation of plant light harvesting by thermal dissipation of excess energy. Biochemical Society transactions. 38 (2), 651-660 (2010).
- Neilson, J. A. D., Durnford, D. G. Structural and functional diversification of the light-harvesting complexes in photosynthetic eukaryotes. Photosynthesis research. 106 (1-2), 57-71 (2010).
- Paulsen, H., Rümler, U., Rüdiger, W. Reconstitution of pigment-containing complexes from light-harvesting chlorophyll a/b-binding protein overexpressed in Escherichia coli. Planta. 181 (2), 204-211 (1990).
- Horn, R., Grundmann, G., Paulsen, H. Consecutive binding of chlorophylls a and b during the assembly in vitro of light-harvesting chlorophyll-a/b protein (LHCIIb). Journal of molecular biology. 366 (3), 1045-1054 (2007).
- Cammarata, K. V., Schmidt, G. W. In vitro reconstitution of a light-harvesting gene product: deletion mutagenesis and analyses of pigment binding. Biochemistry. 31 (10), 2779-2789 (1992).
- Paulsen, H., Hobe, S. Pigment-binding properties of mutant light-harvesting chlorophyll-a/b-binding protein. European journal of biochemistry / FEBS. 205 (1), 71-76 (1992).
- Bassi, R., Croce, R., Cugini, D., Sandonà, D. Mutational analysis of a higher plant antenna protein provides identification of chromophores bound into multiple sites. Proceedings of the National Academy of Sciences of the United States of America. 96 (18), 10056-10061 (1999).
- Paulsen, H., Finkenzeller, B., Kühlein, N. Pigments induce folding of light-harvesting chlorophyll a/b-binding protein. European journal of biochemistry / FEBS. 215 (3), 809-816 (1993).
- Caffarri, S., Croce, R., Cattivelli, L., Bassi, R. A look within LHCII differential analysis of the Lhcb1-3 complexes building the major trimeric antenna complex of higher-plant photosynthesis. Biochemistry. 43 (29), 9467-9476 (2004).
- Giuffra, E., Cugini, D., Croce, R., Bassi, R. Reconstitution and pigment-binding properties of recombinant CP29. European journal of biochemistry / FEBS. 238 (1), 112-120 (1996).
- Rogl, H., Kosemund, K., Kühlbrandt, W., Collinson, I. Refolding of Escherichia coli produced membrane protein inclusion bodies immobilised by nickel chelating chromatography. FEBS letters. (1-2), 21-26 (1998).
- Porra, R. J., Thompson, W. A., Kriedemann, P. E. Determination of accurate extinction coefficients and simultaneous equations for assaying chlorophylls a and b extracted with four different solvents: verification of the concentration of chlorophyll standards by atomic absorption spectroscopy. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 975 (3), 384-394 (1989).
- Davies, B. H. Identification of carotenoids by their absorption characteristics. Biochem J. 103 (2), (1967).
- Gorman, D. S., Levine, R. P. Cytochrome f and plastocyanin their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. Proceedings of the National Academy of Sciences of the United States of America. 54 (6), 1665-1669 (1965).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of bacteriology. 62 (3), 293-300 (1951).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical biochemistry. 72, 248-254 (1976).
- Wientjes, E., Croce, R. The light-harvesting complexes of higher-plant Photosystem I Lhca1/4 and Lhca2/3 form two red-emitting heterodimers. The Biochemical journal. 433 (3), 477-485 (2011).
- Passarini, F., Xu, P., Caffarri, S., Hille, J., Croce, R. Towards in vivo mutation analysis knock-out of specific chlorophylls bound to the light-harvesting complexes of Arabidopsis thaliana - the case of CP24 (Lhcb6). Biochimica et biophysica acta. , (2014).
- Georgakopoulou, S., van der Zwan, G., Bassi, R., van Grondelle, R., van Amerongen, H., Croce, R. Understanding the changes in the circular dichroism of light harvesting complex II upon varying its pigment composition and organization. Biochemistry. 46 (16), 4745-4754 (2007).
- Croce, R., Müller, M. G., Caffarri, S., Bassi, R., Holzwarth, A. R. Energy transfer pathways in the minor antenna complex CP29 of photosystem II a femtosecond study of carotenoid to chlorophyll transfer on mutant and WT complexes. Biophysical journal. 84 (4), 2517-2532 (2003).
- Schmid, V. H., Cammarata, K. V., Bruns, B. U., Schmidt, G. W. In vitro reconstitution of the photosystem I light-harvesting complex LHCI-730 heterodimerization is required for antenna pigment organization. Proceedings of the National Academy of Sciences of the United States of America. 94 (14), 7667-7672 (1997).
- Castelletti, S., Morosinotto, T., Robert, B., Caffarri, S., Bassi, R., Croce, R. Recombinant Lhca2 and Lhca3 subunits of the photosystem I antenna system. Biochemistry. 42 (14), 4226-4234 (2003).
- Storf, S., Jansson, S., Schmid, V. H. R. Pigment binding, fluorescence properties, and oligomerization behavior of Lhca5, a novel light-harvesting protein. The Journal of biological chemistry. 280 (7), 5163-5168 (2005).
- Mozzo, M., Mantelli, M., Passarini, F., Caffarri, S., Croce, R., Bassi, R. Functional analysis of photosystem I light-harvesting complexes (Lhca) gene products of Chlamydomonas reinhardtii. Biochimica et biophysica acta. 1797 (2), 212-221 (2010).
- Remelli, R., Varotto, C., Sandonà, D., Croce, R., Bassi, R. Chlorophyll binding to monomeric light-harvesting complex. A mutation analysis of chromophore-binding residues. The Journal of biological chemistry. 274 (47), 33510-33521 (1999).
- Passarini, F., Wientjes, E., Hienerwadel, R., Croce, R. Molecular basis of light harvesting and photoprotection in CP24 unique features of the most recent antenna complex. The Journal of biological chemistry. 284 (43), 29536-29546 (2009).
- Giuffra, E., et al. Analysis of some optical properties of a native and reconstituted photosystem II antenna complex, CP29 pigment binding sites can be occupied by chlorophyll a or chlorophyll b and determine spectral forms. Biochemistry. 36 (42), 12984-12993 (1997).
- Pagano, A., Cinque, G., Bassi, R. In vitro reconstitution of the recombinant photosystem II light-harvesting complex CP24 and its spectroscopic characterization. The Journal of biological chemistry. 273 (27), 17154-17165 (1998).
- Kleima, F. J., et al. Decreasing the chlorophyll a/b ratio in reconstituted LHCII structural and functional consequences. Biochemistry. 38 (20), 6587-6596 (1999).
- Croce, R., Morosinotto, T., Castelletti, S., Breton, J., Bassi, R. The Lhca antenna complexes of higher plants photosystem I. Biochimica et biophysica acta. 1556 (1), 29-40 (2002).
- Hobe, S., Trostmann, I., Raunser, S., Paulsen, H. Assembly of the major light-harvesting chlorophyll-a/b complex Thermodynamics and kinetics of neoxanthin binding. The Journal of biological chemistry. 281 (35), 25156-25166 (2006).
- Croce, R., Weiss, S., Bassi, R. Carotenoid-binding sites of the major light-harvesting complex II of higher plants. The Journal of biological chemistry. 274 (42), 29613-29623 (1999).
- Hobe, S., Niemeier, H., Bender, A., Paulsen, H. Carotenoid binding sites in LHCIIb. Relative affinities towards major xanthophylls of higher plants. European journal of biochemistry / FEBS. 267 (2), 616-624 (2000).
- Jahns, P., Depka, B., Trebst, A. Xanthophyll cycle mutants from Chlamydomonas reinhardtii indicate a role for zeaxanthin in the D1 protein turnover. Plant Physiology and Biochemistry. 38 (5), 371-376 (2000).
- Wehner, A., Grasses, T., Jahns, P. De-epoxidation of violaxanthin in the minor antenna proteins of photosystemII, LHCB4, LHCB5, and LHCB6. The Journal of biological chemistry. 281 (31), 21924-21933 (2006).
- Croce, R., Canino, G., Ros, F., Bassi, R. Chromophore organization in the higher-plant photosystem II antenna protein CP26. Biochemistry. 41 (23), 7334-7343 (2002).
- Caffarri, S., Passarini, F., Bassi, R., Croce, R. A specific binding site for neoxanthin in the monomeric antenna proteins CP26 and CP29 of Photosystem II. FEBS letters. 581 (24), 4704-4710 (2007).
- Hobe, S., Förster, R., Klingler, J., Paulsen, H. N-proximal sequence motif in light-harvesting chlorophyll a/b-binding protein is essential for the trimerization of light-harvesting chlorophyll a/b complex. Biochemistry. 34 (32), 10224-10228 (1995).
- Kuttkat, A., Hartmann, A., Hobe, S., Paulsen, H. The C-terminal domain of light-harvesting chlorophyll-a/b-binding protein is involved in the stabilisation of trimeric light-harvesting complex. European journal of biochemistry / FEBS. 242 (2), 288-292 (1996).
- Rupprecht, J., Paulsen, H., Schmid, V. H. Protein domains required for formation of stable monomeric Lhca1- and Lhca4-complexes. Photosynthesis research. 63 (3), 217-224 (2000).
- Yang, C., et al. The negatively charged amino acids in the lumenal loop influence the pigment binding and conformation of the major light-harvesting chlorophyll a/b complex of photosystem II. Biochimica et biophysica acta. 1777 (11), 1463-1470 (2008).
- Rogl, H., Kühlbrandt, W. Mutant trimers of light-harvesting complex II exhibit altered pigment content and spectroscopic features. Biochemistry. 38 (49), 16214-16222 (1999).
- Yang, C., Kosemund, K., Cornet, C., Paulsen, H. Exchange of pigment-binding amino acids in light-harvesting chlorophyll a/b protein. Biochemistry. 38 (49), 16205-16213 (1999).
- Morosinotto, T., Castelletti, S., Breton, J., Bassi, R., Croce, R. Mutation analysis of Lhca1 antenna complex. Low energy absorption forms originate from pigment-pigment interactions. The Journal of biological chemistry. 277 (39), 36253-36261 (2002).
- Morosinotto, T., Breton, J., Bassi, R., Croce, R. The nature of a chlorophyll ligand in Lhca proteins determines the far red fluorescence emission typical of photosystem I. The Journal of biological chemistry. 278 (49), 49223-49229 (2003).
- Ballottari, M., Mozzo, M., Croce, R., Morosinotto, T., Bassi, R. Occupancy and functional architecture of the pigment binding sites of photosystem II antenna complex Lhcb5. The Journal of biological chemistry. 284 (12), 8103-8113 (2009).
- Croce, R., et al. Origin of the 701-nm fluorescence emission of the Lhca2 subunit of higher plant photosystem I. The Journal of biological chemistry. 279 (47), 48543-48549 (2004).
- Morosinotto, T., Mozzo, M., Bassi, R., Croce, R. Pigment-pigment interactions in Lhca4 antenna complex of higher plants photosystem I. The Journal of biological chemistry. 280 (21), 20612-20619 (2005).
- Mozzo, M., Morosinotto, T., Bassi, R., Croce, R. Probing the structure of Lhca3 by mutation analysis. Biochimica et biophysica acta. 1757 (12), 1607-1613 (2006).
- Mozzo, M., Passarini, F., Bassi, R., van Amerongen, H., Croce, R. Photoprotection in higher plants the putative quenching site is conserved in all outer light-harvesting complexes of photosystem II. Biochimica et biophysica acta. 1777 (10), 1263-1267 (2008).
- Wientjes, E., Roest, G., Croce, R. From red to blue to far-red in Lhca4 how does the protein modulate the spectral properties of the pigments. Biochimica et biophysica acta. 1817 (5), 711-717 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados