Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Producción y Uso de lentivirus de células selectivamente la transducción primaria Oligodendrocyte precursor para
En este artículo
Resumen
Here we present protocols that offer a flexible and strategic foundation for virally manipulating oligodendrocyte precursor cells to overexpress proteins of interest in order to specifically interrogate their role in oligodendrocytes via the in vitro model of central nervous system myelination.
Resumen
La mielinización es un proceso complejo que implica tanto a las neuronas y las células gliales que forman la mielina, oligodendrocitos en el sistema nervioso central (CNS) y células de Schwann en el sistema nervioso periférico (SNP). Utilizamos un ensayo in vitro en la mielinización, un modelo establecido para el estudio de la mielinización del SNC in vitro. Para ello, se añaden las células precursoras de oligodendrocitos (OPC) a las neuronas purificados dorsal roedor ganglio de la raíz primaria (GRD) para formar co-cultivos myelinating. Con el fin de interrogar específicamente el papel que determinadas proteínas expresadas por los oligodendrocitos ejercen sobre la mielinización hemos desarrollado protocolos que transducen selectivamente OPC utilizando el lentivirus sobreexpresan tipo salvaje, proteínas negativas constitutivamente activos o dominantes antes de ser sembradas en las neuronas DRG. Esto nos permite interrogar específicamente las funciones de estas proteínas oligodendrogliales en la regulación de la mielinización. Los protocolos también pueden aplicarse en el estudio de otsus tipos de células, proporcionando de este modo un enfoque que permite la manipulación selectiva de las proteínas expresadas por un tipo de célula deseada, tales como los oligodendrocitos para el estudio selectivo de señalización y mecanismos de compensación. En conclusión, la combinación de la mielinización ensayo in vitro con los OPC lentiviral infección proporciona una herramienta estratégica para el análisis de los mecanismos moleculares implicados en la mielinización.
Introducción
La mielinización de los axones es crucial para la transmisión rápida y eficaz de los potenciales de acción en los sistemas nerviosos central y periférico. Las células especializadas, las células de Schwann en el sistema nervioso periférico y los oligodendrocitos en el sistema nervioso central, y se envuelven alrededor de los axones en ensheathe mielina, aislantes efectivamente el nervio y que facilitan la conducción saltatoria 1. El proceso de mielinización puede ser estudiada in vitro usando las neuronas ganglionares de la retina 2, nanofibras de ingeniería 3, o neuronas ganglionares de la raíz dorsal co-cultivadas con células de Schwann o 4 oligodendrocitos 5-7. El ensayo de mielinización in vitro es un modelo establecido para el estudio de la mielinización del sistema nervioso y se replica muchos de los procesos fundamentales que ocurren durante la mielinización in vivo 5-8. El ensayo consiste en la co-cultivo de las poblaciones purificadas de Ganglio de raíz dorsal (GRD) neuronas, con los OPC (para CNS mielinización) o células de Schwann (por PNS mielinización). En condiciones específicas de estas células mielinizantes ensheathe axones DRG en la ordenada y ultra verificado estructuralmente, hoja multi-lamelar de aislante membrana plasmática que expresan el mismo complemento de proteínas específicas de mielina presentes in vivo.
El modelo de célula más comúnmente utilizado de estudiar la mielinización del SNC in vitro es el co-cultivos de neuronas DRG y OPC, que han sido utilizados con éxito para estudiar el efecto que los factores exógenos tales como las neurotrofinas ejercen sobre la mielinización del SNC in vitro 5,6. Los factores exógenos tales como factores de crecimiento o inhibidores farmacológicos de moléculas pequeñas se han utilizado ampliamente para estudiar el papel de vías de señalización en la mielinización utilizando el DRG-OPC modelo de cocultivo 7,9. Sin embargo, en la configuración de co-cultivo mixto que contienen tanto las neuronas y oligodendrocitos, se mantuvo formalmente posible que cualquiera de los factores de crecimiento o la pharinhibidores macológico podrían haber ejercido efectos en tanto las neuronas y oligodendrocitos (OL) DRG. Este sí ofrece la capacidad de diseccionar específicamente las funciones que las proteínas expresadas únicamente por GRD o oligodendroglia ejerce sobre la mielinización utilizando este sistema celular dual. Para confirmar inequívocamente que la vía de señalización en oligodendroglial regula directamente la mielinización, la transducción lentiviral de OPC, antes de sembrar en las neuronas DRG para el ensayo de mielinización in vitro, ha demostrado ser una manera elegante de sobreexpresan tanto de tipo salvaje y las proteínas mutantes, así como expresión desmontables de proteínas constitutivamente expresadas por los oligodendrocitos. Así, este enfoque ofrece una vía para interrogar y manipular las vías de señalización dentro de los oligodendrocitos para el estudio de la mielinización 9,10 específicamente.
En este artículo analizamos los métodos que hemos desarrollado para sobreexpresan una proteína de interés de forma selectiva en los oligodendrocitos a través de un lentiviralenfoque para el estudio de la mielinización in vitro. La técnica comienza con la generación de vectores de expresión que contienen el gen de interés, ya sea en un tipo salvaje, forma negativa constitutivamente activa o dominante que luego se clonó posteriormente en el vector pENTR (pENTR L1-L2 pENTR4IRES2GFP). Este vector (que contiene el gen de interés), el donante promotor CMV (pENTR L4-R1 pENTR-pDNOR-CMV) y el lentivector 2K7 se combinan en una reacción enzimática para producir un vector que contiene el promotor CMV 2K7, el gen de interés, una sitio interno de entrada ribosomal y las buenas prácticas agrarias (Figura 1). Este constructo clonado 2K7 puerta de enlace combinada con la envoltura del virus PMD2.G y el paquete de virus pBR8.91 se puede co-transfectadas en células HEK293T para generar lentivirus que posteriormente se puede usar para transducir OPC. Una vez infectado con el lentivirus los OPCs expresan un alto nivel de la proteína de interés. Estos OPC pueden ser sembradas en cultivos de neuronas DRG y el efecto que la expresiónde altos niveles de la proteína deseada ejerce sobre la mielinización puede ser interrogado. Los co-cultivos se evaluaron para la expresión de proteínas de mielina por análisis de transferencia Western y se visualizaron para la formación de segmentos axonales mielinizadas por inmunocitoquímica.
Protocolo
NOTA: Todos los animales utilizados para este estudio eran de sexo mixto y se crió en el Animalario del Departamento de Anatomía y Anatomía Patológica y El Instituto de Neurociencias Florey y la Investigación en Salud Mental de la Universidad de Melbourne. Todos los animales procedimientos fueron aprobados por los Comités de Ética de Experimentación Animal de la Universidad de Melbourne.
1. Clonación de 2K7 LentiVector
- Antes de clonar el gen de interés en el vector lentiviral 2K7, subclonar el gen en el vector pENTR (3637 pb, resistente a la kanamicina) utilizando técnicas moleculares estándar 11. Utilice los sitios de restricción EcoRI y SacII para la subclonación.
- La amplificación de la 2K7 lentivector
- Transformar ADN lentivector 2K7 mezclando suavemente 100 ng de ADN plasmídico con las células competentes y se incuba en hielo durante 30 min.
- ADN de choque térmico / células competentes mezcla a 42 ° C durante 90 segundos y la placa en platos de agar LB que contienen tanto ampiciLLIN (100 g / ml) y cloranfenicol (15 mg / ml) a 37 ° C durante 16-18 h.
NOTA: El cloranfenicol se usa para prevenir la recombinación entre las repeticiones terminales largas. - Al día siguiente, crecer selecciona clones bacterianos en medio LB que contenía ampicilina (100 mg / ml) y cloranfenicol (15 mg / ml) a 37 ° C durante 16-18 h. Extraer el ADN usando un kit DNA maxiprep plásmido comercial según las instrucciones del fabricante.
- Sub-clonación de pENTR vector para 2K7 lentivector (Figura 1)
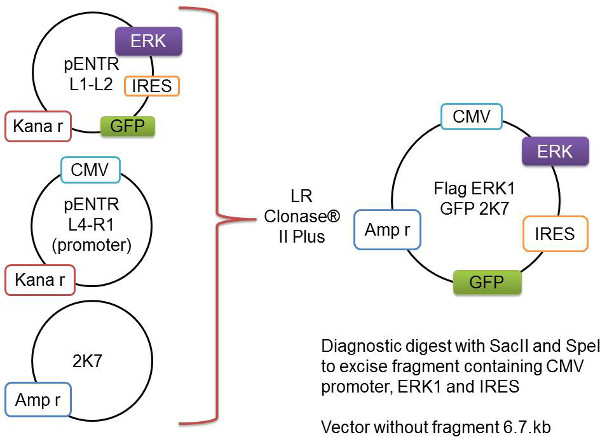
Figura 1:. Representación esquemática del proceso de recombinación de puerta de enlace El gen de interés, aquí representado como Flag-Erk1, se clonó en el vector pENTR L1-L2. Esto se añade al vector pENTR L4-R1 que contiene el promotor CMV y el vector 2K7 columna vertebral. Estos tres vectores se recombinan por la enzima LR Clonase II Plus ainsertar el gen de intereses y el promotor en el 2K7 vector de virus listo.- Agregue lo siguiente a un tubo de 1,5 ml y mezclar suavemente:
L4-R1 pENTR-pDNOR-CMV (promotor) (60 BNG) 1-2,5 l
L1-L2 pENTR4IRES2GFP (con el gen de interés) (80 BNG) 1-2,5 l
K7 lentivector (80 ng / l) 1 l
TE-buffer, pH 82-5 l
Total de 8 l - Eliminar mezcla de enzimas clonase de -80 ° C y descongelar en hielo durante 2 min. Asegúrese de que esta enzima es una parte alícuota de -80 ° C como la enzima ha reducido sustancialmente la eficacia de clonado con los ciclos de congelación-descongelación
- Añadir 2 l de enzima por reacción y mezclar bien a través de vórtice, y se incuba la mezcla de reacción clonasa a 23-25 ° C durante 6-24 h.
- Añadir 1 l de solución de proteinasa K (suministrado con el kit de enzima) a la mezcla de reacción clonase y se incuba durante 10 min a 37 ° C.
- Transformar mezcla de reacción clonasa en células competentes y crecer el selectocolonias en medio LB que contenía ampicilina (100 mg / ml) a 37 ° C durante 16-18 horas.
- Extraer y purificar el ADN utilizando un kit de DNA Miniprep plásmido comercial según las instrucciones del fabricante.
- Confirmar el ADN a través de la digestión usando las enzimas de restricción Spe I y Sac II y un tampón apropiado como por las instrucciones del fabricante para eliminar el fragmento que contiene el promotor y el gen de interés.
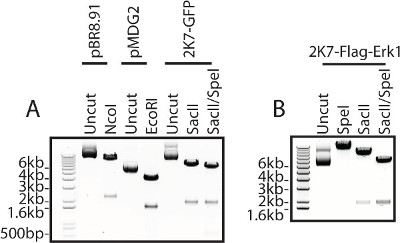
NOTA: Este paso es fundamental para comprobar si el ADN subclonado tiene el gen inserto correcto de intereses con el esqueleto del vector derecha. El tamaño del vector por sí mismo sin fragmento insertado es 6,7 kb. El tamaño del fragmento insertado liberada incluye tanto el promotor y el gen inserto de interés. Calcular los tamaños de los fragmentos esperados de la digestión para el gen específico a través de un programa de edición de secuencia de ADN, por ejemplo, APE - Un editor de plásmido. - Compruebe los tamaños de ambos esqueleto del vector de ADN y el fragmento liberado mediante la ejecución de un gel de agarosa al 1%en tampón TAE 1x (ver Tabla de soluciones de reserva) a 100 V.
- Amplificar el ADN confirmado por el crecimiento de bacterias en 500 ml de medio LB que contenía ampicilina (100 mg / ml) a 37 ° C durante 16-18 h.
- Extraer y purificar el ADN usando un kit DNA maxiprep plásmido comercial según las instrucciones del fabricante.
NOTA: maxiprep normalmente genera suficiente cantidad de ADN necesaria para la producción viral.
- Agregue lo siguiente a un tubo de 1,5 ml y mezclar suavemente:
- Repita los pasos 1.3.9-1.3.10 para amplificar los siguientes ADNs para preparaciones lentiviral: Sobre vector (PMD2.G, 6,1 kb), vector de paquetes (pBR8.91, 12,5 kb) y un vector lentiviral vacío como control (GFP -CMV-2K7, 8,7 kb).
2. 2K7 Producción Virus
NOTA: Día 1:
- En el día de la transfección, placa 32 millones de células HEK293 T en frasco T175 que contiene 25 ml de medio de células HEK293 T (véase la Tabla de soluciones de reserva). Alternativamente, la placa 16 millones de células del díaantes de la transfección se ejecuta si el tiempo ajustado en el día de la transfección.
NOTA: La transfección puede ser el mismo éxito en placas las células en el día de la transfección o antes del día. El uso de cualquiera de las dos alternativas, el punto crítico aquí es hacer que las células seguro de ser pegado hacia abajo en la superficie de placa de cultivo antes de la transfección.
NOTA: Día 2: - La transfección
- Antes de la transfección, diluir ADN a 1 mg / l en tampón TE que contiene Tris 10 mM pH 8, EDTA 1 mM pH 8 en agua desionizada.
- En un tubo de 50 ml, preparar una mezcla maestra (Tabla 1) para la transfección en el matraz T175. Añadir ADN para pre-calentado medio Eagle modificado por Dulbecco (DMEM) y mezclar bien por vortex, a continuación, añadir polietilenimina estéril (PEI) (véase la Tabla de soluciones de reserva) para evitar la precipitación prematura. ">
Tabla 1: Preparación de mezcla de transfección para 2K7 Virus.Vector trong> Concentración Volumen pMDG.2 1 mg / l 5 l pBR8.91 1 mg / l 15 l Vector 2K7 con GFP + gen de interés 1 mg / l 22 l Polietilenimina Estéril (PEI) 1 g / L 500 l DMEM 2100 l - Mezclar bien invirtiendo el 3-4x tubo e incubar durante 15 min a temperatura ambiente (RT).
- Realizar un cambio de medio completo en las células HEK293T. Aspirar los medios de cultivo de las células completamente y alimentar con medios de cultivo celular HEK293T pre-calentado (25 ml por matraz T175).
- Añadir la mezcla de transfección (ADN mezcla / PEI) gota a gota a la monocapa de células. Mueva suavemente para mezclar bien e incubar células transfectadas a 37 ° C, 5% de CO2, durante la noche.
NOTA: Día 3: - Para garantizar la transfección se realiza correctamente, compruebe la expresión de GFP 24 horas después de la transfección a través de un microscopio de fluorescencia.
NOTA: Más del 50% de las células que expresan GFP suele indicar una buena transfección.
NOTA: Día 4:
- Al 48 horas después de la transfección, recoger el sobrenadante viral y reemplazar con medios HEK293 T frescas (25 ml por matraz T175).
- Centrifugar el sobrenadante viral a 1140 xg durante 10 min a 4 ° C para eliminar los desechos de células del sobrenadante. Transferencia aclaró sobrenadante para tubo de 50 ml y se almacena a 4 ° C.
NOTA: Día 5:
- Centrifugar el sobrenadante viral a 1140 xg durante 10 min a 4 ° C para eliminar los desechos de células del sobrenadante. Transferencia aclaró sobrenadante para tubo de 50 ml y se almacena a 4 ° C.
- En 72 horas después de la transfección, recoger el segundo lote de sobrenadante viral. Repita el paso 2.3.1 y piscina 48 y 72 hr borran sobrenadantes.
- Para concentrar el virus, el sobrenadante viral se centrifuga a 170.000 xg durante 90 min a 4 ° C utilizando 30 ml tubos de ultracentrífuga.
- Descartar el sobrenadante y repetir el paso 2.6 hasta que todo el sobrenadante clarificado se ha centrifugado dejando un (invisible) de pellets de virus más precipitado PEI en la base del tubo.
- Para volver a suspender virus, añadir 500 l SATO medios de comunicación (véase la Tabla de soluciones de reserva) a los tubos de ultracentrífuga. Vortex durante 30 segundos unand raspar la base del tubo con una punta de pipeta para aflojar mecánicamente el virus. Repita este paso 6x con el fin de perder pellet viral.
- En común el virus se resuspendió en tubos de microcentrífuga y girar muy brevemente para eliminar PEI insoluble. Se filtra el sobrenadante a través de un filtro de 0,45 micras para eliminar las proteínas.
- Alícuota del virus en 20 l, 50 l, y alícuotas de 100 ly se almacena a -80 ° C.
3. Determinación del título viral en células HEK293T
- Para comprobar la expresión de la proteína de interés y para determinar la concentración viral óptima para los experimentos, añadir una serie de diluciones en serie de acciones viral (por ejemplo, 0, 5, 10, 20, 40, 80 l) para transducir células HEK293 T sembraron en placas de 6 pocillos, y la cultura durante 24 horas a 37 ° C, 5% de CO 2. Este protocolo genera típicamente virus que se puede utilizar en concentraciones que van de 1:50 a 1: 200 que lograr la expresión robusta del gen deinterés.
- 48 hr transducción viral posterior, las células HEK293T lisan en tampón TNE (ver Tabla de solución madre) con inhibidores de la proteasa.
- Enjuague pocillos dos veces con DPBS enfriado y luego añadir tampón TNE 150 l a cada pocillo.
- Lyse células pipeteando arriba y abajo 5-10x. Transferencia de lisados de células enteras para tubo de microcentrífuga de 1,5 ml y se incuba en hielo durante 15-30 min.
- Centrifugar los lisados a 4 ° C durante 30 min a velocidad máxima (20.000 xg) y la transferencia se aclaró el sobrenadante a un tubo nuevo para la determinación de proteínas por Bradford y análisis de transferencia western posterior.
- Determinar el nivel de expresión de la proteína de interés por Western blot estándar, mientras que el sondeo de anticuerpos frente a la proteína en sí y la etiqueta fundido (es decir, de la bandera). Utilice la dilución viral que produce células> 95% GFP + y sólida expresión de la proteína de interés para los experimentos.
4. Aislamiento y cultivo de los GRD (Figura 2 los pasos 1 y 2)
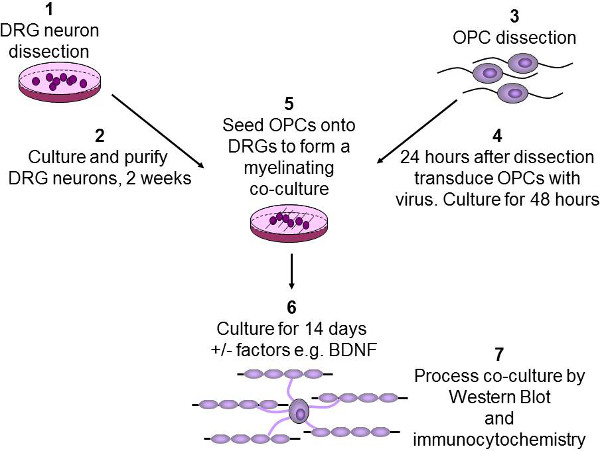
Figura 2: Diagrama esquemático. De la mielinización de ensayo in vitro neuronas DRG se disecan a partir de crías de rata P2-3, a continuación, purificados y cultivados durante dos semanas (1-2). OPCs se purificaron a partir de cerebros de ratas usando P7-9 immunopanning (3). OPCs resultan luego infectados por lentivirus y se cultivaron durante 48 horas (4). OPCs son entonces sembradas en GRD, y se añaden los factores de crecimiento de interés tales como BDNF (5). Co-cultivos se cultivaron durante 2 semanas para permitir que los OPC para diferenciar y mielinizan los axones (6). Por último, co-cultivos se lisan ya sea para el Western Blot o fijos para inmunocitoquímica (7).
NOTA: Día 1- 2 días antes de la disección:
- Escudo autoclave 22 mm x 22 mm cubreobjetos con poli-L-ornitina (0,5 mg / ml) en una placa de 6 pocillos, y se incuba a 37 ° C durante la noche.
- Al día siguiente, cubreobjetos de la capa de nuevo con laminina (20 g / ml en MEM) a 37 ° C durante la noche, aspirar el exceso y seco durante 20 minutos en campana de cultivo de tejido.
NOTA: Día 2: La disección y aislamiento de los GRD: - Cachorros Sacrificio P2-P3 de rata 12x por transección cervical.
- Retire la piel que recubre el músculo de la parte posterior del animal. Utilice Vannas tijeras para abrir agujero vertebral y el uso de fórceps para sacar con cuidado la médula espinal.
- Arranca GRD que se encuentran en medio de las columnas vertebrales y cortar las fibras nerviosas de la médula adjuntos, luego coloque GRD en un plato de Petri de 33 mm que contiene 3 ml L-15 medios de comunicación. Por lo general, es posible reunir entre 8 y 12 DRG de cada lado de la médula espinal
- Transferencia de recogida GRD junto con los medios de comunicación L-15 en un tubo de 15 ml, se centrifuga a 180 xg durante 5 min.
- Aspiradoel sobrenadante, añadir 2 ml 0,25% de tripsina a DRG gránulos y se incuba a 37 ° C durante 30 min.
- Añadir 5 ml de medio M1 (véase la Tabla de solución madre) a los pellets de DRG para detener la tripsina, y se centrifuga a 180 xg durante 3 min a TA.
- Aspirar el sobrenadante, volver a suspender el sedimento en 2 ml de medio M1 precalentado (no con factores de crecimiento). Se tritura el pellet ganglios pipeteando 50x o hasta los ganglios se dispersan. Suspensión de células se centrifuga a 180 xg durante 3 min.
- Resuspender disociado neuronas en medios M1 y la placa en una placa de 6 pocillos a 5 ganglios por 100 l por pocillo.
- Para eliminar la proliferación de células no neuronales, después de un mínimo de 4 h de incubación para facilitar la fijación, retirar el medio M1 y alimentar las neuronas con los medios de comunicación M2 (véase la Tabla de solución madre). Cultura neuronas DRG en presencia de NGF (100 ng / ml) para purificar una cultura de GRD TrkA-expresión de NGF-dependientes. Alternativamente, utilizar BDNF (100 ng / ml) para purificar TrkB-expres BDNF dependientecantar GRD.
- Mantener las neuronas en los medios de comunicación M1 (+ NGF o BDNF a 100 ng / ml) que alternan con antimitótico M2 medios de comunicación durante 2 semanas siguientes.
- Mantener medio M1 (+ NGF o BDNF a 100 ng / ml) en días: 4-6, 8-10, 12-14 y M2 medios de comunicación (+ NGF o BDNF a 100 ng / ml) con FDU y uridina en los días 1 a 4, 6-8, y 10-12. Se requiere un mínimo de 3 ciclos de purificación de los medios de comunicación M2.
- Mantener GRD en M1 solo durante una semana más después de completar el ciclo de antimitótico 2 semanas. Cambie medios M1 cada 2-3 días.
5. Aislamiento y cultivo de los OPC (Figura 2 paso 3)
NOTA: Día 1- 2 días antes de la disección:
- Coat 10 cm placas de cultivo de tejido con poli-D-lisina (PDL, 10 g / ml en agua desionizada esterilizada) a 4 ° C durante la noche.
NOTA: Día 2: 1 día antes de la disección: - Lave las placas de PDL con agua destilada esterilizada para 3x. Deje secar durante ~ 6 h en campana de cultivo de tejidos. Si no se utiliza inmediatamente, envuelva y tiendapara un máximo de 4 semanas a 4 ° C.
- Preparar placas de anticuerpos secundarios para immunopanning. Para 1 disección de cerebro, preparar planchas de IgG 2x (por Ran2 anticuerpo para eliminar astrocitos y anticuerpo O1 para eliminar premielinizantes oligodendrocitos), con 45 l de IgG de cabra α ratón en DPBS (15 ml) por 10 cm placa de Petri; 1x placa de IgM para (anticuerpo O4 para la selección de células precursoras de oligodendrocitos), 45 l de cabra α IgM de ratón en DPBS (15 ml) por 10 cm placa de Petri.
NOTA: Día 3: Día de la disección: - Añadir 200 unidades de papaína en 10 ml de tampón de papaína (Tabla 2), y calentar a 37 ° C hasta que el buffer se vuelve claro.
Tabla 2: Preparación de tampón de papaína.Concentración para 250 ml La concentración final EBSS social 10x 25 ml 1x MgSO 4 100 mM 2,5 ml 1 mM Glucosa 30% 3 ml 0,46% EGTA 0,5 M 1 ml 2 mM NaHCO 3 1 M 6,5 ml 26 mM Aumentar el volumen hasta 250 ml con agua desionizada y se esteriliza con filtro - Lave todas las placas de anticuerpos secundarias con DPBS de 3x.
- Verter el anticuerpo Ran2 y O1 sobre las placas de IgG y el hibridoma O4 a la placa de IgM. Incubar todas estas placas de anticuerpo primario durante más de 2 horas a RT.
- Diseccionar un cerebro de una rata P7. Decapitar a un cachorro con tijeras afiladas y quitar la piel que cubre el cráneo con tijeras.
- Corte alrededor del cráneo del lóbulo occipital, el lóbulo temporal y el lóbulo frontal. Eliminar cerebro de cráneo usando un fórceps y suavemente transferirlo a un 35-mm placa de Petri con 1 ml de DPBS.
- Cortar el cerebro en pedazos pequeños o menos con unas tijeras o una cuchilla estéril.
- Filtro pre-calentado tampón la papaína (10 ml) en un nuevo tubo de 15 ml que contiene unos granos de L-cisteína, a continuación, añadir 200 l de ADNasa (12.500 U / ml) al tampón de la papaína se filtró.
- Vierta el buffer papaína en los tejidos del cerebro en cubitos; incubar a 37 ° C durante 90 min.
- Transferencia Gentlly disociarse los tejidos del cerebro en el tubo de 50 ml con una pipeta 25 ml yse deja reposar.
- Retire búfer papaína, añadir 2 ml Mín Ovo (ovomucoide) a los tejidos cerebrales y titurate 5-10x con la pipeta para romper trozos de tejidos cerebrales, dejar reposar y quitar la parte superior 2 ml de sobrenadante a un nuevo tubo.
- Añadir otra 2 ml Mín Ovo a los tejidos cerebrales y repita el paso 5.11 hasta que no haya trozos de tejido permanecen. La trituración puede obtener cada vez más agresivo.
- Centrifugar la suspensión celular disociada durante 15 min a 200 x g. Aspirar el sobrenadante y resuspender el sedimento celular en 10 ml Hola Ovo y centrifugar durante 15 min a 200 x g.
- Lave primera placa immunopanning (Ran 2 placa) con DPBS de 3x. Aspirar el sobrenadante, Resuspender las células en 10 ml de tampón panning (Tabla 2) y se vierte a la primera placa de immunopanning (Ran 2 plato). Se incuban las células durante 15 min a RT.
- Lave la segunda placa immunopanning (placa O1) con DPBS de 3x.
- Después de la incubación en la primera placa de immunopanning, la punta de la suspensión celular en segundoplaca immunopanning (placa O1), Enjuagar las células sueltas de la superficie de la placa con 1-3 ml de paneo tampón y extraer con la pipeta a la placa O1. Se incuban las células durante 15 min a RT.
- Lavar la placa immunopanning tercero (placa O4). Esta placa es la placa de selección positiva en los OPCs unen su superficie. Lavar la placa O4 con DPBS de 3x.
- Después de la incubación en la segunda placa immunopanning, transferir las células a la placa immunopanning final (placa O4), incubar las células durante 45 min a RT. Este paso será seleccionar O4 + OPC.
- Aspirar el sobrenadante de la última placa immunopanning (placa O4) y enjuagar la placa con EBSS de 6x.
- Para eliminar los OPC de la placa, se incuban las células con 5 ml de tibia 0,05% de tripsina-EDTA diluido 1:10 con EBSS a 37 ° C durante 8 min.
- Añadir 5 ml de 30% de FBS (hecho en EBSS) para neutralizar la tripsina. Eliminar las células fuera de la superficie de la placa por pipeteado durante aproximadamente 50x.
- Transfiera toda celularla suspensión a un tubo nuevo y se centrifuga durante 15 min a 200 x g.
- Desechar el sobrenadante y resuspender el sedimento celular en 1 ml pre-calentado medios SATO, seguido por el recuento de células. Disección de un cerebro puede producir 1,5-2.000.000 OPC.
- Células placa sobre placas recubiertas de PDL seco a una densidad de entre 1 x 10 5 y 5 x 10 5 por 10 cm de placa con los medios de comunicación en SATO (10 ml) que contienen el factor neurotrófico ciliar (CNTF, 10 ng / ml), factor de crecimiento derivado de plaquetas (PDGF, 10 ng / ml,), la neurotrofina 3 (NT-3, 1 ng / ml), y la forskolina (4,2 mg / ml) 2,12. OPCs cultivo a 37 ° C, 8% de CO 2.
6. OPCs de transducción
- OPCs primarios Cultura en SATO medios de comunicación (10 ml / placa de 10 cm) con CNTF (10 ng / ml), PDGF (10 ng / ml), NT3 (1 ng / ml), y la forskolina (4,2 mg / ml) a 37 ° C, 8% de CO 2 durante 24 horas después de la disección.
- Completamente aspirar fuera el medio de cultivo OPC, las células de alimentación con recién hecha medios SATO (10 ml) Con factores de crecimiento (véase más arriba en el paso 6.1).
- Añadir virus a las OPC a la concentración óptima determinada por el paso 3 (Figura 2, etapa 4), los OPC de cultivo para un 48 horas más.
7. Siembra OPC para mielinizantes Co-culturas (Figura 2, los pasos 5 y 6)
- Para eliminar los OPC fuera de la superficie, primero placas OPC enjuague con 8 ml EBSS dos veces, luego incubar las células con 5 ml de agua tibia 0,05% de tripsina-EDTA diluido 1:10 con EBSS a 37 ° C durante 2 min.
- Neutralizar la tripsina con 30% FBS en EBSS (5 ml) y eliminar las células de la placa por pipeteado. Transferir la suspensión celular a un tubo de 15 ml, se centrifuga a 180 xg durante 15 min a TA.
- Aspirar el sobrenadante y resuspender el sedimento celular en 1 ml pre-calentado medios SATO seguido por el recuento de células.
- Antes de la siembra OPC, medios completamente aspirado desde la placa de cultivo DRG. Semilla suavemente 200.000 OPCs gota a gota a las neuronas DRG como anteriormente descritos 4-6.
NOTA:El volumen total de la siembra OPC debe ser inferior a 200 l por 22 mm cubreobjetos. - Deja células para resolver sin mover la placa durante 10 min en la campana de cultivo de tejidos, luego de arriba suavemente con 1 ml pre-calentado medios SATO por pocillo.
- Replate los OPCs hermanas restantes con medios SATO con factores de crecimiento (véase el paso 6.1). Utilice estas OPCs hermanas para verificar la expresión de la proteína de interés.
- Después de 24 h, reemplazar los medios de comunicación SATO con los medios de co-cultivo (2 ml / pocillo) que contiene medios SATO (sin factores) y neurobasal (v / v) con 1% B27. Mantener los co-cultivos durante 14 días con cambio de medio cada 2-3 días.
- Evaluar los co-cultivos para la expresión de proteínas de mielina por análisis de transferencia Western y visualizar para la formación de segmentos axonales mielinizadas por doble inmunotinción con anticuerpos contra marcadores de proteína básica de mielina y los marcadores neuronales 4-6.
Resultados
La bandera de etiquetado de señales relacionadas quinasa 1 (Bandera-Erk1) construcción utilizada para la producción de lentivirus es verificado por enzimas de restricción digerir de las construcciones utilizadas, incluyendo tanto las construcciones 2K7 y las construcciones de embalaje y accesorios requeridos para la producción de virus extracelular (Figura 3) .
 ...
...
Discusión
La mielinización de los axones es un proceso crucial para la función óptima de los sistemas nerviosos central y periférico de vertebrados. La generación y el mantenimiento de los axones mielinizados es un proceso complejo y coordinado de las interacciones moleculares entre neuronal, glial (a partir de células de Schwann o oligodendrocitos) y proteínas de la matriz extracelular. La importancia y la aplicabilidad de este protocolo es que permite la manipulación de las proteínas en un tipo celular específico dent...
Divulgaciones
The authors declare that there is no conflict of interest regarding this research.
Agradecimientos
This work was supported by the Australian National Health and Medical Research Council (NHMRC fellowship #454330 to JX, project grant #628761 to SM and APP1058647 to JX), Multiple Sclerosis Research Australia (MSRA #12070 to JX), the University of Melbourne Research Grant Support Scheme and Melbourne Research CI Fellowship to JX as well as Australia Postgraduate Scholarships to HP and AF. We would like to acknowledge the Operational Infrastructure Scheme of the Department of Innovation, Industry and Regional Development, Victoria Australia.
Materiales
| Name | Company | Catalog Number | Comments |
| 2K7 lentivector | Kind gift from Dr Suter9 | ||
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100mg | |
| Alexa Fluor 488 Goat anti-mouse IgG | Jackson Immunoresearch | 115545205 | |
| Alexa Fluor 488 goat anti-rabbit IgG (H+L) | Life Technologies | A11008 | |
| Alexa Fluor 594 goat anti-mouse IgG (H+L) | Life Technologies | A11005 | |
| Alexa Fluor 594 goat anti-rabbit IgG (H+L) | Life Technologies | A11012 | |
| Ampicillin | Sigma-Aldrich | A9518-5G | |
| B27 - NeuroCul SM1 Neuronal Supplement | Stem Cell Technologies | 5711 | |
| BDNF (Human) | Peprotech | PT450021000 | |
| Biotin (d-Biotin) | Sigma Aldrich | B4639 | |
| Bradford Reagent | Sigma Aldrich | B6916-500ML | |
| BSA | Sigma Aldrich | A4161 | |
| Chloramphenicol | Sigma-Aldrich | C0378-100G | |
| CNTF | Peprotech | 450-13020 | |
| DAKO fluoresence mounting media | DAKO | S302380-2 | |
| DMEM, high glucose, pyruvate, no glutamine | Life Technologies | 10313039 | |
| DNase | Sigma-Aldrich | D5025-375KU | |
| DPBS | Life Technologies | 14190250 | |
| DPBS, calcium, magnesium | Life Technologies | 14040182 | |
| EBSS | Life Technologies | 14155063 | |
| EcoRI-HF | NEB | R3101 | |
| Entry vectors for promoter and gene of interest | Generate as per protocols 1-2 | ||
| Fetal Bovine Serum | Sigma-Aldrich | 12003C | |
| Forskolin | Sigma Aldrich | F6886-50MG | |
| Glucose (D-glucose) | Sigma-Aldrich | G7528 | |
| Glycerol | Chem Supply | GL010-500M | See stock solutions |
| Goat Anti-Mouse IgG | Jackson ImmunoResearch | 115005003 | |
| Goat Anti-Mouse IgM | Jackson ImmunoResearch | 115005020 | |
| Goat Anti-Rat IgG | Jackson ImmunoResearch | 112005167 | |
| Hoechst 33342 | Life Technologies | H3570 | |
| Igepal | Sigma Aldrich | I3021-100ML | |
| Insulin | Sigma Aldrich | I6634 | |
| Kanamycin | Sigma-Aldrich | 60615 | |
| Laminin | Life Technologies | 23017015 | |
| LB Medium | See stock solutions | ||
| LB-Agar | See stock solutions | ||
| L-Cysteine | Sigma-Aldrich | C-7477 | |
| Leibovitz's L-15 Medium | Life Technologies | 11415064 | |
| L-Glutamate | Sigma-Aldrich | G1626 | |
| L-Glutamine- 200 mM (100x) liquid | Life Technologies | 25030081 | |
| LR Clonase II Plus enzyme | Life Technologies | 12538-120 | |
| MEM, NEAA, no Glutamine | Life Technologies | 10370088 | |
| Mouse α βIII Tubulin | Promega | G7121 | |
| Mouse αMBP (monoclonal) | Millipore | MAB381 | |
| Na pyruvate | Life Technologies | 11360-070 | |
| NAC | Sigma Aldrich | A8199 | |
| NcoI-HF | NEB | R3193S | |
| NEBuffer 4 | NEB | B7004S | |
| Neurobasal medium | Life Technologies | 21103049 | |
| NGF (mouse) | Alomone Labs | N-100 | |
| NT-3 | Peprotech | 450-03 | |
| O1 antibody - Mouse anti-O1 | Millipore | MAB344 | Alternative if O1 hybridoma cells are unavailable |
| O1 hybridoma cells | Conditioned medium containing anti-O1 antibody to be used for immunopanning | ||
| O4 antibody - Mouse anti-O4 | Millipore | MAB345 | Alternative if O4 hybridoma cells are unavailable |
| O4 hybridoma cells | Conditioned medium containing anti-O4 antibody to be used for immunopanning | ||
| Competent cells | Life Technologies | A10460 | |
| One Shot Stbl competent cells | Life Technologies | C7373-03 | |
| Papain Suspension | Worthington/Cooper | LS003126 | |
| pBR8.91 | Kind gift from Dr Denham10 | ||
| PDGF-AA (Human) | Peprotech | PT10013A500 | |
| Penicillin-streptomycin 100x solution | Life Technologies | 15140122 | |
| pENTRY4IRES2GFP | Invitrogen | 11818-010 | |
| pMD2.G | Addgene | 12259 | |
| Poly-D-lysine | Sigma | P6407-5MG | |
| Polyethylenimine (PEI) | Sigma-Aldrich | 408727-100ML | |
| Poly-L-ornithine | Sigma Aldrich | P3655 | |
| Progesterone | Sigma Aldrich | P8783 | |
| Protease inhibitor tablet (Complete mini) | Roche | 11836153001 | |
| Proteinase K | Supplied with Clonase enzyme | ||
| Putrescine | Sigma Aldrich | P-5780 | |
| Rabbit α neurofilament | Millipore | AB1987 | |
| Rabbit αMBP (polyclonal) | Millipore | AB980 | |
| Ran2 hybridoma cells | ATCC | TIB-119 | Conditioned medium containing anti-Ran2 antibody to be used for immunopanning |
| Rat anti CD140A/PDGFRa antibody | BD Pharmingen | 558774 | |
| SacII | NEB | R0157 | |
| SOC medium | Supplied with competent bacteria | ||
| Sodium selenite | Sigma Aldrich | S5261 | |
| Spe I | NEB | R0133S | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Buffer | NEB | B0202S | |
| TE buffer pH8 | See stock solutions | ||
| TNE lysis buffer | |||
| Trace Elements B | Cellgro | 99-175-CI | |
| Transferrin (apo-Transferrin human) | Sigma-Aldrich | T1147 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Trypsin | Sigma-Aldrich | T9201-1G | |
| Trypsin Inhibitor From Chicken Egg White | Roche | 10109878001 | |
| Trypsin-EDTA (1x), phenol red (0.05%) | Life Technologies | 25300-054 | |
| Unconjugated Griffonia Simplicifolia Lectin BSL-1 | Vector laboratories | L-1100 | |
| Uridine | Sigma-Aldrich | U3003-5G |
Referencias
- Baumann, N., Pham-Dinh, D. Biology of oligodendrocyte and myelin in the mammalian central nervous system. Physiol Rev. 81 (2), 871-927 (2001).
- Watkins, T. A., Emery, B., Mulinyawe, S., Barres, B. A. Distinct stages of myelination regulated by gamma-secretase and astrocytes in a rapidly myelinating CNS coculture system. Neuron. 60 (4), 555-569 (2008).
- Lee, K., et al. MDGAs interact selectively with neuroligin-2 but not other neuroligins to regulate inhibitory synapse development. Proc Natl Acad Sci U S A. 110 (1), 336-341 (2013).
- Xiao, J., et al. BDNF exerts contrasting effects on peripheral myelination of NGF-dependent and BDNF-dependent DRG neurons. J Neurosci. 29 (13), 4016-4022 (2009).
- Chan, J. R., et al. NGF controls axonal receptivity to myelination by Schwann cells or oligodendrocytes. Neuron. 43 (2), 183-191 (2004).
- Xiao, J., et al. Brain-Derived Neurotrophic Factor Promotes Central Nervous System Myelination via a Direct Effect upon Oligodendrocytes. Neurosignals. 18 (3), 186-202 (2010).
- Lundgaard, I., et al. Neuregulin and BDNF induce a switch to NMDA receptor-dependent myelination by oligodendrocytes. PLoS Biology. 11 (12), e1001743 (2013).
- Kleitman, N., W, P. M., Bunge, R. P. . Tissue culture methodes for the study of myelination. , (1991).
- Xiao, J., et al. Extracellular signal-regulated kinase 1/2 signaling promotes oligodendrocyte myelination in vitro. J Neurochem. 122 (6), 1167-1180 (2012).
- Wong, A. W., Xiao, J., Kemper, D., Kilpatrick, T. J., Murray, S. S. Oligodendroglial expression of TrkB independently regulates myelination and progenitor cell proliferation. The Journal of Neuroscience. 33 (11), 4947-4957 (2013).
- Li, Z., et al. Molecular cloning, Characterization and Expression of miR-15a-3p and miR-15b-3p in Dairy Cattle. Molecular and Cellular Probes. , (2014).
- Emery, B., et al. Myelin gene regulatory factor is a critical transcriptional regulator required for CNS myelination. Cell. 138 (1), 172-185 (2009).
- Murai, K., et al. Nuclear receptor TLX stimulates hippocampal neurogenesis and enhances learning and memory in a transgenic mouse model. Proc Natl Acad Sci U S A. 111 (25), 9115-9120 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados