É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Produção e Uso de Lentivírus a seletivamente transduzem Primária Oligodendrocyte células precursoras para
Neste Artigo
Resumo
Here we present protocols that offer a flexible and strategic foundation for virally manipulating oligodendrocyte precursor cells to overexpress proteins of interest in order to specifically interrogate their role in oligodendrocytes via the in vitro model of central nervous system myelination.
Resumo
Mielinização é um processo complexo que envolve ambos os neurónios e as células formadoras de mielina da glia, oligodendrócitos no sistema nervoso central (SNC) e células de Schwann no sistema nervoso periférico (SNP). Usamos um ensaio in vitro mielinização, um modelo estabelecido para o estudo da mielinização do SNC in vitro. Para fazer isso, as células precursoras de oligodendrócitos (OPCs) são adicionados aos gânglio da raiz primária roedor dorsal (DRG) neurónios purificados para formar myelinating co-culturas. A fim de interrogar especificamente os papéis que determinadas proteínas expressas pelos oligodendrócitos exercer sobre mielinização nós desenvolvemos protocolos que transduzem seletivamente OPCs usando o lentivírus overexpressing tipo selvagem, proteínas negativas constitutivamente ativos ou dominantes antes de serem semeadas em neurônios do GRD. Isso nos permite interrogar especificamente as funções destas proteínas na regulação oligodendrogliais mielinização. Os protocolos podem também ser aplicado no estudo de otos tipos de células, proporcionando assim uma abordagem que permite a manipulação selectiva de proteínas expressas por um tipo de célula desejado, tais como oligodendrócitos orientada para o estudo de mecanismos de sinalização e de compensação. Em conclusão, a combinação de ensaio in vitro com a mielinização lentivirais infectados OPCs fornece uma ferramenta estratégica para a análise dos mecanismos moleculares envolvidos na mielinização.
Introdução
Mielinização de axónios é crucial para a transmissão rápida e eficiente do potencial de acção em ambos os sistemas nervosos central e periférico. As células especializadas, as células de Schwann no sistema nervoso periférico e oligodendrócitos no sistema nervoso central, enrole e embainham axônios em mielina, efetivamente isolamento do nervo e que facilitam a condução saltatory 1. O processo de mielinização pode ser estudada in vitro usando neurónios ganglionares da retina 2, 3, nanofibras modificadas ou neurónios do gânglio da raiz dorsal de co-cultivados quer com células de Schwann 4 ou oligodendrócitos 5-7. O ensaio de mielinização in vitro é um modelo estabelecido para o estudo da mielinização do sistema nervoso e que replica muitos dos processos fundamentais que ocorrem durante a mielinização in vivo 5-8. O teste envolve a co-cultura das populações puras de gânglio de raiz dorsal (DRG) neurônios, com OPCs (para CNS mielinização) ou células de Schwann (por PNS) mielinização. Sob condições específicas destas células mielinizantes embainham axónios DRG na ordenada, ultra-estruturalmente verificado, folha multi-lamelar de isolamento membrana plasmática que expressam o mesmo complemento de proteínas específicas de mielina presentes in vivo.
O modelo de célula mais vulgarmente utilizado de estudar a mielinização do SNC in vitro é a co-culturas de neurónios DRG e OPCs, que têm sido utilizados com êxito para estudar o efeito de que os factores exógenos, tais como as neurotrofinas exercer sobre a mielinização do SNC in vitro 5,6. Factores exógenos, tais como factores de crescimento ou inibidores farmacológicos de moléculas pequenas têm sido amplamente utilizado para estudar o papel das vias de sinalização na mielinização DRG utilizando o modelo de co-cultura OPC-7,9. No entanto, nas configurações mistos de co-cultura que contêm ambos os neurónios e os oligodendrócitos, manteve-se formalmente possível que ambos os factores de crescimento ou a farmacovigilâninibidores macological poderiam ter exercido efeitos sobre ambos os neurônios e oligodendrócitos (OL) DRG. Esta não oferecem a capacidade de dissecar especificamente os papéis que as proteínas expressas apenas por DRGs ou oligodendroglia exerce sobre mielinização usando este sistema de célula dual. Para confirmar inequivocamente que a via de sinalização em oligodendroglial regula directamente a mielinização, transdução lentiviral de OPCs, antes da semeadura sobre neurónios DRG para o ensaio de mielinização in vitro, provou ser uma maneira elegante para sobre-expressar tanto o tipo selvagem e as proteínas mutantes, assim como knockdown expressão de proteínas expressas constitutivamente por oligodendrócitos. Assim, esta abordagem oferece uma avenida para interrogar especificamente e manipular as vias de sinalização dentro oligodendrócitos para estudar myelination 9,10.
Neste artigo, apresentamos métodos que desenvolvemos para superexpressão de uma proteína de interesse seletivamente em oligodendrócitos através de um lentivírusabordagem para estudar a mielinização in vitro. A técnica começa com a geração de vectores de expressão contendo o gene de interesse, quer seja numa tipo selvagem, forma constitutivamente activa ou negativo dominante, que são então subsequentemente clonado no vector pENTR (pENTR L1-L2 pENTR4IRES2GFP). Este vector (contendo o gene de interesse), o promotor de CMV dador (pENTR L4-R1-pENTR pDNOR-CMV) e lentivector 2K7 são combinados numa reacção de enzima para produzir um vector de 2K7 contendo promotor de CMV, o gene de interesse, uma local interno de entrada ribossomal e GFP (Figura 1). Este constructo gateway 2K7 clonado combinado com o envelope do vírus, e o pacote PMD2.G vírus pBR8.91 podem ser co-transfectados em células HEK293T para gerar lentivírus que pode subsequentemente ser utilizado para transduzir OPCs. Uma vez infectadas com lentivírus os OPCs expressam um alto nível da proteína de interesse. Estes OPCs podem então ser semeadas em culturas de neurônios DRG eo efeito que a expressãode níveis elevados da proteína desejada exerce sobre a mielinização pode ser interrogada. As co-culturas são avaliadas para a expressão da proteína de mielina por análise Western blot e visualizados para a formação de segmentos axonais mielinizadas por imunocitoquímica.
Protocolo
NOTA: Todos os animais utilizados para este estudo eram do sexo misto e criados no Biotério do Departamento de Anatomia e Patologia e do Instituto de Neurociências Florey e Pesquisa em Saúde Mental da Universidade de Melbourne. Todos os procedimentos com animais foram aprovados pelos Comitês de Ética em Experimentação Animal da Universidade de Melbourne.
1. Clonagem de 2K7 Lentivector
- Antes da clonagem do gene de interesse no vector lentiviral 2K7, subclonar o gene no vector pENTR (3637 pb, resistente à canamicina) utilizando técnicas de biologia molecular padrão 11. Use os sítios de restrição EcoRI e SacII para a subclonagem.
- A amplificação do 2K7 Lentivector
- Transformação de ADN lentivector 2K7 misturando suavemente 100 ng de DNA plasmídeo com as células competentes e incubar em gelo durante 30 min.
- O choque térmico de ADN / células competentes mistura a 42 ° C durante 90 seg e placa-los em placas de LB-agar contendo tanto ampicillin (100 ug / ml) e cloranfenicol (15? g / ml) a 37 ° C durante 16-18 horas.
NOTA: O cloranfenicol é utilizado para evitar a recombinação entre as repetições terminais longas. - No dia seguinte, cresça clones bacterianos seleccionados em meio LB contendo ampicilina (100 ug / ml) e cloranfenicol (15? G / ml) a 37 ° C durante 16-18 horas. Extrai-se o ADN utilizando um plasmídeo comercial ADN Maxiprep kit, conforme as instruções do fabricante.
- Sub-clonagem do vector pENTR para 2K7 Lentivector (Figura 1)
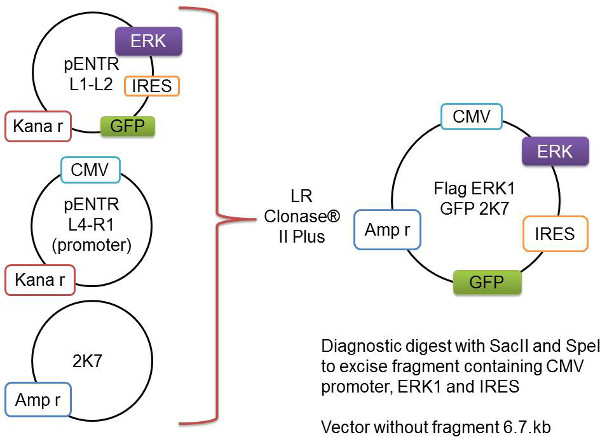
Figura 1:. Representação esquemática do processo de recombinação do gateway O gene de interesse, aqui representada como Flag-Erk1, é clonado no vector pENTR L1-L2. Este é adicionado ao vector pENTR L4-R1 contendo o promotor de CMV e o vector de espinha dorsal 2K7. Estes três vetores são recombinados pela enzima LR clonase II Plus parainserir o gene de interesse e o promotor para o vector de vírus 2K7-pronto.- Adicione o seguinte a um tubo de 1,5 ml e misture delicadamente:
L4-R1 pENTR-pDNOR-CMV (promotor) (60 BNG) 1-2,5 ul
L1-L2 pENTR4IRES2GFP (com gene de interesse) (80 BNG) 1-2,5 ul
K7 lentivector (80 ng / mL) 1 ul
TE-tampão, pH 82-5 ul
Total de 8 ul - Remover mistura de enzima clonase de -80 ° C e descongelar em gelo durante 2 min. Assegure-se que esta enzima é uma nova porção de -80 ° C, tal como a enzima reduziu substancialmente a eficiência da clonagem com ciclos de congelamento e descongelamento repetidos
- Adicionar 2 mL de enzima por reacção e misturar bem por meio de vórtice, e incubar a mistura de reacção em clonase 23-25 ° C durante 6-24 h.
- Adicionar 1 ml de solução de proteinase K (fornecido com o kit de enzima) para a mistura de reacção clonase e incubar durante 10 min a 37 ° C.
- Transforme mistura de reação clonase em células competentes e fazer crescer o selectcolónias em meio de LB contendo ampicilina (100 ug / ml) a 37 ° C durante 16-18 horas.
- Extrair e purificar ADN de plasmídeo utilizando um kit comercial de ADN miniprep de acordo com as instruções do fabricante.
- Confirmar o ADN através de digestão com as enzimas de restrição Spe I e Sac II e um tampão apropriado como por instruções do fabricante, para remover o fragmento que contém o promotor e o gene de interesse.
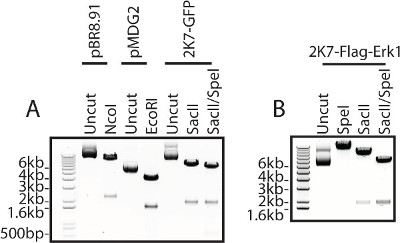
NOTA: Este passo é crítico para verificar se o ADN subclonados tem o gene de interesse inserto correcto com o esqueleto do vector direita. O tamanho do vector sem fragmento inserido em si é de 6,7 kb. O tamanho do fragmento inserido libertado inclui tanto o promotor e o gene da inserção de interesse. Calcular os tamanhos dos fragmentos esperados do Digest para o gene específico através de um programa de edição de sequência de DNA, por exemplo, APE - Um editor de plasmídeo. - Confira os tamanhos de tanto DNA vetor backbone eo fragmento liberado pela execução de um gel de agarose a 1%em tampão 1x TAE (ver Tabela de soluções de ações) a 100 V.
- Amplificar o ADN confirmada por bactérias crescendo em 500 ml de meio LB contendo ampicilina (100 ug / ml) a 37 ° C durante 16-18 horas.
- Extrair e purificar ADN de plasmídeo utilizando um kit comercial de ADN Maxiprep, conforme as instruções do fabricante.
NOTA: Maxiprep gera tipicamente uma quantidade suficiente de ADN necessário para a produção viral.
- Adicione o seguinte a um tubo de 1,5 ml e misture delicadamente:
- Repita os passos 1.3.9-1.3.10 para amplificar os seguintes DNAs para preparações de lentivírus: vetor Envelope (PMD2.G, 6,1 kb), vetor Package (pBR8.91, 12,5 kb), e um vetor lentivírus vazio como um controle (GFP -CMV-2K7, 8,7 kb).
2. 2K7 Virus Produção
NOTA: Dia 1:
- No dia da transfecção, placa 32 milhões de células T HEK293 em frasco contendo 25 ml T175 HEK293 T mídia celular (veja a Tabela das soluções de reserva). Alternativamente, placa 16 milhões de células no diaantes da transfecção se o tempo corre apertado no dia da transfecção.
NOTA: A transfecção pode ser igualmente bem sucedida por cultivo de células no dia da transfecção ou antes do dia. Usando qualquer um dos dois alternativas, o ponto crítico aqui é para tornar as células certeza ser fixado na superfície de placa de cultura antes da transfecção.
NOTA: Dia 2: - Transfection
- Antes da transfecção, dilui-se ADN de 1 ug / ul em tampão TE que contém 10 mM de Tris pH 8, 1 mM EDTA pH 8, em água desionizada.
- Num tubo de 50 ml, preparar uma mistura principal (Tabela 1) para a transfecção no balão T175. Adicionar ADN para pré-aquecido de Eagle Modificado por Dulbecco (DMEM) e misturar bem por vórtice, e depois adicionar estéril polietilenimina (PEI) (ver a Tabela de soluções de reserva) para evitar a precipitação prematura. ">
Tabela 1: Preparando mix transfecção para 2K7 Virus.Vetor trong> Concentração Volume pMDG.2 1 ug / uL 5 ul pBR8.91 1 ug / uL 15 ul Vector 2K7 com GFP + gene de interesse 1 ug / uL 22 ul Estéril polietilenimina (PEI) 1 g / L 500 ul DMEM 2.100 ul - Misturar bem invertendo o tubo 3-4 x e incubar durante 15 min à temperatura ambiente (TA).
- Realize uma mudança de mídia completo em células HEK293T. Aspirar fora do meio de cultura de células completamente e se alimentam com pré-aquecido meio de cultura celular HEK293T (25 ml por frasco T175).
- Adicione a mistura de transfecção (DNA mistura / PEI) gota a gota para as células em monocamada. Mova suavemente para misturar bem e incubar células transfectadas a 37 ° C, 5% de CO 2, durante a noite.
NOTA: Dia 3: - Para garantir a transfecção é bem-sucedida, verifique expressão GFP 24 horas após a transfecção por meio de um microscópio fluorescente.
NOTA: Mais de 50% das células que expressam GFP normalmente indica uma boa transfecção.
NOTA: Dia 4:
- Ao 48 horas após a transfecção, recolher o sobrenadante viral e substituir com media HEK293 T fresco (25 ml por frasco T175).
- Centrifuga-se o sobrenadante virai a 1140 xg durante 10 min a 4 ° C para limpar os detritos celulares a partir do sobrenadante. Transferir o sobrenadante para tubo apagada e armazenamento de 50 ml a 4 ° C.
NOTA: Dia 5:
- Centrifuga-se o sobrenadante virai a 1140 xg durante 10 min a 4 ° C para limpar os detritos celulares a partir do sobrenadante. Transferir o sobrenadante para tubo apagada e armazenamento de 50 ml a 4 ° C.
- Em 72 horas após a transfecção, recolher o segundo lote de sobrenadante viral. Repita o passo 2.3.1 e piscina de 48 e 72 horas apuradas sobrenadantes.
- Para concentrar o vírus, o sobrenadante viral centrifugar a 170.000 xg durante 90 min a 4 ° C, usando 30 ml de tubos de ultracentrífuga.
- Descartar o sobrenadante e repetir o passo 2.6 até que todo o sobrenadante clarificado foi centrifugado deixando um (invisível) pelota de vírus mais precipitado PEI na base do tubo.
- Para voltar a suspender vírus, adicionar 500 mL de mídia SATO (ver quadro das soluções de reserva) para os tubos de ultracentrífuga. Vortex durante 30 segundos and raspar a base do tubo com uma ponta de pipeta para soltar mecanicamente o vírus. Repita este passo 6x, a fim de perder pellet viral.
- A piscina do vírus de novo suspenso em microtubos e girar muito brevemente para remover PEI insolúvel. Filtra-se o sobrenadante através de um filtro de 0,45 um para remover as proteínas.
- Alíquota do vírus em 20 ul, 50 ul, e alíquotas de 100 ul e armazenar a -80 ° C.
3. Determinação Titer viral em células HEK293T
- Para verificar a expressão da proteína de interesse e para determinar a concentração óptima para experiências viral, adicionar uma série de diluições em série de estoque viral (por exemplo, 0, 5, 10, 20, 40, 80 ul) para a transdução de células T, semeadas em HEK293 placas de 6 poços, e a cultura durante 24 horas a 37 ° C, 5% de CO 2. Este protocolo normalmente gera vírus que pode ser utilizada em concentrações que variam de 1:50 a 1: 200, que se conseguir a expressão robusta do gene deinteresse.
- 48 horas após a transdução viral, células HEK293T lisar em tampão TNE (ver Tabela de solução de estoque) com inibidores da protease.
- Lavar os poços duas vezes com DPBS refrigerados e depois adicionar tampão TNE 150 ul a cada poço.
- Lyse células por pipetagem cima e para baixo 5-10x. Transfira os lisados de células inteiras a 1,5 ml tubo de microcentrífuga e incubar em gelo durante 15-30 min.
- Centrifugar os lisados a 4 ° C, durante 30 minutos à velocidade máxima (20000 xg) e o sobrenadante foi afastada transferência para um tubo fresco para a determinação de proteína de Bradford e por subsequente análise de Western blot.
- Determinar o nível de expressão da proteína de interesse por análise de transferência de Western, enquanto padrão de sondagem para anticorpos contra a própria proteína fundida e a etiqueta (isto é, a bandeira). Use a diluição viral que produz células de> 95% GFP + e expressão robusta da proteína de interesse para os experimentos.
4. Isolamento e Cultura de DERs (Figura 2 etapas 1 e 2)
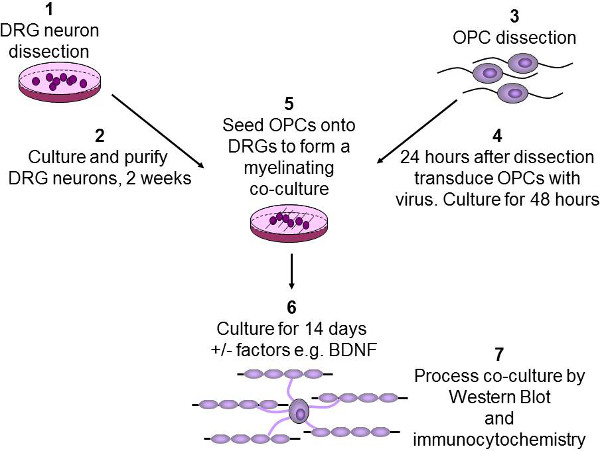
Figura 2: Diagrama esquemático. Da mielinização in vitro ensaio neurónios DRG são dissecados a partir de crias de rato P2-3, em seguida, purificadas e cultivadas durante duas semanas (1-2). OPCs são purificados a partir de cérebros de ratos P7-9 usando immunopanning (3). OPCs são então infectadas com lentivírus e cultivadas durante 48 horas (4). OPCs são então semeadas em GDH, e quaisquer fatores de crescimento de interesse, como BDNF são adicionados (5). Co-culturas são então cultivadas durante 2 semanas para permitir OPCs para diferenciar e mielinizar os axónios (6). Finalmente, co-culturas ou são lisadas por western blotting ou fixo por imunocitoquímica (7).
NOTA: Dia 1- 2 dias antes do dissecção:
- Casaco autoclavada 22 milímetros x 22 milímetros lamelas com poli-L-ornitina (0,5 mg / mL) em uma placa de 6 poços, e incuba-se a 37 ° C durante a noite.
- No dia seguinte, lamelas de revestimento novamente com laminina (20 ug / ml em MEM) a 37 ° C durante a noite, o excesso de aspirado e seco durante 20 minutos no capuz de cultura de tecidos.
NOTA: Dia 2: Dissecção e isolamento de DRGs: - Filhotes Sacrifice P2-P3 ratos 12x por transection cervical.
- Remover a pele que recobre o músculo do dorso do animal. Use Vannas tesoura para abrir forame vertebral e utilize uma pinça para colher cuidadosamente para fora da medula espinhal.
- Arrancam DRGs que se encontram entre as colunas vertebrais e cortar as fibras nervosas da coluna vertebral em anexo, em seguida, coloque em um prato DRGs 33 milímetros Petri contendo 3 ml L-15 mídia. Em geral, é possível reunir entre 8 e 12 DRG de cada lado da medula espinal
- Transferir recolhidos juntamente com os DRGs meio L-15 em um tubo de 15 ml, centrifugar a 180 xg durante 5 min.
- Aspiradoo sobrenadante, adicionar 2 ml de tripsina a 0,25% para DRG peletes e incubar a 37 ° C durante 30 min.
- Adicionar 5 ml de meio M1 (ver Tabela solução de estoque) para as peletes DRG para parar tripsina, e centrifugar a 180 xg durante 3 minutos à temperatura ambiente.
- Aspirar o sobrenadante, re-suspender o sedimento em 2 ml de meio M1 pré-aquecido (não com fatores de crescimento). Tritura-se o sedimento de gânglios pipetando 50 vezes ou até que os gânglios se encontram dispersas. Centrifuga-se a suspensão de células 180 xg durante 3 min.
- Ressuspender neurónios dissociados em meio M1 e placa para baixo em uma placa de 6 cavidades, com 5 gânglios por 100 ul por poço.
- Para remover proliferação de células não-neuronais, após um mínimo de 4 horas de incubação para facilitar a fixação, remova a mídia M1 e alimentam os neurônios com a mídia M2 (ver Tabela de solução de estoque). Culturas neurónios DRG na presença de NGF (100 ng / ml) para purificar uma cultura de NGF-dependentes que expressam TrkA DRGs. Alternativamente, usar BDNF (100 ng / ml) para purificar TrkB-express dependente do BDNFcantar DRGs.
- Manter neurónios em meio M1 (+ NGF ou o BDNF a 100 ng / ml) que alternam com suporte M2 anti-mitótico durante 2 semanas, como mostrado abaixo.
- Manter meio M1 (+ NGF ou o BDNF a 100 ng / mL) nos dias 4-6, 8-10, 12-14 e M2 meios (+ NGF ou o BDNF a 100 ng / ml) com FDU e uridina nos dias 1 a 4, 6-8, e 10-12. É necessário um mínimo de 3 ciclos de M2 purificação mídia.
- Manter DRGs em M1 sozinho por mais um semana depois de completar o ciclo antimitotic duas semanas. Mudança de mídia M1 cada 2-3 dias.
5. Isolamento e Cultura de OPCs (Figura 2 passo 3)
NOTA: Dia 1- 2 dias antes do dissecção:
- O revestimento 10 centímetros placas de cultura de tecido com poli-D-lisina (PDL, 10 ug / ml em água desionizada esterilizada) a 4 ° C durante a noite.
NOTA: Dia 2: 1 dia antes do dissecção: - Lavar pratos PDL com água deionizada esterilizada por 3x. Deixe secar por ~ 6 horas na capa de cultura de tecidos. Se não está sendo utilizado de imediato, enrole e lojaaté 4 semanas a 4 ° C.
- Prepare placas de anticorpos secundários para immunopanning. Para uma dissecção cérebro, para preparar as placas 2x IgG (por Ran2 anticorpo para remover astrócitos e anticorpo O1 para remover premyelinating oligodendrócitos), com 45 ul de IgG de ratinho de cabra α em DPBS (15 ml) por 10 cm numa caixa de Petri; 1x placa para IgM (anticorpo O4 para seleccionar as células precursoras de oligodendrócitos), 45 ul de cabra α IgM de ratinho em DPBS (15 ml) por 10 cm numa caixa de Petri.
NOTA: Dia 3: dia de dissecção: - Adicionar 200 unidades de papaína em 10 ml de tampão de papaína (Tabela 2), e aquecer a 37 ° C, até o tampão ficar transparente.
Tabela 2: Preparação de tampão de papaína.Concentração para 250 ml A concentração final Estoque EBSS 10x 25 ml 1x MgSO4 100 mM 2,5 ml 1 mM Glicose 30% 3 ml 0,46% EGTA 0,5 M 1 mL 2 mM NaHCO3 1 H 6,5 ml 26 mM Levar o volume até 250 ml com água deionizada e esterilizar filtro - Lave todas as placas de anticorpos secundários com DPBS para 3x.
- Verter o anticorpo Ran2 O1 e sobre as placas de IgG e o hibridoma O4 à placa de IgM. Incubar todas as placas destes anticorpos primários durante mais 2 horas à temperatura ambiente.
- Dissecar um cérebro de um rato P7. Decapitar um filhote de cachorro com uma tesoura afiada e retire a pele que recobre o crânio com uma tesoura.
- Cortar em torno do crânio a partir do lobo occipital, lobo temporal e lobo frontal. Remover cérebro de crânio usando uma pinça e gentilmente transferi-lo para uma de 35 mm placa de Petri com 1 ml de DPBS.
- Cortar o cérebro em pedaços pequenos aproximadamente com tesoura ou lâmina estéril.
- Filtrar pré-aquecido tampão papaína (10 ml) para um novo tubo de 15 ml contendo alguns grãos de L-cisteína, em seguida adicionar 200 ul de ADNase (12.500 U / ml) ao tampão de papaína filtrada.
- Despeje o buffer de papaína para os tecidos cerebrais em cubos; incubar a 37 ° C durante 90 min.
- Transferência gentlly dissociado tecidos cerebrais em tubo de 50 ml usando uma pipeta de 25 ml edeixa-se repousar.
- Remover tampão papaína, adicionar 2 ml Lo Ovo (ovomucóide) para os tecidos cerebrais e titurate 5-10x por pipetagem para quebrar pedaços de tecidos cerebrais, deixar repousar e remover o top 2 ml de sobrenadante para um novo tubo.
- Adicionar outro 2 ml Lo Ovo para os tecidos cerebrais e repita o passo 5.11 até que não haja pedaços de tecido permanecem. Trituração pode ficar cada vez mais agressivo.
- Centrifugar a suspensão de células dissociadas durante 15 min a 200 x g. Aspirar fora o sobrenadante e ressuspender o sedimento celular em 10 ml de Ovo Oi e centrifugar durante 15 min a 200 x g.
- Lave primeira placa immunopanning (Ran 2 placa), com DPBS para 3x. Aspirar o sobrenadante, ressuspender as células em 10 ml de tampão de filtração (Tabela 2) e derramar sobre a primeira placa immunopanning (Ran placa 2). Incubar as células durante 15 min a RT.
- Lave a segunda placa immunopanning (placa O1) com DPBS para 3x.
- Após a incubação da primeira placa immunopanning, ponta da suspensão de células em segundoimmunopanning placa (placa O1), Enxaguar quaisquer células soltas para fora da superfície da placa com 1-3 ml de tampão de filtração e, com uma pipeta de transferência para a placa O1. Incubar as células durante 15 min a RT.
- Lava-se a terceira placa immunopanning (placa O4). Esta placa é a placa de selecção positiva em que os OPCs ligar sua superfície. Lave a placa O4 com DPBS para 3x.
- Após a incubação sobre a segunda placa immunopanning, transferência das células para a placa immunopanning final (placa O4), incubar as células durante 45 minutos à TA. Esta etapa irá selecionar O4 + OPCs.
- Aspirar o sobrenadante do último prato immunopanning (placa O4) e lavar a placa com EBSS para 6x.
- Para remover a placa fora OPCs, incubar as células com 5 ml de 0,05% quente Tripsina-EDTA diluída 1:10 com EBSS a 37 ° C durante 8 min.
- Adicionar 5 ml de 30% de FBS (feito em EBSS) para neutralizar a tripsina. Remover as células fora da superfície da placa por pipetagem por cerca de 50x.
- Transferir todas as célulassuspensão para um novo tubo e centrifuga-se durante 15 min a 200 x g.
- Descartar o sobrenadante e ressuspender o sedimento de células em 1 ml de meio pré-aquecido SATO, seguido de contagem de células. Dissecção de um cérebro pode render 1,5-2.000.000 OPCs.
- Células da placa em placas PDL revestidos secos, a uma densidade de 1 x 10 5 e 5 x 10 5 por placa de 10 cm em meios com SATO (10 ml) que contêm o factor neurotrófico ciliar (CNTF, 10 ng / ml), factor de crescimento derivado de plaquetas (PDGF, 10 ng / ml,), a neurotrofina 3 (NT3, 1 ng / ml), e forscolina (4,2 ug / ml) 2,12. OPCs Cultura a 37 ° C, 8% de CO 2.
6. OPCs Transdução
- OPCs Culturas primárias em SATO meios (10 ml / placa de 10 centímetros) com CNTF (10 ng / ml), o PDGF (10 ng / ml), NT3 (1 ng / ml), e forscolina (4,2 ug / ml) a 37 ° C, 8% de CO 2 durante 24 horas após a dissecação.
- Completamente fora de aspirar os meios de cultura OPC, as células de alimentação com media SATO feitos na hora (10 ml) Com fatores de crescimento (veja acima no passo 6.1).
- Adicionar vírus para os OPCs para a concentração óptima determinada pelo passo 3 (Figura 2, passo 4), OPCs cultura durante mais 48 horas.
7. Sementeira OPC para mielinizantes co-culturas (Figura 2, as etapas 5 e 6)
- Para remover OPCs fora da superfície, placas primeira lavagem OPC com 8 mL de EBSS duas vezes, depois incubar as células com 5 ml de 0,05% quente Tripsina-EDTA diluída 1:10 com EBSS a 37 ° C durante 2 min.
- Neutraliza-se a tripsina com 30% de FBS em EBSS (5 ml) e remover as células a partir da placa por pipetagem. Transferir suspensão de células para um tubo de 15 ml, centrifugar a 180 xg durante 15 min à temperatura ambiente.
- Aspirar o sobrenadante e ressuspender o sedimento de células em 1 ml de meio pré-aquecido SATO, seguido por contagem de células.
- Antes da sementeira OPC, meios completamente aspirado a partir da placa de cultura de DRG. Gentilmente semear 200.000 OPCs gota a gota para os neurônios do GRD, como descrito anteriormente 4-6.
NOTA:O volume total de semeadura OPC deve ser inferior a 200 ul por 22 milímetros lamela. - Deixar células para resolver sem mover a placa durante 10 minutos na capa de cultura de tecido, em seguida, o topo suavemente com meios SATO 1 ml de pré-aquecido por poço.
- Replate os restantes OPCs irmãos com a mídia SATO com fatores de crescimento (veja o passo 6.1). Usar estes OPCs irmãos para verificar a expressão da proteína de interesse.
- Após 24 horas, substitui os meios de comunicação com os meios de comunicação SATO co-cultura (2 ml / poço) contendo meios SATO (não há factores) e neurobasal (v / v) com 1% de B27. Manter as co-culturas durante 14 dias com alteração de mídia a cada 2-3 dias.
- Avaliar as co-culturas para a expressão da proteína de mielina por análise Western blot e visualizar para a formação de segmentos axonais mielinizadas por imunocoloração dupla com anticorpos contra marcadores de proteína básica de mielina e marcadores neuronais 4-6.
Resultados
O marcado com bandeira extracelular quinase 1 (Flag-Erk1) relacionado com o sinal de constructo utilizado para a produção de lentivírus é verificada por digestão com enzimas de restrição das construções usadas, incluindo tanto as construções 2K7 e as construções de empacotamento e acessórias necessárias para produção de vírus (Figura 3) .
Discussão
Mielinização de axónios é um processo crucial para a função óptima de ambos os sistemas nervosos central e periférico de vertebrados. A geração e manutenção de axónios mielinizados é um processo complexo e coordenada envolvendo interacções moleculares entre neuronal, glial (a partir de células de Schwann ou oligodendrócitos) e as proteínas de matriz extracelulares. A importância e aplicabilidade deste protocolo é que ele permite a manipulação de proteínas em um tipo específico de célula dentro ...
Divulgações
The authors declare that there is no conflict of interest regarding this research.
Agradecimentos
This work was supported by the Australian National Health and Medical Research Council (NHMRC fellowship #454330 to JX, project grant #628761 to SM and APP1058647 to JX), Multiple Sclerosis Research Australia (MSRA #12070 to JX), the University of Melbourne Research Grant Support Scheme and Melbourne Research CI Fellowship to JX as well as Australia Postgraduate Scholarships to HP and AF. We would like to acknowledge the Operational Infrastructure Scheme of the Department of Innovation, Industry and Regional Development, Victoria Australia.
Materiais
| Name | Company | Catalog Number | Comments |
| 2K7 lentivector | Kind gift from Dr Suter9 | ||
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100mg | |
| Alexa Fluor 488 Goat anti-mouse IgG | Jackson Immunoresearch | 115545205 | |
| Alexa Fluor 488 goat anti-rabbit IgG (H+L) | Life Technologies | A11008 | |
| Alexa Fluor 594 goat anti-mouse IgG (H+L) | Life Technologies | A11005 | |
| Alexa Fluor 594 goat anti-rabbit IgG (H+L) | Life Technologies | A11012 | |
| Ampicillin | Sigma-Aldrich | A9518-5G | |
| B27 - NeuroCul SM1 Neuronal Supplement | Stem Cell Technologies | 5711 | |
| BDNF (Human) | Peprotech | PT450021000 | |
| Biotin (d-Biotin) | Sigma Aldrich | B4639 | |
| Bradford Reagent | Sigma Aldrich | B6916-500ML | |
| BSA | Sigma Aldrich | A4161 | |
| Chloramphenicol | Sigma-Aldrich | C0378-100G | |
| CNTF | Peprotech | 450-13020 | |
| DAKO fluoresence mounting media | DAKO | S302380-2 | |
| DMEM, high glucose, pyruvate, no glutamine | Life Technologies | 10313039 | |
| DNase | Sigma-Aldrich | D5025-375KU | |
| DPBS | Life Technologies | 14190250 | |
| DPBS, calcium, magnesium | Life Technologies | 14040182 | |
| EBSS | Life Technologies | 14155063 | |
| EcoRI-HF | NEB | R3101 | |
| Entry vectors for promoter and gene of interest | Generate as per protocols 1-2 | ||
| Fetal Bovine Serum | Sigma-Aldrich | 12003C | |
| Forskolin | Sigma Aldrich | F6886-50MG | |
| Glucose (D-glucose) | Sigma-Aldrich | G7528 | |
| Glycerol | Chem Supply | GL010-500M | See stock solutions |
| Goat Anti-Mouse IgG | Jackson ImmunoResearch | 115005003 | |
| Goat Anti-Mouse IgM | Jackson ImmunoResearch | 115005020 | |
| Goat Anti-Rat IgG | Jackson ImmunoResearch | 112005167 | |
| Hoechst 33342 | Life Technologies | H3570 | |
| Igepal | Sigma Aldrich | I3021-100ML | |
| Insulin | Sigma Aldrich | I6634 | |
| Kanamycin | Sigma-Aldrich | 60615 | |
| Laminin | Life Technologies | 23017015 | |
| LB Medium | See stock solutions | ||
| LB-Agar | See stock solutions | ||
| L-Cysteine | Sigma-Aldrich | C-7477 | |
| Leibovitz's L-15 Medium | Life Technologies | 11415064 | |
| L-Glutamate | Sigma-Aldrich | G1626 | |
| L-Glutamine- 200 mM (100x) liquid | Life Technologies | 25030081 | |
| LR Clonase II Plus enzyme | Life Technologies | 12538-120 | |
| MEM, NEAA, no Glutamine | Life Technologies | 10370088 | |
| Mouse α βIII Tubulin | Promega | G7121 | |
| Mouse αMBP (monoclonal) | Millipore | MAB381 | |
| Na pyruvate | Life Technologies | 11360-070 | |
| NAC | Sigma Aldrich | A8199 | |
| NcoI-HF | NEB | R3193S | |
| NEBuffer 4 | NEB | B7004S | |
| Neurobasal medium | Life Technologies | 21103049 | |
| NGF (mouse) | Alomone Labs | N-100 | |
| NT-3 | Peprotech | 450-03 | |
| O1 antibody - Mouse anti-O1 | Millipore | MAB344 | Alternative if O1 hybridoma cells are unavailable |
| O1 hybridoma cells | Conditioned medium containing anti-O1 antibody to be used for immunopanning | ||
| O4 antibody - Mouse anti-O4 | Millipore | MAB345 | Alternative if O4 hybridoma cells are unavailable |
| O4 hybridoma cells | Conditioned medium containing anti-O4 antibody to be used for immunopanning | ||
| Competent cells | Life Technologies | A10460 | |
| One Shot Stbl competent cells | Life Technologies | C7373-03 | |
| Papain Suspension | Worthington/Cooper | LS003126 | |
| pBR8.91 | Kind gift from Dr Denham10 | ||
| PDGF-AA (Human) | Peprotech | PT10013A500 | |
| Penicillin-streptomycin 100x solution | Life Technologies | 15140122 | |
| pENTRY4IRES2GFP | Invitrogen | 11818-010 | |
| pMD2.G | Addgene | 12259 | |
| Poly-D-lysine | Sigma | P6407-5MG | |
| Polyethylenimine (PEI) | Sigma-Aldrich | 408727-100ML | |
| Poly-L-ornithine | Sigma Aldrich | P3655 | |
| Progesterone | Sigma Aldrich | P8783 | |
| Protease inhibitor tablet (Complete mini) | Roche | 11836153001 | |
| Proteinase K | Supplied with Clonase enzyme | ||
| Putrescine | Sigma Aldrich | P-5780 | |
| Rabbit α neurofilament | Millipore | AB1987 | |
| Rabbit αMBP (polyclonal) | Millipore | AB980 | |
| Ran2 hybridoma cells | ATCC | TIB-119 | Conditioned medium containing anti-Ran2 antibody to be used for immunopanning |
| Rat anti CD140A/PDGFRa antibody | BD Pharmingen | 558774 | |
| SacII | NEB | R0157 | |
| SOC medium | Supplied with competent bacteria | ||
| Sodium selenite | Sigma Aldrich | S5261 | |
| Spe I | NEB | R0133S | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Buffer | NEB | B0202S | |
| TE buffer pH8 | See stock solutions | ||
| TNE lysis buffer | |||
| Trace Elements B | Cellgro | 99-175-CI | |
| Transferrin (apo-Transferrin human) | Sigma-Aldrich | T1147 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Trypsin | Sigma-Aldrich | T9201-1G | |
| Trypsin Inhibitor From Chicken Egg White | Roche | 10109878001 | |
| Trypsin-EDTA (1x), phenol red (0.05%) | Life Technologies | 25300-054 | |
| Unconjugated Griffonia Simplicifolia Lectin BSL-1 | Vector laboratories | L-1100 | |
| Uridine | Sigma-Aldrich | U3003-5G |
Referências
- Baumann, N., Pham-Dinh, D. Biology of oligodendrocyte and myelin in the mammalian central nervous system. Physiol Rev. 81 (2), 871-927 (2001).
- Watkins, T. A., Emery, B., Mulinyawe, S., Barres, B. A. Distinct stages of myelination regulated by gamma-secretase and astrocytes in a rapidly myelinating CNS coculture system. Neuron. 60 (4), 555-569 (2008).
- Lee, K., et al. MDGAs interact selectively with neuroligin-2 but not other neuroligins to regulate inhibitory synapse development. Proc Natl Acad Sci U S A. 110 (1), 336-341 (2013).
- Xiao, J., et al. BDNF exerts contrasting effects on peripheral myelination of NGF-dependent and BDNF-dependent DRG neurons. J Neurosci. 29 (13), 4016-4022 (2009).
- Chan, J. R., et al. NGF controls axonal receptivity to myelination by Schwann cells or oligodendrocytes. Neuron. 43 (2), 183-191 (2004).
- Xiao, J., et al. Brain-Derived Neurotrophic Factor Promotes Central Nervous System Myelination via a Direct Effect upon Oligodendrocytes. Neurosignals. 18 (3), 186-202 (2010).
- Lundgaard, I., et al. Neuregulin and BDNF induce a switch to NMDA receptor-dependent myelination by oligodendrocytes. PLoS Biology. 11 (12), e1001743 (2013).
- Kleitman, N., W, P. M., Bunge, R. P. . Tissue culture methodes for the study of myelination. , (1991).
- Xiao, J., et al. Extracellular signal-regulated kinase 1/2 signaling promotes oligodendrocyte myelination in vitro. J Neurochem. 122 (6), 1167-1180 (2012).
- Wong, A. W., Xiao, J., Kemper, D., Kilpatrick, T. J., Murray, S. S. Oligodendroglial expression of TrkB independently regulates myelination and progenitor cell proliferation. The Journal of Neuroscience. 33 (11), 4947-4957 (2013).
- Li, Z., et al. Molecular cloning, Characterization and Expression of miR-15a-3p and miR-15b-3p in Dairy Cattle. Molecular and Cellular Probes. , (2014).
- Emery, B., et al. Myelin gene regulatory factor is a critical transcriptional regulator required for CNS myelination. Cell. 138 (1), 172-185 (2009).
- Murai, K., et al. Nuclear receptor TLX stimulates hippocampal neurogenesis and enhances learning and memory in a transgenic mouse model. Proc Natl Acad Sci U S A. 111 (25), 9115-9120 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados