Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Production et utilisation de lentivirus à des cellules sélective transduction primaire Oligodendrocyte précurseur pour
Dans cet article
Résumé
Here we present protocols that offer a flexible and strategic foundation for virally manipulating oligodendrocyte precursor cells to overexpress proteins of interest in order to specifically interrogate their role in oligodendrocytes via the in vitro model of central nervous system myelination.
Résumé
La myélinisation est un processus complexe qui implique à la fois les neurones et la myéline formant des cellules gliales, les oligodendrocytes dans le système nerveux central (SNC) et les cellules de Schwann dans le système nerveux périphérique (SNP). Nous utilisons un test in vitro de la myélinisation, un modèle établi pour étudier myélinisation du SNC in vitro. Pour ce faire, les cellules précurseurs d'oligodendrocytes (OPC) sont ajoutés aux primaires rongeurs ganglion de racine dorsale (DRG) neurones purifiés pour former des co-cultures myélinisantes. Pour interroger spécifiquement les rôles que les protéines exprimées par les oligodendrocytes particuliers exercent sur la myélinisation nous avons développé des protocoles qui transduction sélective OPC à l'aide du lentivirus surexprimant type sauvage, les protéines négatives dominantes ou constitutivement active avant d'être ensemencées sur les neurones DRG. Cela nous permet d'interroger spécifiquement les rôles de ces protéines dans la régulation oligodendrogliales myélinisation. Les protocoles peuvent également être appliqués dans l'étude des OTses types cellulaires, offrant ainsi une approche qui permet la manipulation sélective des protéines exprimées par un type cellulaire souhaité, tels que les oligodendrocytes ciblée pour l'étude de la signalisation et des mécanismes de compensation. En conclusion, combinant le dosage de la myélinisation in vitro avec lentiviraux infectés OPC fournit un outil stratégique pour l'analyse des mécanismes moléculaires impliqués dans la myélinisation.
Introduction
La myélinisation des axones est crucial pour la transmission rapide et efficace des potentiels d'action dans les deux les systèmes nerveux central et périphérique. Des cellules spécialisées, les cellules de Schwann dans le système nerveux périphérique et les oligodendrocytes dans le système nerveux central, se enroulent autour des axones et ensheathe dans la myéline, efficace isolantes du nerf et de faciliter la conduction saltatoire 1. Le procédé de la myélinisation peut être étudiée in vitro en utilisant des neurones ganglionnaires de la rétine, 2 nanofibres par génie 3, ou neurones ganglionnaires de la racine dorsale co-cultivées avec des cellules de Schwann ou des oligodendrocytes 4 7.5. Le dosage in vitro de la myélinisation est un modèle établi pour l'étude du système nerveux myélinisation et il reproduit la plupart des processus fondamentaux qui se produisent pendant la myélinisation in vivo 5-8. Le test implique la co-culture des populations purifiées de ganglions de racine dorsale (DRG) neurones, avec OPC (CNS myélinisation) ou des cellules de Schwann (PNS pour la myélinisation). Dans des conditions spécifiques de ces cellules myélinisantes ensheathe axones DRG dans l'ordre, structurellement vérifié, feuille multi-lamellaire ultra de la membrane plasmique qui expriment la même complément de protéines spécifiques de la myéline présents in vivo isolant.
Le modèle de cellule la plus couramment utilisée de l'étude de myélinisation du SNC in vitro est le co-cultures de neurones DRG et OPC, qui ont été utilisés avec succès pour étudier l'effet que des facteurs exogènes tels que les neurotrophines exercent sur myélinisation du SNC in vitro 5,6. Les facteurs exogènes tels que les facteurs de croissance ou de petites molécules inhibiteurs pharmacologiques ont été largement utilisés pour étudier le rôle des voies de signalisation dans la myélinisation utilisant le DRG-OPC modèle de co-culture 7,9. Cependant, dans les paramètres de co-culture mixte contenant à la fois des neurones et des oligodendrocytes, il est resté formellement possible que soit les facteurs de croissance ou du pharinhibiteurs macologique auraient exercé des effets à la fois les neurones et les oligodendrocytes (OL) DRG. Cela offre la possibilité de disséquer spécifiquement les rôles que les protéines exprimées uniquement par DRG ou oligodendroglie exerce sur la myélinisation utilisant ce système cellulaire double. Pour confirmer sans équivoque que la voie de signalisation dans oligodendrogliale régule directement la myélinisation, transduction lentiviral des OPC, avant le semis sur les neurones de DRG pour le dosage in vitro de la myélinisation, se est avéré être une manière élégante de surexpression de type sauvage et les protéines mutantes, ainsi comme knockdown expression des protéines exprimées de manière constitutive par les oligodendrocytes. Ainsi, cette approche offre une avenue pour interroger et manipuler spécifiquement les voies de signalisation au sein oligodendrocytes pour étudier la myélinisation 9,10.
Dans cet article, nous présentons les méthodes que nous avons développées pour surexprimer une protéine d'intérêt dans les oligodendrocytes sélective via un lentivirusapproche pour étudier la myélinisation in vitro. La technique commence avec la génération de vecteurs d'expression contenant le gène d'intérêt, que ce soit dans un type sauvage, forme négative constitutivement active ou dominant qui sont ensuite ensuite clone dans le vecteur pENTR (pENTR L1-L2 pENTR4IRES2GFP). Ce vecteur (contenant le gène d'intérêt), le donneur CMV promoteur (pENTR L4-R1 pENTR-pDNOR-CMV) et le lentivecteur de 2K7 sont combinés dans une réaction enzymatique pour produire un vecteur de 2K7 contenant le promoteur CMV, le gène d'intérêt, un site interne d'entrée des ribosomes et la GFP (Figure 1). Cette passerelle clone construction d'2K7 combiné avec l'enveloppe du virus PMD2.G et le paquet de virus pBR8.91 peut être co-transfecté dans des cellules HEK293T pour générer lentivirus qui peut ensuite être utilisé pour transduire des OPC. Une fois infectés par le lentivirus les OPC expriment un niveau élevé de la protéine d'intérêt. Ces OPC peuvent ensuite être ensemencées sur les cultures de neurones DRG et l'effet que l'expressionles niveaux des élevés de la protéine désirée exerce sur la myélinisation peut être interrogé. Les co-cultures sont évaluées pour l'expression de la protéine de myéline par analyse Western blot et visualisés pour la formation de segments axonales myélinisées par immunocytochimie.
Protocole
REMARQUE: Tous les animaux utilisés pour cette étude étaient de sexe mixte et a grandi aux installations vétérinaires du Département d'anatomie et de pathologie et Le Florey Institute of Neuroscience et de la recherche en santé mentale à l'Université de Melbourne. Toutes les procédures d'animaux ont été approuvés par les comités d'éthique expérimentation animale à l'Université de Melbourne.
1. Clonage de 2K7 Lentivector
- Avant de cloner le gène d'intérêt dans le vecteur lentiviral 2K7, sous-cloner le gène dans le vecteur pENTR (3637 pb, résistant à la kanamycine) en utilisant des techniques moléculaires classiques 11. Utiliser les sites de restriction EcoRI et SacII pour le sous-clonage.
- Amplification de l'2K7 Lentivector
- Transformer l'ADN lentivector 2K7 mélangeant doucement par 100 ng d'ADN plasmidique avec des cellules compétentes et on incube sur de la glace pendant 30 min.
- ADN choc thermique / cellules compétentes mélanger à 42 ° C pendant 90 secondes et la plaque sur des assiettes LB-agar contenant à la fois ampicillin (100 ug / ml) et du chloramphenicol (15 ug / ml) à 37 ° C pendant 16-18 h.
REMARQUE: Chloramphenicol est utilisé pour empêcher une recombinaison entre les longues répétitions terminales. - Le lendemain, grandir choisi clones bactériens dans un milieu LB contenant de l'ampicilline (100 ug / ml) et du chloramphenicol (15 ug / ml) à 37 ° C pendant 16-18 h. Extraire l'ADN en utilisant un plasmide commerciale kit ADN Maxiprep selon les instructions du fabricant.
- Sous-clonage de pENTR vecteur 2K7 Lentivector (Figure 1)
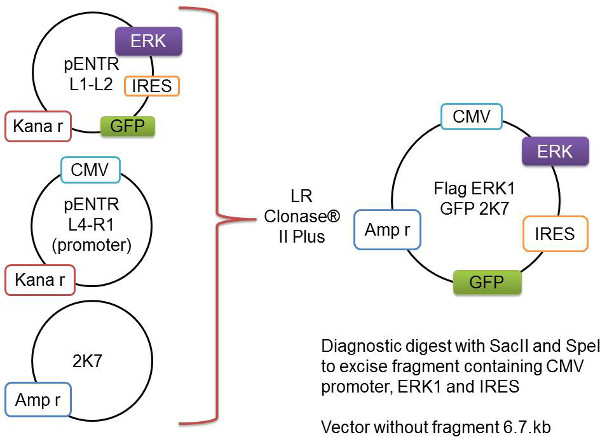
Figure 1:. Représentation schématique du processus de recombinaison passerelle Le gène d'intérêt, ici représentée comme Flag-Erk1, est cloné dans le vecteur pENTR L1-L2. Ceci est ajouté au vecteur pENTR L4-R1 contenant le promoteur CMV et le vecteur squelette 2K7. Ces trois vecteurs sont recombinés par l'enzyme LR Clonase II Plus pourinsérer le gène d'intérêt et le promoteur dans le vecteur du virus 2K7 prêt.- Ajoutez ce qui suit à un tube de 1,5 ml et mélanger doucement:
L4-R1-pENTR pDNOR-CMV (promoteur) (60 bng) 1 à 2,5 pl
L1-L2 pENTR4IRES2GFP (avec le gène d'intérêt) (80 bng) 1 à 2,5 pl
K7 lentivecteur (80 ng / ul) 1 pl
TE-tampon, pH 82-5 ul
Total 8 pi - Retirer mélange d'enzymes clonase de -80 ° C et décongélation sur de la glace pendant 2 min. Assurez-vous que cette enzyme est une autre partie aliquote de -80 ° C, l'enzyme a considérablement réduit l'efficacité de clonage avec des cycles répétés de congélation-décongélation
- Ajouter 2 ul d'enzyme par réaction et bien mélanger par vortex et incuber le mélange réactionnel clonase au 23-25 ° C pendant 6 à 24 h.
- Ajouter 1 pi de solution de proteinase K (fourni avec le kit d'enzyme) au mélange réactionnel clonase et incuber pendant 10 min à 37 ° C.
- Transformer mélange réactionnel clonase dans des cellules compétentes et faire croître l'sélectionnezcolonies dans du milieu LB contenant de l'ampicilline (100 ug / ml) à 37 ° C pendant 16-18 h.
- Extraire et purifier l'ADN en utilisant un plasmide commercial kit ADN Miniprep selon les instructions du fabricant.
- Confirmez l'ADN par digestion en utilisant les enzymes de restriction Spe I et Sac II et un tampon approprié selon les instructions du fabricant pour éliminer le fragment contenant le promoteur et le gène d'intérêt.
NOTE: Cette étape est essentielle pour vérifier si l'ADN sous-cloné le gène a correcte de l'insert d'intérêt avec le squelette du vecteur droite. La taille du vecteur lui-même sans fragment inséré est 6,7 kb. La taille du fragment inséré libéré comprend à la fois le promoteur et le gène d'insert d'intérêt. Calculer les tailles de fragments attendus de l'empreinte pour le gène spécifique via un programme d'édition de séquence d'ADN, par exemple, les APE - un plasmide Editor. - Vérifiez la taille des deux squelette du vecteur d'ADN et le fragment libéré en exécutant un gel d'agarose à 1%dans un tampon TAE 1X (voir tableau de solutions d'achat d'actions) à 100 V.
- Amplifier l'ADN confirmé par des bactéries croissant dans 500 ml de milieu LB contenant de l'ampicilline (100 ug / ml) à 37 ° C pendant 16-18 h.
- Extraire et purifier l'ADN en utilisant un plasmide commercial kit ADN Maxiprep selon les instructions du fabricant.
REMARQUE: Maxiprep génère habituellement une quantité suffisante d'ADN nécessaire à la production virale.
- Ajoutez ce qui suit à un tube de 1,5 ml et mélanger doucement:
- Répétez les étapes 1.3.9-1.3.10 pour amplifier les ADN suivantes pour les préparations: vecteur lentiviraux enveloppe (PMD2.G, 6,1 kb), vecteur de l'emballage (pBR8.91, 12,5 kb), et un vecteur lentiviral vide comme un contrôle (GFP -CMV-2K7, 8,7 kb).
2. 2K7 Virus production
REMARQUE: Jour 1:
- Le jour de la transfection, plaque 32 millions de cellules HEK293 T en flacon contenant 25 ml T175 HEK293 T médias cellulaire (voir la table des solutions d'achat d'actions). Alternativement, plaque 16 millions de cellules le jouravant la transfection si le temps tourne serré sur le jour de la transfection.
REMARQUE: La transfection peut être le même succès par étalement des cellules sur le jour de la transfection ou avant le jour. Utilisation de l'une des deux alternatives, le point critique ici est de se assurer que les cellules soient coincés vers le bas sur la culture surface de la boîte avant la transfection.
REMARQUE: Jour 2: - Transfection
- Avant la transfection, l'ADN dilué à 1 ug / ul dans du tampon TE contenant du Tris 10 mM pH 8, EDTA 1 mM pH 8 dans de l'eau désionisée.
- Dans un tube de 50 ml, préparer un mélange maître (Tableau 1) pour la transfection dans le ballon T175. Ajouter ADN pour préchauffé milieu Eagle modifié de Dulbecco (DMEM) et bien mélanger au vortex, puis ajouter polyéthylènimine stérile (PEI) (voir la table des solutions d'achat d'actions) pour éviter la précipitation prématurée. ">
Tableau 1: Préparation de mélange de transfection pour 2K7 Virus.Vecteur trong> Concentration Volume pMDG.2 1 pg / pl 5 ul pBR8.91 1 pg / pl 15 ul vecteur 2K7 avec la GFP + gène d'intérêt 1 pg / pl 22 pi Polyéthylènimine stérile (PEI) 1 g / L 500 ul DMEM 2100 pi - Bien mélanger en inversant le 3-4x de tube et incuber pendant 15 min à température ambiante (RT).
- Effectuer un changement de support complet sur les cellules HEK293T. Aspirer le milieu de culture à partir de cellules complètement et nourrir avec des milieux de culture de cellules HEK293T préchauffé (25 ml par flacon T175).
- Ajouter le mélange de transfection (ADN / PEI mélange) goutte à goutte aux cellules monocouches. Déplacez doucement pour bien mélanger et incuber des cellules transfectées à 37 ° C, 5% de CO 2, la nuit.
REMARQUE: Jour 3: - Pour assurer la transfection est réussie, vérifiez expression de la GFP 24 heures après la transfection par une microscopie à fluorescence.
NOTE: Plus de 50% des cellules exprimant la GFP indique généralement une bonne transfection.
REMARQUE: Jour 4:
- Au post-transfection 48 h, recueillir le surnageant viral et le remplacer par un milieu frais HEK293 T (25 ml par flacon T175).
- Centrifuger le surnageant viral à 1140 xg pendant 10 min à 4 ° C pour éliminer les débris cellulaires du surnageant. Transfert effacé surnageant à tube de 50 ml et conserver à 4 ° C.
REMARQUE: Jour 5:
- Centrifuger le surnageant viral à 1140 xg pendant 10 min à 4 ° C pour éliminer les débris cellulaires du surnageant. Transfert effacé surnageant à tube de 50 ml et conserver à 4 ° C.
- À 72 heures après la transfection, de recueillir le deuxième lot de surnageant viral. Répétez l'étape 2.3.1 et la piscine 48 et 72 h effacés surnageants.
- Pour concentrer le virus, un surnageant viral centrifugation à 170 000 xg pendant 90 min à 4 ° C en utilisant 30 ml tubes d'ultracentrifugation.
- Jeter le surnageant et répétez l'étape 2.6 jusqu'à ce que le surnageant clarifié a été centrifugé laissant un (invisible) pastille de virus, plus précipité PEI dans la base du tube.
- Pour remettre en suspension virus, ajouter 500 ul SATO médias (voir le tableau des solutions d'achat d'actions) aux tubes d'ultracentrifugation. Vortex pendant 30 secondes unee gratter la base du tube avec un embout de pipette pour desserrer mécaniquement le virus. Répétez cette étape 6x pour perdre culot viral.
- En commun le virus remis en suspension dans des tubes à centrifuger et tourner très brièvement pour enlever insoluble PEI. Filtrer le surnageant à travers un filtre de 0,45 um pour éliminer les protéines.
- Aliquoter le virus dans 20 pi, 50 pi et 100 pi aliquotes et conserver à -80 ° C.
3. détermination du titre viral dans les cellules HEK293T
- Pour vérifier l'expression de la protéine d'intérêt et de déterminer la concentration virale optimale pour des expériences, ajouter une série de dilutions en série de stock virale (par exemple, 0, 5, 10, 20, 40, 80 pi) pour transduire des cellules HEK293 T plaqués en plaques à 6 puits, et la culture pendant 24 heures à 37 ° C, 5% CO 2. Ce protocole génère typiquement virus qui peut être utilisé à des concentrations allant de 1:50 à 1: 200 qui permettent d'atteindre une expression robuste du gène deintérêt.
- 48 heures après la transduction virale, des cellules HEK293T lyse dans un tampon TNE (voir le tableau de la solution mère) avec des inhibiteurs de protéase.
- Rincer puits deux fois avec du DPBS réfrigérés et puis ajouter tampon TNE 150 pi dans chaque puits.
- Lyse cellules par aspiration et refoulement 5-10x. Transfert lysats de cellules entières à 1,5 ml microtube et incuber sur glace pendant 15 à 30 min.
- Centrifuger les lysats à 4 ° C pendant 30 min à vitesse maximale (20 000 xg) et le transfert effacée surnageant dans un nouveau tube pour la détermination des protéines de Bradford et par la suite une analyse Western blot.
- Déterminer le niveau d'expression de la protéine d'intérêt par analyse western blot standard, tout en sondant des anticorps dirigés contre la protéine elle-même et l'étiquette fusionnée (c.-à-Drapeau). Utilisez la dilution virale qui donne cellules> 95% GFP + et l'expression robuste de protéine d'intérêt pour les expériences.
4. Isolement et culture des DRG (Figure 2 étapes 1 et 2)
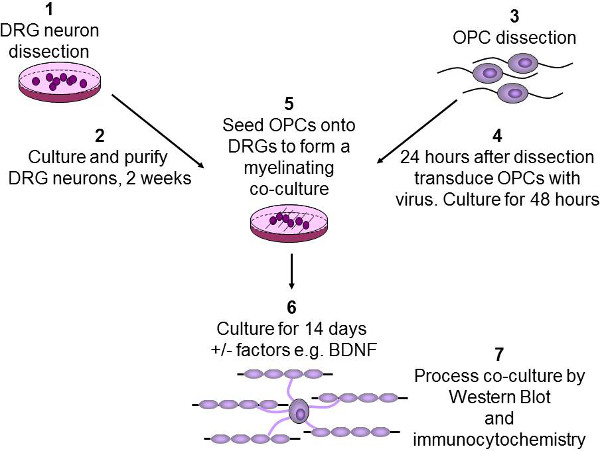
Figure 2:. Schéma de neurones DRG in vitro myélinisation dosage sont disséqués à partir de ratons P2-3, puis purifiés et cultivés sur deux semaines (1-2). Les OPC sont purifiés à partir de cerveaux de rats P7-9 utilisant immunopanning (3). Les OPC sont ensuite infectées par un lentivirus et cultivées pendant 48 heures (4). OPC sont ensuite ensemencées sur DRG, et tous les facteurs de croissance d'intérêt tels que le BDNF sont ajoutés (5). Les co-cultures sont ensuite mises en culture pendant 2 semaines pour permettre de différencier les OPC et myélinisent les axones (6). Enfin, les co-cultures sont soit lysées pendant western blot ou fixés pour immunocytochimie (7).
REMARQUE: Jour 1- 2 jours avant la dissection:
- Manteau autoclave 22 mm x 22 mm avec des lamelles couvre-poly-L-ornithine (0,5 mg / ml) dans une plaque à 6 puits et incuber à 37 ° C pendant une nuit.
- Le jour suivant, des lamelles de revêtement à nouveau avec la laminine (20 pg / ml dans du MEM) à 37 ° C jusqu'au lendemain, aspirer l'excès et sécher pendant 20 minutes dans une culture de tissu capot.
REMARQUE: Jour 2: Dissection et l'isolement des DRG: - Sacrifice chiots P2-P3 rat 12x par transection cervicale.
- Retirer la peau recouvrant le muscle du dos de l'animal. Utilisez des ciseaux pour ouvrir Vannas trou vertébral et utiliser des pinces pour ramasser doucement la moelle épinière.
- Arrache DRG qui se trouvent entre les deux colonnes vertébrales et de couper les fibres nerveuses de la moelle attachés, puis placez DRG dans un plat 33 mm de Pétri contenant 3 ml de L-15 médias. Il est généralement possible de réunir entre 8 et 12 DRG de chaque côté de la moelle épinière
- Transfert recueillies DRG ainsi que les L-15 médias dans un tube de 15 ml, centrifuger à 180 g pendant 5 min.
- Aspirerle surnageant, ajouter 2 ml de trypsine à 0,25% DRG pastilles et incuber à 37 ° C pendant 30 min.
- Ajouter 5 ml de milieu M1 (voir le tableau de la solution mère) pour les pastilles de DRG pour arrêter la trypsine, et centrifuger à 180 g pendant 3 min à température ambiante.
- Aspirer le surnageant, remettre en suspension le culot dans 2 ml de milieu M1 préchauffé (pas de facteurs de croissance). Triturer le culot de ganglions par pipetage 50x ou jusqu'à ce que les ganglions sont dispersés. Centrifuger la suspension de cellules à 180 g pendant 3 min.
- Remettre en suspension dissociée neurones dans les médias M1 et la plaque dans une plaque à 6 puits à cinq ganglions par 100 pi par puits.
- Pour supprimer la prolifération des cellules non neuronales, après un minimum de 4 heures d'incubation pour faciliter la fixation, retirer les supports M1 et M2 nourrir les neurones avec les médias (voir le tableau de la solution mère). Culture neurones DRG en présence de NGF (100 ng / ml) pour purifier une culture de NGF-TrkA __gVirt_NP_NN_NNPS<__ charge DRG exprimant. Vous pouvez également utiliser le BDNF (100 ng / ml) pour purifier TrkB-expres de BDNF-dépendantechanter DRG.
- Maintenir neurones dans les médias M1 (+ NGF ou BDNF à 100 ng / ml) alternant avec des antimitotiques M2 médias pendant 2 semaines que ci-dessous.
- Maintenir milieu M1 (+ NGF ou BDNF à 100 ng / ml) les jours: 4-6, 8-10, 12-14 et M2 médias (+ NGF ou BDNF à 100 ng / ml) avec FDU et uridine les jours 1 à 4, 6-8, et 10-12. Un minimum de trois cycles de purification M2 de média est requise.
- Maintenir les DRG dans M1 seul pendant une semaine supplémentaire après avoir terminé le cycle de antimitotique deux semaines. Changer de support M1 tous les 2-3 jours.
5. Isolement et culture des OPC (figure 2 étape 3)
REMARQUE: Jour 1- 2 jours avant la dissection:
- Manteau de 10 cm des plaques de culture de tissu de poly-D-lysine (PDL, 10 ug / ml dans de l'eau déminéralisée stérilisée) à 4 ° C jusqu'au lendemain.
REMARQUE: Jour 2: un jour avant la dissection: - Laver plaques PDL avec de l'eau déminéralisée stérilisée pour 3x. Laisser sécher pendant ~ 6 h dans une culture tissulaire capot. Si ne étant pas utilisé tout de suite, envelopper et magasinjusqu'à 4 semaines à 4 ° C.
- Préparer des plaques d'anticorps secondaires pour immunopanning. Pour une dissection du cerveau, préparer des plaques IgG 2x (pour RAN2 anticorps pour éliminer les astrocytes et les anticorps O1 à retirer premyelinating oligodendrocytes), avec 45 ul IgG chèvre α de la souris dans du DPBS (15 ml) par 10 cm boîte de Pétri; 1x plaque pour IgM (anticorps O4 de sélection de cellules précurseurs d'oligodendrocytes), 45 ul de chèvre α IgM de souris dans du DPBS (15 ml) par 10 cm de boîte de Pétri.
REMARQUE: Jour 3: jour de dissection: - Ajouter 200 unités de la papaïne dans 10 ml de papaïne tampon (tableau 2), et de se réchauffer à 37 ° C jusqu'à ce que le tampon se claire.
Tableau 2: Préparation du tampon de la papaïne.Concentration pour 250 ml La concentration finale EBSS stocks 10x 25 ml 1x MgSO 4 100 mM 2,5 ml 1 mM Glucose 30% 3 ml 0,46% EGTA 0,5 M 1 ml 2 mM NaHCO 3 M 1 6,5 ml 26 mM Amener le volume jusqu'à 250 ml avec de l'eau déminéralisée et stériliser par filtration - Lavez toutes les plaques d'anticorps secondaires avec DPBS pour 3x.
- Verser l'anticorps RAN2 et O1 sur les plaques d'IgG et l'hybridome O4 à la plaque IgM. Incuber tous ces plaques anticorps primaire pendant plus de 2 heures à température ambiante.
- Disséquer un cerveau d'un rat de P7. Décapiter un chiot avec des ciseaux aiguisés et enlever la peau recouvrant le crâne avec des ciseaux.
- Couper autour du crâne du lobe occipital, le lobe temporal et le lobe frontal. Retirer cerveau de crâne à l'aide d'une pince et doucement le transférer à une boîte de Pétri de 35 mm avec 1 ml DPBS.
- Dés le cerveau en petits morceaux à peu près avec des ciseaux ou la lame stérile.
- Filtre préchauffé tampon de la papaïne (10 ml) dans un nouveau tube de 15 ml contenant quelques grains de L-cystéine, puis ajouter 200 ul de désoxyribonucléase (12 500 U / ml) dans le tampon de la papaïne filtré.
- Verser le tampon de la papaïne sur les tissus du cerveau en dés; incuber à 37 ° C pendant 90 min.
- Transfert Gentlly dissocié tissus cérébraux dans le tube de 50 ml en utilisant une pipette de 25 ml etlaisser reposer.
- Éliminer le tampon de la papaïne, ajouter 2 ml Lo Ovo (ovomucoïde) aux tissus du cerveau et titurate 5-10x par pipetage pour briser des morceaux de tissus cérébraux, laisser reposer et enlever les deux premiers ml de surnageant dans un nouveau tube.
- Ajouter un autre 2 ml Lo Ovo aux tissus du cerveau et répétez l'étape 5.11 jusqu'à ce qu'aucun des morceaux de tissus restent. Trituration peut obtenir de plus en plus agressive.
- Centrifuger la suspension cellulaire dissociée pendant 15 min à 200 x g. Aspirer le surnageant et culot de cellules de remettre en suspension dans 10 ml Salut Ovo et centrifuger pendant 15 min à 200 x g.
- Laver première plaque de immunopanning (Ran 2 de la plaque) avec du DPBS pour 3x. Aspirer le surnageant, remettre les cellules dans 10 ml de tampon de panoramique (tableau 2) et verser sur de la première plaque de immunopanning (Ran 2 de la plaque). Incuber les cellules pendant 15 min à température ambiante.
- Laver la seconde plaque de immunopanning (plaque O1) avec du DPBS pour 3x.
- Après l'incubation sur la première plaque de immunopanning, pencher la suspension cellulaire sur la secondeimmunopanning plaque (plaque O1), rincer les cellules lâches de la surface de la plaque avec 1-3 ml de tampon et panoramique prélever à la pipette à la plaque O1. Incuber les cellules pendant 15 min à température ambiante.
- Laver la troisième plaque de immunopanning (plaque O4). Cette plaque est la plaque de sélection positive où les OPC lient sa surface. Laver la plaque avec du DPBS O4 pour 3x.
- Après l'incubation sur la seconde plaque de immunopanning, transférer les cellules sur la plaque de immunopanning final (plaque O4), incuber les cellules pendant 45 min à température ambiante. Cette étape permet de sélectionner O4 + OPC.
- Aspirer le surnageant de la dernière plaque de immunopanning (plaque O4) et rincer la plaque avec EBSS pour 6x.
- Pour supprimer les OPC de la plaque, incuber les cellules avec 5 ml de chaud 0,05% de trypsine-EDTA dilué 1:10 avec EBSS à 37 ° C pendant 8 min.
- Ajouter 5 ml de 30% de FBS (made in EBSS) pour neutraliser la trypsine. Éliminer les cellules hors surface de la plaque par pipetage pour environ 50x.
- Transférer la totalité des cellulessuspension dans un nouveau tube et centrifuger pendant 15 min à 200 x g.
- Rejeter le surnageant et culot cellulaire de remettre en suspension dans 1 ml de médias SATO préchauffé, suivis par comptage cellulaire. Dissection d'un cerveau peut produire de 1,5 à 2.000.000 OPC.
- cellules de plaque sur des plaques recouvertes PDL sèches à une densité comprise entre 1 x 10 5 et 5 x 10 5 par 10 cm plaque avec de SATO médias (10 ml) qui contiennent facteur neurotrophique ciliaire (CNTF, 10 ng / ml), dérivé des plaquettes facteur de croissance (PDGF, 10 ng / ml,), la neurotrophine 3 (NT3, 1 ng / ml), et de la forskoline (4,2 ug / ml) 2,12. Les OPC de culture à 37 ° C, 8% de CO 2.
6. OPC de transduction
- Culture primaire OPC dans SATO médias (10 ml / 10 cm de plaque) avec du CNTF (10 ng / ml), PDGF (10 ng / ml), NT3 (1 ng / ml), et la forskoline (4,2 pg / ml) à 37 ° C, 8% de CO 2 pendant 24 heures après dissection.
- Complètement hors aspirer les milieux de culture OPC, les cellules d'alimentation avec fraîchement préparé médias SATO (10 ml) Avec des facteurs de croissance (voir ci-dessus à l'étape 6.1).
- Ajouter aux virus OPC à la concentration optimale déterminée par l'étape 3 (figure 2, étape 4), les OPC de culture pendant encore 48 heures.
7. OPC classement pour myélinisantes Co-cultures (Figure 2, les étapes 5 et 6)
- Pour supprimer OPC hors de la surface, premières plaques rinçage OPC avec 8 ml EBSS deux fois, puis incuber les cellules avec 5 ml d'eau tiède 0,05% de trypsine-EDTA dilué 1:10 avec EBSS à 37 ° C pendant 2 min.
- Neutraliser le trypsine avec 30% de FBS dans EBSS (5 ml) et éliminer les cellules de la plaque par pipetage. Transférer la suspension cellulaire à un tube de 15 ml, centrifuger à 180 g pendant 15 min à température ambiante.
- Aspirer le surnageant et remettre le culot cellulaire dans 1 ml de médias SATO préchauffé suivie par la numération des cellules.
- Avant l'ensemencement OPC, les médias aspirer complètement de la plaque de culture de DRG. 200000 semences doucement OPC, goutte à goutte sur les neurones DRG comme précédemment décrits 4-6.
REMARQUE:Le volume total d'ensemencement OPC doit être inférieure à 200 ul par 22 mm lamelle. - Laissez cellules de régler sans bouger la plaque pendant 10 min dans la hotte de culture de tissus, puis de haut doucement avec les médias SATO 1 ml préchauffé par puits.
- Réensemencement les autres OPC sœurs avec les médias SATO avec des facteurs de croissance (voir l'étape 6.1). Utilisez ces OPC sœurs pour vérifier l'expression de la protéine d'intérêt.
- Après 24 heures, remplacer le support SATO avec les médias co-culture (2 ml / puits) contenant les médias SATO (pas de facteurs) et neurobasal (v / v) avec 1% B27. Maintenir les co-cultures pour 14 jours avec changement de milieu tous les 2-3 jours.
- Evaluer les co-cultures d'expression de la protéine de myéline par analyse Western blot et de visualiser à la formation de segments axonales myélinisées par immunocoloration double avec des anticorps contre les marqueurs de protéine basique de myéline et des marqueurs neuronaux 6.4.
Résultats
Le drapeau marqués kinase 1 (Flag-Erk1) liés signal construction utilisé pour la production lentivirus est vérifiée par l'enzyme de restriction digérer des constructions utilisées, y compris les constructions d'2K7 et les emballages et accessoires constructions nécessaires à la production de virus extracellulaire (figure 3) .
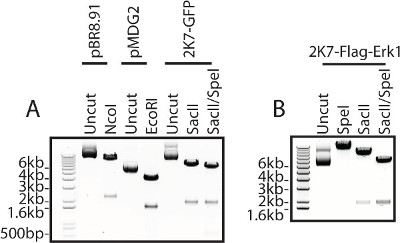
Figure 3:...
Discussion
La myélinisation des axones est un processus crucial pour le fonctionnement optimal des deux systèmes nerveux central et périphérique de vertébrés. La génération et la maintenance des axones myélinisés est un processus complexe et coordonnée impliquant des interactions moléculaires entre neuronale, gliale (à partir de cellules de Schwann ou oligodendrocytes) et protéines de la matrice extra-cellulaire. L'importance et l'applicabilité de ce protocole est qu'il permet la manipulation des protéi...
Déclarations de divulgation
The authors declare that there is no conflict of interest regarding this research.
Remerciements
This work was supported by the Australian National Health and Medical Research Council (NHMRC fellowship #454330 to JX, project grant #628761 to SM and APP1058647 to JX), Multiple Sclerosis Research Australia (MSRA #12070 to JX), the University of Melbourne Research Grant Support Scheme and Melbourne Research CI Fellowship to JX as well as Australia Postgraduate Scholarships to HP and AF. We would like to acknowledge the Operational Infrastructure Scheme of the Department of Innovation, Industry and Regional Development, Victoria Australia.
matériels
| Name | Company | Catalog Number | Comments |
| 2K7 lentivector | Kind gift from Dr Suter9 | ||
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100mg | |
| Alexa Fluor 488 Goat anti-mouse IgG | Jackson Immunoresearch | 115545205 | |
| Alexa Fluor 488 goat anti-rabbit IgG (H+L) | Life Technologies | A11008 | |
| Alexa Fluor 594 goat anti-mouse IgG (H+L) | Life Technologies | A11005 | |
| Alexa Fluor 594 goat anti-rabbit IgG (H+L) | Life Technologies | A11012 | |
| Ampicillin | Sigma-Aldrich | A9518-5G | |
| B27 - NeuroCul SM1 Neuronal Supplement | Stem Cell Technologies | 5711 | |
| BDNF (Human) | Peprotech | PT450021000 | |
| Biotin (d-Biotin) | Sigma Aldrich | B4639 | |
| Bradford Reagent | Sigma Aldrich | B6916-500ML | |
| BSA | Sigma Aldrich | A4161 | |
| Chloramphenicol | Sigma-Aldrich | C0378-100G | |
| CNTF | Peprotech | 450-13020 | |
| DAKO fluoresence mounting media | DAKO | S302380-2 | |
| DMEM, high glucose, pyruvate, no glutamine | Life Technologies | 10313039 | |
| DNase | Sigma-Aldrich | D5025-375KU | |
| DPBS | Life Technologies | 14190250 | |
| DPBS, calcium, magnesium | Life Technologies | 14040182 | |
| EBSS | Life Technologies | 14155063 | |
| EcoRI-HF | NEB | R3101 | |
| Entry vectors for promoter and gene of interest | Generate as per protocols 1-2 | ||
| Fetal Bovine Serum | Sigma-Aldrich | 12003C | |
| Forskolin | Sigma Aldrich | F6886-50MG | |
| Glucose (D-glucose) | Sigma-Aldrich | G7528 | |
| Glycerol | Chem Supply | GL010-500M | See stock solutions |
| Goat Anti-Mouse IgG | Jackson ImmunoResearch | 115005003 | |
| Goat Anti-Mouse IgM | Jackson ImmunoResearch | 115005020 | |
| Goat Anti-Rat IgG | Jackson ImmunoResearch | 112005167 | |
| Hoechst 33342 | Life Technologies | H3570 | |
| Igepal | Sigma Aldrich | I3021-100ML | |
| Insulin | Sigma Aldrich | I6634 | |
| Kanamycin | Sigma-Aldrich | 60615 | |
| Laminin | Life Technologies | 23017015 | |
| LB Medium | See stock solutions | ||
| LB-Agar | See stock solutions | ||
| L-Cysteine | Sigma-Aldrich | C-7477 | |
| Leibovitz's L-15 Medium | Life Technologies | 11415064 | |
| L-Glutamate | Sigma-Aldrich | G1626 | |
| L-Glutamine- 200 mM (100x) liquid | Life Technologies | 25030081 | |
| LR Clonase II Plus enzyme | Life Technologies | 12538-120 | |
| MEM, NEAA, no Glutamine | Life Technologies | 10370088 | |
| Mouse α βIII Tubulin | Promega | G7121 | |
| Mouse αMBP (monoclonal) | Millipore | MAB381 | |
| Na pyruvate | Life Technologies | 11360-070 | |
| NAC | Sigma Aldrich | A8199 | |
| NcoI-HF | NEB | R3193S | |
| NEBuffer 4 | NEB | B7004S | |
| Neurobasal medium | Life Technologies | 21103049 | |
| NGF (mouse) | Alomone Labs | N-100 | |
| NT-3 | Peprotech | 450-03 | |
| O1 antibody - Mouse anti-O1 | Millipore | MAB344 | Alternative if O1 hybridoma cells are unavailable |
| O1 hybridoma cells | Conditioned medium containing anti-O1 antibody to be used for immunopanning | ||
| O4 antibody - Mouse anti-O4 | Millipore | MAB345 | Alternative if O4 hybridoma cells are unavailable |
| O4 hybridoma cells | Conditioned medium containing anti-O4 antibody to be used for immunopanning | ||
| Competent cells | Life Technologies | A10460 | |
| One Shot Stbl competent cells | Life Technologies | C7373-03 | |
| Papain Suspension | Worthington/Cooper | LS003126 | |
| pBR8.91 | Kind gift from Dr Denham10 | ||
| PDGF-AA (Human) | Peprotech | PT10013A500 | |
| Penicillin-streptomycin 100x solution | Life Technologies | 15140122 | |
| pENTRY4IRES2GFP | Invitrogen | 11818-010 | |
| pMD2.G | Addgene | 12259 | |
| Poly-D-lysine | Sigma | P6407-5MG | |
| Polyethylenimine (PEI) | Sigma-Aldrich | 408727-100ML | |
| Poly-L-ornithine | Sigma Aldrich | P3655 | |
| Progesterone | Sigma Aldrich | P8783 | |
| Protease inhibitor tablet (Complete mini) | Roche | 11836153001 | |
| Proteinase K | Supplied with Clonase enzyme | ||
| Putrescine | Sigma Aldrich | P-5780 | |
| Rabbit α neurofilament | Millipore | AB1987 | |
| Rabbit αMBP (polyclonal) | Millipore | AB980 | |
| Ran2 hybridoma cells | ATCC | TIB-119 | Conditioned medium containing anti-Ran2 antibody to be used for immunopanning |
| Rat anti CD140A/PDGFRa antibody | BD Pharmingen | 558774 | |
| SacII | NEB | R0157 | |
| SOC medium | Supplied with competent bacteria | ||
| Sodium selenite | Sigma Aldrich | S5261 | |
| Spe I | NEB | R0133S | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Buffer | NEB | B0202S | |
| TE buffer pH8 | See stock solutions | ||
| TNE lysis buffer | |||
| Trace Elements B | Cellgro | 99-175-CI | |
| Transferrin (apo-Transferrin human) | Sigma-Aldrich | T1147 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Trypsin | Sigma-Aldrich | T9201-1G | |
| Trypsin Inhibitor From Chicken Egg White | Roche | 10109878001 | |
| Trypsin-EDTA (1x), phenol red (0.05%) | Life Technologies | 25300-054 | |
| Unconjugated Griffonia Simplicifolia Lectin BSL-1 | Vector laboratories | L-1100 | |
| Uridine | Sigma-Aldrich | U3003-5G |
Références
- Baumann, N., Pham-Dinh, D. Biology of oligodendrocyte and myelin in the mammalian central nervous system. Physiol Rev. 81 (2), 871-927 (2001).
- Watkins, T. A., Emery, B., Mulinyawe, S., Barres, B. A. Distinct stages of myelination regulated by gamma-secretase and astrocytes in a rapidly myelinating CNS coculture system. Neuron. 60 (4), 555-569 (2008).
- Lee, K., et al. MDGAs interact selectively with neuroligin-2 but not other neuroligins to regulate inhibitory synapse development. Proc Natl Acad Sci U S A. 110 (1), 336-341 (2013).
- Xiao, J., et al. BDNF exerts contrasting effects on peripheral myelination of NGF-dependent and BDNF-dependent DRG neurons. J Neurosci. 29 (13), 4016-4022 (2009).
- Chan, J. R., et al. NGF controls axonal receptivity to myelination by Schwann cells or oligodendrocytes. Neuron. 43 (2), 183-191 (2004).
- Xiao, J., et al. Brain-Derived Neurotrophic Factor Promotes Central Nervous System Myelination via a Direct Effect upon Oligodendrocytes. Neurosignals. 18 (3), 186-202 (2010).
- Lundgaard, I., et al. Neuregulin and BDNF induce a switch to NMDA receptor-dependent myelination by oligodendrocytes. PLoS Biology. 11 (12), e1001743 (2013).
- Kleitman, N., W, P. M., Bunge, R. P. . Tissue culture methodes for the study of myelination. , (1991).
- Xiao, J., et al. Extracellular signal-regulated kinase 1/2 signaling promotes oligodendrocyte myelination in vitro. J Neurochem. 122 (6), 1167-1180 (2012).
- Wong, A. W., Xiao, J., Kemper, D., Kilpatrick, T. J., Murray, S. S. Oligodendroglial expression of TrkB independently regulates myelination and progenitor cell proliferation. The Journal of Neuroscience. 33 (11), 4947-4957 (2013).
- Li, Z., et al. Molecular cloning, Characterization and Expression of miR-15a-3p and miR-15b-3p in Dairy Cattle. Molecular and Cellular Probes. , (2014).
- Emery, B., et al. Myelin gene regulatory factor is a critical transcriptional regulator required for CNS myelination. Cell. 138 (1), 172-185 (2009).
- Murai, K., et al. Nuclear receptor TLX stimulates hippocampal neurogenesis and enhances learning and memory in a transgenic mouse model. Proc Natl Acad Sci U S A. 111 (25), 9115-9120 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon