È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Produzione e l'uso di Lentivirus a celle selettivamente trasdurre primaria Oligodendrocyte precursore
In questo articolo
Riepilogo
Here we present protocols that offer a flexible and strategic foundation for virally manipulating oligodendrocyte precursor cells to overexpress proteins of interest in order to specifically interrogate their role in oligodendrocytes via the in vitro model of central nervous system myelination.
Abstract
Mielinizzazione è un processo complesso che coinvolge sia i neuroni e la mielina che formano le cellule gliali, oligodendrociti nel sistema nervoso centrale (SNC) e le cellule di Schwann nel sistema nervoso periferico (PNS). Usiamo una vitro mielinizzazione test in un modello stabilito per lo studio del sistema nervoso centrale mielinizzazione in vitro. Per fare questo, le cellule precursori degli oligodendrociti (OPC) vengono aggiunti i primari roditore dorsale radice gangli (DRG) neuroni purificati per formare co-culture mielinizzanti. Per interrogare specificamente i ruoli che particolari proteine espresse da oligodendrociti esercitano su di mielinizzazione abbiamo sviluppato protocolli che trasducono selettivamente OPC con il lentivirus overexpressing wild type, proteine negativi costitutivamente attive o dominanti prima di essere testa di serie sui neuroni DRG. Questo ci permette di interrogare in particolare il ruolo di queste proteine nella regolazione oligodendrogliali mielinizzazione. I protocolli possono essere applicati anche nello studio di otsuoi tipi cellulari, fornendo così un metodo che permette la manipolazione selettiva di proteine espresse da un tipo cellulare desiderato, come oligodendrociti per lo studio mirato di segnalazione e meccanismi di compensazione. In conclusione, combinando la mielinizzazione test in vitro con lentivirali infettato OPC fornisce uno strumento strategico per l'analisi dei meccanismi molecolari coinvolti nella mielinizzazione.
Introduzione
Mielinizzazione degli assoni è fondamentale per la trasmissione rapida ed efficiente dei potenziali d'azione sia nel sistema nervoso centrale e periferico. Cellule specializzate, le cellule di Schwann nel sistema nervoso periferico e oligodendrociti nel sistema nervoso centrale, avvolgono e ensheathe assoni in mielina, efficace isolamento del nervo e facilitando la conduzione saltatory 1. Il processo di mielinizzazione può essere studiato in vitro utilizzando retina neuroni gangliari 2, nanofibre ingegnerizzati 3, o neuroni dei gangli delle radici dorsali co-coltura sia con le cellule di Schwann 4 o oligodendrociti 5-7. Il saggio mielinizzazione in vitro è un modello stabilito per lo studio mielinizzazione del sistema nervoso e si replica molti dei processi fondamentali che avvengono durante la mielinizzazione in vivo 5-8. Il test prevede la co-coltura di popolazioni purificate di dorsale Root Ganglion (DRG) neuroni, con OPC (per CNS mielinizzazione) o cellule di Schwann (per PNS mielinizzazione). In condizioni particolari queste cellule mielinizzanti ensheathe assoni DRG in ordinata, ultra strutturalmente verificato, scheda multi-lamellare di isolamento membrana plasmatica che esprimono lo stesso complemento di specifiche proteine della mielina presenti in vivo.
Il modello cellulare più comunemente usato di studiare CNS mielinizzazione in vitro è co-colture di neuroni DRG e OPC, che sono stati usati con successo per studiare l'effetto che i fattori esogeni, quali le neurotrofine esercitano sul sistema nervoso centrale mielinizzazione in vitro 5,6. Fattori esogeni, quali fattori di crescita o piccole molecole inibitori farmacologici sono stati ampiamente utilizzati per studiare il ruolo delle vie di segnalazione nella mielinizzazione utilizzando il DRG-OPC modello coculture 7,9. Tuttavia, nelle impostazioni co-coltura miste contenenti sia i neuroni e oligodendrociti, rimase formalmente possibile che sia i fattori di crescita o il pharinibitori ve- avrebbero potuto esercitare effetti su entrambi i neuroni DRG e oligodendrociti (OL). Questo non offre la possibilità di sezionare specificamente i ruoli che le proteine espresse solo DRG o oligodendroglia esercita su mielinizzazione con questo sistema a doppia cella. Per confermare in modo inequivocabile che il percorso di segnalazione in oligodendrogliale regola direttamente mielinizzazione, trasduzione lentivirale di OPC, prima della semina su neuroni DRG per il saggio mielinizzazione in vitro, ha dimostrato di essere un modo elegante per iperespressione sia wild-type e le proteine mutanti, come pure come espressione atterramento di proteine costitutivamente espresse da oligodendrociti. Così questo approccio offre un viale di interrogare in modo specifico e manipolare vie di segnalazione all'interno oligodendrociti per studiare mielinizzazione 9,10.
In questo lavoro, si segnala metodi che abbiamo sviluppato per iperespressione una proteina di interesse selettivamente in oligodendrociti mediante lentiviralapproccio per studiare mielinizzazione in vitro. La tecnica inizia con la generazione di vettori di espressione contenenti il gene di interesse, sia esso in un ceppo selvatico, forma negativa costitutivamente attiva o dominante che vengono successivamente clonato nel vettore pENTR (pENTR L1-L2 pENTR4IRES2GFP). Questo vettore (contenente il gene di interesse), il donatore CMV promotore (pENTR L4-R1-pENTR pDNOR-CMV) e la lentivector 2K7 sono combinati in una reazione enzimatica per produrre un vettore contenente 2K7 CMV promoter, il gene di interesse, un sito interno ribosomiale ingresso e GFP (Figura 1). Questo Gateway clonato costrutto 2K7 combinata con la busta virus PMD2.G e il pacchetto di virus pBR8.91 può essere co-trasfettato in cellule HEK293T generare lentivirus che può essere successivamente utilizzato per trasdurre OPC. Una volta infettati con il lentivirus le OPC esprimono un alto livello della proteina di interesse. Questi OPC possono essere seminati in colture DRG neuroni e l'effetto che l'espressionedi elevati livelli della proteina desiderata esercita sulla mielinizzazione possono essere interrogato. Le co-colture sono valutati per l'espressione delle proteine della mielina mediante analisi western blot e visualizzati per la formazione di segmenti assonale mieliniche mediante immunocitochimica.
Protocollo
NOTA: Tutti gli animali utilizzati per questo studio erano di sesso misto e allevato presso le strutture animali del Dipartimento di Anatomia e Patologia e l'Istituto di Neuroscienze Florey e Mental Health Research presso l'Università di Melbourne. Tutte le procedure di animali sono state approvate dalla sperimentazione animale comitati etici presso l'Università di Melbourne.
1. Clonazione di 2K7 lentivector
- Prima di clonare il gene di interesse nella 2K7 vettore lentivirale, subclone il gene nel vettore pENTR (3637 bp, Kanamicina resistente) utilizzando tecniche molecolari standard di 11. Utilizzare i siti di restrizione EcoRI e SacII per il subcloning.
- Amplificazione del 2K7 lentivector
- Transform 2K7 lentivector DNA mescolando delicatamente 100 ng di DNA plasmidico con cellule competenti e incubare su ghiaccio per 30 min.
- Scossa di calore DNA / cellule competenti miscela a 42 ° C per 90 secondi e piastra su piastre LB-Agar contenenti sia ampicillin (100 mcg / ml) e cloramfenicolo (15 mg / ml) a 37 ° C per 16-18 ore.
NOTA: cloramfenicolo è utilizzato per prevenire la ricombinazione tra le lunghe ripetizioni terminali. - Il giorno successivo, crescere selezionato cloni batterici in mezzi LB contenenti sia ampicillina (100 ug / ml) e cloramfenicolo (15 mg / ml) a 37 ° C per 16-18 ore. Estrarre il DNA utilizzando un plasmide commerciale kit Maxiprep DNA secondo le istruzioni del produttore.
- Sub-clonazione dal pENTR vettore a 2K7 lentivector (Figura 1)
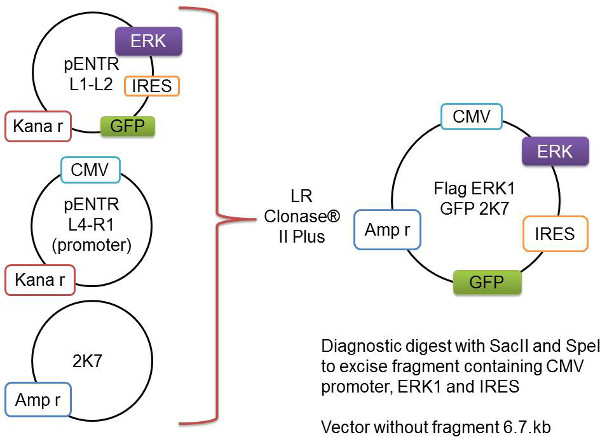
Figura 1:. Rappresentazione schematica del processo di ricombinazione Gateway Il gene di interesse, qui rappresentato come Flag-Erk1, viene clonato nel vettore pENTR L1-L2. Questo viene aggiunto al vettore pENTR L4-R1 contenente il promotore CMV e il backbone 2K7 vettoriale. Questi tre vettori vengono ricombinati dall'enzima LR Clonase II Plusinserire il gene di interessi e il promotore nel 2K7 vettore del virus-ready.- Aggiungere il seguente in una provetta da 1,5 ml e mescolare delicatamente:
L4-R1 pENTR-pDNOR-CMV (promoter) (60 BNG) 1-2,5 ml
L1-L2 pENTR4IRES2GFP (con gene di interesse) (80 BNG) 1-2,5 ml
K7 lentivector (80 ng / ml) 1 ml
TE-buffer, pH 82-5 microlitri
Totale 8 ml - Rimuovere mix enzima clonase da -80 ° C e scongelare il ghiaccio per 2 minuti. Assicurarsi che questo enzima è una nuova aliquota di -80 ° C, come l'enzima ha sostanzialmente ridotto la clonazione efficienza con ripetuti cicli di gelo-disgelo
- Aggiungere 2 ml di enzima per reazione e mescolare bene con vortex e incubare la miscela di reazione clonase a 23-25 ° C per 6-24 ore.
- Aggiungere 1 ml di soluzione di proteinasi K (fornito con il kit enzimatico) alla miscela di reazione clonase e incubare per 10 min a 37 ° C.
- Trasforma miscela di reazione clonase in cellule competenti e far crescere il selectcolonie nei mezzi LB contenente ampicillina (100 ug / ml) a 37 ° C per 16-18 ore.
- Estrarre e purificare il DNA utilizzando un plasmide commerciale kit Miniprep DNA secondo le istruzioni del produttore.
- Conferma il DNA mediante digestione con enzimi di restrizione Spe I e Sac II e un tampone appropriato come da istruzioni del produttore per rimuovere il frammento contenente il promotore e gene di interesse.
NOTA: Questo passaggio è fondamentale per verificare se il DNA subclonato ha il gene corretto inserimento di interessi con il vettore backbone destra. La dimensione del vettore stesso senza frammento inserito è di 6,7 kb. La dimensione del frammento inserito rilasciato include sia il promotore e il gene di interesse inserto. Calcolare le dimensioni dei frammenti attesi dal digest per il gene specifico con un programma di editing sequenza di DNA, ad esempio, APE - Un Editor plasmidi. - Controllare le dimensioni sia vettore backbone DNA e il frammento rilasciato eseguendo un gel di agarosio 1%in 1x tampone TAE (vedi Tabella di soluzioni madre) a 100 V.
- Amplificazione del DNA confermato dalla crescente batteri in 500 ml di mezzi di LB contenenti ampicillina (100 ug / ml) a 37 ° C per 16-18 ore.
- Estrarre e purificare il DNA utilizzando un plasmide commerciale kit Maxiprep DNA secondo le istruzioni del produttore.
NOTA: Maxiprep genera tipicamente quantità sufficiente di DNA necessarie per la produzione virale.
- Aggiungere il seguente in una provetta da 1,5 ml e mescolare delicatamente:
- Ripetere i punti 1.3.9-1.3.10 per amplificare i seguenti DNA per i preparati lentivirali: Envelope vettore (PMD2.G, 6.1 kb), vettore Package (pBR8.91, 12,5 kb), e un vettore lentivirale vuoto come controllo (GFP -CMV-2K7, 8.7 kb).
2. 2K7 Virus Produzione
NOTA: 1 ° giorno:
- Il giorno della trasfezione, piatto 32 milioni di cellule HEK293 T a pallone T175 contenente 25 ml di mezzi di cellule HEK293 T (vedere la tabella delle soluzioni madre). In alternativa, piatto 16 milioni di cellule del giornoprima della trasfezione se il tempo scorre a tenuta il giorno della trasfezione.
NOTA: Transfection può essere ugualmente efficace da placcatura cellule il giorno della trasfezione o prima del giorno. Utilizzando una delle due alternative, il punto critico è quello di assicurarsi che le cellule siano bloccati sul cultura superficiale piatto prima di trasfezione.
NOTA: 2 ° giorno: - Transfection
- Prima di trasfezione, diluire il DNA a 1 mg / mL in TE buffer che contiene 10 mM Tris pH 8, 1 mM EDTA pH 8 in acqua deionizzata.
- In un tubo da 50 ml, preparare una miscela master (Tabella 1) per la trasfezione nel pallone T175. Aggiungi DNA pre-riscaldato di Dulbecco modificato (DMEM) e mescolare bene dal vortice, quindi aggiungere polyethylenimine sterile (PEI) (vedere la tabella delle soluzioni madre) per evitare la precipitazione prematura. ">
Tabella 1: Preparazione mix trasfezione per 2K7 Virus.Vettore trong> Concentrazione Volume pMDG.2 1 mg / mL 5 ml pBR8.91 1 mg / mL 15 microlitri 2K7 vettore con GFP + gene di interesse 1 mg / mL 22 microlitri Polietilenimmina sterile (PEI) 1 g / L 500 microlitri DMEM 2.100 ml - Mescolare bene capovolgendo la 3-4x tubo e incubare per 15 min a temperatura ambiente (RT).
- Eseguire un cambio completo dei media sulle cellule HEK293T. Aspirare fuori il terreno di coltura di cellule completamente e mangimi con mezzi di coltura cellulare HEK293T pre-riscaldato (25 ml per pallone T175).
- Aggiungere la miscela di trasfezione (DNA miscela / PEI) gocciolamento alle cellule monostrato. Spostare delicatamente per amalgamare bene e incubare cellule trasfettate a 37 ° C, 5% CO 2, durante la notte.
NOTA: Day 3: - Per garantire la transfezione è successo, controlla l'espressione della GFP 24 ore dopo la trasfezione tramite un microscopio a fluorescenza.
NOTA: Oltre il 50% delle cellule che esprimono GFP indica in genere un buon trasfezione.
NOTA: Day 4:
- Al 48 ore dopo la trasfezione, raccogliere il surnatante virale e sostituirli con mezzi freschi HEK293 T (25 ml per pallone T175).
- Centrifugare il surnatante virale a 1.140 xg per 10 min a 4 ° C per eliminare i detriti di cellule dal supernatante. Trasferimento eliminato il surnatante in tubo da 50 ml e conservare a 4 ° C.
NOTA: Day 5:
- Centrifugare il surnatante virale a 1.140 xg per 10 min a 4 ° C per eliminare i detriti di cellule dal supernatante. Trasferimento eliminato il surnatante in tubo da 50 ml e conservare a 4 ° C.
- A 72 ore dopo la trasfezione, raccogliere il secondo lotto di surnatante virale. Ripetere il passaggio 2.3.1 e piscina 48 e 72 ore cancellati surnatanti.
- Per concentrare il virus, surnatante virale centrifugare a 170.000 xg per 90 min a 4 ° C con 30 ml provette ultracentrifuga.
- Eliminare il supernatante e ripetere il passaggio 2,6 finché tutto il supernatante è stato eliminato centrifugato lasciando un pellet (invisibile) di virus più precipitato PEI nella base del tubo.
- Per risospendere virus, aggiungere 500 microlitri SATO supporti (vedi Tabella di soluzioni madre) per i tubi ultracentrifuga. Vortex per 30 secondi unnd raschiare la base del tubo con un puntale per allentare meccanicamente il virus. Ripetere questo passaggio 6x per perdere pellet virale.
- Pool il virus risospeso in microprovette e spin brevemente per rimuovere insolubile PEI. Filtrare il surnatante attraverso un filtro da 0,45 micron per rimuovere le proteine.
- Aliquota del virus in 20 microlitri, 50 ml e 100 microlitri aliquote e conservare a -80 ° C.
3. Viral determinazione del titolo in celle HEK293T
- Per controllare l'espressione della proteina di interesse e per determinare la concentrazione virale ottimale per esperimenti, aggiungere una serie di diluizioni seriali di magazzino virale (ad esempio, 0, 5, 10, 20, 40, 80 microlitri) di trasdurre cellule HEK293 T placcati in 6 pozzetti, e la cultura per 24 ore a 37 ° C, 5% CO 2. Questo protocollo genera tipicamente virus che può essere usata a concentrazioni che vanno da 1:50 a 1: 200 che raggiungono robusta espressione del gene diinteresse.
- 48 ore dopo la trasduzione virale, le cellule HEK293T lisi in tampone TNE (vedi Tabella di soluzione) con inibitori della proteasi.
- Sciacquare pozzi due volte con DPBS refrigerati e poi aggiungere del buffer TNE 150 microlitri di ogni bene.
- Lisare le cellule pipettando su e giù 5-10x. Trasferire lisati cellulari totali a 1,5 ml provetta e incubare in ghiaccio per 15-30 min.
- Centrifugare lisati a 4 ° C per 30 min alla massima velocità (20.000 xg) e trasferimento eliminato surnatante in una nuova provetta per la determinazione delle proteine mediante Bradford e successiva analisi Western blot.
- Determinare il livello di espressione della proteina di interesse mediante analisi Western Blot standard mentre sondando per gli anticorpi contro la proteina stessa e il tag fuso (cioè, Bandiera). Utilizzare la diluizione virale che produce le cellule> 95% GFP + e robusta espressione della proteina di interesse per gli esperimenti.
4. Isolamento e la coltura di DRG (FIGURA 2 passaggi 1 & 2)
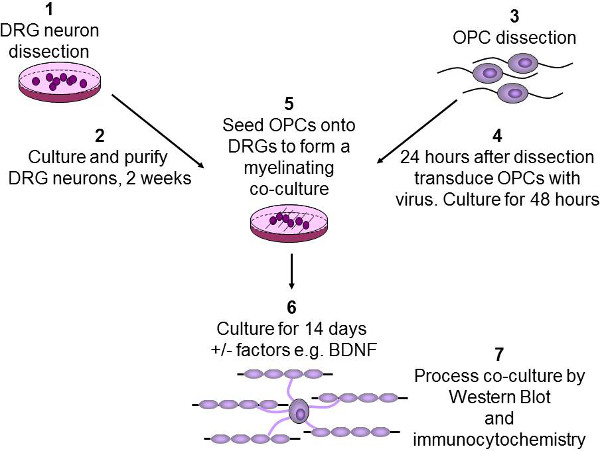
Figura 2:. Schema del test in vitro mielinizzazione neuroni DRG sono sezionati da P2-3 cuccioli di ratto, poi purificate e coltivate più di due settimane (1-2). OPC sono purificati da P7-9 cervello dei topi utilizzando immunopanning (3). OPC vengono quindi infettate con lentivirus e coltivate per 48 ore (4). OPC vengono seminate su DRG, ed eventuali fattori di crescita di interesse, come il BDNF sono aggiunti (5). Co-culture sono poi coltivate per 2 settimane per consentire OPC di differenziare e myelinate gli assoni (6). Infine, co-colture sono o lisate per western blotting o fissati per immunocitochimica (7).
NOTA: Day 1- 2 giorni prima dissezione:
- Coat autoclavato 22 mm x 22 millimetri coprioggetto con Poly-L-ornitina (0,5 mg / ml) in una piastra da 6 pozzetti e incubare a 37 ° C durante la notte.
- Il giorno successivo, vetrini cappotto nuovo con laminina (20 mg / ml di MEM) a 37 ° C durante la notte, aspirare l'eccesso e asciugare per 20 minuti in cappa coltura tissutale.
NOTA: Giorno 2: dissezione e isolamento dei DRG: - Cuccioli Sacrifice P2-P3 ratto 12x da transection cervicale.
- Rimuovere la pelle sovrastante il muscolo dalla parte posteriore dell'animale. Utilizzare Vännäs forbici per aprire forame vertebrale e utilizzare pinze per scavare delicatamente il midollo spinale.
- Pluck out DRG che si trovano tra le colonne vertebrali e tagliare le fibre nervose spinali collegati, luogo DRG in un piatto 33 millimetri Petri contenente 3 ml L-15 media. Di solito è possibile raccogliere tra 8 e 12 DRG da ogni lato del midollo spinale
- Trasferire raccolti DRG insieme alle L-15 supporti in un tubo di 15 ml, centrifugare a 180 xg per 5 min.
- Aspirareil supernatante, si aggiungono 2 ml di 0,25% tripsina per DRG pellets e incubare a 37 ° C per 30 min.
- Aggiungere 5 ml di M1 media (vedi Tabella di soluzione) per il pellet DRG per fermare tripsina e centrifugare a 180 xg per 3 minuti a temperatura ambiente.
- Aspirare il surnatante, risospendere il pellet in 2 ml preriscaldata medio M1 (non con fattori di crescita). Triturare il pellet gangli pipettando 50 volte o fino a quando i gangli sono dispersi. Sospensione cellulare Centrifugare a 180 xg per 3 min.
- Risospendere dissociato neuroni nei media M1 e piastra giù in un 6-pozzetti a 5 gangli per 100 ml per pozzetto.
- Per rimuovere proliferanti cellule non neuronali, dopo un minimo di 4 ore di incubazione per facilitare il fissaggio, rimuovere i supporti M1 e nutrire i neuroni con M2 supporti (vedi Tabella di soluzione). Culture DRG neuroni in presenza di NGF (100 ng / ml) per purificare una cultura di NGF-dipendente-TrkA esprimono DRG. In alternativa, utilizzare BDNF (100 ng / ml) per purificare TrkB-Expres BDNF-dipendentecantare DRG.
- Mantenere i neuroni a M1 supporti (+ NGF o BDNF a 100 ng / ml) si alternano a antimitotica M2 supporto per 2 settimane come sotto.
- Mantenere mezzo M1 (+ NGF o BDNF a 100 ng / ml) a giorni: 4-6, 8-10, 12-14 e M2 supporti (+ NGF o BDNF a 100 ng / ml) con FDU e uridina nei giorni 1 fino 4, 6-8 e 10-12. È richiesto un minimo di 3 cicli di M2 depurazione media.
- Mantenere DRG in M1 solo per un'altra settimana dopo aver completato il ciclo di antimitotici 2 settimane. Cambiare i media M1 ogni 2-3 giorni.
5. Isolamento e la coltura di OPC (figura 2 step 3)
NOTA: Day 1- 2 giorni prima dissezione:
- Coat 10 centimetri piastre di coltura tissutale con Poly-D-Lisina (PDL, 10 mg / ml di acqua deionizzata sterile) a 4 ° C durante la notte.
NOTA: 2 ° giorno: 1 giorno prima dissezione: - Lavare le piastre PDL con acqua deionizzata sterile per 3x. Lasciare asciugare per ~ 6 ore in cappa coltura tissutale. Se non utilizzato immediatamente, avvolgere e conservarefino a 4 settimane a 4 ° C.
- Preparare piatti anticorpi secondari per immunopanning. Per 1 cervello dissezione, preparare piatti IgG 2x (per Ran2 anticorpi per eliminare astrociti e anticorpi O1 per rimuovere premyelinating oligodendrociti), con 45 microlitri IgG topo Capra α in DPBS (15 ml) per 10 cm piastra di Petri; Piastra 1x IgM per (O4 anticorpi per la selezione di cellule precursori degli oligodendrociti), 45 ml di capra α topo IgM in DPBS (15 ml) per 10 cm piastra di Petri.
NOTA: Giorno 3: giorno della dissezione: - Aggiungere 200 unità di papaina in 10 ml di tampone papaina (Tabella 2), e riscaldare a 37 ° C finché il buffer si chiara.
Tabella 2: Preparazione di tampone papaina.Concentrazione per 250 ml Concentrazione finale EBSS magazzino 10x 25 ml 1x MgSO4 100 MM 2,5 ml 1 mM Glucosio 30% 3 ml 0,46% EGTA 0.5 M 1 ml 2 mm NaHCO 3 1 M 6,5 ml 26 mm Portare il volume fino a 250 ml con acqua deionizzata e filtro sterilizzare - Lavare tutti i piatti di anticorpi secondari con DPBS per 3x.
- Versare l'anticorpo Ran2 e O1 sulle piastre IgG e ibridoma O4 alla piastra IgM. Incubare tutti questi piatti anticorpo primario per oltre 2 ore a RT.
- Sezionare un cervello da un ratto P7. Decapitare un cucciolo con le forbici affilate e togliere la pelle sovrastante il cranio con le forbici.
- Tagliare tutto il cranio dal lobo occipitale, lobo temporale e lobo frontale. Togliere dal cervello cranio con una pinza e delicatamente trasferirlo una capsula di Petri da 35 mm con 1 ml DPBS.
- Tagliare a dadini il cervello in piccoli pezzi circa con le forbici o lame sterili.
- Filtro pre-riscaldato tampone papaina (10 ml) in un nuovo tubo da 15 ml contenente alcuni grani di L-cisteina, quindi aggiungere 200 ml DNAase (12.500 U / ml) al tampone papaina filtrato.
- Versare il buffer papaina sui tessuti cerebrali a dadini; incubare a 37 ° C per 90 min.
- Trasferimento Gentlly dissociato tessuti cerebrali in tubo da 50 ml con una pipetta 25 ml elasciar riposare.
- Rimuovere buffer di papaina, aggiungere 2 ml Lo Ovo (ovomucoid) ai tessuti cerebrali e titurate 5-10x pipettando per rompere pezzi di tessuti cerebrali, lasciare riposare e rimuovere gli ultimi 2 ml di surnatante in una nuova provetta.
- Aggiungi un altro 2 ml Lo Ovo ai tessuti cerebrali e ripetere il punto 5.11 fino a quando rimangono senza pezzi di tessuto. Tituration può ottenere sempre più aggressivo.
- Centrifugare dissociato sospensione cellulare per 15 min a 200 x g. Aspirare il surnatante e risospendere pellet cellulare in 10 ml Hi Ovo e centrifugare per 15 minuti a 200 x g.
- Lavare primo piatto immunopanning (Ran 2 piastra) con DPBS per 3x. Aspirare il surnatante, risospendere le cellule in 10 ml di panning tampone (Tabella 2) e versare al primo piatto immunopanning (Ran 2 piastra). Incubare le cellule per 15 minuti a RT.
- Lavare la seconda piastra immunopanning (piatto O1) con DPBS per 3x.
- Dopo l'incubazione sul primo piatto immunopanning, punta la sospensione cellulare sul secondoPiastra immunopanning (piastra O1), risciacqui eventuali cellule sciolto dalla superficie della piastra con 1-3 ml di tampone di panning e trasferimento dalla pipetta alla piastra O1. Incubare le cellule per 15 minuti a RT.
- Lavare la terza piastra immunopanning (piatto O4). Questo piatto è il piatto di selezione positiva dove le OPC legano la sua superficie. Lavare la piastra O4 con DPBS per 3x.
- Dopo l'incubazione sulla seconda piastra immunopanning, trasferire le cellule alla piastra immunopanning finale (piastra O4), incubare le cellule per 45 minuti a RT. Questo passaggio seleziona O4 + OPC.
- Aspirare il surnatante dall'ultimo piastra immunopanning (piatto O4) e lavare la piastra con EBSS per 6x.
- Per rimuovere OPC dalla piastra, incubare le cellule con 5 ml di caldo 0,05% tripsina-EDTA diluito 1:10 con EBSS a 37 ° C per 8 min.
- Aggiungere 5 ml di 30% FBS (made in EBSS) per neutralizzare la tripsina. Rimuovere le cellule dalla superficie della piastra pipettando per circa 50x.
- Trasferire tutto cellsospensione in una nuova provetta e centrifugare per 15 min a 200 x g.
- Gettare il surnatante e risospendere pellet di cellule in 1 ml di media SATO pre-riscaldato, seguiti da conteggio delle cellule. Dissezione di un cervello può produrre 1,5-2.000.000 OPC.
- Cellule piastra su piastre PDL rivestite secco con una densità tra 1 x 10 5 e 5 x 10 5 per 10 cm con piastra in SATO media (10 ml) che contengono fattore neurotrofico ciliare (CNTF, 10 ng / ml), piastrine fattore di crescita derivato (PDGF, 10 ng / ml,), neurotrofine 3 (NT3, 1 ng / ml), e forskolina (4,2 mcg / ml) 2,12. OPC Cultura a 37 ° C, 8% di CO 2.
6. OPC trasduzione
- OPC Culture primarie SATO supporti (10ml / 10 centimetri piatto) con CNTF (10 ng / ml), PDGF (10 ng / ml), NT3 (1 ng / ml), e forskolina (4,2 mcg / ml) a 37 ° C, 8% di CO 2 per 24 ore dopo la dissezione.
- Completamente Aspirare fuori dal terreno di coltura OPC, le cellule di alimentazione con appena fatto i media SATO (10 ml) Con fattori di crescita (vedi sopra al punto 6.1).
- Aggiungere virus alle OPC alla concentrazione ottimale determinato dal passo 3 (Figura 2, passaggio 4), OPC coltura per ulteriori 48 ore.
7. OPC Seeding per mielinizzanti co-culture (Figura 2, punti 5 e 6)
- Per rimuovere OPC dalla superficie, primi piatti risciacquo OPC con 8 ml EBSS due volte, poi incubare cellule con 5 ml di caldo 0,05% tripsina-EDTA diluito 1:10 con EBSS a 37 ° C per 2 min.
- Neutralizzare la tripsina con il 30% FBS in EBSS (5 ml) e rimuovere le cellule dalla piastra pipettando. Trasferimento sospensione cellulare ad un tubo 15 ml, centrifugare a 180 xg per 15 minuti a RT.
- Aspirare il surnatante e risospendere pellet di cellule in 1 ml di media SATO pre-riscaldato, seguiti dal numero delle cellule.
- Prima della semina OPC, supporti completamente aspirato dalla piastra di coltura DRG. Seed delicatamente 200.000 OPC scendono saggio a neuroni DRG come precedentemente descritto 4-6.
NOTA:Il volume totale OPC semina deve essere inferiore a 200 microlitri per 22 millimetri coprioggetto. - Lasciare le cellule di risolvere senza spostare la piastra per 10 minuti nel cofano coltura di tessuti, poi in alto delicatamente con i media SATO 1 ml di pre-riscaldato per bene.
- Replate rimanenti OPC sorella con SATO media con fattori di crescita (vedi punto 6.1). Utilizzare questi OPC sorella per verificare l'espressione della proteina di interesse.
- Dopo 24 ore, sostituire il supporto SATO con i mezzi di co-coltura (2 ml / pozzetto) contenente SATO supporti (senza fattori) e Neurobasal (v / v) con 1% B27. Mantenere le co-colture per 14 giorni con il cambiamento dei media ogni 2-3 giorni.
- Valutare le co-colture per l'espressione delle proteine della mielina mediante analisi western blot e visualizzare per la formazione di segmenti assonale mieliniche doppio immunocolorazione con anticorpi contro la mielina marcatori proteici di base e marcatori neuronali 4-6.
Risultati
Il segnale legate chinasi 1 (Flag-Erk1) costrutto usato per la produzione di lentivirus è verificato da enzima di restrizione digerire dei costrutti utilizzati, inclusi sia i costrutti 2K7 ei costrutti packaging e accessori necessari per la produzione di virus extracellulare (Figura 3) flag-tagged .
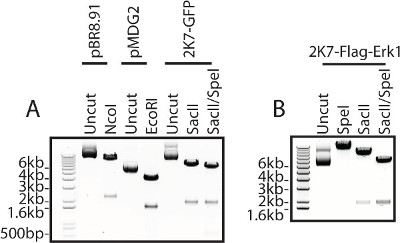
Figura 3: verifica costrutto DNA. Tutti i c...
Discussione
Mielinizzazione di assoni è un processo fondamentale per il funzionamento ottimale di entrambi i sistemi nervoso centrale e periferico di vertebrati. La generazione e la manutenzione di assoni mielinizzati è un processo complesso e coordinato che coinvolge interazioni molecolari tra neuronale, gliale (dalle cellule di Schwann o oligodendrociti) e proteine extracellulari della matrice. Il significato e l'applicabilità di questo protocollo è che permette la manipolazione delle proteine in un tipo di ce...
Divulgazioni
The authors declare that there is no conflict of interest regarding this research.
Riconoscimenti
This work was supported by the Australian National Health and Medical Research Council (NHMRC fellowship #454330 to JX, project grant #628761 to SM and APP1058647 to JX), Multiple Sclerosis Research Australia (MSRA #12070 to JX), the University of Melbourne Research Grant Support Scheme and Melbourne Research CI Fellowship to JX as well as Australia Postgraduate Scholarships to HP and AF. We would like to acknowledge the Operational Infrastructure Scheme of the Department of Innovation, Industry and Regional Development, Victoria Australia.
Materiali
| Name | Company | Catalog Number | Comments |
| 2K7 lentivector | Kind gift from Dr Suter9 | ||
| 5-Fluoro-2′-deoxyuridine | Sigma-Aldrich | F0503-100mg | |
| Alexa Fluor 488 Goat anti-mouse IgG | Jackson Immunoresearch | 115545205 | |
| Alexa Fluor 488 goat anti-rabbit IgG (H+L) | Life Technologies | A11008 | |
| Alexa Fluor 594 goat anti-mouse IgG (H+L) | Life Technologies | A11005 | |
| Alexa Fluor 594 goat anti-rabbit IgG (H+L) | Life Technologies | A11012 | |
| Ampicillin | Sigma-Aldrich | A9518-5G | |
| B27 - NeuroCul SM1 Neuronal Supplement | Stem Cell Technologies | 5711 | |
| BDNF (Human) | Peprotech | PT450021000 | |
| Biotin (d-Biotin) | Sigma Aldrich | B4639 | |
| Bradford Reagent | Sigma Aldrich | B6916-500ML | |
| BSA | Sigma Aldrich | A4161 | |
| Chloramphenicol | Sigma-Aldrich | C0378-100G | |
| CNTF | Peprotech | 450-13020 | |
| DAKO fluoresence mounting media | DAKO | S302380-2 | |
| DMEM, high glucose, pyruvate, no glutamine | Life Technologies | 10313039 | |
| DNase | Sigma-Aldrich | D5025-375KU | |
| DPBS | Life Technologies | 14190250 | |
| DPBS, calcium, magnesium | Life Technologies | 14040182 | |
| EBSS | Life Technologies | 14155063 | |
| EcoRI-HF | NEB | R3101 | |
| Entry vectors for promoter and gene of interest | Generate as per protocols 1-2 | ||
| Fetal Bovine Serum | Sigma-Aldrich | 12003C | |
| Forskolin | Sigma Aldrich | F6886-50MG | |
| Glucose (D-glucose) | Sigma-Aldrich | G7528 | |
| Glycerol | Chem Supply | GL010-500M | See stock solutions |
| Goat Anti-Mouse IgG | Jackson ImmunoResearch | 115005003 | |
| Goat Anti-Mouse IgM | Jackson ImmunoResearch | 115005020 | |
| Goat Anti-Rat IgG | Jackson ImmunoResearch | 112005167 | |
| Hoechst 33342 | Life Technologies | H3570 | |
| Igepal | Sigma Aldrich | I3021-100ML | |
| Insulin | Sigma Aldrich | I6634 | |
| Kanamycin | Sigma-Aldrich | 60615 | |
| Laminin | Life Technologies | 23017015 | |
| LB Medium | See stock solutions | ||
| LB-Agar | See stock solutions | ||
| L-Cysteine | Sigma-Aldrich | C-7477 | |
| Leibovitz's L-15 Medium | Life Technologies | 11415064 | |
| L-Glutamate | Sigma-Aldrich | G1626 | |
| L-Glutamine- 200 mM (100x) liquid | Life Technologies | 25030081 | |
| LR Clonase II Plus enzyme | Life Technologies | 12538-120 | |
| MEM, NEAA, no Glutamine | Life Technologies | 10370088 | |
| Mouse α βIII Tubulin | Promega | G7121 | |
| Mouse αMBP (monoclonal) | Millipore | MAB381 | |
| Na pyruvate | Life Technologies | 11360-070 | |
| NAC | Sigma Aldrich | A8199 | |
| NcoI-HF | NEB | R3193S | |
| NEBuffer 4 | NEB | B7004S | |
| Neurobasal medium | Life Technologies | 21103049 | |
| NGF (mouse) | Alomone Labs | N-100 | |
| NT-3 | Peprotech | 450-03 | |
| O1 antibody - Mouse anti-O1 | Millipore | MAB344 | Alternative if O1 hybridoma cells are unavailable |
| O1 hybridoma cells | Conditioned medium containing anti-O1 antibody to be used for immunopanning | ||
| O4 antibody - Mouse anti-O4 | Millipore | MAB345 | Alternative if O4 hybridoma cells are unavailable |
| O4 hybridoma cells | Conditioned medium containing anti-O4 antibody to be used for immunopanning | ||
| Competent cells | Life Technologies | A10460 | |
| One Shot Stbl competent cells | Life Technologies | C7373-03 | |
| Papain Suspension | Worthington/Cooper | LS003126 | |
| pBR8.91 | Kind gift from Dr Denham10 | ||
| PDGF-AA (Human) | Peprotech | PT10013A500 | |
| Penicillin-streptomycin 100x solution | Life Technologies | 15140122 | |
| pENTRY4IRES2GFP | Invitrogen | 11818-010 | |
| pMD2.G | Addgene | 12259 | |
| Poly-D-lysine | Sigma | P6407-5MG | |
| Polyethylenimine (PEI) | Sigma-Aldrich | 408727-100ML | |
| Poly-L-ornithine | Sigma Aldrich | P3655 | |
| Progesterone | Sigma Aldrich | P8783 | |
| Protease inhibitor tablet (Complete mini) | Roche | 11836153001 | |
| Proteinase K | Supplied with Clonase enzyme | ||
| Putrescine | Sigma Aldrich | P-5780 | |
| Rabbit α neurofilament | Millipore | AB1987 | |
| Rabbit αMBP (polyclonal) | Millipore | AB980 | |
| Ran2 hybridoma cells | ATCC | TIB-119 | Conditioned medium containing anti-Ran2 antibody to be used for immunopanning |
| Rat anti CD140A/PDGFRa antibody | BD Pharmingen | 558774 | |
| SacII | NEB | R0157 | |
| SOC medium | Supplied with competent bacteria | ||
| Sodium selenite | Sigma Aldrich | S5261 | |
| Spe I | NEB | R0133S | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Buffer | NEB | B0202S | |
| TE buffer pH8 | See stock solutions | ||
| TNE lysis buffer | |||
| Trace Elements B | Cellgro | 99-175-CI | |
| Transferrin (apo-Transferrin human) | Sigma-Aldrich | T1147 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Trypsin | Sigma-Aldrich | T9201-1G | |
| Trypsin Inhibitor From Chicken Egg White | Roche | 10109878001 | |
| Trypsin-EDTA (1x), phenol red (0.05%) | Life Technologies | 25300-054 | |
| Unconjugated Griffonia Simplicifolia Lectin BSL-1 | Vector laboratories | L-1100 | |
| Uridine | Sigma-Aldrich | U3003-5G |
Riferimenti
- Baumann, N., Pham-Dinh, D. Biology of oligodendrocyte and myelin in the mammalian central nervous system. Physiol Rev. 81 (2), 871-927 (2001).
- Watkins, T. A., Emery, B., Mulinyawe, S., Barres, B. A. Distinct stages of myelination regulated by gamma-secretase and astrocytes in a rapidly myelinating CNS coculture system. Neuron. 60 (4), 555-569 (2008).
- Lee, K., et al. MDGAs interact selectively with neuroligin-2 but not other neuroligins to regulate inhibitory synapse development. Proc Natl Acad Sci U S A. 110 (1), 336-341 (2013).
- Xiao, J., et al. BDNF exerts contrasting effects on peripheral myelination of NGF-dependent and BDNF-dependent DRG neurons. J Neurosci. 29 (13), 4016-4022 (2009).
- Chan, J. R., et al. NGF controls axonal receptivity to myelination by Schwann cells or oligodendrocytes. Neuron. 43 (2), 183-191 (2004).
- Xiao, J., et al. Brain-Derived Neurotrophic Factor Promotes Central Nervous System Myelination via a Direct Effect upon Oligodendrocytes. Neurosignals. 18 (3), 186-202 (2010).
- Lundgaard, I., et al. Neuregulin and BDNF induce a switch to NMDA receptor-dependent myelination by oligodendrocytes. PLoS Biology. 11 (12), e1001743 (2013).
- Kleitman, N., W, P. M., Bunge, R. P. . Tissue culture methodes for the study of myelination. , (1991).
- Xiao, J., et al. Extracellular signal-regulated kinase 1/2 signaling promotes oligodendrocyte myelination in vitro. J Neurochem. 122 (6), 1167-1180 (2012).
- Wong, A. W., Xiao, J., Kemper, D., Kilpatrick, T. J., Murray, S. S. Oligodendroglial expression of TrkB independently regulates myelination and progenitor cell proliferation. The Journal of Neuroscience. 33 (11), 4947-4957 (2013).
- Li, Z., et al. Molecular cloning, Characterization and Expression of miR-15a-3p and miR-15b-3p in Dairy Cattle. Molecular and Cellular Probes. , (2014).
- Emery, B., et al. Myelin gene regulatory factor is a critical transcriptional regulator required for CNS myelination. Cell. 138 (1), 172-185 (2009).
- Murai, K., et al. Nuclear receptor TLX stimulates hippocampal neurogenesis and enhances learning and memory in a transgenic mouse model. Proc Natl Acad Sci U S A. 111 (25), 9115-9120 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon