Method Article
Identificación de Kinesin-1 Cargos El uso de microscopía de fluorescencia
En este artículo
Resumen
A continuación, se presenta un protocolo para identificar Kinesin-1 cargamentos. Un mutante sin motor de la Kinesin-1 de cadena pesada (KIF5B) agregados en el citoplasma e induce la agregación de sus cargas. Ambos agregados se detectan bajo microscopía de fluorescencia. Una estrategia similar se puede emplear para identificar cargamentos de otras proteínas de motor.
Resumen
Fluorescence microscopy is employed to identify Kinesin-1 cargos. Recently, the heavy chain of Kinesin-1 (KIF5B) was shown to transport the nuclear transcription factor c-MYC for proteosomal degradation in the cytoplasm. The method described here involves the study of a motorless KIF5B mutant for fluorescence microscopy. The wild-type and motorless KIF5B proteins are tagged with the fluorescent protein tdTomato. The wild-type tdTomato-KIF5B appears homogenously in the cytoplasm, while the motorless tdTomato-KIF5B mutant forms aggregates in the cytoplasm. Aggregation of the motorless KIF5B mutant induces aggregation of its cargo c-MYC in the cytoplasm. Hence, this method provides a visual means to identify the cargos of Kinesin-1. A similar strategy can be utilized to identify cargos of other motor proteins.
Introducción
Kinesin-1 es una proteína de motor que media el transporte anterógrado de sus cargos 1,2. Es un heterotetrámero de las dos subunidades de la cadena Kinesin Light 1 (KLC1) y dos subunidades de Cadenas Kinesin pesados (KHCs). KIF5B 1, un KHC, contiene un dominio de motor en su extremo N-terminal, que hidroliza ATP y convierte la energía química en energía mecánica para el movimiento a lo largo de los microtúbulos. Su región C-terminal contiene el dominio de dimerización que interactúa con KLC1, que se asocia con cargas. Kinesin-1 transporta cargas tales como vesículas, orgánulos y mRNAs 3,4. Recientemente, se demostró KIF5B para transportar el factor de transcripción nuclear c-MYC de la degradación en el citoplasma proteosomal 5. Tres metodologías (inhibidor químico, siRNA / shRNA y mutante dominante negativo) fueron utilizados para inhibir la función Kinesin-1. Todos ellos inducen la agregación de c-MYC en el citoplasma. Para la última metodología, c-MYC solamente se vio afectada por la negat dominanteive mutante de KIF5B, pero no por la de otra proteína motor KIF5A relacionada, lo que sugiere que el mutante no ejerce efectos generales sobre los componentes intracelulares (como interrupción de microtúbulos) o en la agregación de proteínas. El mutante dominante negativo de KIF5B también no afectó a otro factor de transcripción, lo que sugiere que no ejerce efectos generales sobre los factores de transcripción. Más bien, se sugiere que el mutante negativo dominante ejerce efectos específicos sobre sus cargas.
El uso de mutantes dominantes negativos es común en el campo de las proteínas de motor. mutantes desprovistos de motor similares de Kinesins y myosins se utilizaron con anterioridad. Se utilizan principalmente para demostrar los efectos de los mutantes en las localizaciones subcelulares de sus cargas o en funciones celulares 6-12. Menos énfasis se puso en la relación espacial entre los mutantes y los cargos afectados por ellos. Sin embargo, en algunas incidencias, se observaron los mutantes para co-localizar con su cargos 6,10.
La interacción entre KIF5B y sus proteínas asociadas se confirmó previamente por la levadura in vivo ensayo de dos híbridos y pull-down ensayos bioquímicos tales como co-inmunoprecipitación y en pull-down ensayos in vitro 13-16. En este artículo, se describe un método mediante microscopía de fluorescencia visual adicional para identificar las proteínas de carga KIF5B. El método hace uso de un mutante KIF5B sin motor que actúa como un mutante negativo dominante. Se agrega en el citoplasma e induce la agregación de sus cargas.
El etiquetado de los de tipo salvaje y mutante KIF5B sin motor con la proteína fluorescente tdTomato 17 permite su visualización por microscopía de fluorescencia. Las proteínas etiquetadas KIF5B pueden co-expresan con una proteína candidata fusionada a una proteína fluorescente diferente con propiedades espectrales adecuadamente separadas de la etiqueta KIF5B. Las proteínas marcadas se observan directamente en células vivasbajo microscopía de fluorescencia. La inducción de la agregación de la proteína candidato por el mutante KIF5B motorless confirmará que la proteína candidata es una carga in vivo de KIF5B. Por otra parte, las proteínas KIF5B tdTomato-etiquetados se pueden expresar por sí solo en las células para estudiar sus efectos sobre las proteínas endógenas de carga. Más tarde, microscopía de inmunofluorescencia se lleva a cabo en el que se fijaron y se tiñeron con un anticuerpo específico contra la proteína candidato endógena las células transfectadas, seguido por un anticuerpo secundario apropiado conjugado con un colorante fluorescente. En este caso, se estudió la proteína endógena candidato en su nivel fisiológico. mutantes desprovistos de motor similares de otras proteínas motoras se pueden preparar para identificar sus cargas.
Protocolo
1. Clonación de la de tipo salvaje tdTomato-etiquetados y sin motor KIF5B proteínas
- Amplificar el ADNc para la de tipo salvaje humana y proteínas KIF5B sin motor utilizando los cebadores en la Tabla 1, la ADN polimerasa Taq (5 unidades de 100 l), mezcla de dNTP (2 mM de cada desoxinucleótido) y su tampón 10X para 30 ciclos. Cada ciclo consta de una etapa de desnaturalización (95 ° C durante 30 seg), una etapa de recocido (45 ° C durante 30 seg) y un paso de extensión (72 ° C durante 3 min).
- Se extrae el producto de ADN amplificado con un volumen igual de fenol / cloroformo (1: 1).
Nota: El fenol es combustible y puede causar quemaduras. El cloroformo es peligroso. Evitar el contacto directo con ellos y utilizarlos en una campana de humos química. Alternativamente, los productos de PCR se pueden purificar por varios kits.- Girar a 18.000 xg en una microcentrífuga a temperatura ambiente durante 1 min. Transferir la solución acuosa a un nuevo tubo y se extrae con un volumen igual de cloroformo.
- Girara 18.000 xg en una microcentrífuga a temperatura ambiente durante 0,5 min. Transferir la solución acuosa a un nuevo tubo.
- Mezclar la solución acuosa con un décimo de volumen de 3 M de acetato sódico y dos volúmenes de etanol absoluto.
- Girar a 18.000 xg en una microcentrífuga a temperatura ambiente durante 5 min. Desechar la solución acuosa.
- Lavar el sedimento de ADN con dos volúmenes de etanol al 75%. Descartar la solución acuosa y el aire seco el sedimento de ADN a temperatura ambiente durante 5 min. Resuspender el sedimento de ADN en 34 l de agua.
- Digerir los productos amplificados en un volumen final de 40 l con las enzimas de restricción SalI (10 unidades) y BamHI (10 unidades) y 4 l de su tampón 10X a 37 ° C durante dos horas. Resolver los productos digeridos en un gel de agarosa (1,0% a 100 V) que contienen bromuro de etidio (0,2 mg / ml).
Nota: El bromuro de etidio es un potente mutágeno y puede ser absorbido por la piel. Por lo tanto, es importante para evitar el contacto directo o indirecto with de bromuro de etidio. - Recortar las bandas correctas para la purificación por columnas. Pesar el gel de agarosa que contiene el fragmento de ADN. A continuación, disolver en el tampón de solubilización (300 l para 0,1 g) a 37 ° C durante aproximadamente 20 min.
- Añadir la solución resultante a una columna y centrifugado en una microcentrífuga a temperatura ambiente durante 5 seg. Desechar el flujo continuo.
- Lavar la columna con tampón de solubilización 0,5 ml mediante la repetición del paso 1.5. Lavar la columna de nuevo con 0,75 ml de tampón de lavado repitiendo el paso 1.5. Girar la columna durante 2 minutos para deshacerse del tampón de lavado restante.
- Eluir el fragmento de ADN con 50 l de agua repitiendo el paso 1.5. Estimar la concentración del fragmento de ADN mediante la ejecución de una parte alícuota de la misma contra una escala de tamaño de ADN en un gel de agarosa (1,0% a 100 V) que contiene bromuro de etidio (0,2 mg / ml).
- Ligar los productos purificados (alrededor de 100 ng) con el vector ptdTomato-C1 17 (alrededor de 100 ng) previamente digerido con THe mismo enzimas de restricción en 10 l utilizando ADN ligasa de T4 (2.000 unidades) y 1 l de tampón de ligación 10 veces a temperatura ambiente durante 16 hr.
Nota: El tipo salvaje y mutante sin motor contiene los aminoácidos de 2 a 963 y 584 a 963, respectivamente. Anteriormente, se utilizó un mutante KIF5B sin motor más grande para identificar su función 9. Un mutante KIF5B sin motor más pequeño se utilizó para los estudios actuales para aumentar sus niveles de expresión.
2. Inmunofluorescencia Microscopía
- Para vivo de células o de imágenes de fluorescencia indirecta con un aumento de 40X en o por debajo, semilla de 0,2-0,3 millones de células HeLa en 1 ml de medio completo [de Eagle modificado por Dulbecco (DMEM) con suero bovino fetal al 10% (FBS)] en cada pocillo de una placa de seis pocillos. Crecer a 37 ° C con 5% de CO2.
- Para los estudios de inmunofluorescencia indirecta con aumentos por encima de 40X, lava cubiertas de vidrio (18 mm x 18 mm; 1,5 de espesor) en etanol absoluto brevemente y aire secarlaslos pocillos de una placa de seis pocillos, dentro de una cabina de bioseguridad para evitar la contaminación.
- Seed 0,2-0,3 millones de células en 1 ml de medio completo DMEM en cada pocillo de una placa de seis pocillos.
- Preparación de complejo de transfección
- El día siguiente, dentro de una cabina de bioseguridad, diluir 0,6 g (total) del plásmido de expresión de tipo salvaje marcada con tdTomato o KIF5B motorless en presencia o ausencia de un plásmido de expresión para una proteína candidata de carga (pTagCFP-c-MYC 5 ) con 0,1 ml de medio de transfección en un tubo de 1,6 ml de microcentrífuga.
Nota: El vector pTagCFP-c-myc expresa el etiquetado TagCFP-c-myc. - Añadir 1,8 l de reactivo de transfección de 0,1 ml de medio de transfección en otro tubo de 1,6 ml de microcentrífuga. Por lo general, preparar una mezcla maestra de reactivo de transfección se diluye suficiente para 12 muestras.
- Incubar durante 5 min, a temperatura ambiente.
- Añadir 0,1 ml del reactivo de transfección se diluyeron a los 0,1 ml diluidos ADN. Mix el contenido invirtiendo los tubos. Girar los tubos durante 5 segundos en una microcentrífuga.
- Incubar durante 45 min, a temperatura ambiente.
- El día siguiente, dentro de una cabina de bioseguridad, diluir 0,6 g (total) del plásmido de expresión de tipo salvaje marcada con tdTomato o KIF5B motorless en presencia o ausencia de un plásmido de expresión para una proteína candidata de carga (pTagCFP-c-MYC 5 ) con 0,1 ml de medio de transfección en un tubo de 1,6 ml de microcentrífuga.
- Lavado de Células
- Se lavan las células sembradas tres veces con solución salina tamponada con fosfato (PBS) durante el período de incubación para la formación del complejo de la transfección.
- Vuelva a colocar el medio de las células sembradas en cada pocillo con 0,8 ml de medio de transfección pre-calentado. Devolver las placas a la incubadora a 37 ° C.
- Transfección de células
- Después de la incubación de 45 min, añadir 0,2 ml del complejo de reactivo de ADN / transfección (preparado en la etapa 2.3) gota a gota a cada pocillo de la placa.
- Rock de la placa de 6 pocillos suavemente durante 5 segundos, antes de que se devuelven a la incubadora a 37 ° C.
- Después de 6-8 horas, sustituir el medio con el medio DMEM completo.
- For Imaging fluorescencia de células en vivo 18
- Después de un 16 horas de incubación adicional de unat 37 ° C, añadir la mancha de intercalación de ADN Hoechst 33342 para el medio a una concentración final de 1 mM.
Nota: Hoechst 33342 es un colorante intercalante de ADN permeable a células que tiñe los núcleos celulares. - Se incuban las células transfectadas durante 10 min en la incubadora a 37 ° C antes de examinar ellos para la expresión de proteínas fluorescentes por microscopía de fluorescencia utilizando un objetivo 40X. Los conjuntos de filtros para DAPI, PPC, FITC y Cy3 son (Ex350 nm / Em460 nm), (Ex436 nm / Em480 nm), (EX470 nm / Em525 nm) y (Ex545 nm / Em605 nm), respectivamente.
Nota: Si el fondo es alto debido a la auto-fluorescencia del medio completo DMEM, se utiliza medio sin rojo fenol.
- Después de un 16 horas de incubación adicional de unat 37 ° C, añadir la mancha de intercalación de ADN Hoechst 33342 para el medio a una concentración final de 1 mM.
3. Para los estudios de inmunofluorescencia indirecta
- Preparación de solución de paraformaldehído
- Pesar polvo de paraformaldehído (4 g por 100 ml de PBS) y añadirlo a PBS.
Nota: El paraformaldehido es un sólido inflamable y un obstáculo de potencial de cáncerre. Se irrita los ojos, las vías respiratorias y la piel. Por lo tanto, es importante para evitar el contacto con o la inhalación de paraformaldehído. - Añadir NaOH a la solución (150 l 10 N NaOH / 100 ml).
- Mantener la solución a 37-42 ° C durante 2-3 horas con agitación ocasional.
- Ajustar el pH a 7,0 mediante la adición de ácido acético glacial a la solución (alrededor de 75 l / 100 ml) después de que el polvo de paraformaldehído se disuelve.
- Pesar polvo de paraformaldehído (4 g por 100 ml de PBS) y añadirlo a PBS.
- Las proteínas diana de tinción
- Después de una incubación adicional durante 16 horas a 37 ° C, se lavan las células transfectadas a temperatura ambiente una vez con PBS haciendo girar la placa de seis pocillos durante 5 segundos.
- Fijar las células con 1 ml de solución recién preparada de paraformaldehído, 4% por pocillo. Incubar a temperatura ambiente durante 30 min.
- Se lavan las células con PBS una vez por agitación durante 5 segundos la placa de seis pocillos. Desechar la solución.
- Se incuban las células con 1 ml de 0,1% Triton X-100 (en PBS) a temperatura ambiente durante 30 min. la detergent Triton-X 100 se permeabilizar la membrana celular para permitir el acceso de los anticuerpos a sus dianas intracelulares.
- Se lavan las células con PBS cuatro veces por agitación de la placa de seis pocillos durante 5 segundos cada vez. Desechar la solución después de cada lavado.
- Se incuban las células con 1 ml de anticuerpos primarios (anticuerpo de conejo c-MYC o anticuerpo p53 de ratón; 0,1 mg / ml) en un FBS 10% en solución de PBS a temperatura ambiente por agitación durante 4 hr.
- Lavar con PBS cuatro veces por agitación de la placa de seis pocillos durante 5 segundos cada vez. Desechar la solución después de cada lavado.
- Incubar con 1 ml de anticuerpos secundarios de tinte conjugado fluorescente (Alexa Fluor 488-conjugado anti-conejo o anti-ratón de anticuerpos IgG; 0,5 mg / ml) en 10% de FBS en PBS a temperatura ambiente en la oscuridad por balanceo durante 2 hr.
- Lavar con PBS cuatro veces por agitación de la placa de seis pocillos durante 5 segundos cada vez. Desechar la solución después de cada lavado.
- La tinción nuclear y de montaje
- incubar las células con el 1 ml de DNA intercalante tinte 4 ', 6-diamidino-2-fenilindol (DAPI; 0,5 mg / ml) en PBS a temperatura ambiente, en oscuridad durante 10 min. Continúe con el paso 3.4 cuando se utilizan no cubiertas de vidrio.
Nota: DAPI es un colorante de intercalación de ADN que tiñe los núcleos celulares. - Aplique 10 l de solución de montaje en cada portaobjetos de microscopio. Montar cada cubierta de vidrio sobre la solución de montaje en un portaobjetos de microscopio.
- Se incuba a temperatura ambiente en la oscuridad durante la noche.
- Sellar los bordes de las cubiertas de vidrio con esmalte de uñas. Coloque en la oscuridad dentro de una campana de humos durante la noche para retirar los gases de esmalte de uñas.
- incubar las células con el 1 ml de DNA intercalante tinte 4 ', 6-diamidino-2-fenilindol (DAPI; 0,5 mg / ml) en PBS a temperatura ambiente, en oscuridad durante 10 min. Continúe con el paso 3.4 cuando se utilizan no cubiertas de vidrio.
- Microscopía de fluorescencia 19
- Al día siguiente, examinar las células por microscopía de fluorescencia utilizando un objetivo 40X. Los conjuntos de filtros para DAPI, PPC, FITC y Cy3 son (Ex350 nm / Em460 nm), (Ex436 nm / Em480 nm), (EX470 nm / Em525 nm) y (Ex545 nm / Em605 nm), respectivamente.
Resultados
Exógena expresada de tipo salvaje KIF5B apareció homogénea en el citoplasma, mientras que c-MYC apareció principalmente en el núcleo (Figura 1A). Sin embargo, el mutante KIF5B motorless formó agregados en el citoplasma (Figura 1A). La agregación de la KIF5B sin motor induce la agregación de c-myc. El porcentaje de células que expresan agregados de etiquetado TagCFP c-myc en las células que expresan de tipo salvaje KIF5B fue baja. Sin embargo, fue significativamente mayor cuando el KIF5B sin motor fue co-expresó (Figura 1A). La co-localización observada de KIF5B mutante con c-MYC (Figura 1) sugiere que KIF5B regula la localización subcelular de c-myc y c-MYC es un cargamento de Kinesin-1. Los controles negativos en los que se expresan las construcciones solo se incluyen en el panel inferior de la Figura 1A. Los resultados muestran que no hubo significativo derrame de EMISIO fluorescencianorte. El KIF5B motorless también indujo la agregación de la endógena c-MYC (Figura 1B) y el factor de transcripción p53 (Figura 2), lo que indica que c-MYC y p53 son a la vez los cargamentos de Kinesin-1 y KIF5B regula la localización subcelular de ambas proteínas endógenas. Junto con los resultados que el inhibidor de Kinesin-1 rose bengal lactona (RBL) 20 induce la formación de especies de alto peso molecular tanto para c-myc y p53 5, p53 es probable una carga de Kinesin-1. Es interesante observar que la p53 nuclear factor de transcripción, como c-MYC, también se exporta desde el núcleo al citoplasma de la degradación proteasomal 21,22. El movimiento de factores de transcripción por KIF5B parece ser específica ya que la expresión del mutante KIF5B motorless no afectó a la localización subcelular c-Fos o hacer que agregados (Figura 3). Los datos anteriores demuestran que el method empleadas en esta publicación permite la identificación de alta especificidad de Kinesin-1 cargamentos. Una estrategia similar se puede aplicar a otras proteínas de motor también.
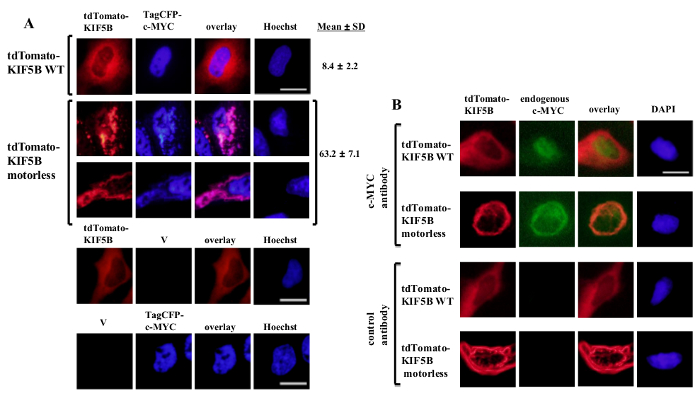
Figura 1: Expresión de la mutante KIF5B motorless induce la agregación c-MYC en el citoplasma de células (A) HeLa fueron transfectadas con etiquetado-tdTomato de tipo salvaje (WT) KIF5B (rojo) y de etiquetado TagCFP c-MYC (azul).. -etiquetados tdTomato WT KIF5B apareció principalmente en el citoplasma, mientras que TagCFP etiquetada-c-myc apareció principalmente en el núcleo. Sin embargo, el mutante KIF5B motorless-etiquetados tdTomato (rojo) formado agregados filamentosos o puntiformes en el citoplasma. Expresión de la KIF5B motorless indujo la agregación de c-MYC. El KIF5B mutante y c-MYC co-localizados juntos (rosa). se muestran los porcentajes de células (%) que indican agregados de CFP-c-MYC (%). Los resultados se muestran comomedia ± SD (n = 3). Los controles negativos con la expresión de las proteínas marcadas con KIF5B tdTomato o etiquetado TagCFP-c-myc, junto con vectores vacíos (V) se muestran en el panel inferior. (B) Resultados similares se obtuvieron con el endógena c-myc (verde) cuando se expresaron etiquetado tdTomato-WT o proteínas KIF5B sin motor (rojo). El mutante KIF5B motorless-etiquetados tdTomato formó agregados en el citoplasma y se indujo la agregación de la endógena c-MYC. Ambos tipos de agregados co-localizados juntos en el citoplasma (naranja). Los núcleos se tiñeron con el colorante Hoechst 33342 o DAPI. La barra de escala; 20 micras. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Expresión de la mutante KIF5B motorless induce la agregación p53 endógeno en tél citoplasma. En las células HeLa, tdTomato-etiquetado de tipo salvaje (WT) KIF5B exógeno (rojo) apareció principalmente en el citoplasma, mientras que el p53 endógeno (verde) apareció principalmente en el núcleo. En contraste, el mutante KIF5B motorless-etiquetados tdTomato (rojo) agregados en el citoplasma formado. Expresión de la KIF5B motorless indujo la agregación de p53, dando como resultado la co-localización de KIF5B y p53 en el citoplasma (amarillo). El experimento se realizó tres veces. Los núcleos se tiñeron con el colorante vital DAPI. La barra de escala; 20 micras. Haga clic aquí para ver una versión más grande de esta figura.
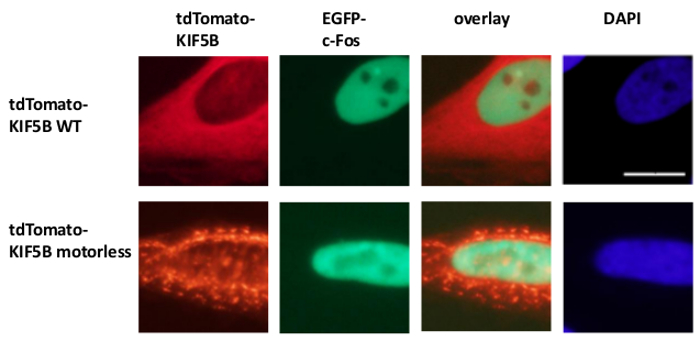
Figura 3:. La expresión de la mutante KIF5B motorless no induce agregación de c-Fos en el citoplasma tdTomato-etiquetado de tipo salvaje (WT) KIF5B (rojo) aparecióprincipalmente en el citoplasma de las células HeLa, mientras EGFP-c-Fos (verde) apareció principalmente en el núcleo. Por el contrario, el mutante KIF5B motorless-etiquetados tdTomato (rojo) agregados en el citoplasma formado. Expresión de la KIF5B motorless no indujo la agregación de c-Fos. El experimento se realizó en cuatro ocasiones. Los núcleos de las células se tiñeron con el tinte DAPI. La barra de escala; 20 micras. Haga clic aquí para ver una versión más grande de esta figura.
| cebador inverso común | 5'-AGAGGATCCTTACACTTGTTTGCCTCCTC-3 ' |
| De tipo salvaje KIF5B cebador directo | 5'-3 AGAGTCGACGCGGACCTGGCCGAGTGCAACATCAAAGT ' |
| cebador directo KIF5B motorless | 5'-3 AGAGTCGACGATGAAGAGTTCACTGTTGC ' |
Tabla 1: Las secuencias de cebador para clonar el de tipo salvaje y las proteínas KIF5B sin motor.
Discusión
El método descrito utiliza las propiedades del mutante KIF5B sin motor, que carece de la capacidad de moverse a lo largo de los microtúbulos, pero conserva la capacidad para formar dímeros con la KIF5B de tipo salvaje y, de este modo, permitir que la proteína tetramérica para interactuar con las mismas proteínas de carga como la de tipo salvaje KIF5B. KIF5B sin motor, por lo tanto, actúa como un mutante negativo dominante y formas mislocalized agregados con sus cargas. Este método se ha demostrado para identificar la Kinesin-1 de carga c-MYC (Figura 1) 5. En este artículo, se usó el mismo mutante KIF5B sin motor para identificar p53 como otra carga potencial de Kinesin-1 (Figura 2). Esto demuestra que la metodología es posible identificar otros cargos de Kinesin-1. Además, la especificidad de la mutante es proporcionado por la falta de efecto de los mutantes de la proteína de control negativo c-Fos (Figura 3).
En este protocolo, el tdTomato-etiquetados salvaje-tyPE o proteína KIF5B sin motor se coexpresan con otra proteína candidata de carga de proteínas marcadas con fluorescencia. En este caso, se llevan a cabo en vivo de células microscopía de fluorescencia y formación de imágenes. La formación de los agregados puede ser rastreado por la imagen de lapso de tiempo. Alternativamente, la proteína tdTomato-etiquetados se expresa solo y la proteína de carga candidato en sus niveles fisiológicos se visualiza mediante microscopía de inmunofluorescencia indirecta utilizando anticuerpos específicos. El tdTomato proteína fluorescente es elegido por su brillo y fotoestabilidad 17. Si el fondo es alto debido a la auto-fluorescencia del medio completo DMEM, se utiliza medio sin rojo fenol.
El mutante KIF5B motorless forma dímeros con el KIF5B de tipo salvaje estequiométricamente. Por lo tanto, es crítico para expresar cantidad suficiente de mutante KIF5B sin motor para formar dímeros con la contraparte de tipo salvaje para inhibir su función y para formar agregados. Para abordar esta cuestión, la optimización de exprESIÓN del mutante sin motor es esencial. Esto se logra mediante el uso de una pequeña mutante sin motor que contiene el dominio de dimerización. Además, la optimización de la transfección reactivo apropiado y el protocolo es también necesaria. La duración de la incubación de las células HeLa con el reactivo de ADN / transfección se optimizó en células HeLa para la expresión de proteínas exógenas y la viabilidad celular. La duración de la incubación puede tener que ser optimizada para otras líneas celulares.
Para aumentos entre 4X a 40X, no se requieren cubiertas de vidrio. Las células pueden ser examinados directamente en los pocillos. Por lo tanto, en este caso, el protocolo es de bajo costo y conveniente. Para aumentos por encima de 40X, las células se cultivan en cubreobjetos en los pozos y, después de la tinción, están montados en portaobjetos de microscopio para examinarlas con objetivos de inmersión en aceite.
Observación de los agregados de la proteína KIF5B sin motor está limitado por el tamaño del citoplasma. Es fácil detectar laagregados en muchas células de mamíferos. Sin embargo, es relativamente difícil de observar los agregados en las células neuronales cuando el volumen del citoplasma es pequeño alrededor de los núcleos y en las neuritas.
El protocolo se utiliza para mostrar la asociación entre KIF5B y sus cargas, además de la in vivo levadura de dos híbridos y pull-down ensayos bioquímicos 13-16. Todos estos ensayos determinan la asociación en diferentes condiciones físicas y sus resultados pueden complementarse entre sí. Por otra parte, la ventaja de este ensayo de fluorescencia con respecto a otros ensayos es que puede mostrar la regulación de la localización subcelular de los cargos por KIF5B (Figuras 1 y 2).
El protocolo no se limita a KIF5B y se puede utilizar para identificar cargamentos de otras proteínas de motor, tales como algunos otros kinesin motores 3 y algunos de los motores de miosina 23,24 utilizados en el transporte intracelular de cargas 3. Estas proteínas motoras también contienen dominios de motor para el movimiento a lo largo de los microtúbulos para kinesin motores y microfilamentos para motores de miosina, y segmentos de doble espiral de oligomerización. La mayoría de ellos forman homodímeros 3,23. Por lo tanto, una estrategia similar se puede aplicar a ellos mediante la creación de sus mutantes negativos dominantes sin motor para identificar sus cargas.
Divulgaciones
publicación de acceso libre y la producción de este artículo es pagado por Roche.
Agradecimientos
Roche's Publication Grant covered the Free Access publication and production of this article. The author also thanks E. Premkumar Reddy, Richard V. Mettus, Stephen C. Cosenza, Sau Ying Yip and Sol D. Gloria for their support and critical reading of the manuscript.
Materiales
| Name | Company | Catalog Number | Comments |
| absolute ethanol (200 proof) | Fisher Scientific | BP2818 | |
| DAPI | Sigma-Aldrich | D9542 | |
| Hoechst 33342 | Sigma-Aldrich | B2261 | |
| Opti-MEM-I (transfection medium) | Life Technologies | 51985 | |
| ProLong Diamond Antifade Mountant | Life Technologies | P36961 | |
| formaldehyde, para | Fisher Scientific | O4042-500 | |
| Triton-X100 | Fisher Scientific | BP151-500 | |
| X-tremeGENE 9 DNA Transfection Reagent | Roche | 6365787001 | |
| Taq DNA polymerase | Life Technologies | 10342020 | |
| PCR Grade Nucleotide Mix (dNTP Mix) | Roche | 12111424 | |
| Microscope cover glass | Fisher Scientific | 12-541A | |
| GeneRuler 1 kb Plus DNA ladder | Life Technologies | SM1333 | |
| PureLink Quick Gel Extraction kit | Life Technologies | K210012 | |
| BamHI | New England Biolab | R0136 | |
| SalI | New England Biolab | R0138 | |
| T4 DNA ligase | New England Biolab | M0202T | |
| Ethidium bromide | Thermo Scientific | 17898 | |
| DMEM | Life Technologies | 11995-065 | |
| c-MYC rabbit antibody | Cell Signaling | 5606 | |
| p53 mouse antibody | Santa Cruz | sc-126 | |
| Alexa Fluor 488-conjugated anti-rabbit IgG antibody | Life Technologies | A11008 | |
| Alexa Fluor 488-conjugated anti-mouse IgG antibody | Life Technologies | A11059 | |
| fluorescent microscope | Olympus IX71_Fluoview | ||
| computer software for imaging | cellSens |
Referencias
- Hirokawa, N., Niwa, S., Tanaka, Y. Molecular motors in neurons: transport mechanisms and roles in brain function, development, and disease. Neuron. 68, 610-638 (2010).
- Yu, Y., Feng, Y. M. The role of kinesin family proteins in tumorigenesis and progression: potential biomarkers and molecular targets for cancer therapy. Cancer. 116, 5150-5160 (2010).
- Verhey, K. J., Hammond, J. W. Traffic control: regulation of kinesin motors. Nat Rev Mol Cell Biol. 10, 765-777 (2009).
- Hirokawa, N., Noda, Y., Tanaka, Y., Niwa, S. Kinesin superfamily motor proteins and intracellular transport. Nat Rev Mol Cell Biol. 10, 682-696 (2009).
- Lee, C. M. Transport of c-MYC by Kinesin-1 for proteasomal degradation in the cytoplasm. Biochim Biophys Acta. 1843, 2027-2036 (2014).
- Bittins, C. M., Eichler, T. W., Hammer, J. A., Gerdes, H. H. Dominant-negative myosin Va impairs retrograde but not anterograde axonal transport of large dense core vesicles. Cell Mol Neurobiol. 30, 369-379 (2010).
- Kimura, T., Watanabe, H., Iwamatsu, A., Kaibuchi, K. Tubulin and CRMP-2 complex is transported via Kinesin-1. J Neurochem. 93, 1371-1382 (2005).
- Lindsay, A. J., McCaffrey, M. W. Myosin Va is required for the transport of fragile X mental retardation protein (FMRP) granules. Biol Cell. 106, 57-71 (2014).
- Rivera, J., Chu, P. J., Lewis, T. L., Arnold, D. B. The role of Kif5B in axonal localization of Kv1 K(+) channels. Eur J Neurosci. 25, 136-146 (2007).
- Roland, J. T., Kenworthy, A. K., Peranen, J., Caplan, S., Goldenring, J. R. Myosin Vb interacts with Rab8a on a tubular network containing EHD1 and EHD3. Mol Biol Cell. 18, 2828-2837 (2007).
- Uchida, A., Alami, N. H., Brown, A. Tight functional coupling of kinesin-1A and dynein motors in the bidirectional transport of neurofilaments. Mol Biol Cell. 20, 4997-5006 (2009).
- Zadeh, A. D., et al. Kif5b is an essential forward trafficking motor for the Kv1.5 cardiac potassium channel. J Physiol. 587, 4565-4574 (2009).
- Su, Y. Y., et al. KIF5B promotes the forward transport and axonal function of the voltage-gated sodium channel Nav1.8. J Neurosci. 33, 17884-17896 (2013).
- Lalioti, V. S., Vergarajauregui, S., Tsuchiya, Y., Hernandez-Tiedra, S., Sandoval, I. V. Daxx functions as a scaffold of a protein assembly constituted by GLUT4, JNK1 and KIF5B. . J Cell Physiol. 218, 416-426 (2009).
- Cho, K. I., et al. Association of the kinesin-binding domain of RanBP2 to KIF5B and KIF5C determines mitochondria localization and function. Traffic. 8, 1722-1735 (2007).
- Diefenbach, R. J., Diefenbach, E., Douglas, M. W., Cunningham, A. L. The ribosome receptor, p180, interacts with kinesin heavy chain, KIF5B. Biochem Biophys Res Commun. 319, 987-992 (2004).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22, 1567-1572 (2004).
- Waters, J. C. Live-cell fluorescence imaging. Methods Cell Biol. 114, 125-150 (2013).
- Sanderson, M. J., Smith, I., Parker, I., Bootman, M. D. Fluorescence microscopy. Cold Spring Harb Protoc. 2014, (2014).
- Hopkins, S. C., Vale, R. D., Kuntz, I. D. Inhibitors of kinesin activity from structure-based computer screening. Biochemistry. 39, 2805-2814 (2000).
- Zhang, Y., Xiong, Y. Control of p53 ubiquitination and nuclear export by MDM2 and ARF. Cell Growth Differ. 12, 175-186 (2001).
- Michael, D., Oren, M. The p53-Mdm2 module and the ubiquitin system. Semin Cancer Biol. 13, 49-58 (2003).
- Kneussel, M., Wagner, W. Myosin motors at neuronal synapses: drivers of membrane transport and actin dynamics. Nat Rev Neurosci. 14, 233-247 (2013).
- Maravillas-Montero, J. L., Santos-Argumedo, L. The myosin family: unconventional roles of actin-dependent molecular motors in immune cells. J Leukoc Biol. 91, 35-46 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados