Method Article
Identifikation von Kinesin-1 Cargos Mit Fluoreszenzmikroskopie
In diesem Artikel
Zusammenfassung
Hier wird ein Protokoll vorgestellt Kinesin-1 Ladungen zu identifizieren. Ein motorlose Mutante des Kinesin-1 schweren Kette (KIF5B) Aggregate im Zytoplasma und induziert die Aggregation der Ladungen. Beide Aggregate werden unter Fluoreszenzmikroskopie nachgewiesen. Eine ähnliche Strategie Ladungen anderer Motor-Proteine zu identifizieren, eingesetzt werden.
Zusammenfassung
Fluorescence microscopy is employed to identify Kinesin-1 cargos. Recently, the heavy chain of Kinesin-1 (KIF5B) was shown to transport the nuclear transcription factor c-MYC for proteosomal degradation in the cytoplasm. The method described here involves the study of a motorless KIF5B mutant for fluorescence microscopy. The wild-type and motorless KIF5B proteins are tagged with the fluorescent protein tdTomato. The wild-type tdTomato-KIF5B appears homogenously in the cytoplasm, while the motorless tdTomato-KIF5B mutant forms aggregates in the cytoplasm. Aggregation of the motorless KIF5B mutant induces aggregation of its cargo c-MYC in the cytoplasm. Hence, this method provides a visual means to identify the cargos of Kinesin-1. A similar strategy can be utilized to identify cargos of other motor proteins.
Einleitung
Kinesin-1 ist ein Protein, das Motor anterograden Transport von seiner Ladungen 1,2 vermittelt. Es ist ein Heterotetramer der beiden Untereinheiten von Kinesin Light Chain 1 (KLC1) und zwei Untereinheiten von Kinesin Schwere Ketten (KHCs). KIF5B 1, a KHC enthält einen Motor Domäne an ihrem N-Terminus, die hydrolysiert ATP und wandelt die chemische Energie in mechanische Energie zur Bewegung entlang Mikrotubuli. Seine C-terminale Region enthält die Dimerisierungsdomäne, die mit KLC1, die mit Ladungen assoziiert interagiert. Kinesin-1 transportiert Ladungen wie Vesikel, Organellen und mRNAs 3,4. Kürzlich wurde KIF5B die Kerntranskriptionsfaktor c-MYC für proteosomalen Abbau im Zytoplasma 5 zu transportieren gezeigt. Drei Methoden (chemischer Inhibitor, siRNA / shRNA und dominant negative Mutante) wurden verwendet, Kinesin-1-Funktion zu hemmen. Alle Aggregation von c-MYC in das Zytoplasma induziert. Für die letzte Methode, c-MYC wurde nur von der herrschenden negat betroffenive Mutante von KIF5B, aber nicht von der eines anderen verwandten KIF5A Motor Protein, was darauf hindeutet, dass die Mutante nicht allgemeine Wirkungen auf die intrazellulären Komponenten ausübt (wie Mikrotubuli Störung) oder Proteinaggregation. Die dominant negative Mutante von KIF5B wirkte sich auch keine andere Transkriptionsfaktor, was darauf hindeutet, dass es keine allgemeinen Auswirkungen auf die Transkriptionsfaktoren ausübt. Vielmehr schlägt er vor, dass die dominante negative Mutante spezifische Auswirkungen auf die Ladungen ausübt.
Die Verwendung von dominant negativen Mutanten ist auf dem Gebiet der Motorproteine gemeinsam. Ähnliche motorlosen Mutanten von kinesins und Myosine wurden bisher verwendeten. Sie wurden hauptsächlich verwendet, 6-12 die Wirkung der Mutanten auf die subzelluläre Lokalisation ihrer Ladungen oder auf zelluläre Funktionen zu demonstrieren. Weniger Wert wurde zwischen den Mutanten auf die räumliche Beziehung setzen und die von ihnen betroffen Ladungen. Jedoch in einigen Fälle wurden die Mutanten beobachtet zusammen lokalisieren mit ihren cargos 6,10.
Die Wechselwirkung zwischen KIF5B und die zugehörigen Proteine wurde zuvor von der in vivo Hefe-Zwei-Hybrid-Assay und biochemische Pull-down-Assays, wie Co-Immunpräzipitation und in vitro-Pulldown-Assays 13-16 bestätigt. In diesem Artikel wird eine zusätzliche visuelle Methode unter Verwendung von Fluoreszenzmikroskopie beschrieben KIF5B Frachtproteine zu identifizieren. Das Verfahren verwendet eine motorlose KIF5B Mutante, die als dominant negative Mutante wirkt. Es vereinigt in das Zytoplasma und induziert die Aggregation der Ladungen.
Die Markierung von Wildtyp und motorlose KIF5B Mutante mit dem fluoreszierenden Protein tdTomato 17 ermöglicht, ihre Visualisierung von Fluoreszenzmikroskopie. Die markierten KIF5B Proteine können aus dem KIF5B Tag getrennt mit spektralen Eigenschaften in geeigneter Weise an einem anderen fluoreszierenden Protein fusioniert mit einem Kandidatenprotein coexprimiert werden. Die markierten Proteine werden direkt in lebenden Zellen beobachtetunter Fluoreszenzmikroskopie. Die Induktion der Aggregation des Kandidatenprotein durch die motorlose KIF5B Mutante wird bestätigen, dass die Kandidaten-Protein ist ein In-vivo-Ladung von KIF5B. Darüber hinaus können die tdTomato-markierten Proteine KIF5B allein in den Zellen exprimiert werden, um ihre Auswirkungen auf die endogene Ladung Proteine zu studieren. Später, Immunfluoreszenz-Mikroskopie durchgeführt wird, in dem die transfizierten Zellen fixiert und mit einem spezifischen Antikörper gegen den endogenen Kandidatenprotein, gefolgt von einem geeigneten sekundären Antikörper konjugiert ist mit einem Fluoreszenzfarbstoff angefärbt. In diesem Fall wird die endogene Kandidatenprotein an seinem physiologischen Ebene untersucht. Ähnliche motorlosen Mutanten von anderen Motorproteine hergestellt werden, ihre Ladungen zu identifizieren.
Protokoll
1. Klonierung des tdTomato-markierten Wildtyp und Motorless KIF5B Proteine
- Erhöhen der cDNAs für das menschliche Wildtyp und motorlose KIF5B Proteine unter Verwendung der Primer in Tabelle 1, Taq-DNA-Polymerase (5 Einheiten pro 100 ul), dNTP-Mix (2 mM je Desoxynukleotid) und dessen 10X Puffer für 30 Zyklen. Jeder Zyklus besteht aus einem Denaturierungsschritt (95 ° C für 30 sec), einem Glühschritt (45 ° C für 30 sec) und einem Verlängerungsschritt (72ºC für 3 min).
- Extrahiere die amplifizierte DNA-Produkt mit einem gleichen Volumen Phenol / Chloroform (1: 1).
Hinweis: Phenol ist brennbar und kann zu Verbrennungen führen. Chloroform ist gefährlich. Vermeiden Sie direkten Kontakt mit ihnen und mit ihnen unter einer chemischen Abzugshaube. Alternativ PCR-Produkte können durch verschiedene Kits gereinigt werden.- Schleudern bei 18.000 xg in einer Mikrozentrifuge bei Raumtemperatur für 1 min. Die wäßrige Lösung in ein neues Röhrchen und Extrahieren mit einem gleichen Volumen an Chloroform.
- Drehenbei 18.000 xg in einer Mikrozentrifuge bei Raumtemperatur für 0,5 min. Die wäßrige Lösung in ein neues Röhrchen.
- Mischen der wässrigen Lösung mit einem Zehntel Volumen 3 M Na-Acetat und zwei Volumina absolutem Ethanol.
- Schleudern bei 18.000 xg in einer Mikrozentrifuge bei Raumtemperatur für 5 min. Verwerfen der wässrigen Lösung.
- Waschen Sie die DNA-Pellet mit zwei Volumen von 75% Ethanol. Verwerfen der wässrigen Lösung und Luft in das DNA-Pellet bei Raumtemperatur für 5 min trocknen. Resuspendieren DNA-Pellet in 34 ul Wasser.
- Verdauen die Amplifikate in einem Endvolumen von 40 ul mit den Restriktionsenzymen SalI (10 Einheiten) und BamHI (10 Einheiten) und 4 & mgr; l der 10X-Puffer bei 37 ° C für zwei Stunden. Die verdauten Produkte auf einem Agarosegel (1,0% bei 100 V), die Ethidiumbromid (0,2 ug / ml).
Hinweis: Ethidiumbromid ein potenter mutagen und kann durch die Haut aufgenommen werden. Daher ist es wichtig, direkten oder indirekten Kontakt zu vermeiden, with Ethidiumbromid. - Schneiden Sie die richtigen Bands zur Reinigung aus durch Spalten. Man wiegt das Agarosegel das DNA-Fragment enthält. Dann löst ihn in der Solubilisierungspuffer (300 & mgr; l für 0,1 g) bei 37 ° C für ca. 20 min.
- Fügen Sie die erhaltene Lösung auf eine Säule und Schleudern in einer Mikrozentrifuge bei Raumtemperatur für 5 sec. Verwerfen Sie den Durchfluss durch.
- Die Säule wird mit 0,5 ml Solubilisierungspuffer durch den Schritt 1.5 wiederholen. Die Säule wird erneut mit 0,75 ml Waschpuffer durch den Schritt 1.5 wiederholen. Drehen Sie die Spalte für 2 Minuten, um loszuwerden, die verbleibende Waschpuffer.
- Man eluiert das DNA-Fragment mit 50 ul Wasser um 1,5 Schritt wiederholen. Schätzung der Konzentration des DNA-Fragments, indem ein Aliquot davon gegen einen DNA-Größenleiter in einem Agarosegel (1,0% bei 100 V) ausgeführt enthalten Ethidiumbromid (0,2 ug / ml).
- Ligation der gereinigten Produkte (etwa 100 ng) mit dem ptdTomato-C1-Vektor 17 (etwa 100 ng), die zuvor mit th verdaute gleichen Restriktionsenzymen in 10 ul T4 DNA-Ligase für 16 Stunden unter Verwendung von (2.000 Einheiten) und 1 ul 10x Ligationspuffer bei Raumtemperatur.
Hinweis: Die Wildtyp und Mutante motorlosen enthält Aminosäuren 2-963 und 584-963, respectively. Zuvor wurde eine größere motorlose KIF5B Mutante verwendet, um seine Funktion 9 zu identifizieren. Eine kleinere motorlose KIF5B Mutante wurde für den vorliegenden Untersuchungen verwendet, um seine Expressionsniveaus zu erhöhen.
2. Immunfluoreszenz-Mikroskopie
- Für Live-Zelle oder indirekten Fluoreszenzbildgebung mit Vergrößerung bei oder unter 40X, Saatgut 0,2-0.300.000 HeLa-Zellen in 1 ml Komplettmedium [Dulbeccos modifiziertes Eagle-Medium (DMEM) mit 10% fötalem Rinderserum (FBS)] in jede Vertiefung einer sechs-Well-Platte. Wachsen bei 37 ° C mit 5% CO 2.
- Für die indirekte Immunfluoreszenz-Studien mit Vergrößerungen über 40x, waschen Deckgläser (18 mm x 18 mm, 1,5 Dicke) in absolutem Ethanol kurz und Luft trocknen sie indie Vertiefungen einer Sechs-Well-Platte in einem biologischen Sicherheitsschrank, Kontamination zu vermeiden.
- Seed ,2-0.300.000 Zellen in 1 ml komplettem DMEM-Medium in jeder Vertiefung einer Sechs-Well-Platte.
- Vorbereitung der Transfektion Complex
- Am nächsten Tag in einem biologischen Sicherheitsschrank, verdünnte 0,6 & mgr; g (gesamt) des Expressionsplasmids für tdTomato-markierten Wildtyp oder motorlose KIF5B in Gegenwart oder Abwesenheit eines Expressionsplasmids für einen Frachtkandidatenprotein (pTagCFP-c-MYC 5 mit 0,1) ml Transfektion Medium in einem 1,6 ml Mikrozentrifugenröhrchen.
Anmerkung: Der Vektor pTagCFP-c-MYC drückt die TagCFP-markierten c-MYC. - In 1,8 ul des Transfektionsreagenz bis 0,1 ml Transfektionsmediums in einem anderen 1,6 ml Mikrozentrifugenröhrchen. Typischerweise einen Master-Mix von verdünnten Transfektionsreagenz genug für 12 Proben vorzubereiten.
- Inkubieren Sie für 5 min bei Raumtemperatur.
- In der verdünnten 0,1 ml Transfektionsreagenz dem verwässerten 0,1 ml DNAs. miden Inhalt X durch die Rohre umgekehrt wird. Drehen Sie die Rohre für 5 Sekunden in einer Mikro.
- Inkubieren für 45 min bei Raumtemperatur.
- Am nächsten Tag in einem biologischen Sicherheitsschrank, verdünnte 0,6 & mgr; g (gesamt) des Expressionsplasmids für tdTomato-markierten Wildtyp oder motorlose KIF5B in Gegenwart oder Abwesenheit eines Expressionsplasmids für einen Frachtkandidatenprotein (pTagCFP-c-MYC 5 mit 0,1) ml Transfektion Medium in einem 1,6 ml Mikrozentrifugenröhrchen.
- Das Waschen der Zellen
- Waschen Sie die beimpften Zellen dreimal mit phosphatgepufferter Salzlösung (PBS) während der Inkubationszeit für die Bildung des Transfektionskomplexes.
- Ersetzen Sie das Medium der ausgesäten Zellen in jeder Vertiefung mit 0,8 ml vorgewärmten Transfektionsmediums. Bringen Sie die Platten in den Inkubator bei 37 ° C.
- Transfektion von Zellen
- Nach einer Inkubation von 45 min tropfenweise zu jeder Vertiefung der Platte 0,2 ml des DNA / Transfektionsreagenz Komplex (in Schritt 2.3 hergestellten) hinzuzufügen.
- Schaukeln der Platte mit 6 Vertiefungen sanft für 5 Sekunden, bevor sie in den Inkubator bei 37 ° C zurückgeführt werden.
- Nach 6-8 Stunden, ersetzen Sie das Medium mit der kompletten DMEM-Medium.
- Lebender Zellen Fluoreszenz-Imaging 18
- Nach weiteren 16 h Inkubation einet 37 ° C, fügen die DNA interkalierende Fleck Hoechst 33342 zu dem Medium bis zu einer Endkonzentration von 1 uM.
Anmerkung: Hoechst 33342 ist ein zelldurchlässig, DNA interkalierenden Farbstoff, der die Zellkerne färbt. - Inkubieren der transfizierten Zellen für 10 min im Brutschrank bei 37 ° C, bevor sie für die Expression von fluoreszierenden Proteinen durch Fluoreszenzmikroskopie unter Verwendung eines 40X-Objektiv untersuchen. Die Filtersätze für DAPI, CFP, FITC und Cy3 sind (EX350 nm / Em460 nm), (Ex436 nm / Em480 nm), (EX470 nm / Em525 nm) und (Ex545 nm / Em605 nm), respectively.
Hinweis: Wenn der Hintergrund durch Autofluoreszenz der kompletten DMEM-Medium hoch ist, Medium ohne Phenolrot verwendet wird.
- Nach weiteren 16 h Inkubation einet 37 ° C, fügen die DNA interkalierende Fleck Hoechst 33342 zu dem Medium bis zu einer Endkonzentration von 1 uM.
3. Für die indirekte Immunfluoreszenz Studies
- Herstellung von Paraformaldehydlösung
- Wiegen Paraformaldehydpulver (4 g pro 100 ml PBS) und fügen Sie ihn in PBS.
Hinweis: Paraformaldehyd ein brennbarer Feststoff ist und eine potenzielle Krebs hazard. Es reizt Augen, Atmungsorgane und die Haut. Daher ist es wichtig, den Kontakt mit oder Inhalation von Paraformaldehyd zu vermeiden. - Hinzufügen NaOH zu der Lösung (150 & mgr; l 10 N NaOH / 100 ml).
- Halten der Lösung bei 37 bis 42 ° C für 2-3 h unter gelegentlichem Schütteln.
- PH-Wert auf 7,0 durch Essigsäure zu der Lösung Zugabe (etwa 75 & mgr; l / 100 ml) nach dem Paraformaldehyd Pulver gelöst ist.
- Wiegen Paraformaldehydpulver (4 g pro 100 ml PBS) und fügen Sie ihn in PBS.
- Zielproteine Färbung
- Nach weiterer Inkubation für 16 h bei 37 ° C, Waschen der transfizierten Zellen bei Raumtemperatur einmal mit PBS von den sechs-Well-Platte für 5 sec wirbeln.
- Fix die Zellen mit 1 ml frisch zubereitet, 4% Paraformaldehydlösung pro Vertiefung. für 30 min bei Raumtemperatur inkubieren.
- Waschen Sie die Zellen einmal mit PBS wurde 5 sec wirbelnden der sechs-Well-Platte. Entsorgen Sie die Lösung.
- Inkubieren der Zellen mit 1 ml von 0,1% Triton X-100 (in PBS) bei Raumtemperatur für 30 min. die detergent Triton-X 100 die Zellmembran permeabilisiert Zugang von Antikörpern auf ihre intrazellulären Ziele zu ermöglichen.
- Waschen der Zellen mit PBS viermal durch jedes Mal die sechs-Well-Platte für 5 sec wirbeln. Entsorgen Sie die Lösung nach jeder Wäsche.
- Inkubieren der Zellen mit 1 ml von primären Antikörpern (c-MYC Kaninchen-Antikörper oder p53-Maus-Antikörper; 0,1 & mgr; g / ml) in einem 10% FBS in PBS-Lösung bei Raumtemperatur für 4 Stunden Schaukel.
- Waschen mit PBS viermal durch die Sechs-Well-Platte für 5 Sekunden wirbeln jedes Mal. Entsorgen Sie die Lösung nach jeder Wäsche.
- Inkubieren mit 1 ml von Fluoreszenzfarbstoff-konjugierten sekundären Antikörper (Alexa Fluor 488-konjugiertem anti-Kaninchen oder Anti-Maus-IgG-Antikörper; 0,5 & mgr; g / ml) in 10% FBS in PBS bei Raumtemperatur im Dunkeln wurde 2 h Schaukel.
- Waschen mit PBS viermal durch die Sechs-Well-Platte für 5 Sekunden wirbeln jedes Mal. Entsorgen Sie die Lösung nach jeder Wäsche.
- Kernfärbung und Montage
- Inkubieren der Zellen mit dem 1 ml DNA interkalierende Farbstoff 4 ', 6-Diamidino-2-phenylindol (DAPI; 0,5 & mgr; g / ml) in PBS bei Raumtemperatur für 10 Minuten in Dunkelheit. Gehen 3.4 zu Schritt, wenn keine Deckgläser verwendet werden.
Anmerkung: DAPI ist ein DNA-interkalierenden Farbstoff, der die Zellkerne färbt. - Bewerben 10 ul Befestigungslösung auf jedem Objektträger. Montieren jedes Deckglas über die Befestigungslösung auf einem Objektträger.
- in der Dunkelheit über Nacht bei Raumtemperatur inkubieren.
- Seal die Ränder der Deckgläser mit Nagellack. Legen Sie in der Dunkelheit im Inneren einer Abzugshaube über Nacht die Nagellack um Abgase zu entfernen.
- Inkubieren der Zellen mit dem 1 ml DNA interkalierende Farbstoff 4 ', 6-Diamidino-2-phenylindol (DAPI; 0,5 & mgr; g / ml) in PBS bei Raumtemperatur für 10 Minuten in Dunkelheit. Gehen 3.4 zu Schritt, wenn keine Deckgläser verwendet werden.
- Fluoreszenzmikroskopie 19
- Am nächsten Tag, überprüfen Sie die Zellen durch Fluoreszenzmikroskopie einen 40X-Objektiv verwenden. Die Filtersätze für DAPI, CFP, FITC und Cy3 sind (EX350 nm / Em460 nm), (Ex436 nm / Em480 nm), (EX470 nm / Em525 nm) und (Ex545 nm / Em605 nm), respectively.
Ergebnisse
Exogen exprimierten Wildtyp KIF5B erschien homogen im Zytoplasma, während c-MYC schien hauptsächlich in dem Kern (1A). Jedoch wird das gebildete motorless KIF5B Mutante Aggregate im Zytoplasma (1A). Die Aggregation des motorlosen KIF5B induziert die Aggregation von c-MYC. Der Prozentsatz der Zellen aggregiert exprimieren TagCFP-markierten c-MYC in den Zellen, die Wildtyp-KIF5B war gering. Es war jedoch signifikant höher, wenn die motorlose KIF5B wurde coexprimiert (1A). Die beobachtete Kolokalisation von mutierten KIF5B mit c-MYC (Figur 1) legt nahe, dass die subzelluläre Lokalisation KIF5B von c-MYC reguliert und c-MYC ist eine Ladung von Kinesin-1. Negative Kontrollen, in denen die Konstrukte wurden allein ausgedrückt werden in der unteren Platte der 1A enthalten. Die Ergebnisse zeigen, dass es keinen signifikanten Durchbluten der Fluoreszenz Emission. Die motorlose KIF5B auch Aggregation des endogenen c-MYC (1B) und den Transkriptionsfaktor p53 (Figur 2) induziert, was anzeigt, dass c-MYC und p53 sind sowohl die Ladungen von Kinesin-1 und KIF5B regelt die subzelluläre Lokalisation von beiden körpereigenen Proteinen. Zusammen mit den Ergebnissen, dass die Kinesin-1-Inhibitor bengal Lacton (RBL) 20 induziert die Bildung von hochmolekularen Spezies sowohl c-MYC und p53 5, ist p53 stieg wahrscheinlich eine Ladung von Kinesin-1. Es ist interessant, dass die Kerntranskriptionsfaktor p53 zu beachten, wie c-MYC, ebenfalls aus dem Zellkern in das Zytoplasma für den proteasomalen Abbau 21,22 exportiert wird. Die Bewegung von Transkriptionsfaktoren durch KIF5B scheint spezifisch zu sein, da die Expression des motorlose KIF5B Mutante nicht die subzelluläre Lokalisation c-Fos nicht beeinträchtigte oder dazu führen, dass (Figur 3) zu aggregieren. Die obigen Daten zeigen, dass die method in dieser Veröffentlichung verwendet ermöglicht die hochspezifische Identifikation von Kinesin-1 Ladungen. Eine ähnliche Strategie kann auch auf andere Motorproteine angewendet werden.
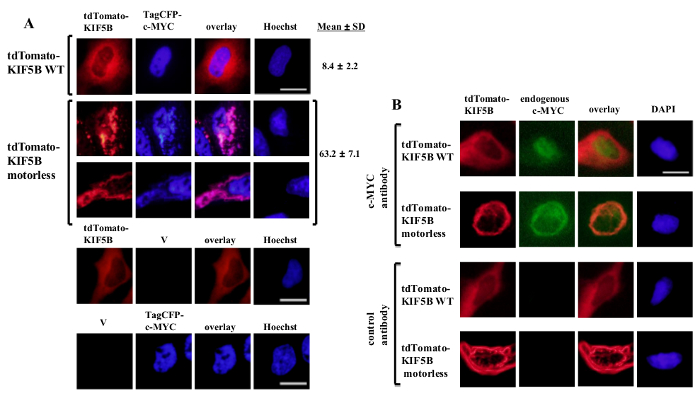
Abbildung 1: Die Expression des motorlosen KIF5B Mutante induziert c-MYC-Aggregation im Zytoplasma (A) wurden HeLa-Zellen, die mit tdTomato-markierten Wildtyp (WT) KIF5B (rot) und TagCFP-markierten c-MYC (blau).. tdTomato-markierten WT KIF5B schien hauptsächlich im Zytoplasma, während TagCFP-markierten c-MYC hauptsächlich im Zellkern erschien. Allerdings ist die tdTomato-markierten motorlose KIF5B Mutante (rot) fadenförmige oder punktförmige Aggregate im Zytoplasma gebildet. Die Expression des motorlose KIF5B induziert die Aggregation von c-MYC. Das mutierte KIF5B und c-MYC co-lokalisiert zusammen (pink). Die Prozentsätze von Zellen (%) Aggregate von GFP-c-MYC (%) sind anzeigt gezeigt. Die Ergebnisse sind alsMittelwert ± Standardabweichung (n = 3). Negative Kontrollen mit der Expression von tdTomato-markierten KIF5B Proteine oder TagCFP-markierten c-MYC zusammen mit leeren Vektoren (V) werden im unteren Bereich angezeigt. (B) Ähnliche Ergebnisse wurden mit dem endogenen c-MYC (grün) erhalten, wenn tdTomato-markierten WT oder motorlose KIF5B Proteine (rot) zum Ausdruck gebracht wurden. Die tdTomato-markierten motorless KIF5B Mutante Aggregate im Zytoplasma gebildet und Aggregation des endogenen c-MYC induziert. Beide Arten von Aggregaten co-lokalisiert gemeinsam in das Zytoplasma (orange). Die Kerne wurden mit dem Farbstoff Hoechst 33342 oder DAPI gefärbt. Maßstabsbalken; 20 & mgr; m. Bitte hier klicken, um eine größere Version dieser Figur zu sehen.

Abbildung 2: Die Expression des motorlosen KIF5B Mutante induziert endogenen p53-Aggregation in ter Zytoplasma. In HeLa-Zellen exogene tdTomato-markierten Wildtyp (WT) KIF5B (rot) erschien hauptsächlich im Zytoplasma, während das endogene p53 (grün) erschien vor allem in den Zellkern. Im Gegensatz dazu bildeten die tdTomato-markierten motorlose KIF5B Mutante (rot) Aggregate im Zytoplasma. Die Expression des motorlose KIF5B induziert die Aggregation von p53, was zu einer Co-Lokalisation von KIF5B und p53 im Zytoplasma (gelb). Das Experiment wurde dreimal durchgeführt. Die Kerne wurden mit dem Farbstoff DAPI gefärbt. Maßstabsbalken; 20 & mgr; m. Bitte hier klicken, um eine größere Version dieser Figur zu sehen.
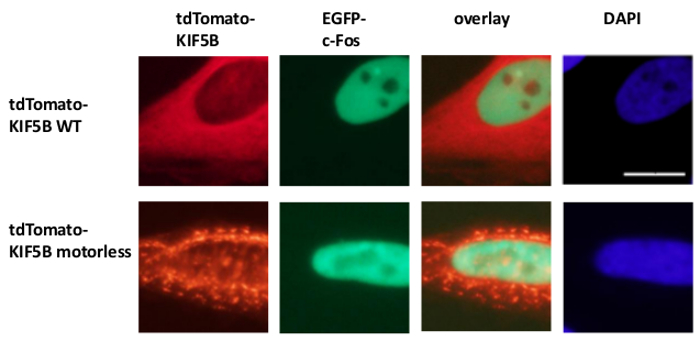
Abb. 3: Die Expression des motorlosen KIF5B Mutante nicht induziert die Aggregation von c-Fos im Zytoplasma tdTomato-markierten Wildtyp (WT) KIF5B (rot) erschienenhauptsächlich im Zytoplasma von HeLa-Zellen, während EGFP-c-Fos (grün) schien hauptsächlich im Zellkern. Im Gegenteil, die tdTomato-markierten motorless KIF5B Mutante (rot) gebildeten Aggregate im Zytoplasma. Die Expression des motorlose KIF5B hat die Aggregation von c-Fos nicht induzieren. Das Experiment wurde viermal durchgeführt. Die Kerne der Zellen wurden mit dem Farbstoff DAPI gefärbt. Maßstabsbalken; 20 & mgr; m. Bitte hier klicken, um eine größere Version dieser Figur zu sehen.
| gemeinsame Rückwärtsprimer | 5'-AGAGGATCCTTACACTTGTTTGCCTCCTC-3 ' |
| Wildtyp-KIF5B Vorwärtsprimer | 5'AGAGTCGACGCGGACCTGGCCGAGTGCAACATCAAAGT-3 ' |
| motorless KIF5B Vorwärtsprimer | 5'AGAGTCGACGATGAAGAGTTCACTGTTGC-3 ' |
Tabelle 1: Primersequenzen für die Wildtyp und motorlose KIF5B Proteine klonen.
Diskussion
Das beschriebene Verfahren nutzt die Eigenschaften des motorlose KIF5B Mutante, die die Fähigkeit, entlang der Mikrotubuli zu bewegen fehlt, aber behält die Fähigkeit Dimere mit dem Wildtyp-KIF5B zu bilden und dadurch ermöglichen, die tetrameres Protein mit den gleichen Ladung Proteinen zu interagieren wie der Wildtyp-KIF5B. Freilaufendes KIF5B wirkt daher als dominant negative Mutante und Formen mislocalized mit ihren Ladungen zusammenfasst. Dieses Verfahren hat sich bewährt, die Kinesin-1 Fracht c-MYC (Abbildung 1) 5 zu identifizieren. In diesem Artikel wurde das gleiche motorlose KIF5B Mutante zu identifizieren p53 als eine weitere potentielle Ladung von Kinesin-1 (2) verwendet. Dies zeigt, dass die Methodik möglich ist, andere Ladungen von Kinesin-1 zu identifizieren. Darüber hinaus wird die Spezifität der Mutante durch das Fehlen der Wirkung der Mutante auf der negativen Kontrollprotein c-Fos (Figur 3) vorgesehen.
In diesem Protokoll, das tdTomato-markierten Wild tyPE oder motorlose KIF5B Protein wird mit einem anderen fluoreszierenden Protein-markierten Kandidaten Fracht Protein koexprimiert. In diesem Fall werden die lebenden Zellen Fluoreszenzmikroskopie und bildgebende durchgeführt. Die Bildung der Aggregate durch Zeitraffer-Bildgebung verfolgt werden. Alternativ wird die tdTomato-markiertes Protein exprimiert allein und der Kandidat Ladung Protein an seine physiologischen Niveaus durch indirekten Immunfluoreszenzmikroskopie unter Verwendung von spezifischen Antikörpern sichtbar gemacht. Das fluoreszierende Protein tdTomato 17 für die Helligkeit und Lichtstabilität gewählt. Wenn der Hintergrund durch Autofluoreszenz der kompletten DMEM-Medium hoch ist, Medium ohne Phenolrot verwendet wird.
Die motorlose KIF5B Mutante bildet Dimere mit dem Wildtyp-KIF5B stöchiometrisch. Daher ist es wichtig, eine ausreichende Menge an motorlose KIF5B Mutante auszudrücken Dimere mit dem Wildtyp-Gegenstück zu bilden, deren Funktion zu inhibieren und Aggregate zu bilden. Zur Bewältigung dieses Problems, die Optimierung der ausdrITZUNG des motorlosen Mutante ist von wesentlicher Bedeutung. Es wird unter Verwendung eines kleinen motorlose Mutante mit dem Dimerisierungsdomäne erreicht. Zusätzlich Optimierung des entsprechenden Transfektionsreagenz und Protokoll ist ebenfalls notwendig. Die Dauer der Inkubation von HeLa-Zellen mit dem DNA / Transfektionsreagenz wurde in HeLa-Zellen für die Expression von exogenen Proteinen und Zelllebensfähigkeit optimiert. Die Inkubationsdauer werden müssen für andere Zelllinien optimiert.
Für Vergrößerungen zwischen 4X bis 40X, werden keine Deckgläser erforderlich. Die Zellen können direkt in den Vertiefungen untersucht werden. Daher wird in diesem Fall ist das Protokoll, preiswert und einfach. Für Vergrößerungen über 40x, werden die Zellen auf Deckgläser in den Vertiefungen gewachsen und, nach dem Färben, montiert auf Objektträger zur Untersuchung unter Öl-Immersionsobjektive.
Beobachtung der Aggregate des motorlose KIF5B Protein wird durch die Größe des Zytoplasmas beschränkt. Es ist leicht, das zu erkennen,Aggregate in vielen Säugerzellen. Es ist jedoch relativ schwierig, die Aggregate in neuronalen Zellen zu beobachten, wenn das Volumen des Zytoplasma und um die Kerne in Neuriten klein ist.
Das Protokoll wird verwendet, um den Zusammenhang zwischen 13-16 KIF5B und ihre Ladungen zusätzlich zu der in vivo Hefe-Zwei-Hybrid und biochemische Pulldown-Assays zu zeigen. Alle diese Tests den Verein unter verschiedenen physikalischen Bedingungen bestimmen und ihre Ergebnisse gegenseitig ergänzen können. Darüber hinaus ist der Vorteil dieses Fluoreszenzassay im Vergleich zu anderen Assays, dass es die Regulierung der subzellulären Lokalisierung der Ladungen durch KIF5B zeigen können (1 und 2).
Das Protokoll ist nicht auf KIF5B beschränkt und kann verwendet werden Ladungen anderer Motor Proteine zu identifizieren, wie einige andere Kinesin Motoren 3 und einige der Myosin Motoren 23,24 in den intrazellulären Transport von Ladungen verwendet 3. Diese Motorproteine auch Motor-Domains für die Bewegung entlang von Mikrotubuli für Kinesin-Motoren und Mikrofilamenten für Myosinmotoren enthalten, und Coiled-Coil-Segmente für die Oligomerisierung. Die meisten von ihnen bilden Homodimere 3,23. Daher kann eine ähnliche Strategie zu ihnen angewendet werden durch ihre motorlose dominant negative Mutanten schaffen ihre Ladungen zu identifizieren.
Offenlegungen
Free Access Publikation und Produktion dieser Artikel ist für die von Roche bezahlt.
Danksagungen
Roche's Publication Grant covered the Free Access publication and production of this article. The author also thanks E. Premkumar Reddy, Richard V. Mettus, Stephen C. Cosenza, Sau Ying Yip and Sol D. Gloria for their support and critical reading of the manuscript.
Materialien
| Name | Company | Catalog Number | Comments |
| absolute ethanol (200 proof) | Fisher Scientific | BP2818 | |
| DAPI | Sigma-Aldrich | D9542 | |
| Hoechst 33342 | Sigma-Aldrich | B2261 | |
| Opti-MEM-I (transfection medium) | Life Technologies | 51985 | |
| ProLong Diamond Antifade Mountant | Life Technologies | P36961 | |
| formaldehyde, para | Fisher Scientific | O4042-500 | |
| Triton-X100 | Fisher Scientific | BP151-500 | |
| X-tremeGENE 9 DNA Transfection Reagent | Roche | 6365787001 | |
| Taq DNA polymerase | Life Technologies | 10342020 | |
| PCR Grade Nucleotide Mix (dNTP Mix) | Roche | 12111424 | |
| Microscope cover glass | Fisher Scientific | 12-541A | |
| GeneRuler 1 kb Plus DNA ladder | Life Technologies | SM1333 | |
| PureLink Quick Gel Extraction kit | Life Technologies | K210012 | |
| BamHI | New England Biolab | R0136 | |
| SalI | New England Biolab | R0138 | |
| T4 DNA ligase | New England Biolab | M0202T | |
| Ethidium bromide | Thermo Scientific | 17898 | |
| DMEM | Life Technologies | 11995-065 | |
| c-MYC rabbit antibody | Cell Signaling | 5606 | |
| p53 mouse antibody | Santa Cruz | sc-126 | |
| Alexa Fluor 488-conjugated anti-rabbit IgG antibody | Life Technologies | A11008 | |
| Alexa Fluor 488-conjugated anti-mouse IgG antibody | Life Technologies | A11059 | |
| fluorescent microscope | Olympus IX71_Fluoview | ||
| computer software for imaging | cellSens |
Referenzen
- Hirokawa, N., Niwa, S., Tanaka, Y. Molecular motors in neurons: transport mechanisms and roles in brain function, development, and disease. Neuron. 68, 610-638 (2010).
- Yu, Y., Feng, Y. M. The role of kinesin family proteins in tumorigenesis and progression: potential biomarkers and molecular targets for cancer therapy. Cancer. 116, 5150-5160 (2010).
- Verhey, K. J., Hammond, J. W. Traffic control: regulation of kinesin motors. Nat Rev Mol Cell Biol. 10, 765-777 (2009).
- Hirokawa, N., Noda, Y., Tanaka, Y., Niwa, S. Kinesin superfamily motor proteins and intracellular transport. Nat Rev Mol Cell Biol. 10, 682-696 (2009).
- Lee, C. M. Transport of c-MYC by Kinesin-1 for proteasomal degradation in the cytoplasm. Biochim Biophys Acta. 1843, 2027-2036 (2014).
- Bittins, C. M., Eichler, T. W., Hammer, J. A., Gerdes, H. H. Dominant-negative myosin Va impairs retrograde but not anterograde axonal transport of large dense core vesicles. Cell Mol Neurobiol. 30, 369-379 (2010).
- Kimura, T., Watanabe, H., Iwamatsu, A., Kaibuchi, K. Tubulin and CRMP-2 complex is transported via Kinesin-1. J Neurochem. 93, 1371-1382 (2005).
- Lindsay, A. J., McCaffrey, M. W. Myosin Va is required for the transport of fragile X mental retardation protein (FMRP) granules. Biol Cell. 106, 57-71 (2014).
- Rivera, J., Chu, P. J., Lewis, T. L., Arnold, D. B. The role of Kif5B in axonal localization of Kv1 K(+) channels. Eur J Neurosci. 25, 136-146 (2007).
- Roland, J. T., Kenworthy, A. K., Peranen, J., Caplan, S., Goldenring, J. R. Myosin Vb interacts with Rab8a on a tubular network containing EHD1 and EHD3. Mol Biol Cell. 18, 2828-2837 (2007).
- Uchida, A., Alami, N. H., Brown, A. Tight functional coupling of kinesin-1A and dynein motors in the bidirectional transport of neurofilaments. Mol Biol Cell. 20, 4997-5006 (2009).
- Zadeh, A. D., et al. Kif5b is an essential forward trafficking motor for the Kv1.5 cardiac potassium channel. J Physiol. 587, 4565-4574 (2009).
- Su, Y. Y., et al. KIF5B promotes the forward transport and axonal function of the voltage-gated sodium channel Nav1.8. J Neurosci. 33, 17884-17896 (2013).
- Lalioti, V. S., Vergarajauregui, S., Tsuchiya, Y., Hernandez-Tiedra, S., Sandoval, I. V. Daxx functions as a scaffold of a protein assembly constituted by GLUT4, JNK1 and KIF5B. . J Cell Physiol. 218, 416-426 (2009).
- Cho, K. I., et al. Association of the kinesin-binding domain of RanBP2 to KIF5B and KIF5C determines mitochondria localization and function. Traffic. 8, 1722-1735 (2007).
- Diefenbach, R. J., Diefenbach, E., Douglas, M. W., Cunningham, A. L. The ribosome receptor, p180, interacts with kinesin heavy chain, KIF5B. Biochem Biophys Res Commun. 319, 987-992 (2004).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22, 1567-1572 (2004).
- Waters, J. C. Live-cell fluorescence imaging. Methods Cell Biol. 114, 125-150 (2013).
- Sanderson, M. J., Smith, I., Parker, I., Bootman, M. D. Fluorescence microscopy. Cold Spring Harb Protoc. 2014, (2014).
- Hopkins, S. C., Vale, R. D., Kuntz, I. D. Inhibitors of kinesin activity from structure-based computer screening. Biochemistry. 39, 2805-2814 (2000).
- Zhang, Y., Xiong, Y. Control of p53 ubiquitination and nuclear export by MDM2 and ARF. Cell Growth Differ. 12, 175-186 (2001).
- Michael, D., Oren, M. The p53-Mdm2 module and the ubiquitin system. Semin Cancer Biol. 13, 49-58 (2003).
- Kneussel, M., Wagner, W. Myosin motors at neuronal synapses: drivers of membrane transport and actin dynamics. Nat Rev Neurosci. 14, 233-247 (2013).
- Maravillas-Montero, J. L., Santos-Argumedo, L. The myosin family: unconventional roles of actin-dependent molecular motors in immune cells. J Leukoc Biol. 91, 35-46 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten