Method Article
Identificazione Kinesin-1 Cargos Utilizzando la microscopia a fluorescenza
In questo articolo
Riepilogo
Qui, un protocollo è presentato per identificare Kinesin-1 carichi. Un mutante senza motore della Kinesin-1 catena pesante (KIF5B) aggrega nel citoplasma e induce l'aggregazione dei suoi carichi. Entrambi gli aggregati vengono rilevate al microscopio a fluorescenza. Una strategia simile può essere impiegato per identificare carichi di altre proteine motrici.
Abstract
Fluorescence microscopy is employed to identify Kinesin-1 cargos. Recently, the heavy chain of Kinesin-1 (KIF5B) was shown to transport the nuclear transcription factor c-MYC for proteosomal degradation in the cytoplasm. The method described here involves the study of a motorless KIF5B mutant for fluorescence microscopy. The wild-type and motorless KIF5B proteins are tagged with the fluorescent protein tdTomato. The wild-type tdTomato-KIF5B appears homogenously in the cytoplasm, while the motorless tdTomato-KIF5B mutant forms aggregates in the cytoplasm. Aggregation of the motorless KIF5B mutant induces aggregation of its cargo c-MYC in the cytoplasm. Hence, this method provides a visual means to identify the cargos of Kinesin-1. A similar strategy can be utilized to identify cargos of other motor proteins.
Introduzione
Kinesin-1 è una proteina motore che media il trasporto anterogrado dei suoi carichi 1,2. Si tratta di un heterotetramer delle due subunità della catena Kinesin Luce 1 (KLC1) e due subunità di catene pesanti (Kinesin KHCs). KIF5B 1, un KHC, contiene un dominio motore al suo N-terminale, che idrolizza ATP e converte l'energia chimica in energia meccanica per il movimento lungo i microtubuli. La regione C-terminale contiene il dominio di dimerizzazione che interagisce con KLC1, che associa con carichi. Kinesin-1 trasporta carichi, come vescicole, organelli e mRNA 3,4. Recentemente, KIF5B stato dimostrato per il trasporto del fattore di trascrizione nucleare c-MYC degradazione proteosomal nel citoplasma 5. Tre metodologie (inibitore chimico, siRNA / shRNA e dominante mutante negativo) sono stati usati per inibire Kinesin-1 funzione. Tutti indotto aggregazione di c-MYC nel citoplasma. Per l'ultima metodologia, c-Myc è stato influenzato solo dal negat dominanteive mutante di KIF5B, ma non da quella di un altro relativo motore di proteine KIF5A, suggerendo che il mutante non esercitare effetti generali sui componenti intracellulari (come microtubuli interruzione) o sull'aggregazione proteica. Il mutante dominante negativo di KIF5B, inoltre, non ha influenzato un altro fattore di trascrizione, suggerendo che non esercita effetti generali sui fattori di trascrizione. Piuttosto, suggerisce che il mutante dominante negativo esercita effetti specifici sui suoi carichi.
L'uso di mutanti dominanti negativi è comune nel campo delle proteine motrici. Simili mutanti prive di motore di kinesins e myosins sono stati utilizzati in precedenza. Essi sono stati utilizzati principalmente per dimostrare gli effetti dei mutanti sulle localizzazioni subcellulari dei loro carichi o sulle funzioni cellulari 6-12. Meno enfasi è stata posta sul rapporto spaziale tra i mutanti e i carichi da esse interessati. Tuttavia, in alcuni incidenze, sono stati osservati i mutanti di co-localizza con la cargos 6,10.
L'interazione tra KIF5B e le sue proteine associate è stato già confermato dal lievito in vivo test doppio ibrido e saggi di pull-down biochimici come co-immunoprecipitazione e in saggi di pull-down in vitro 13-16. In questo articolo, un ulteriore metodo che utilizza la microscopia a fluorescenza visivo è descritto per identificare le proteine cargo KIF5B. Il metodo fa uso di un mutante KIF5B senza motore che agisce come un mutante dominante negativo. Si aggrega nel citoplasma e induce l'aggregazione dei suoi carichi.
Il tagging delle wild-type e mutante senza motore KIF5B con la proteina fluorescente tdTomato 17 consente la loro visualizzazione al microscopio a fluorescenza. Le proteine KIF5B marcati possono essere co-espresso con una proteina candidato fusa ad una proteina fluorescente differente con proprietà spettrali opportunamente separate dal tag KIF5B. Le proteine taggati sono osservati direttamente in cellule viveal microscopio a fluorescenza. Induzione di aggregazione della proteina candidato da parte del mutante KIF5B senza motore confermerà che la proteina candidato è un carico in vivo di KIF5B. Inoltre, le proteine KIF5B tdTomato-tag possono essere espressi solo nelle cellule per studiare gli effetti sulle proteine endogene cargo. Successivamente, immunofluorescenza è condotto in cui le cellule transfettate vengono fissate e colorate con un anticorpo specifico contro la proteina endogena candidato, seguito da un anticorpo secondario coniugato adeguato con un colorante fluorescente. In questo caso, la proteina endogena candidato al livello fisiologico è studiato. Simili mutanti prive di motore delle altre proteine motrici possono essere preparati per identificare i loro carichi.
Protocollo
1. La clonazione di wild-type tdTomato-tag e senza motore KIF5B Proteine
- Amplificazione del cDNA per la wild-type umana e proteine KIF5B senza motore utilizzando i primers in Tabella 1, Taq DNA polimerasi (5 unità per 100 ml), dNTP mix (2 mM per ogni deossinucleotide) e il suo buffer 10X per 30 cicli. Ciascun ciclo consiste di una fase di denaturazione (95 ° C per 30 sec), una fase di ricottura (45 ° C per 30 sec) ed una fase di estensione (72 ° C per 3 min).
- Estrarre il prodotto DNA amplificato a parità di volume di fenolo / cloroformio (1: 1).
Nota: Il fenolo è infiammabile e può causare ustioni. Il cloroformio è pericoloso. Evitare il contatto diretto con loro e li usa sotto una cappa. In alternativa, i prodotti di PCR possono essere purificati mediante vari kit.- Spin a 18.000 xg in una microcentrifuga a temperatura ambiente per 1 min. Trasferire la soluzione acquosa in una nuova provetta ed estrarre con un uguale volume di cloroformio.
- rotazionea 18.000 xg in una microcentrifuga a temperatura ambiente per 0,5 min. Trasferire la soluzione acquosa in una nuova provetta.
- Miscelare la soluzione acquosa con un decimo del volume di 3 M Na-acetato e due volumi di etanolo assoluto.
- Spin a 18.000 xg in una microcentrifuga a temperatura ambiente per 5 min. Eliminare la soluzione acquosa.
- Lavare il pellet di DNA con due volumi di etanolo al 75%. Scartare la soluzione acquosa e asciugare il pellet di DNA a temperatura ambiente per 5 min. Risospendere il pellet di DNA in 34 ml di acqua.
- Digest prodotti amplificati in un volume finale di 40 microlitri con enzimi di restrizione SalI (10 unità) e BamHI (10 unità) e 4 ml di tampone 10X loro a 37 ° C per due ore. Separare i prodotti digeriti in un gel di agarosio (1,0% a 100 V) contenente etidio bromuro (0,2 mcg / ml).
Nota: il bromuro di etidio è un agente mutageno potente e può essere assorbito attraverso la pelle. Pertanto, è importante evitare il contatto diretto o indiretto wesimo bromuro di etidio. - Tagliare le bande corretti per la purificazione da colonne. Pesare il gel contenente il frammento di DNA. Sciogliere quindi nel buffer solubilizzazione (300 microlitri per 0,1 g) a 37 ° C per circa 20 min.
- Aggiungere la soluzione portato ad una colonna e centrifuga in una microcentrifuga a temperatura ambiente per 5 sec. Gettare il flow-through.
- Lavare la colonna con tampone di solubilizzazione 0,5 ml ripetendo il passo 1.5. Lavare la colonna di nuovo con 0,75 ml tampone di lavaggio ripetendo il passo 1.5. Gira la colonna per 2 minuti per sbarazzarsi del tampone di lavaggio residuo.
- Eluire il frammento di DNA con 50 microlitri di acqua ripetendo il passaggio 1.5. Valutare la concentrazione del frammento di DNA eseguendo una aliquota di contro una scala dimensioni DNA in gel di agarosio (1,0% a 100 V) contenente etidio bromuro (0,2 mg / ml).
- Legare i prodotti purificati (circa 100 ng) con il vettore ptdTomato-C1 17 (circa 100 ng) precedentemente digerito con °e stessa enzimi di restrizione in 10 microlitri utilizzando T4 DNA ligasi (2.000 unità) e 1 ml 10X tampone di ligasi a temperatura ambiente per 16 ore.
Nota: La wild-type e mutanti senza motore contiene aminoacidi da 2 a 963 e 584-963, rispettivamente. In precedenza, una più grande mutante KIF5B senza motore è stato utilizzato per identificare la sua funzione 9. Una più piccola senza motore KIF5B mutante è stato utilizzato per gli attuali studi per aumentare i propri livelli di espressione.
2. La microscopia ad immunofluorescenza
- Per live-cell imaging di fluorescenza o indiretto con un ingrandimento pari o inferiore a 40X, semi di 0,2-0,3 milioni di cellule HeLa in 1 ml di mezzo completo [Dulbecco Modified Eagle Medium (DMEM) con il 10% di siero fetale bovino (FBS)] in ciascun pozzetto di una sei pozzetti. Crescere a 37 ° C con 5% di CO 2.
- Per gli studi di immunofluorescenza indiretta con ingrandimenti superiori 40X, lavare bicchieri coperchio (18 mm x 18 mm; 1,5 di spessore) in breve tempo etanolo assoluto e aria asciugarei pozzetti di una piastra ben sei, all'interno di un armadio biosicurezza per evitare la contaminazione.
- Seme 0,2-0,3 milioni di cellule in 1 ml completano terreno DMEM in ciascun pozzetto di una piastra a sei pozzetti.
- Preparazione di Transfection Complex
- Il giorno successivo, all'interno di un armadio biosicurezza, diluire 0,6 mg (totale) del plasmide di espressione per tdTomato-tag wild-type o KIF5B senza motore in presenza o assenza di un plasmide di espressione per una proteina cargo candidato (pTagCFP-c-MYC 5 ) con 0,1 ml di mezzo di transfezione in una provetta 1,6 ml microcentrifuga.
Nota: Il vettore pTagCFP-c-Myc esprime la TagCFP-tag c-myc. - Aggiungere 1,8 microlitri di reagente trasfezione a 0,1 ml di mezzo transfezione in un altro tubo di 1,6 ml microcentrifuga. In genere, preparare un master mix di reagente trasfezione diluito sufficiente per 12 campioni.
- Incubare per 5 minuti, a temperatura ambiente.
- Aggiungere il diluita di 0,1 ml di reagente di trasfezione alle diluiti 0,1 ml DNA. Mix il contenuto invertendo i tubi. Spin le provette per 5 secondi in una microcentrifuga.
- Incubare per 45 minuti, a temperatura ambiente.
- Il giorno successivo, all'interno di un armadio biosicurezza, diluire 0,6 mg (totale) del plasmide di espressione per tdTomato-tag wild-type o KIF5B senza motore in presenza o assenza di un plasmide di espressione per una proteina cargo candidato (pTagCFP-c-MYC 5 ) con 0,1 ml di mezzo di transfezione in una provetta 1,6 ml microcentrifuga.
- Lavaggio delle cellule
- Lavare le cellule seminate tre volte con tampone fosfato isotonico (PBS) durante il periodo di incubazione per la formazione del complesso trasfezione.
- Sostituire il medium delle cellule seminate in ogni pozzetto con 0,8 ml di media trasfezione pre-riscaldato. Riportare le piastre per l'incubatore a 37 ° C.
- Trasfezione delle cellule
- Dopo incubazione di 45 min, aggiungere 0,2 ml del complesso reattivo DNA / trasfezione (preparato al punto 2.3) goccia a goccia in ciascun pozzetto della piastra.
- Rock the piastra 6 bene delicatamente per 5 secondi, prima di essere restituiti al incubatore a 37 ° C.
- Dopo 6-8 ore, sostituire il mezzo con il mezzo DMEM completo.
- Per Live-cell imaging di fluorescenza 18
- Dopo un ulteriore 16 ore di incubazione at 37 ° C, aggiungere la macchia intercalante del DNA Hoechst 33342 al mezzo ad una concentrazione finale di 1 mM.
Nota: Hoechst 33342 è un colorante intercalante del DNA delle cellule permeabili che macchia i nuclei delle cellule. - Incubare le cellule trasfettate per 10 min in incubatore a 37 ° C prima di esaminarli per l'espressione di proteine fluorescenti mediante microscopia a fluorescenza utilizzando un obiettivo 40X. I set di filtri per DAPI, CFP, FITC e Cy3 sono (Ex350 nm / Em460 nm), (Ex436 nm / Em480 nm), (EX470 nm / Em525 nm) e (Ex545 nm / Em605 nm), rispettivamente.
Nota: Se lo sfondo è elevato a causa di auto-fluorescenza del mezzo DMEM completo, viene utilizzato mezzo senza rosso fenolo.
- Dopo un ulteriore 16 ore di incubazione at 37 ° C, aggiungere la macchia intercalante del DNA Hoechst 33342 al mezzo ad una concentrazione finale di 1 mM.
3. Per indiretti studi di immunofluorescenza
- Preparazione della soluzione Paraformaldeide
- Pesare in polvere paraformaldeide (4 g per 100 ml di PBS) e aggiungerlo al PBS.
Nota: Paraformaldeide è un solido infiammabile e un potenziale Hazar cancrod. Si irrita gli occhi, le vie respiratorie e la pelle. Pertanto, è importante evitare il contatto con o inalazione di paraformaldeide. - Aggiungere NaOH alla soluzione (150 microlitri NaOH 10 N / 100 ml).
- Mantenere la soluzione a 37 a 42 ° C per 2-3 ore con agitazione occasionale.
- Regolare il pH a 7,0 mediante aggiunta di acido acetico glaciale alla soluzione (circa 75 microlitri / 100 ml) dopo la polvere paraformaldeide si scioglie.
- Pesare in polvere paraformaldeide (4 g per 100 ml di PBS) e aggiungerlo al PBS.
- Proteine bersaglio di colorazione
- Dopo ulteriore incubazione per 16 ore a 37 ° C, lavare le cellule trasfettate a temperatura ambiente una volta con PBS agitando la piastra sei pozzetti per 5 sec.
- Fissare le cellule con 1 ml di appena preparata, 4% soluzione di paraformaldeide per pozzetto. Incubare a temperatura ambiente per 30 min.
- Lavare le cellule con PBS tempo agitando per 5 secondi il piatto a sei bene. Scartare la soluzione.
- Incubare le cellule con 1 ml di 0,1% Triton-X 100 (in PBS) a temperatura ambiente per 30 min. il detergent Triton-X 100 si permeabilize la membrana cellulare per consentire l'accesso degli anticorpi ai loro bersagli intracellulari.
- Lavare le cellule con PBS quattro volte agitando la piastra sei pozzetti per 5 secondi ogni volta. Scartare la soluzione dopo ogni lavaggio.
- Incubare le cellule con 1 ml di anticorpi primari (c-MYC anticorpo di coniglio o dell'anticorpo p53 del mouse; 0,1 mg / ml) in un FBS 10% in soluzione PBS a temperatura ambiente agitandola per 4 ore.
- Lavare con PBS quattro volte agitando la piastra sei pozzetti per 5 secondi ogni volta. Scartare la soluzione dopo ogni lavaggio.
- Incubare con 1 ml di anticorpi secondari colorante coniugato fluorescenti (Alexa Fluor 488 coniugato anti-coniglio o anticorpi IgG anti-topo; 0,5 mg / ml) in 10% FBS in PBS a temperatura ambiente al buio facendo oscillare per 2 ore.
- Lavare con PBS quattro volte agitando la piastra sei pozzetti per 5 secondi ogni volta. Scartare la soluzione dopo ogni lavaggio.
- Colorazione nucleare e di montaggio
- Incubare le cellule con 1 ml di DNA intercalante dye 4 ', 6-diamidino-2-fenilindolo (DAPI; 0,5 mg / ml) in PBS a temperatura ambiente, al buio per 10 min. Passare al punto 3.4 quando non vetrini vengono utilizzati.
Nota: DAPI è un colorante intercalante del DNA che macchia i nuclei delle cellule. - Applicare 10 ml di soluzione di montaggio su ogni vetrino da microscopio. Montare ciascun vetro di copertura sopra la soluzione di montaggio su un vetrino da microscopio.
- Incubare a temperatura ambiente nella notte scura.
- Sigillare i bordi dei vetrini con smalto. Mettere al buio all'interno di una cappa aspirante durante la notte per rimuovere il chiodo fumi smalto.
- Incubare le cellule con 1 ml di DNA intercalante dye 4 ', 6-diamidino-2-fenilindolo (DAPI; 0,5 mg / ml) in PBS a temperatura ambiente, al buio per 10 min. Passare al punto 3.4 quando non vetrini vengono utilizzati.
- Microscopia a fluorescenza 19
- Il giorno dopo, esaminare le cellule al microscopio a fluorescenza utilizzando un obiettivo 40X. I set di filtri per DAPI, CFP, FITC e Cy3 sono (Ex350 nm / Em460 nm), (Ex436 nm / Em480 nm), (EX470 nm / Em525 nm) e (Ex545 nm / Em605 nm), rispettivamente.
Risultati
Esogenamente espresso wild-type KIF5B apparso omogeneamente nel citoplasma, mentre c-MYC apparso prevalentemente nel nucleo (Figura 1A). Tuttavia, il KIF5B mutante privo di motore formata aggregati nel citoplasma (Figura 1A). Aggregazione del KIF5B senza motore ha indotto l'aggregazione di c-myc. La percentuale di cellule che esprimono aggregato TagCFP-tag c-myc nelle cellule che esprimono wild-type KIF5B è stata bassa. Tuttavia, è stato significativamente più alto quando il KIF5B senza motore è stato co-espressa (Figura 1A). Il osservato co-localizzazione KIF5B mutante c-MYC (Figura 1) suggerisce che KIF5B regola la localizzazione subcellulare di c-myc e c-MYC è un carico di Kinesin-1. I controlli negativi in cui i costrutti sono stati espressi solo sono inclusi nel riquadro inferiore della figura 1A. I risultati mostrano che non vi era alcuna significativa sanguinare-through della fluorescenza emission. Il KIF5B senza motore anche indotto aggregazione della endogena c-MYC (Figura 1B) e il fattore di trascrizione p53 (Figura 2), indicando che c-Myc e p53 sono entrambi i carichi di Kinesin-1 e KIF5B regola la localizzazione subcellulare di entrambe le proteine endogene. Insieme con i risultati che la Kinesin-1 inibitore rosa bengala lattone (RBL) formazione 20 induce di specie ad alto peso molecolare sia per c-myc e p53 5, p53 è probabile un carico di Kinesin-1. È interessante notare che il fattore di trascrizione nucleare p53, come c-MYC, viene esportato anche dal nucleo al citoplasma per proteasoma degradazione 21,22. Il movimento dei fattori di trascrizione da KIF5B sembra essere specifico perché l'espressione del mutante KIF5B senza motore non ha influenzato la localizzazione subcellulare c-Fos o farla aggregare (Figura 3). I dati sopra riportati dimostrano che il metod impiegato in questa pubblicazione consente l'identificazione ad alta specificità della Kinesin-1 carichi. Una strategia simile può essere applicata ad altre proteine motrici pure.
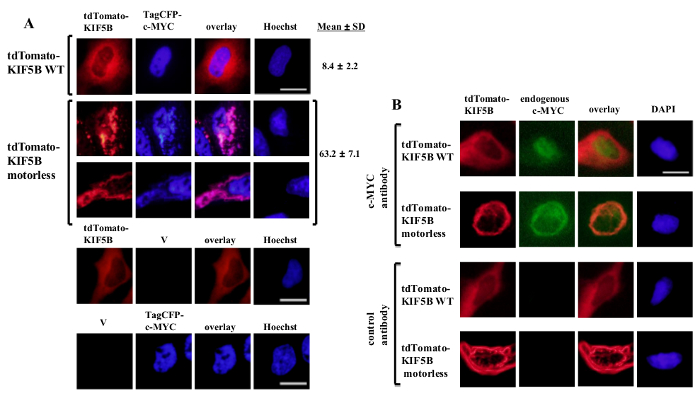
Figura 1: Espressione del mutante KIF5B senza motore induce c-Myc aggregazione nel citoplasma (A) cellule HeLa sono state trasfettate con tdTomato-tag wild-type (WT) KIF5B (rosso) e TagCFP-tag c-Myc (blu).. tdTomato-tag WT KIF5B apparso principalmente nel citoplasma, mentre TagCFP-etichettato c-MYC apparso prevalentemente nel nucleo. Tuttavia, il mutante KIF5B senza motore tdTomato-tag (rosso) formata aggregati filamentosi o puntiformi nel citoplasma. Espressione dei KIF5B senza motore indotta l'aggregazione di c-MYC. Il KIF5B mutante e c-Myc co-localizzate insieme (rosa). Sono mostrati Percentuali di cellule (%) indicanti aggregati di CFP-c-MYC (%). I risultati sono mostrati comemedia ± SD (n = 3). I controlli negativi con l'espressione di proteine KIF5B tdTomato-tag o TagCFP-tag c-Myc con vettori vuoti (V) sono mostrati nel pannello inferiore. (B) Risultati simili sono stati ottenuti con il endogena c-MYC (verde) quando sono stati espressi tdTomato-tag WT o proteine KIF5B senza motore (rossa). Il KIF5B mutante senza motore tdTomato-tag formata aggregati nel citoplasma e l'aggregazione del endogena c-Myc indotta. Entrambi i tipi di aggregati co-localizzate insieme nel citoplasma (arancione). I nuclei sono stati colorati con il colorante Hoechst 33342 o DAPI. Barra di scala; 20 micron. Cliccate qui per vedere una versione più grande di questa figura.

Figura 2: Espressione del mutante KIF5B senza motore produce aggregazione p53 endogena in tha citoplasma. Nelle cellule HeLa, esogeno tdTomato-tag wild-type (WT) KIF5B (rosso) apparso principalmente nel citoplasma, mentre il p53 endogena (verde) apparso principalmente nel nucleo. Al contrario, il mutante KIF5B senza motore tdTomato-tag (rosso) aggregati nel citoplasma formata. Espressione dei KIF5B senza motore indotta l'aggregazione di p53, causando la co-localizzazione KIF5B e p53 nel citoplasma (giallo). L'esperimento è stato eseguito per tre volte. I nuclei sono stati colorati con il DAPI colorante. Barra di scala; 20 micron. Cliccate qui per vedere una versione più grande di questa figura.
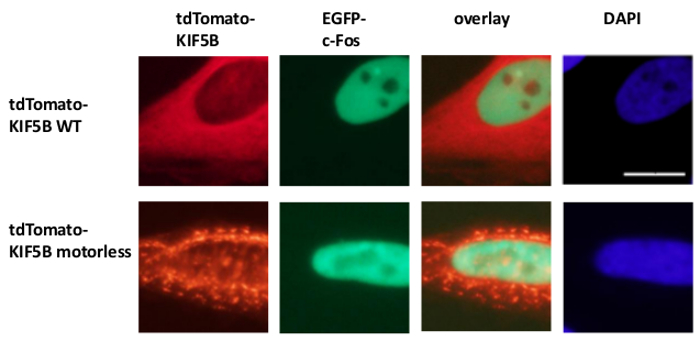
Figura 3:. L'espressione del mutante KIF5B senza motore non induce l'aggregazione di c-Fos nel citoplasma tdTomato-tagged wild-type (WT) KIF5B (rosso) è apparsoprincipalmente nel citoplasma delle cellule HeLa, mentre EGFP-c-Fos (verde) apparso principalmente nel nucleo. Al contrario, il mutante KIF5B senza motore tdTomato-tag (rosso) aggregati nel citoplasma formata. L'espressione della KIF5B senza motore non ha indotto l'aggregazione di c-Fos. L'esperimento è stato eseguito quattro volte. I nuclei delle cellule sono state colorate con DAPI colorante. Barra di scala; 20 micron. Cliccate qui per vedere una versione più grande di questa figura.
| primer reverse comune | 5'-AGAGGATCCTTACACTTGTTTGCCTCCTC-3 ' |
| wild-type KIF5B primer forward | 5'-AGAGTCGACGCGGACCTGGCCGAGTGCAACATCAAAGT-3 ' |
| KIF5B senza motore primer forward | 5'-AGAGTCGACGATGAAGAGTTCACTGTTGC-3 ' |
Tabella 1: sequenze primer per la clonazione del wild-type e le proteine KIF5B senza motore.
Discussione
Il metodo descritto utilizza le proprietà del mutante KIF5B senza motore, che non ha la capacità di muoversi lungo i microtubuli, ma mantiene la capacità di formare dimeri con wild-type KIF5B e, quindi, consentire la proteina tetramerica per interagire con le stesse proteine cargo come wild-type KIF5B. KIF5B senza motore, di conseguenza, agisce come un mutante dominante negativo e forme mislocalized aggrega con i suoi carichi. Questo metodo è dimostrato di identificare il Kinesin-1 cargo c-MYC (Figura 1) 5. In questo articolo, lo stesso mutante KIF5B senza motore è stato utilizzato per identificare p53 come un altro potenziale carico di Kinesin-1 (Figura 2). Questo dimostra che la metodologia sia praticabile individuare altri carichi di Kinesin-1. Inoltre, la specificità del mutante è fornito dalla mancanza di effetto del mutante della proteina controllo negativo c-Fos (Figura 3).
In questo protocollo, il tdTomato-tag wild-tyPE o proteina KIF5B senza motore è coespressi con un'altra proteina carico candidato proteina-tag fluorescente. In questo caso, live-cell microscopia a fluorescenza e di imaging vengono eseguite. La formazione degli aggregati può essere fatta dal time-lapse imaging. In alternativa, la proteina tdTomato-tag si esprime da solo e il candidato del carico di proteine ai suoi livelli fisiologici viene visualizzato al microscopio immunofluorescenza indiretta utilizzando anticorpi specifici. Il tdTomato proteina fluorescente viene scelto per la sua luminosità e fotostabilità 17. Se lo sfondo è elevato a causa di auto-fluorescenza del mezzo DMEM completo, viene utilizzato mezzo senza rosso fenolo.
Il KIF5B mutante senza motore forma dimeri con wild-type KIF5B stechiometricamente. Pertanto, è fondamentale per esprimere quantità sufficiente di senza motore mutante KIF5B per formare dimeri con wild-type controparte di inibire la sua funzione e per formare aggregati. Per risolvere questo problema, l'ottimizzazione di esprESSIONE del mutante senza motore è essenziale. Si è ottenuto utilizzando un piccolo mutante senza motore contenente il dominio di dimerizzazione. Inoltre, l'ottimizzazione del reagente di trasfezione appropriato e protocollo è anche necessario. La durata dell'incubazione delle cellule HeLa con il reagente DNA / trasfezione è stata ottimizzata in cellule HeLa per l'espressione di proteine esogene e vitalità cellulare. La durata di incubazione può essere ottimizzato per altre linee cellulari.
Per ingrandimenti tra 4X a 40X, non sono necessari gli occhiali di copertura. Le celle possono essere esaminate direttamente nei pozzetti. Pertanto, in questo caso, il protocollo è economico e conveniente. Per ingrandimenti superiori 40X, le cellule sono coltivate su vetrini nei pozzi e, dopo la colorazione, sono montati su vetrini da microscopio per l'esame al obiettivi ad immersione in olio.
Osservazione aggregati della proteina KIF5B senza motore è limitata dalla dimensione del citoplasma. È facile rilevare laaggregati in molte cellule di mammifero. Tuttavia, è relativamente difficile osservare aggregati nelle cellule neuronali quando il volume del citoplasma è piccola attorno ai nuclei e neuriti.
Il protocollo viene utilizzato per mostrare l'associazione tra KIF5B ei suoi carichi oltre al in vivo lievito doppio ibrido e saggi di pull-down biochimici 13-16. Tutti questi test determinare l'associazione in diverse condizioni fisiche e loro risultati possono completarsi a vicenda. Inoltre, il vantaggio di questo test fluorescenza nel altri test è che può mostrare la regolazione della localizzazione subcellulare dei carichi da KIF5B (Figure 1 e 2).
Il protocollo non è limitato a KIF5B e può essere utilizzato per identificare carichi di altre proteine motrici, come alcuni altri motori kinesin 3 e alcuni dei motori miosina 23,24 utilizzati nel trasporto intracellulare di carichi 3. Queste proteine motore contengono anche i domini a motore per il movimento lungo i microtubuli per motori kinesin e microfilamenti per motori miosina, e segmenti a doppia spiralizzazione per oligomerizzazione. La maggior parte di essi sotto forma omodimeri 3,23. Pertanto, una strategia simile può essere applicata a loro creando loro senza motore mutanti dominanti negativi per identificare i loro carichi.
Divulgazioni
pubblicazione accesso libero e la produzione di questo articolo è pagato da Roche.
Riconoscimenti
Roche's Publication Grant covered the Free Access publication and production of this article. The author also thanks E. Premkumar Reddy, Richard V. Mettus, Stephen C. Cosenza, Sau Ying Yip and Sol D. Gloria for their support and critical reading of the manuscript.
Materiali
| Name | Company | Catalog Number | Comments |
| absolute ethanol (200 proof) | Fisher Scientific | BP2818 | |
| DAPI | Sigma-Aldrich | D9542 | |
| Hoechst 33342 | Sigma-Aldrich | B2261 | |
| Opti-MEM-I (transfection medium) | Life Technologies | 51985 | |
| ProLong Diamond Antifade Mountant | Life Technologies | P36961 | |
| formaldehyde, para | Fisher Scientific | O4042-500 | |
| Triton-X100 | Fisher Scientific | BP151-500 | |
| X-tremeGENE 9 DNA Transfection Reagent | Roche | 6365787001 | |
| Taq DNA polymerase | Life Technologies | 10342020 | |
| PCR Grade Nucleotide Mix (dNTP Mix) | Roche | 12111424 | |
| Microscope cover glass | Fisher Scientific | 12-541A | |
| GeneRuler 1 kb Plus DNA ladder | Life Technologies | SM1333 | |
| PureLink Quick Gel Extraction kit | Life Technologies | K210012 | |
| BamHI | New England Biolab | R0136 | |
| SalI | New England Biolab | R0138 | |
| T4 DNA ligase | New England Biolab | M0202T | |
| Ethidium bromide | Thermo Scientific | 17898 | |
| DMEM | Life Technologies | 11995-065 | |
| c-MYC rabbit antibody | Cell Signaling | 5606 | |
| p53 mouse antibody | Santa Cruz | sc-126 | |
| Alexa Fluor 488-conjugated anti-rabbit IgG antibody | Life Technologies | A11008 | |
| Alexa Fluor 488-conjugated anti-mouse IgG antibody | Life Technologies | A11059 | |
| fluorescent microscope | Olympus IX71_Fluoview | ||
| computer software for imaging | cellSens |
Riferimenti
- Hirokawa, N., Niwa, S., Tanaka, Y. Molecular motors in neurons: transport mechanisms and roles in brain function, development, and disease. Neuron. 68, 610-638 (2010).
- Yu, Y., Feng, Y. M. The role of kinesin family proteins in tumorigenesis and progression: potential biomarkers and molecular targets for cancer therapy. Cancer. 116, 5150-5160 (2010).
- Verhey, K. J., Hammond, J. W. Traffic control: regulation of kinesin motors. Nat Rev Mol Cell Biol. 10, 765-777 (2009).
- Hirokawa, N., Noda, Y., Tanaka, Y., Niwa, S. Kinesin superfamily motor proteins and intracellular transport. Nat Rev Mol Cell Biol. 10, 682-696 (2009).
- Lee, C. M. Transport of c-MYC by Kinesin-1 for proteasomal degradation in the cytoplasm. Biochim Biophys Acta. 1843, 2027-2036 (2014).
- Bittins, C. M., Eichler, T. W., Hammer, J. A., Gerdes, H. H. Dominant-negative myosin Va impairs retrograde but not anterograde axonal transport of large dense core vesicles. Cell Mol Neurobiol. 30, 369-379 (2010).
- Kimura, T., Watanabe, H., Iwamatsu, A., Kaibuchi, K. Tubulin and CRMP-2 complex is transported via Kinesin-1. J Neurochem. 93, 1371-1382 (2005).
- Lindsay, A. J., McCaffrey, M. W. Myosin Va is required for the transport of fragile X mental retardation protein (FMRP) granules. Biol Cell. 106, 57-71 (2014).
- Rivera, J., Chu, P. J., Lewis, T. L., Arnold, D. B. The role of Kif5B in axonal localization of Kv1 K(+) channels. Eur J Neurosci. 25, 136-146 (2007).
- Roland, J. T., Kenworthy, A. K., Peranen, J., Caplan, S., Goldenring, J. R. Myosin Vb interacts with Rab8a on a tubular network containing EHD1 and EHD3. Mol Biol Cell. 18, 2828-2837 (2007).
- Uchida, A., Alami, N. H., Brown, A. Tight functional coupling of kinesin-1A and dynein motors in the bidirectional transport of neurofilaments. Mol Biol Cell. 20, 4997-5006 (2009).
- Zadeh, A. D., et al. Kif5b is an essential forward trafficking motor for the Kv1.5 cardiac potassium channel. J Physiol. 587, 4565-4574 (2009).
- Su, Y. Y., et al. KIF5B promotes the forward transport and axonal function of the voltage-gated sodium channel Nav1.8. J Neurosci. 33, 17884-17896 (2013).
- Lalioti, V. S., Vergarajauregui, S., Tsuchiya, Y., Hernandez-Tiedra, S., Sandoval, I. V. Daxx functions as a scaffold of a protein assembly constituted by GLUT4, JNK1 and KIF5B. . J Cell Physiol. 218, 416-426 (2009).
- Cho, K. I., et al. Association of the kinesin-binding domain of RanBP2 to KIF5B and KIF5C determines mitochondria localization and function. Traffic. 8, 1722-1735 (2007).
- Diefenbach, R. J., Diefenbach, E., Douglas, M. W., Cunningham, A. L. The ribosome receptor, p180, interacts with kinesin heavy chain, KIF5B. Biochem Biophys Res Commun. 319, 987-992 (2004).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22, 1567-1572 (2004).
- Waters, J. C. Live-cell fluorescence imaging. Methods Cell Biol. 114, 125-150 (2013).
- Sanderson, M. J., Smith, I., Parker, I., Bootman, M. D. Fluorescence microscopy. Cold Spring Harb Protoc. 2014, (2014).
- Hopkins, S. C., Vale, R. D., Kuntz, I. D. Inhibitors of kinesin activity from structure-based computer screening. Biochemistry. 39, 2805-2814 (2000).
- Zhang, Y., Xiong, Y. Control of p53 ubiquitination and nuclear export by MDM2 and ARF. Cell Growth Differ. 12, 175-186 (2001).
- Michael, D., Oren, M. The p53-Mdm2 module and the ubiquitin system. Semin Cancer Biol. 13, 49-58 (2003).
- Kneussel, M., Wagner, W. Myosin motors at neuronal synapses: drivers of membrane transport and actin dynamics. Nat Rev Neurosci. 14, 233-247 (2013).
- Maravillas-Montero, J. L., Santos-Argumedo, L. The myosin family: unconventional roles of actin-dependent molecular motors in immune cells. J Leukoc Biol. 91, 35-46 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon