Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La fabricación de cristal invertida coloidal poli (etilenglicol) Andamios: Una célula Plataforma de cultivo tridimensional para ingeniería de tejidos de hígado
En este artículo
Resumen
This manuscript presents a detailed protocol for the fabrication of an emerging three-dimensional hepatocyte culture platform, the inverted colloidal crystal scaffold, and the concomitant techniques to assess hepatocyte behavior. The size-controllable pores, interconnectivity and ability to conjugate extracellular matrix proteins to the poly(ethylene glycol) (PEG) scaffold enhance Huh-7.5 cell performance.
Resumen
La capacidad para mantener la función de los hepatocitos in vitro, para el propósito de probar la citotoxicidad xenobióticos ', el estudio de la infección por virus y el desarrollo de fármacos dirigidos en el hígado, requiere una plataforma en la que las células reciben señales bioquímicas y mecánicas adecuadas. sistemas recientes de ingeniería de tejidos de hígado han empleado andamios tridimensionales (3D) compuestas de hidrogeles sintéticos o naturales, dado su alta retención de agua y su capacidad de proporcionar los estímulos mecánicos necesarios por las células. Ha habido un creciente interés en el andamio invertida cristal coloidal (ICC), un desarrollo reciente, que permite a la organización espacial alta, homotipica e interacción celular heterotıpica, así como la matriz de interacción célula extracelular (ECM). En este documento, se describe un protocolo para fabricar el andamio ICC utilizando poli diacrilato (etilenglicol) (PEGDA) y el método de lixiviación de partículas. En pocas palabras, una celosía está hecho de partículas de microesferas, después de lo cual un pre-de poSe añade solución r, adecuadamente polimeriza, y las partículas se retiran, o lixiviado, usando un disolvente orgánico (por ejemplo, tetrahidrofurano). La disolución de los resultados de celosía en un andamio altamente poroso con tamaños y interconectividades que permiten los medios de comunicación para llegar a las células más fácilmente de poros controlados. Esta estructura única permite a área de superficie alta para que las células se adhieren a, así como fácil la comunicación entre los poros, y la capacidad de recubrir el andamio ICC PEGDA con proteínas también muestra un efecto marcado en el rendimiento de la célula. Se analiza la morfología del esqueleto, así como el hepatocarcinoma celular (Huh-7.5) el comportamiento en términos de viabilidad y la función de explorar el efecto de la estructura de la CPI y revestimientos de ECM. En general, este documento proporciona un protocolo detallado de un andamio emergente que tiene amplias aplicaciones en la ingeniería de tejidos, especialmente la ingeniería de tejidos del hígado.
Introducción
El hígado es un órgano altamente vascularizado con una multitud de funciones, incluyendo la desintoxicación de la sangre, el metabolismo de xenobióticos, y la producción de proteínas séricas. El tejido del hígado tiene una microestructura tridimensional compleja (3D), que consta de múltiples tipos de células, canalículos biliares, sinusoides, y zonas de diferente composición biomatriz y diferentes concentraciones de oxígeno. Dada esta estructura elaborada, ha sido difícil crear un modelo de hígado adecuado in vitro 1. Sin embargo, hay una creciente demanda de funcional en modelos in vitro de alojamiento hepatocitos humanos como plataformas para la toxicidad del fármaco de prueba 2 y el estudio de enfermedades asociadas con el hígado 3.
Plataformas de ingeniería de tejido hepático actual han simplificado la complejidad del hígado mediante el aislamiento de uno, o concentrarse en unos pocos, de los parámetros del hígado, es decir, co-cultivo de células 4, la composición bioquímica de la zonal microambientes 5, la dinámica de flujo de 6,7 y la configuración de la biomatriz 8. La configuración de la biomatriz se puede dividir en parámetros tales como materiales de andamiaje, la composición de proteínas de la matriz extracelular (ECM), la rigidez de la matriz, así como el diseño y la estructura del andamio. Ha habido un aumento en los estudios de ingeniería de tejidos utilizando hidrogeles sintéticos, especialmente poli (etilenglicol) (PEG) hidrogeles 9, dada la capacidad de sintonizar propiedades mecánicas, bioactividad, y la velocidad de degradación del hidrogel. En cuanto a la investigación relacionada con el hígado, se aplicó el hidrogel biocompatible para estudiar la infección por virus de la enfermedad hepática 3. Como diseño de la plataforma de hepatocitos, numerosos estudios han utilizado cultivos sándwich de hepatocitos 10,11 y encapsulación de células dentro de un hidrogel 12,13 para proporcionar el entorno 3D y célula-ECM y la interacción célula-célula que son esenciales para imitar en el microambiente vivo. Howeembargo, estas plataformas no poseen un alto grado de control y organización espacial, que conduce a propiedades no uniformes a través del andamio 14.
El cristal coloidal invertida (CPI) 14 andamio es un andamio 3D altamente organizada para el cultivo celular que se introdujo por primera vez en la década de 2000. estructura única del andamio se puede atribuir al proceso de fabricación sencillo que utiliza un cristal coloidal, un enrejado ordenado de partículas coloidales de diámetro variable. En pocas palabras, para resumir el proceso, las partículas están bien ordenados y recocidas utilizando calor para formar un enrejado. La lixiviación de esta celosía, por un disolvente orgánico, en un polimerizados resultados de hidrogel en cavidades esféricas hexagonal empaquetados 15 con alta área superficial. Este andamio altamente ordenada se ha hecho previamente con ambos materiales sintéticos y naturales, incluyendo, pero no limitado a, poli (acrilamida) 16-21, poli (láctico-co-ácido glicólico) 15,22-30, Poli (etileno glicol) 31,32, poli (metacrilato de 2-hidroxietilo) 21,33-35, y quitosano 36-39. Andamios ICC hechos de materiales no-incrustantes tienden a promover esferoides celulares dentro de las cavidades 14,23,40. Múltiples tipos de células se ha demostrado que la proliferación de éxito, diferenciar y función dentro de esta configuración, incluyendo condrocitos 41, células estromales de médula ósea 42, y células madre 43,44. En cuanto a los hepatocitos, se han realizado estudios con andamios ICC hechos de Na 2 SiO 3 y poli (acrilamida), pero no PEG. Con las estrategias bioconjugation simples (es decir, a través de acoplamiento de aminas EDC / NHS), andamios a base de PEG ECM proteínas conjugadas se pueden fabricar, que puede resultar más sitios de unión de células a ser más in vivo como el medio ambiente y mejorar la función hepática.
En este manuscrito y el vídeo asociado, detallamos la fabricación del andamio CPIutilizando poli diacrilato de hidrogel (etilenglicol) (PEGDA) y una celosía de poliestireno de microesferas, optimizado para hepatocarcinoma (Huh-7.5) cultura. Se demuestra las diferencias entre los andamios ICC en general no adhesivos desnudos PEGDA y la PEGDA andamio ICC revestido con colágeno en términos de topología de andamio y rendimiento de la célula. La viabilidad celular y la función se miden cualitativa y cuantitativamente para evaluar el comportamiento de células Huh-7.5.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. CPI Andamios Fabricación (Figura 1)
- Preparar el poliestireno (PS), cercados (diámetro = 6 mm; 8-13 capas de perlas).
- Para preparar el molde, cortar las puntas fuera de 0,2 ml tubos de microcentrífuga hervir a prueba al nivel de 40 l. Adherir la parte superior de los tubos de corte de 24 x 60 mm 2 de microscopio de vidrio se desliza la cubierta con pegamento a prueba de agua.
- Poner las esferas de PS (diámetro = 140 mM) contenidos dentro de una suspensión de agua en un vial de 20 ml, pipetear cuidadosamente la suspensión de agua, y añadir 18 ml de 70% de solución de etanol en el vial. Ponga la solución esfera en un baño de ultrasonidos para aflojar las esferas agregados. Repita este paso de lavado varias veces con el fin de eliminar los componentes de agua y solubles en agua por completo.
- Pipetear 100 l de etanol en los moldes.
- Cortar la parte superior de una punta de micropipeta 200 l por 4 mm. Pipetear 25 l de las esferas en el molde dos veces utilizando la micropipeta 200 l para lograr unavolumen total de 50 l en cada molde.
- Colocar los moldes en un agitador oscilante durante la noche a 120 rpm.
- Verificar la disposición de las esferas en cada molde bajo un microscopio óptico. Si las esferas no están clasificadas hexagonal, se añaden 50 l de 70% de etanol y agitar manualmente en la dirección del eje longitudinal y lateral para corregir la disposición.
- Se deja evaporar el etanol a temperatura ambiente (TA) durante dos noches. Colocar el molde y complejo de talón en un horno de 130 ° C durante 6 horas para recocer las perlas de PS.
- Preparación de andamios PEGDA desnudas y recubiertas de ECM.
- Sintetizar macrómeros PEGDA utilizando protocolos establecidos para acrilación 45,46 macrómeros de PEG lineales (M w = 4,6 kDa).
- Preparar 50% (w / v) de solución PEGDA en agua desionizada (DI) y permitir que el macrómero se disuelva adecuadamente por centrifugación a 4.713 xg hasta que se disuelva completamente.
- Para ECM conjugado andamios ICC, disolver un adicional10% (w / v) acriloilo-PEG-NHS (M w = 3,4 kDa) en la solución PEGDA 50%.
- Preparar un 20% (w / v) solución madre de 2-hidroxi-4 '- (2-hidroxietoxi) -2-metilpropiofenona (PI) en 70% de etanol.
- Añadir 50 l de 20% (w / v) de solución de stock PI por 1 ml de 50% (w / v) de PEGDA. Ajustar la cantidad necesaria de solución de PI basado en el peso molecular de PEGDA.
- Vortex la mezcla del tubo de centrífuga durante 1 min para alcanzar una solución homogénea.
- Pelar los moldes de la lámina de vidrio (de la etapa 1.1.7), quitar el pegamento de los moldes, empuje las celosías a cabo con cuidado usando una espátula y colocar cada uno de ellos en un tubo de 1,5 ml. De pipeta 300 l de la solución PEGDA y centrifugar a 845 xg durante 5 min para permitir PEGDA adecuada solución de infiltración en la retícula.
- Retire la celosía del tubo con unas pinzas y secar cuidadosamente el exceso de solución PEGDA seca en los guantes. Coloque la red en un vaso cubierto con película de parafina con el CIR planacular superficie hacia arriba.
- Exponer el andamio infiltrado solución PEGDA a 365 nm ultravioleta (UV) (10,84 mW / cm2) durante 5 min usando una lámpara de luz ultravioleta.
- Colocar redes cristalinas PEGDA-polimerizados en nuevos viales (alrededor de 10 celosías por vial) y se añaden 20 ml de tetrahidrofurano (THF). Agitar los viales en un agitador orbital a 300 rpm. Cambiar THF al menos 3 veces con un intervalo de 1 a 2 hr.
Nota: No eliminar el THF por completo cuando se cambia el THF con el fin de evitar que las burbujas entren en los andamios, que a su vez puede causar la eliminación incompleta del PS. Deje suficiente solución para cubrir las celosías y añadir nueva THF.
Precaución: THF es tóxico. Use guantes, una bata de laboratorio y gafas. Evitar la inhalación operando bajo la campana de humos. - Compruebe si las esferas PS se disuelven poniendo el agua en la solución de THF usado y observando el color de la solución. Repita el paso 1.2.9 Si las esferas de EP no se disuelven adecuadamente.
Nota: El color de la solución cambia ablanco si hay esferas de PS restantes.
- Limpiar los andamios en la cabina de bioseguridad (BSC).
- Para esterilizar los andamios, preparar un tubo de centrífuga de 50 ml con 2 ml de 70% de etanol por andamio y colocar los andamios en el tubo usando una espátula. Permitir a los andamios en remojo en etanol durante 1 hora. A partir de este paso adelante, cabo todos los procedimientos en el BSC.
- Con cuidado, vierta el etanol y reemplazarlo con buffer fosfato salino (PBS) (2 ml por andamios) y se centrifuga a 524 g durante 3 min para eliminar las burbujas. Guarde en el refrigerador y cambiar el PBS unas cuantas veces con un intervalo de 1-2 horas.
- Para colágeno tipo I-andamios recubiertos, preparar otro tubo de centrífuga de 50 ml que contiene colágeno tipo 1 solución madre (1 ml por andamios), la transferencia de los andamios esterilizados a este tubo con una espátula, y se centrifuga a 524 g durante 3 min. Agitar los andamios en 400 rpm en un agitador orbital durante 30 min y mantener el tubo en el equipo fo durante la noche.
- Lavar los andamios con PBS dos veces antes de usar sumergiendo los andamios en PBS fresco y luego aspirar el PBS.
Nota: Otras proteínas de ECM también se pueden utilizar en vez de colágeno de tipo I porque la química NHS requiere un grupo amina para formar el enlace (Figura 2).
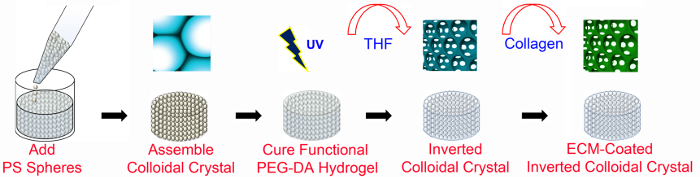
Figura 1. Visión general de la fabricación de la CPI. Andamios ICC basados en PEG se fabrican utilizando técnicas de microfabricación con y sin ECM-funcionalización. Andamios ICC ECM recubierto requieren PEG-NHS, así como PEGDA (como se detalla en la Figura 2). La red PS tiene un diámetro de 6 mm y una altura de 8-13 capas de perlas. PS, poliestireno; PEGDA, poli (etilenglicol) diacrilato; UV, ultravioleta; THF, tetrahidrofurano; ECM, matriz extracelular. Esta cifra ha sido modificada y usada con permiso de conexión inalámbricaLey 47. Haga clic aquí para ver una versión más grande de esta figura.
2. Estructura de la CPI Caracterización
- Para analizar la estructura de ICC con o sin proteínas conjugadas, utilizar la microscopía electrónica de barrido (SEM) 47.
- Fijar los andamios con 4% de paraformaldehído (PFA), en serie deshidratar en 25, 50, 75, 95 y 100% soluciones de etanol, y almacenar a -80 ° C hasta que el etanol se evapora por completo.
- Las muestras secas en un liofilizador durante 48 horas.
- Colocar la muestra en el soporte para la muestra usando cinta de carbono y colocar en un dispositivo de recubrimiento por pulverización catódica.
- Después de pasar la aspiradora automática, cubrir con una película de Pt de 10 nm de espesor mediante pulverización durante 60 segundos a 20 mA.
- Andamios ICC utilizando la imagen SEM a un voltaje de 5 kV (Figura 3A, Figura 4A).
- Para medir el poro ydiámetro interconexión de cavidades, analizar micrografías SEM utilizando software de análisis de imágenes 48 (por ejemplo, ImageJ; Figura 3B, C).
- Para visualizar el colágeno conjugado con el andamio sin células, etiqueta fluorescente el colágeno utilizando anticuerpos (1: 100) contra el colágeno de tipo I y la imagen con láser confocal de barrido microscopía 47 (CLSM; Figura 4B).
3. Huh-7.5 Siembra y Cultivo Celular
- Cultura Huh-7.5 células a una densidad de siembra de 2-2,5 x 10 6 células / ml en placas de cultivo celular de 100 mm con 10 ml de Eagle modificado (DMEM) suplementado con suero bovino fetal al 10% (FBS) y 100 U / ml de Dulbecco penicilina-estreptomicina (medio de crecimiento) a 37 ° C y 5% de CO 2. Cambiar los medios cada tres días en el BSC hasta que hayan alcanzado el 75-80% de confluencia.
- Preparar los andamios para la siembra de células en el BSC.
- Coloque con cuidado la Scaffedad en una placa de 24 pocillos con la superficie plana hacia arriba.
- Para lavar el andamio, pipeta 2 ml de PBS a cada pocillo que contiene un andamio. Aspirar el PBS y pipeta 2 ml de PBS en cada pocillo.
- Aspirar el PBS y pipeta 2 ml de medio de crecimiento (véase el paso 3.1) y dejar actuar durante 30 minutos. Aspirar los medios de comunicación y permitir que el andamio se seque durante 1 hora.
- Separar confluentes Huh-7.5 (células del paso 3.1) de la placa de cultivo en el BSC utilizando el método de digestión con tripsina.
- Aspirar los medios de placa, añaden 4 ml de PBS para lavar las células adherentes y luego aspiran PBS.
- Pipeta de 0,75-1 ml 0,25% de tripsina y colocar en una incubadora a 37 ° C, 5% de CO 2 durante 3 min.
- Retire la placa de la incubadora y la pipeta 5 ml de medio para detener la reacción tripsina. medios pipeta, células desprendidas, y la mezcla de tripsina en un tubo de 15 ml.
- Se centrifuga a 524 g durante 3 min, eliminar el sobrenadante y resuspender el precipitado en 5 ml de medio.
- Contar las células usando un hemocitómetro y calcular el volumen de la suspensión de células que contiene células en el número de destino, N 0, por 25 l (para el experimento estándar, N 0 es 1 x 10 6 células).
Volumen de suspensión de células = (número de destino de las células) / (concentración de la suspensión celular) - Pipetear lentamente 25 l de suspensión celular (que contiene N 0 células) directamente en la parte superior de cada andamio (del paso 3.1.4). Coloque la placa de 24 pocillos en la incubadora.
- Después de 12 h, la transferencia de los andamios con cuidado con el uso de una espátula en una nueva placa de 24 pocillos y la pipeta 2 ml de medio en cada pocillo. Coloque la placa de 24 pocillos en la incubadora.
- Cambiar el soporte de cada 3 días o según el momento en los medios de comunicación se recoge para el análisis de secreción de proteínas (véase el paso 5.1).
4. Viabilidad Celular
- Para analizar cualitativamente la viabilidad celular, utilice las unidades de fluorescentes vivas / muertas de tinción para teñir las células y la imagen usando CLSM.
- Siguiendo instrucciones del kit, preparar una solución con 4 M calceína AM y 8 M de etidio homodímero-1 en los medios de comunicación (véase el paso 3.1.3).
Nota: optimizar en función del número de células. Utilizar el doble de la cantidad de reactivo si las células proliferan y doble en número (alrededor de 2 millones). - En el BSC, los medios de aspirado en cada pocillo con un andamio (paso 3.7) y la pipeta 500 l de la solución preparada. Incubar las muestras en un incubador a 37 ° durante 1 hora.
- Se cubren las placas en papel de aluminio para proteger las muestras de la luz cuando se retira la placa de la incubadora. muestras de imagen utilizando CLSM49.
- Siguiendo instrucciones del kit, preparar una solución con 4 M calceína AM y 8 M de etidio homodímero-1 en los medios de comunicación (véase el paso 3.1.3).
- Para evaluar cuantitativamente la viabilidad celular, medir la actividad enzimática (en células vivas) utilizando ensayos colorimétricos 50 (es decir, 2- (2-metoxi-4-nitrofenil) -3- (4-nitrofenil) -5- (2,4-disulfofenil) 2H-tetrazolio (reactivo monosódico-sal)).
- Crear una curva estándar para la plataforma de ICC (es decir, un gráfico de absorbancia v (OD)ersus determinado número de células).
Nota: la siembra de células no es 100% eficiente en comparación con otras plataformas, ya que las células pueden pasar a través de las cavidades de los andamios.- Determinar los números de la siembra de células, N 0 que serán usados para hacer la curva estándar.
Nota: Elija un intervalo que incluye el número de células que se estiman en el experimento. Por ejemplo, si el número inicial de células es de 5 x 10 5 células y hay un ~ 3 veces de incremento estimado por el último día del experimento, elegir 2,5 x 10 5, 5 x 10 5, 1 x 10 6, y 2 x 10 6 células como N 0. - Realizar la siembra de células en los andamios de la CCI (una N 0 por andamio con un volumen de siembra de 25 l) tal como se describe en los pasos 3.1-3.6.
- Después de 6 horas, la transferencia de los andamios a otra placa de 24 pocillos también. Seleccionar un tiempo que permite la adhesión celular, pero no la proliferación celular.
- Realizar el recuento de células en el andamio CPI transferido.
- Diluir 10 veces reactivo monosódico-sal solución (MSR) a 1x con medios de comunicación en el BSC y la pipeta 500 l solución 1x MSR en cada pocillo con un andamio. Incubar la placa de 24 pocillos a 37 ° C durante 1 hora.
- De cada pocillo, la transferencia de 100 l en una placa de 96 pocillos también. Como en blanco, la pipeta 100 l de solución de reactivo 1x monosódico-sal fresca en distintos pocillos en la placa de 96 pocillos. eliminar manualmente cualquier burbuja presente utilizando una pipeta de punta seca y cubren la placa de 96 pocillos en papel de aluminio para protegerlo de la luz.
- Medida DO a λ = 450 nm usando un espectrofotómetro de lectura. Reste la DO en blanco de otros valores para encontrar el diámetro exterior precisa 51.
- Contar el número de células (N L) restante en el pozo después de transferir el andamio (etapa 4.2.1.3) utilizando un hemocitómetro.
Nota: Utilice 300 l de tripsina para trypsinize las células. - Calcular el número real de la celda, N A.
= realesinicial - quedan en el pozo
N A = N 0 -N L - Hacer curva estándar trazando OD obtenido en el Paso 4.2.1.4.3 frente al número real de la celda (N A) y usar esto para estimar el número de células en los experimentos.
Nota: Para realizar una nueva curva estándar si cualquier parámetro ICC (es decir, el tamaño del generador de poros, dimensiones del andamio, proteínas ECM, etc.) se cambian.
- Determinar los números de la siembra de células, N 0 que serán usados para hacer la curva estándar.
- Crear una curva estándar para la plataforma de ICC (es decir, un gráfico de absorbancia v (OD)ersus determinado número de células).
5. Función de la célula
- Analizar la secreción de proteínas por las células Huh-7.5 (es decir, albúmina, urea) a partir de los medios de comunicación recogidos (de la etapa 3.7) por la enzima ensayo inmunoenzimático (ELISA) 52.
Nota: Diluir los medios de comunicación, en función del número de células sembradas y la cantidad de medios de comunicación recogidos. Para 5 x 10 5 células sembradas en la CPI, use un ~ proporción de 1: 25, antes de introducirlo a los pocillos anticuerpos prerrevestida. - Para analizar cualitativamente la función celular, proteínas intracelulares específicos inmunotinción(Es decir, albúmina), enzimas (es decir, CYP450), componentes estructurales de manchas (es decir, actina celular), así como los núcleos y de imagen utilizando CLSM 49.
- Aspirar los medios de comunicación (desde el paso 3.7) y la pipeta 2 ml de PBS para lavar los andamios ICC-celulares cargados.
- Pipetear 1 ml de PFA al 4% y se incuba durante 5 min a temperatura ambiente durante la fijación.
- Lavar 3 veces con 2 ml de PBS.
- Permeabilizar las membranas mediante la incubación de los andamios en 1 ml de 0,1% de 4- (1,1,3,3-tetrametil-butil) fenil-glicol de polietileno (agente tensioactivo) durante 30 min.
- Lavar 3 veces con 2 ml de PBS para eliminar las proteínas con fugas.
- Pipetear 500 l 1% de albúmina de suero bovino (BSA) y se incuba a RT durante 1 h para bloquear la unión no específica.
- Preparar el anticuerpo primario diluido (es decir, albúmina, CYP450) solución.
- Pipetear 500 l de solución de BSA 1% en un tubo de 15 ml y añadir 4,5 ml de 0,1% de solución de agente tensioactivo para preparar una solución total de BSA 5 ml de 0,1%.
- Pipetear 98 l de la solución de BSA 0,1% en un tubo de microcentrífuga de 200 l y 2 l de anticuerpo primario para producir un 01:50 (anticuerpo primario: 0,1% de BSA) solución de anticuerpo primario.
- De pipeta 40 l de la solución de anticuerpo primario en el andamio y cubrir el sustrato con película de parafina. Envolver la placa de 24 pocillos con papel de aluminio y guardar el plato a 4 ° C durante la noche.
- Lavar 3 veces con 2 ml de PBS y agitar suavemente la placa en el medio de lavado.
- Preparar anticuerpo secundario biotinilado diluido (es decir, anticuerpo anti-ratón) solución madre.
- Pipetear 198 l 0,1% de solución de BSA y 2 l segundo anticuerpo para producir un 1: 100 (anticuerpo secundario: 0,1% de BSA) solución de anticuerpo secundario.
- Preparar una solución madre de 0,1% de rodamina o faloidina marcada con fluoresceína (para teñir los filamentos de actina celular) en la solución de BSA 0,1%.
- Pipeta de 25 l de cada solución en un tubo y mezclar wana.
- Pipeta de 50 l de la solución de anticuerpo secundario de valores en el andamio. Cubrir el andamio con película de parafina, envolver el plato con papel de aluminio y se almacena a temperatura ambiente durante 2 horas.
- Lavar 3 veces con 2 ml de PBS.
- Pipetear 200 l de 0,2% de 4'-6-diamino-2-fenilindol (DAPI; una mancha núcleo) solución en el andamio y mantener a temperatura ambiente durante 2-3 minutos. Cubrir la placa con papel de aluminio.
- Lavar 2 veces con 2 ml de PBS.
- El uso de un cuentagotas, colocar una gota de medio de montaje sobre el sustrato.
- Con cuidado, poner el andamio sobre un portaobjetos de vidrio y la imagen utilizando CLSM 47.
- Evaluar la expresión génica por reacción en cadena de la polimerasa en tiempo real (qPCR). Utilice kits estándar para PCR de la transcriptasa inversa (RT-PCR) 53 y qPCR 54 según las instrucciones del fabricante. Extraer el ARN de las células como se describe a continuación.
- Coloque el andamio (del paso 3,7) en un tubo de microcentrífuga de 1,5 ml.
- Pipeta de 1 ml de una solución de extracción de ARN en el tubo y se mantiene en un baño de ultrasonidos durante 5 minutos a temperatura ambiente.
- Pipetear 200 l de cloroformo en cada tubo de microcentrífuga y agitar vigorosamente el tubo en la mano durante 15-20 seg. Mantener los tubos a TA durante ~ 3 min hasta que las fases se separan.
- Centrifugar la muestra a 13.000 xg, 4 ° C durante 15 min y retirar los tubos cuidadosamente para que las fases no se mezclan.
- pipetear con cuidado 500-600 l de la fase acuosa superior del primer tubo en un segundo tubo de microcentrífuga.
- Añadir un volumen equivalente (500-600 l) de isopropanol a este segundo tubo.
- Invierta el tubo 3-5 veces y dejar el tubo de pie a temperatura ambiente durante 10 min.
- Centrifugar la muestra a 13.000 xg, 4 ° C durante 15 min.
- Si una pastilla no es visible en la parte inferior del tubo, centrifugar de nuevo durante 5 min.
Nota: Si todavía no hay pellet visible, la cantidad de ARN puede ser insuficiente.
- Si una pastilla no es visible en la parte inferior del tubo, centrifugar de nuevo durante 5 min.
- yotubos nvert con la tapa abierta a desechar el sobrenadante y la pipeta en 1 ml etanol al 70% diluido en agua DEPC en el tubo.
- Ligeramente vórtice el tubo de manera que el pellet se separa de la pared del tubo y luego dejar que el aire del tubo seco.
- Añadir 50 l de agua DPEC para volver a suspender el sedimento.
- Mantenga pipeteado hasta que se disuelva de pellets.
- Mantener durante 10 minutos a 55 ° C con el fin de desnaturalizar el ARN de doble cadena en el ARN de una sola hebra.
- Ligeramente tambor dedos en la parte inferior del tubo y centrifugar brevemente los tubos (7.500 xg, 4 min, 4 ° C).
- Mantener los tubos en hielo hasta la realización de la transcriptasa inversa 55 y PCR en tiempo real como se describe en 56.
Access restricted. Please log in or start a trial to view this content.
Resultados
Los resultados representativos para la caracterización estructural del andamio CPI y la comparación de la eficacia de cada condición del andamio de la CPI en el cultivo de hepatocitos se muestran y explican a continuación. Las condiciones de andamio ICC utilizados en estos resultados son recubrimientos de colágeno de 0 g / ml (desnudo), 20 mg / ml (de colágeno 20), 200 mg / ml (de colágeno 200), y 400 mg / ml (de colágeno 400) y la inicial número de células de siembra-7,5 eh es...
Access restricted. Please log in or start a trial to view this content.
Discusión
Andamios de ingeniería de tejidos están evolucionando rápidamente para proporcionar todas las señales físicas y bioquímicas necesarias para regenerar, mantener o reparar los tejidos para la aplicación de la sustitución de órganos, el estudio de la enfermedad, el desarrollo de fármacos, y muchos otros 57. En la ingeniería de tejidos de hígado, los hepatocitos primarios humanos pierden rápidamente sus funciones metabólicas una vez aislados del cuerpo, creando una gran necesidad de andamios de inge...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
The authors have no competing financial interests to disclose.
Agradecimientos
Los autores desean agradecer el apoyo de una beca de la Fundación de Investigación Nacional (NRF -NRFF2011-01) y el Programa de Investigación Competitiva (NRF-CRP10-2012-07).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2 ml PCR tube | Axygen Scientific | PCR-02D-C | Boil-proof |
| Gorilla Glue | Gorilla Glue, Inc. | Depends on vendor. This was purchased from a local store. | |
| Glass slides | VWR | 631-1575 | Dimensions: 24×60 mm2 |
| Polystyrene spheres | Fisher Scientific | TSS#4314A | Diameter = 140 µm; 3x104 particles per milliliter and 1.4% size distribution |
| Ethanol | Merck | 1.00983.1011 | absolute for analysis EMSURE; Dilute to 70% with Milli-Q water |
| Ultrasonic Bath | Elma | S10H | Equiment |
| Furnace | Nabertherm | N7/H | Equipment |
| 200 µl pipette tip | Axygen Scientific | T-210-Y-R-S | |
| Rocking shaker | VWR | 444-0142 | |
| Polyethylene Glycol (PEG) | Merck | 1.09727.0100 | Mw= 4 kDa; acrylation of PEG monomers and purification of the resulting precipitate produces a PEGDA macromer with Mw = 4.6 kDa |
| Centrifuge | Beckman Coulter | 392932 | Equipment |
| Acrylate-Poly(Ethylene Glycol) - Succinimidyl Valerate | Laysan Bio | ACRL-PEG-SVA-3400-1g | Mw = 3.4 kDa |
| 2-hydroxy-4'-(2-hydroxyethoxy)-2-methylpropiophenone | Sigma Aldrich | 410896 | |
| Vortex | VWR | 58816-123 | Equipment |
| Microcentrifuge | Eppendorf | 5404 000.413 | |
| Paraffin Film | Parafilm M | #PM996 | Kept at 9" with allows intensity of 10.84 mW/cm2 |
| Bluewave 200 UV spotlight | Blaze Technology | 120008, 122300 | |
| Tetrahydrofuran (THF) | Merck | 107025 | |
| Orbital shaker | Heidolph | 543-123120-00-5 | |
| Collagen Type I | Sigma Aldrich | C3867-1VL | From rat. 1x, w/o CaCl2 & MgCl2; pH = 7.2 |
| Phosphate Buffered Saline (PBS) | Gibco | 20012-027 | 16% W/V AQ. 10 x 10 ml |
| Paraformaldehyde | VWR | 43368.9M | Equipment |
| Freezone 4.5 freeze drier | Labconco | 7750020 | Equipment |
| Sputter coater | Jeol Ltd. | JFC-1600 | Equipment |
| Scanning Electron Microscope | Jeol Ltd. | JSM 5310 | |
| Anti-mouse primary antibodies against Collagen type I | Abcam | ab6308 | |
| Anti-mouse secondary antibody conjugated with Alexa Fluor 488 | Life Technologies | A21121 | |
| Plate, Tissue Culture 24 Well, Flat Bottom (Nunclon) | Bio-Rev PTE LTD | 3820-024 | |
| Dulbecco's Modified Eagle's Medium(DMEM) 2.5 g/L Glucose w/ L-Gln | Lonza | 12-604F | |
| Fetal Bovine Serum (FBS) | Gibco | A15-151 | |
| Penicillin-Streptomycin (P/S) | Life Tchnologies | 15140-122 E | |
| 100 mm Corning non-treated culture dishes | Sigma Aldrich | CLS430591 | |
| 0.25% Trypsin-EDTA | Gibco | 25200-056 | Equipment; 37 °C, 5% Humidity |
| Forma Steri-Cycle CO2 Incubators | Thermofisher Scientific | 371 | |
| Hausser Bright-Line Phase Hemacytometer | Thermofisher Scientific | 02-671-6 | |
| Live/Dead Viability/Cytotoxicity Kit for mammalian cells | Life Technologies | L3224 | |
| CCK-8 Assay | Dojindo Laboratories | CK04-11 | Monosodium-salt reagent (MSR) |
| Infinite 200 PRO microplate reader | Tecan | ||
| Albumin Human ELISA kit | Abcam | ab108788 | |
| Triton X-100 | Bio-Rad | #1610407 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A2153-50G | |
| Anti-mouse primary antibodies (against CYP3A4, albumin) | Santa Cruz Biotechnology | sc-53850; sc-271605 | |
| DAPI | Life Technologies | D3571 | |
| Alexa Fluor 555 labeled Phalloidin | Life Technologies | A34055 | |
| Trizol | Life Technologies | 15596-026 | |
| Chloroform | VWR | 22706.326 | |
| Isopropanol | Fisher Scientific | 67-63-0 | |
| DPEC water | Thermofisher Scientific | AM9916 | |
| Nanodrop 2000c Spectrophotometer | Thermofisher Scientific | ND-2000 | |
| iScript Reverse Transcription Supermix | Bio-Rad Laboratories | 1708840 | |
| SYBR select Master Mix for CFX | Life Technology | 4472937 | |
| Primers (to be chosen) | |||
| CFX96 Real-Time System, C-1000 Touch Thermal Cycler | Bio Rad Laboratories | SOFT-CFX-31-PATCH |
Referencias
- Yamada, M., et al. Controlled formation of heterotypic hepatic micro-organoids in anisotropic hydrogel microfibers for long-term preservation of liver-specific functions. Biomaterials. 33 (33), 8304-8315 (2012).
- Abboud, G., Kaplowitz, N. Drug-induced liver injury. Drug Safety. 30 (4), 277-294 (2007).
- Cho, N. J., et al. Viral infection of human progenitor and liver-derived cells encapsulated in three-dimensional PEG-based hydrogel. Biomed Mater. 4 (1), (2009).
- Revzin, A., et al. Designing a hepatocellular microenvironment with protein microarraying and poly (ethylene glycol) photolithography. Langmuir. 20 (8), 2999-3005 (2004).
- Sato, A., Kadokura, K., Uchida, H., Tsukada, K. An in vitro hepatic zonation model with a continuous oxygen gradient in a microdevice. Biochem Bioph Res Com. 453 (4), 767-771 (2014).
- Domansky, K., et al. Perfused multiwell plate for 3D liver tissue engineering. Lab Chip. 10 (1), 51-58 (2010).
- Hegde, M., et al. Dynamic interplay of flow and collagen stabilizes primary hepatocytes culture in a microfluidic platform. Lab Chip. 14 (12), 2033-2039 (2014).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat methods. 2 (2), 119-125 (2005).
- Underhill, G. H., Chen, A. A., Albrecht, D. R., Bhatia, S. N. Assessment of hepatocellular function within PEG hydrogels. Biomaterials. 28 (2), 256-270 (2007).
- Dunn, J., Tompkins, R. G., Yarmush, M. L. Hepatocytes in collagen sandwich: evidence for transcriptional and translational regulation. J cell biol. 116 (4), 1043-1053 (1992).
- Dunn, J. C., Tompkins, R. G., Yarmush, M. L. Long-term in vitro function of adult hepatocytes in a collagen sandwich configuration. Biotechnol progr. 7 (3), 237-245 (1991).
- Ling, Y., et al. A cell-laden microfluidic hydrogel. Lab Chip. 7 (6), 756-762 (2007).
- Kim, M., Lee, J. Y., Jones, C. N., Revzin, A., Tae, G. Heparin-based hydrogel as a matrix for encapsulation and cultivation of primary hepatocytes. Biomaterials. 31 (13), 3596-3603 (2010).
- Kotov, N. A., et al. Inverted Colloidal Crystals as Three-Dimensional Cell Scaffolds. Langmuir. 20 (19), 7887-7892 (2004).
- Shanbhag, S., Woo Lee, J., Kotov, N. Diffusion in three-dimensionally ordered scaffolds with inverted colloidal crystal geometry. Biomaterials. 26 (27), 5581-5585 (2005).
- Lee, Y. H., Huang, J. R., Wang, Y. K., Lin, K. H. Three-dimensional fibroblast morphology on compliant substrates of controlled negative curvature. Integr Biol. 5, 1447-1455 (2013).
- da Silva, J., Lautenschlager, F., Kuo, C. H. R., Guck, J., Sivaniah, E. 3D inverted colloidal crystals in realistic cell migration assays for drug screening applications. Integr Biol. 3, 1202-1206 (2011).
- da Silva, J., Lautenschlager, F., Sivaniah, E., Guck, J. R. The cavity-to-cavity migration of leukaemic cells through 3D honey-combed hydrogels with adjustable internal dimension and stiffness. Biomaterials. 31, 2201-2208 (2010).
- Lee, J., Lilly, G. D., Doty, R. C., Podsiadlo, P., Kotov, N. A. In vitro toxicity testing of nanoparticles in 3D cell culture. Small. 5, 1213-1221 (2009).
- Lee, J., Kotov, N. A. Notch ligand presenting acellular 3D microenvironments for ex vivo human hematopoietic stem-cell culture made by layer-by-layer assembly. Small. 5, 1008-1013 (2009).
- Liu, Y., et al. Rapid aqueous photo-polymerization route to polymer and polymer-composite hydrogel 3D inverted colloidal crystal scaffolds. J Biomed Mater Res. Part A. 83, 1-9 (2007).
- Ma, P. X., Choi, J. W. Biodegradable polymer scaffolds with well-defined interconnected spherical pore network. Tissue Eng. 7, 23-33 (2001).
- Cuddihy, M. J., Kotov, N. A. Poly (lactic-co-glycolic acid) bone scaffolds with inverted colloidal crystal geometry. Tissue Eng Part A. 14, 1639-1649 (2008).
- Choi, S. W., Zhang, Y., Xia, Y. Three-dimensional scaffolds for tissue engineering: the importance of uniformity in pore size and structure. Langmuir. 26, 19001-19006 (2010).
- Choi, S. W., Zhang, Y., Thomopoulos, S., Xia, Y. In vitro mineralization by preosteoblasts in poly(DL-lactide-co-glycolide) inverse opal scaffolds reinforced with hydroxyapatite nanoparticles. Langmuir. 26, 12126-12131 (2010).
- Choi, S. W., Zhang, Y., Macewan, M. R., Xia, Y. Neovascularization in biodegradable inverse opal scaffolds with uniform and precisely controlled pore sizes. Adv Healthc Mater. 2, 145-154 (2013).
- Zhang, Y., Choi, S. W., Xia, Y. Modifying the Pores of an Inverse Opal Scaffold With Chitosan Microstructures for Truly Three-Dimensional Cell Culture. Macromol Rapid Commun. 33, 296-301 (2012).
- Cai, X., et al. Investigation of neovascularization in three-dimensional porous scaffolds in vivo by a combination of multiscale photoacoustic microscopy and optical coherence tomography. Tissue Eng. Part C, Meth. 19, 196-204 (2013).
- Zhang, Y. S., Yao, J., Wang, L. V., Xia, Y. Fabrication of Cell Patches Using Biodegradable Scaffolds with a Hexagonal Array of Interconnected Pores (SHAIPs). Polymer. 55, 445-452 (2014).
- Zhang, Y. S., Regan, K. P., Xia, Y. Controlling the Pore Sizes and Related Properties of Inverse Opal Scaffolds for Tissue Engineering Applications. Macromol Rapid Commun. 34, 485-491 (2013).
- Stachowiak, A. N., Bershteyn, A., Tzatzalos, E., Irvine, D. J. Bioactive Hydrogels with an Ordered Cellular Structure Combine Interconnected Macroporosity and Robust Mechanical Properties. Adv Mater. 17, 399-403 (2005).
- Stachowiak, A. N., Irvine, D. J. Inverse opal hydrogel-collagen composite scaffolds as a supportive microenvironment for immune cell migration. J Biomed Mater Res. Part A. 85, 815-828 (2008).
- Liu, Y., Wang, S. 3D inverted opal hydrogel scaffolds with oxygen sensing capability. Colloids and surfaces. B, Biointerfaces. 58, 8-13 (2007).
- Bryant, S. J., Cuy, J. L., Hauch, K. D., Ratner, B. D. Photo-patterning of porous hydrogels for tissue engineering. Biomaterials. 28, 2978-2986 (2007).
- Bhrany, A. D., Irvin, C. A., Fujitani, K., Liu, Z., Ratner, B. D. Evaluation of a sphere-templated polymeric scaffold as a subcutaneous implant. JAMA facial plastic surgery. 15, 29-33 (2013).
- Kuo, Y. C., Chiu, K. H. Inverted colloidal crystal scaffolds with laminin-derived peptides for neuronal differentiation of bone marrow stromal cells. Biomaterials. 32 (3), 819-831 (2011).
- Yang, J. T., Kuo, Y. C., Chiu, K. H. Peptide-modified inverted colloidal crystal scaffolds with bone marrow stromal cells in the treatment for spinal cord injury. Colloids Surf. B, Biointerfaces. 84, 198-205 (2011).
- Kuo, Y. C., Tsai, Y. T. Inverted colloidal crystal scaffolds for uniform cartilage regeneration. Biomacromolecules. 11, 731-739 (2010).
- Choi, S. W., Xie, J., Xia, Y. Chitosan-Based Inverse Opals: Three-Dimensional Scaffolds with Uniform Pore Structures for Cell Culture. Adv Mater. 21, 2997-3001 (2009).
- Long, T. J., Sprenger, C. C., Plymate, S. R., Ratner, B. D. Prostate cancer xenografts engineered from 3D precision-porous poly(2-hydroxyethyl methacrylate) hydrogels as models for tumorigenesis and dormancy escape. Biomaterials. 35, 8164-8174 (2014).
- Kuo, Y. C., Tsai, Y. T. Inverted colloidal crystal scaffolds for uniform cartilage regeneration. Biomacromolecules. 11, 731-739 (2010).
- Kuo, Y. C., Chiu, K. H. Inverted colloidal crystal scaffolds with laminin-derived peptides for neuronal differentiation of bone marrow stromal cells. Biomaterials. 32, 819-831 (2011).
- Lee, J., Cuddihy, M. J., Cater, G. M., Kotov, N. A. Engineering liver tissue spheroids with inverted colloidal crystal scaffolds. Biomaterials. 30 (27), 4687-4694 (2009).
- Galperin, A., et al. Integrated bi-layered scaffold for osteochondral tissue engineering. Adv Healthc Mater. 2, 872-883 (2013).
- Waters, D. J., et al. Morphology of Photopolymerized End-linked Poly(ethylene glycol) Hydrogels by Small Angle X-ray Scattering. Macromolecules. 43 (16), 6861-6870 (2010).
- Elbert, D. L., Hubbell, J. A. Conjugate addition reactions combined with free-radical cross-linking for the design of materials for tissue engineering. Biomacromolecules. 2 (2), 430-441 (2001).
- Kim, M. H., et al. Biofunctionalized Hydrogel Microscaffolds Promote Three-Dimensional Hepatic Sheet Morphology. Macromol Biosci. , (2015).
- Ferreira, T., Rasband, W. ImageJ User Guide. , http://imagej.nih.gov/ij/docs/guide/146-30.html#toc-Subsection-30.1 (2012).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to Fluorescence Microscopy. , JoVE. Cambridge, MA. (2015).
- Tominaga, H., et al. A water-soluble tetrazolium salt useful for colorimetric cell viability assay. Anal Commun. 36 (2), 47-50 (1999).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to the Microplate Reader. , JoVE. Cambridge, MA. (2015).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The ELISA Method. , JoVE. Cambridge, MA. (2015).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat Protoc. 1, 1559-1582 (2006).
- JoVE Science Education Database. Essentials of Environmental Microbiology. RNA Analysis of Environmental Samples Using RT-PCR. , JoVE. Cambridge, MA. (2016).
- JoVE Science Education. Essentials of Environmental Microbiology. , JoVE. (2015).
- Jeong, S., et al. The evolution of gene regulation underlies a morphological difference between two Drosophila sister species. Cell. 132 (5), 783-793 (2008).
- Griffith, L. G., Naughton, G. Tissue engineering--current challenges and expanding opportunities. Science. 295 (5557), 1009-1014 (2002).
- Hegde, M., et al. Dynamic Interplay of Flow and Collagen Stabilizes Primary Hepatocytes Culture in a Microfluidic Platform. Lab Chip. 14, 2033-2039 (2014).
- Kim, Y., Lasher, C. D., Milford, L. M., Murali, T., Rajagopalan, P. A comparative study of genome-wide transcriptional profiles of primary hepatocytes in collagen sandwich and monolayer cultures. Tissue Eng Pt C. 16 (6), 1449-1460 (2010).
- Baimakhanov, Z., et al. Efficacy of multi-layered hepatocyte sheet transplantation for radiation-induced liver damage and partial hepatectomy in a rat model. Cell Transplant. , (2015).
- Li, C. Y., et al. Micropatterned Cell-Cell Interactions Enable Functional Encapsulation of Primary Hepatocytes in Hydrogel Microtissues. Tissue Eng Pt A. 20 (15-16), 2200-2212 (2014).
- Shlomai, A., et al. Modeling host interactions with hepatitis B virus using primary and induced pluripotent stem cell-derived hepatocellular systems. P Natl A Sci USA. 111 (33), 12193-12198 (2014).
- Curcio, E., et al. Mass transfer and metabolic reactions in hepatocyte spheroids cultured in rotating wall gas-permeable membrane system. Biomaterials. 28, 5487-5497 (2007).
- Martinez-Hernandez, A., Amenta, P. The hepatic extracellular matrix. Vichows Archiv A Pathol Anat. 423, 1-11 (1993).
- Liu, Y., Wang, S., Lee, J. W., Kotov, N. A. A Floating Self-Assembly Route to Colloidal Crystal Templates for 3D Cell Scaffolds. Chem Mater. 17 (20), 4918-4924 (2005).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados