Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Fabrication de Inverted colloïdal cristal Poly (éthylène glycol) Échafaudages: Une cellule plate-forme en trois dimensions de la culture pour l'ingénierie tissulaire du foie
Dans cet article
Résumé
This manuscript presents a detailed protocol for the fabrication of an emerging three-dimensional hepatocyte culture platform, the inverted colloidal crystal scaffold, and the concomitant techniques to assess hepatocyte behavior. The size-controllable pores, interconnectivity and ability to conjugate extracellular matrix proteins to the poly(ethylene glycol) (PEG) scaffold enhance Huh-7.5 cell performance.
Résumé
La possibilité de maintenir la fonction des hépatocytes in vitro, dans le but de tester la cytotoxicité des xénobiotiques, l' étude de l' infection virale et le développement de médicaments ciblant le foie, nécessite une plate - forme dans laquelle les cellules reçoivent des signaux biochimiques et mécaniques appropriées. systèmes d'ingénierie tissulaire du foie récents ont utilisé trois dimensions (3D) échafauds composés de hydrogels synthétiques ou naturelles, compte tenu de leur rétention d'eau élevée et leur capacité à fournir les stimuli mécaniques nécessaires par les cellules. Il y a eu un intérêt croissant dans le cristal colloïdal (CPI) échafaudage inversé, un développement récent, qui permet à l'organisation spatiale élevée, homotypique et l'interaction des cellules hétérotypique, ainsi que la matrice (ECM) interaction cellulaire extracellulaire. Nous décrivons ici un protocole pour la fabrication de l'échafaudage ICC à l'aide de poly (éthylène glycol) diacrylate (PEGDA), et le procédé de lixiviation des particules. En bref, un treillis est composé de particules microsphériques, après quoi un pré-polymela solution r est ajoutée proprement polymérisé et les particules sont ensuite éliminés, ou lessivés, à l' aide d' un solvant organique (par exemple le tétrahydrofuranne). La dissolution des résultats du réseau dans un échafaudage très poreuse avec des tailles et des interconnectivités qui permettent aux médias d'atteindre plus facilement les cellules interstitielles contrôlées. Cette structure unique permet une grande surface spécifique pour les cellules à adhérer à ainsi que la communication facile entre les pores, et la capacité de revêtir la CPI échafaudage PEGDA avec des protéines montre également un effet marqué sur la performance des cellules. Nous analysons la morphologie de l'échafaudage ainsi que la cellule d'hépatocarcinome (Huh-7.5) le comportement en termes de viabilité et de la fonction pour explorer l'effet de la structure de la CPI et les revêtements ECM. Dans l'ensemble, ce document fournit un protocole détaillé d'un échafaudage émergent qui a de larges applications dans l'ingénierie tissulaire, en particulier le foie ingénierie tissulaire.
Introduction
Le foie est un organe hautement vascularisé avec une multitude de fonctions, y compris la désintoxication du sang, le métabolisme des xénobiotiques et la production de protéines sériques. Le tissu hépatique a trois dimensions (3D), la microstructure complexe, comprenant plusieurs types cellulaires, les canalicules biliaires, sinusoïdes, et des zones de composition différente biomatrice et différentes concentrations en oxygène. Compte tenu de cette structure complexe, il a été difficile de créer un modèle correct du foie in vitro 1. Cependant, il y a une demande croissante de fonctionnement des modèles in vitro d' hébergement hépatocytes humains comme plates - formes pour les tests de toxicité médicamenteuse 2 et l' étude des maladies associées au foie 3.
Plates - formes d'ingénierie tissulaire du foie actuel ont simplifié la complexité du foie en isolant l' une, ou en se concentrant sur un petit nombre, des paramètres du foie, à savoir la co-culture de cellules 4, la composition biochimique du zonal microenvironnements 5, la dynamique d'écoulement 6,7 et la configuration de la biomatrice 8. La configuration de la biomatrice peut être décomposée en des paramètres tels que les matériaux d'échafaudage, la composition de la matrice extracellulaire (ECM), la raideur des protéines de la matrice, ainsi que la conception et la structure de l'échafaudage. Il y a eu une augmentation dans les études d'ingénierie tissulaire en utilisant des hydrogels synthétiques, en particulier le poly (éthylène glycol) (PEG) hydrogels 9, compte tenu de la capacité d'accorder des propriétés, la bioactivité et de vitesse de dégradation mécanique de l'hydrogel. En ce qui concerne la recherche liée au foie, l'hydrogel biocompatible a été appliquée pour l' étude de l' infection par le virus de la maladie du foie 3. En tant que plate - forme de conception de hépatocytes, de nombreuses études ont utilisé hépatocytes cultures sandwich 10,11 et l' encapsulation des cellules dans un hydrogel 12,13 pour fournir l'environnement 3D et cellule-ECM et l' interaction cellule-cellule qui sont essentiels pour imiter en microenvironnement vivo. However, ces plates - formes ne possèdent pas un degré élevé de contrôle et de l' organisation spatiale, conduisant à des propriétés non uniformes à travers l'échafaudage 14.
Le colloïdal de cristal inversée (ICC) 14 échafaudage est un échafaudage 3D hautement organisée pour la culture cellulaire qui a été introduit dans les années 2000. La structure unique de L'échafaudage peut être attribué au procédé de fabrication simple à l'aide d'un cristal colloïdal, un réseau ordonné de particules colloïdales de diamètre variable. En bref, pour résumer le processus, les particules sont bien rangés et recuites utilisant la chaleur pour former un réseau. La lixiviation de ce réseau, par un solvant organique, on obtient un hydrogel polymérisé dans des cavités sphériques hexagones emballés 15 avec une surface spécifique élevée. Cet échafaudage hautement ordonnée a été préalablement réalisé avec les deux matériaux synthétiques et naturels, y compris , mais sans s'y limiter , le poly (acrylamide) 16-21, le poly (lactique-co-acide glycolique) 15,22-30, Le poly (éthylène glycol) 31,32, le poly (2-hydroxyéthylméthacrylate) 21,33-35, 36-39 et le chitosan. CPI échafauds constituées de matériaux non-salissures ont tendance à favoriser sphéroïdes cellulaires dans les cavités 14,23,40. Plusieurs types de cellules ont été représentés avec succès à proliférer, se différencier et de fonction dans cette configuration, y compris les 41 chondrocytes, les cellules stromales de la moelle osseuse 42, et les cellules souches 43,44. En ce qui concerne les hépatocytes, des études ont été menées avec des échafaudages de la CPI en Na 2 SiO 3 et de poly (acrylamide), mais pas de PEG. Avec des stratégies simples de bioconjugaison (c. -à- couplage amine par EDC / NHS), échafauds à base de PEG ECM protéines conjuguées peuvent être fabriquées, qui peuvent se révéler des sites de liaison plus de cellules pour être un plus in vivo comme l' environnement et d' améliorer la fonction hépatique.
Dans ce manuscrit et la vidéo associée, nous détaillons la fabrication de l'échafaudage de la CPIen utilisant le poly (éthylène glycol) diacrylate (PEGDA) et un réseau d'hydrogel de polystyrène microsphère, optimisé pour une hépatocarcinome (Huh-7.5) la culture. Nous démontrons les différences entre les nues échafauds ICC PEGDA généralement non collantes et le PEGDA ICC échafaudage de collagène revêtu en termes d'échafaudage topologie et la performance des cellules. La viabilité cellulaire et la fonction sont mesurés qualitativement et quantitativement pour évaluer Huh-7,5 comportement cellulaire.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. ICC Échafaudages Fabrication (Figure 1)
- Préparer le polystyrène (PS) lattices (diamètre = 6 mm; 8-13 couches de perles).
- Pour préparer le moule, couper les pointes hors de 0,2 ml microtubes bouillir à l'épreuve au niveau de 40 pi. Adhérer au sommet des coupe-tubes à 24 x 60 mm 2 microscope glisse en verre de couverture avec de la colle imperméable à l' eau.
- Mettez les sphères PS (diamètre = 140 um) contenues dans une suspension de l'eau dans un flacon de 20 ml, soigneusement la pipette sur la suspension de l'eau et ajouter 18 ml de solution à 70% d'éthanol dans le flacon. Mettre la solution de sphère dans un bain à ultrasons pour desserrer les sphères agrégées. Répétez cette étape de lavage à plusieurs reprises afin d'éliminer l'eau et soluble dans l'eau des composants complètement.
- Pipeter 100 pi d'éthanol dans les moules.
- Coupez le haut d'une pointe de micropipette 200 pi par 4 mm. Pipette 25 ul des sphères dans le moule deux fois en utilisant 200 micropipette ul pour obtenir unvolume total de 50 pi dans chaque moule.
- Placer les moules sur un agitateur à bascule à 120 tours par minute pendant la nuit.
- Vérifier l'agencement des sphères dans chaque moule sous un microscope optique. Si les sphères ne sont pas ordonnés en hexagones, ajouter 50 ul d'éthanol à 70% et agiter manuellement dans la direction de l'axe longitudinal et latéral pour corriger l'arrangement.
- Laisser évaporer l'éthanol à la température ambiante (RT) pour deux nuits. Placer le moule et complexe de perles dans un four C 130 ° pendant 6 heures pour recuire les billes de PS.
- Préparation échafauds PEGDA nus et ECM-enduit.
- Synthétiser macromères PEGDA utilisant des protocoles établis pour 45,46 acrylation macromères PEG linéaires (M w = 4,6 kDa).
- Préparer 50% (p / v) dans PEGDA (DI) de l'eau désionisée et laisser le macromère de dissoudre convenablement par centrifugation à 4713 x g jusqu'à ce qu'il soit complètement dissous.
- Pour ECM conjugué échafauds de la CPI, dissoudre une supplémentaire10% (p / v) acryloyl-PEG-NHS (Mp = 3,4 kDa) dans la solution PEGDA 50%.
- Préparer un 20% (p / v) solution mère de 2-hydroxy-4 '- (2-hydroxyéthoxy) -2-méthylpropiophénone (PI) dans 70% d'éthanol.
- Ajouter 50 ul de 20% (p / v) solution PI mère par 1 ml de 50% (p / v) de PEGDA. Ajustez la quantité nécessaire de PI solution mère sur la base du poids moléculaire de PEGDA.
- Vortex le mélange dans un tube de centrifugeuse pour 1 min pour atteindre une solution homogène.
- Peler les moules de la lame de verre (de l'étape 1.1.7), enlever la colle des moules, pousser les grilles à l'aide d'une spatule soigneusement et placer chacun d'eux dans un tube de 1,5 ml. Introduire à la pipette 300 ul de la solution et on centrifuge PEGDA à 845 g pendant 5 min pour permettre une bonne solution PEGDA infiltration dans le réseau.
- Retirer la grille du tube en utilisant des pinces et épongez avec soin solution PEGDA excès sec sur des gants. Placez le treillis sur un verre recouvert d'un film de paraffine avec le cir platsurface culier vers le haut.
- Exposer l'échafaud infiltré de solution PEGDA à 365 nm ultraviolet (UV) (10,84 mW / cm 2) pendant 5 min à l' aide d' une lampe spot UV.
- Placer les réseaux cristallins PEGDA polymérisés dans de nouvelles fioles (environ 10 lattices par flacon) et ajouter 20 ml de tétrahydrofuranne (THF). Agiter les flacons sur un agitateur orbital à 300 rpm. Modifier le THF d'au moins 3 fois avec un intervalle de 1-2 heures.
Note: Ne pas éliminer le THF complètement lors du changement du THF afin d'éviter que des bulles d'entrer dans les échafauds, qui à son tour peut provoquer une élimination incomplète du PS. Laissez suffisamment de solution pour couvrir les réseaux et ajouter de nouveaux THF.
Attention: THF est toxique. Porter des gants, un manteau et des lunettes de laboratoire. Éviter l'inhalation en opérant sous la hotte. - Vérifiez si les sphères PS sont dissous en mettant de l'eau dans la solution de THF utilisé et en observant la couleur de la solution. Répétez l'étape 1.2.9 si les sphères PS ne sont pas correctement dissous.
Remarque: La couleur de la solution passe àblanc si des sphères PS restantes.
- Nettoyez les échafauds dans l'armoire de biosécurité (BSC).
- Pour stériliser les échafauds, préparer un tube centrifuge de 50 ml avec 2 ml d'éthanol à 70% par échafaudage et placer les échafauds dans le tube à l'aide d'une spatule. Autoriser les échafauds à tremper dans de l'éthanol pendant 1 heure. A partir de ce pas en avant, effectuer toutes les procédures du BSC.
- verser doucement l'éthanol et le remplacer par une solution saline de tampon phosphate (PBS) (2 ml par échafaudage) et centrifuger à 524 g pendant 3 min pour éliminer les bulles. Gardez-le dans le réfrigérateur et changer le PBS quelques fois avec un intervalle de 1-2 heures.
- Pour le type de collagène échafauds I-enduit, préparer un autre tube de centrifugeuse de 50 ml contenant du collagène de type 1 solution stock (1 ml par échafaudage), transférer les échafauds stérilisés à ce tube à l'aide d'une spatule, et centrifuger à 524 g pendant 3 min. Agiter les échafauds à 400 rpm sur un agitateur orbital pendant 30 minutes et maintenir le tube dans le équipemou une nuit.
- Laver les échafauds avec PBS deux fois avant de l'utiliser en submergeant les échafauds dans du PBS frais puis aspirer le PBS.
Note: D' autres protéines ECM peuvent également être utilisés à la place du collagène de type I , parce que la chimie NHS a besoin d' un groupe amine pour former la liaison (figure 2).
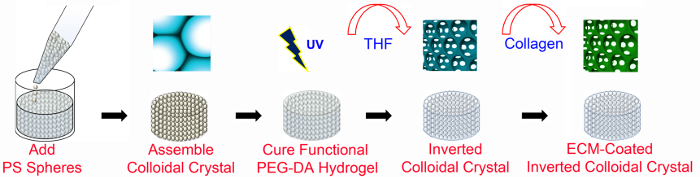
Figure 1. Vue d'ensemble de la CPI fabrication. Échafauds de la CPI à base de PEG sont fabriqués en utilisant des techniques de microfabrication avec et sans ECM-fonctionnalisation. Échafauds ICC ECM enduit nécessitent PEG-NHS ainsi que PEGDA (comme indiqué dans la figure 2). Le réseau PS a un diamètre de 6 mm et une hauteur de 8-13 couches de perles. PS polystyrène; PEGDA, le poly (éthylène glycol) diacrylate; UV, les rayons ultraviolets; THF, tétrahydrofuranne; ECM, la matrice extracellulaire. Ce chiffre a été modifié et utilisé avec la permission de Wiley 47. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
2. Structure ICC Caractérisation
- Pour analyser la structure de la CPI , avec ou sans protéines conjuguées, en utilisant la microscopie électronique à balayage (SEM) 47.
- Fixer les échafauds avec 4% de paraformaldehyde (PFA), en série les déshydrater dans les solutions 25, 50, 75, 95 et 100% d'éthanol, et de les stocker à -80 ° C jusqu'à ce que l'éthanol soit complètement évaporée.
- Des échantillons secs dans un lyophilisateur pendant 48 heures.
- Apposer l'échantillon sur support d'échantillon en utilisant du ruban de carbone et dans une coucheuse par pulvérisation.
- Après l'aspirateur automatique, l'enduire d'un film Pt de 10 nm d'épaisseur par pulvérisation pendant 60 secondes à 20 mA.
- Échafauds image de la CPI à l' aide de SEM à une tension de 5 kV (figure 3A, la figure 4A).
- Pour mesurer les pores etle diamètre d'interconnexion de cavités, analyser micrographies MEB en utilisant un logiciel d'analyse d' image 48 (par exemple ImageJ; la figure 3B, C).
- Pour visualiser le collagène conjugué à l'échafaud sans cellules, le collagène tag fluorescence en utilisant des anticorps (1: 100) contre le collagène de type I et de l' image avec le laser microscopie confocale à balayage 47 (CLSM, figure 4B).
3. Huh-7.5 Culture cellulaire et Semis
- Culture Huh-7.5 cellules à une densité d'ensemencement de 2-2,5 x 10 6 cellules / ml dans 100 mm de boîtes de culture cellulaire avec 10 ml Dulbecco milieu de Eagle modifié (DMEM) supplémenté avec 10% de sérum fœtal bovin (FBS) et 100 U / ml pénicilline-streptomycine (milieu de croissance) à 37 ° C et 5% de CO 2. Changer les médias tous les trois jours dans le BSC jusqu'à ce qu'ils aient atteint 75-80% de confluence.
- Préparer les échafauds pour l'ensemencement des cellules dans le BSC.
- Placez délicatement le Scaffolds dans une plaque de 24 puits avec la surface plate vers le haut.
- Pour laver l'échafaud, pipette 2 ml de PBS à chaque puits contenant un échafaudage. Aspirer le PBS et pipette 2 ml de PBS frais dans chaque puits.
- Aspirer le PBS et pipette 2 ml de milieu de croissance (voir étape 3.1) et laisser reposer pendant 30 min. Aspirer les médias et permettre à l'échafaud à sécher pendant 1 h.
- Détachez confluentes Huh-7,5 (cellules de l'étape 3.1) de la plaque de culture dans le BSC en utilisant la méthode de digestion de la trypsine.
- Aspirer le milieu de la plaque, ajouter 4 ml de PBS pour laver les cellules adhérentes, puis aspirer PBS.
- Pipette 0,75-1 ml 0,25% trypsine et placer dans un incubateur à 37 ° C, 5% de CO 2 pendant 3 min.
- Retirer la plaque de l'incubateur et la pipette 5 ml de milieu pour arrêter la réaction de la trypsine. Pipette support, des cellules isolées, et le mélange de la trypsine dans un tube de 15 ml.
- Centrifuger à 524 xg pendant 3 min, retirer le surnageant et remettre en suspension le culot dans 5 ml de milieu.
- Compter les cellules à l' aide d' un hémocytomètre et de calculer le volume de la suspension cellulaire qui contient des cellules au nombre cible, N 0, par 25 ul (pour l' expérience standard, N 0 est de 1 x 10 6 cellules).
Volume de suspension cellulaire = (nombre cible de cellules) / (concentration de la suspension cellulaire) - Pipette lentement 25 pi de suspension de cellules (contenant du N 0 cellules) directement sur le dessus de chaque échafaudage (de l' étape 3.1.4). Placer la plaque de 24 puits dans l'incubateur.
- Après 12 h, transférer les échafauds soigneusement à l'aide d'une spatule dans une nouvelle plaque de 24 puits et la pipette 2 ml de milieu dans chaque puits. Placer la plaque de 24 puits dans l'incubateur.
- Changer les médias tous les 3 jours ou selon le moment où les médias sont recueillis pour une analyse de sécrétion des protéines (voir l'étape 5.1).
4. Viabilité cellulaire
- Pour analyser qualitativement la viabilité des cellules, utilisez / morts kits de coloration de vivantes fluorescentes pour colorer les cellules et de l'image en utilisant CLSM.
- Après kit instruction, préparer une solution avec 4 uM de calcéine AM et 8 uM éthidium homo-dimère-1 dans les médias (voir étape 3.1.3).
Remarque: optimiser en fonction du nombre de cellules. Utilisez le double de la quantité de réactif si les cellules prolifèrent et double en nombre (environ 2 millions). - Dans le BSC, Aspirer le milieu dans chaque puits avec un échafaudage (étape 3.7) et la pipette 500 ul de la solution préparée. Incuber les échantillons dans un incubateur à 37 ° C pendant 1 heure.
- Couvrir les plaques dans du papier pour protéger les échantillons de la lumière lors du retrait de la plaque de l'incubateur. échantillons d'image en utilisant CLSM49.
- Après kit instruction, préparer une solution avec 4 uM de calcéine AM et 8 uM éthidium homo-dimère-1 dans les médias (voir étape 3.1.3).
- Pour évaluer quantitativement la viabilité des cellules, mesurer l' activité enzymatique (dans des cellules vivantes) en utilisant des dosages colorimétriques 50 ( par exemple, le 2- (2-méthoxy-4-nitrophényl) -3- (4-nitrophényl) -5- (2,4-disulfophényl) -2H-tétrazolium (réactif monosodique-sel)).
- Créer une courbe standard pour la plate - forme de la CPI (ie, un graphique de l' absorbance (DO) versus donné le nombre de cellules).
Note: Cellule ensemencement est pas efficace à 100% par rapport aux autres plates-formes, car les cellules peuvent passer à travers les cavités des échafauds.- Déterminer le nombre d'ensemencement cellulaire, N 0 qui seront utilisés pour rendre la courbe standard.
Remarque: Choisissez une gamme qui comprend le nombre de cellules qui sont estimés dans l'expérience. Par exemple, si le nombre initial de cellules est de 5 x 10 5 cellules , et il existe un ~ 3 fois plus élevée estimée par le dernier jour de l'expérience, pour choisir 2,5 x 10 5 5 x 10 5 1 x 10 6 et 2 x 10 6 cellules en tant que N 0. - Effectuer l' ensemencement des cellules dans les échafauds de la CPI (un N 0 par échafaudage avec un volume d'ensemencement de 25 pi) comme décrit dans les étapes 3.1-3.6.
- Après 6 heures, transférer les échafauds à une autre plaque de 24 puits bien. Sélectionnez un temps qui permet l'adhérence des cellules, mais pas la prolifération cellulaire.
- Effectuer le comptage des cellules sur l'échafaud de la CPI transféré.
- Diluer 10x réactif monosodique-sel (MSR) solution 1x avec les médias dans le BSC et la pipette 500 ul solution 1x MSR dans chaque puits avec un échafaudage. Incuber la plaque à 24 puits à 37 ° C pendant 1 heure.
- De chaque puits, transférer 100 pi dans une plaque de 96 puits bien. En blanc, pipette 100 pi de solution fraîche de réactif 1x monosodique-sel dans différents puits sur la plaque de 96 puits. supprimer manuellement toutes les bulles présentes à l'aide d'une pointe de pipette sèche et couvrent la plaque de 96 puits dans du papier pour le protéger de la lumière.
- Mesure DO à λ = 450 nm lecture à l'aide d'un spectrophotomètre. Soustraire la DO vierge à partir d' autres valeurs pour trouver la DO précise 51.
- Comptez le nombre de cellules (N L) restant dans le puits après le transfert de l'échafaudage (étape 4.2.1.3) en utilisant un hémocytomètre.
Note: Utiliser 300 pi de trypsine pour trypsiniser les cellules. - Calculer le nombre de cellules réelles, N A.
= réelsinitial - à gauche dans le puits
N A = N -N 0 L - Faire courbe standard en reportant OD obtenue à l' étape 4.2.1.4.3 par rapport au nombre réel de la cellule (N A) et l' utiliser pour estimer le nombre de cellules dans les expériences.
Note: Faire une nouvelle courbe standard si tous les paramètres de la CPI (taille de porogène, les dimensions de l'échafaudage, la protéine ECM, etc.) sont modifiés.
- Déterminer le nombre d'ensemencement cellulaire, N 0 qui seront utilisés pour rendre la courbe standard.
- Créer une courbe standard pour la plate - forme de la CPI (ie, un graphique de l' absorbance (DO) versus donné le nombre de cellules).
5. Cellule Fonction
- Analyser la sécrétion de protéines par les Huh-7,5 cellules ( par exemple, l' albumine, urée) des médias collectés (de l' étape 3.7) par l' enzyme linked immunosorbent assay (ELISA) 52.
Note: Diluer les médias, en fonction du nombre de cellules ensemencées et la quantité de médias collectés. Pour 5 x 10 5 cellules ensemencées dans ICC, utiliser un ~ rapport 1: 25, avant de l' introduire dans les puits d'anticorps Prérevêtus. - Pour analyser qualitativement la fonction cellulaire, immunocoloration des protéines intracellulaires spécifiques( Par exemple, l' albumine), des enzymes ( par exemple, le CYP450), des composants de coloration de structure (c. -à- actine cellulaire), ainsi que des noyaux et de l' image à l' aide CLSM 49.
- Aspirer le milieu (de l'étape 3.7) et la pipette 2 ml de PBS pour laver les échafauds de la CPI cellule chargée.
- Introduire à la pipette 1 ml de PFA à 4% et incuber pendant 5 minutes à la température ambiante pendant la fixation.
- Laver 3x avec 2 ml de PBS.
- Perméabiliser les membranes par incubation des échafauds dans 1 ml de 0,1% de 4- (1,1,3,3-tétraméthylbutyl) phényl-polyéthylène glycol (agent tensio-actif) pendant 30 minutes.
- Laver 3 fois avec 2 ml de PBS pour éliminer les protéines qui fuient.
- Introduire à la pipette 500 ul de 1% de sérum albumine bovine (BSA) et on incube à température ambiante pendant 1 heure pour bloquer la liaison non spécifique.
- Préparation de l' anticorps primaire dilué (ie, l' albumine, CYP450) solution.
- Introduire à la pipette 500 ul de solution de BSA à 1% dans un tube de 15 ml et ajouter 4,5 ml de solution à 0,1% d'agent tensio-actif pour préparer une 5 ml de solution à 0,1% au total de BSA.
- Introduire à la pipette 98 ul de la solution de BSA à 0,1% dans un microtube de 200 pi et 2 pi de l'anticorps primaire pour produire un 01:50 (anticorps primaire: 0,1% de BSA), une solution d'anticorps primaire.
- Pipette 40 pl de la solution d'anticorps primaire sur l'échafaud et recouvrir le substrat avec un film de paraffine. Enveloppez la plaque 24 et une feuille d'aluminium et de stocker le plat à 4 ° C pendant la nuit.
- Laver 3x avec 2 ml de PBS et agiter la plaque doucement entre le lavage.
- Préparation de l' anticorps secondaire biotinylé dilué (ie, un anticorps anti-souris) solution mère.
- Introduire à la pipette une solution de BSA à 198 ul de 0,1% et le deuxième anticorps 2 ul pour produire un mélange 1: (anticorps secondaire: 0,1% de BSA), 100 de solution d'anticorps secondaire.
- Préparer une solution mère à 0,1% de rhodamine ou fluorescéine phalloïdine marquée (pour colorer les filaments d'actine cellulaires) dans la solution de BSA à 0,1%.
- Pipette 25 pi de chaque solution dans un tube et mélanger waune.
- Distribuer 50 ul de la solution anticorps secondaire d'actions sur l'échafaud. Couvrir l'échafaud avec un film de paraffine, envelopper le plat avec une feuille d'aluminium et conserver à la température ambiante pendant 2 h.
- Laver 3x avec 2 ml de PBS.
- Pipette 200 ul de 0,2% 4'-6-diamidino-2-phénylindole (DAPI, une tache de noyau) solution sur l'échafaud et de garder à température ambiante pendant 2-3 min. Couvrir la plaque avec une feuille d'aluminium.
- Laver 2x avec 2 ml de PBS.
- L'utilisation d'un compte-gouttes, placer une goutte de milieu de montage sur le substrat.
- Soigneusement mis l'échafaud sur une lame de verre et de l' image en utilisant CLSM 47.
- Évaluer l'expression génique en temps réel par réaction en chaîne par polymérase (PCR quantitative). Utilisez des kits standard pour la transcriptase inverse PCR (RT-PCR) 53 et 54 qPCR selon les instructions du fabricant. Extraire l'ARN à partir des cellules comme décrit ci-dessous.
- Placer l'échafaudage (étape 3.7) dans un microtube de 1,5 ml.
- Pipette 1 ml d'une solution d'extraction d'ARN dans le tube et garder dans un sonicateur pendant 5 min à température ambiante.
- Pipeter 200 pi de chloroforme dans chaque tube à centrifuger et agiter vigoureusement le tube dans la main pendant 15-20 secondes. Gardez les tubes à température ambiante pendant ~ 3 min jusqu'à ce que les phases se séparent.
- Centrifuger l'échantillon à 13 000 xg, 4 ° C pendant 15 minutes et retirer soigneusement les tubes de sorte que les phases ne se mélangent pas.
- pipeter avec soin 500-600 ul de la phase aqueuse supérieure du premier tube dans un deuxième tube de microcentrifugation.
- Ajouter un volume équivalent (500-600 pi) d'isopropanol à ce second tube.
- Inversez le tube 3-5 fois et laisser le tube debout à température ambiante pendant 10 min.
- Centrifuger l'échantillon à 13 000 x g, 4 ° C pendant 15 min.
- Si une pastille ne soit pas visible au fond du tube, centrifuger pendant 5 min.
Remarque: S'il n'y a toujours pas de culot visible, la quantité d'ARN peut être insuffisant.
- Si une pastille ne soit pas visible au fond du tube, centrifuger pendant 5 min.
- jetubes nvert avec le capuchon ouvert à éliminer le surnageant et la pipette dans 1 ml de 70% d'éthanol dilué dans l'eau DEPC dans le tube.
- Légèrement vortex le tube de sorte que le culot se détache de la paroi du tube, puis de laisser l'air de tube sec.
- Ajouter 50 ul d'eau DPEC pour remettre en suspension le culot.
- Gardez pipetage jusqu'à dissolution de granulés.
- Gardez pendant 10 min à 55 ° C pour dénaturer l'ARN double brin dans l'ARN simple brin.
- tambour légèrement les doigts sur le fond du tube, puis centrifuger les tubes brièvement (7500 xg, 4 min, 4 ° C).
- Gardez les tubes dans la glace jusqu'à l' exécution de la transcriptase inverse 55 et PCR en temps réel comme décrit dans 56.
Access restricted. Please log in or start a trial to view this content.
Résultats
Les résultats représentatifs pour la caractérisation structurale de l'échafaudage de la CPI et la comparaison de l'efficacité de chaque CPI échafaudage condition dans la culture des hépatocytes sont présentés et expliqués ci-dessous. Les conditions CPI d'échafaudage utilisés dans ces résultats sont enduits de collagène de 0 pg / ml (nu), 20 pg / ml (collagène 20), 200 ug / ml (collagène 200), et 400 ug / ml (collagène 400) et le premier huh-7,5 cellulaire nom...
Access restricted. Please log in or start a trial to view this content.
Discussion
Échafauds d'ingénierie tissulaire évoluent rapidement pour fournir tous les indices physiques et biochimiques nécessaires pour régénérer, maintenir ou réparer les tissus pour l'application de remplacement d'organes, d' étudier la maladie, le développement de médicaments, et beaucoup d' autres 57. Dans le foie ingénierie tissulaire, les hépatocytes humains primaires perdent rapidement leurs fonctions métaboliques, une fois isolés du corps, créant ainsi un grand besoin d'...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
The authors have no competing financial interests to disclose.
Remerciements
Les auteurs tiennent à remercier le soutien d'une recherche nationale Foundation Fellowship (NRF -NRFF2011-01) et le Programme de recherche concurrentiel (NRF-CRP10-2012-07).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 0.2 ml PCR tube | Axygen Scientific | PCR-02D-C | Boil-proof |
| Gorilla Glue | Gorilla Glue, Inc. | Depends on vendor. This was purchased from a local store. | |
| Glass slides | VWR | 631-1575 | Dimensions: 24×60 mm2 |
| Polystyrene spheres | Fisher Scientific | TSS#4314A | Diameter = 140 µm; 3x104 particles per milliliter and 1.4% size distribution |
| Ethanol | Merck | 1.00983.1011 | absolute for analysis EMSURE; Dilute to 70% with Milli-Q water |
| Ultrasonic Bath | Elma | S10H | Equiment |
| Furnace | Nabertherm | N7/H | Equipment |
| 200 µl pipette tip | Axygen Scientific | T-210-Y-R-S | |
| Rocking shaker | VWR | 444-0142 | |
| Polyethylene Glycol (PEG) | Merck | 1.09727.0100 | Mw= 4 kDa; acrylation of PEG monomers and purification of the resulting precipitate produces a PEGDA macromer with Mw = 4.6 kDa |
| Centrifuge | Beckman Coulter | 392932 | Equipment |
| Acrylate-Poly(Ethylene Glycol) - Succinimidyl Valerate | Laysan Bio | ACRL-PEG-SVA-3400-1g | Mw = 3.4 kDa |
| 2-hydroxy-4'-(2-hydroxyethoxy)-2-methylpropiophenone | Sigma Aldrich | 410896 | |
| Vortex | VWR | 58816-123 | Equipment |
| Microcentrifuge | Eppendorf | 5404 000.413 | |
| Paraffin Film | Parafilm M | #PM996 | Kept at 9" with allows intensity of 10.84 mW/cm2 |
| Bluewave 200 UV spotlight | Blaze Technology | 120008, 122300 | |
| Tetrahydrofuran (THF) | Merck | 107025 | |
| Orbital shaker | Heidolph | 543-123120-00-5 | |
| Collagen Type I | Sigma Aldrich | C3867-1VL | From rat. 1x, w/o CaCl2 & MgCl2; pH = 7.2 |
| Phosphate Buffered Saline (PBS) | Gibco | 20012-027 | 16% W/V AQ. 10 x 10 ml |
| Paraformaldehyde | VWR | 43368.9M | Equipment |
| Freezone 4.5 freeze drier | Labconco | 7750020 | Equipment |
| Sputter coater | Jeol Ltd. | JFC-1600 | Equipment |
| Scanning Electron Microscope | Jeol Ltd. | JSM 5310 | |
| Anti-mouse primary antibodies against Collagen type I | Abcam | ab6308 | |
| Anti-mouse secondary antibody conjugated with Alexa Fluor 488 | Life Technologies | A21121 | |
| Plate, Tissue Culture 24 Well, Flat Bottom (Nunclon) | Bio-Rev PTE LTD | 3820-024 | |
| Dulbecco's Modified Eagle's Medium(DMEM) 2.5 g/L Glucose w/ L-Gln | Lonza | 12-604F | |
| Fetal Bovine Serum (FBS) | Gibco | A15-151 | |
| Penicillin-Streptomycin (P/S) | Life Tchnologies | 15140-122 E | |
| 100 mm Corning non-treated culture dishes | Sigma Aldrich | CLS430591 | |
| 0.25% Trypsin-EDTA | Gibco | 25200-056 | Equipment; 37 °C, 5% Humidity |
| Forma Steri-Cycle CO2 Incubators | Thermofisher Scientific | 371 | |
| Hausser Bright-Line Phase Hemacytometer | Thermofisher Scientific | 02-671-6 | |
| Live/Dead Viability/Cytotoxicity Kit for mammalian cells | Life Technologies | L3224 | |
| CCK-8 Assay | Dojindo Laboratories | CK04-11 | Monosodium-salt reagent (MSR) |
| Infinite 200 PRO microplate reader | Tecan | ||
| Albumin Human ELISA kit | Abcam | ab108788 | |
| Triton X-100 | Bio-Rad | #1610407 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A2153-50G | |
| Anti-mouse primary antibodies (against CYP3A4, albumin) | Santa Cruz Biotechnology | sc-53850; sc-271605 | |
| DAPI | Life Technologies | D3571 | |
| Alexa Fluor 555 labeled Phalloidin | Life Technologies | A34055 | |
| Trizol | Life Technologies | 15596-026 | |
| Chloroform | VWR | 22706.326 | |
| Isopropanol | Fisher Scientific | 67-63-0 | |
| DPEC water | Thermofisher Scientific | AM9916 | |
| Nanodrop 2000c Spectrophotometer | Thermofisher Scientific | ND-2000 | |
| iScript Reverse Transcription Supermix | Bio-Rad Laboratories | 1708840 | |
| SYBR select Master Mix for CFX | Life Technology | 4472937 | |
| Primers (to be chosen) | |||
| CFX96 Real-Time System, C-1000 Touch Thermal Cycler | Bio Rad Laboratories | SOFT-CFX-31-PATCH |
Références
- Yamada, M., et al. Controlled formation of heterotypic hepatic micro-organoids in anisotropic hydrogel microfibers for long-term preservation of liver-specific functions. Biomaterials. 33 (33), 8304-8315 (2012).
- Abboud, G., Kaplowitz, N. Drug-induced liver injury. Drug Safety. 30 (4), 277-294 (2007).
- Cho, N. J., et al. Viral infection of human progenitor and liver-derived cells encapsulated in three-dimensional PEG-based hydrogel. Biomed Mater. 4 (1), (2009).
- Revzin, A., et al. Designing a hepatocellular microenvironment with protein microarraying and poly (ethylene glycol) photolithography. Langmuir. 20 (8), 2999-3005 (2004).
- Sato, A., Kadokura, K., Uchida, H., Tsukada, K. An in vitro hepatic zonation model with a continuous oxygen gradient in a microdevice. Biochem Bioph Res Com. 453 (4), 767-771 (2014).
- Domansky, K., et al. Perfused multiwell plate for 3D liver tissue engineering. Lab Chip. 10 (1), 51-58 (2010).
- Hegde, M., et al. Dynamic interplay of flow and collagen stabilizes primary hepatocytes culture in a microfluidic platform. Lab Chip. 14 (12), 2033-2039 (2014).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat methods. 2 (2), 119-125 (2005).
- Underhill, G. H., Chen, A. A., Albrecht, D. R., Bhatia, S. N. Assessment of hepatocellular function within PEG hydrogels. Biomaterials. 28 (2), 256-270 (2007).
- Dunn, J., Tompkins, R. G., Yarmush, M. L. Hepatocytes in collagen sandwich: evidence for transcriptional and translational regulation. J cell biol. 116 (4), 1043-1053 (1992).
- Dunn, J. C., Tompkins, R. G., Yarmush, M. L. Long-term in vitro function of adult hepatocytes in a collagen sandwich configuration. Biotechnol progr. 7 (3), 237-245 (1991).
- Ling, Y., et al. A cell-laden microfluidic hydrogel. Lab Chip. 7 (6), 756-762 (2007).
- Kim, M., Lee, J. Y., Jones, C. N., Revzin, A., Tae, G. Heparin-based hydrogel as a matrix for encapsulation and cultivation of primary hepatocytes. Biomaterials. 31 (13), 3596-3603 (2010).
- Kotov, N. A., et al. Inverted Colloidal Crystals as Three-Dimensional Cell Scaffolds. Langmuir. 20 (19), 7887-7892 (2004).
- Shanbhag, S., Woo Lee, J., Kotov, N. Diffusion in three-dimensionally ordered scaffolds with inverted colloidal crystal geometry. Biomaterials. 26 (27), 5581-5585 (2005).
- Lee, Y. H., Huang, J. R., Wang, Y. K., Lin, K. H. Three-dimensional fibroblast morphology on compliant substrates of controlled negative curvature. Integr Biol. 5, 1447-1455 (2013).
- da Silva, J., Lautenschlager, F., Kuo, C. H. R., Guck, J., Sivaniah, E. 3D inverted colloidal crystals in realistic cell migration assays for drug screening applications. Integr Biol. 3, 1202-1206 (2011).
- da Silva, J., Lautenschlager, F., Sivaniah, E., Guck, J. R. The cavity-to-cavity migration of leukaemic cells through 3D honey-combed hydrogels with adjustable internal dimension and stiffness. Biomaterials. 31, 2201-2208 (2010).
- Lee, J., Lilly, G. D., Doty, R. C., Podsiadlo, P., Kotov, N. A. In vitro toxicity testing of nanoparticles in 3D cell culture. Small. 5, 1213-1221 (2009).
- Lee, J., Kotov, N. A. Notch ligand presenting acellular 3D microenvironments for ex vivo human hematopoietic stem-cell culture made by layer-by-layer assembly. Small. 5, 1008-1013 (2009).
- Liu, Y., et al. Rapid aqueous photo-polymerization route to polymer and polymer-composite hydrogel 3D inverted colloidal crystal scaffolds. J Biomed Mater Res. Part A. 83, 1-9 (2007).
- Ma, P. X., Choi, J. W. Biodegradable polymer scaffolds with well-defined interconnected spherical pore network. Tissue Eng. 7, 23-33 (2001).
- Cuddihy, M. J., Kotov, N. A. Poly (lactic-co-glycolic acid) bone scaffolds with inverted colloidal crystal geometry. Tissue Eng Part A. 14, 1639-1649 (2008).
- Choi, S. W., Zhang, Y., Xia, Y. Three-dimensional scaffolds for tissue engineering: the importance of uniformity in pore size and structure. Langmuir. 26, 19001-19006 (2010).
- Choi, S. W., Zhang, Y., Thomopoulos, S., Xia, Y. In vitro mineralization by preosteoblasts in poly(DL-lactide-co-glycolide) inverse opal scaffolds reinforced with hydroxyapatite nanoparticles. Langmuir. 26, 12126-12131 (2010).
- Choi, S. W., Zhang, Y., Macewan, M. R., Xia, Y. Neovascularization in biodegradable inverse opal scaffolds with uniform and precisely controlled pore sizes. Adv Healthc Mater. 2, 145-154 (2013).
- Zhang, Y., Choi, S. W., Xia, Y. Modifying the Pores of an Inverse Opal Scaffold With Chitosan Microstructures for Truly Three-Dimensional Cell Culture. Macromol Rapid Commun. 33, 296-301 (2012).
- Cai, X., et al. Investigation of neovascularization in three-dimensional porous scaffolds in vivo by a combination of multiscale photoacoustic microscopy and optical coherence tomography. Tissue Eng. Part C, Meth. 19, 196-204 (2013).
- Zhang, Y. S., Yao, J., Wang, L. V., Xia, Y. Fabrication of Cell Patches Using Biodegradable Scaffolds with a Hexagonal Array of Interconnected Pores (SHAIPs). Polymer. 55, 445-452 (2014).
- Zhang, Y. S., Regan, K. P., Xia, Y. Controlling the Pore Sizes and Related Properties of Inverse Opal Scaffolds for Tissue Engineering Applications. Macromol Rapid Commun. 34, 485-491 (2013).
- Stachowiak, A. N., Bershteyn, A., Tzatzalos, E., Irvine, D. J. Bioactive Hydrogels with an Ordered Cellular Structure Combine Interconnected Macroporosity and Robust Mechanical Properties. Adv Mater. 17, 399-403 (2005).
- Stachowiak, A. N., Irvine, D. J. Inverse opal hydrogel-collagen composite scaffolds as a supportive microenvironment for immune cell migration. J Biomed Mater Res. Part A. 85, 815-828 (2008).
- Liu, Y., Wang, S. 3D inverted opal hydrogel scaffolds with oxygen sensing capability. Colloids and surfaces. B, Biointerfaces. 58, 8-13 (2007).
- Bryant, S. J., Cuy, J. L., Hauch, K. D., Ratner, B. D. Photo-patterning of porous hydrogels for tissue engineering. Biomaterials. 28, 2978-2986 (2007).
- Bhrany, A. D., Irvin, C. A., Fujitani, K., Liu, Z., Ratner, B. D. Evaluation of a sphere-templated polymeric scaffold as a subcutaneous implant. JAMA facial plastic surgery. 15, 29-33 (2013).
- Kuo, Y. C., Chiu, K. H. Inverted colloidal crystal scaffolds with laminin-derived peptides for neuronal differentiation of bone marrow stromal cells. Biomaterials. 32 (3), 819-831 (2011).
- Yang, J. T., Kuo, Y. C., Chiu, K. H. Peptide-modified inverted colloidal crystal scaffolds with bone marrow stromal cells in the treatment for spinal cord injury. Colloids Surf. B, Biointerfaces. 84, 198-205 (2011).
- Kuo, Y. C., Tsai, Y. T. Inverted colloidal crystal scaffolds for uniform cartilage regeneration. Biomacromolecules. 11, 731-739 (2010).
- Choi, S. W., Xie, J., Xia, Y. Chitosan-Based Inverse Opals: Three-Dimensional Scaffolds with Uniform Pore Structures for Cell Culture. Adv Mater. 21, 2997-3001 (2009).
- Long, T. J., Sprenger, C. C., Plymate, S. R., Ratner, B. D. Prostate cancer xenografts engineered from 3D precision-porous poly(2-hydroxyethyl methacrylate) hydrogels as models for tumorigenesis and dormancy escape. Biomaterials. 35, 8164-8174 (2014).
- Kuo, Y. C., Tsai, Y. T. Inverted colloidal crystal scaffolds for uniform cartilage regeneration. Biomacromolecules. 11, 731-739 (2010).
- Kuo, Y. C., Chiu, K. H. Inverted colloidal crystal scaffolds with laminin-derived peptides for neuronal differentiation of bone marrow stromal cells. Biomaterials. 32, 819-831 (2011).
- Lee, J., Cuddihy, M. J., Cater, G. M., Kotov, N. A. Engineering liver tissue spheroids with inverted colloidal crystal scaffolds. Biomaterials. 30 (27), 4687-4694 (2009).
- Galperin, A., et al. Integrated bi-layered scaffold for osteochondral tissue engineering. Adv Healthc Mater. 2, 872-883 (2013).
- Waters, D. J., et al. Morphology of Photopolymerized End-linked Poly(ethylene glycol) Hydrogels by Small Angle X-ray Scattering. Macromolecules. 43 (16), 6861-6870 (2010).
- Elbert, D. L., Hubbell, J. A. Conjugate addition reactions combined with free-radical cross-linking for the design of materials for tissue engineering. Biomacromolecules. 2 (2), 430-441 (2001).
- Kim, M. H., et al. Biofunctionalized Hydrogel Microscaffolds Promote Three-Dimensional Hepatic Sheet Morphology. Macromol Biosci. , (2015).
- Ferreira, T., Rasband, W. ImageJ User Guide. , http://imagej.nih.gov/ij/docs/guide/146-30.html#toc-Subsection-30.1 (2012).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to Fluorescence Microscopy. , JoVE. Cambridge, MA. (2015).
- Tominaga, H., et al. A water-soluble tetrazolium salt useful for colorimetric cell viability assay. Anal Commun. 36 (2), 47-50 (1999).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to the Microplate Reader. , JoVE. Cambridge, MA. (2015).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The ELISA Method. , JoVE. Cambridge, MA. (2015).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat Protoc. 1, 1559-1582 (2006).
- JoVE Science Education Database. Essentials of Environmental Microbiology. RNA Analysis of Environmental Samples Using RT-PCR. , JoVE. Cambridge, MA. (2016).
- JoVE Science Education. Essentials of Environmental Microbiology. , JoVE. (2015).
- Jeong, S., et al. The evolution of gene regulation underlies a morphological difference between two Drosophila sister species. Cell. 132 (5), 783-793 (2008).
- Griffith, L. G., Naughton, G. Tissue engineering--current challenges and expanding opportunities. Science. 295 (5557), 1009-1014 (2002).
- Hegde, M., et al. Dynamic Interplay of Flow and Collagen Stabilizes Primary Hepatocytes Culture in a Microfluidic Platform. Lab Chip. 14, 2033-2039 (2014).
- Kim, Y., Lasher, C. D., Milford, L. M., Murali, T., Rajagopalan, P. A comparative study of genome-wide transcriptional profiles of primary hepatocytes in collagen sandwich and monolayer cultures. Tissue Eng Pt C. 16 (6), 1449-1460 (2010).
- Baimakhanov, Z., et al. Efficacy of multi-layered hepatocyte sheet transplantation for radiation-induced liver damage and partial hepatectomy in a rat model. Cell Transplant. , (2015).
- Li, C. Y., et al. Micropatterned Cell-Cell Interactions Enable Functional Encapsulation of Primary Hepatocytes in Hydrogel Microtissues. Tissue Eng Pt A. 20 (15-16), 2200-2212 (2014).
- Shlomai, A., et al. Modeling host interactions with hepatitis B virus using primary and induced pluripotent stem cell-derived hepatocellular systems. P Natl A Sci USA. 111 (33), 12193-12198 (2014).
- Curcio, E., et al. Mass transfer and metabolic reactions in hepatocyte spheroids cultured in rotating wall gas-permeable membrane system. Biomaterials. 28, 5487-5497 (2007).
- Martinez-Hernandez, A., Amenta, P. The hepatic extracellular matrix. Vichows Archiv A Pathol Anat. 423, 1-11 (1993).
- Liu, Y., Wang, S., Lee, J. W., Kotov, N. A. A Floating Self-Assembly Route to Colloidal Crystal Templates for 3D Cell Scaffolds. Chem Mater. 17 (20), 4918-4924 (2005).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon