É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Fabricação de Inverted coloidal de cristal poli (etileno-glicol) Andaime: Uma Plataforma Cultura celular tridimensional para Liver Engenharia de Tecidos
Neste Artigo
Resumo
This manuscript presents a detailed protocol for the fabrication of an emerging three-dimensional hepatocyte culture platform, the inverted colloidal crystal scaffold, and the concomitant techniques to assess hepatocyte behavior. The size-controllable pores, interconnectivity and ability to conjugate extracellular matrix proteins to the poly(ethylene glycol) (PEG) scaffold enhance Huh-7.5 cell performance.
Resumo
A capacidade para manter a função de hepatócitos in vitro, com a finalidade de testar a citotoxicidade 'xenobióticos, estudar a infecção por vírus e o desenvolvimento de drogas orientadas para o fígado, exige uma plataforma em que as células recebem sinais bioquímicos e mecânicos adequados. sistemas recentes de engenharia de tecidos do fígado têm empregado tridimensionais andaimes (3D) compostas de hidrogéis sintéticas ou naturais, dada a sua alta capacidade de retenção de água e sua capacidade de fornecer os estímulos mecânicos necessários pelas células. Tem havido um interesse crescente no andaime invertida cristal coloidal (ICC), um desenvolvimento recente, que permite alta organização espacial, homot�ica e interação célula heterotípica, bem como matriz (ECM) interação célula-extracelular. Aqui, nós descrevemos um protocolo para fabricar o andaime ICC usando poli diacrilato (etileno glicol) (PEGDA) eo método de partículas de lixiviação. Resumidamente, uma estrutura é feita a partir de partículas de microsferas, após o qual uma pré-polymesolução R é adicionada, adequadamente polimerizadas, e as partículas são, então, removido, ou lixiviado, utilizando um solvente orgânico (por exemplo, tetra-hidrofurano). A dissolução dos resultados grades de um andaime altamente porosa com tamanhos de poro controlado e interconnectivities que permitem que os meios para a obtenção de células mais facilmente. Esta estrutura única permite que a área de superfície elevada para as células a aderir bem como a comunicação fácil entre poros, e a capacidade para revestir o andaime ICC PEGDA com proteínas também mostra um efeito significativo no desempenho da célula. Analisamos a morfologia do suporte, bem como a célula hepatocarcinoma comportamento (Huh-7,5), em termos de viabilidade e função para explorar o efeito da estrutura ICC e revestimentos de ECM. No geral, este artigo fornece um protocolo detalhado de um andaime emergente que tem larga aplicação em engenharia de tecidos, especialmente no fígado engenharia de tecidos.
Introdução
O fígado é um órgão altamente vascularizado com uma multiplicidade de funções, incluindo a desintoxicação do sangue, metabolismo de xenobióticos, e a produção de proteínas séricas. O tecido de fígado tem uma microestrutura complexa tridimensional (3D), que compreende de vários tipos de células, canalículos biliares, sinusóides, e zonas de composição biomatriz diferente e diferentes concentrações de oxigénio. Dada esta estrutura elaborada, tem sido difícil criar um modelo de fígado adequada in vitro 1. No entanto, há uma crescente demanda por funcional em modelos in vitro hospedagem hepatócitos humanos como plataformas para ensaio da toxicidade da droga 2 e estudo de doenças associadas ao fígado 3.
Plataformas de engenharia de tecidos de fígado actual simplificaram a complexidade do fígado através do isolamento de um, ou concentrando-se em alguns, dos parâmetros do fígado, isto é, a co-cultura de células de 4 composição, bioquímica da Zonal microambientes 5, dinâmica de fluxo de 6,7 e a configuração da Biomatriz 8. Configuração da biomatriz pode ser dividida em parâmetros, tais como materiais de andaime, a composição de proteínas da matriz extracelular (ECM), a rigidez da matriz, bem como da concepção e estrutura do andaime. Houve um aumento nos estudos de engenharia de tecidos utilizando hidrogeles sintéticos, especialmente poli (etilenoglicol) (PEG) hidrogéis 9, dada a capacidade de ajustar as propriedades, bioactividade, e taxa de degradação mecânica do hidrogel. Em relação à investigação relacionada com o fígado, o hidrogel biocompatível foi aplicado para estudar a infecção pelo vírus da doença hepática 3. Como um projeto de plataforma de hepatócitos, numerosos estudos têm utilizado hepatócitos sanduíche culturas 10,11 e encapsulamento de células dentro de um hidrogel 12,13 para proporcionar o ambiente 3D e célula-ECM e interação célula-célula, que são essenciais para imitar no microambiente vivo. However, essas plataformas não possuem um alto grau de controle e organização espacial, levando a propriedades não uniformes através do andaime 14.
O coloidal cristal invertido (ICC) 14 scaffold é um arcabouço 3D altamente organizada para cultura de células, que foi introduzido pela primeira vez no início de 2000. estrutura única do andaime pode ser atribuída ao processo de fabricação simples utilizando um cristal coloidal, uma estrutura ordenada de partículas coloidais de diâmetro variável. Resumidamente, para resumir o processo, as partículas são dispostas ordenadamente e recozidos usando o calor para formar uma rede. A lixiviação desta estrutura, por um solvente orgânico, em um polimerizados resultados de hidrogel em cavidades esféricas hexagonal embalados 15 com área de superfície elevada. Este andaime altamente ordenada tenha sido previamente feito com ambos os materiais sintéticos e naturais, incluindo, mas não limitados a poli (acrilamida) 16-21, poli (ácido láctico-co-ácido glicólico) 15,22-30, Poli (etileno-glicol) 31,32, poli (metacrilato de 2-hidroxietilo) 21,33-35, e quitosano 36-39. Andaimes ICC feitas de materiais não-incrustantes tendem a promover esferóides celulares no interior das cavidades 14,23,40. Vários tipos de células têm sido mostrados para proliferar com sucesso, diferenciação e função dentro desta configuração, incluindo condrócitos 41, células do estroma da medula óssea 42, e as células-tronco 43,44. Em relação hepatócito, estudos têm sido realizados com andaimes ICC feitas de Na 2 SiO 3 e poli (acrilamida), mas não PEG. Com estratégias bioconjugação simples (ou seja, o acoplamento de amina através EDC / NHS), scaffolds à base de PEG ECM proteínas conjugadas podem ser fabricados, que podem revelar-se locais de ligação mais celulares para ser um mais in vivo, como o ambiente e melhorar a função hepática.
Neste manuscrito e o vídeo associado, detalhamos a fabricação do andaime ICCusando poli (etileno-glicol) diacrilato de hidrogel (PEGDA) e um látice de poliestireno microesfera, optimizado para hepatocarcinoma (Huh-7,5) cultura. Nós demonstrar as diferenças entre os nus andaimes ICC PEGDA geralmente não aderentes eo PEGDA ICC andaime revestido de colágeno em termos de topologia de andaime e desempenho celular. viabilidade e função celular são medidos qualitativa e quantitativamente avaliar o comportamento das células Huh-7.5.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. ICC andaime Fabrication (Figura 1)
- Prepare o poliestireno (PS) reticulados (diâmetro = 6 mm; 8-13 camadas de grânulos).
- Para preparar o molde, corte as pontas off de 0,2 ml tubos de microcentrífuga à prova de fervura no nível de 40 mL. Aderir ao topo das cut-tubos de 24 x 60 milímetros 2 microscópio lamelas de vidro com cola à prova de água.
- Coloque as esferas PS (diâmetro = 140 um) contidos dentro de uma suspensão de água em um frasco de 20 ml, cuidadosamente pipetar a suspensão em água, e adicionar 18 ml de solução a 70% de etanol para dentro do frasco. Coloque a solução esfera em um banho ultra-sônico para soltar esferas agregados. Repetir esta etapa de lavagem várias vezes a fim de remover componentes de água e solúveis em água completamente.
- Pipetar 100 l de etanol para dentro dos moldes.
- Cortar a parte superior de uma ponta de micropipeta de 200 ul por 4 mm. Pipetar 25 ul das esferas para dentro do molde, duas vezes usando 200 ul micropipeta para atingir umvolume total de 50 ul em cada molde.
- Coloque os moldes em um shaker balançando a 120 durante a noite rpm.
- Verificar o arranjo das esferas em cada molde sob um microscópio óptico. Se as esferas não são ordenados hexagonal, adicionar 50 ul de etanol a 70% e agitar manualmente na direcção do eixo longitudinal e lateral para corrigir o arranjo.
- Deixe a evaporar-se o etanol à temperatura ambiente (RT) durante duas noites. Coloque o molde e complexo grânulo em um forno de 130 ° C durante 6 horas para recozer os grânulos PS.
- Preparando andaimes PEGDA nuas e revestidas de ECM.
- Sintetizar macr�eros PEGDA usando protocolos estabelecidos 45,46 para acrylating macr�eros PEG lineares (M w = 4,6 kDa).
- Prepare a 50% (w / v), solução em PEGDA (DI) de água ionizada-de e permitir que o macrómero para dissolver adequadamente por centrifugação a 4713 xg até que esteja completamente dissolvido.
- Para ECM conjugado andaimes ICC, dissolver um adicional10% (w / v) de acriloilo-PEG-NHS (M w = 3,4 kDa) na solução PEGDA 50%.
- Prepara-se uma a 20% (w / v) de solução de reserva de 2-hidroxi-4 '- (2-hidroxietoxi) -2-metilpropiofenona (PI) em 70% de etanol.
- Adicionar 50 ul de 20% (w / v) de solução de estoque de PI por 1 ml de 50% (w / v) de PEGDA. Ajustar a quantidade necessária de solução-mãe de PI com base no peso molecular de PEGDA.
- Vortex da mistura no tubo de centrífuga de 1 min para chegar a uma solução homogénea.
- Descasque os moldes da lâmina de vidro (do passo 1.1.7), remover a cola dos moldes, empurre os reticulados cuidadosamente com uma espátula e coloque cada um deles em um tubo de 1,5 ml. Pipetar 300 ul da solução PEGDA e centrifugar a 845 xg durante 5 min para permitir a infiltração de solução PEGDA adequada no reticulado.
- Remova a estrutura do tubo, usando uma pinça e secar cuidadosamente solução PEGDA excesso de seca em luvas. Coloque a estrutura em um vidro revestido por uma película de parafina com o cir planasuperfície cular voltado para cima.
- Expor a solução PEGDA andaime infiltrado a 365 nm, radiação ultravioleta (UV) (10,84 mW / cm 2) durante 5 min, utilizando uma lâmpada de UV local.
- Coloque redes cristalinas PEGDA-polimerizados em novos tubos de ensaio (cerca de 10 reticulados por frasco) e adicionar 20 ml de tetra-hidrofurano (THF). Agitar os frascos num agitador orbital a 300 rpm. Mudar THF, pelo menos, 3 vezes com um intervalo de 1-2 horas.
Nota: Não remover o THF completamente ao mudar-se o THF, a fim de evitar que as bolhas de entrar nos andaimes, que por sua vez pode causar a remoção incompleta de PS. Deixar solução suficiente para cobrir as treliças e adicionar novo THF.
Cuidado: THF é tóxico. Usar luvas, jaleco e óculos de proteção. Evitar a inalação, operando sob a coifa. - Verifique se esferas PS são dissolvidos, colocando água na solução THF utilizado e observando a cor da solução. Repita o passo 1.2.9 Se as esferas PS não são devidamente dissolvido.
Nota: A cor da solução mudará parabranco se existem esferas PS restantes.
- Limpe os suportes na cabine de segurança biológica (BSC).
- Para esterilizar os andaimes, preparar um tubo de centrífuga de 50 ml com 2 ml de 70% de etanol por andaime e colocar andaimes no tubo usando uma espátula. Permitir que os andaimes para mergulhar em etanol durante 1 hora. A partir deste passo, a todos os procedimentos no BSC.
- Cuidadosamente verter a etanol e substituir com tampão de fosfatos salino (PBS) (2 ml por andaime) e centrifugar a 524 xg durante 3 minutos para remover as bolhas. Mantê-lo na geladeira e mudar o PBS algumas vezes com um intervalo de 1-2 horas.
- Para o tipo de colágeno andaimes I-revestido, preparar um outro tubo de centrífuga de 50 ml contendo colágeno solução de tipo 1 (1 ml por scaffold), transferir os andaimes esterilizados a este tubo usando uma espátula, e centrifugar a 524 xg por 3 min. Agitar os andaimes a 400 rpm num agitador orbital durante 30 min e manter o tubo na refrigerou durante a noite.
- Lavar os andaimes com PBS duas vezes antes de usar, submergindo os andaimes em PBS fresco e, em seguida, aspirar o PBS.
Nota: Outras proteínas de ECM também pode ser usado em vez de colagénio tipo I, porque a química de NHS requer um grupo amina para formar a ligação (Figura 2).
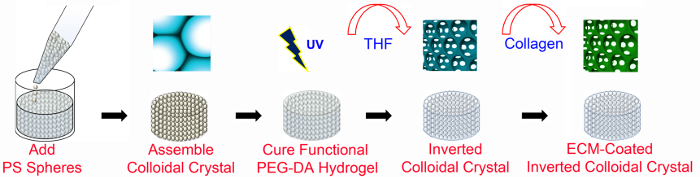
Figura 1. Visão geral da fabricação ICC. Andaimes CCI, sediada em PEG são fabricados usando técnicas de microfabricação com e sem ECM-funcionalização. Andaimes ICC revestidos por ECM requerem PEG-NHS, bem como PEGDA (conforme detalhado na Figura 2). A rede PS tem um diâmetro de 6 mm e uma altura de 8-13 camadas do grânulo. PS, poliestireno; PEGDA, poli (etileno-glicol) diacrilato; UV, ultravioleta; THF, tetra-hidrofurano; ECM, matriz extracelular. Esta figura foi modificada e usada com autorização de Wiley 47. Por favor clique aqui para ver uma versão maior desta figura.
Caracterização Estrutura 2. ICC
- Para analisar a estrutura ICC com ou sem proteínas conjugadas, usar microscopia eletrônica de varredura (SEM) 47.
- Corrigir os andaimes com paraformaldeído a 4% (PFA), desidratar em série-los em 25, 50, 75, 95 e 100% de etanol soluções, e armazená-los a -80 ° C até que o etanol evaporar totalmente.
- As amostras secas em um secador por congelação durante 48 horas.
- Apor a amostra no suporte da amostra usando a fita de carbono e coloque em um revestidor por crepitação.
- Após aspiração automática, bata-a com um filme Pt de 10 nm de espessura por pulverização catódica por 60 seg a 20 mA.
- Andaimes ICC imagem utilizando SEM a uma voltagem de 5 kV (Figura 3A, a Figura 4A).
- Para medir o poro ediâmetro interligação das cavidades, analisar micrografias utilizando imagem software de análise 48 (por exemplo, ImageJ; Figura 3B, C).
- Para visualizar o colagénio conjugado com o andaime sem células, marcação por fluorescência do colagénio utilizando anticorpos (1: 100) contra o colagénio tipo I e com imagem de microscopia confocal de varrimento laser 47 (CLSM; Figura 4B).
3. Huh-7.5 Cultura de Células e Sementeira
- Cultura Huh-7,5 células a uma densidade de sementeira de 2-2,5 x 10 6 culas / ml em placas de cultura de células de 100 mm com 10 ml de Dulbecco meio de Eagle modificado (DMEM) suplementado com 10% de soro fetal de bovino (FBS) e 100 U / ml penicilina-estreptomicina (meio de crescimento) a 37 ° C e 5% de CO 2. Alterar os meios de comunicação a cada três dias no BSC até que tenham atingido 75-80% de confluência.
- Prepare os andaimes para a semeadura célula do BSC.
- Com cuidado, coloque o scaffidade em uma placa de 24 poços, com a superfície plana virada para cima.
- Para lavar o andaime, pipeta de 2 ml PBS a cada poço contendo um andaime. Aspirar o PBS e pipeta de 2 ml de PBS fresco em cada poço.
- Aspirar o PBS e pipeta de 2 ml de meio de crescimento (veja o passo 3.1) e deixe por 30 minutos. Aspirar os meios de comunicação e permitir que o andaime a secar durante 1 hora.
- Retire confluentes células Huh-7,5 (a partir do passo 3.1) da placa de cultura no BSC utilizando o método de digestão com tripsina.
- Aspirar meios de placa, adicionar 4 ml de PBS para lavar as células aderentes e depois aspirar PBS.
- Pipeta 0,75-1 ml de 0,25% de tripsina e colocar numa incubadora a 37 ° C, 5% de CO 2 durante 3 min.
- Retire a placa da incubadora e pipeta de 5 ml de mídia para parar a reacção tripsina. media Pipeta, células isoladas, e uma mistura de tripsina em um tubo de 15 ml.
- Centrifuga-se a 524 xg durante 3 minutos, remover o sobrenadante e ressuspender o sedimento em 5 ml de meio.
- Contagem de células utilizando um hemocitómetro e calcular o volume da suspensão de células que contém células no número de destino, N 0, por 25 ul (para a experiência padrão, n é 0 1 x 10 6 células).
O volume de suspensão de células = (número de células alvo) / (concentração de suspensão de células) - Lentamente Pipetar 25 ul de suspensão de células (células contendo N 0) directamente na parte superior de cada escora (a partir do passo 3.1.4). Colocar a placa de 24 poços na incubadora.
- Após 12 horas, transferir cuidadosamente os suportes com o uso de uma espátula para uma nova placa de 24 poços e pipeta de 2 ml de meio em cada poço. Colocar a placa de 24 poços na incubadora.
- Alterar os meios de comunicação a cada 3 dias ou dependendo quando a mídia é coletada para análise de secreção de proteínas (ver passo 5.1).
Viabilidade 4. celular
- Para analisar qualitativamente a viabilidade celular, use vivo kits de coloração fluorescentes / mortas para corar as células e imagem usando CLSM.
- Seguindo instruções do kit, preparar uma solução com 4 mm AM calceína e 8 mM de etídio homo-dímero-1 em mídia (veja o passo 3.1.3).
Nota: Optimizar dependendo do número de células. Use o dobro da quantidade de reagente se as células proliferam e dupla em número (cerca de 2 milhões). - No BSC, meios de aspirado de cada poço com um andaime (passo 3.7) e pipeta 500 uL da solução preparada. Incubar as amostras em uma incubadora a 37 ° C durante 1 h.
- Cobrir as placas em papel de alumínio para proteger as amostras da luz ao remover a placa fora da incubadora. amostras de imagem usando CLSM49.
- Seguindo instruções do kit, preparar uma solução com 4 mm AM calceína e 8 mM de etídio homo-dímero-1 em mídia (veja o passo 3.1.3).
- Para avaliar quantitativamente a viabilidade celular, medir a actividade enzimática (em células vivas) utilizando ensaios colorimétricos 50 (isto é, 2- (2-metoxi-4-nitrofenil) -3- (4-nitrofenil) -5- (2,4-dissulfofenil) 2H-tetrazólio (reagente monossódico-sal)).
- Criar uma curva padrão para a plataforma TPI (isto é, um gráfico da absorvância (OD) versus determinado número de células).
Nota: sementeira celular não é 100% eficiente em comparação com outras plataformas, uma vez que as células podem passar através das cavidades de andaimes.- Determinar o número de células de sementeira, n 0 que vai ser utilizado para fazer a curva padrão.
Nota: Escolha um intervalo que inclui o número de células que são estimados no experimento. Por exemplo, se o número de células inicial é de 5 x 10 5 células e há uma estimativa de vezes de aumento ~ 3 até ao último dia da experiência, escolher 2,5 x 10 5, 5 x 10 5, 1 x 10 6, e 2 x 10 6 células como N 0. - Realizar a sementeira de células nos andaimes ICC (um N 0 por volume de andaime com sementeira de 25 ul) tal como descrito nos passos 3.1-3.6.
- Após 6 horas, transferir os andaimes para outra placa de 24 poços bem. Escolha um tempo que permite aderência celular, mas não proliferação celular.
- Realizar contagem de células no cadafalso ICC transferidos.
- Diluir 10x monossódico-sal reagente solução (MSR) para 1x com a mídia no BSC e pipeta de 500 mL solução 1x MSR em cada poço com um andaime. Incubar a placa de 24 poços a 37 ° C durante 1 h.
- De cada poço, transferência de 100 ul numa placa de 96 poços bem. Como uma peça em bruto, pipeta 100 uL da solução de reagentes 1x monossódico-sal fresco nos diferentes poços da placa de 96 poços. remover manualmente as bolhas existentes usando uma ponteira seca e cobrir a placa de 96 poços em papel alumínio para protegê-lo da luz.
- Medida OD a λ = 450 nm utilizando um espectrofotómetro de leitura. Subtrair o OD em branco de outros valores para encontrar a OD precisos 51.
- Contar o número de células (N G) remanescente no poço após a transferência do andaime (passo 4.2.1.3), utilizando um hemocitómetro.
Nota: Use 300 ul de tripsina a trypsinize as células. - Calcular o número de células actual, n a.
= reaisinicial - saiu-se bem
Um N = N 0 L -N - Adicione curva padrão através da representação gráfica OD obtido no Passo 4.2.1.4.3 versus o número de células actual (N A) e utilizar isto para estimar o número de células nas experiências.
Nota: Faça uma nova curva padrão, se nenhum parâmetro ICC (ou seja, o tamanho agente porog�ico, dimensões do andaime, proteína ECM, etc.) são alteradas.
- Determinar o número de células de sementeira, n 0 que vai ser utilizado para fazer a curva padrão.
- Criar uma curva padrão para a plataforma TPI (isto é, um gráfico da absorvância (OD) versus determinado número de células).
Função 5. celular
- Analisar a secreção de proteínas pelas células Huh-7,5 (ou seja, albumina, ureia) a partir dos meios colhidos a partir da etapa (3.7) por ensaio de imunoabsorção enzimática (ELISA) 52.
Nota: Dilui-se a meios, dependendo do número de células semeadas e a quantidade de material recolhido. Para 5 x 10 5 células semeadas em ICC, usar um ~ proporção de 1: 25, antes de introduzi-lo aos poços pré-revestidos de anticorpos de. - Para analisar qualitativamente a função celular, imunocoloração proteínas intracelulares específicos(Ou seja, albumina), enzimas (ou seja, do CYP450), componentes estruturais mancha (ou seja, de actina celular), bem como os núcleos e imagem usando CLSM 49.
- media aspirado (do passo 3.7) e pipeta de 2 ml PBS para lavar os andaimes ICC células-laden.
- Pipete 1 mL de PFA a 4% e incubar durante 5 min à temperatura ambiente, durante a fixação.
- Lavar 3x com 2 ml de PBS.
- Permeabilizar as membranas por incubação dos andaimes em 1 ml de 0,1% de 4- (1,1,3,3-tetrametilbutil) fenil-polietileno-glicol (agente tensioactivo) durante 30 min.
- Lavar 3x com 2 ml de PBS para remover quaisquer proteínas de vazamento.
- Pipetar 500 ul 1% de albumina de soro bovino (BSA) e incubar a temperatura ambiente durante 1 h para bloquear a ligação não específica.
- Prepare anticorpo primário diluído (ou seja, albumina, CYP450) solução.
- Pipetar 500 l de solução de BSA a 1% em um tubo de 15 mL e adicionar 4,5 mL de 0,1% solução de agente tensioactivo para preparar uma 5 ml de 0,1% de BSA solução total.
- Pipetar 98 ul de solução de BSA a 0,1% para um tubo de microcentrífuga de 200 uL e 2 ul do anticorpo primário para produzir um 01:50 (anticorpo primário: 0,1% de BSA) solução de anticorpo primário.
- Pipetar 40 ul de solução de anticorpo primário sobre o andaime e cobrir o substrato com a película de parafina. Envolva a placa de 24 poços com folha de alumínio e armazenar o prato a 4 ° C durante a noite.
- Lavar 3x com 2 ml PBS e agitar a placa suavemente no meio de lavagem.
- Prepare anticorpo secundário biotinilado diluído (ou seja, anticorpo anti-ratinho) solução estoque.
- Pipeta solução de BSA a 198 ul de 0,1% e 2 ul segundo anticorpo para produzir uma mistura 1: (anticorpo secundário: 0,1% de BSA) 100 A solução de anticorpo secundário.
- Prepara-se uma solução de estoque de 0,1% de rodamina faloidina marcada com fluoresceína ou (para corar os filamentos de actina celular) em solução de BSA a 0,1%.
- Pipetar 25 ul de cada solução num tubo e misturar Well.
- Pipetar 50 ul de solução-mãe de anticorpo secundário sobre o andaime. Cobrir o andaime com película de parafina, enrole o prato com folha de alumínio e armazenar a temperatura ambiente por 2 horas.
- Lavar 3x com 2 ml de PBS.
- Pipete 200 pi de 0,2% 4'-6-diamidino-2-fenilindole (DAPI; uma mancha núcleo) solução no andaime e manter à temperatura ambiente durante 2-3 minutos. Cubra o prato com papel alumínio.
- Lavar 2x com 2 ml de PBS.
- Usando um conta-gotas, colocar uma gota de meio de montagem sobre o substrato.
- Cuidadosamente coloque o andaime numa lâmina de vidro e imagem usando CLSM 47.
- Avaliar a expressão do gene por reacção em cadeia da polimerase em tempo real (qPCR). Use kits padrão para PCR transcriptase reversa (RT-PCR) 53 e qPCR 54, conforme as instruções do fabricante. Extrai-se a ARN a partir das células, tal como descrito abaixo.
- Colocar o andaime (a partir do passo 3,7) num tubo de microcentrífuga de 1,5 ml.
- Pipeta de 1 ml de uma solução de extração de RNA para dentro do tubo e manter em um sonicador por 5 min a RT.
- Pipetar 200 mL de clorofórmio em cada tubo de microcentrífuga e agitar o tubo vigorosamente na mão por 15-20 seg. Manter os tubos à temperatura ambiente durante ~ 3 min, até à separação das fases.
- Centrifugar a amostra a 13000 xg, a 4 ° C durante 15 min e remover os tubos com cuidado de modo que as fases não se misturam.
- Cuidadosamente pipetar 500-600 ul da fase aquosa superior a partir do primeiro tubo num segundo tubo de microcentrífuga.
- Adicionar um volume equivalente (500-600 uL) de isopropanol para este segundo tubo.
- Inverter o tubo 3-5 vezes e deixar o tubo que está à temperatura ambiente durante 10 min.
- Centrifugar a amostra a 13000 xg, a 4 ° C durante 15 min.
- Se uma pastilha não é visível na parte inferior do tubo, de centrifugação novamente durante 5 min.
Nota: Se ainda não existe sedimento visível, a quantidade de RNA pode ser insuficiente.
- Se uma pastilha não é visível na parte inferior do tubo, de centrifugação novamente durante 5 min.
- Eutubos nvert com a tampa aberta para descartar o sobrenadante e pipeta de 1 ml de etanol a 70% diluído em água com DEPC para dentro do tubo.
- Ligeiramente vortex o tubo de modo que o sedimento se desprende da parede do tubo e deixe o ar seco tubo.
- Adicionar 50 ul de água DPEC para ressuspender o sedimento.
- Mantenha pipetagem até dissolver o pellet.
- Manter durante 10 min a 55 ° C, de modo a desnaturar o ARN de cadeia dupla em RNA de cadeia simples.
- Levemente tambor dedos sobre o fundo do tubo e, em seguida, centrifugar os tubos momentaneamente (7500 xg, 4 min, 4 ° C).
- Manter os tubos em gelo até à realização da transcriptase inversa 55 e PCR em tempo real, tal como descrito em 56.
Access restricted. Please log in or start a trial to view this content.
Resultados
Os resultados representativos para a caracterização estrutural do andaime ICC ea comparação da eficácia de cada condição de andaime ICC em cultura de hepatócitos são mostrados e explicados abaixo. As condições ICC andaime utilizados nestes resultados são revestimentos de colagénio de 0 ug / ml (sem isolamento), 20 ug / ml (colágeno 20), 200 ug / ml (colágeno 200), e 400 ug / ml (colágeno 400) e o inicial huh-7.5 número de semeadura de células é 1x10 6.
<...Access restricted. Please log in or start a trial to view this content.
Discussão
Andaimes de engenharia de tecidos estão a evoluir rapidamente para fornecer todas as pistas físicas e bioquímicas necessárias para regenerar, manter ou reparar tecidos para a aplicação da substituição de órgãos, estudando a doença, o desenvolvimento de drogas, e muitos outros 57. Na engenharia de tecidos do fígado, os hepatócitos primários humanos rapidamente perdem suas funções metabólicas uma vez isolados do corpo, criando uma grande necessidade de andaimes de engenharia e desenvolvimento d...
Access restricted. Please log in or start a trial to view this content.
Divulgações
The authors have no competing financial interests to disclose.
Agradecimentos
Os autores gostariam de agradecer o apoio de um Research Foundation Fellowship Nacional (NRF -NRFF2011-01) e Competitiva Programa de Pesquisa (NRF-CRP10-2012-07).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.2 ml PCR tube | Axygen Scientific | PCR-02D-C | Boil-proof |
| Gorilla Glue | Gorilla Glue, Inc. | Depends on vendor. This was purchased from a local store. | |
| Glass slides | VWR | 631-1575 | Dimensions: 24×60 mm2 |
| Polystyrene spheres | Fisher Scientific | TSS#4314A | Diameter = 140 µm; 3x104 particles per milliliter and 1.4% size distribution |
| Ethanol | Merck | 1.00983.1011 | absolute for analysis EMSURE; Dilute to 70% with Milli-Q water |
| Ultrasonic Bath | Elma | S10H | Equiment |
| Furnace | Nabertherm | N7/H | Equipment |
| 200 µl pipette tip | Axygen Scientific | T-210-Y-R-S | |
| Rocking shaker | VWR | 444-0142 | |
| Polyethylene Glycol (PEG) | Merck | 1.09727.0100 | Mw= 4 kDa; acrylation of PEG monomers and purification of the resulting precipitate produces a PEGDA macromer with Mw = 4.6 kDa |
| Centrifuge | Beckman Coulter | 392932 | Equipment |
| Acrylate-Poly(Ethylene Glycol) - Succinimidyl Valerate | Laysan Bio | ACRL-PEG-SVA-3400-1g | Mw = 3.4 kDa |
| 2-hydroxy-4'-(2-hydroxyethoxy)-2-methylpropiophenone | Sigma Aldrich | 410896 | |
| Vortex | VWR | 58816-123 | Equipment |
| Microcentrifuge | Eppendorf | 5404 000.413 | |
| Paraffin Film | Parafilm M | #PM996 | Kept at 9" with allows intensity of 10.84 mW/cm2 |
| Bluewave 200 UV spotlight | Blaze Technology | 120008, 122300 | |
| Tetrahydrofuran (THF) | Merck | 107025 | |
| Orbital shaker | Heidolph | 543-123120-00-5 | |
| Collagen Type I | Sigma Aldrich | C3867-1VL | From rat. 1x, w/o CaCl2 & MgCl2; pH = 7.2 |
| Phosphate Buffered Saline (PBS) | Gibco | 20012-027 | 16% W/V AQ. 10 x 10 ml |
| Paraformaldehyde | VWR | 43368.9M | Equipment |
| Freezone 4.5 freeze drier | Labconco | 7750020 | Equipment |
| Sputter coater | Jeol Ltd. | JFC-1600 | Equipment |
| Scanning Electron Microscope | Jeol Ltd. | JSM 5310 | |
| Anti-mouse primary antibodies against Collagen type I | Abcam | ab6308 | |
| Anti-mouse secondary antibody conjugated with Alexa Fluor 488 | Life Technologies | A21121 | |
| Plate, Tissue Culture 24 Well, Flat Bottom (Nunclon) | Bio-Rev PTE LTD | 3820-024 | |
| Dulbecco's Modified Eagle's Medium(DMEM) 2.5 g/L Glucose w/ L-Gln | Lonza | 12-604F | |
| Fetal Bovine Serum (FBS) | Gibco | A15-151 | |
| Penicillin-Streptomycin (P/S) | Life Tchnologies | 15140-122 E | |
| 100 mm Corning non-treated culture dishes | Sigma Aldrich | CLS430591 | |
| 0.25% Trypsin-EDTA | Gibco | 25200-056 | Equipment; 37 °C, 5% Humidity |
| Forma Steri-Cycle CO2 Incubators | Thermofisher Scientific | 371 | |
| Hausser Bright-Line Phase Hemacytometer | Thermofisher Scientific | 02-671-6 | |
| Live/Dead Viability/Cytotoxicity Kit for mammalian cells | Life Technologies | L3224 | |
| CCK-8 Assay | Dojindo Laboratories | CK04-11 | Monosodium-salt reagent (MSR) |
| Infinite 200 PRO microplate reader | Tecan | ||
| Albumin Human ELISA kit | Abcam | ab108788 | |
| Triton X-100 | Bio-Rad | #1610407 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A2153-50G | |
| Anti-mouse primary antibodies (against CYP3A4, albumin) | Santa Cruz Biotechnology | sc-53850; sc-271605 | |
| DAPI | Life Technologies | D3571 | |
| Alexa Fluor 555 labeled Phalloidin | Life Technologies | A34055 | |
| Trizol | Life Technologies | 15596-026 | |
| Chloroform | VWR | 22706.326 | |
| Isopropanol | Fisher Scientific | 67-63-0 | |
| DPEC water | Thermofisher Scientific | AM9916 | |
| Nanodrop 2000c Spectrophotometer | Thermofisher Scientific | ND-2000 | |
| iScript Reverse Transcription Supermix | Bio-Rad Laboratories | 1708840 | |
| SYBR select Master Mix for CFX | Life Technology | 4472937 | |
| Primers (to be chosen) | |||
| CFX96 Real-Time System, C-1000 Touch Thermal Cycler | Bio Rad Laboratories | SOFT-CFX-31-PATCH |
Referências
- Yamada, M., et al. Controlled formation of heterotypic hepatic micro-organoids in anisotropic hydrogel microfibers for long-term preservation of liver-specific functions. Biomaterials. 33 (33), 8304-8315 (2012).
- Abboud, G., Kaplowitz, N. Drug-induced liver injury. Drug Safety. 30 (4), 277-294 (2007).
- Cho, N. J., et al. Viral infection of human progenitor and liver-derived cells encapsulated in three-dimensional PEG-based hydrogel. Biomed Mater. 4 (1), (2009).
- Revzin, A., et al. Designing a hepatocellular microenvironment with protein microarraying and poly (ethylene glycol) photolithography. Langmuir. 20 (8), 2999-3005 (2004).
- Sato, A., Kadokura, K., Uchida, H., Tsukada, K. An in vitro hepatic zonation model with a continuous oxygen gradient in a microdevice. Biochem Bioph Res Com. 453 (4), 767-771 (2014).
- Domansky, K., et al. Perfused multiwell plate for 3D liver tissue engineering. Lab Chip. 10 (1), 51-58 (2010).
- Hegde, M., et al. Dynamic interplay of flow and collagen stabilizes primary hepatocytes culture in a microfluidic platform. Lab Chip. 14 (12), 2033-2039 (2014).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat methods. 2 (2), 119-125 (2005).
- Underhill, G. H., Chen, A. A., Albrecht, D. R., Bhatia, S. N. Assessment of hepatocellular function within PEG hydrogels. Biomaterials. 28 (2), 256-270 (2007).
- Dunn, J., Tompkins, R. G., Yarmush, M. L. Hepatocytes in collagen sandwich: evidence for transcriptional and translational regulation. J cell biol. 116 (4), 1043-1053 (1992).
- Dunn, J. C., Tompkins, R. G., Yarmush, M. L. Long-term in vitro function of adult hepatocytes in a collagen sandwich configuration. Biotechnol progr. 7 (3), 237-245 (1991).
- Ling, Y., et al. A cell-laden microfluidic hydrogel. Lab Chip. 7 (6), 756-762 (2007).
- Kim, M., Lee, J. Y., Jones, C. N., Revzin, A., Tae, G. Heparin-based hydrogel as a matrix for encapsulation and cultivation of primary hepatocytes. Biomaterials. 31 (13), 3596-3603 (2010).
- Kotov, N. A., et al. Inverted Colloidal Crystals as Three-Dimensional Cell Scaffolds. Langmuir. 20 (19), 7887-7892 (2004).
- Shanbhag, S., Woo Lee, J., Kotov, N. Diffusion in three-dimensionally ordered scaffolds with inverted colloidal crystal geometry. Biomaterials. 26 (27), 5581-5585 (2005).
- Lee, Y. H., Huang, J. R., Wang, Y. K., Lin, K. H. Three-dimensional fibroblast morphology on compliant substrates of controlled negative curvature. Integr Biol. 5, 1447-1455 (2013).
- da Silva, J., Lautenschlager, F., Kuo, C. H. R., Guck, J., Sivaniah, E. 3D inverted colloidal crystals in realistic cell migration assays for drug screening applications. Integr Biol. 3, 1202-1206 (2011).
- da Silva, J., Lautenschlager, F., Sivaniah, E., Guck, J. R. The cavity-to-cavity migration of leukaemic cells through 3D honey-combed hydrogels with adjustable internal dimension and stiffness. Biomaterials. 31, 2201-2208 (2010).
- Lee, J., Lilly, G. D., Doty, R. C., Podsiadlo, P., Kotov, N. A. In vitro toxicity testing of nanoparticles in 3D cell culture. Small. 5, 1213-1221 (2009).
- Lee, J., Kotov, N. A. Notch ligand presenting acellular 3D microenvironments for ex vivo human hematopoietic stem-cell culture made by layer-by-layer assembly. Small. 5, 1008-1013 (2009).
- Liu, Y., et al. Rapid aqueous photo-polymerization route to polymer and polymer-composite hydrogel 3D inverted colloidal crystal scaffolds. J Biomed Mater Res. Part A. 83, 1-9 (2007).
- Ma, P. X., Choi, J. W. Biodegradable polymer scaffolds with well-defined interconnected spherical pore network. Tissue Eng. 7, 23-33 (2001).
- Cuddihy, M. J., Kotov, N. A. Poly (lactic-co-glycolic acid) bone scaffolds with inverted colloidal crystal geometry. Tissue Eng Part A. 14, 1639-1649 (2008).
- Choi, S. W., Zhang, Y., Xia, Y. Three-dimensional scaffolds for tissue engineering: the importance of uniformity in pore size and structure. Langmuir. 26, 19001-19006 (2010).
- Choi, S. W., Zhang, Y., Thomopoulos, S., Xia, Y. In vitro mineralization by preosteoblasts in poly(DL-lactide-co-glycolide) inverse opal scaffolds reinforced with hydroxyapatite nanoparticles. Langmuir. 26, 12126-12131 (2010).
- Choi, S. W., Zhang, Y., Macewan, M. R., Xia, Y. Neovascularization in biodegradable inverse opal scaffolds with uniform and precisely controlled pore sizes. Adv Healthc Mater. 2, 145-154 (2013).
- Zhang, Y., Choi, S. W., Xia, Y. Modifying the Pores of an Inverse Opal Scaffold With Chitosan Microstructures for Truly Three-Dimensional Cell Culture. Macromol Rapid Commun. 33, 296-301 (2012).
- Cai, X., et al. Investigation of neovascularization in three-dimensional porous scaffolds in vivo by a combination of multiscale photoacoustic microscopy and optical coherence tomography. Tissue Eng. Part C, Meth. 19, 196-204 (2013).
- Zhang, Y. S., Yao, J., Wang, L. V., Xia, Y. Fabrication of Cell Patches Using Biodegradable Scaffolds with a Hexagonal Array of Interconnected Pores (SHAIPs). Polymer. 55, 445-452 (2014).
- Zhang, Y. S., Regan, K. P., Xia, Y. Controlling the Pore Sizes and Related Properties of Inverse Opal Scaffolds for Tissue Engineering Applications. Macromol Rapid Commun. 34, 485-491 (2013).
- Stachowiak, A. N., Bershteyn, A., Tzatzalos, E., Irvine, D. J. Bioactive Hydrogels with an Ordered Cellular Structure Combine Interconnected Macroporosity and Robust Mechanical Properties. Adv Mater. 17, 399-403 (2005).
- Stachowiak, A. N., Irvine, D. J. Inverse opal hydrogel-collagen composite scaffolds as a supportive microenvironment for immune cell migration. J Biomed Mater Res. Part A. 85, 815-828 (2008).
- Liu, Y., Wang, S. 3D inverted opal hydrogel scaffolds with oxygen sensing capability. Colloids and surfaces. B, Biointerfaces. 58, 8-13 (2007).
- Bryant, S. J., Cuy, J. L., Hauch, K. D., Ratner, B. D. Photo-patterning of porous hydrogels for tissue engineering. Biomaterials. 28, 2978-2986 (2007).
- Bhrany, A. D., Irvin, C. A., Fujitani, K., Liu, Z., Ratner, B. D. Evaluation of a sphere-templated polymeric scaffold as a subcutaneous implant. JAMA facial plastic surgery. 15, 29-33 (2013).
- Kuo, Y. C., Chiu, K. H. Inverted colloidal crystal scaffolds with laminin-derived peptides for neuronal differentiation of bone marrow stromal cells. Biomaterials. 32 (3), 819-831 (2011).
- Yang, J. T., Kuo, Y. C., Chiu, K. H. Peptide-modified inverted colloidal crystal scaffolds with bone marrow stromal cells in the treatment for spinal cord injury. Colloids Surf. B, Biointerfaces. 84, 198-205 (2011).
- Kuo, Y. C., Tsai, Y. T. Inverted colloidal crystal scaffolds for uniform cartilage regeneration. Biomacromolecules. 11, 731-739 (2010).
- Choi, S. W., Xie, J., Xia, Y. Chitosan-Based Inverse Opals: Three-Dimensional Scaffolds with Uniform Pore Structures for Cell Culture. Adv Mater. 21, 2997-3001 (2009).
- Long, T. J., Sprenger, C. C., Plymate, S. R., Ratner, B. D. Prostate cancer xenografts engineered from 3D precision-porous poly(2-hydroxyethyl methacrylate) hydrogels as models for tumorigenesis and dormancy escape. Biomaterials. 35, 8164-8174 (2014).
- Kuo, Y. C., Tsai, Y. T. Inverted colloidal crystal scaffolds for uniform cartilage regeneration. Biomacromolecules. 11, 731-739 (2010).
- Kuo, Y. C., Chiu, K. H. Inverted colloidal crystal scaffolds with laminin-derived peptides for neuronal differentiation of bone marrow stromal cells. Biomaterials. 32, 819-831 (2011).
- Lee, J., Cuddihy, M. J., Cater, G. M., Kotov, N. A. Engineering liver tissue spheroids with inverted colloidal crystal scaffolds. Biomaterials. 30 (27), 4687-4694 (2009).
- Galperin, A., et al. Integrated bi-layered scaffold for osteochondral tissue engineering. Adv Healthc Mater. 2, 872-883 (2013).
- Waters, D. J., et al. Morphology of Photopolymerized End-linked Poly(ethylene glycol) Hydrogels by Small Angle X-ray Scattering. Macromolecules. 43 (16), 6861-6870 (2010).
- Elbert, D. L., Hubbell, J. A. Conjugate addition reactions combined with free-radical cross-linking for the design of materials for tissue engineering. Biomacromolecules. 2 (2), 430-441 (2001).
- Kim, M. H., et al. Biofunctionalized Hydrogel Microscaffolds Promote Three-Dimensional Hepatic Sheet Morphology. Macromol Biosci. , (2015).
- Ferreira, T., Rasband, W. ImageJ User Guide. , http://imagej.nih.gov/ij/docs/guide/146-30.html#toc-Subsection-30.1 (2012).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to Fluorescence Microscopy. , JoVE. Cambridge, MA. (2015).
- Tominaga, H., et al. A water-soluble tetrazolium salt useful for colorimetric cell viability assay. Anal Commun. 36 (2), 47-50 (1999).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to the Microplate Reader. , JoVE. Cambridge, MA. (2015).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The ELISA Method. , JoVE. Cambridge, MA. (2015).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat Protoc. 1, 1559-1582 (2006).
- JoVE Science Education Database. Essentials of Environmental Microbiology. RNA Analysis of Environmental Samples Using RT-PCR. , JoVE. Cambridge, MA. (2016).
- JoVE Science Education. Essentials of Environmental Microbiology. , JoVE. (2015).
- Jeong, S., et al. The evolution of gene regulation underlies a morphological difference between two Drosophila sister species. Cell. 132 (5), 783-793 (2008).
- Griffith, L. G., Naughton, G. Tissue engineering--current challenges and expanding opportunities. Science. 295 (5557), 1009-1014 (2002).
- Hegde, M., et al. Dynamic Interplay of Flow and Collagen Stabilizes Primary Hepatocytes Culture in a Microfluidic Platform. Lab Chip. 14, 2033-2039 (2014).
- Kim, Y., Lasher, C. D., Milford, L. M., Murali, T., Rajagopalan, P. A comparative study of genome-wide transcriptional profiles of primary hepatocytes in collagen sandwich and monolayer cultures. Tissue Eng Pt C. 16 (6), 1449-1460 (2010).
- Baimakhanov, Z., et al. Efficacy of multi-layered hepatocyte sheet transplantation for radiation-induced liver damage and partial hepatectomy in a rat model. Cell Transplant. , (2015).
- Li, C. Y., et al. Micropatterned Cell-Cell Interactions Enable Functional Encapsulation of Primary Hepatocytes in Hydrogel Microtissues. Tissue Eng Pt A. 20 (15-16), 2200-2212 (2014).
- Shlomai, A., et al. Modeling host interactions with hepatitis B virus using primary and induced pluripotent stem cell-derived hepatocellular systems. P Natl A Sci USA. 111 (33), 12193-12198 (2014).
- Curcio, E., et al. Mass transfer and metabolic reactions in hepatocyte spheroids cultured in rotating wall gas-permeable membrane system. Biomaterials. 28, 5487-5497 (2007).
- Martinez-Hernandez, A., Amenta, P. The hepatic extracellular matrix. Vichows Archiv A Pathol Anat. 423, 1-11 (1993).
- Liu, Y., Wang, S., Lee, J. W., Kotov, N. A. A Floating Self-Assembly Route to Colloidal Crystal Templates for 3D Cell Scaffolds. Chem Mater. 17 (20), 4918-4924 (2005).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados