Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Fagosoma Migración y velocidad medida en vivo primaria macrófagos humanos infectados con VIH-1
En este artículo
Resumen
We describe a method to measure the velocity of phagosomes moving towards the cell center in living cells infected with or without the human immunodeficiency virus (HIV) type 1, using spinning disk confocal fluorescence microscopy to identify fluorescent infected cells and bright field microscopy to detect phagosomes.
Resumen
Macrophages are phagocytic cells that play a major role at the crossroads between innate and specific immunity. They can be infected by the human immunodeficiency virus (HIV)-1 and because of their resistance to its cytopathic effects they can be considered to be persistent viral reservoirs. In addition, HIV-infected macrophages exhibit defective functions that contribute to the development of opportunistic diseases.
The exact mechanism by which HIV-1 impairs the phagocytic response of macrophages was unknown. We had previously shown that the uptake of various particulate material by macrophages was inhibited when they were infected with HIV-1. This inhibition was only partial and phagosomes did form within HIV-infected macrophages. Therefore, we focused on analyzing the fate of these phagosomes. Phagosome maturation is accompanied by migration of these compartments towards the cell center, where they fuse with lysosomes, generating phagolysosomes, responsible for degradation of the ingested material. We used IgG-opsonized Sheep Red Blood Cells as a target for phagocytosis. To measure the speed of centripetal movement of phagosomes in individual HIV-infected macrophages, we used a combination of bright field and fluorescence confocal microscopy. We established a method to calculate the distance of phagosomes towards the nucleus, and then to calculate the velocity of the phagosomes. HIV-infected cells were identified thanks to a GFP-expressing virus, but the method is applicable to non-infected cells or any type of infection or treatment.
Introducción
Macrophages play a major role in the innate immune system and in homeostasis. They are professional phagocytes that internalize and eliminate pathogens and debris by a process called phagocytosis 1,2. The phagosome, the closed compartment that forms after the engulfment of particulate material, undergoes a series of fusion and fission events with endocytic compartments, leading to a degradative compartment called the phagolysosome. This compartment has an acidic pH, due to the acquisition of proton-pumping v-ATPases, contains hydrolytic enzymes and is enriched in lysosomal-associated membrane proteins (LAMPs). The maturation of phagosomes is accompanied by their migration on microtubules 3,4 towards the cell center to reach a perinuclear location where lysosomes are accumulated.
Many pathogens have been reported to hijack phagosome maturation, including bacteria with intracellular lifestyles that modify the vacuolar environment where they reside 5. The Human Immunodeficiency Virus (HIV)-1 targets macrophages in addition to T cells. As macrophages are resistant to the cytopathic effects of the virus, unlike T cells, they can be considered as a reservoir for the virus. In addition, macrophages infected with HIV-1 show defective phagocytic functions and contribute to the emergence of opportunistic diseases. In particular, severe invasive non-typhoidal Salmonella disease caused by Salmonella Typhimurium ST313 has been prevalent for the last three decades in sub-Saharan African children or adults infected with HIV 6. It has been estimated that the risk of developing tuberculosis is more than 20 times greater in people living with HIV than among those without HIV infection.
For all these reasons, it is important to better define the molecular mechanisms underlying the phagocytic defects in HIV-infected macrophages. We have shown that the uptake of particulate material, opsonized particles, bacteria or fungi, was inhibited in HIV-infected macrophages 7. Given that this inhibition is partial, we then set out to analyze the fate of the internalized phagosomes in HIV infected human macrophages 8. Because phagosome maturation is tightly connected with migration to the cell center and fusion with lysosomes, a defect in phagosomal maturation can be due to modifications of the trafficking modalities in the infected cell. The method described here uses IgG-opsonized Sheep Red Blood Cells (IgG-SRBCs) as a model to target receptor-mediated phagocytosis and in particular receptors for the Fc portion of immunoglobulins (FcR). These particles are easier to image in bright field (BF) than latex beads because extracellular and intracellular SRBCs show different refraction properties 9. To measure the velocity of phagosomes moving towards the nucleus in HIV-infected macrophages, we used a fluorescent virus 10 and set up a simple manual tracking method that is described here. The method does not require advanced programming and simply uses ImageJ. It is amenable to adherent cells and any type of particle or pathogen that can be visualized with bright field microscopy or with fluorescent imaging.
Protocolo
El protocolo tiene que ser llevado a cabo en estricta conformidad con las legislaciones nacionales e internacionales y reglamentos locales. La sangre de donantes sanos que dieron su consentimiento para donar sangre con fines de investigación se ha obtenido a partir de los centros de transfusión de sangre con la que las instituciones han firmado un acuerdo. protecciones especiales se deben tomar cuando se utiliza la sangre humana. Los experimentos con VIH-1 deberán efectuarse en un nivel de bioseguridad 3 o 2 (BSL-3 o 2) de laboratorio de acuerdo con la legislación local.
1. Preparación de macrófagos derivados de monocitos humanos (hMDMs) por centrifugación en gradiente de densidad y selección por adherencia
- Comience con la sangre fresca de donantes sanos (9 ml). Diluir todo el volumen de la sangre fresca con 1x estéril salina tamponada con fosfato (PBS) sin Ca2 + y Mg2 + para obtener un volumen final de 70 ml y añadir suavemente la sangre diluida en dos tubos de 50 ml cónicos (35 ml por tubo) , en la parte superior de 15 ml de unaeutral, muy ramificado, de gran masa, polisacárido hidrófilo en la solución ya está en cada tubo.
- Centrifugar los dos tubos de sangre en 537 xg durante 20 min a 20 ° C sin freno. Luego recoger las células mononucleares de sangre periférica (PBMCs) contenidos en el anillo de células nublado en la interfase y transferirlos a un nuevo tubo de 50 ml que contiene 15 ml de 1x PBS sin Ca2 + y Mg2 +.
- Centrifugar las células a 218 xg durante 5 min a 20 ° C y resuspender el precipitado en 45 ml de 1x PBS sin Ca2 + y Mg2 +.
- Centrifugar las células a 218 xg durante 5 min a 20 ° C, resuspender el precipitado en 10 ml de 1x PBS sin Ca2 + y Mg2 +, y recuento de las células mediante la dilución a una dilución final de 1/200 en Trypan Blue.
- Centrifugar las células a 218 xg durante 5 min a 20 ° C y resuspender el sedimento en medio RPMI (Roswell Park Memorial Institute) 1640 suplementado con 2 mM L-glutamina y 100 mg / ml Penicillin-estreptomicina tener 7 x 10 6 PBMCs por pocillo en 2 ml de medio en cada pocillo de una placa de pocillos 6.
- Incubar las placas a 37 ° C con 5% de CO2 durante 2 hr. Después de 2 horas los monocitos se han adherido al plástico.
- Para permitir aislados recién monocitos de diferenciarse en hMDMs, aspirar el medio y lo reemplazan con 2 ml HMDM medio (RPMI 1640, 10% descomplementado suero de ternera fetal (FCS), 2 mM de L-glutamina y 1% de penicilina-estreptomicina) suplementado con colonias de macrófagos factor estimulante (RHM-CSF) a una concentración final de 10 ng / ml.
- Se incuban las células a 37 ° C con 5% de CO 2 durante 11 días (Figura 1).
- En el día 11 se elimina el medio y se lava 2 veces con 2 ml de medio HMDM frío por pocillo. A continuación lavar cada pocillo 2 veces con 1 ml de PBS 1x frío.
- Para separar hMDMs completamente diferenciadas, lavar cada vez que el pozo 1 con 1 ml de PBS frío 1x con ácido ethylenediaminetetraacetatic 2 mM (EDTA) e incubar las células en 2 ml de PBS frío 1x con EDTA 2 mM por pocillo durante 15 a 60 min a 4 ° C.
NOTA: Los métodos alternativos para desprender las células existen (por ejemplo, tripsina o actividades similares a la tripsina) que podrían dar buenos resultados 11. - Después de la separación (terminado pipeteando suavemente hacia arriba y hacia abajo en el pozo), recoger las células y los puso en un tubo de 50 ml en hielo que contiene 10 ml de medio HMDM frío.
- Centrifugar las células a 218 xg durante 5 min a 20 ° C, resuspender el precipitado en 10 ml de medio HMDM frío y el recuento de las células mediante la dilución de 1/20 en Trypan Blue.
- Sembrar las células a 1 x 10 6 hMDMs por microscopía de cristal del grado 35 mm placa de fondo y se incuban las placas a 37 ° C con 5% de CO 2 durante 1 día.
2. Producción y cuantificación de VIH-1 reservas virales
NOTA: NLR4.3 VIH-1 Gag-IGFP (proteína verde fluorescente) que lleva un sobre R5-trópico, obsequio de M. Schindler 10 se usa para infectar a los macrófagos y para ver las células infectadas en tiempo real.
- Producir reservas virales mediante la transfección de las células humanas embrionarias de riñón 293T (2 x 10 6 en 100 mm de plato) con 6 g de ADN proviral correspondiente usando un reactivo de transfección comercial.
- Cuantificar la infectividad de las reservas de virus usando las células indicadoras HeLa TZM-bl (que llevan el gen de ß-galactosidasa bajo el control del VIH-1 LTR) utilizando diluciones seriadas de las reservas de virus, seguido de una coloración ß-galactosidasa de las células y contando de células azules 12.
3. La infección de hMDMs con VIH-1
- Añadir el virus a una multiplicidad de infección (MOI) 0,02 a 0,03 en 1 ml de medio de HMDM a hMDMs (células en placas en 1,13). Para los pocillos de control sólo añadir 1 ml de medio de HMDM e incubar los platos a 37 ° C con 5% de CO 2 durante 2 días (Figura 1).
- En el día 2 se lavan las células con HMDM medIUM 3 veces y se añade 1 ml de medio fresco HMDM por plato. Se incuban las células a 37 ° C con 5% de CO 2 durante 6 días (Figura 1).
4. La opsonización de ovejas Glóbulos rojos
- Para la preparación de 7 x 10 6 SRBCs por placa, lavar los SRBC dos veces en 100 l de solución que contenía 0,1% albúmina de suero bovino (BSA) en PBS 1x con centrifugación a 600 xg durante 4 min.
- Resuspender las SRBC lavadas en 500 l de 1x PBS / BSA 0,1% con IgG de conejo anti-SRBC a una concentración sub-aglutinantes por 5 l de SRBC y se incuba con la rotación a TA durante 30 min.
NOTA: Para determinar la concentración sub-aglutinantes de IgG anti-SRBC, se preparan diluciones seriadas de la IgG (concentración de reserva a 13,1 mg / ml) a partir de 1/50 a 1 / 25.600 en 20 l en una placa de 96 pocillos. Añadir 2 x 10 6 SRBCs en 20 l en cada pocillo y colocar la placa en una habitación oscura durante varias horas. La concentración sub-aglutinante es ladilución de la bien justo antes de la aglutinación bien con (+ SRBCs IgG formación de una red). - Después de la rotación, centrifugar los eritrocitos de cordero-opsonized-IgG a 600 xg durante 4 minutos y se lava con 100 l de 1x PBS / BSA 0,1% con centrifugación a 600 g durante 4 min.
- Resuspender las-SRBC opsonizadas-IgG en un medio de pre-calentado libre de rojo fenol RPMI suplementado con 2 mM L-glutamina y 1% de penicilina-estreptomicina (1 ml / placa).
5. Ensayo en vivo de la célula microscopía de vídeo fagocitosis
- Utilice un sistema de imagen confocal, tal como un sistema de disco giratorio equipado con una cámara de calentamiento a 37 ° C con CO 2 pasa a través de una botella con agua para la humidificación.
- Encienda la cámara de calentamiento antes del experimento para tener la platina del microscopio a 37 ° C antes del comienzo del ensayo de fagocitosis. Encienda el microscopio y el ordenador, y cargar el software de imágenes.
- Optimizar los ajustes de imagen como la velocidad de exploración, magnífication, la resolución, etc. para tener al menos una célula por campo y a la imagen un fotograma cada minuto entre 60 a 120 min.
NOTA: En este caso, la muestra se forma la imagen de un marco de cada minuto durante 60 min con 63x lente. - Coloque el recipiente de formación de imágenes en la platina del microscopio. Ajustar el enfoque y la ubicación para encontrar sólo un conjunto de VIH-1 de macrófagos infectados en el campo. Use la configuración de excitación / emisión apropiados basados en el sistema de imagen utilizado y la sonda. Incluir un canal brillante campo (BF) para observar los fagosomas (Figura 1II). Optimizar la aparición de las diferentes imágenes del canal ajustando el porcentaje de tiempo de la luz y la exposición transmitida.
NOTA: En este caso, NLR4.3 VIH-1 Gag-IGFP estaba excitada usando un láser de 491 nm con 50 ms de tiempo de exposición con 20% de láser (Figura 1i). - Retirar la cápsula de imágenes y añadir 1 ml de suspensión de SRBC en 7 x 10 6 / ml a SRBC el plato (Figura 1).
- Centrifugar a 500 xg durante2 min a TA para sincronizar la fagocitosis, registrar el tiempo al final de esta centrifugación y devolver el plato a la etapa.
- Optimizar la GFP enfoque y la captura y las imágenes BF en Z-pilas (en todo el espesor de la celda con un paso a la distancia de 0,3 m - por lo general 20 aviones) cada minuto durante al menos 1 hr. Guardar el vídeo de lapso de tiempo en el formato de archivo nativo del sistema de imagen utilizado.
NOTA: En este caso, las películas a intervalos fueron salvados en el formato nativo, archivos * .stk.
6. Análisis de las películas a intervalos
- Para la edición de vídeo, haga clic en el menú desplegable "Aplicaciones" y en la ficha "Revisión multidimensional de datos" en el software de edición de vídeo.
- Para abrir el archivo, haga clic en "Seleccionar archivo de la base", luego en "Seleccionar directorio". En el "conjuntos de datos", seleccione la adquisición de analizar (en formato .nd) y haga clic en "Ver". Los datos se representan en una tabla con el tiempo en una columnad Z de planta en la línea.
- Representar a la infección en un Z-proyección (Figura 2, paneles de la izquierda), seleccione 491 nm de longitud de onda en el cuadro de "Las longitudes de onda" y haga clic en la pestaña "proyección Z" con "todos los planos".
- Para analizar una secuencia de tiempo, seleccione la longitud de onda de BF en el cuadro de "Las longitudes de onda" y seleccione el plano óptima en el eje Z para distinguir los eritrocitos de cordero externos (Figura 2, punta de flecha roja), SRBC internos (Figura 2, círculo rojo) y el núcleo ( La figura 2, círculo azul).
- Para guardar los montajes de vídeo, haga clic en la pestaña "Selección [X]" y después en "Load Image (s)". Por último, guardar las imágenes cargadas en formato tif y abrirlos al lado en el software ImageJ.
- Utilizar el plugin "Tracking Manual" en el software ImageJ para medir la posición del núcleo y las diferentes fagosomas observados en el canal BF (Figura 3).
- Download el plugin "Manual de seguimiento" en la página web ImageJ. En ImageJ, abren el plugin y la secuencia de imágenes a analizar (figura 3, paso 1 y 2).
- Introduzca los ajustes tales como "Intervalo de tiempo", que representa la cantidad de tiempo entre tramas adyacentes, y la "x / y de calibración", que representa la distancia por píxel (figura 3, paso 3).
NOTA: Aquí, guardar la secuencia de imágenes con un intervalo de tiempo de 1 min entre cada fotograma y x / y de calibración de 0,205 micras porque se utilizan el zoom 63X y una cámara con tamaño de píxel de 6,45 x 6,45 m 2. - Para iniciar el seguimiento, haga clic en "Añadir pista" (figura 3, paso 4) y hacer clic en un centro de SRBC en el primer momento en que se internaliza. La siguiente trama aparece automáticamente.
- Continúe haciendo clic en el centro de SRBC en todos los marcos que tienen diferentes posiciones durante el tiempo (figura 3, paso 6 en el cuadro rojo).
- Comenzará a realizar el seguimiento del núcleo que su posición en todos los marcos haciendo clic en su centro. Tema siguiente los fagosomas (haciendo clic en su centro), uno por uno en el momento (marco) de su internalización, diferentes en función de los eritrocitos de cordero. Para una mayor comodidad para ver SRBC en el canal BF, utilice la ventana de "Brillo y contraste" durante el seguimiento (figura 3, paso 5).
- Entre cada SRBC de seguimiento, haga clic en "Añadir pista" (figura 3, paso 4) para tener una nueva pista. El número de seguimiento será cambiado en la segunda columna, "Pista n °" de la tabla de resultados (figura 3, paso 6).
- Guardar los datos en una hoja de cálculo para continuar con el siguiente paso del análisis.
- Utilice el software de hoja de cálculo para calcular la distancia recorrida de los fagosomas que contienen SRBCs hacia el núcleo y la velocidad de los fagosomas durante los primeros 5 minutos después de la internalización de los eritrocitos de cordero (Fifigura 4).
- Abra la tabla de hoja de cálculo y un nuevo archivo de hoja de cálculo. Transferencia en este nuevo archivo sólo los siguientes parámetros, el tiempo, la X e Y de coordenadas del núcleo y todos los SRBC (figura 4A).
- Para calcular la distancia entre SRBC y el núcleo con sólo sus coordenadas, considere el núcleo y un SRBC en un plano de coordenadas xy (Figura 4B).
NOTA: La distancia entre dos puntos es la longitud de la trayectoria de conectarlos. En el plano, la distancia entre SRBC y el núcleo está dado por el teorema de Pitágoras.- Si c (la distancia entre el núcleo y el SRBC) denota la longitud de la hipotenusa y a y b indican las longitudes de los otros dos lados, expresar el teorema de Pitágoras como la ecuación de Pitágoras:

NOTA: Al mismo tiempo, en la ortonormal, la distancia horizontala es (x -x núcleo SRBC) y la distancia vertical b es (y núcleo -y SRBC). - Por lo tanto, calcular la distancia entre el núcleo y el SRBC (Figura 4A, caja púrpura) por:
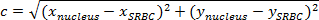
NOTA: Multiplicar el valor de la distancia obtenida (en píxeles) por x / y de calibración para tener la distancia en micras. Aquí, el x / y de calibración es 0,205 m. - En cada tiempo, se resta la distancia entre el núcleo y el SRBC a la distancia inicial (Figura 4C).
- Trazar la medición en función del tiempo para los primeros 5 minutos. Aplique una línea de tendencia lineal (Figura 5A) a la trama y determinar la pendiente de la línea de tendencia lineal que representa la velocidad fagosoma hacia el núcleo durante los primeros 5 minutos después de la internalización SRBC.
- Cotejar los datos para calcular el error medio y estadístico delos valores de velocidad y representarlos en una forma apropiada con el software adecuado (Figura 5B).
- Si c (la distancia entre el núcleo y el SRBC) denota la longitud de la hipotenusa y a y b indican las longitudes de los otros dos lados, expresar el teorema de Pitágoras como la ecuación de Pitágoras:
Resultados
Fagocitosis mediada por FcR hMDMs infectados por el VIH-1 y no infectados se describe aquí usando SRBCs opsonized-IgG como objetivos modelo (Figura 1). Los pasos críticos de este protocolo son la preparación de hMDMs y la infección con el VIH-1. De hecho, el rendimiento y la calidad de macrófagos diferenciados varía entre los donantes, así como la tasa de infección con eficiencias en el rango de 10-40%. Además, la preparación de IgG-opsonizado-SRBC también es ...
Discusión
Esta técnica tiene varios pasos críticos. En primer lugar, la preparación de hMDMs y su infección por el VIH-1 es muy importante ya que el porcentaje de infección depende de los donantes. Es de destacar, que hemos decidido utilizar los macrófagos que no están polarizados in vitro antes de la infección, debido a que el estado de los macrófagos potencialmente encontradas por el virus in vivo no ha sido bien caracterizado hasta ahora. Se verificó la expresión de varios marcadores de la superfic...
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
We thank Dr Jamil Jubrail for reading the manuscript. This work was supported by grants from CNRS, Inserm, Université Paris Descartes, Agence Nationale de la Recherche (2011 BSV3 025 02), Fondation pour la Recherche Médicale (FRM DEQ20130326518 including a doctoral fellowship for GLB) and Agence Nationale de Recherche sur le SIDA et les hépatites virales (ANRS, including a post-doctoral fellowship for CD) to FN. A. Dumas was supported by doctoral fellowships from Université Paris Descartes and Sidaction.
Materiales
| Name | Company | Catalog Number | Comments |
| Falcon 100mm TC-Treated Cell Culture Dish | Corning | 353003 | For viral production |
| Glass Bottom Dishes 35 mm uncoated 1.5 | MatTek corporation | P35G-1.5-14-C Case | For acquisition |
| Falcon Tissue Culture Plates 6-well | Thermo Fischer Scientific | Corning. Inc. 353934 | For human monocyte-derived macrophages |
| Ficoll-Plaque PLUS | Dominique Dutscher | 17-1440-03 | a neutral, highly branched, high-mass, hydrophilic polysaccharide in solution for density centrifugation |
| DPBS, no calcium, no magnesium | Thermo Fischer Scientific | 14190-094 | Room temperature |
| Dulbecco's Modified Eagle Medium (DMEM) 1X, liquid (High Glucose) | GIBCO, Molecular probes | 31966-021 | Conserved at 4°C ; for HEK cells culture |
| RPMI 1640 medium GLUTAMAX Supplement | Life technologies | 61870-010 | Conserved at 4°C; for hMDMs culture |
| Fœtal Calf Serum (FCS) | Eurobio | CVFSVF0001 | Conserved at -20°C ; decomplemented |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fischer Scientific | 15140-122 | Conserved at -20°C ; for hMDMs culture |
| RPMI 1640 medium, no phenol red (10x500 ml) | Life technologies | 11835-105 | Warm in 37°C water bath before use ; for phagocytosis assay |
| FuGENE6 Transfection Reagent | Promega | E2692 | Conserved at 4°C ; for viral production |
| Sheep red blood cells (SRBCs) | Eurobio | DSGMTN00-0Q | Conserved in Alsever buffer at 4°C before use |
| Anti-sheep red blood cells IgG | MP Biomedicals | 55806 | Conserved at 4°C |
| Bovine Serum Albumin heat shock fraction, pH 7, ≥98% | Sigma | A7906 | Conserved at -20°C |
| Inverted microscope DMI600 | Leica | ||
| Confocal Spinning Disk Unit CSU-X1M1 | Yokogawa | ||
| 491 nm 50mW laser | COBOLT CALYPSO | ||
| HCX PL APO CS Objectif | Leica | Objective lens ; Magnification 100x ; Numerical aperture 1.40 ; Immersion oil | |
| CoolSnap HQ2 (FireWire) Camera | Photometrics | Pixel size 6.45 x 6.45 µm ; Definition 1392 x 1040 ; Encoding the image in 14 Bit | |
| Metamorph 7.7.5 software | Molecular Devices | For the control of the microscope | |
| GraphPad Prism software | For the statistics analysis |
Referencias
- Flannagan, R. S., Jaumouille, V., Grinstein, S. The cell biology of phagocytosis. Annu. Rev. Pathol. 7, 61-98 (2012).
- Niedergang, F. . Encyclopedia of Cell Biology. 2, 751-757 (2016).
- Blocker, A., Griffiths, G., Olivo, J. C., Hyman, A. A., Severin, F. F. A role for microtubule dynamics in phagosome movement. J Cell Sci. 111 (Pt 3), 303-312 (1998).
- Blocker, A., et al. Molecular requirements for bi-directional movement of phagosomes along microtubules. J Cell Biol. 137, 113-129 (1997).
- Flannagan, R. S., Cosio, G., Grinstein, S. Antimicrobial mechanisms of phagocytes and bacterial evasion strategies. Nat Rev Microbiol. 7, 355-366 (2009).
- Feasey, N. A., Dougan, G., Kingsley, R. A., Heyderman, R. S., Gordon, M. A. Invasive non-typhoidal salmonella disease: an emerging and neglected tropical disease in Africa. Lancet. 379, 2489-2499 (2012).
- Mazzolini, J., et al. Inhibition of phagocytosis in HIV-1-infected macrophages relies on Nef-dependent alteration of focal delivery of recycling compartments. Blood. 115, 4226-4236 (2010).
- Dumas, A., et al. The HIV-1 protein Vpr impairs phagosome maturation by controlling microtubule-dependent trafficking. J Cell Biol. 211, 359-372 (2015).
- Greenberg, S., el Khoury, J., Kaplan, E., Silverstein, S. C. A fluorescence technique to distinguish attached from ingested erythrocytes and zymosan particles in phagocytosing macrophages. J. Immunol. Methods. 139, 115-122 (1991).
- Koppensteiner, H., Banning, C., Schneider, C., Hohenberg, H., Schindler, M. Macrophage internal HIV-1 is protected from neutralizing antibodies. J Virol. 86, 2826-2836 (2012).
- Gartner, S. The macrophage and HIV: basic concepts and methodologies. Methods Mol Biol. 1087, 207-220 (2014).
- Wei, X., et al. Emergence of resistant human immunodeficiency virus type 1 in patients receiving fusion inhibitor (T-20) monotherapy. Antimicrob Agents Chemother. 46, 1896-1905 (2002).
- Harrison, R. E., Bucci, C., Vieira, O. V., Schroer, T. A., Grinstein, S. Phagosomes fuse with late endosomes and/or lysosomes by extension of membrane protrusions along microtubules: role of Rab7 and RILP. Mol Cell Biol. 23, 6494-6506 (2003).
- Toyohara, A., Inaba, K. Transport of phagosomes in mouse peritoneal macrophages. J Cell Sci. 94 (Pt 1), 143-153 (1989).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados