È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Migrazione phagosome e velocità misurata in Live primarie macrofagi umani infettati con HIV-1
In questo articolo
Riepilogo
We describe a method to measure the velocity of phagosomes moving towards the cell center in living cells infected with or without the human immunodeficiency virus (HIV) type 1, using spinning disk confocal fluorescence microscopy to identify fluorescent infected cells and bright field microscopy to detect phagosomes.
Abstract
Macrophages are phagocytic cells that play a major role at the crossroads between innate and specific immunity. They can be infected by the human immunodeficiency virus (HIV)-1 and because of their resistance to its cytopathic effects they can be considered to be persistent viral reservoirs. In addition, HIV-infected macrophages exhibit defective functions that contribute to the development of opportunistic diseases.
The exact mechanism by which HIV-1 impairs the phagocytic response of macrophages was unknown. We had previously shown that the uptake of various particulate material by macrophages was inhibited when they were infected with HIV-1. This inhibition was only partial and phagosomes did form within HIV-infected macrophages. Therefore, we focused on analyzing the fate of these phagosomes. Phagosome maturation is accompanied by migration of these compartments towards the cell center, where they fuse with lysosomes, generating phagolysosomes, responsible for degradation of the ingested material. We used IgG-opsonized Sheep Red Blood Cells as a target for phagocytosis. To measure the speed of centripetal movement of phagosomes in individual HIV-infected macrophages, we used a combination of bright field and fluorescence confocal microscopy. We established a method to calculate the distance of phagosomes towards the nucleus, and then to calculate the velocity of the phagosomes. HIV-infected cells were identified thanks to a GFP-expressing virus, but the method is applicable to non-infected cells or any type of infection or treatment.
Introduzione
Macrophages play a major role in the innate immune system and in homeostasis. They are professional phagocytes that internalize and eliminate pathogens and debris by a process called phagocytosis 1,2. The phagosome, the closed compartment that forms after the engulfment of particulate material, undergoes a series of fusion and fission events with endocytic compartments, leading to a degradative compartment called the phagolysosome. This compartment has an acidic pH, due to the acquisition of proton-pumping v-ATPases, contains hydrolytic enzymes and is enriched in lysosomal-associated membrane proteins (LAMPs). The maturation of phagosomes is accompanied by their migration on microtubules 3,4 towards the cell center to reach a perinuclear location where lysosomes are accumulated.
Many pathogens have been reported to hijack phagosome maturation, including bacteria with intracellular lifestyles that modify the vacuolar environment where they reside 5. The Human Immunodeficiency Virus (HIV)-1 targets macrophages in addition to T cells. As macrophages are resistant to the cytopathic effects of the virus, unlike T cells, they can be considered as a reservoir for the virus. In addition, macrophages infected with HIV-1 show defective phagocytic functions and contribute to the emergence of opportunistic diseases. In particular, severe invasive non-typhoidal Salmonella disease caused by Salmonella Typhimurium ST313 has been prevalent for the last three decades in sub-Saharan African children or adults infected with HIV 6. It has been estimated that the risk of developing tuberculosis is more than 20 times greater in people living with HIV than among those without HIV infection.
For all these reasons, it is important to better define the molecular mechanisms underlying the phagocytic defects in HIV-infected macrophages. We have shown that the uptake of particulate material, opsonized particles, bacteria or fungi, was inhibited in HIV-infected macrophages 7. Given that this inhibition is partial, we then set out to analyze the fate of the internalized phagosomes in HIV infected human macrophages 8. Because phagosome maturation is tightly connected with migration to the cell center and fusion with lysosomes, a defect in phagosomal maturation can be due to modifications of the trafficking modalities in the infected cell. The method described here uses IgG-opsonized Sheep Red Blood Cells (IgG-SRBCs) as a model to target receptor-mediated phagocytosis and in particular receptors for the Fc portion of immunoglobulins (FcR). These particles are easier to image in bright field (BF) than latex beads because extracellular and intracellular SRBCs show different refraction properties 9. To measure the velocity of phagosomes moving towards the nucleus in HIV-infected macrophages, we used a fluorescent virus 10 and set up a simple manual tracking method that is described here. The method does not require advanced programming and simply uses ImageJ. It is amenable to adherent cells and any type of particle or pathogen that can be visualized with bright field microscopy or with fluorescent imaging.
Protocollo
Il protocollo deve essere effettuata in stretta conformità con le normative nazionali ed internazionali e le normative locali. Il sangue da donatori sani che hanno dato il loro consenso a donare il sangue per scopi di ricerca è stato ottenuto dal sangue centri trasfusionali con la quale le istituzioni hanno firmato un accordo. protezioni speciali devono essere prese quando si utilizza il sangue umano. Esperimenti con HIV-1 devono essere eseguite in un livello di biosicurezza 3 o 2 (BSL-3 o 2) di laboratorio in base alla legislazione locale.
1. Preparazione di macrofagi umani derivate da monociti (hMDMs) per gradiente di densità centrifugazione e selezione per adesione
- Inizia con sangue fresco da donatori sani (9 ml). Diluire l'intero volume di sangue fresco con 1x tampone fosfato sterile (PBS) senza Ca 2 + e Mg 2+ a ottenere un volume finale di 70 ml e aggiungere delicatamente il sangue diluito in due provette da 50 ml coniche (35 ml per provetta) , in cima a 15 ml di unaeutral, altamente ramificata, di massa elevata, polisaccaride idrofila in soluzione già in ciascun tubo.
- Centrifuga entrambi i tubi di sangue al 537 xg per 20 minuti a 20 ° C senza freno. Poi raccogliere le cellule mononucleate del sangue periferico (PBMC) contenuti nel ring cellule nuvoloso a livello di interfaccia e trasferirli in un nuovo tubo da 50 ml contenente 15 ml di 1x PBS senza Ca 2+ e Mg 2+.
- Centrifugare le cellule a 218 xg per 5 minuti a 20 ° C e risospendere il pellet in 45 ml di 1x PBS senza Ca 2+ e Mg 2+.
- Centrifugare le cellule a 218 xg per 5 min a 20 ° C, risospendere il pellet in 10 ml di PBS 1x senza Ca 2 + e Mg 2+, e contare le cellule diluendo ad una diluizione finale di 1/200 in Trypan Blue.
- Centrifugare le cellule a 218 xg per 5 min a 20 ° C e risospendere il pellet in RPMI (Roswell Park Memorial Institute) 1640 integrato con 2 mM L-glutammina e 100 ug / ml PenicIllin-streptomicina avere 7 x 10 6 PBMC per pozzetto in 2 ml di mezzo in ogni pozzetto di una piastra a pozzetti 6.
- Incubare le piastre a 37 ° C con 5% di CO 2 per 2 ore. Dopo 2 ore monociti avranno aderito alla plastica.
- Per consentire monociti appena isolati di differenziarsi in hMDMs, aspirare il terreno e sostituirlo con 2 ml hMDM media (RPMI 1640, il 10% decomplemented siero fetale di vitello (FCS), 2 mM L-glutammina e l'1% di penicillina-streptomicina) integrato con ricombinante umano macrofagi fattore stimolante le colonie (RHM-CSF) ad una concentrazione finale di 10 ng / ml.
- Incubare le cellule a 37 ° C con 5% di CO 2 per 11 giorni (Figura 1).
- Al giorno 11 rimuovere il supporto e lavare 2 volte con 2 ml di terreno hMDM freddo per pozzetto. Successivo lavare i pozzetti 2 volte con 1 ml di freddo 1x PBS.
- Per staccare hMDMs completamente differenziate, lavare ogni tempo ben 1 con 1 ml di freddo 1x PBS con 2 mm di acido ethylenediaminetetraacetatic (EDTA) e incubare le cellule in 2 ml di PBS freddo 1x con 2 mM EDTA per pozzetto per 15-60 minuti a 4 ° C.
NOTA: Metodi alternativi per staccare esistono le cellule (ad esempio, tripsina o attività tripsina-simile) che potrebbe dare buoni risultati 11. - Dopo il distacco (completato pipettando gentilmente su e giù nel pozzo), raccogliere le cellule e metterli in un tubo da 50 ml in ghiaccio contenente 10 ml di terreno hMDM freddo.
- Centrifugare le cellule a 218 xg per 5 min a 20 ° C, risospendere il pellet in 10 ml di terreno hMDM fredda, e contare le cellule diluendo 1/20 in Trypan Blue.
- Seme le cellule a 1 x 10 6 hMDMs a 35 millimetri di vetro microscopia grade piatto fondo e incubare le piastre a 37 ° C con 5% di CO 2 per 1 giorni.
2. Produzione e quantificazione di HIV-1 Stocks virali
NOTA: NLR4.3 HIV-1 gag-iGFP (Green Fluorescent Protein) che trasportano una busta R5-tropico, dono di M. Schindler 10 viene utilizzato per infettare i macrofagi e le cellule infette vedere in tempo reale.
- Produrre stock virali mediante trasfezione delle cellule embrionali umane del rene 293T (2 x 10 6 in 100 millimetri piatto) con 6 ug del corrispondente DNA provirale utilizzando un reagente di trasfezione commerciale.
- Quantificare l'infettività delle riserve di virus utilizzando le cellule indicatore HeLa TZM-bl (recanti il gene beta-galattosidasi sotto il controllo di HIV-1 LTR) utilizzando diluizioni seriali dei titoli virali seguita da una colorazione ß-galattosidasi delle cellule e il conteggio le cellule di blu 12.
3. L'infezione di hMDMs con HIV-1
- Aggiungere il virus ad una molteplicità di infezione (MOI) 0,02-0,03 in 1 ml di terreno hMDM per hMDMs (cellule placcati in 1.13). Per controllare pozzetti aggiungere solo 1 ml di terreno hMDM e incubare le piastre a 37 ° C con 5% di CO 2 per 2 giorni (Figura 1).
- Al giorno 2 Lavare le cellule con hMDM medIUM 3 volte e aggiungere 1 ml di terreno hMDM fresco per ogni piatto. Incubare le cellule a 37 ° C con 5% di CO 2 per 6 giorni (Figura 1).
4. Opsonizzazione di pecore Globuli rossi
- Per la preparazione di 7 x 10 6 GRP che per piatto, lavare i GRP che due volte con 100 ml di soluzione contenente 0,1% di sieroalbumina bovina (BSA) in PBS 1x con centrifugazione a 600 xg per 4 min.
- Risospendere le GRP che lavata in 500 microlitri 1x PBS / BSA 0,1% con IgG di coniglio anti-GRP che ad una concentrazione sub-agglutinante per 5 ml di GRP che e incubare con rotazione a temperatura ambiente per 30 min.
NOTA: per determinare la concentrazione sub-agglutinante di anti-GRP che IgG, preparare diluizioni seriali di IgG (magazzino di concentramento di 13,1 mg / ml) da 1/50 a 1 / 25.600 in 20 microlitri in una piastra da 96 pozzetti. Aggiungere 2 x 10 6 GRP che in 20 microlitri di ogni bene e mettere il piatto in una stanza buia durante diverse ore. La concentrazione sub-agglutinante è ildiluizione del pozzo poco prima del bene con agglutinazione (IgG + GRP che formano una rete). - Dopo la rotazione, centrifugare le-GRP che IgG-opsonizzati a 600 xg per 4 minuti e lavare con 100 ml di 1x PBS / BSA 0,1% con centrifugazione a 600 xg per 4 minuti.
- Risospendere le-GRP che IgG-opsonizzati in media pre-riscaldato rosso fenolo-libero RPMI integrato con 2 mM L-glutammina e l'1% di penicillina-streptomicina (1 ml / piatto).
5. cellulare dal vivo microscopio video fagocitosi Assay
- Utilizzare un sistema di imaging confocale come un sistema disco rotante dotato di una camera di riscaldamento a 37 ° C con CO 2 passa attraverso una bottiglia con acqua per umidificazione.
- Accendere la camera di riscaldamento prima dell'esperimento di avere palco microscopio a 37 ° C prima dell'inizio del saggio fagocitosi. Accendere il microscopio e il computer, e caricare il software di imaging.
- Ottimizzare le impostazioni di imaging come la velocità di scansione, magnification, risoluzione, ecc avere almeno una cella per campo e all'immagine un fotogramma ogni minuto tra 60 a 120 min.
NOTA: Qui, il campione viene ripreso un fotogramma ogni minuto durante 60 minuti con 63X lente. - Porre la capsula di imaging sul palco microscopio. Regolare la messa a fuoco e la posizione per trovare una sola intera HIV-1 macrofagi infettati nel campo. Utilizzare le impostazioni di eccitazione / emissione appropriate in base al sistema di imaging utilizzata e sonda. Includere un canale luminoso campo (BF) per osservare phagosomes (Figura 1ii). Ottimizzare l'aspetto delle diverse immagini del canale regolando la percentuale di tempo di luce e l'esposizione trasmessa.
NOTA: Qui, NLR4.3 HIV-1 gag-iGFP è stato eccitato con un laser a 491 nm con 50 msec di tempo di esposizione con il 20% di laser (Figura 1i). - Rimuovere il piatto di imaging e aggiungere 1 ml di sospensione SRBC a 7 x 10 6 / ml GRP che al piatto (Figura 1).
- Centrifugare a 500 xg per2 min a RT per sincronizzare fagocitosi, registrare il tempo alla fine di questa centrifugazione e tornare il piatto allo stadio.
- Ottimizzare la messa a fuoco e cattura GFP e immagini BF in Z-stack (tutto lo spessore della cella con un passo distanza di 0,3 micron - solitamente 20 aerei) ogni minuto per almeno 1 ora. Salvare il video time-lapse nel file-formato nativo del sistema di imaging utilizzato.
NOTA: Qui, filmati time-lapse sono stati salvati nel formato nativo, i file * .stk.
6. L'analisi dei filmati time-lapse
- Per l'editing video, fare clic sul menu a discesa "Apps" e nella scheda "Review Multi Dimensional Data" nel software di editing video.
- Per aprire il file, cliccare su "Seleziona file base" e poi su "Seleziona Directory". Nella casella "Set di dati", selezionare l'acquisizione da analizzare (in formato .nd) e cliccare su "Visualizza". I dati sono rappresentati in una tabella nel tempo in una colonnad Z-piano in linea.
- Per rappresentare l'infezione in un Z-proiezione (Figura 2, i pannelli di sinistra), selezionare 491 nm di lunghezza d'onda nella casella "lunghezze d'onda" e fare clic sulla scheda "proiezione Z" con "tutti i piani".
- Per analizzare una sequenza temporale, selezionare la lunghezza d'onda BF nella casella "lunghezze d'onda" e scegliere il piano ottimale su l'asse Z per distinguere GRP che esterni (Figura 2, punta di freccia rossa), GRP che interni (Figura 2, cerchio rosso) e il nucleo ( Figura 2, cerchio blu).
- Per salvare i montaggi video, cliccare su "Selezione [di X] scheda" e poi su "Load Image (s)". Infine, salvare le immagini caricate in formato tif e aprirli prossima sul software ImageJ.
- Utilizzare il plug "Manual Tracking" sul software ImageJ per misurare la posizione del nucleo e le diverse phagosomes osservate nel canale BF (Figura 3).
- Download il plugin "Allineamento manuale" sul sito web ImageJ. Su ImageJ, aprire il plugin e la sequenza di immagini da analizzare (Figura 3, punti 1 e 2).
- Inserire le impostazioni quali "Intervallo di tempo", che rappresenta quantità di tempo tra i fotogrammi adiacenti, e la "taratura x / y", che rappresenta la distanza per pixel (Figura 3, punto 3).
NOTA: Qui, salvare la sequenza di immagini con un intervallo di tempo di 1 min tra ogni fotogramma e x / y calibrazione di 0,205 micron perché lo zoom 63X e una fotocamera con dimensioni pixel di 6,45 x 6,45 micron 2 vengono utilizzati. - Per avviare il monitoraggio, cliccare su "Add Track" (Figura 3, punto 4) e fare clic su un centro di SRBC a la prima volta quando viene interiorizzato. Il fotogramma successivo appare automaticamente.
- Continuare a fare clic sul centro SRBC in tutti i fotogrammi di avere posizioni diverse nel corso del tempo (figura 3, passo 6 nel riquadro rosso).
- Iniziare a monitorare il nucleo che la sua posizione in tutti i fotogrammi facendo clic sul suo centro. Traccia successiva le phagosomes (cliccando sul suo centro) uno per uno, al momento (frame) della loro internalizzazione, diversa in funzione della GRP che. Per comodità di vedere SRBC sul canale BF, utilizzare la finestra "Luminosità e contrasto" durante l'inseguimento (Figura 3, punto 5).
- Tra ogni monitoraggio SRBC, cliccare su "Add Track" (Figura 3, punto 4) per avere una nuova traccia. Il numero di inseguimento sarà cambiata nella seconda colonna, "Track n °" della tabella dei risultati (Figura 3, punto 6).
- Salvare i dati in un foglio di continuare alla fase successiva dell'analisi.
- Utilizzare il software foglio di calcolo per calcolare la distanza percorsa di fagosomi contenenti GRP che verso il nucleo e la velocità dei phagosomes durante i primi 5 minuti dopo l'internalizzazione di GRP che (Fifigura 4).
- Aprire la tabella foglio di calcolo e un nuovo file di foglio di calcolo. Trasferire in questo nuovo file solo i seguenti parametri, il tempo, gli assi X e Y coordinate del nucleo e tutti i GRP che (figura 4a).
- Per calcolare la distanza tra GRP che il nucleo e con solo loro coordinate, considerare il nucleo e un SRBC in un piano XY coordinata (Figura 4B).
NOTA: La distanza tra due punti è la lunghezza del percorso che li collega. Nel piano, la distanza tra SRBC e il nucleo è dato dal teorema di Pitagora.- Se c (la distanza tra il nucleo e il SRBC) indica la lunghezza dell'ipotenusa e ae b indicano le lunghezze degli altri due lati, esprimere teorema di Pitagora 'come equazione di Pitagora:

NOTA: Allo stesso tempo, il ortonormale, la distanza orizzontalea è (x nucleo -x SRBC) e la distanza verticale b è (y nucleo -y SRBC). - Così, calcolare la distanza tra il nucleo e il SRBC (Figura 4A, scatola viola) da:
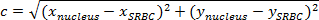
NOTA: Moltiplicare il valore della distanza ottenuto (in pixel) mediante calibrazione x / y di avere distanza in micron. Qui, la calibrazione x / y è 0,205 micron. - Ad ogni volta, sottrarre la distanza tra il nucleo e il SRBC alla distanza iniziale (Figura 4C).
- Tracciare la misura contro il tempo per il primo 5 min. Applicare un trend-line lineare (Figura 5A) per la trama e determinare la pendenza della linea di tendenza lineare che rappresenta la velocità phagosome verso il nucleo durante i primi 5 minuti dopo l'internalizzazione SRBC.
- Collazionare i dati per calcolare l'errore medio e statistica dii valori di velocità e li trama in forma adeguata utilizzando il software appropriato (Figura 5B).
- Se c (la distanza tra il nucleo e il SRBC) indica la lunghezza dell'ipotenusa e ae b indicano le lunghezze degli altri due lati, esprimere teorema di Pitagora 'come equazione di Pitagora:
Risultati
FCR-mediata fagocitosi da parte hMDMs HIV-1-infetti e non infetti è descritto qui utilizzando GRP che IgG-opsonizzati come bersagli modello (Figura 1). I passaggi critici di questo protocollo sono la preparazione del hMDMs e l'infezione da HIV-1. Infatti, la resa e la qualità dei macrofagi differenziati varia tra i donatori, così come il tasso di infezione con efficienze nell'intervallo 10-40%. Inoltre, la preparazione di IgG-opsonizzati-GRP che è anche impor...
Discussione
Questa tecnica ha diversi passaggi critici. In primo luogo, la preparazione di hMDMs e l'infezione da HIV-1 è critico poiché la percentuale di infezione è donatore dipendente. Da notare, abbiamo deciso di utilizzare macrofagi che non sono polarizzati in vitro prima dell'infezione, perché lo stato macrofagi potenzialmente incontrate dal virus in vivo non è stata ben caratterizzata finora. Abbiamo controllato l'espressione di diversi marcatori di superficie, il che indica che i macrofagi...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
We thank Dr Jamil Jubrail for reading the manuscript. This work was supported by grants from CNRS, Inserm, Université Paris Descartes, Agence Nationale de la Recherche (2011 BSV3 025 02), Fondation pour la Recherche Médicale (FRM DEQ20130326518 including a doctoral fellowship for GLB) and Agence Nationale de Recherche sur le SIDA et les hépatites virales (ANRS, including a post-doctoral fellowship for CD) to FN. A. Dumas was supported by doctoral fellowships from Université Paris Descartes and Sidaction.
Materiali
| Name | Company | Catalog Number | Comments |
| Falcon 100mm TC-Treated Cell Culture Dish | Corning | 353003 | For viral production |
| Glass Bottom Dishes 35 mm uncoated 1.5 | MatTek corporation | P35G-1.5-14-C Case | For acquisition |
| Falcon Tissue Culture Plates 6-well | Thermo Fischer Scientific | Corning. Inc. 353934 | For human monocyte-derived macrophages |
| Ficoll-Plaque PLUS | Dominique Dutscher | 17-1440-03 | a neutral, highly branched, high-mass, hydrophilic polysaccharide in solution for density centrifugation |
| DPBS, no calcium, no magnesium | Thermo Fischer Scientific | 14190-094 | Room temperature |
| Dulbecco's Modified Eagle Medium (DMEM) 1X, liquid (High Glucose) | GIBCO, Molecular probes | 31966-021 | Conserved at 4°C ; for HEK cells culture |
| RPMI 1640 medium GLUTAMAX Supplement | Life technologies | 61870-010 | Conserved at 4°C; for hMDMs culture |
| Fœtal Calf Serum (FCS) | Eurobio | CVFSVF0001 | Conserved at -20°C ; decomplemented |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fischer Scientific | 15140-122 | Conserved at -20°C ; for hMDMs culture |
| RPMI 1640 medium, no phenol red (10x500 ml) | Life technologies | 11835-105 | Warm in 37°C water bath before use ; for phagocytosis assay |
| FuGENE6 Transfection Reagent | Promega | E2692 | Conserved at 4°C ; for viral production |
| Sheep red blood cells (SRBCs) | Eurobio | DSGMTN00-0Q | Conserved in Alsever buffer at 4°C before use |
| Anti-sheep red blood cells IgG | MP Biomedicals | 55806 | Conserved at 4°C |
| Bovine Serum Albumin heat shock fraction, pH 7, ≥98% | Sigma | A7906 | Conserved at -20°C |
| Inverted microscope DMI600 | Leica | ||
| Confocal Spinning Disk Unit CSU-X1M1 | Yokogawa | ||
| 491 nm 50mW laser | COBOLT CALYPSO | ||
| HCX PL APO CS Objectif | Leica | Objective lens ; Magnification 100x ; Numerical aperture 1.40 ; Immersion oil | |
| CoolSnap HQ2 (FireWire) Camera | Photometrics | Pixel size 6.45 x 6.45 µm ; Definition 1392 x 1040 ; Encoding the image in 14 Bit | |
| Metamorph 7.7.5 software | Molecular Devices | For the control of the microscope | |
| GraphPad Prism software | For the statistics analysis |
Riferimenti
- Flannagan, R. S., Jaumouille, V., Grinstein, S. The cell biology of phagocytosis. Annu. Rev. Pathol. 7, 61-98 (2012).
- Niedergang, F. . Encyclopedia of Cell Biology. 2, 751-757 (2016).
- Blocker, A., Griffiths, G., Olivo, J. C., Hyman, A. A., Severin, F. F. A role for microtubule dynamics in phagosome movement. J Cell Sci. 111 (Pt 3), 303-312 (1998).
- Blocker, A., et al. Molecular requirements for bi-directional movement of phagosomes along microtubules. J Cell Biol. 137, 113-129 (1997).
- Flannagan, R. S., Cosio, G., Grinstein, S. Antimicrobial mechanisms of phagocytes and bacterial evasion strategies. Nat Rev Microbiol. 7, 355-366 (2009).
- Feasey, N. A., Dougan, G., Kingsley, R. A., Heyderman, R. S., Gordon, M. A. Invasive non-typhoidal salmonella disease: an emerging and neglected tropical disease in Africa. Lancet. 379, 2489-2499 (2012).
- Mazzolini, J., et al. Inhibition of phagocytosis in HIV-1-infected macrophages relies on Nef-dependent alteration of focal delivery of recycling compartments. Blood. 115, 4226-4236 (2010).
- Dumas, A., et al. The HIV-1 protein Vpr impairs phagosome maturation by controlling microtubule-dependent trafficking. J Cell Biol. 211, 359-372 (2015).
- Greenberg, S., el Khoury, J., Kaplan, E., Silverstein, S. C. A fluorescence technique to distinguish attached from ingested erythrocytes and zymosan particles in phagocytosing macrophages. J. Immunol. Methods. 139, 115-122 (1991).
- Koppensteiner, H., Banning, C., Schneider, C., Hohenberg, H., Schindler, M. Macrophage internal HIV-1 is protected from neutralizing antibodies. J Virol. 86, 2826-2836 (2012).
- Gartner, S. The macrophage and HIV: basic concepts and methodologies. Methods Mol Biol. 1087, 207-220 (2014).
- Wei, X., et al. Emergence of resistant human immunodeficiency virus type 1 in patients receiving fusion inhibitor (T-20) monotherapy. Antimicrob Agents Chemother. 46, 1896-1905 (2002).
- Harrison, R. E., Bucci, C., Vieira, O. V., Schroer, T. A., Grinstein, S. Phagosomes fuse with late endosomes and/or lysosomes by extension of membrane protrusions along microtubules: role of Rab7 and RILP. Mol Cell Biol. 23, 6494-6506 (2003).
- Toyohara, A., Inaba, K. Transport of phagosomes in mouse peritoneal macrophages. J Cell Sci. 94 (Pt 1), 143-153 (1989).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon