É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Migração fagossomo e velocidade medida no Live primárias macrófagos humanos infectados com o HIV-1
Neste Artigo
Resumo
We describe a method to measure the velocity of phagosomes moving towards the cell center in living cells infected with or without the human immunodeficiency virus (HIV) type 1, using spinning disk confocal fluorescence microscopy to identify fluorescent infected cells and bright field microscopy to detect phagosomes.
Resumo
Macrophages are phagocytic cells that play a major role at the crossroads between innate and specific immunity. They can be infected by the human immunodeficiency virus (HIV)-1 and because of their resistance to its cytopathic effects they can be considered to be persistent viral reservoirs. In addition, HIV-infected macrophages exhibit defective functions that contribute to the development of opportunistic diseases.
The exact mechanism by which HIV-1 impairs the phagocytic response of macrophages was unknown. We had previously shown that the uptake of various particulate material by macrophages was inhibited when they were infected with HIV-1. This inhibition was only partial and phagosomes did form within HIV-infected macrophages. Therefore, we focused on analyzing the fate of these phagosomes. Phagosome maturation is accompanied by migration of these compartments towards the cell center, where they fuse with lysosomes, generating phagolysosomes, responsible for degradation of the ingested material. We used IgG-opsonized Sheep Red Blood Cells as a target for phagocytosis. To measure the speed of centripetal movement of phagosomes in individual HIV-infected macrophages, we used a combination of bright field and fluorescence confocal microscopy. We established a method to calculate the distance of phagosomes towards the nucleus, and then to calculate the velocity of the phagosomes. HIV-infected cells were identified thanks to a GFP-expressing virus, but the method is applicable to non-infected cells or any type of infection or treatment.
Introdução
Macrophages play a major role in the innate immune system and in homeostasis. They are professional phagocytes that internalize and eliminate pathogens and debris by a process called phagocytosis 1,2. The phagosome, the closed compartment that forms after the engulfment of particulate material, undergoes a series of fusion and fission events with endocytic compartments, leading to a degradative compartment called the phagolysosome. This compartment has an acidic pH, due to the acquisition of proton-pumping v-ATPases, contains hydrolytic enzymes and is enriched in lysosomal-associated membrane proteins (LAMPs). The maturation of phagosomes is accompanied by their migration on microtubules 3,4 towards the cell center to reach a perinuclear location where lysosomes are accumulated.
Many pathogens have been reported to hijack phagosome maturation, including bacteria with intracellular lifestyles that modify the vacuolar environment where they reside 5. The Human Immunodeficiency Virus (HIV)-1 targets macrophages in addition to T cells. As macrophages are resistant to the cytopathic effects of the virus, unlike T cells, they can be considered as a reservoir for the virus. In addition, macrophages infected with HIV-1 show defective phagocytic functions and contribute to the emergence of opportunistic diseases. In particular, severe invasive non-typhoidal Salmonella disease caused by Salmonella Typhimurium ST313 has been prevalent for the last three decades in sub-Saharan African children or adults infected with HIV 6. It has been estimated that the risk of developing tuberculosis is more than 20 times greater in people living with HIV than among those without HIV infection.
For all these reasons, it is important to better define the molecular mechanisms underlying the phagocytic defects in HIV-infected macrophages. We have shown that the uptake of particulate material, opsonized particles, bacteria or fungi, was inhibited in HIV-infected macrophages 7. Given that this inhibition is partial, we then set out to analyze the fate of the internalized phagosomes in HIV infected human macrophages 8. Because phagosome maturation is tightly connected with migration to the cell center and fusion with lysosomes, a defect in phagosomal maturation can be due to modifications of the trafficking modalities in the infected cell. The method described here uses IgG-opsonized Sheep Red Blood Cells (IgG-SRBCs) as a model to target receptor-mediated phagocytosis and in particular receptors for the Fc portion of immunoglobulins (FcR). These particles are easier to image in bright field (BF) than latex beads because extracellular and intracellular SRBCs show different refraction properties 9. To measure the velocity of phagosomes moving towards the nucleus in HIV-infected macrophages, we used a fluorescent virus 10 and set up a simple manual tracking method that is described here. The method does not require advanced programming and simply uses ImageJ. It is amenable to adherent cells and any type of particle or pathogen that can be visualized with bright field microscopy or with fluorescent imaging.
Protocolo
O protocolo tem de ser efectuada em estrita conformidade com as legislações nacionais e internacionais e regulamentos locais. O sangue de doadores saudáveis que deram o seu consentimento para doar sangue para fins de pesquisa foi obtido a partir de centros de transfusão de sangue com o qual as instituições assinaram acordo. protecções especiais devem ser tomados ao usar sangue humano. As experiências com o HIV-1 deve ser realizado em um nível de segurança biológica, ou 2 3 (BSL-3 ou 2) laboratório de acordo com a legislação local.
1. Preparação de macrófagos humanos Monócitos-derivados (hMDMs) por centrifugação em gradiente de densidade e por adesão Selecção
- Comece com o sangue fresco de dadores saudáveis (9 mL). Dilui-se todo o volume de sangue fresco com 1x estéril salina tamponada com fosfato (PBS) sem Ca 2+ e Mg 2+ para se obter um volume final de 70 ml e adicionar cuidadosamente o sangue diluído em dois tubos de 50 mL cónicos (35 mL por tubo) , no topo de 15 ml de umaeutral, altamente ramificado, de elevada massa, polissacárido hidrófilo na solução já em cada tubo.
- Centrífuga ambos os tubos de sangue a 537 xg durante 20 min a 20 ° C sem travão. Em seguida, recolher as células mononucleares do sangue periférico (PBMCs) contidos no anel célula nebuloso na interface e transferi-las para um novo tubo de 50 ml contendo 15 ml de PBS 1x sem Ca 2+ e Mg 2+.
- Centrifugar as células a 218 xg durante 5 min a 20 ° C e ressuspender o sedimento em 45 ml de PBS 1x sem Ca 2+ e Mg 2+.
- Centrifugar as células a 218 xg durante 5 min a 20 ° C, ressuspender o sedimento em 10 ml de PBS 1x sem Ca 2+ e Mg 2+, e contar as células por diluição para uma diluição final de 1/200 em azul de tripano.
- Centrifugar as células a 218 xg durante 5 min a 20 ° C e ressuspender o sedimento em meio RPMI (Roswell Park Memorial Institute) 1640 suplementado com 2 mM de L-glutamina e 100 ug / ml Penicillin-estreptomicina a ter 7 x 10 6 PBMC por cavidade em 2 ml de meio em cada poço de uma placa de 6 bem.
- Incubar as placas a 37 ° C com 5% de CO 2 durante 2 h. Após 2 h os monócitos vai aderiram ao plástico.
- Para permitir que os recém-isolados monócitos de se diferenciar em hMDMs, Aspirar o meio e substitui-la com 2 ml hMDM meio (RPMI 1640, 10% descomplementado Soro Fetal de Vitela (FCS), 2 mM de L-glutamina e 1% de penicilina-estreptomicina) suplementado com factor de estimulação do macrófago humano recombinante Colony (RHM-CSF), a uma concentração final de 10 ng / ml.
- Incubar as células a 37 ° C com 5% de CO 2 durante 11 dias (Figura 1).
- No dia 11, remover o meio e lava-se 2 vezes com 2 ml de meio por poço hMDM frio. De seguida, lavar cada poço 2 vezes com 1 ml de PBS frio 1x.
- Para retirar hMDMs totalmente diferenciadas, lavar cada poço 1 hora com 1 ml de PBS frio 1x com ácido ethylenediaminetetraacetatic 2 mM (EDTA) e incuba-se as células em 2 ml de PBS frio 1x com EDTA 2 mM por poço, durante 15-60 min a 4 ° C.
NOTA: Os métodos alternativos para separar existem as células (por exemplo, tripsina ou atividades de tripsina-like) que possa dar bons resultados 11. - Depois de descolamento (concluído com cuidado pipetando cima e para baixo no poço), recolher as células e colocá-los em um tubo de 50 ml de gelo contendo 10 ml de meio hMDM frio.
- Centrifugar as células a 218 xg durante 5 min a 20 ° C, ressuspender o sedimento em 10 ml de meio frio hMDM, e contar as células por diluição de 1/20 em azul de tripano.
- Semear as células a 1 x 10 6 hMDMs por 35 milímetros de vidro da classe microscopia prato inferior e incubar as placas a 37 ° C com 5% de CO 2 durante 1 dia.
2. Produção e quantificação do VIH-1 reservas virais
NOTA: NLR4.3 HIV-1 Gag-IGFP (Green Fluorescent Protein) carregando um envelope R5-trópico, presente de M. Schindler 10 é utilizado para infectar macrófagos e células infectadas para ver em tempo real.
- Produção de reservas virais através de transfecção de células humanas embrionárias de rim 293T (2 x 10 6 em 100 milímetros prato) com 6 ug do ADN pró-viral correspondente usando um reagente de transfecção comercial.
- Quantificar a infecciosidade das reservas de vírus com as células indicadoras HeLa TZM-bl (possuindo o gene ß-galactosidase sob o controlo do HIV-1 LTR) utilizando diluições em série dos stocks virais, seguido de uma coloração ß-galactosidase das células e contagem células de azul 12.
3. A infecção de hMDMs com HIV-1
- Adicionar o vírus a uma multiplicidade de infecção (MOI) 0,02-0,03 em 1 ml de meio para hMDM hMDMs (células plaqueadas em 1,13). Para controlar poços adicionar apenas 1 ml de meio hMDM e incubar os pratos a 37 ° C com 5% de CO 2 durante 2 dias (Figura 1).
- No dia 2 lavar as células com hMDM medio 3 vezes e adicionar 1 ml de meio hMDM fresco por prato. Incubar as células a 37 ° C com 5% de CO 2 durante 6 dias (Figura 1).
4. Opsonização de ovelhas Glóbulos vermelhos
- Para a preparação de 7 x 10 6 por placa de SRBCs, lavar os SRBCs duas vezes em 100 ul de solução contendo 0,1% de Albumina de Soro Bovino (BSA) em PBS 1x com centrifugação a 600 x g durante 4 min.
- Ressuspender as SRBCs lavadas com 500 ul de 1x PBS / BSA a 0,1% com IgG de coelho anti-SRBCs a uma concentração sub-aglutinação por 5 ul de SRBCs e incubar com rotação à temperatura ambiente durante 30 min.
NOTA: Para determinar a concentração sub-aglutinação de anti-IgG de SRBCs, preparar diluições em série de IgG (concentração de estoque de 13,1 mg / ml) a partir de 1/50 a 1 / 25.600 em 20 ul numa placa de 96 poços. Adicionar 2 x 10 6 SRBCs em 20 ul em cada cavidade e colocar a placa de um quarto escuro durante várias horas. A concentração sub-aglutinação é adiluição do poço imediatamente antes do poço com aglutinação (IgG + SRBCs formando uma rede). - Após rotação, centrifugar os SRBCs-IgG-opsonizadas a 600 xg durante 4 min e lava-se com 100 ul de 1x PBS / BSA a 0,1%, com centrifugação a 600 x g durante 4 min.
- Ressuspender as SRBCs-IgG-opsonizados em meio pré-aquecido Fenol RPMI sem vermelho suplementado com 2 mM de L-glutamina e 1% de penicilina-estreptomicina (1 ml / prato).
5. células vivas Vídeo Microscopy fagocitose Assay
- Usar um sistema de imagiologia confocal, tais como um sistema de disco giratório equipado com uma câmara de aquecimento a 37 ° C com CO 2 passando através de um frasco com água para a humidificação.
- Ligar a câmara de aquecimento antes da experiência ter a platina do microscópio a 37 ° C antes do início do ensaio de fagocitose. Ligue o microscópio e computador e carregar o software de imagem.
- Otimizar as configurações de imagem, como velocidade de digitalização, Magnificatião, resolução, etc, para ter pelo menos uma célula por campo e a imagem de um quadro de cada minuto entre 60 a 120 min.
NOTA: Aqui, a amostra é fotografada um quadro a cada minuto durante 60 min com 63x lente. - Coloque o prato de imagem no palco microscópio. Ajustar o foco e o local para encontrar apenas um conjunto de macrófagos infectados pelo HIV-1 no campo. Use as configurações de excitação / emissão adequadas com base no sistema de imagem usados e sonda. Incluir um canal brilhante campo (BF) para observar phagosomes (Figura 1ii). Optimizar a aparência das diferentes imagens de canal, ajustando a percentagem de tempo de exposição a luz e transmitidos.
NOTA: Aqui, NLR4.3 HIV-1 Gag-IGFP estava animado usando um laser de 491 nm com 50 ms de tempo de exposição com 20% do laser (Figura 1-I). - Remover o prato de imagem e adicionar 1 ml de uma suspensão SRBC a 7 x 10 6 / ml SRBCs ao prato (Figura 1).
- Centrifugar a 500 xg durante2 min à temperatura ambiente para sincronizar a fagocitose, registar o tempo no final desta centrifugação e devolver o prato para a fase.
- Optimizar a captura de foco e GFP e imagens BF em z-pilhas (em toda a espessura da célula com um passo a distância de 0,3 um - 20 geralmente planos) a cada minuto durante pelo menos 1 h. Salvar o vídeo de lapso de tempo no arquivo de formato nativo do sistema de imagem utilizado.
NOTA: Aqui, os filmes de lapso de tempo foram salvos no formato nativo, arquivos * .stk.
6. Análise da Time-lapse Filmes
- Para edição de vídeo, clique no menu drop-down "Apps" e na guia "Análise multidimensional de dados" no software de edição de vídeo.
- Para abrir o arquivo, clique em "Selecionar Arquivo Base", em seguida, em "Selecionar Directory". No "Conjuntos de dados", selecione a aquisição de analisar (em formato .nd) e clique em "Ver". Os dados são representados numa tabela com o tempo em uma colunad Z-plano na linha.
- Para representar a infecção em um Z-projeção (Figura 2, painéis da esquerda), selecione 491 nm de comprimento de onda na caixa "comprimentos de onda" e clique na aba "Z projecção" com "todos os planos".
- Para analisar uma seqüência de tempo, selecione o comprimento de onda BF na caixa "comprimentos de onda" e escolha o plano ideal no eixo Z para distinguir SRBCs externos (Figura 2, seta vermelha), SRBCs internos (Figura 2, círculo vermelho) e o núcleo ( Figura 2, círculo azul).
- Para salvar as montagens de vídeo, clique em "Selecção [do X]" e depois em "Load Image (s)". Por fim, salve as imagens carregadas em formato .tif e abri-los em seguida em software ImageJ.
- Use o plugin "Rastreio Manual" no software ImageJ para medir a posição do núcleo e os diferentes phagosomes observadas no canal BF (Figura 3).
- DowNload o plug-in "Manual de Rastreamento" no site do ImageJ. No ImageJ, abra o plugin e a sequência de imagens a serem analisadas (Figura 3, passo 1 e 2).
- Introduzir as definições tais como "intervalo de tempo" que representa quantidade de tempo entre quadros adjacentes, eo "calibração x / y", que representa a distância per pixel (Figura 3, passo 3).
NOTA: Aqui, guardar a sequência de imagens com um intervalo de tempo de 1 minuto entre cada armação e a calibração x / y de 0,205 uM porque o zoom 63X e uma câmara com tamanho de pixel de 6,45 x 6,45? M 2 são usados. - Para iniciar o rastreamento, clique em "Adicionar faixa" (Figura 3, passo 4) e clique em um centro de SRBC no primeiro tempo quando é internalizado. O próximo quadro aparece automaticamente.
- Continue a clicar sobre o centro SRBC em todos os quadros de ter posições diferentes durante o tempo (Figura 3, Passo 6 na caixa vermelha).
- Começar a controlar o núcleo que a sua posição em todos os quadros, clicando em seu centro. Próximo acompanhar os phagosomes (clicando em seu centro), um por um na época (frame) de sua interiorização, diferente em função do SRBCs. Para maior comodidade para ver SRBC no canal BF, use a janela "Brilho e Contraste" durante o acompanhamento (Figura 3, passo 5).
- Entre cada rastreamento SRBC, clique em "Adicionar faixa" (Figura 3, passo 4) para ter uma nova pista. O número de rastreamento será alterado na segunda coluna, "Track n °" da tabela de resultados (Figura 3, passo 6).
- Salvar os dados em uma planilha para continuar para a próxima etapa da análise.
- Use um software de planilha para calcular a distância percorrida de phagosomes contendo SRBCs para o núcleo e a velocidade dos phagosomes durante os primeiros 5 minutos após a internalização de SRBCs (Fifigura 4).
- Abra a tabela de folha de cálculo e um novo arquivo de planilha. Transferir para este novo arquivo os seguintes parâmetros somente, tempo, o X e Y coordenadas do núcleo e todos os SRBCs (Figura 4A).
- Para calcular a distância entre o núcleo e SRBCs com apenas as suas coordenadas, considerar o núcleo e uma SRBC num plano XY de coordenadas (Figura 4B).
NOTA: A distância entre dois pontos é o comprimento do caminho de os ligar. No plano, a distância entre o núcleo e SRBC é dada pelo teorema de Pitágoras.- Se C (a distância entre o núcleo e o SRBC) indica o comprimento da hipotenusa e a e b representam os comprimentos dos outros dois lados, expressar o teorema de Pitágoras como a equação de Pitágoras:

NOTA: Ao mesmo tempo, no ortonormal, a distância horizontalum é (x núcleo -x SRBC) e a distância vertical b é (Y-Y núcleo SRBC). - Assim, o cálculo da distância entre o núcleo e o SRBC (Figura 4A, a caixa roxo) por:
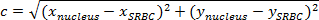
NOTA: multiplicar o valor de distância obtida (em pixels) por x / y de calibragem ter a distância em uM. Aqui, a calibração x / y é 0,205 uM. - Em cada tempo, subtrair a distância entre o núcleo e o SRBC à distância inicial (Figura 4C).
- Traça-se a medição em função do tempo para os primeiros 5 min. Aplicar uma linha de tendência linear (Figura 5A) para a trama e determinar a inclinação da linha de tendência linear que representa a velocidade fagossoma para o núcleo durante os primeiros 5 minutos após a internalização SRBC.
- Agrupar os dados para calcular o erro médio e de estatísticaos valores de velocidade e traçar-los em uma forma adequada, utilizando o software apropriado (Figura 5B).
- Se C (a distância entre o núcleo e o SRBC) indica o comprimento da hipotenusa e a e b representam os comprimentos dos outros dois lados, expressar o teorema de Pitágoras como a equação de Pitágoras:
Resultados
Fagocitose mediada por FcR-hMDMs HIV-1 infectadas e não-infectadas é descrito aqui usando SRBCs IgG-opsonizadas como alvos modelo (Figura 1). Os passos críticos deste protocolo são a preparação de hMDMs ea infecção com HIV-1. Com efeito, o rendimento e a qualidade dos macrófagos diferenciados varia entre dadores, bem como a taxa de infecção com eficiências na gama de 10-40%. Além disso, a preparação de IgG-opsonizado-SRBCs também é importante para evitar...
Discussão
Esta técnica tem várias etapas críticas. Em primeiro lugar, a preparação de hMDMs e a sua infecção por HIV-1 é crítica, porque a percentagem de infecção é dependente do dador. De nota, decidimos usar macrófagos que não são polarizados in vitro antes da infecção, pois o estado de macrófagos potencialmente encontradas pelo vírus in vivo não foi bem caracterizada medida. Nós verificamos a expressão de vários marcadores de superfície, o que indica que os macrófagos são nem nem M1 ...
Divulgações
The authors have nothing to disclose.
Agradecimentos
We thank Dr Jamil Jubrail for reading the manuscript. This work was supported by grants from CNRS, Inserm, Université Paris Descartes, Agence Nationale de la Recherche (2011 BSV3 025 02), Fondation pour la Recherche Médicale (FRM DEQ20130326518 including a doctoral fellowship for GLB) and Agence Nationale de Recherche sur le SIDA et les hépatites virales (ANRS, including a post-doctoral fellowship for CD) to FN. A. Dumas was supported by doctoral fellowships from Université Paris Descartes and Sidaction.
Materiais
| Name | Company | Catalog Number | Comments |
| Falcon 100mm TC-Treated Cell Culture Dish | Corning | 353003 | For viral production |
| Glass Bottom Dishes 35 mm uncoated 1.5 | MatTek corporation | P35G-1.5-14-C Case | For acquisition |
| Falcon Tissue Culture Plates 6-well | Thermo Fischer Scientific | Corning. Inc. 353934 | For human monocyte-derived macrophages |
| Ficoll-Plaque PLUS | Dominique Dutscher | 17-1440-03 | a neutral, highly branched, high-mass, hydrophilic polysaccharide in solution for density centrifugation |
| DPBS, no calcium, no magnesium | Thermo Fischer Scientific | 14190-094 | Room temperature |
| Dulbecco's Modified Eagle Medium (DMEM) 1X, liquid (High Glucose) | GIBCO, Molecular probes | 31966-021 | Conserved at 4°C ; for HEK cells culture |
| RPMI 1640 medium GLUTAMAX Supplement | Life technologies | 61870-010 | Conserved at 4°C; for hMDMs culture |
| Fœtal Calf Serum (FCS) | Eurobio | CVFSVF0001 | Conserved at -20°C ; decomplemented |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fischer Scientific | 15140-122 | Conserved at -20°C ; for hMDMs culture |
| RPMI 1640 medium, no phenol red (10x500 ml) | Life technologies | 11835-105 | Warm in 37°C water bath before use ; for phagocytosis assay |
| FuGENE6 Transfection Reagent | Promega | E2692 | Conserved at 4°C ; for viral production |
| Sheep red blood cells (SRBCs) | Eurobio | DSGMTN00-0Q | Conserved in Alsever buffer at 4°C before use |
| Anti-sheep red blood cells IgG | MP Biomedicals | 55806 | Conserved at 4°C |
| Bovine Serum Albumin heat shock fraction, pH 7, ≥98% | Sigma | A7906 | Conserved at -20°C |
| Inverted microscope DMI600 | Leica | ||
| Confocal Spinning Disk Unit CSU-X1M1 | Yokogawa | ||
| 491 nm 50mW laser | COBOLT CALYPSO | ||
| HCX PL APO CS Objectif | Leica | Objective lens ; Magnification 100x ; Numerical aperture 1.40 ; Immersion oil | |
| CoolSnap HQ2 (FireWire) Camera | Photometrics | Pixel size 6.45 x 6.45 µm ; Definition 1392 x 1040 ; Encoding the image in 14 Bit | |
| Metamorph 7.7.5 software | Molecular Devices | For the control of the microscope | |
| GraphPad Prism software | For the statistics analysis |
Referências
- Flannagan, R. S., Jaumouille, V., Grinstein, S. The cell biology of phagocytosis. Annu. Rev. Pathol. 7, 61-98 (2012).
- Niedergang, F. . Encyclopedia of Cell Biology. 2, 751-757 (2016).
- Blocker, A., Griffiths, G., Olivo, J. C., Hyman, A. A., Severin, F. F. A role for microtubule dynamics in phagosome movement. J Cell Sci. 111 (Pt 3), 303-312 (1998).
- Blocker, A., et al. Molecular requirements for bi-directional movement of phagosomes along microtubules. J Cell Biol. 137, 113-129 (1997).
- Flannagan, R. S., Cosio, G., Grinstein, S. Antimicrobial mechanisms of phagocytes and bacterial evasion strategies. Nat Rev Microbiol. 7, 355-366 (2009).
- Feasey, N. A., Dougan, G., Kingsley, R. A., Heyderman, R. S., Gordon, M. A. Invasive non-typhoidal salmonella disease: an emerging and neglected tropical disease in Africa. Lancet. 379, 2489-2499 (2012).
- Mazzolini, J., et al. Inhibition of phagocytosis in HIV-1-infected macrophages relies on Nef-dependent alteration of focal delivery of recycling compartments. Blood. 115, 4226-4236 (2010).
- Dumas, A., et al. The HIV-1 protein Vpr impairs phagosome maturation by controlling microtubule-dependent trafficking. J Cell Biol. 211, 359-372 (2015).
- Greenberg, S., el Khoury, J., Kaplan, E., Silverstein, S. C. A fluorescence technique to distinguish attached from ingested erythrocytes and zymosan particles in phagocytosing macrophages. J. Immunol. Methods. 139, 115-122 (1991).
- Koppensteiner, H., Banning, C., Schneider, C., Hohenberg, H., Schindler, M. Macrophage internal HIV-1 is protected from neutralizing antibodies. J Virol. 86, 2826-2836 (2012).
- Gartner, S. The macrophage and HIV: basic concepts and methodologies. Methods Mol Biol. 1087, 207-220 (2014).
- Wei, X., et al. Emergence of resistant human immunodeficiency virus type 1 in patients receiving fusion inhibitor (T-20) monotherapy. Antimicrob Agents Chemother. 46, 1896-1905 (2002).
- Harrison, R. E., Bucci, C., Vieira, O. V., Schroer, T. A., Grinstein, S. Phagosomes fuse with late endosomes and/or lysosomes by extension of membrane protrusions along microtubules: role of Rab7 and RILP. Mol Cell Biol. 23, 6494-6506 (2003).
- Toyohara, A., Inaba, K. Transport of phagosomes in mouse peritoneal macrophages. J Cell Sci. 94 (Pt 1), 143-153 (1989).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados