Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Phagosome Migration et Velocity Mesurée en direct primaire des macrophages humains infectés par le VIH-1
Dans cet article
Résumé
We describe a method to measure the velocity of phagosomes moving towards the cell center in living cells infected with or without the human immunodeficiency virus (HIV) type 1, using spinning disk confocal fluorescence microscopy to identify fluorescent infected cells and bright field microscopy to detect phagosomes.
Résumé
Macrophages are phagocytic cells that play a major role at the crossroads between innate and specific immunity. They can be infected by the human immunodeficiency virus (HIV)-1 and because of their resistance to its cytopathic effects they can be considered to be persistent viral reservoirs. In addition, HIV-infected macrophages exhibit defective functions that contribute to the development of opportunistic diseases.
The exact mechanism by which HIV-1 impairs the phagocytic response of macrophages was unknown. We had previously shown that the uptake of various particulate material by macrophages was inhibited when they were infected with HIV-1. This inhibition was only partial and phagosomes did form within HIV-infected macrophages. Therefore, we focused on analyzing the fate of these phagosomes. Phagosome maturation is accompanied by migration of these compartments towards the cell center, where they fuse with lysosomes, generating phagolysosomes, responsible for degradation of the ingested material. We used IgG-opsonized Sheep Red Blood Cells as a target for phagocytosis. To measure the speed of centripetal movement of phagosomes in individual HIV-infected macrophages, we used a combination of bright field and fluorescence confocal microscopy. We established a method to calculate the distance of phagosomes towards the nucleus, and then to calculate the velocity of the phagosomes. HIV-infected cells were identified thanks to a GFP-expressing virus, but the method is applicable to non-infected cells or any type of infection or treatment.
Introduction
Macrophages play a major role in the innate immune system and in homeostasis. They are professional phagocytes that internalize and eliminate pathogens and debris by a process called phagocytosis 1,2. The phagosome, the closed compartment that forms after the engulfment of particulate material, undergoes a series of fusion and fission events with endocytic compartments, leading to a degradative compartment called the phagolysosome. This compartment has an acidic pH, due to the acquisition of proton-pumping v-ATPases, contains hydrolytic enzymes and is enriched in lysosomal-associated membrane proteins (LAMPs). The maturation of phagosomes is accompanied by their migration on microtubules 3,4 towards the cell center to reach a perinuclear location where lysosomes are accumulated.
Many pathogens have been reported to hijack phagosome maturation, including bacteria with intracellular lifestyles that modify the vacuolar environment where they reside 5. The Human Immunodeficiency Virus (HIV)-1 targets macrophages in addition to T cells. As macrophages are resistant to the cytopathic effects of the virus, unlike T cells, they can be considered as a reservoir for the virus. In addition, macrophages infected with HIV-1 show defective phagocytic functions and contribute to the emergence of opportunistic diseases. In particular, severe invasive non-typhoidal Salmonella disease caused by Salmonella Typhimurium ST313 has been prevalent for the last three decades in sub-Saharan African children or adults infected with HIV 6. It has been estimated that the risk of developing tuberculosis is more than 20 times greater in people living with HIV than among those without HIV infection.
For all these reasons, it is important to better define the molecular mechanisms underlying the phagocytic defects in HIV-infected macrophages. We have shown that the uptake of particulate material, opsonized particles, bacteria or fungi, was inhibited in HIV-infected macrophages 7. Given that this inhibition is partial, we then set out to analyze the fate of the internalized phagosomes in HIV infected human macrophages 8. Because phagosome maturation is tightly connected with migration to the cell center and fusion with lysosomes, a defect in phagosomal maturation can be due to modifications of the trafficking modalities in the infected cell. The method described here uses IgG-opsonized Sheep Red Blood Cells (IgG-SRBCs) as a model to target receptor-mediated phagocytosis and in particular receptors for the Fc portion of immunoglobulins (FcR). These particles are easier to image in bright field (BF) than latex beads because extracellular and intracellular SRBCs show different refraction properties 9. To measure the velocity of phagosomes moving towards the nucleus in HIV-infected macrophages, we used a fluorescent virus 10 and set up a simple manual tracking method that is described here. The method does not require advanced programming and simply uses ImageJ. It is amenable to adherent cells and any type of particle or pathogen that can be visualized with bright field microscopy or with fluorescent imaging.
Protocole
Le protocole doit être effectué en stricte conformité avec les législations nationales et internationales et les réglementations locales. Le sang provenant de donneurs sains qui ont donné leur consentement au don de sang à des fins de recherche a été obtenu à partir de transfusion sanguine centres avec lesquels les institutions ont signé un accord. protections spéciales doivent être prises lors de l'utilisation du sang humain. Les expériences avec le VIH-1 doivent être réalisés dans un niveau de biosécurité 3 ou 2 (BSL-3 ou 2) Laboratoire conformément à la législation locale.
1. Préparation de macrophages humains dérivés des monocytes (hMDMs) par gradient de densité Centrifugation et sélection par Adhesion
- Commencez avec du sang frais provenant de donneurs sains (9 ml). Diluer tout le volume de sang frais avec 1x stérile tamponnée au phosphate (PBS) sans Ca2 + et Mg2 + afin d' obtenir un volume final de 70 ml et ajouter doucement le sang dilué dans 50 ml de deux tubes coniques (35 ml par tube) , au-dessus de 15 ml d'uneeutral, très ramifié, de masse élevée, polysaccharide hydrophile en solution déjà dans chaque tube.
- Centrifuger les deux tubes de sang à 537 xg pendant 20 min à 20 ° C sans frein. Ramassez ensuite les cellules mononucléaires du sang périphérique (CMSP) contenus dans l'anneau cellulaire trouble à l'interface et de les transférer dans un nouveau tube de 50 ml contenant 15 ml de PBS 1x sans Ca 2+ et Mg 2+.
- Centrifuger les cellules à 218 xg pendant 5 min à 20 ° C et à remettre en suspension le culot dans 45 ml de PBS 1x sans Ca 2+ et Mg 2+.
- Centrifuger les cellules à 218 xg pendant 5 min à 20 ° C, de remettre en suspension le culot dans 10 ml de PBS 1x sans Ca 2+ et Mg 2+, et compter les cellules par dilution à une dilution finale de 1/200 dans le bleu Trypan.
- Centrifuger les cellules à 218 xg pendant 5 min à 20 ° C et remettre en suspension le culot dans RPMI (Roswell Park Institute Memorial) 1640 complété avec 2 mM de L-glutamine et 100 pg / ml penicIllin-streptomycine avoir 7 x 10 6 PBMC par puits dans 2 ml de milieu dans chaque puits d'une plaque 6.
- Incuber les plaques à 37 ° C avec 5% de CO2 pendant 2 heures. Après 2 heures les monocytes auront adhéré à la matière plastique.
- Pour permettre monocytes fraîchement isolés à se différencier en hMDMs, aspirer le moyen et le remplacer par un milieu de 2 ml (RPMI 1640, 10% décomplémenté sérum foetal de veau (FCS), 2 mM de L-glutamine et 1% de pénicilline-streptomycine) additionné de colonie recombinante humaine du facteur de stimulation des macrophages (rhM-CSF) à une concentration finale de 10 ng / ml.
- Incuber les cellules à 37 ° C avec 5% de CO 2 pendant 11 jours (figure 1).
- Au jour 11 éliminer le milieu et laver 2 fois avec 2 ml de milieu de hMDM froid par puits. Laver ensuite chaque puits 2 fois avec 1 ml de froid 1x PBS.
- Pour détacher hMDMs complètement différenciées, lavez chaque puits 1 fois avec 1 ml de froid 1x PBS avec de l'acide ethylenediaminetetraacetatic 2 mM (EDTA) et incuber les cellules dans 2 ml de PBS froid 1x avec 2 mM d'EDTA par puits pendant 15 à 60 min à 4 ° C.
NOTE: Les méthodes alternatives pour détacher les cellules existent (par exemple, la trypsine ou les activités de trypsine) qui pourraient donner de bons résultats 11. - Après le détachement (complété par pipetage doucement monter et descendre dans le puits), recueillir les cellules et les mettre dans un tube de 50 ml sur de la glace contenant 10 ml de milieu de hMDM froid.
- Centrifuger les cellules à 218 xg pendant 5 min à 20 ° C, de remettre en suspension le culot dans 10 ml de milieu de hMDM froid, et compter les cellules par dilution au 1/20 dans du bleu Trypan.
- Ensemencer les cellules à 1 x 10 6 hMDMs par 35 mm de verre de qualité en microscopie plat fond et incuber les plaques à 37 ° C avec 5% de CO 2 pour 1 jour.
2. Production et Quantification du VIH-1 Stocks virales
NOTE: NLR4.3 HIV-1 Gag-IGFP (Green Fluorescent Protein) portant une enveloppe R5-tropique, don de M. Schindler 10 est utilisé pour infecter les macrophages et de voir les cellules infectées en temps réel.
- Produire des stocks viraux par transfection de cellules embryonnaires humaines de rein 293T (2 x 10 6 à 100 mm plat) avec 6 pg de l'ADN proviral correspondant en utilisant un réactif de transfection du commerce.
- Quantifier l'infectivité des stocks de virus en utilisant des cellules indicatrices HeLa TZM-BL (portant le gène de la ß-galactosidase sous le contrôle du VIH-1 LTR) en utilisant des dilutions en série des souches virales, suivie d'une coloration de ß-galactosidase des cellules et le comptage des cellules bleues 12.
3. L'infection de hMDMs avec le VIH-1
- Ajouter le virus à une multiplicité d'infection (MOI) de 0,02 à 0,03 dans 1 ml de milieu hMDM à hMDMs (cellules étalées à 1,13). Ajouter aux puits témoins seulement 1 ml de milieu hMDM et incuber les boîtes à 37 ° C avec 5% de CO 2 pendant 2 jours (figure 1).
- Au jour 2 se laver les cellules avec hMDM medium 3 fois et ajouter 1 ml de milieu de hMDM frais par plat. Incuber les cellules à 37 ° C avec 5% de CO2 pendant 6 jours (figure 1).
4. Opsonisation de mouton Red Blood Cells
- Pour la préparation de 7 x 10 6 globules rouges par boîte, laver les globules rouges à deux reprises dans 100 pl de solution contenant 0,1% de sérum - albumine bovine (BSA) dans du PBS 1X avec une centrifugation à 600 x g pendant 4 minutes.
- Remettre en suspension les globules rouges lavées dans 500 ul de 1 x PBS / BSA 0,1%, avec une IgG de lapin anti-globules rouges à une concentration sous-agglutinante pour 5 pi de globules rouges et on incube avec une rotation à température ambiante pendant 30 min.
REMARQUE: Pour déterminer la concentration de sous-agglutination d'anti-GRM IgG, préparer des dilutions en série d'IgG (concentration des stocks à 13,1 mg / ml) de 1/50 à 1 / 25.600 dans 20 pl dans une plaque de 96 puits. Ajouter 2 x 10 6 GRM dans 20 pl dans chaque puits et mettre la plaque dans une pièce sombre pendant plusieurs heures. La concentration de sous-agglutinant est lala dilution du puits juste avant le puits avec agglutination (IgG + GRM formation d'un réseau). - Après rotation, centrifuger les-GRM IgG-opsonisées à 600 xg pendant 4 min et laver avec 100 pi de PBS 1x / BSA 0,1% avec centrifugation à 600 g pendant 4 min.
- Remettre en suspension les globules rouges-IgG opsonisées dans un milieu préchauffé Phénol anérythre RPMI supplémenté avec 2 mM de L-glutamine et 1% de pénicilline-streptomycine (1 ml / boîte).
5. Live Cell Video Microscopy phagocytose Assay
- Utiliser un système d'imagerie confocale tel qu'un système à disque tournant muni d'une chambre de chauffage à 37 ° C avec du CO 2 en passant par une bouteille d'eau pour l' humidification.
- Allumer la chambre de chauffage avant l'expérience pour avoir la platine du microscope à 37 ° C avant le début de l'essai de phagocytose. Allumez le microscope et l'ordinateur, et charger le logiciel d'imagerie.
- Optimiser les paramètres d'imagerie telles que la vitesse de balayage, magnification, résolution, etc. pour avoir au moins une cellule par champ et à l' image d' une image chaque minute entre 60 à 120 min.
NOTE: Ici, l'échantillon est imagée d'une image toutes les minutes pendant 60 min avec 63X lentille. - Placer le plat d'imagerie sur la platine du microscope. Réglez la mise au point et l'emplacement pour trouver un seul ensemble de VIH-1 macrophage infecté dans le domaine. Utiliser les paramètres d'excitation / d'émission appropriés basés sur le système d'imagerie utilisé et de la sonde. Inclure un canal lumineux champ (BF) pour observer phagosomes (Figure 1ii). Optimiser l'apparence des différentes images de canal en ajustant le pourcentage de lumière transmise et le temps d'exposition.
NOTE: Ici, NLR4.3 HIV-1 Gag-IGFP était excité à l' aide d' un laser 491 nm avec 50 msec de temps d' exposition avec 20% de laser (Figure 1i). - Retirer le plat d'imagerie et ajouter 1 ml de suspension de SRBC à 7 x 10 6 GRM / ml à plat (figure 1).
- Centrifuger à 500 xg pour2 min à température ambiante pour synchroniser la phagocytose, enregistrer le temps à la fin de cette centrifugation et retourner le plat de la scène.
- Optimiser la mise au point et la capture GFP et images BF dans Z-piles (toute l'épaisseur de la cellule avec une étape-distance de 0,3 um - habituellement 20 avions) chaque minute pendant au moins 1 h. Enregistrez la vidéo time-lapse dans le format de fichier natif du système d'imagerie utilisé.
NOTE: Ici, les films en accéléré ont été enregistrés dans le format natif, les fichiers * .stk.
6. Analyse des time-lapse Films
- Pour le montage vidéo, cliquez sur le menu déroulant "Apps" et dans l'onglet «Revue multi dimensionnelle de données" dans le logiciel de montage vidéo.
- Pour ouvrir le fichier, cliquez sur "Fichier de base Sélectionnez" puis sur "Sélectionner le répertoire". Dans le «ensembles de données», sélectionnez l'acquisition à analyser (en format .ND) et cliquez sur "View". Les données sont représentées sous forme de tableau dans le temps en une colonne ded Z-plan de la ligne.
- Pour représenter l'infection dans un Z-projection (Figure 2, panneaux de gauche), sélectionnez 491 nm de longueur d' onde dans la zone "Longueurs d' onde" et cliquez sur l'onglet "Z de projection" avec "tous les plans".
- Pour analyser une séquence de temps, sélectionnez BF longueur d' onde dans la zone "Longueurs d' onde" et choisissez le plan optimal sur l'axe Z pour distinguer GRM externes (figure 2, flèche rouge), GRM internes (figure 2, cercle rouge) et le noyau ( Figure 2, cercle bleu).
- Pour enregistrer les montages vidéo, cliquez sur l'onglet "Sélection de [X]" puis sur "Image (s) de charge". Enfin, enregistrez les images chargées en format .tif et les ouvrir à côté sur le logiciel ImageJ.
- Utilisez le plug - in "Tracking Manuel" sur le logiciel ImageJ pour mesurer la position du noyau et les différentes phagosomes observées dans le canal BF (figure 3).
- Download le "suivi manuel" plug-in sur le site ImageJ. Sur ImageJ, ouvrez le plugin et la séquence d'images à analyser (figure 3, étape 1 et 2).
- Entrez les paramètres tels que "Intervalle de temps" qui représente quantité de temps entre les images adjacentes, et le "x / y calibration" qui représente la distance par pixel (Figure 3, étape 3).
NOTE: Ici, enregistrer la séquence d'images avec un intervalle de temps de 1 min entre chaque trame et x / y calibration de 0,205 pm parce que le zoom 63X et un appareil photo avec une taille de pixel de 6,45 x 6,45 um 2 sont utilisés. - Pour démarrer le suivi, cliquez sur "Ajouter piste" (figure 3, étape 4) et cliquez sur un centre SRBC à la première fois quand il est internalisé. L'image suivante apparaît automatiquement.
- Continuez à cliquer sur le centre SRBC dans tous les cadres d'avoir des positions différentes pendant le temps (figure 3, étape 6 dans la case rouge).
- Commencer à suivre le noyau pour avoir sa position dans tous les cadres en cliquant sur son centre. suivre ensuite les phagosomes (en cliquant sur son centre), un par un à l'époque (cadre) de leur intériorisation, différente en fonction GRM. Pour plus de commodité pour voir SRBC sur le canal BF, utilisez la fenêtre "Luminosité et contraste" pendant le suivi (figure 3, étape 5).
- Entre chaque suivi SRBC, cliquez sur "Ajouter piste" (figure 3, étape 4) d'avoir une nouvelle piste. Le numéro du suivi sera changé dans la deuxième colonne, "Track n °" de la table des résultats (figure 3, étape 6).
- Sauvegardez les données dans un tableur pour passer à l'étape suivante de l'analyse.
- Utilisez un logiciel de feuille de calcul pour calculer la distance parcourue de phagosomes contenant GRM vers le noyau et la vitesse des phagosomes pendant les 5 premières minutes après intériorisation de GRM (Figurer 4).
- Ouvrez la table de feuille de calcul et un nouveau fichier de feuille de calcul. Transfert dans ce nouveau fichier les paramètres suivants seulement, le temps, la x- et y- coordonnées du noyau et tous les GRM (figure 4A).
- Pour calculer la distance entre GRM et le noyau avec seulement leurs coordonnées, considérons le noyau et un SRBC dans un plan XY de coordonnées (figure 4B).
REMARQUE: La distance entre deux points est la longueur du chemin qui les relie. Dans le plan, la distance entre le noyau et GRM est donnée par le théorème de Pythagore.- Si c (la distance entre le noyau et le SRBC) désigne la longueur de l'hypoténuse et a et b désignent les longueurs des deux autres côtés, d' exprimer le théorème de Pythagore que l'équation de Pythagore:

REMARQUE: Dans le même temps, au repère orthonormé, la distance horizontalea est (x -x noyau SRBC) et la distance b est verticale (y -y noyau SRBC). - Ainsi, calculer la distance entre le noyau et le GRM (figure 4A, boîte violette) par:
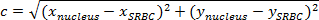
NOTE: Multiplier la valeur de distance obtenue (en pixels) par étalonnage x / y pour avoir la distance en um. Ici, le calibrage x / y est de 0,205 um. - À chaque fois, on soustrait la distance entre le noyau et le SRBC à la distance initiale (figure 4C).
- Tracer la mesure contre le temps pour les 5 premières minutes. Appliquer une ligne de tendance linéaire (Figure 5A) à la parcelle et de déterminer la pente de la ligne de tendance linéaire qui représente la vitesse de phagosome vers le noyau pendant les 5 premières minutes après intériorisation SRBC.
- Regroupez les données pour calculer l'erreur moyenne et statistique desles valeurs de vitesse et de tracer les sous une forme appropriée à l' aide du logiciel approprié (figure 5B).
- Si c (la distance entre le noyau et le SRBC) désigne la longueur de l'hypoténuse et a et b désignent les longueurs des deux autres côtés, d' exprimer le théorème de Pythagore que l'équation de Pythagore:
Résultats
Phagocytose FcR-médiée par hMDMs VIH-1-infectés et non infectés est décrite ici en utilisant GRM IgG-opsonisées comme cibles de modèle (figure 1). Les étapes essentielles de ce protocole sont la préparation de hMDMs et l'infection par le VIH-1. En effet, le rendement et la qualité des macrophages différenciés varie entre les donateurs, ainsi que le taux d'infection avec des rendements de l'ordre de 10-40%. En outre, la préparation d'IgG opson...
Discussion
Cette technique présente plusieurs étapes critiques. Tout d'abord, la préparation d'hMDMs et leur infection par le VIH-1 est critique parce que le pourcentage d'infection est dépendante donneur. Il convient de noter, nous avons décidé d'utiliser les macrophages qui ne sont pas polarisées in vitro avant l'infection, car le statut des macrophages potentiellement rencontrés par le virus in vivo n'a pas été bien caractérisé jusqu'à présent. Nous avons vérifié l&#...
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
We thank Dr Jamil Jubrail for reading the manuscript. This work was supported by grants from CNRS, Inserm, Université Paris Descartes, Agence Nationale de la Recherche (2011 BSV3 025 02), Fondation pour la Recherche Médicale (FRM DEQ20130326518 including a doctoral fellowship for GLB) and Agence Nationale de Recherche sur le SIDA et les hépatites virales (ANRS, including a post-doctoral fellowship for CD) to FN. A. Dumas was supported by doctoral fellowships from Université Paris Descartes and Sidaction.
matériels
| Name | Company | Catalog Number | Comments |
| Falcon 100mm TC-Treated Cell Culture Dish | Corning | 353003 | For viral production |
| Glass Bottom Dishes 35 mm uncoated 1.5 | MatTek corporation | P35G-1.5-14-C Case | For acquisition |
| Falcon Tissue Culture Plates 6-well | Thermo Fischer Scientific | Corning. Inc. 353934 | For human monocyte-derived macrophages |
| Ficoll-Plaque PLUS | Dominique Dutscher | 17-1440-03 | a neutral, highly branched, high-mass, hydrophilic polysaccharide in solution for density centrifugation |
| DPBS, no calcium, no magnesium | Thermo Fischer Scientific | 14190-094 | Room temperature |
| Dulbecco's Modified Eagle Medium (DMEM) 1X, liquid (High Glucose) | GIBCO, Molecular probes | 31966-021 | Conserved at 4°C ; for HEK cells culture |
| RPMI 1640 medium GLUTAMAX Supplement | Life technologies | 61870-010 | Conserved at 4°C; for hMDMs culture |
| Fœtal Calf Serum (FCS) | Eurobio | CVFSVF0001 | Conserved at -20°C ; decomplemented |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fischer Scientific | 15140-122 | Conserved at -20°C ; for hMDMs culture |
| RPMI 1640 medium, no phenol red (10x500 ml) | Life technologies | 11835-105 | Warm in 37°C water bath before use ; for phagocytosis assay |
| FuGENE6 Transfection Reagent | Promega | E2692 | Conserved at 4°C ; for viral production |
| Sheep red blood cells (SRBCs) | Eurobio | DSGMTN00-0Q | Conserved in Alsever buffer at 4°C before use |
| Anti-sheep red blood cells IgG | MP Biomedicals | 55806 | Conserved at 4°C |
| Bovine Serum Albumin heat shock fraction, pH 7, ≥98% | Sigma | A7906 | Conserved at -20°C |
| Inverted microscope DMI600 | Leica | ||
| Confocal Spinning Disk Unit CSU-X1M1 | Yokogawa | ||
| 491 nm 50mW laser | COBOLT CALYPSO | ||
| HCX PL APO CS Objectif | Leica | Objective lens ; Magnification 100x ; Numerical aperture 1.40 ; Immersion oil | |
| CoolSnap HQ2 (FireWire) Camera | Photometrics | Pixel size 6.45 x 6.45 µm ; Definition 1392 x 1040 ; Encoding the image in 14 Bit | |
| Metamorph 7.7.5 software | Molecular Devices | For the control of the microscope | |
| GraphPad Prism software | For the statistics analysis |
Références
- Flannagan, R. S., Jaumouille, V., Grinstein, S. The cell biology of phagocytosis. Annu. Rev. Pathol. 7, 61-98 (2012).
- Niedergang, F. . Encyclopedia of Cell Biology. 2, 751-757 (2016).
- Blocker, A., Griffiths, G., Olivo, J. C., Hyman, A. A., Severin, F. F. A role for microtubule dynamics in phagosome movement. J Cell Sci. 111 (Pt 3), 303-312 (1998).
- Blocker, A., et al. Molecular requirements for bi-directional movement of phagosomes along microtubules. J Cell Biol. 137, 113-129 (1997).
- Flannagan, R. S., Cosio, G., Grinstein, S. Antimicrobial mechanisms of phagocytes and bacterial evasion strategies. Nat Rev Microbiol. 7, 355-366 (2009).
- Feasey, N. A., Dougan, G., Kingsley, R. A., Heyderman, R. S., Gordon, M. A. Invasive non-typhoidal salmonella disease: an emerging and neglected tropical disease in Africa. Lancet. 379, 2489-2499 (2012).
- Mazzolini, J., et al. Inhibition of phagocytosis in HIV-1-infected macrophages relies on Nef-dependent alteration of focal delivery of recycling compartments. Blood. 115, 4226-4236 (2010).
- Dumas, A., et al. The HIV-1 protein Vpr impairs phagosome maturation by controlling microtubule-dependent trafficking. J Cell Biol. 211, 359-372 (2015).
- Greenberg, S., el Khoury, J., Kaplan, E., Silverstein, S. C. A fluorescence technique to distinguish attached from ingested erythrocytes and zymosan particles in phagocytosing macrophages. J. Immunol. Methods. 139, 115-122 (1991).
- Koppensteiner, H., Banning, C., Schneider, C., Hohenberg, H., Schindler, M. Macrophage internal HIV-1 is protected from neutralizing antibodies. J Virol. 86, 2826-2836 (2012).
- Gartner, S. The macrophage and HIV: basic concepts and methodologies. Methods Mol Biol. 1087, 207-220 (2014).
- Wei, X., et al. Emergence of resistant human immunodeficiency virus type 1 in patients receiving fusion inhibitor (T-20) monotherapy. Antimicrob Agents Chemother. 46, 1896-1905 (2002).
- Harrison, R. E., Bucci, C., Vieira, O. V., Schroer, T. A., Grinstein, S. Phagosomes fuse with late endosomes and/or lysosomes by extension of membrane protrusions along microtubules: role of Rab7 and RILP. Mol Cell Biol. 23, 6494-6506 (2003).
- Toyohara, A., Inaba, K. Transport of phagosomes in mouse peritoneal macrophages. J Cell Sci. 94 (Pt 1), 143-153 (1989).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon