Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Phagosom Migration und Geschwindigkeit gemessen in Live-primären humanen Makrophagen mit HIV-1 infizierten
In diesem Artikel
Zusammenfassung
We describe a method to measure the velocity of phagosomes moving towards the cell center in living cells infected with or without the human immunodeficiency virus (HIV) type 1, using spinning disk confocal fluorescence microscopy to identify fluorescent infected cells and bright field microscopy to detect phagosomes.
Zusammenfassung
Macrophages are phagocytic cells that play a major role at the crossroads between innate and specific immunity. They can be infected by the human immunodeficiency virus (HIV)-1 and because of their resistance to its cytopathic effects they can be considered to be persistent viral reservoirs. In addition, HIV-infected macrophages exhibit defective functions that contribute to the development of opportunistic diseases.
The exact mechanism by which HIV-1 impairs the phagocytic response of macrophages was unknown. We had previously shown that the uptake of various particulate material by macrophages was inhibited when they were infected with HIV-1. This inhibition was only partial and phagosomes did form within HIV-infected macrophages. Therefore, we focused on analyzing the fate of these phagosomes. Phagosome maturation is accompanied by migration of these compartments towards the cell center, where they fuse with lysosomes, generating phagolysosomes, responsible for degradation of the ingested material. We used IgG-opsonized Sheep Red Blood Cells as a target for phagocytosis. To measure the speed of centripetal movement of phagosomes in individual HIV-infected macrophages, we used a combination of bright field and fluorescence confocal microscopy. We established a method to calculate the distance of phagosomes towards the nucleus, and then to calculate the velocity of the phagosomes. HIV-infected cells were identified thanks to a GFP-expressing virus, but the method is applicable to non-infected cells or any type of infection or treatment.
Einleitung
Macrophages play a major role in the innate immune system and in homeostasis. They are professional phagocytes that internalize and eliminate pathogens and debris by a process called phagocytosis 1,2. The phagosome, the closed compartment that forms after the engulfment of particulate material, undergoes a series of fusion and fission events with endocytic compartments, leading to a degradative compartment called the phagolysosome. This compartment has an acidic pH, due to the acquisition of proton-pumping v-ATPases, contains hydrolytic enzymes and is enriched in lysosomal-associated membrane proteins (LAMPs). The maturation of phagosomes is accompanied by their migration on microtubules 3,4 towards the cell center to reach a perinuclear location where lysosomes are accumulated.
Many pathogens have been reported to hijack phagosome maturation, including bacteria with intracellular lifestyles that modify the vacuolar environment where they reside 5. The Human Immunodeficiency Virus (HIV)-1 targets macrophages in addition to T cells. As macrophages are resistant to the cytopathic effects of the virus, unlike T cells, they can be considered as a reservoir for the virus. In addition, macrophages infected with HIV-1 show defective phagocytic functions and contribute to the emergence of opportunistic diseases. In particular, severe invasive non-typhoidal Salmonella disease caused by Salmonella Typhimurium ST313 has been prevalent for the last three decades in sub-Saharan African children or adults infected with HIV 6. It has been estimated that the risk of developing tuberculosis is more than 20 times greater in people living with HIV than among those without HIV infection.
For all these reasons, it is important to better define the molecular mechanisms underlying the phagocytic defects in HIV-infected macrophages. We have shown that the uptake of particulate material, opsonized particles, bacteria or fungi, was inhibited in HIV-infected macrophages 7. Given that this inhibition is partial, we then set out to analyze the fate of the internalized phagosomes in HIV infected human macrophages 8. Because phagosome maturation is tightly connected with migration to the cell center and fusion with lysosomes, a defect in phagosomal maturation can be due to modifications of the trafficking modalities in the infected cell. The method described here uses IgG-opsonized Sheep Red Blood Cells (IgG-SRBCs) as a model to target receptor-mediated phagocytosis and in particular receptors for the Fc portion of immunoglobulins (FcR). These particles are easier to image in bright field (BF) than latex beads because extracellular and intracellular SRBCs show different refraction properties 9. To measure the velocity of phagosomes moving towards the nucleus in HIV-infected macrophages, we used a fluorescent virus 10 and set up a simple manual tracking method that is described here. The method does not require advanced programming and simply uses ImageJ. It is amenable to adherent cells and any type of particle or pathogen that can be visualized with bright field microscopy or with fluorescent imaging.
Protokoll
Das Protokoll muss in strikter Übereinstimmung mit nationalen und internationalen Gesetzen und örtlichen Vorschriften durchgeführt. Blut von gesunden Spendern, die ihre Zustimmung Blut für Forschungszwecke zu spenden gab wurde von Bluttransfusionszentren erhalten, mit denen die Institutionen Vereinbarung unterzeichnet haben. Besondere Schutzmaßnahmen getroffen werden müssen, wenn menschliches Blut verwendet wird. Experimente mit HIV-1 muss in einem Biosicherheitsstufe 3 oder 2 (BSL-3 oder 2) Labor nach den örtlichen Vorschriften durchgeführt werden.
1. Herstellung von Human-Monozyten abgeleiteten Makrophagen (hMDMs) durch Dichtegradientenzentrifugation und Auswahl durch Adhesion
- Beginnen Sie mit frischem Blut von gesunden Spendern (9 ml). Verdünne das gesamte Volumen von frischem Blut mit sterilem 1x phosphatgepufferter Kochsalzlösung (PBS) ohne Ca 2+ und Mg 2+ ein Endvolumen von 70 ml zu erhalten , und sanft das verdünnte Blut in zwei 50 ml konische Röhrchen (35 ml pro Röhrchen) hinzuzufügen , oben auf 15 ml eineutral, stark verzweigten, hoher Masse, hydrophile Polysaccharid in Lösung bereits in jedem Röhrchen.
- Zentrifuge beide Rohre von Blut bei 537 xg für 20 min bei 20 ° C ohne Bremse. Dann sammeln Sie die peripheren mononukleären Blutzellen (PBMCs) in den bewölkten Zelle Ring an der Schnittstelle enthalten sind, und übertragen sie in eine neue 50 - ml - Röhrchen mit 15 ml 1x PBS ohne Ca 2+ und Mg 2+.
- Zentrifuge die Zellen bei 218 · g für 5 min bei 20 ° C und das Pellet in 45 ml 1x PBS ohne Ca 2+ und Mg 2+.
- Zentrifuge die Zellen bei 218 · g für 5 min bei 20 ° C, Resuspendieren des Pellets in 10 ml 1x PBS ohne Ca 2+ und Mg 2+ und die Zellen zählen , indem zu einer Endverdünnung von 1/200 in Trypan Blau verdünnt.
- Zentrifugieren der Zellen bei 218 xg für 5 min bei 20 ° C und das Pellet in RPMI (Roswell Park Memorial Institute) 1640-Medium, ergänzt mit 2 mM L-Glutamin und 100 ug / ml Penicillin-Streptomycin 7 x 10 6 PBMCs in 2 ml Medium in jeder Vertiefung einer 6 - Well-Platte pro Vertiefung zu haben.
- Inkubiere die Platten bei 37 ° C mit 5% CO 2 für 2 Std. Nach 2 Stunden wird die Monozyten an den Kunststoff eingehalten.
- Damit frisch isolierten Monozyten in hMDMs zu unterscheiden, aspirieren das Medium und ersetzen Sie es mit 2 ml hMDM Medium (RPMI 1640, 10% decomplemented Fetale Kälberserum (FCS), 2 mM L-Glutamin und 1% Penicillin-Streptomycin), ergänzt mit rekombinantes humanes M-CSF (RHM-CSF) in einer Endkonzentration von 10 ng / ml.
- Inkubieren der Zellen bei 37 ° C mit 5% CO 2 für 11 Tage (Abbildung 1).
- Am Tag 11 das Medium abgenommen und gewaschen werden 2 mal mit 2 ml kaltem hMDM Medium pro Vertiefung. Weiter waschen jeder Vertiefung 2 mal mit 1 ml kaltem 1x PBS.
- Zum Abnehmen ausdifferenzierten hMDMs, waschen Sie sich gut 1 mal mit 1 ml kaltem 1x PBS mit 2 mM ethylenediaminetetraacetatic Säure (EDTA) und die Zellen in 2 ml kaltem 1x PBS mit 2 mM EDTA pro Vertiefung für 15-60 min bei 4 ° C inkubieren.
HINWEIS: Alternative Methoden die Zellen existieren (zB Trypsin oder Trypsin-ähnliche Aktivitäten) zu lösen , die gute Ergebnisse 11 geben könnte. - Nach Ablösung (wird von sanft bis Pipettieren und nach unten in das Bohrloch), um die Zellen zu sammeln und sie in einem 50-ml-Röhrchen auf Eis 10 ml kaltem hMDM Medium enthält.
- Zentrifuge die Zellen bei 218 · g für 5 min bei 20 ° C, das Pellet in 10 ml kaltem hMDM Medium und die Zellen zählen, indem 1/20 in Trypan Blau verdünnt.
- Seed die Zellen bei 1 x 10 6 hMDMs pro 35 mm Mikroskopie Glasbodenschale und Inkubation der Platten bei 37 ° C mit 5% CO 2 für 1 Tag.
2. Herstellung und Quantifizierung von HIV-1-Virus Stocks
HINWEIS: NLR4.3 HIV-1 Gag-iGFP (Green Fluorescent Protein) trägt eine R5-trop Umschlag, Geschenk von M. Schindler 10 wird verwendet , Makrophagen zu infizieren und infizierte Zellen in Echtzeit zu sehen.
- Produzieren viralen Bestände durch Transfektion von humanen embryonalen Nieren 293T Zellen (2 x 10 6 in 100 - mm - Schale) mit 6 ug des entsprechenden provirale DNA Kommerzielle Transfektionsreagenz verwenden.
- Quantifizierung der Infektiosität der Virusstämme mit den Indikatorzellen HeLa TZM-bl (das ß-Galactosidase-Gen unter der Kontrolle des HIV-1 LTR trägt) unter Verwendung von seriellen Verdünnungen der viralen durch eine ß-Galactosidase Färbung der Zellen und das Zählen gefolgt Bestände der blauen Zellen 12.
3. Die Infektion von hMDMs mit HIV-1
- Füge das Virus bei einer Multiplizität der Infektion (MOI) von 0,02 bis 0,03 in 1 ml Medium zu hMDM hMDMs (plattierten Zellen in 1,13). Um zu steuern , Brunnen hinzufügen nur 1 ml hMDM Medium und Inkubation das Geschirr bei 37 ° C mit 5% CO 2 für 2 Tage (Abbildung 1).
- Am Tag 2 waschen Sie die Zellen mit hMDM medium 3 Mal und 1 ml frisches hMDM Medium pro Schale. Inkubieren der Zellen bei 37 ° C mit 5% CO 2 für 6 Tage (Abbildung 1).
4. Opsonisierung von roten Blutkörperchen vom Schaf
- Zur Herstellung von 7 x 10 6 pro Schale SRBCs, wäscht den SRBCs zweimal in 100 ul Lösung , die 0,1% Rinderserumalbumin (BSA) in 1x PBS mit Zentrifugation bei 600 xg für 4 min enthält.
- Resuspendieren der gewaschenen SRBCs in 500 ul 1x PBS / BSA 0,1% mit Kaninchen-IgG anti-SRBCs bei einer sub-agglutinierende Konzentration pro 5 ul SRBCs und Inkubation für 30 min mit Rotation bei RT.
HINWEIS: Um die Unter agglutinierend Konzentration von anti-SRBCs IgG, bereiten serielle Verdünnungen von IgG (Stammkonzentration bei 13,1 mg / ml) von 1/50 bis 1 / 25.600 in 20 & mgr; l in einer 96-Well-Platte bestimmen. In 2 x 10 6 SRBCs in 20 & mgr; l in jede Vertiefung und stellte den Teller in einem dunklen Raum während mehrerer Stunden. Der Teil agglutinierende Konzentration ist dieVerdünnung der gut, kurz bevor die gut mit Agglutination (IgG + SRBCs ein Netzwerk bilden). - Nach der Drehung Zentrifugation der IgG-opsonisierter-SRBCs bei 600 × g für 4 min und Waschen mit 100 ul 1x PBS / BSA 0,1% mit Zentrifugation bei 600 × g für 4 min.
- Resuspendieren der IgG-opsonisiertes-SRBCs in vorgewärmten Phenolrot-freiem RPMI-Medium mit 2 mM L-Glutamin und 1% Penicillin-Streptomycin (1 ml / Schale).
5. Live Cell Videomikroskopie Phagozytose Assay
- Verwenden eines konfokalen Abbildungssystems , wie beispielsweise einem Spinnplattensystem mit einer Heizkammer ausgerüstet bei 37 ° C mit CO 2 mit Wasser für die Befeuchtung durch eine Flasche vorbei.
- Schalten Sie die Heizkammer vor dem Experiment das Mikroskoptisch zu haben, bei 37 ° C vor dem Beginn des Phagocytose-Assay. Schalten Sie das Mikroskop und Computer, und laden Sie die Bildbearbeitungssoftware.
- Optimieren Sie die Imaging-Einstellungen wie Scangeschwindigkeit, magnificatIon, Auflösung usw. mindestens eine Zelle pro Feld zu haben und Bild eine jede Minute zwischen 60 bis 120 min - Rahmen.
HINWEIS: Hier wird die Probe ein Rahmen jede Minute während 60 min mit 63X Objektiv abgebildet wird. - Setzen Sie die Druckschale auf dem Mikroskoptisch. Stellen Sie den Fokus und die Lage zu finden, nur eine ganze HIV-1-infizierten Makrophagen in das Feld ein. Verwenden Sie geeignete Anregungs- / Emissions-Einstellungen auf der Basis des verwendeten Abbildungssystems und der Sonde. Fügen Sie ein helles Feld (BF) Kanal phagosomes (Abbildung 1II) zu beobachten. Optimieren das Aussehen der verschiedenen Kanalbilder, indem der Prozentsatz von durchgelassenem Licht und die Belichtungszeit eingestellt wird.
HINWEIS: Hier NLR4.3 HIV-1 Gag-iGFP war begeistert , eine 491 - nm - Laser mit 50 ms Zeitbelichtung mit 20% der Laser (Abbildung 1 i) verwendet wird . - Nehmen Sie die Bildschale und 1 ml SRBC - Suspension bei 7 x 10 6 SRBCs / ml in die Schale (Abbildung 1).
- Zentrifuge bei 500 × g für2 min bei RT Phagozytose, notieren Sie die Zeit am Ende dieser Zentrifugation und Rück die Schale auf die Bühne zu synchronisieren.
- Optimierung des Fokus und capture GFP und BF Bilder in Z-Stapel (über die Dicke der Zelle mit einem Schritt-Abstand von 0,3 um - in der Regel 20-Ebenen) mindestens 1 Stunde jede Minute für. Speichern Sie die Zeitraffer-Video im nativen Dateiformat des verwendeten Abbildungssystems.
HINWEIS: Hier Zeitraffer-Filme im nativen Format gespeichert wurden, * .STK Dateien.
6. Die Analyse der Zeitraffer-Filme
- Für Videobearbeitung, klicken Sie auf das Dropdown-Menü "Apps" und auf der Registerkarte "Bewertungs Multi Dimensional Data" in der Video-Editing-Software.
- Um die Datei zu öffnen, klicken Sie auf "Select Base-Datei" und dann auf "Select Directory". In der "Datensätze" Feld, wählen Sie den Erwerb (in .ND-Format) zu analysieren, und klicken Sie auf "Ansicht". Die Daten werden in einer Tabelle mit der Zeit in der Spalte ein repräsentiertd Z-Plan in der Leitung.
- Um die Infektion in einer Z-Projektion darstellen (Abbildung 2, links Platten), wählen Sie 491 nm Wellenlänge im "Wavelengths" und klicken Sie auf den "Z Projektion" Tab mit "allen Ebenen".
- Um eine zeitliche Abfolge analysieren, BF Wellenlänge im "Wavelengths" ein und wählen Sie die optimale Ebene , auf der Z-Achse wählen zu externen SRBCs unterscheiden (Abbildung 2, roten Pfeilspitze), interne SRBCs (Abbildung 2, roter Kreis) und den Kern ( Abbildung 2, blauer Kreis).
- Um die Videomontagen speichern, klicken Sie auf "Auswahl [X]" und dann auf "Load Image (s)". Schließlich die geladenen Bilder in .tif-Format speichern und Software als nächstes auf ImageJ öffnen.
- Verwenden Sie das Plugin "Manuelle Spur" auf der ImageJ Software die Position des Kerns und die verschiedenen phagosomes in BF Kanal (Abbildung 3) beobachtet zu messen.
- Download das "Manual-Tracking" Plugin auf der ImageJ Website. Auf ImageJ, öffnen Sie das Plugin und die Bildfolge analysiert werden (Abbildung 3, Schritt 1 und 2).
- Geben Sie die Einstellungen wie "Time Interval" , die zwischen benachbarten Rahmen Höhe der Zeit darstellt und die "x / y - Kalibrierung" , die den Abstand pro Pixel darstellt (Abbildung 3, Schritt 3).
HINWEIS: Hier speichern Sie die Bildfolge mit einem Zeitintervall von 1 Minute zwischen jedem Rahmen und x / y Kalibrierung von 0,205 & mgr; m , da die 63X - Zoom und eine Kamera mit Pixelgröße von 6,45 x 6,45 & mgr; m 2 verwendet werden. - Um die Verfolgung zu starten, klicken Sie auf "Add Track" (Abbildung 3, Schritt 4) und klicken Sie auf ein SRBC Zentrum beim ersten Mal , wenn er internalisiert wird. Das nächste Bild erscheint automatisch.
- Weiter auf der SRBC - Center in allen Frames klicken verschiedenen Positionen während der Zeit zu haben (Abbildung 3, Schritt 6 im roten Kasten).
- Starten Sie den Kern zu verfolgen seine Position durch einen Klick auf das Zentrum in allen Frames zu haben. Spur folgen die phagosomes (durch seine Mitte zu klicken) nacheinander zu der Zeit (Rahmen) ihrer Verinnerlichung, unterscheidet sich in Abhängigkeit von SRBCs. Der Einfachheit halber SRBC auf BF - Kanal zu sehen, verwenden Sie die "Helligkeit und Kontrast" Fenster während der Tracking (Abbildung 3, Schritt 5).
- Zwischen jedem SRBC - Tracking, klicken Sie auf "Add Track" (Abbildung 3, Schritt 4) eine neue Spur zu haben. Die Anzahl der Tracking wird in der zweiten Spalte "Track n °" der Ergebnistabelle (Abbildung 3, Schritt 6) geändert werden.
- Speichern der Daten in einer Tabelle mit dem nächsten Schritt der Analyse fortzusetzen.
- Verwenden , Tabellenkalkulationssoftware , die zurückgelegte Strecke von Phagosomen enthält SRBCs Richtung Kern und der Geschwindigkeit der Phagosomen während der ersten 5 min nach der Internalisierung von SRBCs zu berechnen (FiAbbildung 4).
- Öffnen Sie die Tabelle Tabelle und eine neue Tabellenkalkulationsdatei. Transfer in diese neue Datei nur die folgenden Parameter, die Zeit, die x- und y- Koordinate des Kerns und alle SRBCs (4A).
- Um den Abstand zwischen SRBCs berechnen und den Kern nur mit ihren Koordinaten betrachten den Kern und einen SRBC in einem XY-Koordinatenebene (4B).
HINWEIS: Der Abstand zwischen zwei Punkten ist die Länge der Strecke, die sie verbinden. In der Ebene wird der Abstand zwischen SRBC und dem Kern durch den Satz des Pythagoras gegeben.- Wenn c (der Abstand zwischen dem Kern und dem SRBC) , um die Länge der Hypotenuse und A und b die Längen der beiden anderen Seiten bezeichnen exprimieren Pythagoras 'Theorem als pythagoreischen Gleichung:

HINWEIS: In der gleichen Zeit auf der orthonormal, der horizontale Abstanda (x - x Kern SRBC) und der vertikale Abstand b (y -y Kern SRBC). - So berechnen Sie den Abstand zwischen dem Kern und dem SRBC (4A, lila Box) von:
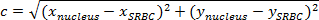
HINWEIS: Multiplizieren der Wert, der Abstand (in Pixeln) von x / y-Kalibrierung den Abstand in & mgr; m zu haben. Hier wird die x / y-Kalibrierung ist 0,205 & mgr; m. - Zu jedem Zeitpunkt, zu subtrahieren , den Abstand zwischen dem Kern und dem SRBC auf den Anfangsabstand (4C).
- Zeichnen Sie die Messung gegen die Zeit für den ersten 5 min. Tragen Sie eine lineare Trendlinie (5A) auf dem Grundstück und bestimmen die Steigung der linearen Trend-Linie, die die phagosome Geschwindigkeit in Richtung des Kerns während der ersten 5 Minuten nach der SRBC Internalisierung darstellt.
- Collate die Daten den Durchschnitt und die statistischen Fehler von zu berechnendie Geschwindigkeitswerte und sie in geeigneter Form zeichnen die entsprechende Software (5B) verwendet wird .
- Wenn c (der Abstand zwischen dem Kern und dem SRBC) , um die Länge der Hypotenuse und A und b die Längen der beiden anderen Seiten bezeichnen exprimieren Pythagoras 'Theorem als pythagoreischen Gleichung:
Ergebnisse
FcR-vermittelte Phagozytose durch HIV-1-infizierten und nicht-infizierten hMDMs wird hier beschrieben unter Verwendung von IgG-opsonisiertes SRBCs als Modellzielen (Abbildung 1). Die kritischen Schritte dieses Protokolls sind die Herstellung von hMDMs und die Infektion mit HIV-1. Tatsächlich variiert die Ausbeute und die Qualität der differenzierten Makrophagen unter den Gebern sowie die Infektionsrate mit einem Wirkungsgrad im Bereich von 10-40%. Darüber hinaus ist d...
Diskussion
Diese Technik hat mehrere wichtige Schritte. Zum einen ist die Herstellung von hMDMs und ihre Infektion durch HIV-1 wichtig, da der Prozentsatz der Infektion Spender abhängt. Bemerkenswert ist , haben wir beschlossen , Makrophagen zu verwenden , die in vitro vor der Infektion nicht polarisiert sind, weil der Status der Makrophagen durch das Virus in vivo angetroffen möglicherweise nicht gut , so weit charakterisiert. Wir überprüften die Expression verschiedener Oberflächenmarker, was darauf hinwei...
Offenlegungen
The authors have nothing to disclose.
Danksagungen
We thank Dr Jamil Jubrail for reading the manuscript. This work was supported by grants from CNRS, Inserm, Université Paris Descartes, Agence Nationale de la Recherche (2011 BSV3 025 02), Fondation pour la Recherche Médicale (FRM DEQ20130326518 including a doctoral fellowship for GLB) and Agence Nationale de Recherche sur le SIDA et les hépatites virales (ANRS, including a post-doctoral fellowship for CD) to FN. A. Dumas was supported by doctoral fellowships from Université Paris Descartes and Sidaction.
Materialien
| Name | Company | Catalog Number | Comments |
| Falcon 100mm TC-Treated Cell Culture Dish | Corning | 353003 | For viral production |
| Glass Bottom Dishes 35 mm uncoated 1.5 | MatTek corporation | P35G-1.5-14-C Case | For acquisition |
| Falcon Tissue Culture Plates 6-well | Thermo Fischer Scientific | Corning. Inc. 353934 | For human monocyte-derived macrophages |
| Ficoll-Plaque PLUS | Dominique Dutscher | 17-1440-03 | a neutral, highly branched, high-mass, hydrophilic polysaccharide in solution for density centrifugation |
| DPBS, no calcium, no magnesium | Thermo Fischer Scientific | 14190-094 | Room temperature |
| Dulbecco's Modified Eagle Medium (DMEM) 1X, liquid (High Glucose) | GIBCO, Molecular probes | 31966-021 | Conserved at 4°C ; for HEK cells culture |
| RPMI 1640 medium GLUTAMAX Supplement | Life technologies | 61870-010 | Conserved at 4°C; for hMDMs culture |
| Fœtal Calf Serum (FCS) | Eurobio | CVFSVF0001 | Conserved at -20°C ; decomplemented |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fischer Scientific | 15140-122 | Conserved at -20°C ; for hMDMs culture |
| RPMI 1640 medium, no phenol red (10x500 ml) | Life technologies | 11835-105 | Warm in 37°C water bath before use ; for phagocytosis assay |
| FuGENE6 Transfection Reagent | Promega | E2692 | Conserved at 4°C ; for viral production |
| Sheep red blood cells (SRBCs) | Eurobio | DSGMTN00-0Q | Conserved in Alsever buffer at 4°C before use |
| Anti-sheep red blood cells IgG | MP Biomedicals | 55806 | Conserved at 4°C |
| Bovine Serum Albumin heat shock fraction, pH 7, ≥98% | Sigma | A7906 | Conserved at -20°C |
| Inverted microscope DMI600 | Leica | ||
| Confocal Spinning Disk Unit CSU-X1M1 | Yokogawa | ||
| 491 nm 50mW laser | COBOLT CALYPSO | ||
| HCX PL APO CS Objectif | Leica | Objective lens ; Magnification 100x ; Numerical aperture 1.40 ; Immersion oil | |
| CoolSnap HQ2 (FireWire) Camera | Photometrics | Pixel size 6.45 x 6.45 µm ; Definition 1392 x 1040 ; Encoding the image in 14 Bit | |
| Metamorph 7.7.5 software | Molecular Devices | For the control of the microscope | |
| GraphPad Prism software | For the statistics analysis |
Referenzen
- Flannagan, R. S., Jaumouille, V., Grinstein, S. The cell biology of phagocytosis. Annu. Rev. Pathol. 7, 61-98 (2012).
- Niedergang, F. . Encyclopedia of Cell Biology. 2, 751-757 (2016).
- Blocker, A., Griffiths, G., Olivo, J. C., Hyman, A. A., Severin, F. F. A role for microtubule dynamics in phagosome movement. J Cell Sci. 111 (Pt 3), 303-312 (1998).
- Blocker, A., et al. Molecular requirements for bi-directional movement of phagosomes along microtubules. J Cell Biol. 137, 113-129 (1997).
- Flannagan, R. S., Cosio, G., Grinstein, S. Antimicrobial mechanisms of phagocytes and bacterial evasion strategies. Nat Rev Microbiol. 7, 355-366 (2009).
- Feasey, N. A., Dougan, G., Kingsley, R. A., Heyderman, R. S., Gordon, M. A. Invasive non-typhoidal salmonella disease: an emerging and neglected tropical disease in Africa. Lancet. 379, 2489-2499 (2012).
- Mazzolini, J., et al. Inhibition of phagocytosis in HIV-1-infected macrophages relies on Nef-dependent alteration of focal delivery of recycling compartments. Blood. 115, 4226-4236 (2010).
- Dumas, A., et al. The HIV-1 protein Vpr impairs phagosome maturation by controlling microtubule-dependent trafficking. J Cell Biol. 211, 359-372 (2015).
- Greenberg, S., el Khoury, J., Kaplan, E., Silverstein, S. C. A fluorescence technique to distinguish attached from ingested erythrocytes and zymosan particles in phagocytosing macrophages. J. Immunol. Methods. 139, 115-122 (1991).
- Koppensteiner, H., Banning, C., Schneider, C., Hohenberg, H., Schindler, M. Macrophage internal HIV-1 is protected from neutralizing antibodies. J Virol. 86, 2826-2836 (2012).
- Gartner, S. The macrophage and HIV: basic concepts and methodologies. Methods Mol Biol. 1087, 207-220 (2014).
- Wei, X., et al. Emergence of resistant human immunodeficiency virus type 1 in patients receiving fusion inhibitor (T-20) monotherapy. Antimicrob Agents Chemother. 46, 1896-1905 (2002).
- Harrison, R. E., Bucci, C., Vieira, O. V., Schroer, T. A., Grinstein, S. Phagosomes fuse with late endosomes and/or lysosomes by extension of membrane protrusions along microtubules: role of Rab7 and RILP. Mol Cell Biol. 23, 6494-6506 (2003).
- Toyohara, A., Inaba, K. Transport of phagosomes in mouse peritoneal macrophages. J Cell Sci. 94 (Pt 1), 143-153 (1989).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten