Method Article
Protocolo para la diferenciación de células madre pluripotentes inducidas por humanos en cultivos mixtos de neuronas y glia para pruebas de neurotoxicidad
En este artículo
Resumen
Las células madre pluripotentes inducidas por el hombre (HiPSCs) se consideran una herramienta poderosa para la detección de drogas y químicos y para el desarrollo de nuevos modelos in vitro para pruebas de toxicidad, incluyendo neurotoxicidad. Aquí, se describe un protocolo detallado para la diferenciación de hiPSCs en neuronas y glia.
Resumen
Las células madre pluripotentes humanas pueden diferenciarse en varios tipos de células que pueden aplicarse a ensayos de toxicidad in vitro basados en humanos. Una ventaja importante es que la reprogramación de células somáticas para producir células madre pluripotentes inducidas por humanos (hiPSCs) evita las cuestiones éticas y legislativas relacionadas con el uso de células madre embrionarias humanas (hESCs). Los HiPSCs pueden ampliarse y diferenciarse eficazmente en diferentes tipos de células neuronales y gliales, sirviendo como sistemas de prueba para pruebas de toxicidad y, en particular, para la evaluación de diferentes vías implicadas en la neurotoxicidad. Este trabajo describe un protocolo para la diferenciación de hiPSCs en cultivos mixtos de células neuronales y gliales. Se definen las vías de señalización que son reguladas y / o activadas por diferenciación neuronal. Esta información es fundamental para la aplicación del modelo celular al nuevo paradigma de pruebas de toxicidad, en el que se evalúan los productos químicos en base a su capacidad paraRturb vías biológicas. Como prueba de concepto, la rotenona, un inhibidor del complejo respiratorio mitocondrial I, se utilizó para evaluar la activación de la vía de señalización Nrf2, un regulador clave del mecanismo de defensa celular impulsado por el elemento antioxidante-respuesta (ARE) contra el estrés oxidativo .
Introducción
El informe 1 del Consejo Nacional de Investigación de EE. UU. Preveía un nuevo paradigma de pruebas de toxicidad en el que las pruebas de toxicidad reglamentarias cambiarían de un enfoque basado en cambios fenotípicos observados en animales a un enfoque centrado en ensayos in vitro mecánicos utilizando células humanas. Los derivados de células madre pluripotenciales (PSC) pueden representar alternativas a los modelos de células cancerosas, ya que las células obtenidas pueden parecerse más a las condiciones fisiológicas de los tejidos humanos y proporcionar herramientas más relevantes para estudiar los efectos adversos inducidos por productos químicos. Los dos tipos principales de cultivos PSC que son más prometedores para las pruebas de toxicidad son las células madre embrionarias humanas (hESCs) y las células madre pluripotentes inducidas por humanos (hiPSCs), que actualmente se utilizan ampliamente en las áreas de investigación básica y medicina regenerativa 2 , 3 . Esta experiencia puede ahora ser aprovechada para el desarrollo de una nueva clase de toxicoloPruebas in vitro in vitro dirigidas a identificar las vías fisiológicas perturbadas implicadas en el desarrollo de efectos adversos in vivo . Sin embargo, es improbable que todos los Estados miembros de la UE y los países de todo el mundo apliquen métodos de prueba para las evaluaciones de seguridad reglamentarias basadas en CCEL debido a posibles preocupaciones éticas ya diversas políticas legislativas nacionales que regulan el uso de células derivadas de embriones.
HiPSCs comparten características similares a las hESCs 4 , 5 y poseen un gran potencial para los métodos in vitro , tanto para la identificación de objetivos terapéuticos, así como para las evaluaciones de seguridad. Además, la tecnología hiPSC mitiga las limitaciones de un grupo limitado de donantes y las preocupaciones éticas asociadas con las células derivadas de embriones. Un desafío importante para los hiPSCs es la demostración de que estas células pueden generar de forma reproducible una gama significativa de derivados de células toxicológicamente relevantes,Con características y respuestas típicas de tejidos humanos. Los niveles predefinidos de los marcadores seleccionados se utilizan generalmente para caracterizar las poblaciones de células después del proceso de diferenciación y para proporcionar conocimientos sobre la estabilidad del proceso de diferenciación.
Los trabajos previos evaluaron la idoneidad de los hiPSC para generar cultivos mixtos de células neuronales y gliales y evaluar los efectos de la rotenona, un inhibidor del complejo respiratorio mitocondrial I, sobre la activación de la vía Nrf2, un regulador clave de los mecanismos de defensa antioxidante en Muchos tipos de células 6 , 7 .
Este trabajo describe un protocolo utilizado para la diferenciación de hiPSCs en cultivos mixtos neuronales y gliales, proporcionando detalles sobre las vías de señalización (nivel de genes y proteínas) que se activan tras la diferenciación neuronal / glial. Además, el trabajo muestra resultados representativos que demuestranHiPSC derivados neuronal y glial modelo de la célula se puede utilizar para evaluar Nrf2 señalización activación inducida por aguda (24 h) de tratamiento con rotenona, lo que permite la evaluación de la inducción del estrés oxidativo.
IMR90 fibroblastos fueron reprogramados en hiPSCs en I-Stem (Francia) por la transducción viral de 2 factores de transcripción (Oct4 y Sox2) utilizando pMIG vectores [ 6] . También se pueden aplicar modelos análogos de hiPSC. Los protocolos que se describen a continuación resumen todas las etapas de diferenciación de los hiPSCs en células madre neuronales (NSCs) y aún más en cultivos mixtos de neuronas postmitoticas y células gliales (pasos 1 y 2). El protocolo) 8 .
En los pasos 3 y 4 se describe un protocolo adicional para el aislamiento, expansión, crioconservación y una mayor diferenciación de NSC en neuronas mixtas y células gliales (también se refieren al EURL ECVAM DBALM weB sitio para una descripción detallada de este protocolo) 9 . El paso 5 describe los análisis que se pueden hacer para evaluar la identidad fenotípica de las células durante las diversas etapas de compromiso y diferenciación.
Protocolo
1. Expansión de células madre pluripotentes inducidas por humanos (hiPSC)
NOTA: Los hiPSCs pueden cultivarse en un sustrato de mezcla de proteínas adecuado en presencia de medio mTeSR1 que contiene suplementos de mTeSR1 5x (preparados siguiendo las instrucciones del fabricante, placa ~ 100 fragmentos de colonia / placa de Petri de 60 mm). Cuando las colonias de hiPSC alcanzan un tamaño apropiado (véase un ejemplo de una colonia en la Figura 2A ), pasan las células como se describe a continuación (una vez a la semana).
- Los platos de recubrimiento con una matriz de membrana basal calificada con hESC (en lo sucesivo denominada "matriz calificada") o cualquier otro sustrato proteico adecuado.
- Almacene la matriz calificada (véase la tabla de materiales ) a -80 ° C en alícuotas de 200 μl en tubos fríos de 1,5 ml y pipetas frías de 5 ó 10 ml.
- Antes de pasar, descongelar 200 μl de matriz calificada en hielo.
- Diluir 200 μl de matriz calificada en 20 mL de medio DMEM / F12 (dilución 1: 100).
- Recubrir placas de Petri de 60 mm con esta solución (5 ml / placa).
- Incubar platos recubiertos a 37 ° C durante al menos 1 h.
- Criopreservación y descongelación del fragmento de colonia HiPSC
- Después de cortar las colonias de hiPSC (ver paso 1.3 para el procedimiento de corte de colonia hiPSC), resuspender suavemente y lentamente los fragmentos de colina hiPSC en medio de congelación de células madre, sim 100 fragmentos / 250 mu l (véase la Tabla de Materiales).
- Alícuota de los fragmentos de colonias en viales adecuados para crioconservación (250 μl / vial).
- Coloque los viales en un recipiente lleno de 2-propanol y coloque el recipiente a -80 ° C durante un mínimo de 2 hy hasta 2 semanas.
- Transferir los viales a la fase de vapor de un tanque de nitrógeno líquido.
- Para reiniciar el cultivo, descongelar 1 vial congelado en baño de agua a 37 ° C.
- Recoger suavemente los fragmentos de colonia hiPSC en 7 mL de hiPSC completo precalentadoMedio (véase la Tabla de Materiales ) en un tubo de 15 ml usando una pipeta de 1, 2 o 5 ml.
- Centrifugar los fragmentos de colonia hiPSC a 130 xg durante 3 min.
- Se retira el sobrenadante y se resuspenden suavemente los fragmentos de colonia hiPSC en 1 ml de medio hiPSC completo usando una pipeta de 1, 2 o 5 ml.
- Se diluye adicionalmente la suspensión celular en 3 o 4 ml de medio hiPSC completo.
- Placa los fragmentos de colonia hiPSC en una placa de Petri de 60 mm de matrices calificada (~ 100 fragmentos / plato; cubrir los platos como se describe en el paso 1.1).
- Incubar los hiPSCs a 37 ° C y 5% de CO 2 .
- Realice un cambio medio total cada día.
- El paso de las colonias de hiPSC
NOTA: Los hiPSC indiferenciados deben ser de forma redonda, con nucleolos grandes y sin citoplasma abundante. Las colonias indiferenciadas deben caracterizarse por una morfología plana y bien compacta. Sólo las colonias indiferenciadas (alrededor de 1 mm iN de diámetro) se deben cortar para el paso adicional.- Cortar las colonias de células madre en cuadrados de aproximadamente 200 μm x 200 μm usando una jeringa de 1 ml con una aguja de 30 G o cualquier otra herramienta disponible comercialmente (vea la Tabla de Materiales). Utilice un microscopio estereoscópico con un aumento de 4 veces en un gabinete de flujo laminar a temperatura ambiente.
- Separar los fragmentos de la colonia de la superficie del plato usando una pipeta de 200 μl pipeteando suavemente el medio debajo para levantar las piezas.
- Transferir los fragmentos de la colonia (~ 100 piezas) a una placa calificada con DMEM / F12 revestida con 4 mL de medio hiPSC completo (véase la Tabla de Materiales, cubrir los platos como se describe en el paso 1.1).
- Incubar la (s) nueva (s) placa (s) a 37 ° C y 5% de CO 2 .
- Realice un cambio medio total cada día e inspeccione la morfología de las colonias usando un microscopio de contraste de fase en aumentos 4X y 10X.
2. HiPSC DiffereEn Neuronas Mixtas y Glía
NOTA: El procedimiento tarda aproximadamente 28 días en completarse, con los pasos principales descritos en la Figura 1 (parte superior).
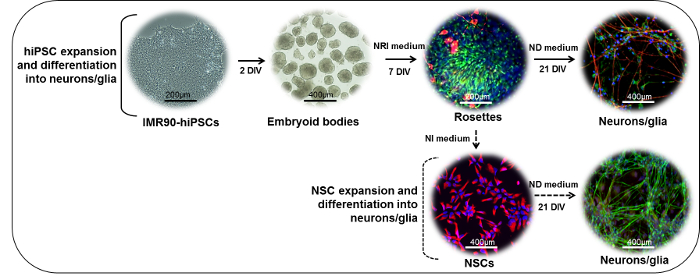
Figura 1: Representación Esquemática del Protocolo de Diferenciación Neuronal. (Parte superior) Las colonias IMR90-hiPSC pueden cortarse en fragmentos para formar cuerpos embrioides (EBs). Después de 2 días in vitro (DIV), los EB pueden ser colocados en platos revestidos con laminina o matrices convencionales y cultivados en presencia de un medio de inducción neuroepitelial (NRI) para generar derivados neuroectodérmicos (rosetas, aquí teñidas para nestina (verde) y β -III-tubulina (rojo)). Los rosetones se pueden disociar, recolectar, replantar en platos revestidos con laminina o matrices estándar, y diferenciarse adicionalmente en neuronas maduras (NF200, rojo) y glial(GFAP, verde) en presencia de un medio de diferenciación neuronal (ND). Se pueden expandir en presencia de medio de inducción neural (NI), crioconservado, o diferenciarse adicionalmente en presencia de medio ND para formar neuronas mixtas (NF200, verde) y glial ( GFAP, rojo) culturas. Haga clic aquí para ver una versión más grande de esta figura.
- Generación de cuerpos embrioides (EBs) (Días 0 → 1)
NOTA: Este procedimiento requiere buenas habilidades manuales y precisión. Los fragmentos de colonia HiPSC deben ser de igual tamaño para obtener cuerpos embrionarios homogéneos (EBs) en los próximos pasos. Las colonias morfológicamente diferenciadas (con grandes fracciones citoplasmáticas y nucleolos pequeños) deben ser descartadas.- Refrescar el medio hiPSC (placa de Petri de 3 ml / 60 mm) antes de cortar las colonias de hiPSC indiferenciadas (aproximadamente 1 mm en diamEter, véase la Figura 2A ) en condiciones estériles (como se describe en el paso 1).
- Cortar las colonias no diferenciadas (como se muestra en la Figura 2A y la Figura 2B ) en fragmentos de aproximadamente 200 μm x 200 μm usando una jeringuilla de 1 ml con una aguja 30G. Utilice un microscopio estereoscópico con una ampliación de 4X en un gabinete de flujo laminar a temperatura ambiente.
- Separar los fragmentos de la colonia de la superficie del plato usando una pipeta de 200 μl por pipeteado suavemente debajo del medio para levantar las piezas.
- Transferir todos los fragmentos desprendidos y el medio a un tubo de 15 ml usando una pipeta de 1, 2 o 5 ml.
- Enjuague el plato con 2 mL de medio hiPSC completo para recuperar todos los fragmentos.
- Centrifugar a 112 xg durante 1 min.
- Aspirar el sobrenadante y resuspender suavemente los fragmentos en 5 ml de medio completo de EB hiPSC (véase la Tabla de Materiales ).
- Plato el coLony en una placa de Petri de 60 mm de capacidad de inserción ultrabaja (placa Petri de 5 ml / 60 mm).
- Incubar la cápsula de Petri durante la noche a 37 ° C y 5% de CO 2 .
- Al día siguiente (día 1), recoja el EB y su medio en un tubo de 15 ml usando una pipeta de 1, 2 o 5 ml.
- Centrifugar los EBs a 112 xg durante 1 min.
- Aspirar cuidadosamente el sobrenadante y resuspender suavemente los EBs en 5 ml de medio completo de EB hiPSC usando una pipeta de 1, 2 o 5 ml.
- Vuelva a colocar los EB en una nueva placa de Petri de 60 mm de anclaje ultrabajo (placa de Petri de 5 ml / 60 mm).
- Incubar la cápsula de Petri durante la noche a 37 ° C y 5% de CO 2 .
- El día 1, se cubren los platos con una matriz de membrana basal ( por ejemplo, matrigel, denominada en adelante "matriz estándar") o cualquier otro sustrato de proteína adecuado ( por ejemplo , laminina).
- Almacene la matriz estándar (véase la tabla de materiales ) a -80 ° C enAlícuotas de 200 μL usando tubos fríos de 1,5 ml y pipetas frías de 5 ó 10 ml.
- Descongele 200 μl de matriz estándar sobre hielo.
- Diluir 200 μl de matriz estándar en 20 ml de medio DMEM / F12 (dilución 1: 100).
- Recubrir placas de Petri de 60 mm con esta solución (5 ml / placa).
- Incubar las placas recubiertas a 37 ° C durante la noche.
NOTA: Estos platos se utilizarán para planchar los EB (aproximadamente 50 EBs / plato) y generar agregados neuroepiteliales (rosetas); Ver paso 2.3.
- Generación de agregados neuroepiteliales (rosetas) (Días 2 → 7)
- El día 2, retire la solución de revestimiento de matriz estándar de las placas de Petri de 60 mm (no es necesario enjuagar las placas) y llénelas con 5 ml / plato de medio de inducción neuroepitelial completo (NRI); Ver la Tabla de Materiales .
- Transferir los EBs flotantes (desde el paso 2.1.14) a platos recubiertos (~ 50 EBs / plato) usando una pipeta de 200 μl bajo un filtro estereoscópicoCroscope a 4x de aumento y se coloca en un gabinete de flujo laminar.
NOTA: Es fundamental seleccionar EBs de tamaño medio y homogéneo (~ 200-300 μm de diámetro). EBs demasiado pequeño puede no sobrevivir bien durante la diferenciación neuroectodérmica, mientras que demasiado grande EBs tienden a sufrir núcleo necrosis. - Incubar las vajillas a 37 ° C y 5% de CO 2 .
- Al día siguiente (Día 3), revise los platos bajo el microscopio con un aumento de 10x para asegurarse de que todos los EB estén conectados.
- Efectúe suavemente un cambio medio total con medio NRI completo.
- Cambiar el medio NRI todos los días hasta el día 7, cuando los agregados neuroepiteliales (rosetas) deben ser visibles.
- En el día 7, se cubre la matriz estándar (o laminina), como se describe en el paso 2.2, en cualquier placa o formato de plato requerido: placas de 96 pocillos (100 μl / pocillo), placas de 24 pocillos (250 μl / pocillo) (500 μL / pocillo), chips MEA (para actividad eléctrica, 1 ml / chip de un solo pocillo), o 60 mm Petri disheS (4 ml / placa).
- Incubar las placas recubiertas durante al menos 2 horas a 37 ° C y 5% de CO 2 .
- La disociación de rosetas y la diferenciación neuronal (Días 8 → 28)
NOTA: Este procedimiento requiere buenas habilidades manuales y precisión. Para evitar la recolección de células mesodérmicas y endodérmicas, sólo se deben disociar y recolectar estructuras parecidas a rosetas de ectodermo.- El día 8, cortar las estructuras tipo roseta en fragmentos bajo un microscopio estereoscópico en una ampliación 10X en condiciones estériles. Utilice una jeringa de 1 ml con una aguja de 30 grados. Obsérvese que las rosetas tienden a separarse fácilmente del plato cuando se tocan con la aguja.
- Completar el desprendimiento de los fragmentos de roseta con una pipeta de 200 μl.
- Transferir el plato bajo la campana de flujo laminar y recoger los fragmentos de roseta y su medio en un tubo cónico de 15 mL usando una pipeta de 1, 2 o 5 mL. Enjuague el plato con 2 mL de medio NRI para recuperarTodos los fragmentos.
- Girar los fragmentos de roseta a 112 xg durante 2 min.
- Aspirar el sobrenadante.
- Resuspender suavemente el gránulo en 1 mL de 1x DPBS (sin calcio y magnesio) y pipetear suavemente los fragmentos de roseta hacia arriba y hacia abajo usando una pipeta de 1.000 l para disociarlos parcialmente.
- Agregue 4 mL de medio NRI completo y cuente las células usando azul de tripano y un contador de células automatizado (vea la Tabla de Materiales )
NOTA: Diluir 20 μl de suspensión celular en 20 μl de azul Trypan. Esta etapa puede omitirse si las células no pueden ser llevadas a una suspensión de una sola célula. Si los fragmentos de roseta no parecen estar completamente disociados, se pueden resuspender fragmentos de rosetas derivados de aproximadamente 50 platos de EBs / 60 mm en 50 mL de medio NRI completo y se plaquean, como se indica en la Tabla 1 . - Aspirar la solución de revestimiento de matriz estándar (o laminina) de las placas Petri, placas y / o chips MEA (del paso 2.3.7). No dejes que se sequen.
- Plantear las células en medio NRI completo de acuerdo con el plan de estudio (aproximadamente 15.000 células / cm2, ver Tabla 1 para las indicaciones de volumen de placas).
- Incubar las placas durante la noche a 37 ° C y 5% de CO 2 .
- El día 10, realizar un cambio de medio total usando medio de diferenciación neuronal completo (ND); Ver la Tabla de Materiales.
- Refresque el medio ND completo dos veces por semana hasta el día 28.
- Caracterizar los derivados de células neuronales / glia- les, como se describe en el paso 5 (ver Tabla 2 para criterios de aceptación general).
3. Expansión y diferenciación de células madre neuronales derivadas de HiPSC (NSC) en neuronas mixtas y Glia
NOTA: Los NSC derivados de fragmentos de roseta se pueden expandir y mantener siguiendo el procedimiento descrito a continuación ( Figura 1 , parte inferior). Esto permite incrementar laE número de células para la diferenciación y las pruebas químicas.
- Se cubre una placa de Petri de 60 mm (o un matraz T-25) con 5 ml de solución de revestimiento DMEM / F12 de matriz estándar y se incuba durante al menos 2 h a 37ºC y 5% de CO 2 (como se describe en el paso 2.2).
- Hacer girar los fragmentos de roseta derivados de los pasos 1-2 (ver paso 2.4.) En un tubo cónico de 15 ml a 112 xg durante 2 min.
- Resuspender suavemente el pellet en 5 ml de medio de inducción neural (NI); Ver la Tabla de Materiales.
- Transferir las células a una placa de Petri de 60 mm de matriz estándar (o matraz T-25).
- Cultura NSCs derivado de roseta en presencia de medio NI, refrescando el medio cada dos días hasta que las células llegan a la confluencia.
- Cuando se confluyen, pasan los NSC como se describe en los siguientes pasos.
NOTA: Pasar los NSCs aproximadamente una vez a la semana; Considere el uso de platos recién recubiertos, frascos o placas, dependiendo del plan de estudio. - Eliminar el medio NI completo y gentlY enjuague los NSC con DPBS (sin calcio y magnesio).
- Añadir 1,5 ml de tripsina-EDTA al 0,05% precalentado a 37 ° C a la placa de Petri de 60 mm (o matraz T-25) que contiene las células y colocarlo en la incubadora durante 1 min.
- Golpee suavemente el plato (o frasco) para separar las células.
- Añadir 1,5 ml de inhibidor de tripsina precalentado a 37 ° C y transferir las células a un tubo de 15 ml.
- Enjuague la placa de Petri (o el matraz T-25) con un volumen igual de medio NI (1,5 ml) y recoja el volumen en el mismo tubo de 15 ml.
- Centrifugar las células a 130 xg durante 3 min.
- Elimine el sobrenadante y resuspenda suavemente las células en 1 mL de medio NI completo usando una pipeta de 1.000 μl.
- Se diluye adicionalmente la suspensión de células en 3 ó 4 ml de medio NI completo y se cuentan las células usando azul de tripano y un contador de células automatizado.
- Se colocan los NSC en la placa de Petri de 60 mm (o en el matraz T-25) desde la etapa 3.1 a una densidad de aproximadamente 50.000 células / cm2.
- Realice un cambio medio total con el medio NI completo cada dos días.
- Caracterizar las células para la presencia de neuronal / glial células derivados, como se describe en el paso 5.
NOTA: Los NSC se pueden diferenciar en cultivos mixtos de neuronas y glia en medio ND completo (como se describe en los pasos 2.4.11-2.4.13), refrescando el medio ND completo dos veces por semana durante 21 días.
4. Crioconservación y descongelación de NSC derivada de HiPSC
NOTA: Al pasar, los NSC se pueden congelar y volver a descongelar siguiendo este procedimiento.
- Centrifugar pasó NSCs (de la etapa 3.12) a 130 xg durante 3 min.
NOTA: Las celdas deben contarse en el paso 3.14. - Suavemente y lentamente resuspenda los NSCs a 3 x 10 6 / mL de medio de congelación (véase la Tabla de Materiales ).
- Alícuota de las células en viales adecuados para crioconservación (aproximadamente 0,5 ml = 1,5 x 10 6 / vial).
- Coloque los viales en un recipiente fiLled con 2-propanol y colocar el recipiente a -80 ° C durante un mínimo de 2 hy hasta 2 semanas.
- Transferir los viales a la fase de vapor de un tanque de nitrógeno líquido.
- Para reiniciar el cultivo celular, descongelar 1 vial congelado en un baño de agua a 37 ° C.
- Recoja suavemente las células en 7 ml de medio NI completo precalentado en un tubo de 15 ml usando una pipeta de 1.000 μl.
- Centrifugar las células a 130 xg durante 3 min.
- Se retira el sobrenadante y se resuspende suavemente las células en 1 ml de medio NI completo usando una pipeta de 1.000 μl.
- Diluir adicionalmente la suspensión celular en 3 o 4 ml de medio NI completo y contar las células utilizando azul de tripano y un contador de células automatizado (nota: diluir 20 μl de suspensión celular en 20 μl de azul de tripano, la viabilidad después de la descongelación debe ser ≥ 80 %).
- Placa los NSC en una placa de Petri de 60 mm recubierta (o matraz T25) a una densidad de aproximadamente 50.000 células / cm2.
5. CharaCterización de las células neuronales y glial derivadas de HiPSC
NOTA: Tras la diferenciación, los derivados neuronales y gliales pueden caracterizarse utilizando diferentes técnicas, como las descritas en las siguientes secciones.
- Análisis cuantitativo en tiempo real PCR (qPCR) 10
- Girar los fragmentos de colonia hiPSC, EBs y / o NSCs a 130 xg durante 3 min.
- Resuspender el sedimento celular en 100 μl de tampón de lisis de ARN frío proporcionado en un kit adecuado para la extracción de ARN.
- Alternativamente, recoger derivado neuronal / glial directamente de las placas por aspiración del medio y adición de tampón de lisis de ARN frío a los pocillos para recoger las células.
- Aísle el ARN siguiendo las instrucciones del fabricante.
- Reverse-transcribe 500 ng del ARN total utilizando un kit adecuado para el ARN a la retrotranscripción de cDNA.
- Ejecutar las reacciones de qPCR por duplicado utilizando mezcla maestra apropiada y cebador(Véase la tabla de materiales ).
- Registre la emisión fluorescente en tiempo real: 45 ciclos con recocido de primers a 60 ° C.
- Normalizar las cantidades relativas de ARN a GAPDH y β-actina como genes de referencia y utilizar hiPSC indiferenciados o células no tratadas para las condiciones de calibración (método ΔΔCt). Alternativamente, utilice otro método adecuado.
- Immunocytochemistry y de alto contenido de imágenes (HCI) 6 , 11
- Fijar las colonias hiPSC, NSCs y / o derivados neuronales / glial con paraformaldehído frío al 4% durante 15 min a temperatura ambiente.
- Lavar suavemente las células en 1X PBS y almacenar las placas a 4 ° C durante un máximo de 1 mes.
- Cuando esté listo para la tinción, permeabilice las células en tampón de permeabilización (1x DPBS que contiene 0,1% de triton-X-100 y BSA al 3%) durante 15 minutos a temperatura ambiente.
- Quitar la permeabilización buFfer e incubar las células en tampón de bloqueo (3% BSA / 1X DPBS) durante 15 min a temperatura ambiente para evitar la unión no específica de anticuerpos.
- Se retira el tampón de bloqueo e incuba las células durante la noche a 4 ° C en tampón de bloqueo que contiene anticuerpos primarios adecuados (véase la Tabla de Materiales ).
- Lavar las células 3 veces con 1x PBS.
- Incubar las células durante 45 min a temperatura ambiente en tampón de bloqueo que contiene anticuerpos secundarios conjugados con fluorocromo (véase la Tabla de Materiales ), counterstaining los núcleos con DAPI tinte.
- Cuantificar la intensidad de fluorescencia media y los porcentajes relativos de tipos de células utilizando una plataforma de imágenes de alto contenido adecuado, si está disponible (véase la Tabla de Materiales ).
NOTAS: Para determinar el nivel de intensidad del fondo fluorescente, incubar algunas células / pocillos con anticuerpos secundarios solos. El análisis de citometría de flujo de vivo (no fijo), undIfferentiated hiPSCs puede ser realizado para evaluar la expresión de PSC-marcadores específicos, como SSEA4 (véase la tabla de materiales ). Las colonias hiPSC indiferenciadas se pueden analizar para determinar la actividad de la fosfatasa alcalina usando kits BCIP / NBT comercialmente disponibles, siguiendo las instrucciones del fabricante (véase la Tabla de Materiales ). Además, se pueden realizar ensayos y análisis de la matriz de proteínas de fase inversa (RPPA), como se describe en la referencia 12 (para una lista de anticuerpos probados, véase la tabla de materiales ).
- Mediciones electrofisiológicas 13
- Placa los fragmentos de roseta disociada (después de 7 DIV) o NSC derivados de rosetas en matrices de multielectrodos recubiertos (MEAs, véase la Tabla de Materiales ) en medio ND completo (~ 1 x 10 5 células / chip MEA de pozo único).
- Diferenciar las células durante 3 semanas en medio ND completa, refrescanteMedia dos veces por semana.
- Al final de la diferenciación, sellar los chips MEA con una membrana semi-permeable bajo una campana de flujo laminar para mantener los cultivos estériles para las mediciones repetidas.
- Reemplace uno de los electrodos con una referencia de tierra, permitiendo grabaciones de los electrodos restantes.
- Registre la velocidad media de disparo (MFR, número de picos / min) usando un amplificador MEA con el control de temperatura integrado ajustado a 37 ° C y 5% CO 2 .
- Detectar picos de los datos brutos MEA usando el umbral de -4.7σ (σ representa la desviación estándar del ruido basal).
- Procesar los datos posteriores a la grabación con un software adecuado.
Resultados
Caracterización de los hiPSC indiferenciados
Para evaluar el fenotipo de los hiPSCs, el análisis de la morfología de colonias / células, la determinación de marcadores específicos de PSC, y el examen de la expresión génica y la actividad de fosfato alcalino debe realizarse. Los hiPSC indiferenciados deben ser redondos, con nucleolos grandes y sin citoplasma abundante. La mayoría de las colonias deben ser caracterizadas por una morfología plana y fuertemente empaquetada, indicativa de un fenotipo indiferenciado ( Figura 2A y Figura 2B ). Adicionalmente, más del 80% de las colonias deben ser positivas para la tinción con actividad de fosfatasa alcalina ( Figura 2C ).
Alrededor del 80% de las células deben ser positivas para los marcadores clásicos relacionados con la pluripotencia, como Oct4, SSEA3, SSEA4 y Tra1-60 ( figura 2D-H ), como se muestra por inmunocitoquímica y citometría de flujo, mientras que los porcentajes de las células nestina + y β-III-tubulina + deberían ser significativamente bajos (aproximadamente 8% y 3%, respectivamente, Como se muestra en la Figura 2H ). Estos resultados deben ser reproducibles sobre pasajes.
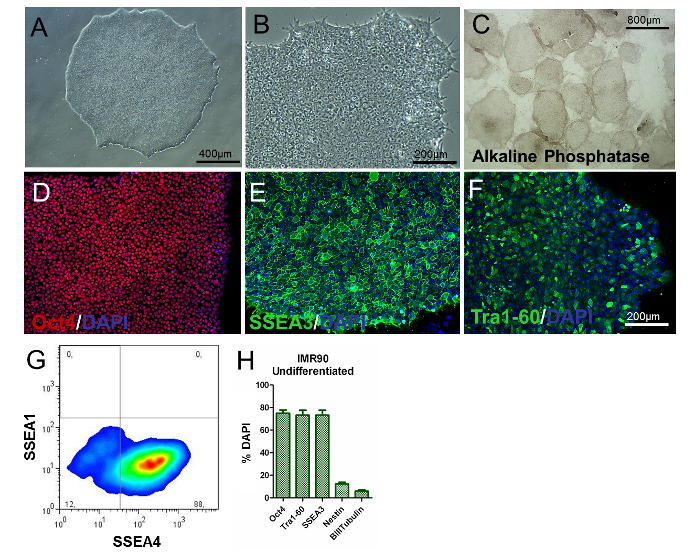
Figura 2. Caracterización de IMR90-hiPSC indiferenciadas. (A y B) Imágenes de contraste de fase representativas (aumentos de 10X y 20X) de colonias IMR90-hiPSC no diferenciadas. (C) imágenes representativas de colonias teñidas con fosfatasa alcalina (aumento 4X). (DF) Imágenes inmunocitoquímicas representativas de (D) Oct4 (rojo), (E) SSEA3 (verde) y (F) TrA1-60 (verde). (G) Diagrama de puntos representativo de tinción SSEA1 (CD15) y SSEA4, analizado por citometría de flujo. (H) El gráfico de barras muestra los porcentajes de Oct4 + (~ 75-80%), Tra1-60 + (~ 75-80%), SSEA3 + (~ 75-80%), ), Y células β-III-tubulina + (~ 3 - 7%), contrastadas con DAPI y cuantificadas por HCl, con una media de 3 a 5 repeticiones biológicas ± el SEM (gráfico modificado a partir de la Referencia 6). Haga clic aquí para ver una versión más grande de esta figura.
Evaluación de la pluripotencia mediante la formación de EB
Los HiPSCs son pluripotentes, lo que significa que expresan tres genes relacionados con la capa germinal en condiciones apropiadas. Para evaluar la pluripotencia hiPSC, es posible aplicar unaEnfoque basado en la formación espontánea EB, que induce la formación de las tres capas germinales [ 14] . Los análisis de los genes específicos de la capa germinal deben indicar un aumento en el tiempo del endodermo (α-fetoproteína (AFP) y Cytokeratin 18 (KRT18)), ectodermo (Nestin, SRY-box 1 (Sox1) ), Y el mesodermo (péptido natriurético A (NPPA) y Brachyury-T) relacionados con la expresión génica ( Figura 3A y Figura 3B ]; Ver la Tabla de Materiales.
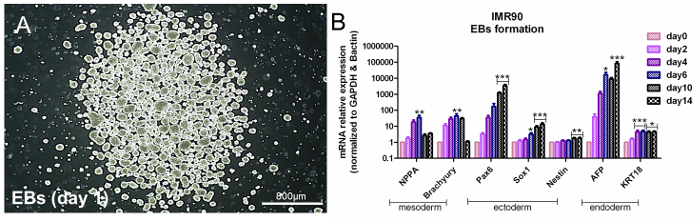
Figura 3. Evaluación de Pluripotencia por formación de EB. ( B) El gráfico de barras muestra los análisis qPCR de mesodermal (NPPA y braquianuria), ectodérmico (Pax6, Sox1 y nestina) y endodérmico (AFP y KRT18 ), Normalizado a los genes de referencia, β-actina y GAPDH, y calibrado a las células indiferenciadas (Día 0). Este es el método ΔΔCt, con una media de 5 análisis independientes ± el SEM * p <0,05, ** p <0,01, *** p <0,001; Gráfico modificado de la referencia 6. Haga clic aquí para ver una versión más grande de esta figura.
Inducción de la diferenciación neuronal y glial
Los IMR90-iPSCs pueden diferenciarse en cultivos mixtos de neuronas postmitoticas y células gliales siguiendo los pasos resumidos en la Figura 1 y en las secciones del protocolo. 5-8 días después de depositar los EB en placas estándar revestidas con matriz o laminina en presencia de un medio NRI completo, las estructuras tipo roseta deberían comenzar a ser visibles (= "Xfig"> Figura 4A). Las rosetas se caracterizan por la presencia de células nestin + (precursores neuronales, ~ 90%), con pocas células β-III-tubulina + (células neuronales comprometidas, ~ 5 - 10%), generalmente localizadas principalmente en la periferia de la Rosetas ( Figura 4B , rosetas el día 12).
Tras la disociación de la roseta y la repoblación en placas o placas laminadas o matriciales estándar en presencia de un medio ND completo, las células comienzan a diferenciarse en cultivos mixtos de neuronas y glias, formando progresivamente grupos de cuerpos celulares neuronales conectados por haces de neuritas . 4C y Figura 4D ). Se deben obtener resultados similares cuando se analizan las poblaciones neuronales obtenidas mediante la expansión de las NSC derivadas de rosetas y diferenciándolas en neuronas y glia. Los NSC expandidos de rosetas deben ser nEstina + ( Figura 4E , inserto que muestra nestina + células).
Después de 21 días de diferenciación, las células deben ser positivas para β-III-tubulina; NF200; Tau; Y MAP2, el marcador tardío de dendritas ( Figura 4D , 4F y Figura 4H), con al menos 10-15% de las células positivas para la proteína ácida fibrilar glial (GFAP), un marcador astroglial ( Figura 4G y Figura 4H) . Además, ~ 20 - 30% de las células deben conservar la expresión de nestin después de la diferenciación ( Figura 4H ]. Es importante considerar que el porcentaje de cada tipo de célula ( es decir, neurona, astrocyte, nestin + células) puede variar en los pasajes, y se puede observar variabilidad dependiente del usuario.
Al analizar determinadas subpoblaciones neuronales, las neuronas GABAérgicas representanLas neuronas dopaminérgicas ~ 13 - 20%, y las neuronas glutamatérgicas ~ 35 - 42%, como se muestra por la inmunotinción para el ácido gamma - aminobutírico (GABA), la tirosina hidroxilasa (TH) y el glutamato vesicular Transportador 1 (VGlut1), respectivamente (véase la cuantificación representativa en la Figura 4H ). La inducción de la diferenciación también puede evaluarse mediante el análisis de marcadores relacionados con la pluripotencia ( p. Ej., Oct4, Tra1-60 y SSEA3), que deberían estar significativamente regulados negativamente en células diferenciadas frente a hiPSCs no diferenciadas (no se muestra en la referencia 6). Esto también se puede confirmar a través del análisis de la expresión génica por qPCR, que debería indicar una disminución de Oct4 y Nanog y la regulación positiva de los genes neuronales, como la molécula de adhesión de células neurales 1 (NCAM1) y los microtúbulos asociados a la proteína 2 (MAP2); El gen presináptico, la sinaptofisina (SYP); Y el gen post-sináptico, proteína tau asociada a los microtúbulos (MAPT), como se muestra en < Strong class = "xfig"> Figura 4I. Además, los genes relacionados con la dopaminérgica (TH y NR4A1), noradrenérgicos (PHOX2A y PHOX2B), glutamatérgicos (NARG2, GRIA1 y GAP43), GABAérgicos (GABRA1 y GABRA3), neuronas motoras (ISL1 y LHX3) y colinérgicos (SLC5A7 y SLC18A13) Resultan en upregulated células neuronales en comparación con las células indiferenciadas ( Figura 4 ].
El análisis de la actividad eléctrica espontánea, por medio de MEA, es una valiosa lectura para evaluar la funcionalidad de la red neuronal en diferenciadas hiPSCs. Al final del período de diferenciación, los derivados neuronales se caracterizan generalmente por una tasa de disparo promedio (MFR) de al menos 60 picos / min (véase el gráfico raster representativo en la Figura 4K ). Sin embargo, no se observan ráfagas.
55702fig4.jpg "/>
Figura 4. Diferenciación de IMR90-hiPSCs en cultivos mixtos de neuronas y glia. (A y B) Imágenes representativas de rosetas después de 7 DIV (A) y después de 12 DIV (B) , teñidas para nestin (verde) y β-III-tubulina (rojo)). (C y D) Imágenes representativas de células diferenciadas después de 22 DIV (C) y 28 DIV (D) , teñidas para β-III-tubulina (rojo) y NF200 (verde)). (E) Imagen representativa de NSC derivadas de la disociación y expansión de la roseta (la inserción muestra células nestin + , de color rojo). (F y G) Imágenes representativas de células neuronales ( F , manchadas para NF200 (rojo) y Tau (verde)) y células gliales ( G , teñidas para GFAP (rojo)) diferenciadas de NSCs (después de 21 DIV). (H) Cuantificación de nestina, MAP2, GFAP, ácido gamma-aminobutírico (GABA), transportador de glutamato vesicular 1 (VGlut1), y las células positivas de tirosina hidroxilasa (TH) por HCl, comparando derivados de IMR90-hiPSC y células diferenciadas de NSC derivadas de IMR90-hiPSC (gráfico modificado de la Referencia 7). (I y J) Gráficos de barras que muestran los análisis qPCR de genes de pluripotencia (Oct4 y Nanog) y genes neuronales (NCAM1, MAP2, SYP y MAPT) (I) y de dopaminérgicos (TH y NR4A1), noradrenérgicos (PHOX2A y PHOX2B) Glutamatérgicos (NARG2, GRIA1 y GAP43), GABAérgicos (GABRA1 y GABRA3), neuronas motoras (ISL1 y LHX3) y colinérgicos (SLC5A7 y SLC18A13) (J) . Todos los análisis se normalizaron a los genes de referencia, β-actina y GAPDH, y calibrado a las células indiferenciadas (barras verdes). Se trata del método ΔΔCt, con una media de 5 análisis independientes ± el SEM * p <0,05, ** p <0,01, *** p <0,001. Las gráficas en I y J se modificaron a partir de la Referencia 6. (K) Gráfica raster representativa de neur derivada de IMR90-NSCOns (la grabación se realizó durante un mínimo de 600 s, las barras verticales representan picos individuales). Haga clic aquí para ver una versión más grande de esta figura.
Una firma específica de marcadores neuronales es upregulated en diferenciado IMR90-hiPSCs
En el nuevo paradigma de la prueba de la toxicidad, es esencial definir los acontecimientos moleculares y celulares que ocurren dentro de una célula después de la exposición a un tóxico dado. Por lo tanto, es relevante para caracterizar qué vías de señalización son activados y / o upregulated dentro del modelo celular bajo investigación.
Las matrices comercialmente disponibles para los análisis de la expresión del gen de la proteína quinasa pueden usarse para comparar los hiPSC no diferenciados frente a las células diferenciadas. DiferenciarLos receptores de neurotrófica, la regulación de la orientación del axón, la modulación del crecimiento de las neuritas, los receptores del factor neurotrófico derivado de glial (GDNF), la proteína de la proteína morfogenética ósea (BMP) / TGF-beta, (PDGF) ( Figura 5A y Figura 5B ).
El análisis de RPPA muestra la regulación positiva de una firma neuronal específica en diferenciadas IMR90-hiPSCs. En particular, las vías de señalización Erk / CREB, Akt / PDK1 / mTOR y Notch1 se activan tras la diferenciación ( Figura 5C ).
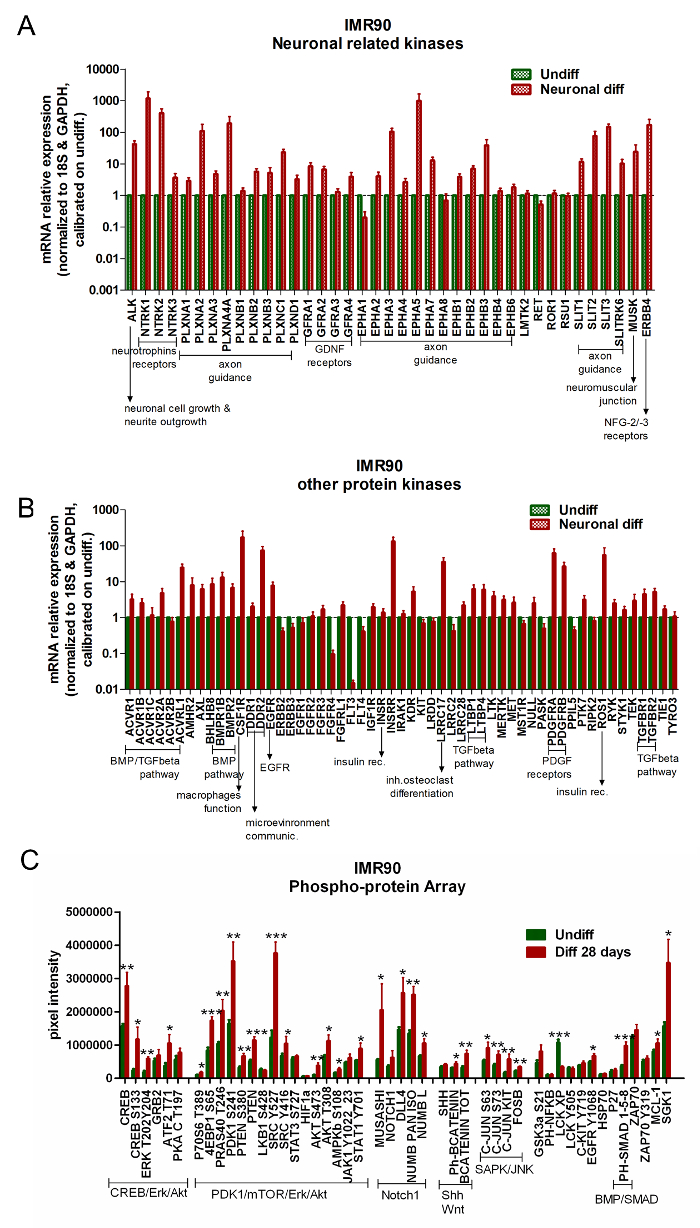
Figura 5. Células diferenciadas neuronales y glialas muestran la activación de las vías relacionadas con las neuronas. (UNY B) Los gráficos de barras informan de los análisis de qPCR de quinasas relacionadas con neuronas (A) y otros genes relacionados con la quinasa (B) . Los datos de expresión génica se normalizaron a los genes de referencia 18S y GAPDH (proporcionados en el conjunto) y se calibraron a células no diferenciadas. Para estos análisis, un gen se consideró significativamente upregulated cuando su expresión fue al menos 2 veces superior a las indiferenciadas en las células (2- ΔΔCt ≥ 2); Media de 3 análisis independientes ± el SEM). (C) El gráfico de barras muestra las cuantificaciones de proteínas absolutas mediante análisis RPPA, comparando las células diferenciadas (barras rojas) y no diferenciadas (barras verdes). Las proteínas pertenecientes a las mismas cascadas de la vía de señalización se agrupan de la siguiente manera: CREB / Erk / Akt, PDK1 / mTOR / Erk / Akt, Notch1, Shh y Wnt, SAPK / JNK y BMP / SMAD. Media ± SEM de 4 análisis independientes. * P <0,05, ** p <0,01, *** p <0,001; Gráfico modificado fRom Referencia 6. Haga clic aquí para ver una versión más grande de esta figura.
Los cultivos neuronales / glial derivados de IMR90-hiPSC pueden usarse para evaluar los efectos de la rotenona
Se sabe que la rotenona, un inhibidor del complejo I de la cadena respiratoria mitocondrial, causa estrés oxidativo al activar la activación de la vía Nrf2. Bajo condiciones de reposo, Nrf2 está anclado en el citoplasma por Keap1 (Kelch-like ECH-associated protein 1), un Nrf2 represor, lo que facilita Nrf2 ubiquitination y proteólisis [ 15] . Tras la inducción de estrés oxidativo, Nrf2 translocates en el núcleo y activa la expresión de Nrf2-ARE genes objetivo [ 16] .
Neuronas derivadas de IMR90-hiPSC yD para evaluar los efectos de la rotenona sobre la activación de Nrf2 mediante la exposición de las células a diferentes concentraciones de rotenona ( por ejemplo, 1, 10 y 100 nM) durante 24 h. Estas concentraciones se establecieron de acuerdo con estudios previos 17 , 18 .
A estas concentraciones y tiempos de exposición, la rotenona no dio lugar a citotoxicidad, como se muestra por la cuantificación de núcleos vivos de células DAPI + ( Figura 6A ). Rotenone inducida Nrf2 translocación nuclear, especialmente después de exponer las células a 100 nM rotenona ( Figura 6B y Figura 6E ]. En la misma concentración, se observó una disminución significativa en el Keap1 citoplasmático ( Figura 6C ), junto con un aumento tanto de NAD (P) H quinona oxidorreductasa 1 (NQO1) y Sulfiredoxina 1 (SRXN1), dos Nrf2-objetivo enZymes 19 , 20 ( Figura 6D ).
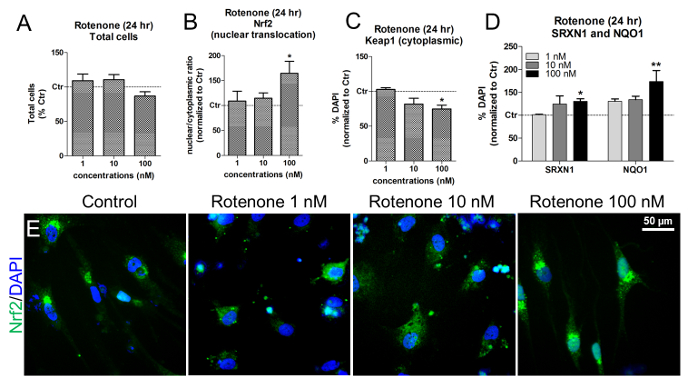
Figura 6. Efectos de la rotenona sobre la translocación nuclear de Nrf2, los niveles de proteína Keap1, SRXN1 y NQO1. (A) Cuantificación de células DAPI + vivas ( es decir, núcleos no picnóticos) después de 24 h de tratamiento con 1, 10, 100 nM de rotenona y normalizado a células no tratadas (Control, Ctr). (B) translocación nuclear de proteína Nrf2 ( es decir, relaciones nucleares / citoplásmicas) después de 24 h de exposición a rotenona, evaluada mediante mediciones de intensidad de fluorescencia usando análisis HCI. (C) Cuantificación de los niveles de proteína Keap1 citoplásmica tras el tratamiento con rotenona, evaluado mediante análisis de HCl. (D) Cuantificación de NAD (P) H quinona oxidorreductasa 1 (NQO1) y SulfiRedoxina 1 (SRXN1) por medio de inmunofluorescencia e HCl después de 24 h de tratamiento con rotenona. (E) Imágenes representativas de la localización de la proteína Nrf2 (verde). Todos los valores se muestran como la media pm SEM de 3 repeticiones biológicas. * P <0,05, ** p & lt; 0,01; Figura modificada de la referencia 7. Haga clic aquí para ver una versión más grande de esta figura.
A estas concentraciones y tiempos de tratamiento, la rotenona también provocó un aumento dependiente de la concentración del porcentaje de células astroglial (GFAP + ) ( Figura 7A y Figura 7B ), sin afectar las proporciones de NSCs (nestin + ) y neuronas (MAP2 + ) . 7B ). Observando las proporciones de los subtipos neuronales específicos, el tratamiento con rotenona (10 nM y 100 nM)Disminuyó significativamente el número de neuronas dopaminérgicas (TH + ) ( Figura 7C y D ), mientras que los porcentajes de GABAergic (GABA + ) y glutamatergic (VGlut1 + ) las neuronas no cambiaron ( Figura 7D ). De forma análoga, estudios previos in vivo e in vitro han descrito una muerte neuronal dopaminérgica dependiente de la rotenona 21 , 22 , 23 .
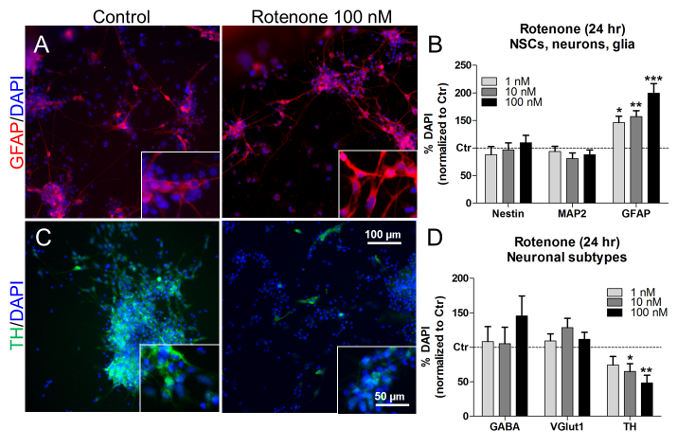
Figura 7. Efectos de la Rotenona sobre las Células Glándulas y las Neuronas Dopaminérgicas. (A) Imágenes representativas de células GFAP + (rojo), con un aumento de 40X en las inserciones, no tratadas o tratadas con rotenona 100 nM durante 24 h. (B) Cuantificación De las células nestin + , MAP2 + y GFAP + , normalizadas a las células no tratadas (control, Ctr). (C) Imágenes representativas de las neuronas TH + dopaminérgicas (verde), con aumento de 40X en las inserciones, no tratadas o tratadas con rotenona 100 nM durante 24 h. (D) Cuantificación de células neuronales GABA + , VGlut1 + y TH + , normalizadas a células no tratadas (Ctr). Todos los valores se muestran como la media pm SEM de 3 repeticiones biológicas. * P <0,05, ** p <0,01, *** p <0,001; Figura modificada de la referencia 7. Haga clic aquí para ver una versión más grande de esta figura.
La significancia estadística se evaluó mediante ANOVA de una vía con la prueba de comparación múltiple de Dunnett como una prueba posterior (comparando todas las columnas con la columna de control)Xref "> 24 o por t-test pareado o no pareado de dos colas según el tipo de análisis.Todos los datos representan el promedio de al menos tres repeticiones biológicas ± el error estándar de la media (SEM) Un asterisco sobre una barra indica Una diferencia significativa con el grupo control. * P <0,05, ** p <0,01, *** p <0,001.
Tabla 1.
Nota sobre la densidad de revestimiento de roseta disociada: si los fragmentos de roseta no parecen estar completamente disociados, para alcanzar una densidad de recubrimiento celular de aproximadamente 15.000 células / cm2, se pueden resuspender fragmentos de rosetas disociadas que se derivan de aproximadamente 50 EBs / 1 x 60 mm 50 ml de medio NRI completo y se cubre de la siguiente manera (dependiendo del formato de la placa):
| Placas multipozo / MEA | Área de crecimiento (cm ^ { 2 } / pozo) | Volumen de la suspensión celular a la placa por pocillo (o chip MEA) | Número máximo de placas que pueden ser chapadas (con 50 ml de suspensión celular) |
| 96 pozos | 0,3 | 100 μl | 5 |
| 48 pozos | 0,7 | 220 ul | 4 |
| 24 pozos | 2 | 625 ul | 3 |
| 12 pozos | 4 | 1,25 ml | 3 |
| 6 pozos | 10 | 3.125 ml | 2 |
| Single well MEA chip | 3,5 | 1,1 ml | 45 |
Tabla 2: Criterios de aceptación
| Marcador /Anticuerpo | Porcentaje (en células DAPI + (vivas) después de 28 DIV |
| B - III - tubulina (Tuj1) | 35-45% |
| MAP2 | 50-60% |
| NF200 | 45-55% |
| GFAP | 10-25% |
| Nestin | 15-25% |
Discusión
Este trabajo describe un protocolo robusto y relativamente rápido para la diferenciación de IMR90-HiPSCs en neuronas post-mitótico y células gliales. Los protocolos de diferenciación neuronal previamente publicados basados en hESCs y hiPSCs suelen producir altos porcentajes de precursores neuronales 25 , 26 y un número significativo de células diana neuronales 27 , 28 , 29 , 30 , 31 , 32 , 33 . Análogamente, el protocolo de diferenciación descrito aquí es adecuado para generar cultivos heterogéneos de células neuronales GABAérgicas, glutamatérgicas y dopaminérgicas, junto con glia y una discreta proporción de células nestina + . La presencia de glutamatergic (~ 35-42%) y GABAérgico (~ 15-20%) las células neurales sugiere queEsta cultura posee prosencéfalo, rasgos corticales y la presencia de un número discreto de neuronas dopaminérgicas (~ 13-20%) también puede indicar la especificidad del mesencéfalo. Además, la permanencia de una proporción modesta de células nestin + puede resultar adecuado para el estudio de la neurogénesis y los posibles efectos de los productos químicos en NSCs, que se limitan principalmente al hipocampo y la zona subventricular (SVZ) del prosencéfalo 34 . Otros análisis inmunocitoquímicos y de expresión génica ayudarían a definir mejor la especificidad regional de los derivados celulares diferenciados.
Los dos pasos más críticos en el protocolo de diferenciación descritos en este documento son: (i) el corte de colonias hiPSC en fragmentos homogéneos (que es crítico para la generación de EBs con tamaños homogéneos) y (ii) el corte de estructuras neuroectodérmicas ) Para la diferenciación NSC, que requiere una habilidad manual significativaY precisión para evitar la recolección de células mesodérmicas y endodérmicas que pueden reducir las proporciones de neuronas y células gliales obtenidas tras la diferenciación.
Es crucial caracterizar los fenotipos de las células durante la expansión (como colonias indiferenciadas o NSCs) y durante todas las etapas de diferenciación. En particular, los perfiles de expresión de genes y proteínas de los derivados de células neuronales / glial deberían mostrar una regulación positiva y activación de vías de señalización relacionadas con las neuronas, mientras que la expresión de marcadores de pluripotencia debería disminuir.
La generación de EBs y derivados neuroectodérmicos (rosetas) puede ser manualmente desafiante y propenso a la variabilidad. Por esta razón, hemos desarrollado un protocolo para la expansión de roseta derivados NSCs y su posterior diferenciación en células neuronales / glial.
Las posibles limitaciones de este protocolo de diferenciación son principalmente (i) el relativamente bajo porcentaje de dDerivaciones glial ifferenciadas y (ii) la falta de funciones de red neuronal maduras (como se muestra por la falta de ráfagas). Además, subpoblaciones específicas de astrocitos pueden funcionar como progenitores primarios o NSC 35 . Aunque nestin / GFAP doble positivo de las células no se observaron en este cultivo celular diferenciado (datos no presentados), se plantea la hipótesis de que las células GFAP + en estos cultivos mixtos son progenitores astrocíticos y astrocitos. Es plausible que al extender el tiempo de diferenciación, el número de astrocitos pueda aumentar, y su morfología puede llegar a ser más madura, como ya se indicó en trabajos previos del grupo de Zhang 36 , 37 .
En el nuevo paradigma de pruebas de toxicidad, el conocimiento sobre las perturbaciones químicas de las vías biológicas es de suma importancia cuando se evalúa la adversidad química. Por lo tanto, los sistemas de ensayo in vitro debenRelacionan los efectos adversos con los trastornos de las vías de señalización, de acuerdo con el concepto de la vía de resultado adverso (AOP). Como prueba de concepto, la rotenona puede usarse para evaluar la activación de la vía Nrf2, que está implicada en la defensa celular contra el estrés oxidativo o electrofílico 38 , y el estrés oxidativo es un evento clave importante y común en varios AOPs relevantes para Neurotóxica del desarrollo y del adulto 39 .
La rodamona debería provocar la activación de la vía Nrf2, que puede demostrarse mediante la translocación nuclear de la proteína Nrf2 y el aumento de la expresión de las enzimas diana Nrf2, incluyendo NQO1 y SRXN1. Se ha encontrado que la rotenona induce un aumento dependiente de la dosis de los niveles de proteína GFAP, indicativo de la activación de astrocitos 40 , 41 . Rotenona también disminuye el número de células dopaminérgicas (TH + ), lo que está de acuerdo con previIn vitro e in vivo que muestran la muerte celular dopaminérgica dependiente de rotenona, ya que este tipo de neurona es particularmente sensible al estrés oxidativo 21 , 22 , 23 .
En conclusión, este hiPSC derivado neuronal y glial modelo de cultivo celular es una valiosa herramienta para evaluar los efectos neurotóxicos de los productos químicos que provocan el estrés oxidativo resultante en la activación de la vía Nrf2. Dado que este protocolo de diferenciación permite la generación de cultivos mixtos de células neuronales (neuronas GABAérgicas, dopaminérgicas y glutamatérgicas) y astrocitos, puede resultar adecuado para estudiar la diafonía entre neuronas y glía en condiciones fisiológicas y patológicas, como en las enfermedades neurodegenerativas Por ejemplo, enfermedad de Parkinson). Por otra parte, la presencia de una proporción significativa de NSCs puede ayudar a evaluar los posibles efectos de los productos químicos en el prog neuralEn los que se sabe que son el principal objetivo de las mutaciones inducidas químicamente o infecciones virales [ 42] .
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores desean agradecer al Dr. Marc Peschanski (I-Stem, Évry, Francia), por proporcionar los IMR90-hiPSCs; La Dra. Giovanna Lazzari y la Dra. Silvia Colleoni (Avantea srl, Cremona, Italia); Dra. Simone Haupt (Universidad de Bonn, Alemania); Dr. Tiziana Santini (Instituto Italiano de Tecnología, Roma), por brindar asesoramiento sobre evaluación de tinción por inmunofluorescencia; La Dra. Benedetta Accordi, la Dra. Elena Rampazzo y el Dr. Luca Persano (Universidad de Padua, Italia), por sus contribuciones al análisis RPPA ya la validación de anticuerpos. Financiación: este trabajo fue apoyado por el proyecto financiado por la UE "SCR & Tox" (Convenio de subvención N ° 266753).
Materiales
| Name | Company | Catalog Number | Comments |
| Complete hiPSC medium: | |||
| mTeSR1 Basal Medium | Stem Cell Technologies | 05851 | (Step 1.2.6). Complete mTeSR1 is stable when stored at 2 - 8°C for up to 2 weeks. 5X Supplements can be dispensed into working aliquots and stored at -20°C. Use frozen aliquots within 3 months. |
| mTeSR1 5X Supplements | Stem Cell Technologies | 05852 | |
| Matrigel hESC-qualified Matrix | Corning | 354277 | 1:100 (Step 1.1). Thaw Matrigel on ice, prepare 200 ul aliquots and store them in -80°C. For coating, dilute 200ul aliquot in 20 ml of DMEM/F12 medium. |
| CryoStem Freezing Medium | Stemgent | 01-0013-50 | Freeze ~ 100 fragments/250 ul/vial (Step 1.2.1) |
| Name | Company | Catalog Number | Comments |
| hiPSC EB medium: | |||
| Knockout DMEM | Thermo-Fisher | 10829-018 | (Step 2.1.7) |
| Knockout Serum Replacement (KOSR) | Thermo-Fisher | 10828-028 | 20% final concentration (Step 2.1.7) |
| Non-Essential Amino Acids | Thermo-Fisher | 11140-035 | (Step 2.1.7) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 2.1.7) |
| L-Glutamine 200 mM Solution | Thermo-Fisher | 25030-081 | 2 mM final concentration (Step 2.1.7) |
| β-Mercaptoethanol | Thermo-Fisher | 31350-010 | 50 µM final concentration (Step 2.1.7) |
| Name | Company | Catalog Number | Comments |
| Complete neuroepithelial induction medium (NRI): | |||
| DMEM/F12 | Thermo-Fisher | 3133-038 | (Step 2.3.1) |
| Non-Essential Amino Acids | Thermo-Fisher | 11140-035 | (Step 2.3.1) |
| N2 Supplement | Thermo-Fisher | 17502-048 | (Step 2.3.1) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 2.3.1) |
| Heparin Grade I-A, ≥180 USP units/mg | Sigma-Aldrich | H3149-100KU | 2 µg/ml final concentration (Step 2.3.1) |
| bFGF | Thermo-Fisher | 13256-029 | 20 ng/ml final concentration added before use (Step 2.3.1) |
| Matrigel Basement Membrane Matrix | Corning | 354234 | 1:100 (Step 2.2). Thaw Matrigel on ice, prepare 200 ul aliquots and store them in -80°C. For coating, dilute 200 ul aliquot in 20 ml of cold DMEM/F12 medium. |
| Laminin | Sigma-Aldrich | L2020 | 1:100 (Step 2.2). Dilute in PBS 1X. |
| Name | Company | Catalog Number | Comments |
| Complete Neuronal Differentiation medium (ND): | |||
| Neurobasal Medium | Thermo-Fisher | 21103049 | (Step 2.4.11) |
| B-27 Supplements (50x) | Thermo-Fisher | 17504044 | (Step 2.4.11) |
| N2 Supplement | Thermo-Fisher | 17502-048 | (Step 2.4.11) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 2.4.11) |
| GDNF | Thermo-Fisher | PHC7045 | 1 ng/ml final concentration. Added before use. (Step 2.4.11) |
| BDNF | Thermo-Fisher | PHC7074 | 2.5 ng/ml final concentration. Added before use. (Step 2.4.11) |
| Name | Company | Catalog Number | Comments |
| Neural induction medium (NI): | |||
| DMEM/F12 | Thermo-Fisher | 3133-038 | (Step 3.3) |
| Non-Essential Amino Acids | Thermo-Fisher | 11140-035 | (Step 3.3) |
| N2 Supplement | Thermo-Fisher | 17502-048 | (Step 3.3) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 3.3) |
| Heparin Grade I-A, ≥180 USP units/mg | Sigma-Aldrich | H3149-100KU | 2 µg/ml final concentration (Step 3.3) |
| B-27 Supplement (50X), minus vitamin A | Thermo-Fisher | 12587010 | (Step 3.3) |
| L-Glutamine 200 mM Solution | Thermo-Fisher | 25030-081 | 2 mM final concentration (Step 3.3) |
| bFGF | Thermo-Fisher | 13256-029 | 10 ng/ml final concentration. Added before use (Step 3.3) |
| EGF | Thermo-Fisher | PHG6045 | 10 ng/ml final concentration. Added before use (Step 3.3) |
| BDNF | Thermo-Fisher | PHC7074 | 2.5 ng/ml final concentration. Added before use (Step 3.3) |
| Defined Trypsin Inhibitor (DTI) | Thermo-Fisher | R007-100 | Pre-warm at 37°C. Add an equal amount of DTI to Trypsin-EDTA (Step 3.10) |
| Trypsin-EDTA (0.5%), no phenol red | Thermo-Fisher | 15400054 | 1:10. Dilute Trypsin-EDTA in PBS 1x (without calcium and magnesium), pre-warm the solution at 37°C (Step 3.8) |
| CryoStor cell cryopreservation medium | Sigma-Aldrich | C2874-100ML | (Step 4.2) |
| Trypan Blue (0.4%) | Sigma-Aldrich | T8154-100ML | multiple manufacturers/suppliers |
| Name | Company | Catalog Number | Comments |
| TaqMan Probesets and reagents for gene expression analysis: | |||
| RNAqueous-Micro kit | Thermo-Fisher | AM1931 | (Step 5.1.6) |
| High Capacity cDNA Reverse Transcription Kits | Thermo-Fisher | 4368814 | |
| TaqMan Gene Expression Master Mix | Thermo-Fisher | 4369016 | |
| GFAP | Thermo-Fisher | Hs00909233_m1 | |
| MAP2 | Thermo-Fisher | Hs00258900_m1 | |
| NQO1 | Thermo-Fisher | Hs02512143_s1 | |
| SRXN1 | Thermo-Fisher | Hs00607800_m1 | |
| HMOX1 | Thermo-Fisher | Hs01110250_m1 | |
| GSR | Thermo-Fisher | Hs00167317_m1 | |
| PAX6 | Thermo-Fisher | Hs01088112_m1 | |
| NES | Thermo-Fisher | Hs00707120_s1 | |
| GRIA1 | Thermo-Fisher | Hs00181348_m1 | |
| GAP43 | Thermo-Fisher | Hs00967138_m1 | |
| GABRA3 | Thermo-Fisher | Hs00968132_m1 | |
| GABRA1 | Thermo-Fisher | Hs00168058_m1 | |
| NR4A2 | Thermo-Fisher | Hs00428691_m1 | |
| TH | Thermo-Fisher | Hs00165941_m1 | |
| GAPDH | Thermo-Fisher | Hs02758991_g1 | |
| ACTB | Thermo-Fisher | Hs99999903_m1 | |
| MAPT | Thermo-Fisher | Hs00902194_m1 | |
| SYP | Thermo-Fisher | Hs00300531_m1 | |
| NANOG | Thermo-Fisher | Hs04260366_g1 | |
| POU5F1 (OCT4) | Thermo-Fisher | Hs04195369_s1 | |
| SOX1 | Thermo-Fisher | Hs01057642_s1 | |
| AFP | Thermo-Fisher | Hs00173490_m1 | |
| KRT18 | Thermo-Fisher | Hs01941416_g1 | |
| NPPA | Thermo-Fisher | Hs00383230_g1 | |
| T | Thermo-Fisher | Hs00610080_m1 | |
| NCAM1 | Thermo-Fisher | Hs00941821_m1 | |
| NR4A1 | Thermo-Fisher | Hs00374226_m1 | |
| PHOX2A | Thermo-Fisher | Hs00605931_mH | |
| PHOX2B | Thermo-Fisher | Hs00243679_m1 | |
| NARG2 | Thermo-Fisher | Hs00973298_g1 | |
| SLC18A3 | Thermo-Fisher | Hs00268179_s1 | |
| SLC5A7 | Thermo-Fisher | Hs00222367_m1 | |
| ISL1 | Thermo-Fisher | Hs00158126_m1 | |
| LHX3 | Thermo-Fisher | Hs01033412_m1 | |
| TaqMan Human Protein Kinase Array | Thermo-Fisher | 4418721 | |
| Name | Company | Catalog Number | Comments |
| Antibodies and reagents for immunostaining: | |||
| B-III-tubulin (Tuj1) | Covance | MMS-435P | 1:500 (Step 5.2.5). Other antibodies may also be used. |
| MAP2 | Sigma Aldrich | M4403 | 1:500 |
| NF200 | Sigma Aldrich | N4142 | 1:1000 |
| GFAP | Acris Antibodies GmbH | AP02002SU-N | 1:500 |
| Nestin | Sigma-Aldrich | N5413 | 1:200 |
| synaptophysin (SYN) | Abcam | AB14692 | 1:200 |
| Tau | Thermo-Fisher | MA5-12808 | 1:100 |
| Nrf2 | Abcam | AB62352 | 1:200 |
| Keap1 | Abcam | AB66620 | 1:200 |
| sulfiredoxin1 (SRXN1) | Abcam | AB92298 | 1:200 |
| NAD(P)H quinone oxidoreductase 1 (NQO1) | Abcam | AB2346 | 1:200 |
| OCT4 | Millipore | MAB4401 | 1:100 |
| SSEA3 | Millipore | MAB4303 | 1:100 |
| Tra1-60 | Millipore | MAB4360 | 1:250 |
| Tyrosine hydroxylase (TH) | Millipore | AB152 | 1:200 |
| Gamma-aminobutyric acid (GABA) | Sigma-Aldrich | A0310 | 1:100 |
| Vesicular glutamate transporter 1 (VGlut1) | Abcam | AB72311 | 1:500 |
| Paraformaldehyde | Sigma-Aldrich | P6148-500G | 4% (4% formaldehyde can also be used) |
| DPBS, no calcium, no magnesium | Thermo-Fisher | 14190144 | |
| Triton-X-100 Solution | Sigma-Aldrich | 93443-100ML | 0.1% |
| BSA 35% | Sigma-Aldrich | A7979-50ML | 3.5% |
| Donkey anti-Rabbit IgG (H+L) Cross Adsorbed Secondary Antibody, DyLight 594 conjugate | Thermo-Fisher | SA5-10040 | 1:500. (Step 5.2.7) Other fluorochrome-conjugated secondary antibodies may also be used. In this case, appropriate dilutions should be tested by the enduser. |
| Donkey anti-Mouse IgG (H+L) Cross Adsorbed Secondary Antibody, DyLight 488 conjugate | Thermo-Fisher | SA5-10166 | 1:500 |
| Donkey anti-Goat IgG (H+L) Cross Adsorbed Secondary Antibody, DyLight 488 conjugate | Thermo-Fisher | SA5-10086 | 1:500 |
| DAPI Solution (1 mg/ml) | Thermo-Fisher | 62248 | 1:1000 (Step 5.2.7) |
| Name | Company | Catalog Number | Comments |
| Antibodies for Reverse Phase Protein Array (RPPA): | |||
| 4E-BP1 (S65) | Abcam | AB81297 | 1:250 (Note after step 5.2.8) |
| Akt (T308) | Cell Signaling | 9275 | 1:100 |
| Akt (S473) | Cell Signaling | 9271 | 1:100 |
| AMPKalpha (T172) | Cell Signaling | 2531 | 1:100 |
| AMPKbeta1 (S108) | Cell Signaling | 4181 | 1:100 |
| ATF-2 (T71) | Cell Signaling | 9221 | 1:100 |
| c-Jun (S63) | Cell Signaling | 9261 | 1:200 |
| c-Jun (S73) | Cell Signaling | 9164 | 1:200 |
| c-Kit (Y719) | Cell Signaling | 3391 | 1:250 |
| CREB (S133) | Cell Signaling | 9191 | 1:100 |
| EGFR (Y1068) | Cell Signaling | 2234 | 1:50 |
| ErbB2/HER2 (Y1248) | Cell Signaling | 2247 | 1:100 |
| ERK 1/2, p44/42 (T202/Y204) | Cell Signaling | 9101 | 1:2000 |
| GSK-3alpha (S21) | Cell Signaling | 9337 | 1:50 |
| Jak1 (Y1022/1023) | Cell Signaling | 3331 | 1:100 |
| Lck (Y505) | Cell Signaling | 2751 | 1:500 |
| LKB1 (S428) | Cell Signaling | 3051 | 1:100 |
| mTOR (S2448) | Cell Signaling | 5536 | 1:100 |
| NFkB p65 (S536) | Cell Signaling | 3031 | 1:50 |
| p70 S6 Kinase (T389) | Cell Signaling | 9205 | 1:200 |
| PDK1 (S241) | Cell Signaling | 3061 | 1:100 |
| PKA C (T197) | Cell Signaling | 4781 | 1:250 |
| PRAS40 (T246) | BioSource | 44-1100 | 1:2000 |
| PTEN (S380) | Cell Signaling | 9551 | 1:500 |
| Smad1 (S463/465), Smad5 (S463/465), Smad8 (S426/428) | Cell Signaling | 9511 | 1:500 |
| Src (Y527) | Cell Signaling | 2105 | 1:500 |
| Src Family (Y416) | Cell Signaling | 2101 | 1:200 |
| Stat1 (Y701) | Cell Signaling | 9171 | 1:200 |
| Stat3 (S727) | Cell Signaling | 9134 | 1:200 |
| Zap-70 (Y319) | Enogene | E011159 | 1:100 |
| βCatenin (S33/37/T41) | Cell Signaling | 9561 | 1:250 |
| CREB | Upstate Biotechnologies | 06-863 | 1:100 |
| Fos B | Cell Signaling | 2251 | 1:200 |
| GRB2 | Cell Signaling | 3972 | 1:2000 |
| HSP70 | Stressgen | SPA-810 | 1:100 |
| c-Jun | Cell Signaling | 9165 | 1:100 |
| Kip1/p27 | BD | 610241 | 1:100 |
| Lck | Cell Signaling | 2984 | 1:250 |
| Mcl-1 | Cell Signaling | 4572 | 1:80 |
| Musashi | Cell Signaling | 2154 | 1:100 |
| NOTCH1 | Cell Signaling | 3439 | 1:100 |
| PTEN | Cell Signaling | 9552 | 1:500 |
| SGK1 | Abnova | PAB4590 | 1:250 |
| Zap-70 | Cell Signaling | 2705 | 1:250 |
| β-Catenin | Abcam | AB32572 | 1:1000 |
| Dll4 | Abcam | AB7280 | 1:500 |
| Shh | Abcam | AB53281 | 1:250 |
| HIF-1α | BD | 610958 | 1:50 |
| NUMB PAN-ISO | Upstate Biotechnologies | 07-207 | 1:400 |
| NUMB-L | Chemicon | AB15145 | 1:750 |
| Cyclin B | BD | 610220 | 1:75 |
| c-Myc | Calbiochem | OP-10 | 1:100 |
| BCIP/NBT Kit | Thermo-Fisher | 002209 | (Note after step 5.2.8). Kit used to measure alkaline phosphatase activity, similar kits can be used. |
| Name | Company | Catalog Number | Comments |
| Antibodies for Flow Cytometry: | |||
| SSEA1 Antibody, Pacific Blue conjugate | Thermo-Fisher | MHCD1528 | 1:100 (Note after step 5.2.8) |
| SSEA4 Antibody (MC813-70), Alexa Fluor 647 | Thermo-Fisher | SSEA421 | 1:100 |
| Name | Company | Catalog number | Comments |
| Specific instruments, tools and softwares: | |||
| Countess Automated Cell Counter | Thermo-Fisher | C10227 | Neubauer chamber or other suitable glass hemocytometer can be used. |
| MEA1060-Inv-BC | Multichannel Systems | MEA1060-Inv-BC | (Step 5.3) |
| MEA1060-BC control software | Multichannel Systems | MEA1060-BC | (Step 5.3) |
| NeuroExplorer | Multichannel Systems | NeuroExplorer (NE) | (Step 5.3) For post-processing of MEA data |
| Multielectrode arrays (MEA) | Multichannel Systems | 60MEA100/10iR-Ti-gr | (Step 5.3) Single-well MEA chip |
| ArrayScan XTI High Content Platform | Thermo-Fisher | ASN00002P | (Step 5.2.8) Mean fluorescence can be quantified by using specific ArrayScan algorithms (e.g., Cytotoxicity V.4 and NucTrans V.4 bioapplications). It is recommended to take minimum 20 pictures/well, and have 7-8 internal replicates per condition |
| 7900HT Fast Real-Time PCR System | Thermo-Fisher | 4351405 | (Step 5.1.6) |
| BD ULTRA-FINE Needle Insulin Syringe (with 30G needle) | BD | 328280 | (Steps 1.3.1, 2.1.2, and 2.4.1) |
| StemPro EZPassage Disposable Stem Cell Passaging Tool | ThermoFisher | 23181010 | This colony cutting tool can be used as an alternative to the use of 30G needle 1 mL syringes (Step 1.3.1) |
| Ultra-Low attachment Petri dish (60 mm) | Corning | 10010582 | (Step 2.1.8) Also other brands can be used. |
| Mr. Frosty Freezing container | Sigma-Aldrich | C1562-1EA | |
Referencias
- NRC. . Toxicity Testing in the 21st Century: A Vision and a Strategy. , (2007).
- Drews, K., Jozefczuk, J., Prigione, A., Adjaye, J. Human induced pluripotent stem cells--from mechanisms to clinical applications. J Mol Med (Berl). 90 (7), 735-745 (2012).
- Ho, P. J., Yen, M. L., Yet, S. F., Yen, B. L. Current applications of human pluripotent stem cells: possibilities and challenges. Cell Transplant. 21 (5), 801-814 (2012).
- Krueger, W. H., Swanson, L. C., Tanasijevic, B., Rasmussen, T. P. Natural and artificial routes to pluripotency. Int J Dev Biol. 54 (11-12), 1545-1564 (2010).
- Pistollato, F., Bremer-Hoffmann, S., Healy, L., Young, L., Stacey, G. Standardization of pluripotent stem cell cultures for toxicity testing. Expert Opin Drug Metab Toxicol. 8 (2), 239-257 (2012).
- Pistollato, F., et al. Development of a pluripotent stem cell derived neuronal model to identify chemically induced pathway perturbations in relation to neurotoxicity: effects of CREB pathway inhibition. Toxicol Appl Pharmacol. 280 (2), 378-388 (2014).
- Zagoura, D., Canovas-Jorda, D., Pistollato, F., Bremer-Hoffmann, S., Bal-Price, A. Evaluation of the rotenone-induced activation of the Nrf2 pathway in a neuronal model derived from human induced pluripotent stem cells. Neurochem Int. , (2016).
- Standard operating procedure for differentiation of human induced pluripotent stem cells into post-mitotic neurons and glial cells. EURL ECVAM Available from: https://ecvam-dbalm.jrc.ec.europa.eu/methods-and-protocols/protocol/standard-operating-procedure-for-differentiation-of-human-induced-pluripotent-stem-cells-into-post-mitotic-neurons-and-glial-cells-%28mixed-culture%29-protocol-no.-165/key/p_1570 (2016)
- Standard operating procedure for expansion of rosette-derived neural stem cells. EURL ECVAM Available from: https://ecvam-dbalm.jrc.ec.europa.eu/methods-and-protocols/protocol/standard-operating-procedure-for-expansion-of-rosette-derived-neural-stem-cells-protocol-no.-166/key/p_1571 (2016)
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Brien, P. J., et al. High concordance of drug-induced human hepatotoxicity with in vitro cytotoxicity measured in a novel cell-based model using high content screening. Arch Toxicol. 80 (9), 580-604 (2006).
- Accordi, B., et al. Functional protein network activation mapping reveals new potential molecular drug targets for poor prognosis pediatric BCP-ALL. PLoS One. 5 (10), e13552 (2010).
- Vassallo, A., et al. A multi-laboratory evaluation of microelectrode array-based measurements of neural network activity for acute neurotoxicity testing. Neurotoxicology. , (2016).
- Shamblott, M. J., et al. Human embryonic germ cell derivatives express a broad range of developmentally distinct markers and proliferate extensively in vitro. Proc Natl Acad Sci U S A. 98 (1), 113-118 (2001).
- Bryan, H. K., Olayanju, A., Goldring, C. E., Park, B. K. The Nrf2 cell defence pathway: Keap1-dependent and -independent mechanisms of regulation. Biochem Pharmacol. 85 (6), 705-717 (2013).
- Tufekci, K. U., Civi Bayin, E., Genc, S., Genc, K. The Nrf2/ARE Pathway: A Promising Target to Counteract Mitochondrial Dysfunction in Parkinson's Disease. Parkinsons Dis. , 314082 (2011).
- Kovac, S., et al. Nrf2 regulates ROS production by mitochondria and NADPH oxidase. Biochim Biophys Acta. 1850 (4), 794-801 (2015).
- Lee, J. M., Shih, A. Y., Murphy, T. H., Johnson, J. A. NF-E2-related factor-2 mediates neuroprotection against mitochondrial complex I inhibitors and increased concentrations of intracellular calcium in primary cortical neurons. J Biol Chem. 278 (39), 37948-37956 (2003).
- Itoh, K., et al. An Nrf2/small Maf heterodimer mediates the induction of phase II detoxifying enzyme genes through antioxidant response elements. Biochem Biophys Res Commun. 236 (2), 313-322 (1997).
- Li, L., et al. Nrf2/ARE pathway activation, HO-1 and NQO1 induction by polychlorinated biphenyl quinone is associated with reactive oxygen species and PI3K/AKT signaling. Chem Biol Interact. , 56-67 (2014).
- Cannon, J. R., et al. A highly reproducible rotenone model of Parkinson's disease. Neurobiol Dis. 34 (2), 279-290 (2009).
- Sherer, T. B., Kim, J. H., Betarbet, R., Greenamyre, J. T. Subcutaneous rotenone exposure causes highly selective dopaminergic degeneration and alpha-synuclein aggregation. Exp Neurol. 179 (1), 9-16 (2003).
- Testa, C. M., Sherer, T. B., Greenamyre, J. T. Rotenone induces oxidative stress and dopaminergic neuron damage in organotypic substantia nigra cultures. Brain Res Mol Brain Res. 134 (1), 109-118 (2005).
- Zhou, J., et al. High-efficiency induction of neural conversion in human ESCs and human induced pluripotent stem cells with a single chemical inhibitor of transforming growth factor beta superfamily receptors. Stem Cells. 28 (10), 1741-1750 (2010).
- Topol, A., Tran, N. N., Brennand, K. J. A Guide to Generating and Using hiPSC Derived NPCs for the Study of Neurological Diseases. J Vis Exp. (96), e52495 (2015).
- Jiang, Y., Zhang, M. J., Hu, B. Y. Specification of functional neurons and glia from human pluripotent stem cells. Protein Cell. 3 (11), 818-825 (2012).
- Parsons, X. H., et al. Efficient derivation of human neuronal progenitors and neurons from pluripotent human embryonic stem cells with small molecule induction. J Vis Exp. (56), e3273 (2011).
- Shi, Y., Kirwan, P., Livesey, F. J. Directed differentiation of human pluripotent stem cells to cerebral cortex neurons and neural networks. Nat Protoc. 7 (10), 1836-1846 (2012).
- Zeng, H., et al. Specification of region-specific neurons including forebrain glutamatergic neurons from human induced pluripotent stem cells. PLoS One. 5 (7), e11853 (2010).
- Zeng, X., et al. An in vitro model of human dopaminergic neurons derived from embryonic stem cells: MPP+ toxicity and GDNF neuroprotection. Neuropsychopharmacology. 31 (12), 2708-2715 (2006).
- Paşca, A. M., et al. Functional cortical neurons and astrocytes from human pluripotent stem cells in 3D culture. Nat Methods. 12 (7), 671-678 (2015).
- Almeida, S., et al. Modeling key pathological features of frontotemporal dementia with C9ORF72 repeat expansion in iPSC-derived human neurons. Acta Neuropathol. 126 (3), 385-399 (2013).
- Urbán, N., Guillemot, F. Neurogenesis in the embryonic and adult brain: same regulators, different roles. Front Cell Neurosci. 8, 396 (2014).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annu Rev Neurosci. 32, 149-184 (2009).
- Krencik, R., Weick, J. P., Liu, Y., Zhang, Z. J., Zhang, S. C. Specification of transplantable astroglial subtypes from human pluripotent stem cells. Nat Biotechnol. 29 (6), 528-534 (2011).
- Krencik, R., Zhang, S. C. Directed differentiation of functional astroglial subtypes from human pluripotent stem cells. Nat Protoc. 6 (11), 1710-1717 (2011).
- Nguyen, T., Nioi, P., Pickett, C. B. The Nrf2-antioxidant response element signaling pathway and its activation by oxidative stress. J Biol Chem. 284 (20), 13291-13295 (2009).
- Bal-Price, A., et al. Putative adverse outcome pathways relevant to neurotoxicity. Crit Rev Toxicol. 45 (1), 83-91 (2015).
- Cabezas, R., El-Bacha, R. S., Gonzalez, J., Barreto, G. E. Mitochondrial functions in astrocytes: neuroprotective implications from oxidative damage by rotenone. Neurosci Res. 74 (2), 80-90 (2012).
- Swarnkar, S., et al. Astrocyte activation: a key step in rotenone induced cytotoxicity and DNA damage. Neurochem Res. 37 (10), 2178-2189 (2012).
- Canovas-Jorda, D., Louisse, J., Pistollato, F., Zagoura, D., Bremer, S. Regenerative toxicology: the role of stem cells in the development of chronic toxicities. Expert Opin Drug Metab Toxicol. 10 (1), 39-55 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados